中国科学院微生物研究所、中国微生物学会主办
文章信息
- 付立霞, 冀德君, 卢徐斌, 韩先干, 魏文志
- Fu Lixia, Ji Dejun, Lu Xubin, Han Xian'gan, Wei Wenzhi
- 金黄色葡萄球菌核酸酶A与不同外源DNA片段融合表达的作用比较
- Comparison of effects of staphylococcal nuclease A fused with different exogenous DNA fragments
- 生物工程学报, 2016, 32(12): 1654-1663
- Chin J Biotech, 2016, 32(12): 1654-1663
- 10.13345/j.cjb.160194
-
文章历史
- Received: May 9, 2016
- Accepted: September 18, 2016
2 中国农业科学院上海兽医研究所,上海 200241
2 Shanghai Veterinary Research Institute, Chinese Academy of Agricultural Sciences, Shanghai 200241, China
菌蜕是革兰氏阴性菌被噬菌体PhiX174裂解基因E的诱导表达产物裂解后所形成的不含胞质内含物的完整细菌空壳。由于保留了细菌原有的表面结构和特性,菌蜕具有疫苗和佐剂的双重功能,可刺激机体产生更强的免疫反应,因而较常规疫苗具有更好的免疫保护效果[1-4],其更进一步的优点是可以作为抗原、核酸和药物的高级递送载体[5-7]。正是由于具有这些优点,使得菌蜕日益成为当前疫苗研究的热点。在菌蜕的制备过程中,尽管大多数实验细菌可被裂解基因E所介导的裂解作用完全灭活,但也有一些细菌尤其是大肠杆菌不易被完全灭活,而且在溶菌诱导后期会出现反弹性生长[7-9]。为了解决这一问题,具有核酸降解能力的金黄色葡萄球菌核酸酶被作为辅助致死蛋白引入菌蜕疫苗的制备之中,以使宿主菌进一步被灭活,同时去除残存的遗传物质,消除潜在的生物安全风险[10-13]。
由于金黄色葡萄球菌核酸酶在基因组成上除了结构基因之外,前端还包括前体序列和信号肽序列[14],在将其应用于菌蜕制备时为避免可能的外泌作用影响效果,只克隆表达其结构基因。但Lee等在相关研究中却发现由λ噬菌体启动子pR指导的SNA的基因表达产物不能降解细菌基因组DNA,而通过融合Cro蛋白N端26个氨基酸片段可成功解决这一问题,他因此推测SNA可能在缺失信号肽的情况下仍能被分泌至胞外[12],与之相应的是Recchi等在将金黄色葡萄球菌核酸酶用于耻垢分枝杆菌外泌蛋白筛选时也观察到类似现象[15]。不过,Fu等在后续研究中却再次证实SNA基因的单纯表达产物即可成功降解细菌基因组DNA,并推断SNA并非必须与其他基因融合,基因的表达效率才是关键[16]。
鉴于目前关于SNA在菌蜕制备中应用形式方面存在的上述争议,本研究通过对争议中所涉及的SNA结构基因及其相关融合片段进行克隆及温控表达,并对相应表达产物的核酸酶活性及对宿主菌的作用效果进行比较,以期厘清这一争议,为高效安全菌蜕疫苗的制备奠定基础。
1 材料与方法 1.1 菌株、质粒及试剂大肠杆菌DH5α感受态细胞购自南京诺唯赞生物科技有限公司,质粒pFLX107、pBV220-SNA由本实验构建保存[16-17]。限制性内切酶XcmⅠ为New England BioLabs产品,KpnⅠ、PstⅠ、T4 DNA连接酶、PrimeSTAR HS (Premix)、Premix ExTaq、DL5000、DL10000等均购自于TaKaRa。DNA Mini Kit、High-Speed Plasmid Mini Kit和Gel/PCR DNA等为Geneaid产品。甲苯胺蓝DNA琼脂购自杭州滨和微生物试剂有限公司。所有引物(表 1)均由赛默飞世尔(Thermo Fisher)旗下英潍捷基(上海)贸易有限公司合成。
| Primer name | Primer sequence (5'-3') | Restriction site |
| SNA-F | CGGGGTACCATGGCAACTTCAACTAAAAA | KpnⅠ |
| SNA-R | AAACTGCAGTTATTGACCTGAATCAGCGT | PstⅠ |
| tSNA-F | ATGGCAACTTCAACTAAAAA | |
| tSNA-R | TTATTGACCTGAATCAGCGT | |
| Cro-F | CGGGGTACCATGGAACAACGCATAACCCT | KpnⅠ |
| Cro-R | AGTTGCATATACGCCGAGATCTTTAGCTGTCTTGG | |
| cSNA-F | ATCTCGGCGTATATGCAACTTCAACTAAAAAATT | |
| cSNA-R | AAACTGCAGTTATTGACCTGAATCAGCGT | PstⅠ |
| Ure-F | CGGGGTACCATGCGCCTGACGCCGCAC | KpnⅠ |
| Ure-R | GTTGAAGTTGCGCGGGCCCGACGCCGGCG | |
| uSNA-F | CGCGCAACTTCAACTAAAAAATTAC | |
| uSNA-R | AAACTGCAGTTATTGACCTGAATCAGC | PstⅠ |
| M13-47 | CGCCAGGGTTTTCCCAGTCACGAC | |
| M13-48 | GAGCGGATAACAATTTCACACAGG |
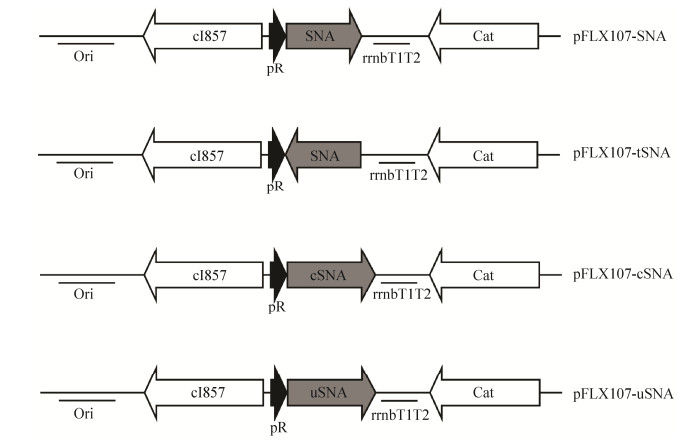
以质粒pBV220-SNA为模板,利用引物SNA-F/SNA-R扩增SNA。反应体系为50 μL,其中模板和正反向引物各1 μL,PrimeSTAR HS (Premix) 25 μL,灭菌双蒸水22 μL。PCR反应程序为:98 ℃变性30 s,55 ℃复性30 s,72 ℃延伸30 s,30个循环,最后72 ℃延伸5 min。PCR产物经1%琼脂糖电泳后切胶回收。用KpnⅠ和PstⅠ对回收目的片段进行双酶切,然后连入同样经KpnⅠ/PstⅠ双酶切的pFLX107质粒中,最终构建成功的质粒命名为pFLX107-SNA (图 1)。
|
| 图 1 温控表达质粒图谱 Figure 1 Maps of thermo-inducible plasmids. Cat: chloramphenicol acetyltransferase gene; Ori: origins of replication; pR: the rightward lambda promoter; cI857: a temperature sensitive repressor cI857; rrnBT1T2: ribosomal RNA operon T1T2 terminator; SNA: staphylococcal nuclease A gene; cSNA: SNA fused with Cro; uSNA: SNA fused with UreA. |
| |
质粒pFLX107-tSNA为通过TA克隆的方式构建。以质粒pBV220-SNA为模板,利用引物tSNA-F/tSNA-R和Premix ExTaq扩增SNA基因,然后回收连入经XcmⅠ酶切回收的pFLX107中,将经测序验证SNA为反向插入的质粒命名为pFLX107-tSNA,作为后续实验的空白对照(图 1)。
1.4 质粒pFLX107-cSNA的构建以质粒pBV220-SNA为模板,先通过引物Cro-F/Cro-R和cSNA-F/cSNA-R分别扩增cro 5′端序列和SNA基因,然后以回收的Cro片段和SNA片段为混合模板、以Cro-F/cSNA-R为引物,通过重叠PCR将Cro片段与SNA融合,最后经KpnⅠ/PstⅠ酶切后连入pFLX107相应酶切位点,构建成质粒pFLX107-cSNA (图 1)。
1.5 质粒pFLX107-uSNA的构建根据已发布序列委托合成结核分枝杆菌脲酶A基因(ureA) 5′端序列[18],然后利用引物Ure-F/Ure-R扩增ureA基因5′端序列。同时,以质粒pBV220-SNA为模板,利用引物uSNA-F/uSNA-R扩增SNA基因。最后通过上述方法将ureA序列与SNA融合并克隆至质粒pFLX107,构建成质粒pFLX107-uSNA (图 1)。
1.6 不同SNA基因的诱导表达所有构建成功的质粒均转化至大肠杆菌DH5α。诱导表达前,将含有相应质粒的大肠杆菌DH5α,接种至100 mL含氯霉素(35 μg/mL)的LB液体培养基中,于28 ℃振荡培养。当培养液OD600达到0.3-0.4时,将温度升至42 ℃诱导基因的表达,同时向培养液中加入CaCl2和MgCl2,使终浓度分别为10 mmol/L和1 mmol/L,以发挥SNA的最大活性。每隔一定时间取样测定培养物的OD600,监测细菌的生长情况。对诱导后0、2和4 h的菌液做适当稀释后,进行平板活菌计数,计算SNA所介导的灭活效率。计算公式为:
灭活率=(1−诱导后CFU/诱导前CFU)×100%。
1.7 基因组检测诱导后每隔1 h取1 mL细菌培养液提取总基因组,确认核酸酶的降解活性。基因组的提取使用Genomic DNA Mini Kit,整个操作按照产品说明书进行。核酸酶对基因组的降解活性通过0.8%琼脂糖电泳分析。
1.8 核酸酶活性测定核酸酶活性的测定参照唐俊妮等[19]的方法,通过甲苯胺蓝DNA琼脂平板进行并略有改动。具体操作为:先配置甲苯胺蓝DNA琼脂,灭菌后倒入平皿中,待凝固后,用直径3 mm的打孔器均匀打孔,取出孔中琼脂。然后待升温诱导后每隔一定时间取样离心,取10 μL上清加入孔中以测定培养液上清中核酸酶的活性,然后弃上清,以等量灭菌LB液体对沉淀菌体进行重悬,煮沸15 min,再离心取10 μL上清加入其他孔中以测定胞内核酸酶的活性,加样后的平板均置于28 ℃过夜培养。上样孔周围蓝色消褪出现粉红色圆圈者为阳性反应,无变色为阴性反应,通过测量粉红圈的直径(不含孔径)以判定核酸酶活性,阴性反应则记录为零。
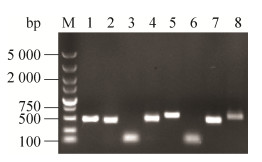
2 结果与分析 2.1 pFLX107系列衍生质粒的构建以质粒pBV220-SNA为模板,SNA-F/SNA-R、tSNA-F/tSNA-R、Cro-F/Cro-R、cSNA-F/cSNA-R和uSNA-F/uSNA-R,引物对成功扩增出大小分别为468 bp (含限制性酶切位点和保护碱基,后同)、450、93、473和462 bp的预期目的片段(图 2);以人工合成ureA基因为模板,引物Ure-F/ Ure-R成功扩增出大小为98 bp的目的片段(图 2)。运用重叠PCR技术,分别以Cro-F/cSNA-R和Ure-F/uSNA-R为引物,均成功扩增出大小约为546 bp的融合片段(图 2)。
|
| 图 2 目的基因的PCR扩增 Figure 2 PCR amplification of target gene. M: DNA marker DL5000; 1: SNA; 2: tSNA; 3: cro; 4: SNA for constructing cSNA; 5: cSNA; 6: ureA; 7: SNA for constructing uSNA; 8: uSNA. |
| |
对构建成功的质粒pFLX107-SNA、pFLX107-tSNA、pFLX107-cSNA、pFLX107-uSNA以M13-47/M13-48引物进行PCR鉴定均能扩增出预期目的片段,分别为673、571、748和748 bp (图 3),测序结果表明,成功构建上述质粒。

|
| 图 3 构建质粒的PCR鉴定 Figure 3 PCR identification of constructed plasmids. M: DNA marker DL5000; 1: pFLX107-SNA; 2: pFLX107-tSNA; 3: pFLX107-cSNA; 4: pFLX107-uSNA. |
| |
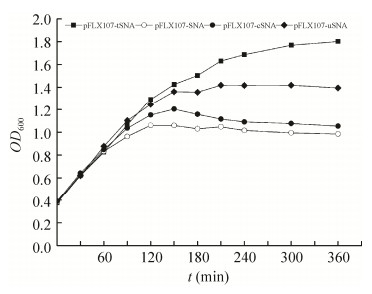
转化有上述构建质粒的大肠杆菌DH5α在28 ℃培养至OD600值约为0.3-0.4后,升温至42 ℃并加入MgCl2/CaCl2进行SNA基因的诱导表达,在核酸酶的作用下其OD600表现出不同的变化规律。
含质粒pFLX107-SNA、pFLX107-cSNA、pFLX107-uSNA的大肠杆菌DH5α经升温诱导后,OD600均经历一段快速增长后缓慢下降最后趋于平稳,其中大肠杆菌DH5α (pFLX107-SNA) OD600在120 min达到最大值后开始缓慢下降,180 min后稳定于约1.0左右;大肠杆菌DH5α (pFLX107-cSNA)的OD600于150 min达到最大值后缓慢下降,210 min后稳定于1.1左右;大肠杆菌DH5α (pFLX107-uSNA)的OD600于210 min达到最大值后一直稳定维持于1.4左右。作为对照的大肠杆菌DH5α (pFLX107-tSNA) OD600则在整个实验期间一直持续增长直至达到平台期(OD600=1.8) (图 4)。
|
| 图 4 大肠杆菌DH5α的生长曲线 Figure 4 Growth curve of E.coli DH5α carrying pFLX107-SNA, pFLX107-tSNA, pFLX107-cSNA and pFLX107-uSNA, respectively. The cells were grown at 28 ℃ until the OD600 reached 0.3-0.4, and then was induced by the upshift of temperature from 28 ℃ to 42 ℃. Mg2+ and Ca2+were added to the culture simultaneously. |
| |
平板活菌计数从另一方面反映了SNA的作用效果。诱导后2 h和4 h,转化有质粒pFLX107-SNA或pFLX107-cSNA的大肠杆菌DH5α活菌数均较诱导前下降了2-3个数量级,大肠杆菌DH5α (pFLX107-SNA)的灭活率分别为99.7%和99.9%,大肠杆菌DH5α (pFLX107-cSNA)的灭活率分别为99.5%和99.8%;而转化有质粒pFLX107-uSNA的大肠杆菌DH5α活菌数则只较诱导前降低了不到1个数量级,灭活率分别为55.7%和74.2%。相较之下,大肠杆菌DH5α (pFLX107-tSNA)活菌数在实验期间一直处于增长之中,诱导后2 h和4 h的活菌数分别为诱导前的1.6倍和3.5倍(表 2)。
| Bacteria (plasmid) | Number of CFU (Inactivation rate) | ||
| 0 h | 2 h | 4 h | |
| E. coli DH5α (pFLX107-tSNA) | 8.6×107 | 1.4×108 | 3.0×108 |
| E. coli DH5α (pFLX107-SNA) | 1.4×108 | 3.8×105(99.7%) | 2.1×105(99.9%) |
| E. coli DH5α (pFLX107-cSNA) | 1.1×108 | 5.1×105(99.5%) | 1.9×105(99.8%) |
| E. coli DH5α (pFLX107-uSNA) | 9.7×107 | 4.3×107(55.7%) | 2.5×107(74.2%) |
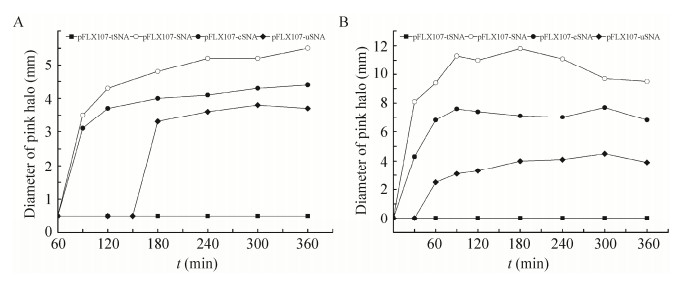
为了解不同重组质粒中金黄色葡萄球菌核酸酶基因的诱导表达及可能存在的外泌情况,对诱导后不同时段的培养液上清和胞内核酸酶进行活性检测。活性检测阳性反应会在点样孔周围形成粉红色圆圈,其直径大小可在一定程度上反映核酸酶的活性。
升温诱导90 min后,大肠杆菌DH5α (pFLX107-SNA)和大肠杆菌DH5α (pFLX107-cSNA)培养液上清中可首次检测到核酸酶,此后呈缓慢增加趋势,以最后4组测量值计算的平均粉红圈直径分别为(4.7±0.3) mm和(3.7±0.2) mm (后同);大肠杆菌DH5α (pFLX107-uSNA)则在诱导180 min后方在其培养液上清中检测到核酸酶,平均粉红圈直径为(3.1±0.2) mm (图 5A)。与之相对的是,大肠杆菌DH5α (pFLX107-SNA)和大肠杆菌DH5α (pFLX107-cSNA)胞内核酸酶在诱导30 min后即可被检测到,大肠杆菌DH5α (pFLX107-uSNA)则在诱导60 min后可检测到胞内核酸酶,三者的平均粉红圈直径分别为(10.5±1.1) mm、(7.2±0.4) mm和(4.1±0.3) mm (图 5B)。而作为空白对照的大肠杆菌DH5α (pFLX107-tSNA)则在历次核酸酶活性检测中均为阴性(图 5)。
|
| 图 5 大肠杆菌DH5α培养液上清(A)和胞内(B)核酸酶活性 Figure 5 Nuclease activity of culture supernatant (A) and intracellular nuclease activity (B) of E. coli DH5α carrying pFLX107-SNA, pFLX107-tSNA, pFLX107-cSNA and pFLX107-uSNA, respectively. |
| |
在钙、镁离子存在时,升温诱导2 h后,大肠杆菌DH5α (pFLX107-SNA)和大肠杆菌DH5α (pFLX107-cSNA)的基因组即被成功降解(图 6),而大肠杆菌DH5α (pFLX107-uSNA)和大肠杆菌DH5α (pFLX107-tSNA)的基因组在整个实验期间则始终可被检测到,但通过电泳条带的明暗强度可看出前者基因组浓度诱导后期较诱导前期有所下降(图 6C),而后者基因组浓度则不断增加(图 6D)。

|
| 图 6 大肠杆菌DH5α总基因组电泳分析 Figure 6 Electrophoretic analysis of total DNA of E. coli DH5α harboring pFLX107-SNA (A), pFLX107-cSNA (B), pFLX107-uSNA (C) and pFLX107-tSNA (D), respectively. M: DNA marker DL10000; Lane 1-6: samples prepared from the induced culture every one hour after induction. |
| |
作为一种新型的疫苗技术,菌蜕较传统疫苗的优势已在各类实验中一再得到证实[1-4],但在菌蜕的制备过程中,单纯基因E所介导的裂解作用并不足以使宿主菌完全灭活[7-9]。出于生物安全考虑,SNA被作为辅助致死基因引入菌蜕的制备之中,但如前所述,目前关于SNA在菌蜕制备中的应用形式存在争议,与之对应的理论解释也截然相反[10-13],因此厘清这一争议对于高效安全菌蜕疫苗的生产或制备无疑具有十分重要的理论意义和经济价值。
本研究通过对争议中所涉及的SNA结构基因及其相关融合片段进行克隆及温控表达,并从不同方面对其作用效果进行了比较。本次研究中,无论是SNA的单纯表达产物还是其与Cro或脲酶N端氨基序列的融合产物均具有核酸酶活性,而且综合比较之下单纯SNA的活性最强,cSNA次之,uSNA则活性最低,显示外源片段的融入反而会在一定程度上降低SNA的活性,降低程度则因融入的片段而异,这可能与融入片段会影响到SNA的折叠并进而影响到其活性有关。此外,升温诱导后三者在宿主菌胞内被检出的时间均要早于其在培养液上清中被检出的时间,且对应胞内核酸酶活性均显著高于培养液上清中的核酸酶活性,这表明三者可能皆如Recchi等对uSNA所描述的那样不能外泌,只是由于过表达和宿主菌灭活致死才被动释放至胞外。
另一方面,在菌蜕的制备过程中,裂解基因E会在宿主菌表面形成平均直径约40-200 nm的跨膜通道,导致胞质内容物的释放[20],这意味着将SNA应用于菌蜕制备时无论其自身能否外泌均会被释放至胞外,因此Lee等认为核酸酶只有在胞内才能发挥作用的解释值得商榷。根据其描述通过软件重构相关质粒发现,当SNA基因被直接置于pR控制之下时,SD盒与起始密码子之间的距离为57 bp,包含有许多起始构建质粒pGEM-T easy vector自身酶切位点及居间序列,并非如之前所分析的那样只是增加了一个限制性酶切位点[16],而这一距离的最适范围通常为5-13 bp,超过20 bp就会阻碍后续基因的表达[21],因此两者之间距离过长可能才是导致SNA诱导后不能发挥作用的真正原因。进一步分析显示,当Lee等进行Cro和SNA融合时,并没如其所言用pR-Cro元件替换质粒中的pR,而是直接将这一组合元件插入在前述57 bp序列中的SalⅠ和NdeⅠ酶切位点之间,对应于Cro蛋白N端26个氨基酸片段的核酸序列实际上也只包含65 bp的Cro 5′端序列,鉴于此后这一存在移码的Cro和SNA的表达产物仍能正常发挥作用,一种可能的解释是这65 bp序列中存在着能影响基因转录或表达的序列元件,类似现象曾在裂解基因E的体外DNA重组(DNA shuffling)中出现过,缺失了1个起始碱基的基因E反而具有更高的裂解效率[22]。此时,Cro和SNA的融合表达产物实际上仍为单纯SNA。
最后,本次研究中uSNA在整个实验期间尽管表现出了对DNA的降解能力但并不能使宿主基因组完全降解,对宿主菌的灭活也不到一个数量级,而SNA和cSNA在升温诱导2 h后即将宿主基因组成功降解,对宿主菌的灭活效果也较uSNA高出1-2个数量级,则从另一方面反映了一个值得注意的问题,即核酸酶的活性才是决定对宿主菌的灭活水平和对基因组降解能力的关键。
| [1] | Mayr UB, Kudela P, Atrasheuskaya A, et al. Rectal single dose immunization of mice withEscherichia coli O157: H7 bacterial ghosts induces efficient humoral and cellular immune responses and protects against the lethal heterologous challenge. Microb Biotechnol, 2012, 5 (2) : 283–294. DOI: 10.1111/mbt.2012.5.issue-2 |
| [2] | Jawale CV, Lee JH. Evaluation of immunogenicity and protective efficacy of adjuvanted Salmonella typhimuriumghost vaccine against salmonellosis in chickens. Vet Quart, 2016, 36 (3) : 130–136. DOI: 10.1080/01652176.2016.1138248 |
| [3] | Liu J, Li Y, Sun Y, et al. Immune responses and protection induced by Brucella suis S2 bacterial ghosts in mice. Vet Immunol Immunopathol, 2015, 166 (3/4) : 138–144. |
| [4] | Hajam IA, Dar PA, Appavoo E, et al. Bacterial ghosts of Escherichia colidrive efficient maturation of bovine monocyte-derived dendritic cells. PLoS ONE, 2015, 10 (12) : e0144397. DOI: 10.1371/journal.pone.0144397 |
| [5] | Muhammad A, Champeimont J, Mayr UB, et al. Bacterial ghosts as carriers of protein subunit and DNA-encoded antigens for vaccine applications. Expert Rev Vaccines, 2012, 11 (1) : 97–116. DOI: 10.1586/erv.11.149 |
| [6] | Ganeshpurkar A, Ganeshpurkar A, Pandey V, et al. Harnessing the potential of bacterial ghost for the effective delivery of drugs and biotherapeutics. Int J Pharm Investig, 2014, 4 (1) : 1–4. DOI: 10.4103/2230-973X.127733 |
| [7] | Langemann T, Koller VJ, Muhammad A, et al. The bacterial ghost platform system: production and applications. Bioeng Bugs, 2010, 1 (5) : 326–336. DOI: 10.4161/bbug.1.5.12540 |
| [8] | Haidinger W, Szostak MP, Beisker W, et al. Green fluorescent protein (GFP)-dependent separation of bacterial ghosts from intact cells by FACS. Cytometry, 2001, 44 (2) : 106–112. DOI: 10.1002/(ISSN)1097-0320 |
| [9] | Haidinger W, Mayr UB, Szostak MP, et al. Escherichia coli ghost production by expression of lysis gene E and staphylococcal nuclease. Appl Environ Microbiol, 2003, 69 (10) : 6106–6113. DOI: 10.1128/AEM.69.10.6106-6113.2003 |
| [10] | Mayr UB, Haller C, Haidinger W, et al. Bacterial ghosts as an oral vaccine: a single dose of Escherichia coliO157: H7 bacterial ghosts protects mice against lethal challenge. Infect Immun, 2005, 73 (8) : 4810–4817. DOI: 10.1128/IAI.73.8.4810-4817.2005 |
| [11] | Liu J, Wang WD, Liu YJ, et al. Mice vaccinated with enteropathogenicEscherichia coli ghosts show significant protection against lethal challenges. Lett Appl Microbiol, 2012, 54 (3) : 255–262. DOI: 10.1111/lam.2012.54.issue-3 |
| [12] | Lee DJ, Kwon SR, Zenke K, et al. Generation of safety enhanced Edwardsiella tarda ghost vaccine. Dis Aquat Organ, 2008, 81 (3) : 249–254. |
| [13] | Kwon SR, Kang YJ, Lee DJ, et al. Generation of Vibrio anguillarum ghost by coexpression of phiX174 lysis E gene and staphylococcal nuclease A gene. Mol Biotechnol, 2009, 42 (2) : 154–159. DOI: 10.1007/s12033-009-9147-y |
| [14] | Shortle D. A genetic system for analysis of staphylococcal nuclease. Gene, 1983, 22 (2-3) : 181–189. DOI: 10.1016/0378-1119(83)90102-6 |
| [15] | Recchi C, Rauzier J, Gicquel B, et al. Signal-sequence-independent secretion of the staphylococcal nuclease inMycobacterium smegmatis. Microbiology, 2002, 148 (Pt 2) : 529–536. |
| [16] | Fu LX, Lu CP. A novel dual vector coexpressing phiX174 lysis E gene and staphylococcal nuclease A gene on the basis of lambda promoter pR and pL, respectively. Mol Biotechnol, 2013, 54 (2) : 436–444. DOI: 10.1007/s12033-012-9581-0 |
| [17] |
Fu LX, Wei WZ, Wang XY. Temperature-controlled zero background T-vector precursor and construction method and application thereof: China, CN 201310488129.7. 2015-01-07 (in Chinese). 付立霞, 魏文志, 王秀英.一种温控零背景T载体前体及其构建方法和应用:中国, CN 201310488129.7. 2015-01-07. |
| [18] | Clemens DL, Lee BF, Horwitz MA. Purification, characterization, and genetic analysis of Mycobacterium tuberculosis urease, a potentially critical determinant of host-pathogen interaction. J Bacteriol, 1995, 177 (19) : 5644–5652. DOI: 10.1128/jb.177.19.5644-5652.1995 |
| [19] |
Tang JN, Shi XM, Zhang R, et al. The influences of different conditions on thermonuclease activity in Staphylococcus aureus.
J Southwest Univ Nationalities: Nat Sci Ed, 2010, 36 (6) : 589–592.
(in Chinese). 唐俊妮, 史贤明, 张荣, 等. 不同条件对金黄色葡萄球菌耐热核酸酶活性影响的研究. 西南民族大学学报:自然科学版, 2010 , 36 (6): 589-592. |
| [20] | Witte A, Wanner G, Bl si U, et al. Endogenous transmembrane tunnel formation mediated by phiX174 lysis protein E. J Bacteriol, 1990, 172 (7) : 4109–4114. DOI: 10.1128/jb.172.7.4109-4114.1990 |
| [21] | Ma Y, Li AX, Wang YC, et al. Construction and experimental application of a highly efficient temperature-selection T-vector. Biotechnol Appl Biochem, 2009, 53 (4) : 247–251. |
| [22] | Yu SY, Peng W, Si W, et al. Enhancement of bacteriolysis of shuffled phage phiX174 gene E. Virol J, 2011, 8 (1) : 1–6. DOI: 10.1186/1743-422X-8-1 |
 2016, Vol. 32
2016, Vol. 32




