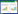服务
文章信息
- 桂海娈, 金庆日, 张亚军, 王晓杜, 杨永春, 邵春艳, 程昌勇, 卫芳芳, 杨杨, 杨梦华, 宋厚辉
- Gui Hailuan, Jin Qingri, Zhang Yajun, Wang Xiaodu, Yang Yongchun, Shao Chunyan, Cheng Changyong, Wei Fangfang, Yang Yang, Yang Menghua, Song Houhui
- 基于荧光染料PicoGreen 和核酸适配体的伏马毒素B1 检测方法
- Development of an aptamer/fluorescence dye PicoGreen-based method for detection of fumonisin B1
- 生物工程学报, 2015, 31(9): 1393-1400
- Chin J Biotech, 2015, 31(9): 1393-1400
- 10.13345/j.cjb.140590
-
文章历史
- Received: November 30, 2014
- Accepted: January 30, 2015
伏马毒素 (Fumonisin,FB) 是一种对啮齿动物具有致癌作用的真菌毒素,主要破坏肝肾功能[1],诱发多种疾病,如马脑白质软化症和猪肺水肿综合症[2],已被证明对多种家畜、实验动物有害[3]。目前FB有11种,主要由轮状、层状镰刀霉菌产生,其中FB1最常见[4]。FB1主要存在于玉米及玉米制品中,小麦、牛奶等食品及饲料易被污染[5]。世界卫生组织对FB限量标准因各地的玉米及玉米制品的产量不同而异,美国食品与药物管理局规定以玉米为原料的食品中 FB的最高限量为2 mg/kg,动物饲料的限量范围为1−50 mg/kg。
目前检测FB1的方法主要是薄层色谱法[6]、高效液相色谱法[7]、酶联免疫吸附试验 (ELISA)[8]等。这些方法对检测仪器及操作人员要求较高,因此不宜进行现场检测;其中,基于抗体的ELISA方法所需的抗体制备繁琐[9],因此亟待建立一种快速检测FB1的方法。
核酸适配体 (Aptamer) 是一种功能与抗体相似的单链寡核苷酸。经指数富集配体的系统进化技术[10]筛选得到的核酸适配体可与目标物质特异性结合[11],如蛋白质[12]和药物[13]等。核酸适配体可直接合成使用,经济实用,易修饰且无免疫原性[14]。现已报道的基于核酸适配体的检测方法有:磁珠法[15]、胶体金法[16]、荧光标记法[17]等。
本文选用已报道的亲和度最高的FB1特异性核酸适配体,利用非标记荧光染料PicoGreen识别双链核酸的特异性,建立了一种快速、高效检测FB1的方法。该方法的原理[18]是:当核酸适配体与FB1结合后,多余的、未结合FB1的核酸适配体可以与反应体系中核酸适配体的互补链杂交生成双链DNA。由于PicoGreen不结合单链核苷酸,仅识别并结合双链DNA的螺旋沟,继而可以释放荧光信号[19]。当不同浓度的FB1与特定浓度的核酸适体结合时,反应液中的荧光强度不同,从而可以实现快速定量检测FB1的目的。
1 材料与方法 1.1 仪器与试剂96孔板购自Conning公司 (Costar2592);荧光检测仪购自BioTek公司 (Synergy™ H1);超纯水仪购自Thermo Fisher Scientific公司 (D11921)。
FB1、赭曲霉毒素A (Ochratoxin A,OTA)、黄曲霉毒素B1 (Aflatoxin B1,AFB1)、桔毒素 (Citrinin,CTN) 和玉米赤霉烯酮 (Zearalenone,ZEN) 购自Sigma公司;PicoGreen购自 Life Technologies公司 (P7581);伏马毒素ELISA检测试剂盒购自Reagen公司 (RNM98009,检测线性范围:1−50 ppb)。
FB1核酸适配体 (FB1 39号)[20]、核酸适配体互补链Seq1 (表1) 由生工生物工程 (上海) 股份有限公司合成。
| Oligonucleotide | Sequence (5¢-3¢) |
| FB1 aptamer | AATCGCATTACCTTATACCAGCTTATTCAATTACGTCTGCACATACCAGCTTATTCAATT |
| Seq1 | AATTGAATAAGCTGGTATGTGCAGACGTAATTGAATAAGCTGGTATAAGGTAATGCGATT |
PicoGreen是一种定量检测双链DNA的染料,灵敏度可达pg级,与双链DNA结合后荧光增强1 000倍以上,且不与单链DNA结合。当FB1核酸适配体结合FB1后,适配体的空间构象发生变化,不能再与其互补链Seq1结合。未与FB1结合的核酸适配体可与其互补链杂交形成双链DNA。由此产生的双链DNA可通过由PicoGreen检测,通过荧光信号的变化确定样品中的FB1浓度[21] (图1)。
|
| 图1 PicoGreen和适配体检测FB1的原理图 Fig.1 Schematic presentation of the PicoGreen/aptamer- based method for detection of FB1 |
所有反应试剂以10 mmol/L Tris、120 mmol/L NaCl、5 mmol/L KCl、20 mmol/L CaCl2 (pH 8.5) 组成的缓冲液[22]为稀释溶液。分别取50 μL 10 nmol/L核酸适配体与50 μL 10 nmol/L 互补链Seq1,于95 ℃变性5 min,静置于冰上10 min[23]。将核酸适配体与互补链于孔内混匀,于25 ℃反应5 min[24],再加入10 μL 10×PicoGreen溶液避光反应,检测荧光强度 (参数设置:激发波长为480 nm,发射波长为520 nm)。
1.4 PicoGreen识别双链DNA的时间优化取50 μL核酸适配体和50 μL互补链Seq1于孔内混匀 (1∶1),于25 ℃反应5 min,再加入10 μL 10×PicoGreen后立即放入荧光检测仪内,连续检测30 min。
1.5 FB1检测的灵敏度试验将FB1稀释成质量浓度为0−1 μg/L的溶液,取50 μL FB1溶液与25 μL 20 nmol/L核酸适配体混匀,25 ℃反应20 min,加入25 μL 20 nmol/L互补链Seq1溶液反应5 min,再加入10 μL 10×PicoGreen,混匀避光,检测荧光强度。
1.6 核酸适配体特异性试验稀释FB1、OTA、AFB1、CTN和ZEN质量浓度为1 μg/L,分别取50 μL霉菌毒素溶液与25 μL 20 nmol/L FB1核酸适配体混匀,25 ℃反应20 min,加入25 μL 20 nmol/L互补链Seq1溶液反应5 min,再加入10 μL 10×PicoGreen,混匀避光,检测荧光强度。
1.7 检测样品试验在含1%牛奶的溶液内添加FB1,其中28份样品内含有FB1,浓度分别为1、2、2.5、5、10、50和100 μg/L (样品重复数n =4),10份样品无FB1,使用本文建立的方法检测,计算实际检测值与理论值的比值。同时用商品化的ELISA试剂盒方法进行检测,计算Kappa值,并评估两种方法是否一致。本研究方法:将样品稀释为 1 μg/L,取50 μL样品溶液与25 μL 20 nmol/L核酸适配体混匀,25 ℃反应20 min,加入25 μL 20 nmol/L互补链Seq1溶液反应5 min,再加入10 μL 10×PicoGreen,混匀避光,检测荧光强度。ELISA检测试剂盒操作方法:在包被有FB1抗原的微孔板中,每孔加入50 μL样品溶液,再依次加入50 μL HRP标记的二抗,50 μL FB1抗体,室温孵育30 min,用300 μL洗涤缓冲液清洗3次,每孔加入100 μL TMB溶液,室温孵育15 min,再加入100 μL终止液终止反应,读取OD450,根据制作的标准曲线计算样品中OTA含量。计算ELISA方法和本试验建立的核酸适体-荧光染料检测法之间的Kappa值[25]。
2 结果与分析 2.1 PicoGreen对FB1核酸适配体形成的双链DNA的检测建立基于PicoGreen和核酸适配体方法检测FB1的前提是确定PicoGreen分别与适配体单链核苷酸、适配体和互补链之间形成的双链核苷酸、缓冲液、FB1反应后是否释放荧光,该荧光是否可以被仪器识别并能用于后续的检测。
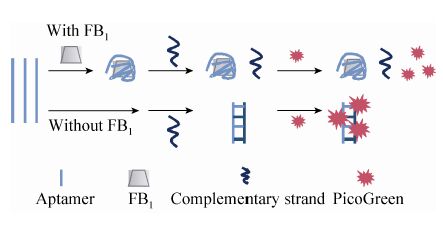
结果显示,核酸适配体与互补链Seq1的杂交产物双链DNA结合PicoGreen后能够释放荧光信号,可以被荧光检测仪捕捉。单链核苷酸或缓冲液本身不与PicoGreen结合,不能释放出荧光信号 (图2)。这说明PicoGreen和核酸适配体可以用于FB1毒素检测技术的开发。
|
| 图2 FB1核酸适配体形成的双链DNA与PicoGreen结合后的荧光检测 Fig.2 Detection of PicoGreen fluorescence in the presence of FB1 aptamer hybridizer |
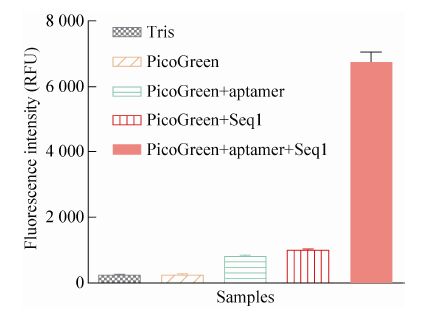
PicoGreen结合双链DNA后会被瞬间激发产生荧光,随着时间推移荧光强度不断增强,直到反应体系中PicoGreen与双链DNA的结合达到饱和为止。对于FB1特异性检测技术的开发,加入反应体系中的PicoGreen和核酸适配体浓度均为固定值,因此需要确定在特定浓度下荧光强度达到峰值的时间。由图3可知,反应体系内加入PicoGreen后,PicoGreen与FB1核酸适配体形成的双链DNA结合后随即释放出荧光,随着反应时间的推移,荧光强度在15 min时趋于稳定,并达到峰值。当PicoGreen浓度固定时,荧光强度随双链DNA的增多而增强,直到PicoGreen完全结合双链DNA为止。因此,在后续的检测技术的开发中,选择15 min 作为PicoGreen与样品的反应时间。
|
| 图3 PicoGreen与双链DNA最佳反应时间的确定 Fig.3 Determination of the optimal reaction time of PicoGreen and double-stranded DNA. F0 and F stand for the fluorescence intensity with or without DNA |
灵敏度又称敏感性或最低检测限,是检测技术开发的一个关键参数。根据上述确定的最佳反应时间,利用PicoGreen和核酸适配体方法对不同浓度的FB1进行检测。结果表明,在反应体系中随着FB1质量浓度的增加,PicoGreen释放的荧光强度逐渐减小 (图4A)。这说明FB1结合的核酸适配体增多,导致与后加入的互补链Seq1结合的核酸适配体减少,因此与PicoGreen结合的双链DNA量减少,激发产生的荧光强度减小。该方法的检测下限为0.1 μg/L (0.1 ppb),线性范围为0.1−1 μg/L (0.1−1 ppb) (图4B)。

|
| 图4 FB1的灵敏度检测 Fig.4 Sensitivity test of the aptamer/PicoGreen-based method. (A) Sensitivity determination at FB1 concentrations ranging from 0.1 to 1 μg/L. (B) Standard curve plotted with F/F0 against the concentration of FB1. F0 and F stand for the fluorescence intensity with or without FB1 |
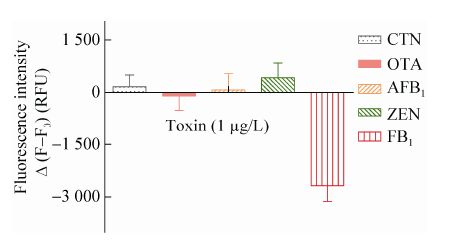
特异性又称交叉反应性,是评价检测技术好坏的一个重要标志。为了证实本检测技术采用的核心试剂FB1适配体和PicoGreen是否只能用于检测FB1,不能和其他毒素发生交叉反应。我们对食品中常见的霉菌毒素FB1、OTA、ZEN、CTN、AFB1进行了检测。结果表明,毒素质量浓度相同时,PicoGreen检测OTA、ZEN、CTN、AFB1反应体系的荧光强度最大,而FB1反应体系的荧光强度最小 (图5)。这说明在有FB1的反应体系内,核酸适配体与FB1发生了特异性结合,少量的核酸适配体与互补链Seq1结合,产生与PicoGreen结合的双链DNA,因此激发产生少许荧光;其他毒素的反应体系中的核酸适配体与互补链Seq1完全结合,产生大量双链DNA,激发产生更多的荧光。即当样品中有FB1时,荧光强度较小,反之荧光强度较大。由此可知,本文选择的核酸适配体可特异性识别并结合FB1,与OTA、AFB1、CTN或ZEN不发生交叉反应。
|
| 图5 基于核酸适配体与PicoGreen检测FB1的特异性试验 Fig.5 Specificity test of the aptamer/PicoGreen-based method |
为了比较基于核酸适体的荧光检测法与基于抗体的商业化ELISA检测试剂盒方法之间的符合度,以验证本方法检测样品的准确度。本实验室对38份牛奶样品进行了检测,计算出本方法的回收率 (表2),低浓度条件下,回收率较低;当浓度大于10 μg/L时,回收率高达99%。利用统计方法计算Kappa值为0.857 (检测样品浓度为0、1、2、2.5、5、10 μg/L),两种方法的检测结果具有良好的一致性 (表3),可用于样品中FB1的检测。
| FB1 concentration (μg/L) | Recovery rate (%) | |||
| Spiked (n=4) | Aptamer/ PicoGreen | ELISA | Aptamer/ PicoGreen | ELISA |
| 0 | 0 | 0 | 0 | 0 |
| 1.00 | 0.05 | 0.28 | 4.92 | 28.45 |
| 2.00 | 0.26 | 1.07 | 13.11 | 53.33 |
| 2.50 | 0.23 | 1.33 | 9.06 | 53.19 |
| 5.00 | 0.47 | 3.80 | 9.36 | 75.95 |
| 10.00 | 9.93 | 10.26 | 99.26 | 102.64 |
| 50.00 | 48.75 | 97.50 | ||
| 100.00 | 112.01 | 112.01 | ||
| Aptamer/PicoGreen | ELISA | Total | |
| + | - | ||
| + | 18 | 0 | 18 |
| - | 2 | 10 | 12 |
| Total | 20 | 10 | 30 |
| Kappa value | 0.857 | ||
| +: positive; -: negative; n=4 |
食品安全问题一直是世界关注的焦点,其中霉菌毒素污染最为常见。FB1主要污染玉米和玉米制品,严重危害人类和动物的健康[26]。食品和饲料行业的检测目前主要是免疫法和色谱分析法,存在设备与试剂昂贵、样品处理繁琐等不足之处[27]。
核酸适配体是完全不同于抗体的一种单链核苷酸,可形成茎环、发夹和G-四聚体 (G-quadruplex) 等结构并与靶分子特异性结合,目前已被广泛应用于生物样品的检测[28]。
由本研究建立的基于核酸适配体的荧光检测法选择的FB1核酸适配体具有高特异性,与常见的霉菌毒素不发生交叉反应。该荧光检测法的检测灵敏度为:0.1 μg/L,比商业化的检测灵敏度高10倍 (美国Reagen公司,RNM98009,检测灵敏度为:1.0 μg/L)。
本研究建立的检测方法线性范围0.1−1 μg/L是根据伏马毒素标准品 (纯品) 进行的检测。回收率是根据人工污染牛奶样品进行的检测。对于人工污染的样品,需要利用甲醇萃取等步骤进行回收,然后才能按照标准步骤进行FB1含量检测。原始样品中伏马毒素含量越少,回收时损失越大。当样品中伏马毒素含量超过10 μg/L (μg/kg) 时,本方法的回收率可以达到97%以上,而国际上对食品和动物性饲料中允许的最大残留量分别是1 000−2 000 μg/kg,因此可以用于实际样品的检测。所以本实验针对的原始样品中伏马毒素的回收率是符合要求的,检测线性范围和回收率不矛盾。为保证检测结果的准确性,当样品中FB1含量超过1 μg/L时,需要将样品稀释到线性范围内。
本研究建立的基于核酸适配体与荧光染料PicoGreen的新方法将反应体系内未知浓度的FB1转换为可以定量检测的荧光信号,达到检测FB1的目的,特异性强,灵敏度高。整个检测过程可在40 min内完成,具备快速、易操作的特点。本研究建立的基于核酸适配体与PicoGreen检测FB1的方法为霉菌毒素的检测提供了新的思路,具有广阔的应用前景。
| [1] | Bryla M, Roszko M, Szymczyk K, et al. Fumonisins in plant-origin food and fodder-a review. Food Addit Contam Part A Chem Anal Control Expo Risk Assess, 2013, 30(9): 1626-1640. |
| [2] | Gelderblom WC, Jaskiewicz K, Marasas WF, et al. Fumonisins-novel mycotoxins with cancer-promoting activity produced by Fusarium moniliforme. Appl Environ Microbiol, 1988, 54(7): 1806-1811. |
| [3] | Gelderblom WCA, Kriek NPJ, Marasas WFO, et al. Toxicity and carcinogenicity of the Fusarium moniliforme metabolite, fumonisin B1 in rats. Carcinogenesis, 1991, 12(7): 1247-1251. |
| [4] | Queiroz B, Pereyra CM, Keller KM, et al. Fungal contamination and determination of fumonisins and aflatoxins in commercial feeds intended for ornamental birds in Rio de Janeiro, Brazil. Lett Appl Microbiol, 2013, 57(5): 405-411. |
| [5] | Chu FS, Li GY. Simultaneous occurrence of fumonisin B1 and other mycotoxins in moldy corn collected from the People's Republic of China in regions with high incidences of esophageal cancer. Appl Environ Microbiol, 1994, 60(3): 847-852. |
| [6] | Shelby RA, Rottinghaus GE, Minor HC. Comparison of thin-layer chromatography and competitive immunoassay methods for detecting fumonisin on maize. J Agric Food Chem, 1994, 42(9): 2064-2067. |
| [7] | Shephard GS, Sydenham EW, Thiel PG, et al. Quantitative determination of fumonisins B1 and B2 by high-performance liquid chromatography with fluorescence detection. J Liq Chromatogr, 1990, 13(10): 2077-2087. |
| [8] | Quan Y, Zhang Y, Wang S, et al. A rapid and sensitive chemiluminescence enzyme-linked immunosorbent assay for the determination of fumonisin B1 in food samples. Anal Chim Acta, 2006, 580(1): 1-8. |
| [9] | De Girolamo A, Solfrizzo M, Visconti A, et al. Comparison of different extraction and clean-up procedures for the determination of fumonisins in maize and maize-based food products. Food Addit Contam, 2001, 18(1): 59-67. |
| [10] | Ellington AD, Szostak JW. In vitro selection of RNA molecules that bind specific ligands. Nature, 1990, 346(30): 818-822. |
| [11] | Ellington AD, Szostak JW. Selection in vitro of single-stranded DNA molecules that fold into specific ligand-binding structures. Nature, 1992, 335(27): 850-852. |
| [12] | Xu W, Ellington AD. Anti-peptide aptamers recognize amino acid sequence and bind a protein epitope. Proc Natl Acad Sci USA, 1996, 93(15): 7475-7480. |
| [13] | Cerchia L, Hamm J, Libri D, et al. Nucleic acid aptamers in cancer medicine. FEBS Lett, 2002, 528(1/3): 12-16. |
| [14] | Clark SL, Remcho VT. Aptamers as analytical reagents. Electrophoresis, 2002, 23(9): 1335-1340. |
| [15] | Nguyen BH, Tran LD, Do QP, et al. Label-free detection of aflatoxin M1 with electrochemical Fe3O4 polyaniline-based aptasensor. Mater Sci Eng C Mater Biol Appl, 2013, 33(4): 2229-2234. |
| [16] | Wang L, Ma W, Chen W, et al. An aptamer-based chromatographic strip assay for sensitive toxin semi-quantitative detection. Biosens Bioelectron, 2011, 26(6): 3059-3062. |
| [17] | Zhao Q, Geng X, Wang H. Fluorescent sensing ochratoxin A with single fluorophore-labeled aptamer. Anal Bioanal Chem, 2013, 405(19): 6281-6286. |
| [18] | Lv ZZ, Chen AL, Liu JC, et al. A simple and sensitive approach for ochratoxin A detection using a label-free fluorescent aptasensor. PLoS ONE, 2014, 9(1): e85968. |
| [19] | Singer VL, Jones LJ, Yue ST, et al. Characterization of PicoGreen reagent and development of a fluorescence-based solution assay for double-stranded DNA quantitation. Anal Biochem, 1997, 249(2): 228-238. |
| [20] | McKeague M, Bradley CR, Girolamo AD, et al. Screening and initial binding assessment of fumonisin B1 aptamers. Int J Mol Sci, 2010, 11(12): 4864-4881. |
| [21] | Lv ZZ, Liu JC, Zhou Y, et al. Highly sensitive fluorescent detection of small molecules, ions, and proteins using a universal label-free aptasensor. Chem Commun (Camb), 2013, 49(48): 5465-5467. |
| [22] | Cruz-Aguado JA, Penner G. Determination of ochratoxin A with a DNA aptamer. J Agric Food Chem, 2008, 56(22): 10456-10461. |
| [23] | Chen X, Huang Y, Duan N, et al. Selection and identification of ssDNA aptamers recognizing zearalenone. Anal Bioanal Chem, 2013, 405(20): 6573-6581. |
| [24] | Chen J, Fang Z, Liu J, et al. A simple and rapid biosensor for ochratoxin A based on a structure-switching signaling aptamer. Food Control, 2012, 25(2): 555-560. |
| [25] | Landis JR, Koch GG. The measurement of observer agreement for categorical data. Biometrics, 1977, 33(1): 159-174. |
| [26] | Visconti A, Doko MB. Survey of fumonisin production by Fusarium isolated from cereals in Europe. J AOAC Int, 1993, 77(2): 546-550. |
| [27] | Scott PM, Trucksess MW. Application of immunoaffinity columns to mycotoxin analysis. J AOAC Int, 1996, 80(5): 941-949. |
| [28] | Song S, Wang L, Li J, et al. Aptamer-based biosensors. TrAC-Trend Anal Chem, 2008, 27(2): 108-117. |
 2015, Vol. 31
2015, Vol. 31