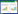服务
文章信息
- 冯万军, 邢国芳, 牛旭龙, 窦晨, 韩渊怀
- Feng Wanjun, Xing Guofang, Niu Xulong, Dou Chen, Han Yuanhuai
- 植物谷氨酰胺合成酶研究进展及其应用前景
- Progress and application prospects of glutamine synthase in plants
- 生物工程学报, 2015, 31(9): 1301-1312
- Chin J Biotech, 2015, 31(9): 1301-1312
- 10.13345/j.cjb.140585
-
文章历史
- Received: November 27, 2014
- Accepted: March 6, 2015
2. 山西农业大学信息学院,山西 太谷 030800
2. College of Information, Shanxi Agricultural University, Taigu 030800, Shanxi, China
氮素是制约作物产量的主要营养元素之一。近几十年来,作物产量的迅速增加在很大程度上依赖于氮肥的大量施用,但是施加的氮素仅有30%−40%可以最终被作物吸收利用[1]。目前,中国也面临农民盲目施肥和作物对氮素的利用效率低下的问题。氮肥过量施加不仅造成了经济浪费,更重要的是导致农田土壤酸化,从而加速土壤营养元素流失[2]。基于此,深入了解新的作物品种对氮素吸收和再利用的效率,对于土壤中的氮肥施用量具有指导意义,从而保证作物能在有限的氮肥条件下产出最大量的食物,为粮食安全提供保障。研究表明,环境中的高浓度CO2抑制小麦对氮素的吸收[3],所以通过分子的手段,有针对性地设计环境适应型的作物新品种也是解决目前作物氮素利用效率不高的有效方式。
在高等植物中,谷氨酰胺合成酶 (Glutamine synthase,GS; EC6.3.1.2) 是氮素代谢途径中的关键酶之一,它联合谷氨酸合成酶 (Glutamine synthase,GOGAT) 催化谷氨酸转化为谷氨酰胺 (Glutamine synthase,Gln) (图1),参与光呼吸氨、还原氨 (初级氮)、循环氨的再同化等代谢过程,对植物的生长和发育至关重要[4]。本文就植物中谷氨酰胺合成酶的分类、遗传进化关系、与氮素的关系以及组织表达特异性、生物学功能以及分子调控机理进行了综述,以期通过对GS基因的调控来提高植物氮素利用效率。
到目前为止,在真核生物和原核生物中共鉴定出了3类GS形态,包括GSI、GSII和GSIII[5]。通过全基因组查找发现,不管单子叶植物还是双子叶植物仅存在GSI和GSII两种形态,且家族成员数不多[5]。基于亚细胞定位结果,谷氨酰胺合成酶被分为2类:胞液型GS1,分布于细胞质中;胞质型GS2,定位在叶绿体或质体中[6]。在植物中,GS2一般均只有1个成员,而不同植物的GS1则包含3到5个同工酶[5]。在双子叶植物拟南芥中,GS1由5个同源基因编码产生[7];而在禾本科作物中,一般含有3到5个GS1基因,如水稻有3个 (GS1;1,GS1;2和 GS1;3),玉米有5个 (Gln1-1、Gln1-2、Gln1-3、Gln1-4和Gln1-5),短柄草有3个 (Gln1-1、Gln1-2和Gln1-3),而小麦则包含3类 (GS1、GSr和GSe),每类又有2个同源基因[6]。
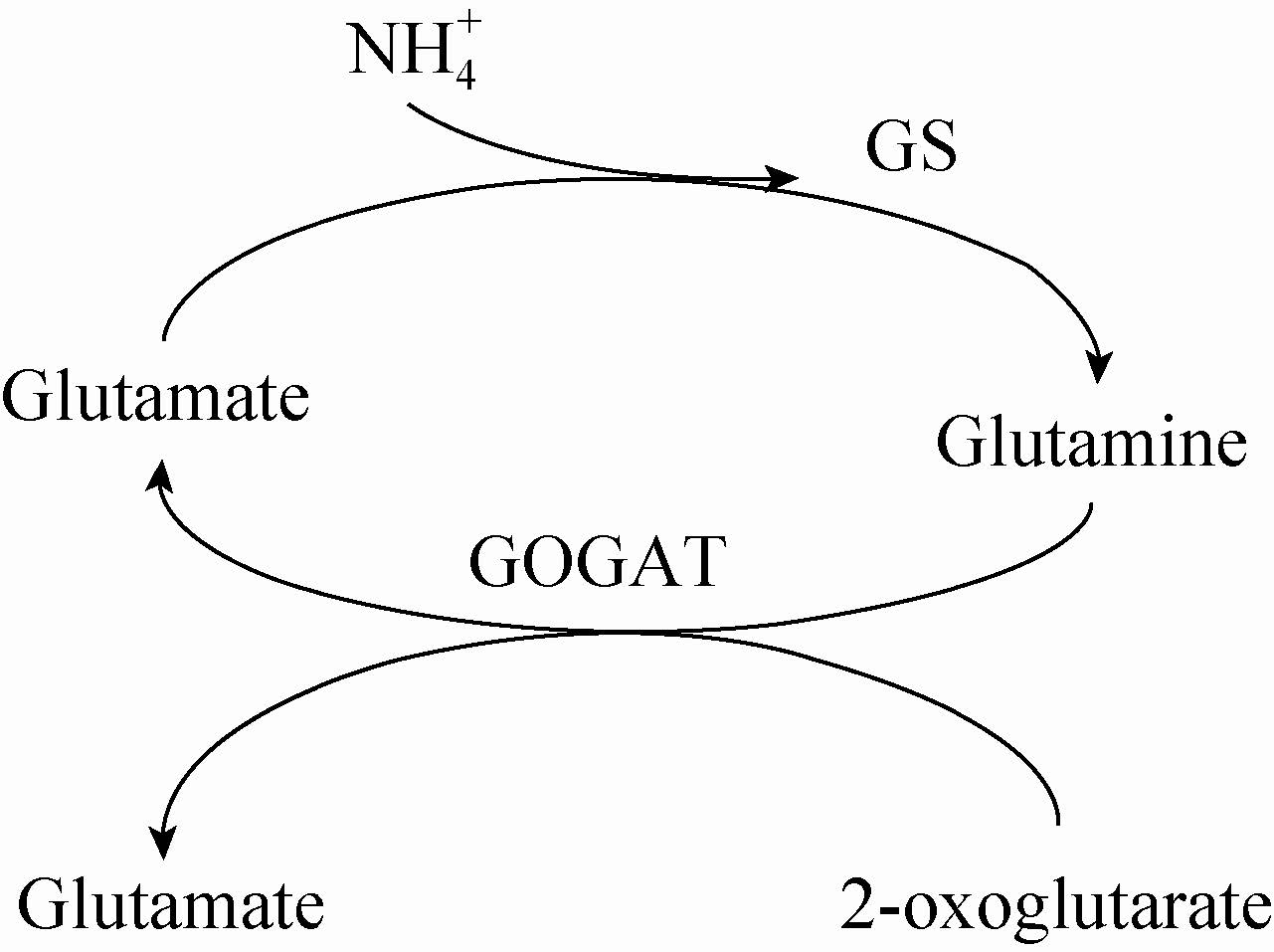
1.2 植物GS基因的遗传进化关系通过序列比对发现,高等植物的GS1基因的核苷酸的序列一致性在76.5%-82.3%之间,而氨基酸的序列一致性在78.7%-83.1%之间,平均同一性达88.1%-93%[5]。相对来说,拟南芥等双子叶植物和禾本科单子叶植物在基因序列上的变异程度要远大于禾本科之间。另外研究发现,小麦GS1的基因序列与GSr的序列一致性要高于与GSe的序列比对结果[7-8]。利用GS蛋白序列进行遗传进化分析,结果显示,GS2和GS1基因的出现发生在单子叶和双子叶分化之前[8]。禾本科包括众多的亚科,如果仅仅选取一两个亚科的作物进行分析,很难确定胞液型GS1基因的进化关系。Swarbreck等[5]选取禾本科中黍亚科Panicoideae、早熟禾亚科Pooideae和稻亚科3个亚科的14个物种,利用不同的进化分析方法对胞液型GS基因的同源进化关系进行了分析,结果显示,这些作物的胞液型GS基因依照在小麦上的分类方式大致可以归为GS1、GSr和GSe三个亚组,这与之前研究相似[9, 10]。有趣的是,进化分析结果显示GS1和GSe亚组的进化相对于GSr亚组亲缘关系更近一些,这与序列比对的结果有所不同。最近,Thomsen等[6]选取拟南芥和6种禾谷类 (短柄草、水稻、玉米、大麦、小黑麦和黑麦小麦杂交种),对其GS蛋白序列进行了遗传进化分析,结果显示拟南芥和禾谷类的GS蛋白聚在不同的进化分支上,印证了GS2和GS1基因的出现发生在单子叶和双子叶分化之前的结论[8],而禾谷类GS1蛋白可分为GS1-1、GS1-2、GS1-3三个亚组 (图2),这与之前研究报道相同[8, 9]。综上分析发现,对于同一亚组的GS1,在黍亚科 (玉米)、早熟禾亚科 (短柄草、小麦、小黑麦和黑麦小麦杂交种) 和稻亚科 (水稻) 中处于不同的进化树分支[6, 7, 8],说明胞液型GS1基因的进化发生在禾本科分化之前。
采用QTL的方法,在玉米、水稻、小麦、油菜等农作物中均定位到一些与氮素利用有关的QTL位点,其中部分位点与GS基因关联。在玉米中研究发现,玉米籽粒产量与GS酶活性呈正相关,位于玉米1、4和5号染色体的4个GS基因能够影响有关籽粒灌浆酶的活性[11]。通过对玉米施加高浓度氮肥和低浓度氮肥,数量遗传分析结果显示,ZmGln1-3基因与有关产量和千粒重的两个QTL位点重合[12],ZmGln1-4基因与1个千粒重QTL位点重合[13]。在水稻中,单个小穗产量的QTL区域含有OsGS1基因,这与玉米的研究结果相似,说明GS同工酶能够在穗部将氨态氮同化,进而影响籽粒数目及籽粒大小,从而直接与产量相关[14]。在小麦中,小麦旗叶GS酶活性QTL位点定位在TaGSr位点,并与1个籽粒氮浓度QTL重合[15],而另外1个小麦GS1基因 (TaGSe) 与籽粒蛋白含量QTL位点关联[16]。另外,在衰老的叶片中,1个小麦GS同工酶基因TaGS1.1和油菜的两个胞液型GS1基因 (BnaGLN1.1和BnaGLN1.4) 上调表 达[17, 18],说明GS1蛋白在叶片衰老过程中将NH4+同化,并将其从筛管伴细胞转移到叶肉细胞,从而在植物的生殖器官的氮素再同化过程中发挥重要的作用。
3 植物GS基因对氮素响应及其组织表达特异性性状变异的分子基础在于基因表达的变化。谷氨酰胺合成酶作为氮素同化途径中的关键酶,在不同氮素浓度处理前后GS基因的表达变化和植物体内氮素含量这一生理性状的关系成为研究的焦点。El Omari等[19]研究表明,对高粱施加NH4+会诱导两种不同GS1多肽 (等电点较小的a和较大的b) 的表达,然而施加硝酸盐却不能诱导a和b两个GS1多肽的富集。有关根系中GS蛋白异构体受NH4+特异诱导的研究在多种植物中均有报道,包括大豆[20]、玉 米[21, 22]、拟南芥[23]和水稻[24]。在大豆根系中,胞液型GS基因在外源施加NH4+或者共生固氮作用下表达水平发生改变[20]。水稻终生处于水生环境,它的根系中胞液型GS1的不同成员对氮素的响应并不一致[15]。Sakakibara等[21]通过施加NH4+和硝酸盐对玉米根系处理3 d后,RNA杂交结果显示,GS1-1和GS2受硝酸盐诱导,而家族其他成员对硝酸盐并未作出响应。另外,Sakakibara等[ 22]研究发现,在施加NH4+处理不同时间后,玉米根系中Gln1-1和Gln1-5两个基因的表达量在处理1 h后开始增加,到6 h的时候丰度达到最大值,随后逐渐降低,而Gln1-4和Gln1-3两个基因在处理后转录水平则不断降低。与之相反,拟南芥中4个GS1成员中只有1个对NH4+有响应[24]。综上可以看出,不同作物的GS基因确实对氮素有响应,但是不同成员对氮素的反应并不一致。
基因的时空表达分析可以为解析基因的生物学功能及其调控机理提供重要参考依据。在植物中,GS基因家族成员存在明显的组织表达特异性[7, 8]。一般在C3植物的叶片中,GS2表达水平较GS1高,它主要在叶绿体的合成以及叶片的衰老过程中发挥重要作用,但是比例会随着叶片衰老而逐渐降低[25]。拟南芥GS1基因也表现出明显的组织表达特异性,并与NH4+和谷氨酸的水平有密切的关系[7]。最近,Guan 等[26]将拟南芥Gln1;1和Gln1;2两个基因启动子融合GFP转化到拟南芥中显微观察发现,在根系伸长区能检测到Gln1;1启动子融合GFP蛋白的荧光信号,说明Gln1;1基因在初生根发育过程中促进氮素的吸收利用;而转Gln1;2启动子融合GFP蛋白的拟南芥中,在根系成熟区和次生根维管束基部检测到很强的荧光信号,另外,在地上部的叶肉细胞和维管束鞘细胞内也有表达,甚至在萼片、花瓣、雄蕊的维管组织以及果柄和果荚的节点部位均检测到荧光信号,说明Gln1;2基因在花的维管束组织和生殖器官的氨吸收过程中发挥重要作用,进而影响种子发育。在大麦中存在3个GS1基因 (HvGS1-1、HvGS1-2和HvGS1-3),其中HvGS1-1蛋白主要分布在不同组织的维管组织中;HvGs1-2蛋白主要分布在叶片的表皮细胞中,在根部的皮质和中柱鞘中也有少量表达;而HvGS1-3蛋白主要分布在麦穗中[27]。水稻也包含3个GS1基因,其中GS1;1在所有器官 (根系、叶片、叶鞘和花的小穗) 中均有表达[28];GS1;2在苗期根系中表达水平较高;而GS1;3则在小穗中特异表 达[28, 29]。在玉米中,GS1基因的5个成员也表现出明显的组织表达特异性,如Gln1-3和Gln1-4分别在叶肉细胞和叶片维管束鞘细胞中表达丰度较高,并决定穗粒数和子粒大小;Gln1-1和Gln1-2分别在根系和种皮等组织器官中表达水平较高;而Gln1-5在任何 组织器官中的表达水平均较低[30]。最近,我们以玉米Gln1-1基因启动子融合GUS报告基因转化拟南芥,对转基因后代不同发育时期的组织器官进行了GUS染色。结果显示,在苗期根系的成熟和伸长区维管束组织、幼嫩的花药、花丝末端及珠柄基部等部位检测到较强的GUS信号 (结果未发表)。
综上所述,GS基因对氮素有响应,且在生殖器官和营养器官均有表达,意味着GS基因在这些组织器官的生长发育过程中通过改变其氮素利用效率来发挥重要的作用。上述结果为进一步鉴定GS基因的生物学功能丰富了信息和资料。
4 植物GS基因的生物学功能谷氨酰胺合成酶是氮素代谢途径中的关键酶,在植物的生长和发育过程中发挥重要的作用[4]。在高等植物中,谷氨酰胺合成酶分为胞液型GS1和叶绿体型GS2两类,其中GS2基因一般只有1个成员,在叶片等能进行光合作用的组织器官中通过氨吸收途径在光呼吸和硝酸还原过程中发挥重要作用,同时在豆科根系中也有GS2基因表达,在氨吸收和硝酸还原过程中也具有重要作用[31-32]。烟草中超表达GS2基因能够提高叶片中自由氨基酸 (谷氨酸和谷氨酰胺) 的含量,表型分析发现,GS2基因超表达后能促进幼苗生长,转基因幼苗鲜重较野生型提高20%-30%[33]。
鉴于植物GS1成员数有3-5个,且表现出明显的组织表达特异性,所以针对GS1基因不同成员进行生物学功能研究,对于明晰植物的特定组织或器官的发育机制有重要意义。众多研究报道,超表达GS1基因后能够提高GS酶的活性,且在植物生物量和产量等重要农业性状上具有决定性作用[34, 35, 36]。目前,超表达转基因手段是研究基因生物学功能的主要方式。在转基因手段选择上,通常采用组成型启动子连接GS1基因的方法[37, 38],少量研究采用的是组织特异型的启动子,有针对性地在某些组织或器官中提高GS酶活性,如在根系特异表达的[36]和叶片中特异表达的[34],但是不管选取哪种启动子,不同植物在GS1同源基因超表达后表现并不完全一致。早期在松树上研究发现,将松树GS1a基因异位表达到杨树中,不管是温室培养还是田间种植,转基因株系的生长特性均有所提高和改善[39, 40]。Oliveira等[41]将大豆的GS1基因超表达到烟草中研究发现,转基因株系的干重和鲜重较对照显著提高,同时分析发现GS酶活力和生物量显著相关。将根系特异表达启动子 (rolD) 连接GS15基因转化大豆后,转基因植株的生物量和氮含量较非转基因分别提高了17%和54%,但是二者并不相关,表明GS1蛋白可能受翻译后调控[36]。在水稻中,超表达2个GS1同源基因 (Gln1;1和Gln1;2) 和一个大肠杆菌glnA基因,温室培养条件下,转基因植株体内可溶蛋白和氮含量均显著提高[42],但是不管采用温室培养还是田间种植的方式,与对照相比,超表达GS1基因的水稻植株在表型上未表现出任何差异[38]。这可能与氮肥的施用量和种植过程中的环境控制有关。
在玉米、小麦、水稻和拟南芥上研究表明,GS1基因对籽粒产量有重要贡献[6]。关于玉米GS1基因的生物学功能研究,目前主要集中在Gln1-3和Gln1-4两个基因上。Martin等[37]对Gln1-3和Gln1-4单突变体和双突变体及野生型进行了研究,发现Gln1-4突变体的粒重变小,Gln1-3突变体表现为穗粒数减少,而Gln1-3/Gln1-4的双突变体有累加效应,导致单株产量明显降低。另外,Gln1-3在玉米中超表达后,转基因株系的穗粒数增加了30%,这与突变体的研究结果相印证[37]。与野生型相比,Gln1-3和Gln1-3/Gln1-4突变体中具有更高的氨基酸含量和低C/N比,同时自由铵的浓度也更高,并且ZmAS3和ZmAS4两个天冬氨酸合成酶基因的转录水平也明显提高[43]。在小麦中,超表达菜豆Glna基因能够提高穗粒 数和粒重,但是利用同样的小麦自交系[34, 38],Thomsen等[6]却未能重复出这些实验结果,暗示基因生物学功能鉴定过程的复杂。最近,在拟南芥上,Guan等[26]利用gln1-1和gln1-2单突变体以及双突变体研究发现,gln1-2单突变体和gln1;1:gln1;2双突变体较野生型的单果荚籽粒数目减少了约17%,种子干重分别降低了10%和18%,最终导致产量分别减少了20%和35%,而gln1-1单突变体产量并未显著降低,不过结合双突变体的研究结果可以看出,gln1-2基因在拟南芥种子产量构成上发挥关键作用,而gln1-1和gln1-2两个基因具有一定的累加效应。
总之,到目前为止,不同植物上超表达GS1同源基因的植株在表型上表现并不一致,这说明GS1基因上游或下游的序列可能影响GS酶活性的提高,从而在一定程度上影响其生物学功能的发挥。基于此,深入研究GS基因的分子调控机制对利用其提高氮素利用效率则十分必要。
5 植物GS基因的分子调控机制植物的GS基因表现出明显的组织表达特异性,即使同一器官或组织的不同时期其表达水平也有所不同,意味着同一器官中GS基因的功能及代谢过程在不同组织、细胞和亚细胞中会发生变化[6]。研究发现,谷氨酰胺合成酶基因的表达受到基因、转录后以及蛋白等多个水平的调控[24]。
5.1 转录水平的调控基因序列分析发现,在谷氨酰胺合成酶基因的5′端启动子区含有光响应、生长素响应以及MYB (v-myb avian myeloblastosis viral oncogene homolog)、Dof (DNA binding with one finger) 等转录因子特异结合的元件[44]。最近,El-Kereamy等[45]研究发现,在转OsMYB55基因的水稻中氨基酸含量显著提高,原因在于包括OsGS1;2基因在内的一些氨基酸合成酶的表达水平发生了改变,这一结果说明OsMYB55基因能够调控OsGS1;2基因的表达。最近,我们利用生物信息学分析发现,在6个玉米GS基因的启动子区域均含有MYB结合位点,其中ZmGS1-2和ZmGS1-2两个基因含有5个MYB结合位点,ZmGS1-2和ZmGS1-5各含有3个,而ZmGS1-3和ZmGS2则分别含有2个和1个 (结果未发表)。这部分印证了OsMYB55基因能够调控OsGS1;2基因表达的研究结论[45],但是存在多个MYB结合位点也暗示其调控机理的复杂。有关松树Dof5的研究表明,Dof5转录因子可同时调控GS1a和GS1b的表达,它能促进GS1b的表达,而抑制GS1a的转录,从而调控松树GS1同工酶的空间分布[46]。最新研究发现,NLP7转录因子能够诱导GS2基因的表达[47]。另外,LBD (Lateral organ boundaries domain) 基因家族一些成员在氮素代谢过程中具有调控作用。Rubin等[48]研究发现,拟南芥3个LBD基因的过量表达转基因株系中一些氮素代谢基因的表达量降低3倍以上,其中就包括GS基因,且体内氮素及谷氨酸等氨基酸含量显著降低。
研究表明,NO3-作为信号分子参与诱导根系中激素代谢基因表达,这些基因在供NO3-的根系中的表达量远远高于不供NO3-的根系中,说明这些激素代谢基因对氮素有一定响应[49]。激素是植物生长发育的重要调控物质,其中生长素与根系的发育存在密切关系[50]。有关生长素和GS的关系仅在木本植物中有少数报道。如Man等[51]研究发现,将松树中的谷氨酰胺合成酶异源表达到杨树后,其转基因植株中自由IAA的含量显著增加。此外,在转录后水平上,谷氨酰胺合成酶基因的表达也会受到一定的调控,可能源于GS基因的3′非编码区影响转录本的稳定性[52, 53]。
5.2 翻译后调控在翻译后水平上,GS蛋白也会发生修饰,包括磷酸化、氧化和硝酸化等。在高等植物中,有研究表明GS可被磷酸化,且可与14-3-3蛋白互作,通过GS磷酸化水平的增加或降低,能够改变与14-3-3蛋白的反应程度[54, 55]。对苜蓿和烟草的GS2研究发现,GS2经过磷酸化修饰后等电点会有所变化[56]。El Omari等[19]发现高粱中的胞液型GS1也存在等电点的变化,预示着磷酸化修饰的可能。最近,利用在线软件NetPhos 2.0,我们对玉米GS的磷酸化位点进行了预测,结果发现,6个玉米GS均具有多个可能的磷酸化位点,其中以ZmGS2为最多,有38个,ZmGS1-5最少,有19个 (结果未发表)。这些结果说明玉米GS蛋白可能会发生磷酸化修饰,进而影响GS酶的活性,从而在玉米各组织器官的不同发育阶段发挥重要的作用 。
6 应用前景展望氮素是制约作物产量的主要营养元素之一。近几十年,全球范围内普遍存在氮素施用不合理的现象,一部分地区氮素匮乏,氮肥施加却不足,而另一些地区则过量施加氮肥,从而导致农田土壤酸化,加速了土壤营养元素流失[2]。所以,明确作物对氮素的利用效率,培育出氮素高效利用、环境适应型作物新品种是解决目前在作物氮素利用上存在问题的有效方法。
在高等植物中,谷氨酰胺合成酶是氮素代谢途径中的关键酶之一,对植物氮素吸收和利用有直接作用,影响植物的生长发育和产量[4]。到目前为止,通过转基因和基因沉默手段,在很多作物上已经开展了GS1基因生物学功能的研究,结果表明,GS1基因对于产量这一重要农艺性状有重要贡献[37, 38]。然而,对不同作物GS1同源基因的研究结果却并不一致,甚至存在矛盾的地方。基于此,若想通过GS1基因来改变作物的氮素利用效率,则需要综合考虑组织特异性或植物的特定发育时期。另外,氮素代谢过程涉及多个酶,所以为了平衡这些基因间的效应,让GS基因最终能够在氮素吸收和利用上发挥最大作用,共转化手段也是一种策略,如超表达GS基因时,同时超表达提供反应底物的谷氨酸合成酶 (Glutamine synthase,GOGAT) 基因和天冬酰氨合成酶基因。
谷氨酰胺合成酶基因的5′端启动子区含有光响应、生长素响应以及MYB、Dof等转录因子特异结合的元件,表明GS基因的表达会受到多个基因的调控。研究发现,一些基因可促进GS基因的表达,而另外一些起抑制作用或者对GS的活性造成影响[45, 46, 47, 48]。在烟草中超表达拟南芥的Dof1.7和Gln1;3两个基因后,与野生型相比,转基因植株氮素利用效率显著提高[57]。水稻和小麦分别超表达玉米ZmDof1和小麦TaDof1基因后,转基因植株的GS活性较野生型显著增加,从而提高了转基因植株的氮素利用效率。在松树中,Dof5转录因子能促进GS1b的表达,而抑制GS1a的转录[46]。在水稻中,OsMYB55基因能够调控OsGS1;2基因的表 达[45]。NLP7转录因子能够诱导GS2基因的表达[47]。此外,LBD基因家族一些成员在氮素代谢过程中具有调控作用[48]。综上所述,一些基因在碳氮代谢方面发挥重要作用,且对GS活性具有调控功能,所以将这些氮素代谢调控基因与GS基因共表达也是提高作物氮素利用效率的一种有效手段。
| [1] | Godfray HCJ, Beddington JR, Crute IR, et al. Food security: the challenge of feeding 9 billion people. Science, 2010, 327(5967): 812-818. |
| [2] | Guo J, Liu X, Zhang Y, et al. Significant acidification in major Chinese croplands. Science, 2010, 327(5968): 1008-1010. |
| [3] | Bloom AJ, Burger M, Asensio JSR, et al. Carbon dioxide enrichment inhibits nitrate assimilation in wheat and Arabidopsis. Science, 2010, 328(5980): 899-903. |
| [4] | Kusano M, Tabuchi M, Fukushima A, et al. Metabolomics data reveal a crucial role of cytosolic glutamine synthetase 1; 1 in coordinating metabolic balance in rice. Plant J, 2011, 66(3): 456-466. |
| [5] | Swarbreck SM, Defoin-Platel M, Hindle M, et al. New perspectives on glutamine synthetase in grasses. J Exp Bot, 2011, 62(4): 1511-1522. |
| [6] | Thomsen HC, Eriksson D, M?ller IS, et al. Cytosolic glutamine synthetase: a target for improvement of crop nitrogen use efficiency? Trends Plant Sci, 2014, 19(10): 656-662. |
| [7] | Li RJ, Hua W, Lu YT. Arabidopsis cytosolic glutamine synthetase AtGLN1,1 is a potential substrate of AtCRK3 involved in leaf senescence. Biochem Biophys Res Commun, 2006, 342(1): 119-126. |
| [8] | Bernard SM, M?ller ALB, Dionisio G, et al. Gene expression, cellular localisation and function of glutamine synthetase isozymes in wheat (Triticum aestivum L.). Plant Mol Biol, 2008, 67(1/2): 89-105. |
| [9] | Hirel B, Chardon F, Durand J. The contribution of molecular physiology to the improvement of nitrogen use efficiency in crops. J Crop Sci Biotech, 2007, 10(3): 129-136. |
| [10] | Bernard SM, Habash DZ. The importance of cytosolic glutamine synthetase in nitrogen assimilation and recycling. New Phytol, 2009, 182(3): 608-620. |
| [11] | Gallais A, Hirel B. An approach to the genetics of nitrogen use efficiency in maize. J Exp Bot, 2004, 55(396): 295-306. |
| [12] | Hirel B, Bertin P, Quilleré I, et al. Towards a better understanding of the genetic and physiological basis for nitrogen use efficiency in maize. Plant Physiol, 2001, 125(3): 1258-1270. |
| [13] | Masclaux C, Quilleré I, Gallais A, et al. The challenge of remobilisation in plant nitrogen economy. a survey of physio-agronomic and molecular approaches. Ann Appl Biol, 2001, 138(1): 69-81. |
| [14] | Obara M, Kajiura M, Fukuta Y, et al. Mapping of QTLs associated with cytosolic glutamine synthetase and NADH-glutamate synthase in rice (Oryza sativa L.). J Exp Bot, 2001, 52(359): 1209-1217. |
| [15] | Habash DZ, Bernard S, Schondelmaier J, et al. The genetics of nitrogen use in hexaploid wheat: N utilisation, development and yield. Theor Appl Genet, 2007, 114(3): 403-419. |
| [16] | Gadaleta A, Nigro D, Marcotuli I, et al. Isolation and characterisation of cytosolic glutamine synthetase (GSe) genes and association with grain protein content in durum wheat. Crop Pasture Sci, 2014, 65(1): 38-45. |
| [17] | Christiansen MW, Gregersen PL. Members of the barley NAC transcription factor gene family show differential co-regulation with senescence- associated genes during senescence of flag leaves. J Exp Bot, 2014, 65(14): 4009–4022. |
| [18] | Orsel M, Moison M, Clouet V, et al. Sixteen cytosolic glutamine synthetase genes identified in the Brassica napus L. genome are differentially regulated depending on nitrogen regimes and leaf senescence. J Exp Bot, 2014, 65(14): 3927-3947. |
| [19] | El Omari R, Rueda-López M, Avila C, et al. Ammonium tolerance and the regulation of two cytosolic glutamine synthetases in the roots of sorghum. Funct Plant Biol, 2010, 37(1): 55-63. |
| [20] | Hirel B, Bouet C, King B, et al. Glutamine synthetase genes are regulated by ammonia provided externally or by symbiotic nitrogen fixation. EMBO J, 1987, 6(5): 1167-1171. |
| [21] | Sakakibara H, Kawabata S, Hase T, et al. Differential effects of nitrate and light on the expression of glutamine synthetases and ferredoxin-dependent glutamate synthase in maize. Plant Cell Physiol, 1992, 33(8): 1193-1198. |
| [22] | Sakakibara H, Shimizu H, Hase T, et al. Molecular identification and characterization of cytosolic isoforms of glutamine synthetase in maize roots. J Biol Chem, 1996, 271(47): 29561-29568. |
| [23] | Ishiyama K, Inoue E, Tabuchi M, et al. Biochemical background and compartmentalized functions of cytosolic glutamine synthetase for active ammonium assimilation in rice roots. Plant Cell Physiol, 2004, 45(11): 1640-1647. |
| [24] | Ishiyama K, Inoue E, Watanabe-Takahashi A, et al. Kinetic properties and ammonium-dependent regulation of cytosolic isoenzymes of glutamine synthetase in Arabidopsis. J Biol Chem, 2004, 279(16): 16598-16605. |
| [25] | Lothier J, Gaufichon L, Sormani R, et al. The cytosolic glutamine synthetase GLN1;2 plays a role in the control of plant growth and ammonium homeostasis in Arabidopsis rosettes when nitrate supply is not limiting. J Exp Bot, 2011, 62(4): 1375-1390. |
| [26] | Guan M, M?ller I, Schjoerring J. Two cytosolic glutamine synthetase isoforms play specific roles for seed germination and seed yield structure in Arabidopsis. J Exp Bot, 2015, 66(1): 203-212. |
| [27] | Goodall AJ, Kumar P, Tobin AK. Identification and expression analyses of cytosolic glutamine synthetase genes in barley (Hordeum vulgare L.). Plant Cell Physiol, 2013, 54(4): 492-505. |
| [28] | Tabuchi M, Sugiyama K, Ishiyama K, et al. Severe reduction in growth rate and grain filling of rice mutants lacking OsGS1, 1, a cytosolic glutamine synthetase1; 1. Plant J, 2005, 42(5): 641-651. |
| [29] | Tabuchi M, Abiko T, Yamaya T. Assimilation of ammonium ions and reutilization of nitrogen in rice (Oryza sativa L.). J Exp Bot, 2007, 58(9): 2319-2327. |
| [30] | Li MG, Villemur R, Hussey PJ, et al. Differential expression of six glutamine synthetase genes in Zea mays. Plant Mol Biol, 1993, 23(2): 401-407. |
| [31] | Woodall J, Forde B. Glutamine synthetase polypeptides in the roots of 55 legume species in relation to their climatic origin and the partitioning of nitrate assimilation. Plant Cell Environ, 1996, 19(7): 848-858. |
| [32] | Zozaya-Hinchliffe M, Potenza C, Ortega JL, et al. Nitrogen and metabolic regulation of the expression of plastidic glutamine synthetase in alfalfa (Medicago sativa). Plant Sci, 2005, 168(4): 1041-1052. |
| [33] | Migge A, Carrayol E, Hirel B, et al. Leaf-specific overexpression of plastidic glutamine synthetase stimulates the growth of transgenic tobacco seedlings. Planta, 2000, 210(2): 252-260. |
| [34] | Habash D, Massiah A, Rong H, et al. The role of cytosolic glutamine synthetase in wheat. Ann Appl Biol, 2001, 138(1): 83-89. |
| [35] | Fuentes SI, Allen DJ, Ortiz-Lopez A, et al. Over-expression of cytosolic glutamine synthetase increases photosynthesis and growth at low nitrogen concentrations. J Exp Bot, 2001, 52(358): 1071-1081. |
| [36] | Fei H, Chaillou S, Hirel B, et al. Effects of theoverexpression of a soybean cytosolic glutamine synthetase gene (GS15) linked to organ-specific promoters on growth and nitrogen accumulation of pea plants supplied with ammonium. Plant Physiol Biochem, 2006, 44(10): 543-550. |
| [37] | Martin A, Lee J, Kichey T, et al. Two cytosolic glutamine synthetase isoforms of maize are specifically involved in the control of grain production. Plant Cell, 2006, 18(11): 3252-3274. |
| [38] | Brauer EK, Rochon A, Bi YM, et al. Reappraisal of nitrogen use efficiency in rice overexpressing glutamine synthetase1. Physiol Plant, 2011, 141(4): 361-372. |
| [39] | Fu J, Sampalo R, Gallardo F, et al. Assembly of a cytosolic pine glutamine synthetase holoenzyme in leaves of transgenic poplar leads to enhanced vegetative growth in young plants. Plant Cell Environ, 2003, 26(3): 411-418. |
| [40] | Gallardo F, Fu J, Cantón FR, et al. Expression of a conifer glutamine synthetase gene in transgenic poplar. Planta, 1999, 210(1): 19-26. |
| [41] | Oliveira IC, Brears T, Knight TJ, et al. Overexpression of cytosolic glutamine synthetase, relation to nitrogen, light, and photorespiration. Plant Physiol, 2002, 129(3): 1170-1180. |
| [42] | Cai H, Zhou Y, Xiao J, et al. Overexpressed glutamine synthetase gene modifies nitrogen metabolism and abiotic stress responses in rice. Plant Cell Rep, 2009, 28(3): 527-537. |
| [43] | Ca?as RA, Quilleré I, Lea PJ, et al. Analysis of amino acid metabolism in the ear of maize mutants deficient in two cytosolic glutamine synthetase isoenzymes highlights the importance of asparagine for nitrogen translocation within sink organs. Plant Biotechnol J, 2010, 8(9): 966-978. |
| [44] | Castro-Rodríguez V, García-Gutiérrez A, Canales J, et al. The glutamine synthetase gene family in Populus. BMC Plant Biol, 2011, 11(1): 119-134. |
| [45] | El-Kereamy A, Bi YM, Ranathunge K, et al. The rice R2R3-MYB transcription factor OsMYB55 is involved in the tolerance to high temperature and modulates amino acid metabolism. PLoS ONE, 2012, 7(12): e52030. |
| [46] | Rueda-López M, Crespillo R, Cánovas FM, et al. Differential regulation of two glutamine synthetase genes by a single Dof transcription factor. Plant J, 2008, 56(1): 73-85. |
| [47] | Marchive C, Roudier F, Castaings L, et al. Nuclear retention of the transcription factor NLP7 orchestrates the early response to nitrate in plants. Nat Commun, 2013, 4(4): 1713-1713 . |
| [48] | Rubin G, Tohge T, Matsuda F, et al. Members of the LBD family of transcription factors repress anthocyanin synthesis and affect additional nitrogen responses in Arabidopsis. Plant Cell, 2009, 21(11): 3567-3584. |
| [49] | Wang R, Xing X, Wang Y, et al. A genetic screen for nitrate regulatory mutants captures the nitrate transporter gene NRT1.1. Plant Physiol, 2009, 151(1): 472-478. |
| [50] | Jones B, Ljung K. Subterranean space exploration: the development of root system architecture. Curr Opin Plant Biol, 2012, 15(1): 97-102. |
| [51] | Man H, Pollmann S, Weiler EW, et al. Increased glutamine in leaves of poplar transgenic with pine GS1a caused greater anthranilate synthetase α-subunit (ASA1) transcript and protein abundances: an auxin-related mechanism for enhanced growth in GS transgenics?. J Exp Bot, 2011, 62(13): 4423-4431. |
| [52] | Ortega JL, Moguel-Esponda S, Potenza C, et al. The 3′ untranslated region of a soybean cytosolic glutamine synthetase (GS1) affects transcript stability and protein accumulation in transgenic alfalfa. Plant J, 2006, 45(5): 832-846. |
| [53] | Simon B, Sengupta-Gopalan C. The 3′ untranslated region of the two cytosolic glutamine synthetase (GS1) genes in alfalfa (Medicago sativa) regulates transcript stability in response to glutamine. Planta, 2010, 232(5): 1151-1162. |
| [54] | Finnemann J, Schjoerring JK. Post-translational regulation of cytosolic glutamine synthetase by reversible phosphorylation and 14-3-3 protein interaction. Plant J, 2000, 24(2): 171-181. |
| [55] | Lima L, Seabra A, Melo P, et al. Post-translational regulation of cytosolic glutamine synthetase of Medicago truncatula. J Exp Bot, 2006, 57(11): 2751-2761. |
| [56] | Riedel J, Tischner R, Mack G. The chloroplastic glutamine synthetase (GS-2) of tobacco is phosphorylated and associated with 14-3-3 proteins inside the chloroplast. Planta, 2001, 213(3): 396-401. |
| [57] | Wang Y, Fu B, Pan L, et al. Overexpression of Arabidopsis Dof1, GS1 and GS2 enhanced nitrogen assimilation in transgenic tobacco grown under low-nitrogen conditions. Plant Mol Biol Rep, 2013, 31(4): 886-900. |
 2015, Vol. 31
2015, Vol. 31