服务
文章信息
- 张白雪, 孙其信, 李海峰
- Zhang Baixue, Sun Qixin, Li Haifeng
- 基因修饰技术研究进展
- Advances in genetic modification technologies
- 生物工程学报, 2015, 31(8): 1162-1174
- Chin J Biotech, 2015, 31(8): 1162-1174
- 10.13345/j.cjb.140479
-
文章历史
- Received: October 10, 2014
- Accepted: November 26, 2014
2. 新疆农业职业技术学院,新疆 昌吉 831100
2. Xinjiang Agricultural Vocational Technical College, Changji 831100, Xinjiang, China
随着越来越多不同物种完成基因组测序,探索基因组的功能尤为重要。近年来,基因组靶向修饰技术逐渐成为基因组改造与基因功能研究的一个重要手段,并且于2012年被Science评为年度十大重要科学进展之一。人工核酸内切酶 (Engineered endonuclease,EEN) 是完成基因组定向修饰的重要工具[1]。自上世纪80年代末,基于人工核酸内切酶的基因修饰技术开始发展,目前主要包括:第一代人工核酸内切酶锌指核酸酶 (ZFN) 技术、第二代人工核酸内切酶转录激活子样效应物核酸酶 (TALEN) 技术、第三代人工核酸内切酶CRISPR-Cas核酸酶 (CRISPR-Cas RGNs) 技术。人工核酸内切酶能够特异识别目的DNA序列,对靶标单链或双链进行精准切割,形成双链断裂 (DSB),诱发细胞内源性的修复机制,激活体内非同源末端连接修复 (NHEJ) 或同源重组修复 (HR),在该位点实现敲除、插入、碱基替换、点突变等定点修饰[2, 3]。
T-DNA插入、转座因子、化学诱变及辐射诱变等传统技术早已被广泛用于产生突变[4, 5, 6, 7, 8],但是这些技术除了不能特异地针对某个目的基因外,同时会引起多个基因突变,需要进一步用互补方法验证表型和目的基因的连锁关系;反向遗传学方法如RNAi 技术可以对基因定点敲除[9],但无法彻底敲除基因的表达且无法在后代中恢复。与传统转基因技术相比,基因修饰技术具有针对性强、效率高、构建时间短、应用广泛等特点,显示了极大的基因编辑优势。
近年来,基因修饰技术发展迅速,不断得到改进,在细菌[10, 11]、酵母Saccharomyces cerevisiae[12, 13]、人类细胞[13, 14, 15, 16, 17, 18, 19, 20]、果蝇Drosophila melanogaster[16, 21, 22]、斑马鱼Danio rerio[16, 23, 24, 25, 26, 27, 28, 29, 30, 31]、爪蟾Xenopus laevis[16, 32]、小鼠Mus musculus[20, 33, 34]、大鼠Rattus norvegicus[35, 36]、猪Sus scrofa、牛Bos taurus[37]、拟南芥Arabidopsis thaliana[12, 38, 39, 40, 41, 42, 43, 44]、水稻Oryza sativa[42, 44, 45, 46, 47]、烟草Nicotiana tabacum[42, 43, 48, 49]、玉米Zea mays[50]、高粱Sorghum bicolor[42]、小麦Triticum aestivum[47]、大麦Hordeum vulgare[51]等多种生物体中得到了广泛的研究和应用。本文对基因修饰技术原理及取得的研究进展作一总结和概述。
1 锌指核酸酶 (ZFN) 技术1983年,锌指蛋白 (Zinc finger protein,ZFP) 首次在非洲爪蟾的转录因子IIIA中被发现[52]。第一种经过改造的人工核酸内切酶——锌指核酸酶(Zinc finger nueleases,ZFN) 在20世纪90年代末首次得到研究和应用[53],实现了基因的高效定点修饰,具有划时代的意义。
1.1 ZFN的结构和作用机制锌指核酸酶 (ZFN) 由锌指蛋白 (ZFP) 结构域和切割结构域 (FokⅠ) 两部分构成[54]。锌指 (Zinc finger,ZF) 是构成ZFP结构域的DNA结合基序 (Motif),含有30个左右的氨基酸残 基[55]。锌指折叠形成 α-β-β (C端—N端) 类型的二级结构,α螺旋能够插入到DNA双螺旋结构的大沟中。α螺旋的-1- + 6位的7个氨基酸残基 (+ 4位通常固定为亮氨酸残基) 决定靶标序列的特异性识别,通过特异识别DNA双螺旋中某一条单链上连续的3个核苷酸 (称为一个三联子,Triplet),完成靶标 (图1A)。
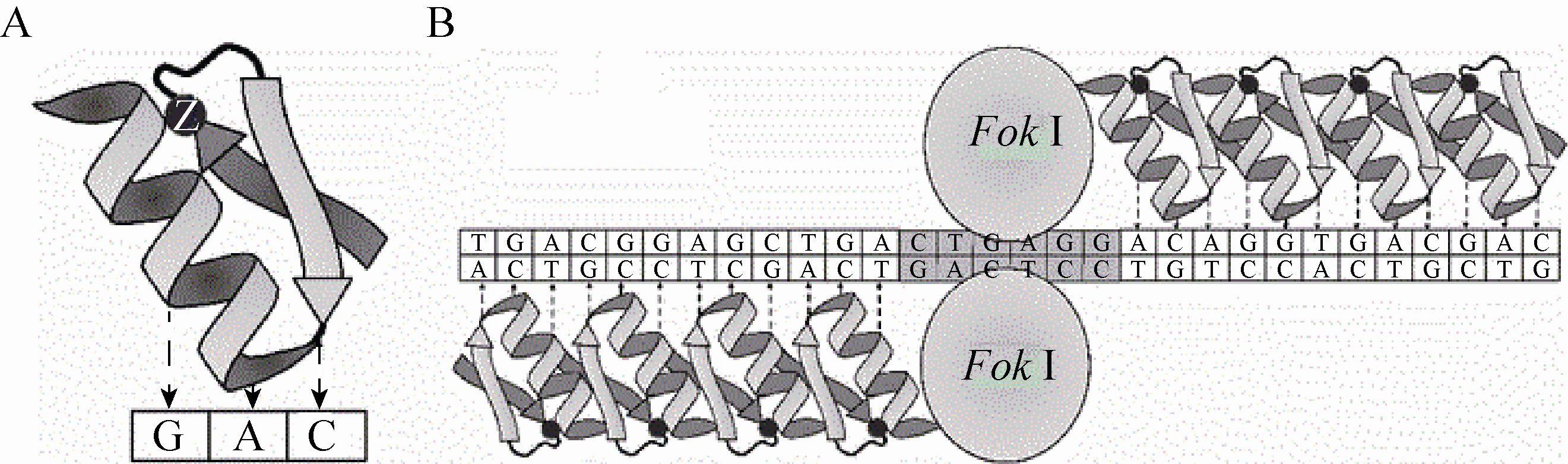
|
| 图1 锌指蛋白核酸酶定点突变示意图[57] Fig.1 The diagram of targeted mutagenesis via engineered ZFNs[57]. (A) Structure of the zinc-finger molecule which consists of one α-helix and two β-sheets and binds to 3 bp recognized by residues in theα-helix. (B) The diagram of targeted mutagenesis via ZFNs. Two ZFNs bind to the targeted region in tail to tail direction and two FokⅠ nuclease domains form dimmer to create double strand break. |
多个锌指结构串联可以识别更长的DNA目标片段,同时保证能够精确地特异识别靶标序列。因此,通常把3-6个C2H2类型的锌指结构域重复串联构成ZFP结构域,它们在DNA上沿3′到5′的方向依次同其靶序列结合。FokⅠ核酸酶主要来源于海床黄杆菌Flavobacterium okeanokoites,能够通过二聚化发挥核酸内切酶活性[56],同ZFP的C端融合构成ZFN单体。靶标DNA链分别由两个ZFN单体的锌指结构域识别其5′到3′方向和3′到5′方向,两个FokⅠ切割结构域形成二聚体的活性形式发挥剪切作用 (图1B)。因此,构建识别不同碱基的ZFP只需要改变这些氨基酸的组成即可。
将ZFN的质粒或mRNA通过转染或注射细胞后,核定位信号引导ZFN进入细胞核,两个ZFN分子的FokⅠ结构域与目标位点结合,于两个结合位点的间隔区切割产生DSB切口。细胞可通过NHEJ或HR等方式可能出现错误修复或者引入改变,造成DNA序列改变,从而实现基因的定向修饰操作。
ZFN的构建包括:选择靶位点、构建ZFP结构域、选择FokⅠ切割域、优化筛选等步骤。由于三联子靶序列中的3个碱基同锌指结构的-1、+3、+6位氨基酸残基能够相互作用,而ZFP 的结合特性和效率同样也会受到其他氨基酸残基及相邻串联的锌指结构氨基酸序列的影响,这种上下文依赖效应 (Context dependent effects)有可能对ZFP与靶位点的结合产生增强或减弱作用。因此,ZFN应用的关键技术是构建筛选出能够高效、特异结合靶标位点的ZFP结构。
除了根据已知的ZFP结合位点直接构建ZFN外,Joung等成立的“锌指联合会”提供在线软件ZiFiT (www.zincfinger.org) 用于开发易于操作的,标准化的ZFN构建平台,主要包括:模块组装 (Modular assembly,MA),OPEN (Oligomerized pool engineering) 和CoDA (Context dependent assembly) 方法。除此之外,还有利用ZFN技术分析目标基因的ZFN靶位点免费的在线ZFN设计软件ZiFiT:http://bindr.gdcb.iastate.edu/ZiFiT/;Zinc Finger Tools:http://www. zincfingertools.org[58]。
1.2 ZFN技术的研究进展在医学研究方面,Urnov等在人的体细胞基因组上利用ZFN技术对人内源基因IL2Rγ完成定点切割,同源重组效率达20%[14]。Perez等对CD4+ T细胞CCR5基因实现定点敲除,获得HIV抗性基因型细胞以抑制艾滋病毒的繁殖和传播,达到了基因治疗的目的[15]。在动物基因组研究方面,Bibikova等利用ZFN技术对果蝇X性染色体的yellow基因完成定点修饰,结果发现50%的雄性果蝇发生颜色的改变[21]。研究者通过向斑马鱼单细胞胚胎中注人不同目的基因设计的ZFN mRNA,特定基因位点实现突变并表现出预期的性状。实验中突变存在一定的脱靶现象,但是实验结果表明通过构建ZFN直接注射mRNA到动物胚胎,能够完成基因的定点修饰[23, 24, 25]。在植物体的研究中,研究者通过把ZFN基因及其靶位点转入拟南芥、玉米,获得了定点突变的突变体植株,并且证明了由ZFN介导的基因突变可以遗传[38, 50]。
ZFN技术优势明显,可以应用到很多种生物中定点修饰基因。然而,在ZF模块设计中,由于上下文依赖效应使得ZFNs特异识别任意靶标序列的能力较差[59],导致一些ZF结构域缺乏特异性,结果出现脱靶现象,引起其他目的基因突变和染色体畸变[60, 61]。此外,ZFN的设计筛选耗时费力,成本高,因此限制了其更加广泛的应用。
2 转录激活子样效应物核酸酶 (TALEN) 技术2007年,植物黄单胞菌Xanthomonas Campestris分泌的转录激活子样(Transcription activator-like effectors,TALEs) 被发现[62, 63],同理构建的转录激活因子样效应物核酸酶 (Transcription activator-like effector nucleases,TALENs) 脱靶几率小,细胞毒性小,成为另一种能够高效简便地靶向基因组的新技术[64, 65]。
2.1 TALEN的结构和作用机制TALE由N端转运信号、中部DNA特异识别结合域、C端核定位信号和转录激活结构域3部分构成。其中,DNA特异识别结合域由一串连续排列、序列高度同源的重复单元组成,能够识别特异的DNA序列。每个重复单元通常由33−35个氨基酸组成,第12、13位的氨基酸高度可变,被称为重复序列可变的双氨基酸残基 (Repeat variable diresidues,RVD),并且能够与A、T、C、G碱基相互对应,即:NI特异识别A,HD特异识别C,NG特异识别T,NH特异识别G,NN对应G或A,NS可以识别A、T、G、C中的任一种[66, 67]。此外,除了第12、13位氨基酸外,其余氨基酸序列高度保守。每个TALEN识别的靶标序列长度通常为14-20 bp[68]。因此,识别某一特定氨基酸序列,只需设计相应TALE单元串联克隆即可,其中RVD是TALEs特异识别DNA序列的关键。不同于存在上下文依赖效应的ZFs,TALE构成的重复氨基酸序列模块具有蛋白与DNA识别码和模块自然属性,可以和任何单碱基发生特异性结合,因此理论上可以设计并识别任意靶DNA的序 列[69]。FokⅠ结构域切割位点位于两个靶标序列的间隔区,间隔距离一般在15-30 bp之间[70] (图2A)。

|
| 图2 利用TALEN诱变目的基因[46] Fig.2 Targeted gene mutagenesis induced by engineered TALENs[46]. (A) Structure of a TALEN binding to target gene (OsBRII). The colored boxes denote the TAL effector repeats. Each color represents a different RVD. (B) TALEN-induced DSBs can be repaired by either NHEJ or HR. As a result,mutagenesissuch as deletion,replacement and insertion are occurred. |
将设计的特异识别靶标序列的TALEs与FokⅠ结构域融合,即构建得到一对TALEN质粒。将TALEN质粒对共转化到细胞中,表达的融合蛋白将分别和靶位点结合[71],再由二聚体化的FokⅠ对其进行切割,形成DSB,诱发DNA损伤修复机制,细胞则激发体内产生修复机制。若细胞内不存在修复模板,可激发NHEJ方式修复DNA,可能发生敲除或者插入碱基等错配修复,获得基因敲除突变体;如果在DSB周围存在同源DNA模板,细胞则采用HR方式进行修复,可能会发生碱基增加或者替换等 (图2B )。研究表明,NHEJ方式是高等植物进行自发修复的主要方式[72]。
TALEN的构建主要依靠分子克隆的手段,目前已形成了几种快速有效的方法:Golden gate分子克隆法、连续克隆组装法、高通量法、长黏末端的LIC (Ligation-indepent cloning) 组装法等。其中,连续克隆组装法包括:限制性酶切-连接法 (Restriction enzyme and ligation,REAL)、单元组装法 (Unit assembly,UA) 和idTALE一步酶切次序连接法;高通量法包括FLASH和ICA (Iterative capped assembly)[73]。为优化设计,一些实验室构建了预测设计TALEN靶点的服务器可供使用,如TALENT (https://tale-nt.cac.cornell. edu)、ZiFiT (http://zifit.partners.org/ZiFiT) 和idTALE (http://idtate.kaust.edu.sa) 等。
2.2 TALEN技术的研究进展在动物方面,Sakuma等使用该项技术对HEK293T细胞、人类诱导性多功能干细胞 (iPS)、果蝇、斑马鱼以及非洲爪蟾等模式动物中完成了基因突变[16]。Liu等利用TALEN技术在果蝇中对yellow基因成功打靶,并得到了可以遗传突变的种系[22]。Sander等利用TALEN技术在斑马鱼体细胞中成功对hey2和gria3a两个基因成功打靶,突变效率为11%-33%[26];Huang等利用UA法构建TALEN在斑马鱼中分别得到了tnikb和dip2a两个基因的突变,结果证明斑马鱼突变体可以稳定遗传[27]。Moore等利用TALEN技术在斑马鱼中进行研究,结果表明TALEN产生突变的效率高于ZFN[28]。Zu等使用该技术对斑马鱼的7个内源基因成功打靶,其中大部分基因的突变效率大于30%,有3个基因的突变效率接近100%[29]。Lei等通过构建TALEN转入两种不同的爪蟾中进行研究,结果表明TALEN技术都能完成基因定向敲除[32]。Sung等在小鼠中利用该技术对特定基因成功突变[33]。Tong等在大鼠细胞中通过构建TALEN成功敲除了BMPR2基因[35]。Carlson等利用TALEN技术对牛和猪的基因成功定点打靶[37]。
在植物中,Christian等首次运用TALEN在拟南芥中实验,组装若干对针对AtADH1基因的TALENs,通过使用LacZ作为报告基因的酵母系统验证,结果表明构建的TALENs能够发挥功能[12]。Cermak等构建了一种有自定义重复单元构成的能够高效组装TALEN的方法——Golden gate分子克隆的方法,以AtADH1作为靶标,用拟南芥的原生质体进行验证[39]。Mahfouz等利用TALE-SRDX抑制拟南芥基因的表达[40]。Christian等在拟南芥中研究发现TALEN技术产生的突变体在下代的遗传效率在1.5%-12%之间[41]。Li等首次运用TALENs技术对水稻OsSWEET14基因的启动子区域进行插入修饰,获得了抗白叶枯病的水稻株系[45]。Chen等利用TALEN技术定向敲除了水稻的52个基因,并且建立了一个大规模的TALEN突变体平台。研究者通过运用Golden Gate方法进行TALEN组装、构建植物表达载体、在水稻原生质体中进行TALEN活性验证、TALEN转化水稻、筛选基因敲出,最终得到转基因植株,突变率达30%,并发现突变片段常包含小的删除和插入,删除长度为1-20 bp,通常发生在TALEN结构域的间隔区[46]。Zhang等在烟草原生质体中利用TALEN技术成功进行基因置换[48]。Wendt等运用TALEN技术以大麦磷酸酶基因HvPAPhy的启动子区域一组控制元件作为靶标[51]。
3 CRISPR-Cas核酸酶 (CRISPR-Cas RGNs) 技术1987年,Ishino等首次在大肠杆菌Escherichia coli K12的碱性磷酸酶基因附近区域发现了成簇的规律间隔的短回文重复序 列[82]。随着研究的不断深入,2002年科学家将其正式命名为CRISPR (Clustered regularly interspaced short palindromic repeat)。2013年,人工改造的另外一种全新的人工核酸酶CRISPR-Cas系统迅速在更多的动植物物种中得到广泛应用。基因编辑操作由于Ⅱ型CRISPR-Cas9系统的出现变得更加简便、高效,可同时沉默任意数量的基因,成本变得更低。
3.1 CRISPR-Cas的结构和作用机制CRISPR是成簇的规律性间隔的短回文重复序列[75],其序列由一个前导区 (Leader)、多个短而高度保守的重复序列区 (Repeat) 和多个间隔区 (Spacer) 组成[76]。前导区长度一般为300-500 bp,富含A、T碱基,启动CRISPR序列转录[77]。 重复序列通常是包含回文序列长度为21-48 bp的区域,可以形成发卡结构[78]。间隔序列将各个重复序列隔开[79, 80],长度一般是26-72 bp,它与一些质粒或噬菌体的序列存在同源性[81, 82],使得宿主细胞能够抵抗外源基因的入侵。
Cas (CRISPR associated) 位于CRISPR位点附近的保守区域,通常与CRISPR结构的重复序列相连。Cas基因编码的蛋白具有核酸相关的结构域,Cas蛋白能够在向导RNA指导下通过位点特异性对靶位点进行特异性切割,例如目前研究较为深入的Cas9。Cas9是一种多结构域蛋白,由1 409个氨基酸组成,包含2个核酸酶结构域:氨基末端的RuvC-1ike结构域以及位于蛋白中间位置的HNH核酸酶结构域。
在CRISPR-Cas9系统中,长度约20 bp的外源DNA可以作为短的回文重复序列整合在CRISPR基因组中,转录加工成CRISPR RNA (crRNA)。这些crRNA会与trans-activating crRNA (tracrRNA) 形成一种双链二级结构[83]。在这两种RNA的引导下,Cas9蛋白的HNH能够特异性识别与crRNA互补配对的模板链并实现切割,切割位点位于原型间隔序列毗邻基序 (Protospacer adjacent motif,PAM) 上游3 nt处;RuvC-1ike参与另一条链特定位点的切割,切割位点位于PAM上游3-8 nt处 (即NGG位点)[84]。因此,可设计不同的crRNA使得 CRISPR-Cas9能够剪切不同的DNA序列。Jinek等研究发现,这两种RNA可以被“改装”成一个向导RNA (single-guide RNA,sgRNA)[85]。因此,设计不同的sgRNA足以指导Cas9内切酶完成对DNA的定点切割,形成DSB (图3)。
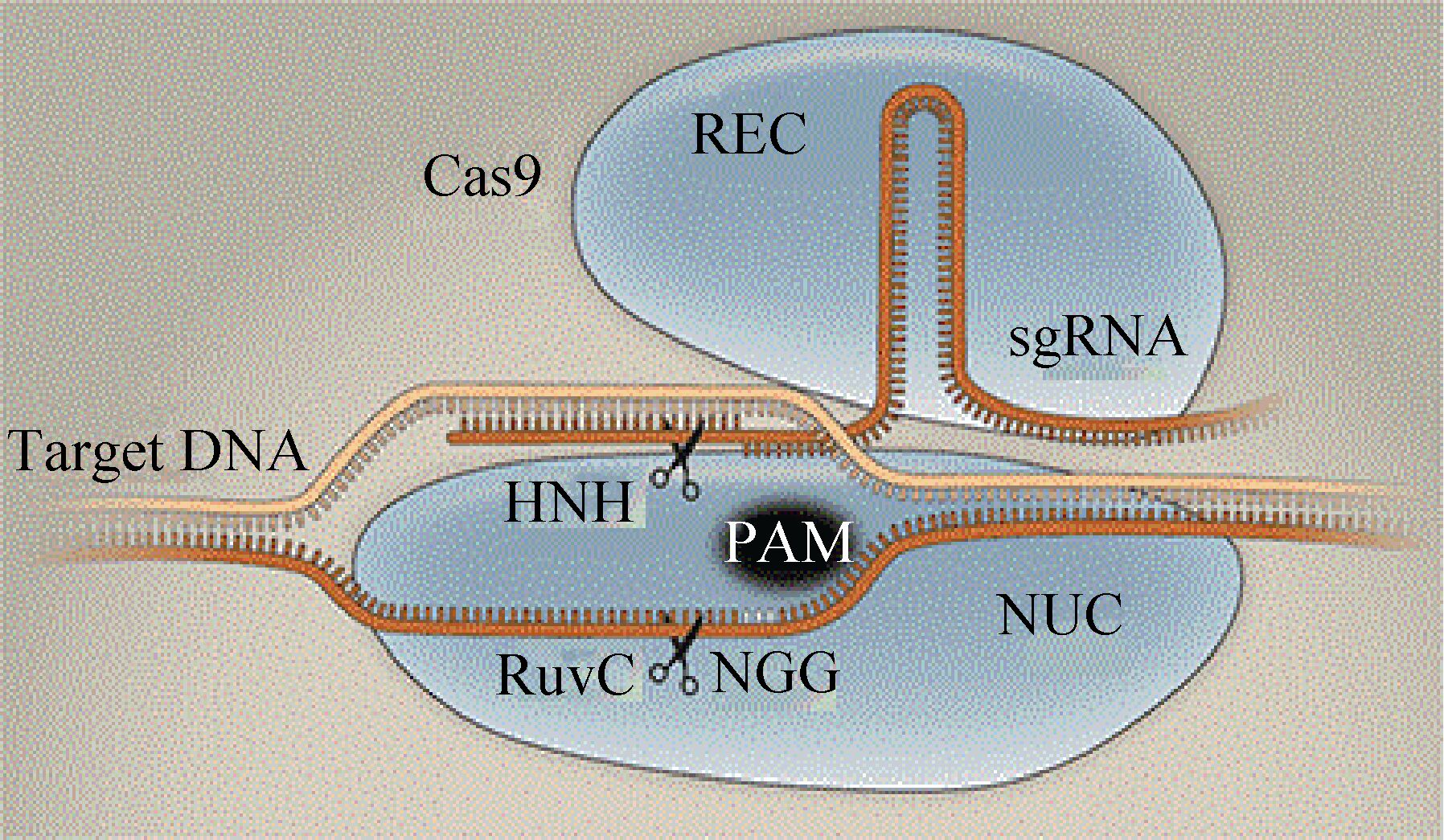
|
| 图3 CRISPR-Cas9定点诱变示意图[86] Fig.3 The diagram of targeted mutagenesis induced by engineered CRISPR-Cas9[86]. The Cas9 recognition (REC) domain interacts with the sgRNA,while the nuclease (NUC) lobe drives interaction with the PAM and target DNA. Two nuclease domains (RuvC,HNH) each nick one DNA strand,generating a double-strand break. |
在细菌方面,Jiang等利用CRISPR-Cas系统成功对细菌基因组进行了定向改造,获得了肺炎链球菌Diplococcus pneumoniae以及大肠杆菌的突变体,突变效率分别达到100%和65%[10]。在动物方面,Hwang等利用构建的sgRNA靶向的CRISPR/Cas9系统对斑马鱼胚胎fh1、apoea等基因实现定点打靶 ,突变率与使用TALENs技术在该位点引起突变的效率相近。另外,利用sgRNA靶向的CRISPR-Cas9系统成功获得了drd3、gsk3b基因的突变体[30]。Chang等在斑马鱼中分别完成了etsrp、gata4和gata5三个基因的靶向突变,结果表明突变效率约为35%[31]。Wang等利用CRISPR-Cas9系统已经获得了打靶的小鼠[34]。Li等利用CRISPR-Cas技术同时敲除大鼠Tet1、Tet2、Tet3基因,获得了双等位基因纯合突变的单基因突变体和三基因同时敲除的突变体,突变效率分别为100%、60%;并且证明CRISPR-Cas系统引入的基因修饰可以通过生殖细胞传递到下一代[36]。
Cho等研究表明改造后的化脓性链球菌Streptococcus编码的Cas9内切酶可以在人类细胞的细胞核被活化,通过将构建的特异性识别人体DNA序列的长约20 bp的双RNA复合体或者sgRNA导入到细胞中,实现对人体基因组定点打靶和修饰。该系统已经在多种人体细胞包括诱导多能干细胞中完成对预定的DNA位点进行基因组双链DNA切割,突变率约为38%[17]。Ding等分别利用TALENs和CRISPR-Cas技术在人类多功能干细胞基因组中完成了一个基因的定点打靶,结果表明利用CRISPR-Cas技术的突变效率高,并且还更容易生成纯合子突变克隆 (总克隆的7%-25%)[18]。Mali等通过构建Ⅱ型CRISPR-Cas系统对多种人类细胞系中进行基因定点突变,成功获得了以NHEI方式修复的基因突变,突变效率为2%-38%,研究同时表明突变效率可能与细胞类型和RNA表达载体有关[19]。Cong等构建了两种不同类型的Ⅱ型CRISPR-Cas系统,完成了Cas9核酸酶在短RNAs指导下对人类细胞及小鼠细胞基因组位点特异性切割。通过错配实验证明crRNA的5′端碱基错配对剪切效率影响不大;在EMX1基因位点CRISPR-Cas9引起的基因定点突变效率等于甚至高于TALENs在该位点引起突变的效率[20]。
同时,CRISPR-Cas系统在植物基因修饰方面的应用和研究也取得了重大突破。
Jiang等运用Ⅱ型CRISPR-Cas系统成功利用农杆菌将基因编码的Cas9、sgRNA和一个突变的无功能的绿色荧光蛋白 (GFP) 转入到拟南芥、烟草叶片细胞内。突变的GFP基因在其5′编码区域内存在靶标位点,Ⅱ型CRISPR-Cas系统成功完成定点切割,经过编辑获得了有功能的GFP[42]。研究结果同时证实了CRISPR-Cas9用来定点诱变高粱和水稻基因的可行性[42]。其他研究人员也用CRISPR-Cas系统成功定点突变了拟南芥、水稻多个基因[43, 44, 45, 46]。除此之外,该技术在编辑和定点突变小麦基因方面也取得了成功[87]。研究发现该系统还可以将单链寡核苷酸DNA (ssDNA) 转入植物细胞中作为模板,通过同源重组DNA修复途径在基因特定位点精确插入12 bp两个限制性内切酶识别序列[47]。
4 展望随着多种生物基因测序的完成,利用基因定向修饰技术对目的基因进行定点突变实现重要基因功能的鉴定越来越重要。与传统的遗传学方法相比,基因修饰技术能够精确、高效地对特定基因实现编辑修饰,必将得到越来越广泛的应用。ZFP构建的复杂性使ZFN技术的应用受到很大的限制。为提高效率,减少工作量,高特异性的ZFP筛选以及最优的ZFP模块组合应该是将来ZFN的研究方向。TALEN技术效率高、细胞毒性小、脱靶率低、操作简便,一出现就得到了广泛的研究和应用。利用该技术,科学家已经成功地同时定点突变异源六倍体普通小麦中分别位于A、B、D基因组上的3个MILDEW-RESISTANCE LOCUS (MLO) 同源基因,创制了抗白粉病的转基因小麦种质资源[88]。而近年发展起来的CRISPR-Cas系统具有更加高效、操作简便、实验周期短、成本低,利于在实验室普遍发展等特点,为基因工程提供了一个更加强有力的应用新工具。2014年1月,Cell在线报道利用CRISPR-Cas9系统首次在灵长类动物食蟹猴的单细胞阶段的胚胎中获得了两个靶基因同时发生突变的株系,实现了精准的基因修饰[89]。CRISPR-Cas系统将给基因组定向修饰的研究和应用领域带来突破性的技术革命,特别是在基因功能解析、人类疾病靶向治疗、生物能源及生物制药等应用中具有巨大的潜力和广阔的前景。同时,CRISPR-Cas系统在水稻、小麦等农作物的性状改良与分子定向育种方面也将发挥重要的作用。
| [1] | Xiao A, Wu YD, Yang ZP, et al. EENdb: A database and knowledge base of ZFNs and TALENs for endonuclease engineer. Nucleic Acids Res, 2013, 41(D1): D415-D422. |
| [2] | Sanjana NE, Cong L, Zhou Y, et al. A transcription activator-like effector toolbox for genome engineering. Nat Protoc, 2012, 7(1): 171-192. |
| [3] | Wyman C, Kanaar R. DNA double-strand break repair: all’s well that ends well. Annu Rev Genet, 2006, 40: 363-383. |
| [4] | Tzfwa T, White C. Towards targeted mutagenesis and gene replacement in plants. Trends Biotechnol, 2005, 23(12): 567-569. |
| [5] | Sallaud C, Gay C, Larmande P, et al. High throughput T-DNA insertion mutagenesis in rice. Plant J, 2004, 39(3): 450-464. |
| [6] | Jeon JS, Lee S, Jung KH, et al. T-DNA insertional mutagenesis for functional genomics in rice. Plant J, 2000, 22(6): 561-570. |
| [7] | Greco R, Ouwerkerk PB, Sallaud C, et al. Transposon insertional mutagenesis in rice. Plant Physiol, 2001, 125(3): 1175-1177. |
| [8] | Till BJ, Cooper J, Tai TH, et al. Discovery of chemically induced mutations in rice by TILLING. BMC Plant Biol, 2007, 7: 19. |
| [9] | Mohr SE, Perrimon N. RNAi screening: new approaches understandings, and organisms. Wiley Interdiscip Rev RNA, 2012, 3(2): 145-158. |
| [10] | Jiang W, Bikard D, Cox D, et al. RNA-guided editing of bacterial genomes using CRISPR-Cas systems. Nat Biotechnol, 2013, 31(3): 233-239. |
| [11] | Qi LS, Larson MH, Gilbert LA, et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell, 2013, 152(5): 1173-1183. |
| [12] | Christian M, Cermak T, Doyle EL, et al. Targeting DNA double-strand breaks with TAL effector nucleases. Genetics, 2010, 186(2): 757-761. |
| [13] | Gilbert LA, Larson MH, Morsut L, et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell, 2013, 154(2): 442-451. |
| [14] | Urnov FD, Miller JC, Lee YL, et al. Highly efficient endogenous human gene correction using designed zinc-finger nucleases. Nature, 2005, 435(7042): 646-651. |
| [15] | Perez EE, Wang J, Miller JC, et al. Establishment of HIV-1 resistance in CD4+ T cells by genome editing using zinc-finger nucleases. Nat Biotechnol, 2008, 26(7): 808-816. |
| [16] | Sakuma T, Hosoi S, Woltjen K, et a1. Efficient TALEN construction and evaluation methods for human cell and animal applications. Genes Cells, 2013, 18(4): 315-326. |
| [17] | Cho SW, Kim S, Kim J M, et al. Targeted genome engineering in human cells with the Cas9 RNA-guided endonuclease. Nat Biotechnol, 2013, 31(3): 230-232. |
| [18] | Ding Q, Regan SN, Xia Y, et al. Enhanced efficiency of human pluripotent stem cell genome editing through replacing TALENs with CRISPRs. Cell Stem Cell, 2013, 12(4): 393-394. |
| [19] | Mali P, Yang L, Esvelt KM, et al. RNA-guided human genome engineering via Cas9. Science, 2013, 339(6121): 823-826. |
| [20] | Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems. Science, 2013, 339(6121): 819-823. |
| [21] | Bibikova M, Golic M, Golic KG, et al. Targeted chromosomal cleavage and mutagenesis in Drosophila using zinc-finger nucleases. Genetics, 2002, 161(3): 1169-1175. |
| [22] | Liu J, Li C, Yu Z, et al. Efficient and specific modifications of the Drosophila genome by means of an easy TALEN strategy. J Genet Genomics, 2012, 39(5): 209-215. |
| [23] | Doyon Y, McCammon JM, Miller JC, et al. Heritable targeted gene disruption in zebrafish using designed zinc-finger nucleases. Nat Biotechnol, 2008, 26(6): 702-708. |
| [24] | Meng X, Noyes MB, Zhu LJ, et al. Targeted gene inactivation in zebrafish using engineered zinc-finger nucleases. Nat Biotechnol, 2008, 26(6): 695-701. |
| [25] | Foley JE, Yeh JR, Maeder ML, et al. Rapid mutation of endogenous zebrafish genes using zinc finger nucleases made by oligomerized pool engineering (OPEN). PLoS ONE, 2009, 4(2): e4348. |
| [26] | Sander JD, Cade L, Khader C, et al. Targeted gene disruption in somatic zebrafish cells using engineered TALENs. Nat Biotechnol, 2011, 29(8): 697-698. |
| [27] | Huang P, Xiao A, Zhou M, et al. Heritable gene targeting in zebrafish using customized TALENs. Nat Biotechnol, 2011, 29(8): 699-700. |
| [28] | Moore FE, Reyon D, Sander JD, et al. Improved somatic mutagenesis in zebrafish using transcription activator-like effector nucleases (TALENs). PLoS ONE, 2012, 7(5): e37877. |
| [29] | Zu Y, Tong X, Wang Z, et al. TALEN-mediated precise genome modification by homologous recombination in zebrafish. Nat Methods, 2013, 10(4): 329-331. |
| [30] | Hwang WY, Fu Y, Reyon D, et a1. Efficient genome editing in zebrafish using a CRISPR-Cas system. Nat Biotechnol, 2013, 31(3): 227-229. |
| [31] | Chang N, Sun C, Gao L, et al. Genome editing with RNA-guided Cas9 nuclease in zebrafish embryos. Cell Res, 2013, 23(4): 465-472. |
| [32] | Lei Y, Guo X, Liu Y, et al. Efficient targeted gene disruption in Xenopus embryos using engineered transcription activator-like effector nucleases (TALENs). Proc Natl Acad Sci USA, 2012, 109(43): 17484-17489. |
| [33] | Sung YH, Baek IJ, Kim DH, et al. Knockout mice created by TALEN-mediated gene targeting. Nat Biotechnol, 2013, 31(1): 23-24. |
| [34] | Wang H, Yang H, Shivalila CS, et al. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas mediated genome engineering. Cell, 2013, 153(4): 910-918. |
| [35] | Tong C, Huang G, Ashton C, et al. Rapid and cost-effective gene targeting in rat embryonic stem cells by TALENs. J Genet Genomics, 2012, 39(6): 275-280. |
| [36] | Li W, Teng F, Li T, et al. Simultaneous generation and germline transmission of multiple gene mutations in rat using CRISPR-Cas systems. Nat Biotechnol, 2013, 31(8): 684-686. |
| [37] | Carlson DF, Tan W, Lillico SG, et al. Efficient TALEN-mediated gene knockout in livestock. Proc Natl Acad Sci USA, 2012, 109(43): 17382-17387. |
| [38] | Lloyd A, Plaisier CL, Carroll D, et al. Targeted mutagenesis using zinc-finger nucleases in Arabidopsis. Proc Natl Acad Sci USA, 2005, 102(6): 2232-2237. |
| [39] | Cermak T, Doyle EL, Christian M, et al. Efficient design and assembly of custom TALEN and other TAL effector-based constructs for DNA targeting. Nucleic Acids Res, 2011, 39(12): e82. |
| [40] | Mahfouz MM, Li L, Piatek M, et al. Targeted transcriptional repression using a chimeric TALE-SRDX repressor protein. Plant Mol Biol, 2012, 78(3): 311-321. |
| [41] | Christian M, Qi Y, Zhang Y, et al. Targeted mutagenesis of Arabidopsis thaliana using engineered TAL effector nucleases. G3 (Bethesda), 2013, 3(10): 1697-1705. |
| [42] | Jiang W, Zhou H, Bi H, et al. Demonstration of CRISPR/Cas9/sgRNA-mediated targeted gene modification in Arabidopsis, tobacco, Sorghum and rice. Nucleic Acids Res, 2013, 41(20): e188. |
| [43] | Li JF, Norville JE, Aach J, et al. Multiplex and homologous recombination-mediated genome editing in Arabidopsis and Nicotiana benthamiana using guide RNA and Cas9. Nat Biotechnol, 2013, 31(8): 688-691. |
| [44] | Feng Z, Zhang B, Ding W, et al. Efficient genome editing in plants using a CRISPR/Cas system. Cell Res, 2013, 23(10): 1229-1232. |
| [45] | Li T, Liu B, Spalding MH, et al. High-efficiency TALEN-based gene editing produces disease-resistant rice. Nat Biotechnol, 2012, 30(5): 390-392. |
| [46] | Chen K, Shan Q, Gao C. An efficient TALEN mutagenesis system in rice. Methods, 2014, 69(1): 2-8. |
| [47] | Shan Q, Wang Y, Li J, et al. Targeted genome modification of crop plants using a CRISPR-Cas system. Nat Biotechnol, 2013, 31(8): 686-688. |
| [48] | Zhang Y, Zhang F, Li X, et al. Transcription activator-like effector nuclesses enable plant genome engineering. Plant Physiol, 2013, 161(1): 20-27. |
| [49] | Nekrasov V, Staskawicz B, Weigel D, et al. Targeted mutagenesis in the model plant Nicotiana benthamiana using Cas9 RNA-guided endonuclease. Nat Biotechnol, 2013, 31(8): 691-693. |
| [50] | Shukla VK, Doyon Y, Miller JC, et al. Precise genome modification in the crop species Zea mays using zinc-finger nucleases. Nature, 2009, 459(7245): 437-441. |
| [51] | Wendt T, Holm PB, Starker CG, et al. TAL effector nucleases induce mutation at a pre-selected location in the genome of primary barley transformants. Plant Mol Biol, 2013, 83(3): 279-285. |
| [52] | Mani M, Kandavelou K, Dy FJ, et al. Design, engineering and characterization of zinc finger nucleases. Biochem Biophys Res Commun, 2005, 335(2): 447-457. |
| [53] | Bibikova M, Beumer K, Trautman JK, et al. Enhancing gene targeting with designed zinc finger nucleases. Science, 2003, 300(5620): 764. |
| [54] | Kim YG, Cha J, Chandrasegaran S. Hybrid restriction enzymes: zinc finger fusions to FokⅠ cleavage domain. Proc Natl Acad Sci USA, 1996, 93(3): 1156-1160. |
| [55] | Klug A. The discovery of zinc fingers and their development for practical applications in gene regulation and genome manipulation. Q Rev Biophys, 2010, 43(1): 1-21. |
| [56] | Palpant NJ, Dudzinski D. Zinc finger nucleases: looking toward translation. Gene Ther, 2013, 20(2): 121-127. |
| [57] | Hauschild-Quintern J, Petersen B, Cost GJ, et al. Gene knockout and knockin by zinc-finger nucleases: current status and perspectives. Cell Mol Life Sci, 2013, 70(16): 2969-2983. |
| [58] |
Zhang HY, Zhang XH, Zhang CJ. The use of zinc finger protein nucleases for plant site-directed mutagenesis and replacement. Chin Biotechnol, 2008, 28(11): 110-115 (in Chinese). 张余洋, 张晓辉, 张婵娟. 利用人工锌指蛋白核酸酶进行植物基因定点突变和置换. 中国生物工程杂志, 2008, 28(11): 110-115. |
| [59] | Ramirez CL, Foley JE, Wright DA, et al. Unexpected failure rates for modular assembly of engineered zinc fingers. Nat Methods, 2008, 5(5): 374-375. |
| [60] | Pattanayak V, Ramirez CL, Joung JK, et al. Revealing off-target cleavage specificities of zinc-finger nucleases by in vitro selection. Nat Methods, 2011, 8(9): 765-770. |
| [61] | Radecke S, Radecke F, Cathomen T, et al. Zinc-finger nuclease-induced gene repair with oligodeoxynucleotides: wanted and unwanted target locus modifications. Mol Ther, 2010, 18(4): 743-753. |
| [62] | Moscou MJ, Bogdanove AJ. A simple cipher governs DNA recognition by TAL effectors. Science, 2009, 326(5959): 1501. |
| [63] | Boch J, Bonas U. Xanthomonas AvrBs3 family-type III effectors: discovery and function. Annu Rev Phytopathol, 2010, 48: 419-436. |
| [64] | Ding Q, Lee YK, Schaefer EA, et al. A TALEN genome editing system for generating human stem cell-based disease models. Cell Stem Cell, 2013, 12(2): 238-251. |
| [65] | Mussolino C, Morbitzer R, Lütge F, et al. A novel TALE nuclease scaffold enables high genome editing activity in combination with low toxicity. Nucleic Acids Res, 2011, 39(21): 9283-9293. |
| [66] | Boch J, Scholze H, Schomack S, et al. Breaking the code of DNA binding specificity of TAL-type Ⅲ effectors. Science, 2009, 326(5959): 1509-1512. |
| [67] | Moscou MJ, Bogdanove AJ. A simple cipher governs DNA recognition by TAL effectors. Science, 2009, 326(5959): 1501. |
| [68] | Reyon D, Tsai SQ, Khayter C, et al. FLASH assembly of TALENs for high-throughput genome editing. Nat Biotechnol, 2012, 30(5): 460-465. |
| [69] | Sun N, Zhao H. Transcription activator-like effector nucleases (TALENs): a highly efficient and versatile tool for genome editing. Biotechnol Bioeng, 2013, 110(7): 1811-1821. |
| [70] | Mahfouz MM, Li L, Shamimuzzama M, et al. de novo-engineered transcription activator-like effector (TALE) hybrid nuclease with novel DNA binding specificity creates double-strand breaks. Proc Natl Acad Sci USA, 2011, 108(6): 2623-2628. |
| [71] | Li T, Huang S, Zhao X, et al. Modularly assembled designer TAL effeetor nucleases for targeted gene knockout and gene replacement in eukaryotes. Nucleic Acids Res, 2011, 39(14): 6315-6325. |
| [72] | Lieberman-Lazarovich M, Levy AA. Homologous recombination in plants: an antireview. Methods Mol Biol, 2011, 701: 51-65. |
| [73] |
Zhang DB, Shi P, Pang XN. Research progress in engineered nucleases for genome site-specific editing. Basic Med Sci Clin, 2013, 33(12): 1634-1637 (in Chinese). 张殿宝, 施萍, 庞希宁. 人工核酸酶用于基因组定点编辑的研究进展. 基础医学与临床, 2013, 33(12): 1634-1637. |
| [74] | Godde JS, Bickerton A. The repetitive DNA elements called CRISPRs and their associated genes: evidence of horizontal transfer among prokaryotes. J Mol Evol, 2006, 62(6): 718-729. |
| [75] | Mojica FJ, Diez-Villaseñor C, Soria E, et al. Biological significance of a family of regularly spaced repeats in the genomes of Archaea, Bacteria and mitochondria. Mol Microbiol, 2000, 36(1): 244-246. |
| [76] | Makarova KS, Grishin NV, Shabalina SA, et al. A putative RNA-interference-based immune system in prokaryotes: computational analysis of the predicted enzymatic machinery, functional analogies with eukaryotic RNAi and hypothetical mechanisms of action. Biol Direct, 2006, 1: 7. |
| [77] | Sorek R, Kunin V, Hugenholtz P. CRISPR: a widespread system that provides acquired resistance against phages in bacteria and archaea. Nat Rev Microbiol, 2008, 6(3): 181-186. |
| [78] | Grissa I, Vergnaud G, Pourcel C. The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats. BMC Bioinformatics, 2007, 8: 172. |
| [79] | Karginov FV, Hannon GJ. The CRISPR system: small RNA-guided defense in bacteria and archaea. Mol Cell, 2010, 37(1): 7-19. |
| [80] | Deltcheva E, Chylinski K, Sharma CM, et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase Ⅲ. Nature, 2011, 471(7340): 602-607. |
| [81] | Villion M, Moineau S. The double-edged sword of CRISPR-Cas systems. Cell Res, 2013, 23(1): 15-17. |
| [82] | Ishino Y, Shinagawa H, Makino K, et al. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product. J Bacteriol, 1987, 169(12): 5429-5433. |
| [83] | Marraffini LA, Sontheinmer EJ. CRISPR interference: RNA-directed adaptive immunity in bacteria and archaea. Nat Rev Genet, 2010, 11(3): 181-190. |
| [84] | Friedland AE, Tzur YB, Esvelt KM, et al. Heritable genome editing in Celegans via a CRISPR-Cas9 system. Nat Methods, 2013, 10(8): 741-743. |
| [85] | Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337(6096): 816-821. |
| [86] | Barrangou R. RNA events. Cas9 targeting and the CRISPR revolution. Science, 2014, 344(6185): 707-708. |
| [87] | Shan Q, Wang Y, Li J, et al. Genome editing in rice and wheat using the CRISPR/Cas system. Nat Protoc, 2014, 9(10): 2395-2410. |
| [88] | Wang YP, Cheng X, Shan QW, et al. Simultaneous editing of three homoeoalleles in hexaploid bread wheat confers heritable resistance to powdery mildew. Nat Biotechnol, 2014, 32(9): 947-951. |
| [89] | Hsu PD, Lander ES, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering. Cell, 2014, 157(6): 1262-1278. |
 2015, Vol. 31
2015, Vol. 31




