服务
文章信息
- 陆吉学, 王世珍, 方柏山
- Lu Jixue, Wang Shizhen, Fang Baishan
- 生物分子机器--甲烷单加氧酶的研究进展
- Advances in biomolecular machine: methane monooxygenases
- 生物工程学报, 2015, 31(7): 1015-1023
- Chin J Biotech, 2015, 31(7): 1015-1023
- 10.13345/j.cjb.140517
-
文章历史
- Received: October 28, 2014
- Accepted: January 19, 2015
2厦门市合成生物技术重点实验室,福建 厦门 361005
2. Xiamen Key Laboratory of Synthetic Biotechnology, Xiamen 361005, Fujian, China
1959年,第29届美国物理学会年会上美国著名物理学家理查德•费曼 (Richard Feynman) 演讲中首次提出人工分子机器的概念[1]。分子机器被定义为一种纳米水平的装置,它可以把化学能、电能和光能转化成机械能,人体则可以看成纳米分子机器的集合体[2]。而生物分子机器 (Biological molecular machine) 被定义为能够利用ATP水解释放的化学能转变为机械能的蛋白分子[1]。这种对生物分子机器的定义比较狭窄,实际上Balzani等认为分子机器可定义为把一些分散的分子聚集到一起,在外部驱动力的作用下,它的组成部分有相对位置的变化,表现出类似于机械一样的运动的分子[2]。生物催化涉及到的小分子底物反应,通常由自然界中大分子的蛋白和蛋白复合物来催化完成[3]。甲烷单加氧酶是一种复杂的大分子蛋白复合物,它在催化甲烷生成甲醇的过程中伴随着外部驱动力化学能的输入,促使甲烷单加氧酶亚基之间或亚基中的氨基酸侧链之间发生相对运动[3],Lippard 教授把甲烷单加氧酶称为神奇的生物分子机器[4]。
生物分子机器甲烷单加氧酶(Methanemonooxygenases,MMO) 来自于甲烷氧化菌 (Methanotrophs)。这些细菌是甲基营养型革兰氏阴性菌,特点是能以甲烷为唯一碳源和能源进行生长繁殖,甲烷氧化菌可用于生产虾青素、单细胞蛋白、聚β-羟基丁酸等附加值高的产品;此外由于甲烷氧化菌特有的甲烷单加氧酶底物谱较广使得甲烷氧化菌在重金属、有机物污染的生物修复方面具有潜在的应用价值[5],如甲烷单加氧酶在降解卤代烃过程中起着关键作用[6];甲烷单加氧酶也能够在温和条件下巧妙地催化具有极高键能的甲烷C-H键 (104 kcal/mol) 氧化生成甲醇[7];甲烷氧化菌和甲烷单加氧酶的这些特性使甲烷氧化菌和甲烷单加氧酶成为研究的热点[8]。
1 甲烷单加氧酶产生菌甲烷氧化菌是生物分子机器甲烷单加氧酶的产生菌。荷兰微生物学家Söhngen于1906年首先发现了一株可以利用甲烷的细菌,后被命名为甲烷芽胞杆菌Bacillus methanicus[9]。Whittenbury等通过对100多株甲烷氧化菌的研究,初步建立了甲烷氧化菌的分类方案,并根据形态学、细胞质内膜等的精细结构、细菌休眠期的形式等把甲烷氧化菌分成甲基弯曲菌属Methylosinus、甲基孢囊菌属Methylocysti、甲基单胞菌属Methylomona、甲基杆菌属Methylobacte、甲基球菌属Methylococcus 5个属[10]。这种分类法提供了甲烷氧化菌的分类框架,它与目前被广泛接受的分类法很相似。Bowman等利用 16S rRNA系统分析法解决了许多菌种命名的难题[11]。现在主要根据碳源同化路径、细胞质内膜的显微结构、脂肪酸碳链长度、系统发育、DNA mol% G+C含量和细菌休眠期形式等把甲烷氧化菌分为Ⅰ型、Ⅱ型和X型[11, 12]。Ⅰ型甲烷氧化菌属于变形杆菌纲γ亚纲,此类甲烷氧化菌的特征是:细胞质内的膜贯穿细胞形成多气泡碟片状束;碳的同化路径是磷酸核糖路径;特征性脂肪酸的碳链长度为14C和16C[11, 12, 13, 14]。II型甲烷氧化菌属于变形杆菌纲的α亚纲,其特征为:细胞质内的膜成对齐的线型排布在细胞的外围;利用丝氨酸路径同化碳源;特征性脂肪酸的碳链长度为18C[11, 12, 13, 14, 15, 16]。X型甲烷氧化菌包括Methylococcus和甲基暖菌属Methylocaldum,此类甲烷氧化菌兼有Ⅰ型、Ⅱ型甲烷氧化菌的特性,甲醛的主要代谢途径是RuMP路径且有Serine路径,此类型细菌耐热性较强[13, 14]。
2 甲烷单加氧酶生物分子机器甲烷的C-H键能高,性质稳定,工业生产甲烷的条件非常苛刻,反应温度和压力甚至分别高达900 ℃和1.1×107 Pa[17]。甲烷氧化菌特有的甲烷单加氧酶则能在温和条件下无污染地实现甲烷到甲醇的转化,并在一系列脱氢酶的作用下生成二氧化碳和水。Lippard教授曾在Nature杂志上发表过数篇有关MMO研究方面的文章,在他的主页介绍中称MMO为“Anamazing biomolecular machine” (神奇的生物分子机器)[4]。MMO是甲烷氧化菌的特征酶,可分为2种类型:细胞质内可溶性甲烷单加氧酶 (Soluble methane monooxygenase,sMMO) 和膜结合的颗粒状甲烷单加氧酶 (Particulate methane monooxygenase,pMMO),这2类酶都能够催化甲烷生成甲醇,sMMO可利用的底物范围较广泛可以氧化包括芳香族类的化合物,pMMO底物专一性较强,只能氧化短碳链化合物[18]。
2.1 可溶性甲烷单加氧酶sMMO底物范围宽,不仅能够氧化甲烷,还可以氧化短链烷烃、烯烃及芳香族化合物[19]。sMMO组成复杂,由羟化酶 (MMOH)、蛋白B (MMOB) 和还原酶 (MMOR) 3个部分蛋白组成。其中MMOH分子量为251 kDa,以α、β、γ三个亚基构成的二聚体 (αβγ)2 形式存在。羟化酶的α亚基具有μ-氧桥双铁活性中心。蛋白B分子量为16 kDa,可以调解sMMO的催化反应,它在MMOH的双铁活性中心催化底物羟基化时具有耦合电子的作用;MMOR分子量为 39 kDa,含有双铁-双硫[2Fe-2S]簇和一个辅酶FAD,此部分酶具有还原作用,还原力的来源由NAD(P)H提供[3, 17]。
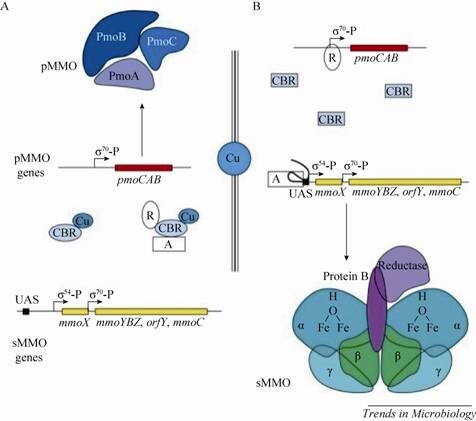
sMMO的基因簇大小为5.5 kb,其排列顺序是mmoXYBZDC,它们分别编码羟化酶的α亚基、羟化酶的β亚基、调控蛋白B、羟化酶的γ亚基、MMOD 蛋白和还原酶C[19, 20]。D基因又称orfY基因,它编码未知功能的MMOD蛋白,Lippard等发现MMOD蛋白可以紧密地结合于sMMO上,可能起着传感器的作用,从而调节sMMO的酶活性[21]。Csaki等通过研究荚膜甲基球菌Methylococcus capsulatus (Bath) sMMO基因,发现sMMO的转录依赖位于mmoX基因区的5′位置的σN启动子,相对pMMO基因来说,sMMO基因的表达需要在低浓度铜离子条件,其机理不能简单地认为是受铜离子浓度的影响,可能是mmoX基因的启动子需要铜离子作为激活剂[22]。实际上关于sMMO的调控存在2种假设 (图1):负控调节假设,游离态抑制剂R具有抑制作用,但在低Cu2+条件下,可结合Cu2+的调控子 (CBR) 不能结合Cu2+,因此不能与抑制剂R结合,不能形成Cu-CBR-A复合物,R起抑制作用,mmoXYBZDC 的转录受到抑制;正控调节假设,游离态激活剂A具有激活作用,但在高浓度Cu2+条件下,CBR结合Cu2+,并与A形成Cu-CBR-A复合物,A没有激活作用,mmoXYBZDC不能转录[17, 23]。
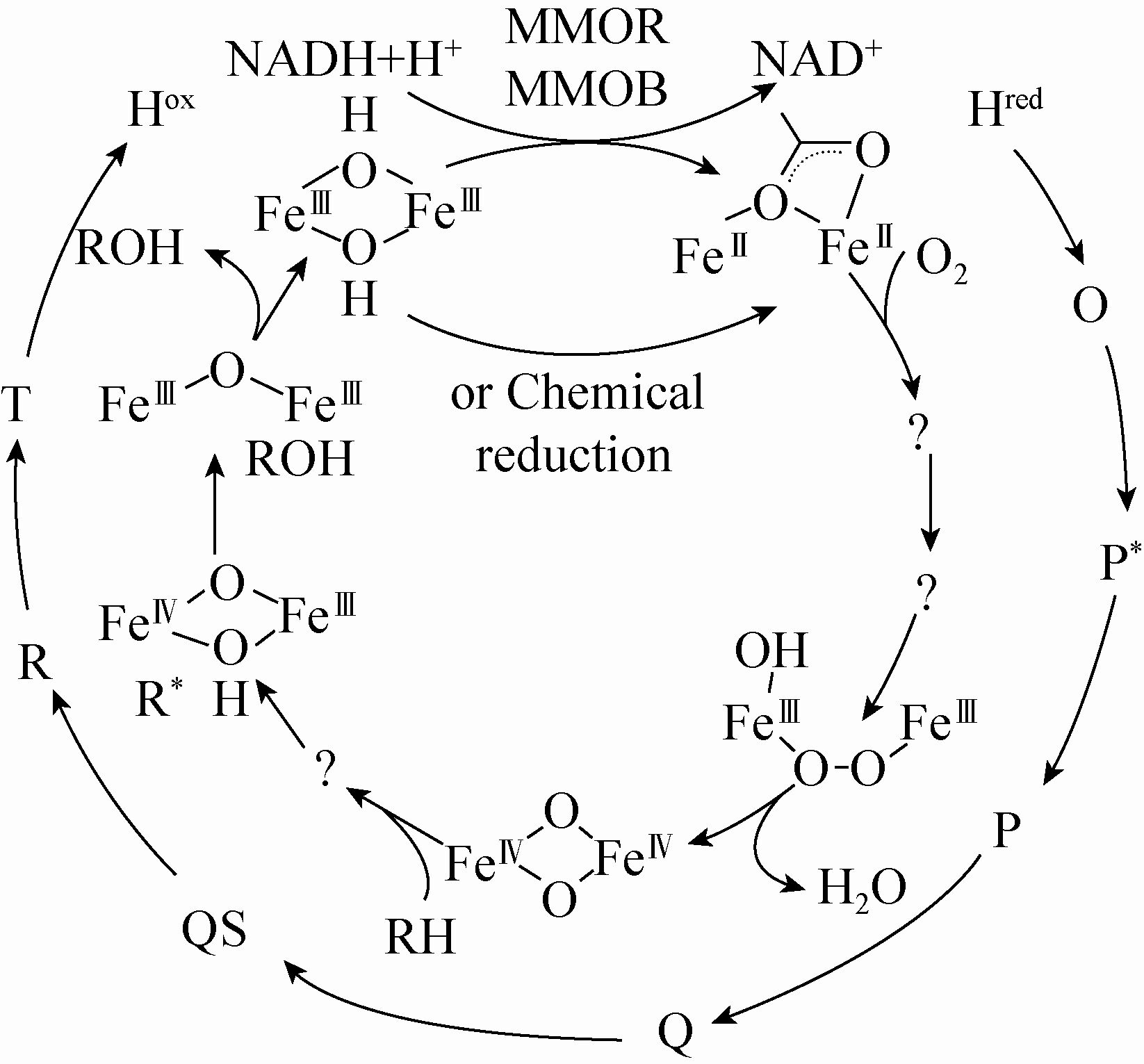
sMMO催化甲烷氧化的过程如图2所示。sMMO在静息态Hox时,羟化酶MMOH的双铁中心以Fe2Ⅲ形式存在,在此状态下不能键合氧气分子,在还原酶MMOR和蛋白B的存在下,通过NAD(P)H,提供2个电子使之变成还原态Hred才能与O2结合,形成O中间态,然后转变成P*中间态,并迅速地衰变为Q态,双铁中心以Fe2IV形式存在,在此状态下参与CH4的氧化[3, 24, 25, 26, 27]。甲烷氧化时关于C-H键的断裂机制有3种假设:C-H键均裂,形成自由基;C-H键异裂,生成碳负离子中间体,碳负离子为了保持稳定,它需要结合到双铁中心的一个铁原子上;C-H键断裂是CH4和Fe-O通过协同反应的断裂机理断裂[17]。至今难以确切判定这3种假设中哪个是C-H键断裂的机理,也有可能是多种键断裂方式并存。Huang等通过密度泛函理论 (Density functional theory) 进行sMMO羟基化甲烷反应机理的研究,并建立了5个双铁模型催化反应机理,发现4个协同的双铁模型是甲烷羟基化反应最有效的模型,此模型涉及到自由基和非自由基中间体反应机理[28]。Lippard等研究了MMOB在sMMO催化甲烷氧化的作用机制,揭示了MMOB的调控原理:MMOB通过其Asp36-Leu129氨基酸残基,锚定在2个MMOH的由α2β2形成的峡谷区,MMOB的长N-末端尾巴链接在MMOH的α亚基上,形成MMOH-MMOB复合体,MMOB的Tyr8和Ser111氨基酸残在空间上诱导MMOH的E α-螺旋的侧链移近双铁活性中心,引起了MMOH构象的改变,因此可以控制质子、甲烷和氧气接近活性位点[3, 27]。MMO这种精巧细致的多亚基协同催化机制,给我们对体内外生物分子机器的构建提供了重要的借鉴意义。
2.2 颗粒性甲烷单加氧酶颗粒性甲烷单加氧酶 (pMMO) 是一种含铜原子的膜结合蛋白[29, 30]。pMMO除了能氧化甲烷外,还可以氧化C2-C5的C-2位置的C-H键生成相应的醇或氧化相应的烯烃生成环氧乙烷[31]。pMMO存在于绝大多数甲烷氧化菌中,其编码基因在高铜离子浓度下转录。关于pMMO的首 次报道是在20世纪70年代中期,相对sMMO来说pMMO底物范围窄,只能氧化碳链长度小于5C的烷烃和烯烃,关于它的研究相对较少,原因是pMMO酶活性比较低且暴露在空气中极不稳定[17, 23]。pMMO由羟化酶 (pMMOH) 和还原酶 (pMMOR) 2部分组成,羟化酶含有3个亚基分别为α (PmoB)、β (PmoA) 和 γ (PmoC),3个亚基的分子量分别为45、27和23 kDa。pMMOR由分子量分别为63和8 kDa的蛋白构成,pMMO以三聚体 (αβγ)3形式存 在[23, 32]。pMMO的活性中心已经证实包含金属元素,一般认为pMMO含有2个金属位点:一个是位于PmoB亚基的双铜中心,一个是位于PmoC亚基的可变金属位点中心[33],但是人们对于金属元素的种类 (Cu,Fe,Zn) 和数目存在很大的争议[17, 19, 34]。Bollinger等综述了pMMO中可能存在的金属种类和数目,从pMMO的晶体结构分析,活性中心的金属元素可能存在于PmoA亚基的三铜簇,或是存在于PmoC亚基的双铁簇,或是存在于PmoB亚基的三铜簇[35]。
pMMO酶的编码基因是pmoCAB,对应编码pMMO的γ、β和α亚基,此操纵子的转录需要σ70作为启动子[23]。研究者已经完成2种不同类型的甲烷氧化菌M. capsu latus (Bath) 和M. trichosporium (OB3b) 的pMMO基因测序,其相似度>99%[23, 36]。MMO的表达都受铜离子浓度的影响,铜离子的浓度不仅决定了sMMO或pMMO基因是否表达,而且对2种不同类型的甲烷单加氧酶的表达量也具有影响[37]。相对于sMMO而言,pMMO的表达需要在较高铜离子浓度的条件。调控机理示意图如图1所示,关于pMMO转录调控有2个假说:负控调节假设,游离态抑制剂R具有抑制活性,但低Cu2+浓度条件下,CBR不能结合Cu2+,因此不能与抑制剂R结合,不能形成Cu-CBR-R复合物,R结合到pmoCAB上游的操纵基因区,pmoCAB的转录受到抑制;正控调节假设,激活剂在形成Cu-CBR-A复合物时具有激活作用,高Cu2+浓度下,激活剂A与Cu-CBR结合后,形成Cu-CBR-A复合物,具有激活作用,pmoCAB可以转录[17, 23]。
研究者们认为pMMO的活性位点位于pmoB亚基的可溶性N末端区域[38]。关于pMMO的催化反应机理,一些学者提出了自己的观点。Wang等通过比较pMMO和重组的pmoB亚基与氧化剂O2或H2O2的催化反应,通过谱峰分析等证明pMMO的氧气结合位点在pmoB的可溶部分的双铜离子中心[39]。Li等认为pMMO催化的甲烷羟基化反应是自由基重排反应,活性中心在双铜离子中心,C-H键的断裂是速度限制步骤,然而Li等、Shiota等认为在双铜离子中心附近的苏氨酸残基提供一个氢原子配位过氧化二铜,形成一个氧化和一个羟基化的铜,从而羟基化甲 烷[40, 41]。Sirajuddin等对pMMO晶体结构衍射分析,在分辨率为2.6Å条件下发现pMMO结构中很可能存在2个Zn2+抑制位点,一个在pmoC亚基中,另一个在pmoC亚基的胞质侧,他们的研究结果表明Zn2+可能通过抑制质子的传递来抑制pMMO活性[42]。pMMO催化甲烷生成甲醇的反应机理争议较大,原因可能是一方面由于pMMO暴露在空气中极不稳定给研究带来困难,另一方面由于pMMO结构比较复杂。
3 展望生物分子机器甲烷单加氧酶,能够在温和的条件下将甲烷氧化成甲醇,如果能够有效地抑制甲醇脱氢酶的活性,如添加氯化铵、EDTA等[43],减弱甲烷氧化菌中甲醇的进一步氧化,则可能实现生物法生产甲醇,节约工业甲醇合成时的能耗。Furuto等利用甲基弯菌Methylosinus trichosporium OB3b发酵生产甲醇,发酵液甲醇的浓度达到6 mmol/L[44];邢新会课题组利用膜反应器进一步将发酵液甲醇的浓度提高到35 mmol/L[45]。甲烷氧化菌也可用于生产虾青素、单细胞蛋白、聚β-羟基丁酸等附加值高的产品,此外由于甲烷单加氧酶底物谱较广使得甲烷氧化菌在重金属、有机物污染的生物修复方面具有潜在的应用价值[5]。虽然用生物分子机器甲烷单加氧酶氧化甲烷生产甲醇还存在许多难题,但是我们可能寻找到一些策略来增加甲醇的生产效率。例如找到甲烷单加氧酶的关键基因,通过构建工程菌高效表达甲烷单加氧酶,在体内外进行催化反应;通过生物信息学分析,定点突变基因,筛选出高酶活菌株等。生物分子机器甲烷单加氧酶复杂精巧的催化反应机制巧妙地催化甲烷氧化成甲醇,也体现了具有生物活性的大分子催化剂的复杂和精巧的特点[3],这些特性对合成生物学、催化剂或分子生物学中生物分子机器设计具有启发和借鉴意义。一些科学家通过模仿MMO的催化机理设计出具有类似MMO活性位点的人造催化剂如3N配体二铁复合物、铜激活的Fe-ZSM-5、Cu-ZSM-5、μ-氮二铁酞菁、三铜复合物等,它们可在较温和的条件下催化烷烃生成醇或降解卤代烃[7, 46, 47, 48, 49, 50, 51, 52],这对于生产甲醇的工艺开发或卤代烃污染物的治理提供了新的方法和思路。
| [1] | Huang TJ, Juluri BK. Biological and biomimetic molecular machines. Nanomedicine, 2008, 3(1): 107-124. |
| [2] | Balzani V, Credi A, Raymo FM, et al. Artificial molecular machines. Angew Chem Int Ed, 2000, (19): 3349-3391. |
| [3] | Lee SJ, McCormick MS, Lippard SJ, et al. Control of substrate access to the active site in methane monooxygenase. Nature, 2013, 494(21): 380-385. |
| [4] | MIT Chemistry Directory: Stephen J. Lippard [EB/OL]. [2014-12-31]. http://chemistry.mit.edu/ people/lippard-stephen. |
| [5] | Pandey VC, Singh JS, Singh DP, et al. Methanotrophs: promising bacteria for environmental remediation. Int J Environ Sci Te, 2014, 11(1): 241-250. |
| [6] |
Xing ZL, Zhang LJ, Zhao TT. Advances in degradation of chlorinated hydrocarbons by obligate and facultative methanotrophs. Chin J Biotech, 2014, 30(4): 531-544 (in Chinese). 邢志林, 张丽杰, 赵天涛. 专一营养与兼性甲烷氧化菌降解氯代烃的研究现状、动力学分析及展望. 生物工程学报, 2014, 30(4): 531-544. |
| [7] | Chan SI, Lu YJ, Nagababu P, et al. Efficient oxidation of methane to methanol by dioxygen mediated by tricopper clusters. Angew Chem Int Edit, 2013, 52(13): 3731-3735. |
| [8] | Lee JH, Kim TG, Cho KS. Isolation and characterization of a facultative methanotroph degrading malodor-causing volatile sulfur compounds. J Hazard Mater, 2012, 235(10): 224-229. |
| [9] | Theisen AR, Murrell JC. Facultative methanotrophs revisited. J Bacteriol, 2005, 187(13): 4303-4305. |
| [10] | Whittenbury R, Phillips KC, Wilkinson JF. Enrichment, isolation and some properties of methane-utilizing bacteria. J Gen Microbiol, 1970, 61(2): 205-218. |
| [11] | Bowman J. The methanotrophs-the families Methylococcaceae and Methylocystaceae. Prokaryotes, 2006, 5(1): 266-289. |
| [12] | Bowman JP, Sly LI, Nichols PD, et al. Revised taxonomy of the methanotrophs-description of Methylobacter Gen-Nov, emendation of Methylococcus, validation of Methylosinus and Methylocystis species, and a proposal that the family Methylococcaceae includes only the Group-I methanotrophs. Int J Syst Bacteriol, 1993, 43(4): 735-753. |
| [13] | Semrau JD, DiSpirito AA, Yoon S. Methanotrophs and copper. FEMS Microbiol Rev, 2010, 34(4): 496-531. |
| [14] | Murrell JC. Handbook of Hydrocarbon and Lipid Microbiology: the Aerobic Methane Oxidizing Bacteria (Methanotrophs). Berlin Heidelberg: Springer-Verlag, 2010: 1953-1966. |
| [15] | Park D, Lee J. Biological conversion of methane to methanol. Korean J Chem Eng, 2013, 30(5): 977-987. |
| [16] | Jiang H, Chen Y, Murrell JC, et al. Methanotrophs: multifunctional bacteria with promising applications in environmental bioengineering. Biochem Eng J, 2010, 49(3): 277-288. |
| [17] | Dalton H. The leeuwenhoek lecture 2000-the natural and unnatural history of methane-oxidizing bacteria. Phil Trans R Soc B, 2005, 360(1458): 1207-1222. |
| [18] |
Xu N, Xin JY, Wang Y, et al. Research advances of methane monooxygenase. Lett Biotechnol, 2013, 24(1): 130-133 (in Chinese). 徐宁, 辛嘉英, 王艳, 等. 甲烷单加氧酶的研究进展. 生物技术通讯, 2013, 24(1): 130-133. |
| [19] | Trotsenko YA, Murrell JC. Metabolic aspects of aerobic obligate methanotrophy. Adv Appl Microbiol, 2008, 63(1): 183-229. |
| [20] | Hua SF, Song YY, Li SB, et al. Sequencing analysis of 16S rDNA and soluble methane monooxygenase genes from a methanotroph Methylosinus trichosporium IMV 3011. Ann Microbiol, 2011, 61(2): 391-396. |
| [21] | Merkx M, Lippard SJ. Why OrfY? characterization of MMOD, a long overlooked component of the soluble methane monooxygenase from Methylococcus capsulatus (Bath). J Biol Chem, 2002, 277(8): 5858-5865. |
| [22] | Csaki R, Bodrossy L, Kovacs KL, et al. Genes involved in the copper-dependent regulation of soluble methane monooxygenase of Methylococcus capsulatus (Bath): cloning, sequencing and mutational analysis. Microbiol-SGM, 2003, 149(7): 1785-1795. |
| [23] | Murrell JC, McDonald IR, Gilbert B. Regulation of expression of methane monooxygenases by copper ions. Trends Microbiol, 2000, 8(5): 221-225. |
| [24] | Banerjee R, Meier KK, Munck E, et al. Intermediate P* from soluble methane monooxygenase contains a diferrous cluster. Biochemistry-Us, 2013, 52(25): 4331-4342. |
| [25] | Tinberg CE, Lippard SJ. Revisiting the Mechanism of Dioxygen Activation in Soluble Methane Monooxygenase from M. capsulatus (Bath): Evidence for a Multi-Step, Proton-Dependent Reaction Pathway. Biochemistry-Us, 2009, 48(51): 12145-12158. |
| [26] | Gherman BF, Dunietz BD, Lippard SJ, et al. Activation of the C-H bond of methane by intermediate Q of methane monoozygenase: a theoretical study. J Am Chem Soc, 2001, 123(16): 3836-3837. |
| [27] | Wang WX, Iacob RE, Lippard SJ, et al. Electron transfer control in soluble methane monooxygenase. J Am Chem Soc, 2014, 136(27): 9754-9762. |
| [28] | Huang SP, Shiota Y, Yoshizawa K. DFT study of the mechanism for methane hydroxylation by soluble methane monooxygenase (sMMO): effects of oxidation state, spin state, and coordination number. Dalton Trans, 2013, 42(4): 1011-1123. |
| [29] | Culpepper MA, Cutsail GE, Rosenzweig AC, et al. Identification of the valence and coordination environment of the particulate methane monooxygenase copper centers by advanced EPR characterization. J Am Chem Soc, 2014, 136(33): 11767-11775. |
| [30] | Tavormina PL, Ussler W, Orphan VJ, et al. Abundance and distribution of diverse membrane-bound monooxygenase (Cu-MMO) genes within the Costa Rica oxygen minimum zone. Env Microbiol Rep, 2013, 5(3): 414-423. |
| [31] | Chen YS, Luo WI, Chiang CH, et al. Controlled oxidation of aliphatic C-H bonds in metallo-monooxygenases: mechanistic insights derived from studies on deuterated and fluorinated hydrocarbons. J Inorg Biochem, 2014, 134(5): 118-133. |
| [32] | Dispirito A, Gulledge J, Krema C, et al. Trichloroethylene oxidation by the membrane-associated methane monooxygenase in type I, type II and type X methanotrophs. Biodegradation, 1991, 2(3): 151-164. |
| [33] | Liew EF, Tong DC, Holmes AJ, et al. Mutagenesis of the hydrocarbon monooxygenase indicates a metal centre in subunit-C, and not subunit-B, is essential for copper-containing membrane monooxygenase activity. Microbiol-SGM, 2014, 160(6): 1267-1277. |
| [34] | Lieberman RL, Rosenzweig AC. Biological methane oxidation: regulation, biochemistry, and active site structure of particulate methane monooxygenase. Crit Rev Biochem Mol, 2004, 39(3): 147-164. |
| [35] | Bollinger JM. Biochemistry getting the metal right. Nature, 2010, 465(6): 40-41. |
| [36] | Murrell JC, Gilbert B, McDonald IR. Molecular biology and regulation of methane monooxygenase. Arch Microbiol, 2000, 173(5): 325-332. |
| [37] | Ho A, Luke C, Frenzel P, et al. Selective stimulation in a natural community of methane oxidizing bacteria: effects of copper on pmoA transcription and activity. Soil Biol Biochem, 2013, 65(10): 211-216. |
| [38] | Lawton TJ, Ham J, Rosenzweig AC, et al. Structural conservation of the B subunit in the ammonia monooxygenase/particulate methane monooxygenase superfamily. Proteins, 2014, 82(9): 2263-1167. |
| [39] | Wang WX, Lippard SJ. Diiron oxidation state control of substrate access to the active site of soluble methane monooxygenase mediated by the regulatory component. J Am Chem Soc, 2014, 136(6): 2244-2247. |
| [40] | Li CQ, Yang HQ, Hu CW, et al. Hydroxylation mechanism of methane and its derivatives over designed methane monooxygenase model with peroxo dizinc core. Org Biomol Chem, 2012, 10(19): 3924-3931. |
| [41] | Shiota Y, Juhasz G, Yoshizawa K. Role of tyrosine residue in methane activation at the dicopper site of particulate methane monooxygenase: a density functional theory study. Inorg Chem, 2013, 52(14): 7907-7917. |
| [42] | Sirajuddin S, Barupala D, Rosenzweig AC, et al. Effects of zinc on particulate methane monooxygenase activity and structure. J Biol Chem, 2014, 289(31): 21782-21794. |
| [43] | Han JS, Ahn CM, Kim CG, et al. Partial oxidative conversion of methane to methanol through selective inhibition of methanol dehydrogenase in methanotrophic consortium from landfill cover soil. Appl Biochem Biotech, 2013, 171(6): 1487-1499. |
| [44] | Furuto T, Takeguchi M, Okura I. Semicontinuous methanol biosynthesis by Methylosinus trichosporium OB3b. J Mol Catal A-Chem, 1999, 144(2): 257-261. |
| [45] | Duan CH, Luo MF, Xing XH. High-rate conversion of methane to methanol by Methylosinus trichosporium OB3b. Bioresour Technol, 2011, 102(15): 7349-7353. |
| [46] | Hammond C, Forde MM, Jenkins RL, et al. Direct catalytic conversion of methane to methanol in an aqueous medium by using copper-promoted fe-zsm-5. Angew Chem Int Ed, 2012, 51(21): 5129-5133. |
| [47] | Shan JJ, Huang WX, Li YY, et al. Conversion of methane to methanol with a bent mono (mu-oxo) dinickel anchored on the internal surfaces of micropores. Langmuir, 2014, 30(28): 8558-8569. |
| [48] | Chen PPY, Nagababu P, Chan SI, et al. Development of the tricopper cluster as a catalyst for the efficient conversion of methane into MeOH. Chemcatchem, 2014, 6(2): 429-437. |
| [49] | Sankaralingam M, Palaniandavar M. Diiron(III) complexes of tridentate 3N ligands as functional models for methane monooxygenases: effect of the capping ligand on hydroxylation of alkanes. Polyhedron, 2014, 67(1): 171-180. |
| [50] | Colomban C, Kudrik EV, Sorokin AB, et al. Degradation of chlorinated phenols in water in the presence of H2O2 and water-soluble mu-nitrido diiron phthalocyanine. Catal Today, 2014, 235(10): 14-19. |
| [51] | Stoian SA, Xue GQ, Munck E, et al. Spectroscopic and theoretical investigation of a complex with an [O=Fe-IV-O-Fe-IV=O] core related to methane monooxygenase intermediate Q. J Am Chem Soc, 2014, 136(4): 1545-1558. |
| [52] | Sheppard T, Hamill CD, Thompson JM, et al. A low temperature, isothermal gas-phase system for conversion of methane to methanol over Cu-ZSM-5. Chem Commun, 2014, 50(75): 11053-11055. |
 2015, Vol. 31
2015, Vol. 31