服务
文章信息
- 陶正生, 王业民, 郑华亮, 陶美凤
- Tao Zhengsheng, Yemin Wang, Hualiang Zheng, Tao Meifeng
- 异源表达afsRScla全局性调控基因提高工业菌株纳他霉素产量
- Improvement of natamycin production in an industrial strain by heterologous expression of the afsRScla global regulatory genes
- 生物工程学报, 2015, 31(5): 744-751
- Chin J Biotech, 2015, 31(5): 744-751
- 10.13345/j.cjb.140173
-
文章历史
- Received: March 22, 2014
- Accepted: May 5, 2014
纳他霉素 (Natamycin) 又称匹马菌素 (Pimaricin),是一种二十六元多烯大环内酯类抗生素[1],具有很强的抗真菌活性,能特异性地抑制酵母菌和霉菌的生长[2]。目前发现的纳他霉素产生菌主要有3种:纳他链霉菌Streptomyces natalensis[3],恰塔努加链霉菌Streptomyces chattanoogensis[4]及褐黄孢链霉菌Streptomyces gilvosporeus[5]。
纳他霉素通过与真菌细胞膜上的甾醇化合物发生反应,改变细胞膜结构,从而使细胞破裂,导致细胞死亡[6]。由于纳他霉素对真菌作用的广谱性,并具有良好的理化稳定性及对人体无毒害,已被全球多个国家作为食品防腐剂批准使用。除了作为食品防腐剂广泛应用于食品行业,纳他霉素还被应用于临床医疗,如治疗真菌性角膜炎等[7]。纳他霉素应用领域广泛,但是较高生产成本是限制其广泛使用的主要因素,因此提高纳他霉素工业生产菌株的产量,降低其生产成本,有利于进一步推广应用。
纳他霉素生物合成基因簇含有19个开放阅读框,其中包括2个途径特异性调节基因 (pimR和pimM)、5个负责纳他霉素聚酮骨架合成的聚酮合酶基因 (pimS0−pimS4) 和12个负责后修饰和其他功能的基因[1, 8] (图1)。pimI合成一种Ⅱ型硫脂酶,可能具有编辑校正功能,使纳他霉素得以积累[9]。pimJ为GDP-甘露糖脱水酶,参与海藻糖胺糖部分的形成[10];pimK为糖基转移酶,负责C-15位上甘露糖的转移[11]。
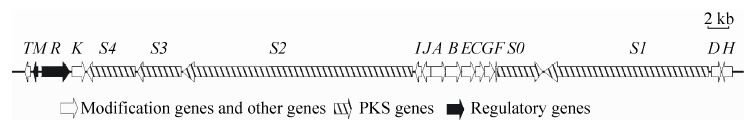
|
| 图1 褐黄孢链霉菌的纳他霉素生物合成基因簇 Fig.1 The natamycin biosynthetic gene cluster from S. gilvosporeus. |
链霉菌的次级代谢受到许多因素的影响,其中转录水平的调控受到多种转录调节蛋白的控制。AfsR是一种较早在链霉菌中发现的转录正调控蛋白,含有993个氨基酸,在其N端有SARP类家族的两个特征结构域,还含有可以结合ATP的保守基元[12]。AfsR是一个全局性调控因子,通过磷酸化介导的信号转导作用将生理和环境信号进行整合,对多种抗生素合成具有调控作用[13]。蛋白激酶AfsK、PkaG和AfsL可以磷酸化AfsR[14],AfsR蛋白的丝氨酸和苏氨酸残基磷酸化使AfsR的DNA结合能力增强。被磷酸化后AfsR可结合在紧邻afsR下游的afsS启动子区,从而启动afsS转录。天蓝色链霉菌afsS编码64个氨基酸的小蛋白AfsS,虽然其功能尚未确定,但它的存在可以显著增强放线紫红素、钙依赖抗生素和十一烷基灵菌红素的合成[15]。本课题组陈黎等从棒状链霉菌Streptomyces clavuligerus克隆得到afsRScla,包含afsR和afsS两个基因,发现afsRScla不但可以提高棒状链霉菌中克拉维酸的产量并激活沉默的全霉素的合成,同时还可以激活异源宿主变铅青链霉菌Streptomyces lividans 中沉默的放线紫红素和钙依赖抗生素的产生[16]。本研究通过把全局性调控基因afsRScla导入工业生产菌株褐黄孢链霉菌TZ1401中,考察afsRScla对纳他霉素生物合成基因转录的影响,通过基因工程的方法提高纳他霉素工业生产菌株的产量,获得高产菌株。
1 材料与方法 1.1 材料 1.1.1 菌株与质粒大肠杆菌ET12567/pUZ8002为大肠杆菌-链霉菌属间接合转移供体菌[17],本实验室保存。纳他霉素工业生产菌株褐黃孢链霉菌TZ1401由工厂提供。质粒pSET152含接合转移起始位点oriTRK2、阿泊拉霉素抗性基因aac(3)Ⅳ、放线菌噬菌体FC31整合位点attP以及整合酶基因int[18]。pHL851为pSET152衍生质粒,含有棒状链霉菌afsRScla基因,由Chen等构建[16]。
1.1.2 主要试剂DNA聚合酶rTaq及相关试剂购自TaKaRa公司;抗生素购自Sigma公司;其余试剂购自国药集团或Oxoid公司。发酵纳他霉素的种子培养基和发酵培养基粉末由纳他霉素生产厂家提供。
1.1.3 培养基LB培养基 (g/L):酵母抽提物5,胰蛋白胨10,NaCl 5,pH 7.0,用于液体培养大肠杆菌。
2×YT培养基 (g/L):酵母抽提物10,胰蛋白胨16,NaCl 5,pH 7.2,用于接合转移时链霉菌孢子的热激处理。
2CMY培养基 (g/L):可溶性淀粉10,胰蛋白胨2,NaCl 1,(NH4)2SO4 2,CaCO3 2,MgSO4·7H2O 2,K2HPO4 1,无机盐溶液 (1 mL),琼脂20,pH 7.2,用于褐黄孢链霉菌的接合转移。其中无机盐溶液 (g/L):FeSO4·7H2O 1,MgCl2·6H2O 1,ZnSO4·7H2O 1。
COM培养基(g/L):玉米淀粉10,燕麦粉5,麦芽提取物5,酵母抽提物2,琼脂15,pH 7.2,用于褐黄孢链霉菌培养和产孢。
纳他霉素种子培养基 (g/L):种子培养基粉末43,pH 7.2。
纳他霉素发酵培养基 (g/L):发酵培养基粉末88.5,pH 7.2。
1.1.4 引物用于PCR验证重组菌株以及qRT-PCR的引物序列如表1所示,由生工生物工程 (上海) 股份有限公司合成。引物afsRS-F、afsRS-R、Apr-F和Apr-R根据pHL851序列设计,两对引物的序列分别位于阿泊拉霉素抗性基因aac(3)Ⅳ和afsRScla基因[16],用于验证pSET152和pHL851是否存在于重组链霉菌菌株。其他引物用于qRT-PCR,本研究中褐黄孢链霉菌纳他霉素基因簇序列已部分测序,和纳他链霉菌纳他霉素基因簇序列 (基因序列号为AJ278573) 相同,qRT-PCR引物序列参考AJ278573序列,引物名称与所扩增的基因名称一一对应。
| Primer | Nucleotide sequence (5′-3′) |
| afsRS-F | GTCCGTGTACGCCTCCTTGT |
| afsRS-R | CGACCTCGATGTGATGTCCC |
| Apr-F | TGGGCAGGTACTTCTCCTCG |
| Apr-R | GCATCGCATTCTTCGCATC |
| hrdB-F | GCACATCCCGTACGCTCCCG |
| hrdB-R | CTCGAAGGCCCGACGCACGT |
| pimS1-F | GCTACGACTTCCACACCGAG |
| pimS1-R | CTGGAAGTGGGGACCGTAGT |
| pimS3-F | GCAGGGCTGATCAAGATGAT |
| pimS3-R | GCCACTCCTGAGGCTCCGTC |
| pimM-F | ATGACGGGCGTGACGAATC |
| pimM-R | CGAGCCTTGATAGAACATTGACC |
| pimI-F | ACGACCCCAAGACCACGCTG |
| pimI-R | TTCCCGCAGCAGTGCCAGTA |
| pimJ-F | GGTCAGGCGAACCCCAGGAA |
| pimJ-R | CTGCTGCCAGGACATCGGTA |
| pimK-F | CCGCTGTGGGTGGACTGCTA |
| pimK-R | GAAGGACGGGGTGCCGAGAA |
链霉菌与大肠杆菌属间接合转移参考文献[17]。将酶切验证正确的质粒pHL851和空载质粒pSET152分别电转到ET12567/pUZ8002感受态细胞,获得的转化子ET12567/pUZ8002/pHL851和ET12567/pUZ8002/pSET152,经过质粒提取和酶切验证正确后,作为接合转移供体菌;褐黃孢链霉菌TZ1401作为受体菌。大肠杆菌和热激后的链霉菌孢子混合后涂布2CMY平板,30 ℃倒置培养20 h后用阿泊拉霉素 (终浓度50 μg/mL) 和三甲氧苄啶 (终浓度50 μg/mL) 覆盖,继续培养直至接合转移子长出。各随机挑取12个形态正常的接合转移子,在COM平板 (含50 μg/mL的阿泊拉霉素和25 μg/mL萘啶酮酸) 划菌块进行抗性验证。选取抗性验证正确的重组菌株TZ1401/pSET152和TZ1401/pHL851,提取总DNA,PCR验证菌株含正确的质粒。
1.2.2 褐黃孢链霉菌的摇瓶发酵将重组菌TZ1401/pSET152和TZ1401/pHL851在COM培养基上培养至产孢,用1 mL蓝色塑料枪头粗口端打菌块 (约1 cm2/块),接种至30 mL种子培养基中 (250 mL摇瓶),在30 ℃、220 r/min振摇 (5 cm振幅) 培养43 h。转接3 mL种子培养物至25 mL发酵培养基中 (250 mL摇瓶),30 ℃、220 r/min振摇 (5 cm振幅) 继续培养5 d,发酵结束。
1.2.3 褐黃孢链霉菌发酵液的处理方法纳他霉素的提取:取发酵液0.5 mL加入到2 mL离心管中,7 000 r/min离心,弃上清,菌渣中加入1.5 mL 5%冰乙酸甲醇,充分打散后超声处理20 min,7 000 r/min离心,取上清50 μL加入到950 μL 5%冰乙酸甲醇中,充分混匀后过滤,取150 μL于内衬管中,进行HPLC检测。
菌浓测定:取3 mL发酵液,7 000 r/min离心,收集菌体,称量菌体;菌浓 (g/mL)=菌体质量/3。
1.2.4 检测纳他霉素产量的HPLC方法色谱柱(Agilent ZORBAX SB-C18 4.6 mm× 250 mm,5 μm);色谱仪:Agilent1260;流动相:甲醇:水:冰乙酸 (60:40:5);流速:0.5 mL/min;检测波长:303 nm;上样量:10 μL。纳他霉素标品 (含量89.9%),配制成0.1 mg/mL作为标准品对照;纳他霉素发酵效价计算公式:发酵效价 (g/L)=样品峰面积/标准品峰面积×稀释倍数×标准品浓度。
1.2.5 qRT-PCR分析基因表达量的方法取在发酵培养基生长96 h的TZ1401、TZ1401/pSET152和TZ1401/pHL851各3个平行样品发酵液,参照RNA提取试剂盒 (赛百盛基因公司) 说明书抽取菌体总RNA,残余的基因组总DNA用DNaseⅠ进行消化。DNA消化后用Nanodrop (Thermo) 定量RNA的浓度。参照反转录试剂盒 (Fermentas) 说明书进行RNA的反转录。以反转录后的cDNA为模板,选用6个具有代表性的纳他霉素基因簇内的基因进行qRT-PCR,以hrdB (基因序列号为X52983) 作为内参基因。扩增程序:95 ℃预变性10 min;随后95 ℃变性15 s,60 ℃退火和延伸1 min,共40个循环。
qRT-PCR采用2-ΔΔCT相对基因表达分析方法[19],以hrdB作为内参基因。设计7个基因的qRT-PCR引物,扩增片段大小200 bp以内,引物见表1。7个基因的qRT-PCR反应完成后的溶解曲线均呈现单一峰,扩增产物只有一条预期大小的目标条带,说明7对引物均能特异扩增,没有引物二聚体的形成,同时反转录合成的cDNA也没有受到基因组DNA污染。确保qRT-PCR数据具有可靠性。
2 结果与分析 2.1 afsRScla对工业菌株S. gilvosporeus TZ1401生长的影响将pSET152和pHL851分别接合转移到褐黄孢链霉菌TZ1401,各随机挑取12个接合转移子,转接培养验证菌株具有阿泊拉霉素抗性;再分别随机挑取其中抗性正确的菌株提取总DNA,进行PCR验证。PCR结果如图2所示,TZ1401/pSET152重组菌 (8株) 均可扩增出阿泊拉霉素抗性基因 (345 bp),但不能扩增afsRScla基因 (934 bp);TZ1401/pHL851重组菌 (7株) 则可以扩增出两个基因。PCR表明质粒pSET152和pHL851分别正确导入了TZ1401。把重组菌株TZ1401/pSET152和TZ1401/pHL851分别传代培养,与出发工业菌株TZ1401对照观察生长、产孢状况,发现3种菌株单菌落形态基本相似,但是含有afsRScla的菌株TZ1401/pHL851产气生菌丝和孢子时间均有约12 h延迟,TZ1401/pHL851在第4天产白色气生菌丝,第10天出现灰色孢子丝。

|
| 图2 PCR检测重组菌株的afsRScla (A) 和aac(3)Ⅳ (B) Fig.2 Detection of afsRScla (A) and aac(3)Ⅳ (B) in the recombinant strains by PCR. M: DNA marker; 1−8: 8 strains TZ1401/pSET152; 9−15: 7 strains TZ1401/pHL851; 16: control TZ1401; 17: control plasmid pSET152; 18: control plasmid pHL851. |
分别挑取TZ1401/pSET152和TZ1401/pHL851的3株平行菌株进行摇瓶发酵,工厂菌株TZ1401作为对照,每个菌株各重复发酵4瓶。在发酵结束后,测量菌体浓度 (图3A),TZ1401的平均菌浓为0.38 g/mL,TZ1401/pSET152菌浓略低于TZ1401,TZ1401/pHL851平均菌浓与TZ1401/pSET152相当,暗示菌浓改变系由载体引入菌株引起。HPLC检测纳他霉素发酵产量,结果见图3B,工厂菌株TZ1401的纳他霉素平均效价为2.58 g/L;导入空载质粒pSET152的3株菌株纳他霉素产量降低4%−10%;而导入含有afsRScla的pHL851的3株重组菌的纳他霉素产量平均效价达到 (3.56±0.04) g/L,与出发工业菌株TZ1401比提高34%−40% (平均提高38%),而与TZ1401/pSET152比则提高50%。

|
| 图3 工业菌株TZ1401及含pSET152或pHL851的重组菌摇瓶发酵液的菌浓 (A) 和发酵效价 (B) Fig.3 Biomass concentration (A) and fermentation titers (B) of the industrial strain TZ1401 and recombinants carrying pSET152 or pHL851. Each data bar represents $\bar x$±s from four independent samples. |
选取纳他霉素基因簇内6个基因进行转录水平分析,研究导入全局性调控基因afsRScla对工业菌株TZ1401纳他霉素生物合成基因转录水平的影响。选取的6个基因包括调控基因pimM和结构基因pimS1、pimS3、pimI、pimJ和pimK。前期实验表明在96 h时,TZ1401纳他霉素合成基因表达量达到最大值 (未发表)。因此选择在发酵培养96 h时进行基因转录水平分析,考察6个基因在TZ1401、TZ1401/pSET152和TZ1401/pHL851三种菌株的相对转录水平。每个菌株平行发酵3瓶作为重复,qRT-PCR结果如图4所示。导入空载质粒pSET152对工业菌株TZ1401纳他霉素基因簇的6个基因表达几乎没有影响,而导入pHL851后纳他霉素基因簇的6个基因表达量都有提高,最低的pimJ有1.9倍的提高,最高的pimS1则有2.7倍的提高。表明纳他霉素合成基因受到了全局性调控基因afsRScla的正调控作用,这和纳他霉素产量提高的结果是一致的,但是转录水平比产量提高的更多,可能因为次级代谢产物的产量受到多种因素影响,比如前体物供应及翻译效率等。

|
| 图4 qRT-PCR分析afsRScla对纳他霉素基因簇转录的影响 Fig.4 Analysis of the effect of afsRScla on transcription of the natamycin gene cluster by qRT-PCR. TZ1401 was used as calibrator. hrdB was used as the internal control. Each data bar represents $\bar x$±s from three independent samples. |
在链霉菌中,afsR是研究较清楚的与次级代谢相关的全局性调控基因,它首先在天蓝色链霉菌Streptomyces coelicolor中被发现,能够引起A-因子、放线紫红素和十一烷基灵菌红素合成基因的高表达[20, 21]。在波赛链霉菌Streptomycespeucetius中发现了afsR的同源基因,过表达该afsR同源基因时,能够引起波赛链霉菌中阿霉素(Doxorubicin)、变铅青链霉菌中放线紫红素、棒状链霉菌中克拉维酸和灰色链霉菌Streptomyces griseus 中链霉素 (Streptomycin) 的高表达[22],表明afsR可能对多种链霉菌中的多种抗生素都有正调节作用。我们早期研究中发现来源于棒状链霉菌的afsRScla能够激活变铅青链霉菌沉默的放线紫红素、钙依赖性抗生素和棒状链霉菌全霉素的生物合成,并大幅度提高克拉维酸的产量[16]。这是在野生型链霉菌中的情况。
本研究运用基因工程的方法改造了纳他霉素工业菌株褐黄孢链霉菌TZ1401,把棒状链霉菌的全局性调控基因afsRScla导入到TZ1401中,摇瓶发酵发现afsRScla使纳他霉素产量提高38%,表明afsRS对工业菌株的抗生素合成也具有促进作用。同时,qRT-PCR结果表明,含有afsRScla的TZ1401/pHL851菌株纳他霉素基因簇的6个代表性基因其转录水平都有2倍左右的提高,说明全局性调控基因afsRScla通过正调控纳他霉素基因簇的表达水平,从而提高纳他霉素的产量。
在前人研究基础上[15, 23],推测afsRScla提高纳他霉素合成的机理为:afsRcla产物AfsR被蛋白激酶磷酸化,磷酸化的AfsR的DNA结合能力和ATPase活性都增强,磷酸化的AfsR结合到下游的afsScla的启动子上形成封闭型的转录复合物,利用ATP水解释放的能量,将封闭型的转录复合物转变为开放的转录复合物,从而诱导afsScla的转录,AfsS能够激活调控基因的转录,包括途径特异性调控基因pimM的转录,进而促进纳他霉素生物合成基因的转录,引起纳他霉素的高产。
基因工程育种可以有目的地改造菌株,进行定向选育。随着对菌株遗传背景和代谢途径的充分了解,基因工程育种势必成为菌株选育重要手段。据本研究结果,全局性正调控基因afsRScla可以应用于工业生产菌株,这对于使用afsRScla来提高其他抗生素工业菌株的产量有重要借鉴意义。
| [1] | Aparicio JF, Colina AJ, Ceballos E, et al. The biosynthetic gene cluster for the 26-membered ring polyene macrolide Pimaricin. J Biol Chem, 1999, 274(15): 10133-10139. |
| [2] | Pedersen JC. Natamycin as a fungicide in agar media. Appl Environ Microbiol, 1992, 58(3): 1064-1066. |
| [3] | el-Enshasy HA, Farid MA, el-Sayed el-SA. Influence of inoculum type and cultivation conditions on natamycin production by Streptomyces natalensis. J Basic Microbiol, 2000, 40(5/6): 333-342. |
| [4] | Du YL, Chen SF, Cheng LY, et al. Identification of a novel Streptomyces chattanoogensis L10 and enhancing its natamycin production by overexpressing positive regulator ScnRII. J Microbiol, 2009, 47(4): 506-513. |
| [5] | Chen GQ, Lu FP, Du LX. Natamycin production by Streptomyces gilvosporeus based on statistical optimization. J Agric Food Chem, 2008, 56(13): 5057-5061. |
| [6] | te Welscher YM, ten Napel HH, Balagué MM, et al. Natamycin blocks fungal growth by binding specifically to ergosterol without permeabilizing the membrane. J Biol Chem, 2008, 283(10): 6393-6401. |
| [7] | O'Day DM, Head WS. Advances in the management of keratomycosis and Acanthamoeba keratitis. Cornea, 2000, 19(5): 681-687. |
| [8] | Vicente CM, Santos-Aberturas J, Guerra SM, et al. PimT, an amino acid exporter controls polyene production via secretion of the quorum sensing pimaricin-inducer PI-factor in Streptomyces natalensis. Microb Cell Fact, 2009, 8(1): 33. |
| [9] | Kim BS, Cropp TA, Beck BJ, et al. Biochemical evidence for an editing role of thioesterase II in the biosynthesis of the polyketide pikromycin. J Biol Chem, 2002, 277(50): 48028-48034. |
| [10] | Aparicio JF, Caffrey P, Gil JA, et al. Polyene antibiotic biosynthesis gene clusters. Appl Microbiol Biotechnol, 2003, 61(3): 179-188. |
| [11] | Martín JF, Aparicio JF. Enzymology of the polyenes pimaricin and candicidin biosynthesis. Methods Enzymol, 2009, 459: 215-242. |
| [12] | Tanaka A, Takano Y, Ohnishi Y, et al. AfsR recruits RNA polymerase to the afsS promoter: a model for transcriptional activation by SARPs. J Mol Biol, 2007, 369(2): 322-333. |
| [13] | Matsumoto A, Hong SK, Ishizuka H, et al. Phosphorylation of the AfsR protein involved in secondary metabolism in Streptomyces species by a eukaryotic-type protein kinase. Gene, 1994, 146(1): 47-56. |
| [14] | Sawai R, Suzuki A, Takano Y, et al. Phosphorylation of AfsR by multiple serine/threonine kinases in Streptomyces coelicolor A3(2). Gene, 2004, 334: 53-61. |
| [15] | Lee PC, Umeyama T, Horinouchi S. afsS is a target of AfsR, a transcriptional factor with ATPase activity that globally controls secondary metabolism in Streptomyces coelicolor A3(2). Mol Microbiol, 2002, 43(6): 1413-1430. |
| [16] | Chen L, Wang Y, Guo H, et al. High-throughput screening for Streptomyces antibiotic biosynthesis activators. Appl Environ Microbiol, 2012, 78(12): 4526-4528. |
| [17] | Kieser T, Bibb M, Buttner M, et al. Practical Streptomyces Genetics. Norwich: John Innes Foundation Press, 2000. |
| [18] | Bierman M, Logan R, O'Brien K, et al. Plasmid cloning vectors for the conjugal transfer of DNA from Escherichia coli to Streptomyces spp.. Gene, 1992, 116(1): 43-49. |
| [19] | Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method. Methods, 2001, 25(4): 402-408. |
| [20] | Horinouchi S, Hara O, Beppu T. Cloning of a pleiotropic gene that positively controls biosynthesis of A-factor, actinorhodin, and prodigiosin in Streptomyces coelicolor A3(2) and Streptomyces lividans. J Bacteriol, 1983, 155(3): 1238-1248. |
| [21] | Horinouchi S, Beppu T. Production in large quantities of actinorhodin and undecyl-prodigiosin induced by afsB in Streptomyces lividans. Agric Biol Chem, 1984, 48(8): 2131-2133. |
| [22] | Parajuli N, Viet HT, Ishida K, et al. Identification and characterization of the afsR homologue regulatory gene from Streptomyces peucetius ATCC 27952. Res Microbiol, 2005, 156(5): 707-712. |
| [23] | Horinouchi S. AfsR as an integrator of signals that are sensed by multiple serine/threonine kinases in Streptomyces coelicolor A3(2). J Ind Microbiol Biotechnol, 2003, 30(8): 462-467. |
 2015, Vol. 31
2015, Vol. 31




