服务
文章信息
- 张宇琪, 张洪斌, 甘微苇, 胡雪芹
- Zhang Yuqi, Zhang Hongbin, Gan Weiwei, Hu Xueqin
- 右旋糖酐酶研究进展
- Research progress in dextranase
- 生物工程学报, 2015, 31(5): 634-647
- Chin J Biotech, 2015, 31(5): 634-647
- 10.13345/j.cjb.140475
-
文章历史
- Received: October 2, 2014
- Accepted: December 1, 2014
右旋糖酐及其衍生物被广泛应用于医药、食品、化工等众多行业,市场需求巨大 (全球每年的需求近几百亿美金)。但是目前临床级右旋糖酐的生产方法多采用酸水解,不仅需要消耗大量的能源,而且由于含有大量的氯离子而严重影响产品质量。近年来国内外学者都在致力于研究一种低能耗、高品质的右旋糖酐生产方法,研究发现利用右旋糖酐酶能够水解右旋糖酐聚合物得到各种分子量的右旋糖酐[1]以及功能性低聚糖[2],因此许多学者都在研究右旋糖酐酶制备与催化性能,以提高右旋糖酐酶的酶活、稳定性与工业应用等。右旋糖酐酶催化降解高分子右旋糖酐,能够生产出适用于食品、化工、医药等领域的中、小分子量的多糖,酶催化反应条件温和、能耗低,从而能实现低分子量多糖生产工艺的绿色、环保、低碳目标,尤其有利于生物多糖在医药领域中的应用。本文结合各国学者对右旋糖酐酶的研究以及本实验室研究成果对其产生菌、酶学性质及工业应用等进行系统论述,并对当前有关该酶研究的热点和重点、国内右旋糖酐酶研究存在的问题以及未来的研究趋势提出了见解。
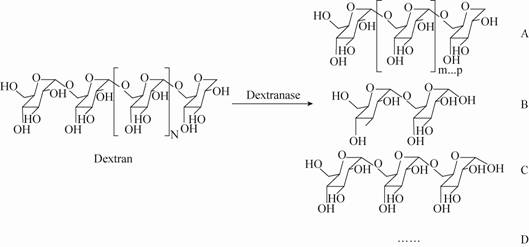
1 右旋糖酐酶的分类、产生菌以及酶分子结构 1.1 右旋糖酐酶的分类右旋糖酐酶属于糖苷水解酶[3],通常根据对右旋糖酐的水解方式不同将该酶分为内切右旋糖酐酶 (Endodextranase EC 3.2.1.11,α-1,6- glucan 6-glucanohydrolase;也被称作右旋糖酐酶) 和外切右旋糖酐酶 (Exodextranase EC 3.2.1.70,glucan 1,6-α-glycosidase;也被称作右旋糖酐葡萄糖苷酶)。外切右旋糖酐酶主要从右旋糖酐链的非还原端降解α-(1→6) 键,释放出葡萄糖。内切右旋糖酐酶主要水解右旋糖酐链内部的α- (1→6) 键,生成系列中低分子量多糖 (图1)。大多数内切右旋糖酐酶只有在多个连贯的α-(1→6) 葡萄糖苷键存在时才能发挥其催化作用[4],但也有不需要多个连贯的α-(1→6) 葡萄糖苷键存在即能发挥水解作用的内切右旋糖酐酶,例如Wynter等[5]报道的一株厌氧嗜热细菌-Rt364,它产生的右旋糖酐酶能水解淀粉中单一的α-(1→6) 葡萄糖苷键。外切右旋糖酐酶主要由肠道内的一些类杆菌属的细菌产生,而内切右旋糖酐酶的产生菌比较多,常见的有青霉菌[6]、芽胞杆菌和节杆菌。
|
| 图1 内切型右旋糖酐酶水解方式 Fig.1 The hydrolysis reaction of endodextranase. A: series of low molecular and middle molecular weight polysaccharide; B: isomaltose; C: isomaltotriose; D: other isomaltooligosacharide. |
另外,也有报道过右旋糖酐蔗糖酶、糊精右旋糖酐酶以及异麦芽糖右旋糖酐酶,其中右旋糖酐蔗糖酶 (EC 2.4.1.5,dextransucrase) 催化D-吡喃葡萄糖基从蔗糖转移到葡聚糖,能以蔗糖为底物合成右旋糖酐,并生成副产物果 糖[7],Robyt等[8]的研究证实了该过程中存在受体反应且符合两点嵌入机制;Yamamoto等[9]的研究显示能够产生糊精右旋糖酐酶 (EC 2.4.1.2,dextrin dextranase),该酶通过催化供体非还原性末端上的α-1,4-吡喃葡萄糖基转移到受体的非还原性末端上,形成α-1,6-糖苷键来生成高分子右旋糖酐;Tochihara等[10]的研究显示球形节杆菌能够产生异麦芽糖右旋糖酐酶 (EC 3.2.1.94,1,6-α-D-glucanisomaltohydrolase),该酶以外切方式从非还原端水解右旋糖酐,释放出连续的异麦芽糖单元,最适作用温度约65 ℃,最适pH约5.3,其催化方式是一种异头碳的保留机制[11]。迄今报道的右旋糖酐酶都属于诱导酶,产生菌的发酵液必须要有诱导物的存在才能保证右旋糖酐酶的形成,主要的诱导物有:右旋糖酐,改良的底物酮基右旋糖酐 (Ketodextran) 和改良的产物异麦芽糖棕榈酸单酯或二酯 (Isomaltosemo nopalmitate或Dipalmitate)。
近年来,由于国际生物化学和分子生物学联合会 (The International Union of Biochemistry and Molecular Biology,IUB-MB) 对糖苷酶基于酶底物特异性和催化反应类型分类具有一定的局限性,1991年Henrissat等将糖苷水解酶和糖基转移酶按家族分类并建立了CAZy (Carbohydrate active enzymes) 数据库[12],该法基于蛋白质结构域中的氨基酸序列和空间结构的相似性,结合酶分子的序列、结构与催化机制对其结构域进行定义,将内切右旋糖酐酶被划分到49和66家族,外切右旋糖酐酶归属于13和15家族[13]。
1.2 右旋糖酐酶产生菌右旋糖酐酶的产生菌种类繁多,如绳状青霉、点青霉、淡紫拟青霉、芽胞杆菌、链球菌、申克孢子丝菌、双歧杆菌、节杆菌等。总的来说,右旋糖酐酶产生菌大体可以分为真菌类、细菌类以及其他类目,表1列举了文献记载的不同来源的右旋糖酐酶产生菌及酶学性质。
| Type | Name of producing strains | Enzymatic properties | Reference sources | |
| Fungi | Penicillium | Penicillium funiculosum | 80 ℃,pH 6.0 | [14] |
| Penicillium notatum | 50 ℃,pH 5.0 | [15] | ||
| Isoenzyme with similar temperature (50 ℃) and pH (4.8) | [16] | |||
| Penicillium minioluteum | Mw 67 kDa pH 5.0 Isoelectric point 3.88 | [17] | ||
| Penicillium lilacinum | Mw 26.5 kDa 40-45 ℃ pH 5.0-5.8 | [18] | ||
| Penicillium aculeatum | Mw 66 kDa 35℃ pH 5.0 | [19] | ||
| Paecilomyces | Paecilomyces lilacinus | 50-55℃ pH 5.0-5.5 | [20] | |
| 65 ℃ pH 5.4 | [21] | |||
| Trichoderma | Trichoderma harzianum | Mw 62 kDa 25 ℃ pH 5.0 | [22, 23] | |
| Sporothrix | Sporothrix schenckii | Mw 79 kDa 30 ℃ pH 5.0 | [24] | |
| Chaetomium | Chaetomium erraticum | 30 ℃ pH 5.4 | [25] | |
| Aspergillus | Aspergillus ustus | 50-55 ℃ pH 5.0-5.5 | [20] | |
| Bacteria | Bacillus | Paenibacillus illinoisensis | IsoformⅠ76 kDa IsoformⅡ89 kDa Isoform Ⅲ 110 kDa 50 ℃ pH 6.8 | [26] |
| Paenibacillus sp. mutant | 60 ℃ pH 6.0 | [27] | ||
| Bacillus licheniformis | Non-pathogenic | [28] | ||
| Streptococcus | Streptococcus rattus | Mw 100 kDa 40 ℃ pH 5.0 | [29] | |
| Catenovulum | Catenovulum sp. | 40 ℃ pH 8.0 | [30] | |
| Others | Anaerobic thermophilic bacterium-Rt364 | Mw 140 kDa 80 ℃ pH 5.5 | [5] | |
| Altermonas espejiana YSN412 | 30 ℃ pH 8.0 | [31] | ||
| Actinomycetes | Streptomyces | Streptomyces sp. NK458 | Mw 410 kDa 60 ℃ pH 9.0 | [32] |
| Arthrobacter | Arthrobacter oxydans KQ11 | Mw 66.2 kDa 50 ℃ pH 7.0 | [33] | |
| Arthrobacter sp. | Mw 64 kDa 45 ℃ pH 5.5 | [34] | ||
由表1可以看出,右旋糖酐酶产生菌多数来源于真菌,特别是青霉菌,其次来源较广泛的为细菌,而放线菌来源以节杆菌属为主。2012年Purushe等[32]报道出一株放线菌来源的右旋糖酐酶产生菌,所产右旋糖酐酶同时具有嗜碱 (最适pH 9.0) 与嗜热 (最适温度60 ℃) 的特性,对于制糖业右旋糖酐污染的消除具有很好的效果,但是其热稳定性有待于进一步提高。细菌来源的右旋糖酐酶普遍具有良好的耐热性,最适温度多在40-60 ℃,最适pH约为5.0-7.0,但是酶活普遍较低,需要通过诱变筛选或者基因克隆的手段获取高活力的右旋糖酐酶[27, 28, 35]。从总体上分析,真菌来源的右旋糖酐酶最适温度多为45-70 ℃,pH约为5.0-5.5,活力较高,稳定性较好,因此从真菌中筛选得到高活力、高稳定性的右旋糖酐酶对于该酶的工业应用具有很高的价值。
本实验室近两年已经成功从土壤中筛选出若干株右旋糖酐酶产生菌,并完成了其中两株的分离纯化及酶学性质研究[19, 22, 23],一株为棘孢青霉Penicillium aculeatum,GenBank登录号HQ647326,所产右旋糖酐酶与本实验室构建表达的基因工程菌Escherichia coli BL21产生的右旋糖酐蔗糖酶进行协同催化反应,可制得不同分子量的低聚右旋糖酐,通过结构分析推断其产物具有很高的临床应用价值且可用于制备右旋糖酐衍生物[36];另外一株为哈茨木霉Trichoderma harzianum,GenBank登录号HQ647325,所产右旋糖酐酶具有优先降解高分子量右旋糖酐的特性,非常有利于药用右旋糖酐 (70 kDa & 40 kDa & 20 kDa) 的合成,且与上述重组右旋糖酐蔗糖酶联合作用可合成出功能性糖分含量高、益生元特性好的低聚糖[37]。
1.3 右旋糖酐酶的结构由表1可知,不同来源的右旋糖酐酶的分子量总体偏大 (低至26.5 kDa,高达410 kDa),使得这种大分子酶蛋白的结构鉴定显得非常困难,因而关于右旋糖酐酶的分子结构及催化机理的研究成为该领域的一个难点。据文献记载,口腔链球菌右旋糖酐酶的分子结构主要由5部分组成[38]:N-末端信号肽 (Sp)、C-末端膜结合区域 (MAR)、一个不变区 (CR) 和两个可变区 (VR)。不变区约位于整个肽链的中间位置,其两侧是两个可变区。右旋糖酐酶的不变区具有高度同源性,约由540个氨基酸残基组成,同源性49%-90%。不变区含有两个功能结构域:催化部位 (Catalytic site) 和葡聚糖结合部位 (Dextran-binding domain)。
截止到2014年8月,CAZy数据库已收录25种内切右旋糖酐酶和5种外切右旋糖酐酶,其中3种右旋糖酐酶的结构已经获得解析,酶蛋白的分子信息如表2所示。
| GH family | Molecule | Organism | Gene name | PDB ID | Length (aa) | Structure weight (Da) | Mechanism | References |
| 49 | Dextranase | Talaromyces minioluteus | dex | P48845 | 574 | 62 445.60 | Inverting | [13] |
| 15 | Glucodextranase | Arthrobacter globiformis | g1d | Q9LBQ9 | 1 020 | 106 860.57 | Inverting | [35] |
| 66 | Dextranase | Streptococcus mutans | dexA SMU_2042 | Q54443 | 643 | 72 692.07 | Retaining | [36] |
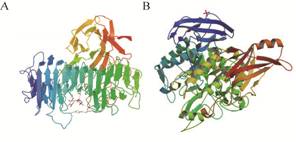
2003年,Larsson等[17]报道了一种朱黄青霉右旋糖酐酶的晶体结构以及催化机理。如图2所示,该酶也主要含有两个结构域:一个右手平行的β螺旋结构与N末端的β夹层结构域;通过核磁共振波谱追踪分析,该酶的反应过程伴随着异头碳的反转,意味着存在一个单一的置换机制[13]。在酶分子的表面有一个键合异麦芽糖的缝隙,构成酶分子与产物的复合物,葡萄糖单元的糖苷氧在+1亚位点处与Asp395形成一个氢键,Asp376和Asp396两个氨基酸残基被用于活化水分子,该水分子被用于亲和攻击右旋糖酐α-1,6-糖苷键上的异头碳,而酸提供的质子用于糖苷键的质子化,两者在聚合物链的同侧同时进行,最终实现α-糖苷键的断裂,使得右旋糖酐被降解、分子量降低、粘度下降。
2012年,Suzuki等[40]报道了一种变形链球菌右旋糖酐酶的晶体结构及催化机理,如图2所示,该酶由至少5个氨基酸序列区域组成,其不变区含有3个结构域:具有一个免疫球蛋白样的β-夹层结构域N;含有一个 (β/α) 8-桶状结构的结构域A为该酶的催化部位;包含两个希腊钥匙图案的β夹层结构域C。通过分析酶-产物复合结构发现:Asp385是催化反应的亲核试剂,Glu453是双置换机制的酸/碱,且该酶表现出保留型催化性质。这是66家族糖苷水解酶中首个阐明结构、酶促反应机理及底物识别位点的报道。此外,近年来利用定点诱变技术还发现Asp243对栖热袍菌TMO右旋糖酐酶的催化反应也是必需的[35]。
2 右旋糖酐酶编码基因的进化分析右旋糖酐酶的编码基因主要来自于真菌和细菌。其中,真菌来源的基因主要来自青霉菌,细菌来源的种类比较多,主要有大鼠链球菌、变形链球菌、唾液链球菌和类芽胞杆菌等。表3列举了已经报道的不同菌株来源的右旋糖酐酶基因。
| Original strain | Gene | Expression host | Gene size | MW of recombinase (kDa) | GenBank Accession No. |
| Penicillium minioluteum HI-4[30] | dex | Escherichia coli T7 | 2 109 bp 608 aa | 67.0 | L41562 |
| Penicillium minioluteum IMI068219[42] | dex1 | Saccharomyces cerevisiae pYES2/CT | 1 821 bp | 63.0 | DQ394070 |
| Streptococcus sobrinus UAB66[11] | dex | Escherichia coli 2831 | 3 999 bp 1 337 aa | 80.0-130.0 | M96978 |
| Streptococcus mutans[43] | dexA | Escherichia coli | 2 553 bp 850 aa | 94.5 | BAA08409 |
| Streptococcus suis R735[44] | dexS | Phage λZapⅡ | 2 417 bp | 62.0 | U35633 |
| Arthrobacter oxydans[45] | dex2 | Escherichia coli | 1 882 bp 620 aa | 137.0 | DQ497801 |
| Paenibacillus strains[46] | Dex40-8 dex1 | Escherichia coli TOP10F | 716 aa | 80.8 | AY326309 |
| Dex50-2 dex1 | 905 aa | 100.1 | AY326310 | ||
| Dex50-2 dex2 | 596 aa | 68.3 | AY326311 | ||
| Lipomyces starkey strain[47, 48] | dex | Pichia pastoris GS115 | 1 824 bp 608 aa | 70.0 | AY280636 |
| Saccharomyces cerevisiae | 67.6 |
由表3可知,学者们已从大量的右旋糖酐酶天然菌株中克隆得到相应的编码基因,重组酶的分子量由62 kDa到128.1 kDa不等。图3为青霉菌、类芽胞杆菌及链球菌右旋糖酐酶基因的系统发育树。通过比对分析发现,青霉菌的dex与dex1同源性高达89.18%;类芽胞杆菌的Dex70-1B和Dex70-34同源性为59.84%;Dex50-2 dex2与Dex70-1B、Dex70-34比对同源性分别为48.41%和46.21%;Dex40-8 dex1与Dex50-2 dex1比对的同源性为65.33%;Dex40-8 dex1与Dex70-1B比对的同源性为40.31%;Dex50-2 dex1与Dex70-34比对同源性仅为32.57%。变形链球菌dexA与鼠链球菌dex同源性为66.85%;而唾液链球菌dex与变形链球菌dexA、鼠链球菌dex同源性分别为53.98%和52.57%;仓鼠链球菌dex与汗毛链球菌dex同源性为66.83%。

|
| 图3 青霉菌、类芽孢杆菌及链球菌右旋糖酐酶基因的系统发育树 Fig.3 The phylogram of Penicillium,Paenibacillus & Streptococcus dextranase gene. The phylogram was constructed using a Clustal W alignment package (European Bioinformatics Institute,http://www.ebi.ac.uk). Branch lengths and associated numerals indicate the fraction of positions that differ between amino acid sequences. |
总体来讲,真菌来源的右旋糖酐酶编码基因的研究相对较少;链球菌来源的右旋糖酐酶基因同源性较高;其次为类芽胞杆菌,而来自不同类芽胞杆菌的基因同源性相对较低;并且来自不同菌属的右旋糖酐酶基因同源性相差较大。本实验室在基因克隆方面也进行了深入研究,2008年成功对右旋糖酐蔗糖酶基因进行了克隆和测序并成功表达[49, 50],其GenBank登录号为DQ345760;目前正在进行右旋糖酐酶的基因克隆研究。当然,国际上关于右旋糖酐酶的基因克隆研究还处在发展阶段;而国内有关右旋糖酐酶基因克隆的研究主要集中于朱黄青 霉[51]、细丽毛壳菌[52]和斯氏油脂酵母来源的右旋糖酐酶,因此仍然有很多未知的右旋糖酐酶编码基因有待于研究人员的深入研究。
3 右旋糖酐酶的应用右旋糖酐的分子质量对其性质和应用的影响很大,许多学者对于生物合成高分子右旋糖酐及酶解法制备不同分子量右旋糖酐的工艺进行了研究,尤其是膜工艺、基因克隆技术以及固定化技术的发展,为右旋糖酐的生产提供了新的理论和技术支持。Kim等[53]研究了右旋糖酐蔗糖酶浓度、pH、温度对产物右旋糖酐分子量及结构的影响;Falconer等[54]进一步揭示了一定条件下右旋糖酐分子量与体系中的酶含量、蔗糖浓度及发酵温度的关系;Kubik等[55]联合使用右旋糖酐酶和右旋糖酐蔗糖酶,通过控制二者比例可获得不同分子量的右旋糖酐或低聚糖等产物。近年来,国内学者也对于制备右旋糖酐及其分子量调控等研究进行了探索。姚日生等[56]利用固定化技术,探讨了酶法制备右旋糖酐并有效控制其相对分子质量的工艺。许朝芳等[57]研究了酶解法制备特微分子右旋糖酐的工艺;常国炜等[58]对右旋糖酐酸水解和酶水解产物的成分和分子量分布进行了比较研究,对酶解法取代酸解法调控右旋糖酐分子量具有指导意义。本实验室利用右旋糖酐蔗糖酶和右旋糖酐酶先后成功制备出不同分子量的右旋糖酐 (MW 70 kDa、40 kDa、20 kDa、10 kDa、7 kDa、5 kDa) 以及聚合度为2-15的低聚糖,为酶法生产右旋糖酐及低聚糖的工业化奠定了坚实的基础[22, 36, 37, 59],并对低聚右旋糖酐的结构进行了初步探索。
在医药工业领域,右旋糖酐酶制备的药用右旋糖酐 (70 kDa & 40 kDa & 20 kDa) 具有补充血容量和改善微循环的作用,小分子右旋糖酐 (10 kDa) 具有渗透性利尿作用。右旋糖酐酶用于制备葡聚糖凝胶药物缓释体系[60],可以增强药物化学稳定性和生物利用度,具有延长药效、减少毒性、增强靶向性及降低免疫原性等作用,已知可载运的有抗肿瘤药、抗生素、抗炎药和蛋白质类药物等[61]。分子量6-8 kDa的右旋糖酐常被用于制成右旋糖酐铁,用于治疗严重的贫血症。微分子量 (3-5 kDa) 的硫酸葡聚糖则具有抗凝血特性和抗病毒特性,具有发展成为抗HIV药物的潜力。右旋糖酐1 (Dextran 1,MW 1 kDa) 已于1984年在英国上市,主要用于抗血栓,同时可以大大降低中分子右旋糖酐诱发人体变态反应 (Dextrain-induced anaphyactic reations,DIAR) 的发生。此外,研究发现细菌来源的右旋糖酐酶对于龋齿的防 治[62]、开发龋齿疫苗具有重要意义,近年来已引起很多学者的关注。
在食品工业中,右旋糖酐酶深度催化水解高分子右旋糖酐制备的功能性低聚异麦芽 糖[2, 63]具有益生元特性。右旋糖酐酶制备的右旋糖酐可用作生物保鲜剂,对于我国绿色农业的发展有着深远的意义。经右旋糖酐酶不同程度水解的葡聚糖用作食品添加剂[64],可以提高食品的柔软度,增大食品的体积。在制糖工业[65]中,右旋糖酐酶能减少甚至避免葡聚糖带来的经济问题,改善糖产品的滤过性、设备平滑性、增加糖的回收率,降低黏度。
在化工领域,右旋糖酐酶可用于制备分离大分子物质的交联葡聚糖凝胶等色谱填料[66]、乳化剂和增稠剂、高粘度胶、成像试剂、土壤改良剂[67]以及油井钻泥添加剂[68]等,同时对阐明右旋糖酐以及其他多糖的结构亦具有重要 作用[69]。
总之,右旋糖酐酶在医药、食品、化工工业的运用十分广泛,随着各种新技术、新产品的出现,右旋糖酐酶的应用定会带来大的经济和社会效益。
4 结语根据文献记载,近年来国内外对右旋糖酐酶的研究逐渐增多,且引起了各行业更为广泛的关注,其研究方向涉及酶学、药理学、化学、遗传学、微生物学、免疫学等多个学科,研究重点主要围绕该酶的产生菌、分离纯化及其酶学性质、基因的克隆表达、固定化技术等。目前已有各种来源的60余种右旋糖酐酶被分离并得到纯化。在右旋糖酐酶的众多产生菌中,真菌尤其是青霉菌产生的右旋糖酐酶的酶活、温度稳定性、pH稳定性都较好,具有很高的工业应用价值。但就工业生产而言,来源于真菌的右旋糖酐酶制备周期长、能耗大,并且单纯的游离酶反应使得产品的纯化困难,同时存在安全问题[33]。因此右旋糖酐酶的固定化技术以及基因克隆技术[70, 71]显得尤为重要,日益成为该领域的研究热点,大大增加了该酶工业化运用的可能性。
然而,目前国内关于右旋糖酐酶的研究多集中在产生菌的筛选及分离纯化阶段,对酶的构效关系、固定化以及基因克隆研究还有很长一段距离。酶的构效关系研究需要解析出右旋糖酐酶以及该酶与底物复合物的结构,结合酶分子的序列、结构与催化机制对其结构域进行定义,确定相关基因功能结构域的组合方式[13],进一步认识右旋糖酐酶的作用机制可以大大提高功能预测的准确度和理性设计的成功率,对其分子改造的进一步应用提供了有力的技术支持。利用该酶分析多糖结构,有利于研究以及开发生物多糖的特性,使其在多个领域的应用得到更为广阔的发展。酶的固定化技术需要改善与克服的问题也有很多,如固定化酶的渗漏、重复使用批次及催化效率等,基因克隆研究也需要进一步深入以阐明右旋糖酐酶各家族之间的复杂关联,从而方便人们更好地开发右旋糖酐酶的商业价值。
近年来右旋糖酐酶在生物合成多糖及低聚糖、食品加工、防治龋齿、药物控释体系等多个方面的应用引起了更多学者的深入研究,为实现其工业应用价值奠定了坚实的基础。相信在不久的将来,固定化技术和基因工程技术的继续发展将会生产出性能良好的新酶,以满足医药、化工、食品工业的不同需求。
| [1] | Goulas AK, Fisher DA, Grimble GK, et al. Synthesis of isomaltooligosaccharides and oligodextrans by the combined use of dextransucrase and dextranase. Enzyme Microb Technol, 2004, 35(4): 327-338. |
| [2] | Aslana Y, Tanrisevenb A. Immobilization of Penicillium lilacinum dextranase to produce isomaltooligo saccharides from dextran. Biochem Eng J, 2007, 34(1): 8-12. |
| [3] | Coutinho P, Henrissat B. Carbohydrate-active enzymes database [EB/OL]. [2014-08-20]. http:// afmb.cnrs-mrs.fr/cazy/CAZY/index.html. |
| [4] | Walker GJ, Pulkownik A. Action of alpha-1,6- glucan glucohydrolase on oligosaccharides derived from dextran. Carbohyd Res, 1974, 36(1): 53-66. |
| [5] | Cynter CVA, Hang M, De Jersey J, et al. Isolation and characterization of a thermostable dextranase. Enzyme Microb Tech, 1997, 20(4): 242-247. |
| [6] | Bailey RW, Clarke RT. A bacterial dextranase. Biochem J, 1959, 72(1): 49-54. |
| [7] | Robyt JF. Dextran. Ency Polym Sci, 1986, 4(2): 753−767. |
| [8] | Robyt JF, Yoon SH, Mukerjea R. Dextransucrase and the mechanism for dextran biosynthesis. Carbohyd Res, 2008, 343(18): 3039-3048. |
| [9] | Yamamoto K, Yoshikawa K, Okada S. Effective production of glycosyl-steviosides by α-1,6 transglucosylation of dextrin dextranase. Biosci Biotechnol Biochem, 1994, 58(9): 1657-1661. |
| [10] | Tochihara T, Sasak K, Araki O, et al. Site-directed mutagenesis establishes aspartic acids-227 and -342 as essential for enzyme activity in an isomalto-dextranase from Arthrobacter globiformis. Biotechnol Lett, 2004, 26(8): 659-664. |
| [11] | Khalikova E, Susi P, Korpela T. Microbial dextran-hydrolyzing enzymes: fundamentals and applications. Microbiol Mol Biol Rev, 2005, 69(2): 306-325. |
| [12] | Henrissat B. A classification of glycosyl hydrolases based on amino acid sequences similarities. Biochem J, 1991, 280(2): 309-316. |
| [13] |
Wang S, Chen GJ, Zhang HQ, et al. Carbohydrate- active enzyme (CAZy) database and its new prospect. Chin J Bioproc Eng, 2014, 12(1): 102−108 (in Chinese). 王帅, 陈冠军, 张怀强, 等. 碳水化合物活性酶数据库(CAZy)及其研究趋势. 生物加工过程, 2014, 12(1): 102−108. |
| [14] | Abdel-Naby MA, Ismail Abdel-Mohsen S, Abdel-Fattah AM, et al. Preparation and some properties of immobilized Penicillium funiculosum 258 dextranase. Process Biochem, 1999, 34(4): 391-398. |
| [15] | Szczodrak J, Pleszczynska M, Fiedurek J. Penici llium notatum 1 a new source of dextranase. J Ind Microbio, 1994, 13(5): 315-320. |
| [16] | Pleszczynska M, Rogalski J, Szczodrak J, et al. Purification and some properties of an extracellular dextranase from Penicillium notatum. Mycol Res, 1996, 100(6): 681-686. |
| [17] | Larsson AM, Andersson R, Stahlberg J, et al. Dextranase from Penicillium minioluteum: reaction course, crystal structure, and product complex. Struct, 2003, 11(9): 1111-1121. |
| [18] | Das DK, Dutta SK. Purification, biochemical characterization and mode of action of an extra cellular endo-dextranase from the culture filtrate of Penicillium lilacinum. Int J Biochem Cell Biol, 1996, 28(1): 107-113. |
| [19] |
Zhang HB, Wu DT, Huang LJ, et al. Purification, characterization of an extracellular dextranase from an isolated Penicillium sp.. Acta Microbiol Sin, 2011, 51(4): 495-503 (in Chinese). 张洪斌, 吴定涛, 黄丽君, 等. 一株产右旋糖酐酶青霉的分离及酶的纯化和性质. 微生物学报, 2011, 51(4): 495-503. |
| [20] |
Sun JW, Cheng XL, Yan ZZ, et al. Screening of dextranase-producing strains and comparative study on enzymatic properties. Acta Microbiol Sin, 1988, 28(1): 45-55 (in Chinese). 孙晋武, 程秀兰, 严自正, 等. 右旋糖酐酶产生菌的筛选及其酶学性质的比较研究. 微生物学报, 1988, 28(1): 45-55. |
| [21] | Galvez-Mariscal A, Lopez-Munguia A. Production and characterization of a dextranase from an isolated Paecilomyces lilacinus strain. Appl Microbiol Biot, 1991, 36(3): 327-331. |
| [22] | Wu DT, Zhang HB, Huang LJ, et al. Purification and characterization of extracellular dextranase from a novel producer, Hypocrea lixii F1002, and its use in oligodextran production. Process Biochem, 2011, 46(10): 1942-1950. |
| [23] |
Zhang HB, Wu DT, Huang LJ, et al. Fermentation method for producing extracellular dextranase with Hypocrea lixii: CN, 102220244A. 2011-10-19 (in Chinese). 张洪斌, 吴定涛, 黄丽君, 等. 一种哈茨木霉菌株及其用该菌株制备右旋糖酐酶的方法: 中国, 102220244A. 2011-10-19. |
| [24] | Arnold WN, Nguyen TB, Mann LC, et al. Purification and characterization of a dextranase from Sporothrix schenckii. Arch Microbiol, 1998, 170(2): 91-98. |
| [25] | Erhardt FA, Jordening Hans-Joachim. Immobilization of dextranase from Chaetomium erraticum. J Biotechnol, 2007, 131(4): 440-447. |
| [26] | Khalikova E, Susi P, Usanov N, et al. Purification and properties of extracellular dextranase from a Bacillus sp. J Chromatogr B, 2003, 796(2): 315-326. |
| [27] | Hild E, Brumbley SM, O'Shea MG, et al. A Paenibacillus sp. dextranase mutant pool with improved thermostability and activity. Appl Microbiol Biotechnol, 2007, 75(5): 1071-1078. |
| [28] | Zohra RR, Aman A, Zohra RR, et al. Dextranase: hyper production of dextran degrading enzyme from newly isolated strain of Bacillus licheniformis. Carbohyd Polym, 2013, 92(2): 2149-2153. |
| [29] | Igarashi T, Morisaki H, Goto N. Molecular characterization of dextranase from Streptococcus rattus. Microbiol Immunol, 2004, 48(3): 155-162. |
| [30] | Cai RH, Lu MS, Fang YW, et al. Screening, production, and characterization of dextranase from Catenovulum sp. Ann Microbiol, 2014, 64(1): 147-155. |
| [31] |
Jiao YL, Wang SJ, Lü MS, et al. Screening, identification and activity analysis of a dextranase-producing Alteromonas espejiana strain. Sci Tech Food Ind, 2013, 34(2): 171-178 (in Chinese). 焦豫良, 王淑军, 吕明生, 等. 产右旋糖酐酶埃氏交替单胞菌的筛选、鉴定及酶活分析. 食品工业科技, 2013, 34(2): 171-178. |
| [32] | Purushe S, Prakash D, Nawani NN, et al. Biocatalytic potential of an alkalophilic and thermophilic dextranase as a remedial measure for dextran removal during sugar manufacture. Bioresour Technol, 2012, 115: 2-7. |
| [33] | Wang DL, Lu MS, Wang SJ, et al. Purification and characterization of a novel marine Arthrobacter oxydans KQ11 dextranase. Carbohyd Polym, 2014, 106: 71-76. |
| [34] | Jiao YL, Wang SJ, Lu MS, et al. Characterization of a marine-derived dextranase and its application to the prevention of dental caries. J Ind Microbiol Biotechnol, 2014, 41(1): 17-26. |
| [35] | Kim YM, Kim DM. Characterization of novel thermostable dextranase from Thermotoga lettingae TMO. Appl Microbiol Biotechnol, 2010, 85(3): 581-587. |
| [36] | Gan WW, Zhang HB, Zhang YQ, et al. Biosyn thesis of oligodextrans with different Mw by synergistic catalysis of dextransucrase and dextranase. Carbohyd Polym, 2014, 112: 387-395. |
| [37] | Zhang HB, Gan WW, Zhang YQ, et al. Synthesis of isomalto-oligosaccharides by using recombinant dextransucrase and Hypocrea lixii dextranase. J Chem Pharm Res, 2013, 5(11): 49-57. |
| [38] |
Jiang D, Qiu YX, Hu T, et al. Dextranase of oral Streptococci: molecular structure and function. Int J Stomatol, 2008, 35(3): 249-251 (in Chinese). 蒋丹, 仇元新, 胡涛, 等. 口腔链球菌右旋糖酐酶分子结构和功能的研究进展. 国际口腔医学杂志, 2008, 35(3): 249-251. |
| [39] | Mizuno M, Tonozuka T, Suzuki S, et al. Structural insights into substrate specificity and function of glucodextranase. J Biol Chem, 2004, 279(11): 10575-10583. |
| [40] | Suzuki N, Kim YM, Fujimoto Z, et al. Structural elucidation of dextran degradation mechanism by Streptococcus mutans dextranase belonging to glycoside hydrolase family 66. J Biol Chem, 2012, 287(24): 19916-19926. |
| [41] | RCSB protein data bank-RCSB PDB-3VMN structure summary [EB/OL]. [2014-08-20]. http://www.rcsb.org/pdb/explore/explore.do?structureId=3VMN. |
| [42] | Li XM, Millson SH, Coker RD, et al. Cloning and expression of Penicillium minioluteum dextranase in Saccharomyces cerevisiae and its exploitation as a reporter in the detection of mycotoxins. Biotechnol Lett, 2006, 28(23): 1955-1964. |
| [43] | Igarashi T, Yamamoto A, Goto N. Sequence analysis of the Streptococcus mutans ingbritt dexA gene encoding extracellular dextranase. Microbiol Immunol, 1995, 39(11): 853-860. |
| [44] | Serhir B, Dugourd D, Jacques Mario, et al. Cloning and characterization of a dextranase gene (dexS) from Streptococcus suis. Gene, 1997, 190(2): 257-261. |
| [45] | Lee JH, Nam SH, Park HJ, et al. Biochemical characterization of dextranase from Arthrobacter oxydans and its cloning and expression in Escherichia coli. Food Sci Biotechnol, 2010, 19(3): 757-762. |
| [46] | Finnegan PM, Brumbley SM, O'Shea MG, et al. Diverse dextranase genes from Paenibacillus species. Arch Microbiol, 2005, 183(2): 140-147. |
| [47] | Kang HK, Kim SH, Park JY, et al. Cloning and characterization of a dextranase gene from Lipomyces starkeyi and its expression in Saccharomyces cerevisiae. Yeast, 2005, 22(15): 1239-1248. |
| [48] | Chen L, Zhou XS, Fan WM, et al. Expression, purification and characterization of a recombinant Lipomyces starkey dextranase in Pichia pastoris. Protein Expres Purif, 2008, 58(1): 87-93. |
| [49] | Zhang HB, Mao XQ, Wang YJ, et al. Optimization of culture conditions for high-level expression of dextransucrase in Escherichia coli. J Food Agric Environ, 2009, 7(3/4): 75-78. |
| [50] |
Zhang HB, Zhu CB, Hu YJ, et al. Construction and culture conditions of dextransucrase-secreting engineered strain. Acta Microbiol Sin, 2008, 48(4): 492-497 (in Chinese). 张洪斌, 朱春宝, 胡又佳, 等. 右旋糖酐蔗糖酶工程菌株的构建及其培养条件的研究. 微生物学报, 2008, 48(4): 492-497. |
| [51] |
Wang Y, Zhang XQ, Wang FQ. Heteroexpression of α-1,6-dextranase from Penicillium minioluteum in Pichia pastoris. Food Drug, 2014, 16(2): 77-80 (in Chinese). 王赟, 章晓庆, 王风清. 朱黄青霉α-1,6-葡聚糖酶在毕赤酵母GS115中的异源表达. 食品与药品, 2014, 16(2): 77-80. |
| [52] |
Wang Y. Cloning and heteroexpression of α-dextranase from Chaetomiun gracile[D]. Shanghai: East China University of Science and Technology, 2012 (in Chinese). 王赟. 细丽毛壳α-葡聚糖酶的基因克隆及异源表达[D]. 上海: 华东理工大学, 2012. |
| [53] | Kim D, Robyt JF, Lee So-Young, et al. Dextran molecular size and degree of branching as a function of sucrose concentration, pH, and temperature of reaction of Leuconostoc mesenteroides B-512FMCM dextransucrase. Carbohyd Res, 2003, 338(11): 1183-1189. |
| [54] | Falconer DJ, Mukerjea R, Robyt JF. Biosynthesis of dextrans with different molecular weights by selecting the concentration of Leuconostoc mesenteroides B-512FMC dextransucrase, the sucrose concentration, and the temperature. Carbohyd Res, 2011, 346(2): 280-284. |
| [55] | Kubik C, Sikora B, Bielecki S. Immobilization of dextransucrase and its use with soluble dextranase for glucooligosaccharides synthesis. Enzyme Microb Tech, 2004, 34(6): 555-560. |
| [56] |
Yao RS, Dong MH, Zhu HX. Control of molecular weight of dextran produced by enzymatic synthesis. Fine Chem Icals, 2007, 24(10): 964-967 (in Chinese). 姚日生, 董明辉, 朱慧霞. 酶法合成右旋糖酐相对分子质量的控制. 精细化工, 2007, 24(10): 964-967. |
| [57] |
Xu CF, Zhang LJ, Ruan H, et al. Preoaration of super micro-moecular dextran by degradation of small molecular dextran with dextran enzyme. Technol Develop Chem Ind, 2012, 41(12): 10-12 (in Chinese). 许朝芳, 张丽娟, 阮恒, 等. 右旋糖酐酶降解小分子右旋糖酐制备特微分子右旋糖酐. 化工技术与开发, 2012, 41(12): 10-12. |
| [58] |
Chang GW, Lin RZ, Zeng LQ, et al. Comparison of dextran hydrolyzates between acidic and enzymatic hydrolysis. Sugarcane Canesugar, 2012, 12(6): 33-38 (in Chinese). 常国炜, 林荣珍, 曾练强, 等. 右旋糖酐酸解与酶解产物比较. 甘蔗糖业, 2012, 12(6): 33-38. |
| [59] |
Zhang HB, Gan WW, Wu DT, et al. Double-enzyme method for preparing medical dextran with controlled molecular weight: CN, 102676615A. 2012-09-19 (in Chinese). 张洪斌, 甘微苇, 吴定涛, 等. 双酶法制备分子量可控的药用右旋糖酐: 中国, 102676615A. 2012-09-19. |
| [60] |
Li LC, Li MY. Recent Biochemical Drugs Preparation Technology. Beijing: China Medical Science Press, 2003: 299−301 (in Chinese). 李良铸, 李明晔. 最近生化药物制备技术. 北京:中国医药科技出版社, 2003: 299−301. |
| [61] |
Chen FM, Wu ZF, Jin Y, et al. Preparation of enamel matrix proteins controlled release micro spheres and their biological effects on the proli feration and differentiation of human periodontal ligament cells in vitro. West Chin J Stomatol, 2005, 23(6): 529-533 (in Chinese). 陈发明, 吴只芬, 金岩, 等. 釉基质蛋白控释微球的研制及其生物学性能的初步研究. 华西口腔医学杂志, 2005, 23(6): 529-533. |
| [62] | Staat RH, Schachtele CF. Characterization of a dextranase produced by an oral strain of Actinomyces israelii. Inject Immun, 1975, 12(3): 556-563. |
| [63] | Oelcer Z, Tanriseven A. Co-immobilization of dextransucrase and dextranase in alginate. Process Biochem, 2010, 45(10): 1645-1651. |
| [64] |
National Health Ministry of the People's Republic of China. Approval announcement about new varieties of five kinds of food additives such as trisodium monohydrogen diphosphate (National Health Ministry, 15th 2012) [EB/OL]. [2014-08- 20]. http://www.nhfpc.gov.cn/sps/s7891/201208/ 799b8bac60e64fb39ce72f995c3ed03f.shtml (in Chinese). 中华人民共和国卫生部. 关于批准焦磷酸一氢三钠等5种食品添加剂新品种的公告 (卫生部公告2012年第15号) [EB/OL]. [2014-08-20]. http:// www.nhfpc.gov.cn/sps/s7891/201208/799b8bac60e64fb39ce72f995c3ed03f.shtml. |
| [65] | Cuddihy JA, Rauh JS, Mendez F, et al. Dextranase in Sugar Production: Factory Experience. 29th Annual Joint Meeting American Society of Sugar Cane Technologists, 2000: 95-103. |
| [66] | Porath J. From gel filtration to adsorptive size exclusion. J Protein Chem, 1997, 16(5): 463-468. |
| [67] | Alsop RM. Industrial production of dextrans. Prog Ind Microbiol Microbial Polysaccharides, 1983, 18: 1−44. |
| [68] |
Wang XY. The development of petroleum microbiology in Institute of Microbiology, Chinese Academy of Sciences. Microbiol China, 2008, 35(12): 1851-1861 (in Chinese). 王修垣. 石油微生物学在中国科学院微生物研究所的发展. 微生物学通报, 2008, 35(12): 1851-1861. |
| [69] | Hayacibara MF, Koo H, Vacca-Smith AM, et al. The influence of mutanase and dextranase on the production and structure of glucans synthesized by streptococcal glucosyltransferases. Carbohyd Res, 2004, 339(12): 2127–2137. |
| [70] | Kim YM, Seo MY, Kang HK, et al. Construction of a fusion enzyme of dextransucrase and dextranase: application for one-step synthesis of isomalto-oligosaccharides. Enzyme Microb Technol, 2009, 44(3): 159-164. |
| [71] | Ryu HJ, Jin XJ, Lee JH, et al. Optimal expression and characterization of a fusion enzyme having dextransucrase and dextranase activities. Enzyme Microb Technol, 2010, 47(5): 212-215. |
 2015, Vol. 31
2015, Vol. 31