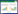服务
文章信息
- 袁家代, 陈贵英, 程世君, 葛方兰, 王琼, 李维, 李江
- Yuan Jiadai, Chen Guiying, Cheng Shijun, Ge Fanglan, Qiong Wang, Li Wei, Li Jiang
- 3-甾酮-9α-羟基化酶基因在分枝杆菌中的异源表达与9α-羟基雄烯二酮的制备
- Accumulation of 9α-hydroxy-4-androstene-3,17-dione by co-expressing kshA and kshB encoding component of 3-ketosteroid-9α-hydroxylase in Mycobacterium sp. NRRL B-3805
- 生物工程学报, 2015, 31(4): 523-533
- Chin J Biotech, 2015, 31(4): 523-533
- 10.13345/j.cjb.140411
-
文章历史
- Received: August 12, 2014
- Accepted: November 15, 2014
甾醇化合物又称固醇,为细胞膜的重要组成成分,广泛存在于动植物体内,它们种类繁多,资源极为丰富,但多作为工农业生产中的副产物而被废弃,未能得到有效利用。从上世纪50年代以来,微生物降解植物甾醇和胆固醇而制备甾体药物重要中间体的研究逐渐受到人们广泛关注,微生物转化被认为是取代传统工业中化学分解方法生产甾体类药物的重要途径[1]。研究表明,甾醇化合物经微生物细胞吸收进入胞内,通过类似β-氧化的循环途径降解甾醇的侧链,获得雄烯二酮 (AD),后者经过各种转化修饰,可以进一步合成睾酮、雌二醇、炔雌醇、睾酮内酯、可的松、皮质醇、泼尼松等多种重要的类固醇衍生物。但多数甾醇降解菌株如马红球菌、诺卡氏菌等能彻底分解甾醇化合物而无法积累上述甾体药物中间体。
3-甾酮-9α-羟基化酶 (KSH) 是甾体微生物转化中的一个关键酶[2],它可在AD(D) 多元环的第9位引入一个羟基 (9α-OH),将AD(D) 转化为9-OH-AD(D)[3]。9-OH-AD在化学结构上由于9α-羟基的存在,可利用化学方法形成C9,11-双键体系,进而在C9-位引入一个卤族原子,同时在C11β-位上形成重要的功能羟基,该中间体可进一步合成糖皮质激素抗炎药和高效含卤 (氟,氯) 的皮质激素。该工艺流程的实现可根本解决目前工业生产中存在的C11α-羟基化转化率低、副产物多的问题,具有极高的商业价值[4]。
3-甾酮-9α-羟基化酶广泛存在于甾醇化合物降解菌中,属于单加氧酶IA家族,由kshA编码的Rieske型加氧酶和kshB编码的还原酶两个亚基构成[5]。2002年,van der Geize等首先对红平红球菌Rhodococcus erythropolis SQ1的3-甾酮-9α-羟基化酶编码基因kshA和kshB进行了分子鉴定[2]。2006年,Andor等发现了耻垢分枝杆菌M. smegmatis mc2155中的kshA基因并获得异源表达[6]。2009年,Petrusma等在大肠杆菌中表达了来自紫红红球菌Rhodococcus rhodochrous DSM 43269的kshA和kshB基因,并证明kshB将来自NADH的还原力传递给kshA,后者催化底物的羟基化反应[7]。2009年,范书玥等对分枝杆菌Mycobacterium sp. NwIB-01中的kshA进行了异源表达和表达蛋白的纯化[8]。2011年,van der Geize所在课题组分析了存在于紫红红球菌R. rhodochrous DSM 43269基因组中的5个kshA基因产物的酶学 特性[9]。
本研究在戈登氏菌G. neofelifaecis NRRL B-59395和耻垢分枝杆菌M. smegmatis mc2 155的转录组测序的基础上,克隆受胆固醇诱导的ksh候选基因,构建kshA和kshB基因共表达质粒,将其导入甾醇转化的模式菌-分枝杆菌Mycobacterium sp. NRRL B-3805,构建了kshA和kshB基因共表达的重组菌,并利用该重组菌转化甾醇化合物,制备获得了9-OH-AD。本研究成功利用基因工程手段对甾醇转化模式菌株的3-甾酮-9α-羟基化酶基因进行改造,为甾体药物中间体9α-雄烯二酮的工业生产奠定基础。
1 材料与方法 1.1 菌株和质粒戈登氏菌G.neofelifaecis NRRL B-59395为胆固醇高效降解菌,由本实验室从肉食动物肠道中分离获得[10];耻垢分枝杆菌M. smegmatis mc2155、大肠杆菌Escherichia coli DH5α、分枝杆菌Mycobacterium sp. NRRL B-3805,均保存于本实验室。分枝杆菌表达质粒pNIT由Christopher M. Sassettia教授 (University of Massachusetts Medica l School) 构建、西南大学谢建平教授转赠。
1.2 培养基和主要试剂基础培养基:MgSO4·7H2O 0.25 g,NH4NO3 1.0 g,K2HPO4 0.25 g,FeSO4·7H2O 1.0 mg,酵母粉5 g,葡萄糖1 g,加水至1 000 mL,pH调为7.2。
大肠杆菌LB培养基:胰蛋白胨10 g,酵母粉5 g,氯化钠10 g,琼脂粉10 g,加水至1 000 mL。
限制性内切酶及T4 DNA连接酶购自TaKaRa公司;质粒提取试剂盒、琼脂糖凝胶回收试剂盒购自OMEGA公司;卡那霉素购自Amresco公司。
1.3 DNA的常规操作方法质粒提取、DNA限制性酶切、DNA片段连接以及大肠杆菌的转化等分子生物学常规操作方法参照分子克隆实验指南[11]以及相关产品说明书。
1.4 总DNA的提取戈登氏菌G. neofelifaecis NRRL B-59395和耻垢分枝杆菌M. smegmatis mc2155的总DNA提取方法参照文献[12]。
1.5 目的DNA片段的PCR扩增本研究所用引物如表1所示,由北京华大基因服务技术有限公司合成。PCR反应体系 (50 μL):模板1 μL,上下引物各0.5 μL,10×缓冲液5 μL,Taq酶0.6 μL,MgCl2 3 μL,加水补足50 μL。反应条件:94 ℃变性5 min,扩增反应36个循环 (94 ℃变性50 s,52 ℃复性50 s,72 ℃延伸1 min),72 ℃保温10 min。
| Primer name | Primer sequence (5′−3′) | Size (bp) |
| MSMEG_5925-F | ACTCATATGGCTACCGAGACTGTCGG (the underline indicates NdeⅠ) | 26 |
| MSMEG_5925-R | AGCGAATTCAGCTCGACTGCTCCGCCTTC (the underline indicates EcoRⅠ) | 29 |
| MSMEG_6039-F | CTAGAATTCGGAGGAATCACTTCGCAATGGTGACTGATGAGCCCCTGG (the underline indicates EcoRⅠ) | 48 |
| MSMEG_6039-R | GCAAAGCTTCTATTCGTCGTAGGTGACTTCG (the underline indicates Hind Ⅲ) | 31 |
| SCNU_16633-F | CAGCATATGATGGCTGACACCGACATCCG (the underline indicates NdeⅠ) | 29 |
| SCNU_16633-R | ATCGGATCCTTCGTCGGCGCGAGTCATCAGAC (the underline indicates BamHⅠ) | 32 |
| SCNU_18062-F | ATAGGATCCGGAGGAATCACTTCGCAATGACCGAACTGACCCCGCAC (the underline indicates BamHⅠ) | 47 |
| SCNU_18062-R | GCTAAGCTTCAGAACTCGATCTTGAGCTTGTCGG (the underline indicates Hind Ⅲ) | 34 |
分枝杆菌Mycobacterium sp. NRRL B-3805电转化感受态细胞的制作参照文献[13]。
1.7 分枝杆菌的电转化取100 μL分枝杆菌Mycobacterium sp. NRRL B-3805感受态细胞与2 μL重组质粒混匀,冰上放置30 min,吸取感受态细胞与质粒混合液垂直加入电转杯中,设定电压为2 500 V进行电转化,吸出转化混合液,加入500 μL液体基础培养基,于30 ℃、180 r/min条件下恢复培养3 h后,5 000 r/min离心2 min,倒掉培养液,用100 μL无菌水重悬细胞,将其涂布于含20 μg/mL卡那霉素的基础培养基平板上,28 ℃倒置培养8-10 d,统计菌落数。挑取平板上长出的抗性单菌落进行PCR快速鉴定[14]。
1.8 转化产物化学结构的鉴定转化产物经分离纯化后,采用13C NMR、1H NMR、高分辨质谱、红外光谱进行分析,鉴定其结构,得到的波谱数据如下:TOF-MS m/z 303 [M+H]+;IR vmax(cm-1):3304,1735,1655;13C NMR (CDCl3,400 MHz)δ:29.2(C-1),33.4(C-2),180.36(C-3),125.1(C-4),172.0(C-5),31.6(C-6),23.0(C-7),35.8(C-8),76.1(C-9),37.1(C-10),26.8(C-11),28.2(C-12),44.2(C-13),42.0(C-14),21.1(C-15),35.3(C-16),200.8(C-17),11.8(C-18),18.9(C-19)。1H NMR (CDCl3) 0.93(3H,s,H-18),1.38(3H,s,H-19),2.03 (1H,ddd,H-8),6.05 (1H,s,H-4)。
1.9 甾醇的生物转化及定量分析将重组菌株接种于100 mL分别含植物甾醇、胆固醇、谷甾醇 (0.5 g/L) 的液体基础培养基中 (加入0.05% Tween-80乳化底物),于30 ℃、180 r/min振荡培养。当菌液OD600约为0.8时加入己内酰胺 (终浓度为28 mmol/L),诱导表达24 h[15]。每隔12 h取样,乙酸乙酯振荡萃取,薄层层析检测,展层剂为环己烷:乙酸乙酯=6∶4。将萃取样品真空干燥后,甲醇溶解,用于HPLC分析,计算其转化率[16]。采用岛津Essentia LC-15C高效液相色谱仪进行HPLC分析;色谱柱为岛津WondaSil C18色谱柱 (4.6 mm×150 mm,5 μm),柱温35 ℃,检测波长 254 nm,流动相为甲醇∶水=65∶35,流速: 1 mL/min,进样量:20 μL。转化率计算方法为:转化率=(9-OH-AD产生量/底物量)×100%。
1.10 重组分枝杆菌稳定性分析将重组菌株接种于50 mL液体基础培养基中,振荡培养,分别于48、96、144 h吸取菌液,用无菌水稀释,浓度梯度为10-3、10-4、10-5,分别涂布于不含卡那霉素和含20 μg/mL卡那霉素的基础培养基平板上,28 ℃培养7 d,对单菌落数进行计数[1]。
2 结果与分析 2.1 3-甾酮-9α-羟基化酶候选基因转录与同源性分析对耻垢分枝杆菌M. smegmatis mc2155基因组进行搜索,结果显示MSMEG_2870和MSMEG_5925为kshA候选基因MSMEG_2893和MSMEG_6039为kshB候选基因;而戈登氏菌G.neofelifaecis NRRL B-59395基因组中存在着3个kshA候选基因,分别是SCNU_16633、SCNU_18995、SCNU_20231;同时有2个kshB候选基因,它们分别是SCNU_18062、SCNU_05586。在前期研究中,我们分别对耻垢分枝杆菌和戈登氏菌在不同甾醇化合物为唯一碳源条件下的转录组进行了测序 (该研究结果另文报道),对所得数据进行分析,结果表明耻垢分枝杆菌M. smegmatis mc2155的2个kshA候选基因中,MSMEG_5925在3种碳源条件下表达量均相对较高,RPKM值为分别为101.47、288.50、100.33,而且该基因在胆固醇条件下表达明显上调,如表2所示;其两个kshB候选基因表达量均较低,其中MSMEG_6039在胆固醇条件下表达上调。戈登氏菌G. neofelifaecis NRRL B-59395转录组测序表明,kshA候选基因SCNU_16633和SCNU_20231的表达均受到胆固醇的诱导,其中SCNU_16633表达量较高,RPKM值为214.60;kshB候选基因SCNU_18062在雄烯二酮条件下表达上调。综合上述分析,我们推测差异表达的候选基因MSMEG_5925、MSMEG_6039、SCNU_16633、SCNU_18062参与了甾醇化合物的羟基化,因此对其进行克隆并用于重组菌株的构建。
| Gene ID | Encoding subunit of KSH | AD (RPKM) | Cholesterol (RPKM) | Glycerin (RPKM) |
| MSMEG_2870 | KshA | 13.13 | 10.67 | 1.42 |
| MSMEG_5925 | KshA | 101.47 | 288.50 | 100.33 |
| MSMEG_2893 | KshB | 0 | 6.2 | 0 |
| MSMEG_6039 | KshB | 0 | 28.2 | 0 |
| SCNU_16633 | KshA | 54.87 | 214.60 | 113.7 |
| SCNU_18995 | KshA | 70.12 | 11.23 | 2.33 |
| SCNU_20231 | KshA | 23.62 | 104.63 | 12.39 |
| SCNU_18062 | KshB | 88.22 | 61.095 | 40.52 |
| SCNU_05586 | KshB | 3.22 | 5.23 | 3.10 |
| RPKM: reads per kilobase of coding sequence,per million mapped reads. | ||||
氨基酸同源性分析表明耻垢分枝杆菌 M. smegmatis mc2155中的MSMEG_5925与结核分枝杆菌Mycobacterium tuberculosis H37RV的KshA[17]编码基因Rv3526在氨基酸水平的同源性高达82%,同红平红球菌R. erythropolis SQ1中KshA[2]同源性为61%,同紫红红球菌 R. rhodochrous DSM 43269中的KshA[7]同源性为58%。MSMEG_6039与结核分枝杆菌 M. tuberculosis H37RV编码KshB的基因Rv3571在氨基酸水平的同源性为73%,同红平红球菌R. erythropolis SQ1中KshB[2]的氨基酸同源性为60%,同紫红红球菌R. rhodochrous DSM 43269中的KshB同源性为58%。
戈登氏菌G. neofelifaecis NRRL B-59395中的SCNU_16633同结核分枝杆菌M. tuberculosis H37RV的Rv3526 (kshA) 基因在氨基酸水平的同源性达到53%,同红平红球菌R. erythropolis SQ1中KshA[2]的氨基酸同源性为61%,与紫红红球菌R. rhodochrous DSM 43269中KshA[6]同源性为55%。SCNU_18062与结核分枝杆菌 M. tuberculosis H37RV编码KshB的Rv3571在氨基酸水平的同源性达到52%,与红平红球菌R. erythropolis SQ1中的KshB[2]同源性为64%,同紫红红球菌R. rhodochrous DSM 43269中的KshB[7]同源性为65%。
2.2 重组质粒的构建与转化构建的共表达质粒图谱如图1所示。以耻垢分枝杆菌M. smegmatis mc2 155总DNA为模板,分别用引物MSMEG_5925-F/R和MSMEG_6039-F/R进行PCR扩增,得到目的片段MSMEG_5925 (大小为1 151 bp) 和MSMEG_6039 (大小为1 061 bp),如图2A泳道1、2所示。用NdeⅠ/EcoRⅠ双酶切DNA片段MSMEG_5925,将其插入经过同样酶切处理的表达载体pNIT中,获得的中间质粒再经EcoRⅠ/Hind Ⅲ双酶切,与经过同样酶切处理的MSMEG_6039连接,从而与MSMEG_5925融合,获得含有MSMEG_5925和MSMEG_6039融合基因的表达质粒,命名为 pNIT-Mksh (图1A)。

|
| 图1 表达质粒pNIT-Mksh和pNIT-Gksh示意图 Fig.1 The construction of expression plasmids pNIT-Mksh and pNIT-Gksh. |

|
| 图2 共表达重组质粒琼脂糖电泳分析 Fig.2 Agarose gel electrophoresis analysis of recombinant plasmids of pNIT-Mksh and pNIT-Gksh. (A) 1: the PCR product of MSMEG_5925; 2: the PCR product of MSMEG_6039; 3: pNIT; 4: pNIT-Mksh /NdeI,Hind Ⅲ; M: DNA marker. (B) 1: the PCR product of SCNU_16633; 2: the PCR product of SCNU_18062; 3: the pNIT; 4: pNIT-Gksh/NdeⅠ,Hind Ⅲ; M: DNA marker. |
同时,以戈登氏菌G. neofelifaecis NRRL B-59395的总DNA为模板,用引物SCNU_16633-F/R和SCNU_18062-F/R分别扩增得到DNA片段SCNU_16633 (图2B泳道1,1 170 bp) 和SCNU_18062 (图2B泳道2,1 056 bp)。经 NdeⅠ/BamHⅠ双酶切将SCNU_16633重组入pNIT载体,获得的中间质粒再经BamHⅠ/Hind Ⅲ双酶切,与经过同样酶切的DNA片段SCNU_18062连接,获得含有SCNU_16633和SCNU_18062融合的表达质粒,命名为pNIT-Gksh (图1B)。
用限制性内切酶NdeⅠ和Hind Ⅲ对两重组质粒pNIT-Mksh和pNIT-Gksh分别进行双酶切,琼脂糖电泳显示,pNIT-Mksh经双酶切产生两条DNA条带 (图2A泳道4),其中较大的条带大小为6 261 bp,与原载体pNIT相符,大小约为2 kb的DNA条带是MSMEG_5925、MSMEG_6039融合片段。图2B泳道4为pNIT-Gksh双酶切的结果,产生的条带为SCNU_16633、SCNU_18062融合片段 (2 226 bp) 和原载体pNIT (6 261 bp),同样与预期相符。
采用电转化方法将上述表达质粒分别导入分枝杆菌Mycobacterium sp. NRRL B-3805感受态细胞中,涂布于含卡那霉素的培养基中培养,挑取单菌落进行菌落PCR扩增,阳性菌落即为构建的基因重组菌株。质粒pNIT-Mksh电转入分枝杆菌Mycobacterium sp. NRRL B-3805得到的重组菌株命名为3805-Mksh,质粒pNIT-Gksh电转入分枝杆菌Mycobacterium sp. NRRL B-3805得到的重组菌株命名为3805-Gksh。
2.3 重组菌株转化产物的鉴定分别利用原始菌株和重组菌株对胆固醇、植物甾醇和谷甾醇进行生物转化,用薄层层析 (图3A) 和HPLC (图3B) 检测转化产物的变化。原始菌株分枝杆菌Mycobacterium sp. NRRL B-3805转化产物是大量AD和极少量ADD (图3A泳道1,图3B曲线c),而导入kshA-kshB共表达质粒的重组菌株的转化产物中,AD产量变低甚至消失,并产生了新的转化产物 (图3A泳道7-12,图3B曲线a)。图3B (曲线a、c) 还显示,原始菌株Mycobacterium sp. NRRL B-3805和重组菌株的转化产物中除目的产物外均含有少量副产物或杂质,但并未对其做进一步分析和鉴定。从薄层层析板上刮取产物,乙酸乙酯萃取,减压蒸馏,获得纯化的样品,进行光谱学分析以鉴定其化学结构。红外光谱出现了数值为3 304的吸收峰,说明转化产物中含有羟基基团;TOF-MS谱显示其[M+H]+处于m/z 303,说明该产物分子量为302,与9-OH-AD的分子质量302.4相符合;对比AD的13C NMR谱,C9的吸收峰值从δ53.8 ppm位置化学位移到δ76.1 ppm,验证了C9出现1个羟基基团,这与文献[18]报道的相符。1H NMR光谱分析表明存在两个甲基基团,其δ值分别为0.93(s) 和 1.35(s) ppm,6.05(d) ppm表示存在1个双键;未出现δ3.5-4.5范围的化学位移,推测羟基应该存在于一个三级碳原子上,这与Horinouchi等的报道一致[19],确定该基团应存在于9α-位上。因此重组菌株转化甾醇底物获得产物9-OH-AD,证明导入的基因kshA、kshB成功表达出了有活性的KSH,重组菌株直接将胆固醇、植物甾醇和谷甾醇转化为9-OH-AD。
|
| 图3 转化产物的薄层层析 (A) 和HPLC (B) 分析 Fig.3 TLC and HPLC analysis of products. (A) TLC analysis of products. 1: the products of strain NRRL B-3805; 2: the products of NRRL B-3805 with pNIT; 3−6: the standard samples of AD,ADD,ADD and AD; 7−9: the products of strain 3805-Mksh cultured in phytosterol,sitosterol or cholesterol; 10−12: the products of strain 3805-Gksh cultured in phytosterol,sitosterol or cholesterol. (B) HPLC analysis for the transformation products. a−c: HPLC analysis for the products of the recombination strains,the standard sample of 9-OH-AD and the products of the strain NRRL B-3805. |
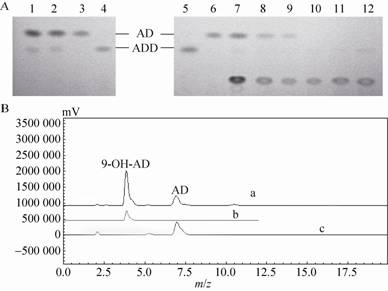
分别以植物甾醇、胆固醇、谷甾醇为底物,利用构建的重组菌株进行转化,取样,HPLC检测,结果表明,重组菌株转化36 h时,3种不同底物的转化液中均开始积累9-OH-AD。菌株3805-Mksh在转化108 h时,以植物甾醇或以胆固醇为底物的培养液中,9-OH-AD积累量达到最大,分别为453.1 μg/mL和34 μg/mL。其中,植物甾醇的最高转化率达到了90.6% (图4A);而以谷甾醇为底物转化,转化120 h后,培养液中9-OH-AD积累量达到最高,为183.3 μg/mL。随着转化时间的延长,9-OH-AD积累量逐渐降低。
|
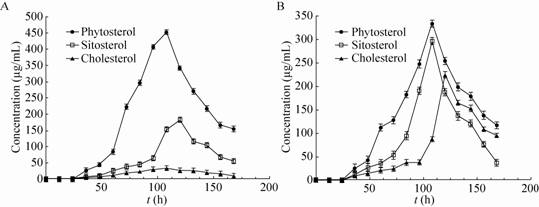
| 图4 菌株3805-Mksh (A) 和3805-Gksh (B) 对几种底物的转化 Fig.4 The transformation of the several substrates by 3805-Mksh (A) and 3805-Gksh (B). |
重组菌3805-Gksh以植物甾醇或谷甾醇为底物转化,9-OH-AD最大积累量出现在108 h,达到335 μg/mL、296 μg/mL;而以胆固醇为底物转化,则出现在120 h,达到225.7 μg/mL,底物的最大转化率为67% (图4B)。其9-OH-AD积累量同样随着转化时间延长而降低。
2.5 重组分枝杆菌稳定性分析选取转化能力较强的重组分枝杆菌 3805-Mksh进行质粒稳定性分析,结果如表3所示,经过144 h的发酵培养,含有质粒的菌落由98.97%降到了85%,因而随着培养时间的延长,质粒稳定性略有下降,但总体稳定性良好。
| Time (h) | Dilution factor | CFU Kan(−) | CFU Kan(+) | CFU Kan(+)/kan(−) |
| 48 | 10-3 | 291 | 288 | 98.97% |
| 10-4 | 29 | 28 | 96.55% | |
| 96 | 10-4 | 273 | 252 | 92.31% |
| 10-5 | 23 | 21 | 91.30% | |
| 144 | 10-4 | 236 | 209 | 88.56% |
| 10-5 | 20 | 17 | 85.00% |
KSH为双组分酶,由KshA和KshB组成。KshA是9α-羟基化起主体作用的酶,而KshB为铁氧还蛋白还原酶,对KshA起还原和电子循环传递作用。目前,仅有耻垢分枝杆菌M. smegmatis mc2155的kshA、红平红球菌R. erythropolis SQ1的kshA和kshB,来自紫红红球菌R. rhodochrous DSM 43269的kshA和kshB[20]以及分枝杆菌Mycobacterium sp. NwIB-01中的kshA获得了功能验证。我们依据基因在胆固醇条件下基因表达上调的情况,从耻垢分枝杆菌M.smegmatis mc2155的2个kshA候选基因和2个kshB候选基因中选择了MSMEG_5925 (kshA候选基因)、MSMEG_6039 (kshB候选基因) 进行克隆并用于基因重组菌的构建;从戈登氏菌G. neofelifaecis NRRL B-59395 基因组的3个kshA候选基因、2个kshB候选基因中,选择了SCNU_16633 (kshA候选基因)、SCNU_18062 (kshB候选基因) 进行克隆并用于基因重组菌的构建。
为了实现kshA和kshB的共表达和增加翻译效率,构建表达质粒时,在kshB基因上游引物中设计了核糖体结合位点SD序列和间隔序列 (CGGAGGAATCACTTCGCA),依次将kshA和kshB克隆到分枝杆菌表达质粒pNIT中时,从而在两个基因之间引入了核糖体结合位点序列 (SD) 和间隔序列。分枝杆菌Mycobacterium sp. NRRL B-3805是研究甾醇微生物转化的模式菌,能够转化胆固醇、植物甾醇积累大量雄烯二酮和少量雄二烯二酮。将kshA和kshB的共表达质粒转入分枝杆菌Mycobacterium sp. NRRL B-3805,构建重组菌株,利用该重组菌株转化胆固醇、植物甾醇和谷甾醇,进而诱导kshA和kshB进行表达,但可能由于表达载体在宿主细胞中拷贝数过低,表达量较少,因而在蛋白质电泳中并未检测到目的蛋白质条带,但是通过分离纯化转化产物,进行光谱学鉴定,确定该转化产物为9α-羟基雄烯二酮,可表明导入的kshB和kshA获得了有效表达,从而证明将出发菌株积累的雄烯二酮转化为9α-羟基雄烯二酮。同时也证明了来自耻垢分枝杆菌的MSMEG_5925、MSMEG_6039和戈登氏菌的SCNU_16633、SCNU_18062分别为有功能的kshB和kshA编码基因。
各重组菌株分别以3种甾醇化合物为底物进行转化时,以植物甾醇为底物较胆固醇和谷甾醇的转化率要高;利用重组分枝杆菌 3805-Mksh以植物甾醇为底物转化时,转化率最高达到90.6%,而重组分枝杆菌 3805-Gksh最大转化率仅为67%,显示来自来耻垢分枝杆菌M. smegmatis mc2155的kshA和kshB基因产物具有更高的羟基化活性。
在生物转化的过程中,9-OH-AD会随着转化时间的延长而降解,其可能的原因是分枝杆菌Mycobacterium sp. NRRL B-3805大量积累AD的同时,也能产生少量的ADD,说明其细胞中仍有少量的类固醇脱氢酶 (3-ketosteroid D1-dehydrogenase,KsdD) 活性,将AD转变ADD,ADD在羟基化酶的催化作用下,打开核心环,从而进一步被降解,因而9-OH-AD会随着转化时间的延长而减少。因此,要进一步提高9-OH-AD产率,需要敲除ksdD基因,消除其脱氢酶活性。本研究在甾醇化合物转化模式菌株中实现了3-甾酮-9α-羟基化酶基因共表达,使其由积累AD变为积累9-OH-AD,因而成功地进行了分枝杆菌生物转化特性的改造,为探索各种甾体药物中间体的工业生产奠定了基础。
| [1] | Wei W, Fan SY, Wang FQ, et al. Accumulation of androstadiene-dione by overexpression of heterologous 3-ketosteroid Δ1-dehydrogenase in Mycobacterium neoaurum NwIB-01. World J Microbiol Biotechnol, 2014, 30(7): 1947-1954. |
| [2] | van der Geize R, Hessels GI, Van Gerwen R, et al. Molecular and functional characterization of kshA and kshB, encoding two components of 3-ketosteroid-9α-hydroxylase, a class IA monooxygenase, in Rhodococcus erythropolis strain SQ1. Mol Microbiol, 2002, 45(4): 1007-1018. |
| [3] | Sukhodolskaya GV, Nikolayeva VM, Khomutov SM, et al. Steroid-1-dehydrogenase of Mycobacterium sp. VKM-Ac-1817D strain producing 9α-hydroxy- androst-4-ene-3,17-dione from sitosterol. Appl Microbiol Biotechnol, 2007, 74(4): 867-873. |
| [4] |
Yang SK, Yang YL, Wu ZL, et al. Microbial fermentation of phytosterol side chain cleaved for production of 17-keto steroids. Chin J Bioproc Eng, 2010, 8(5): 69-77 (in Chinese). 杨顺楷, 杨亚力, 吴中柳, 等. 微生物发酵降解植物甾醇侧链生产17-酮甾体研究进展. 生物加工过程, 2010, 8(5): 69-77. |
| [5] | Hu Y, van der Geize R, Besra GS, et al. 3-ketosteroid 9α-hydroxylase is an essential factor in the pathogenesis of Mycobacterium tuberculosis. Mol Microbial, 2010, 75(5): 107-121. |
| [6] | Andor A, Jekkel A, Hopwood DA, et al. Generation of useful insertionally blocked sterol degradation pathway mutants of fast-growing mycobacteria and cloning, characterization, and expression of the terminal oxygenase of the 3-ketosteroid 9α-hydroxylase in Mycobacterium smegmatis mc2155. Appl Environ Microbiol, 2006, 72(10): 6554-6559. |
| [7] | Petrusma M, Dijkhuizen L, van der Geize R. Rhodococcus rhodochrous DSM 43269 3-ketosteroid- 9α-hydroxylase, a two-component iron-sulfur-containing monooxygenase with subtle steroid substrate specificity. Appl Environ Microbiol, 2009, 75(16): 5300-5307. |
| [8] |
Fan SY, Wei W, Wang FQ, et al. Cloning, heterologous expression and purification of a 3-ketosteroid-9α-hydroxylase (KSH) from Mycobacterium sp. NwIB-01. Chin J Biotech, 2009, 25(12): 2014-2021 (in Chinese). 范书玥, 魏巍, 王风清, 等. 分枝杆菌Mycobacterium sp. NwIB-01 3-甾酮-9α-羟基化酶基因的克隆、异源表达及分离纯化. 生物工程学报, 2009, 25(12): 2014-2021. |
| [9] | Mirjan P, Gerda H, Lubbert D, et al. Multiplicity of 3-ketosteroid-9α-hydroxylase enzymes in Rhodococcus rhodochrous DSM 43269 for specific degradation of different classes of steroids. J Bacteriol, 2011, 193(15): 3931-3940. |
| [10] | Liu YC, Chen GY, Li W, et al. Efficient biotransformation of cholesterol to androsta-1,4-diene-3,17-dione by a newly isolated actinomycete Gordonia neofelifaecis. World J Microbiol Biotechnol, 2011, 27(4): 759-765. |
| [11] | Sambrook J, Fritsch EF, Maniatis T. Molecular Cloning: A Laboratory Manual. 2nd ed. New York: Cold Spring Harbor Laboratory Press, 1989: 20-25. |
| [12] | Liu YC, Ge F, Li W, et al. Gordonia neofelifaecis sp. nov., a cholesterol side-chain-cleaving actinomycete isolated from the faeces of Neofelis nebulosa. Internat J System Evolut Microbiol, 2011, 61(1): 1-5. |
| [13] | Veiga-Crespo P, Feijoo-Siota L, de Miguel T, et al. Proposal of a method for the genetic transformation of Gordonia jacobaea. J Appl Microbiol, 2006, 100(3): 608-614. |
| [14] | Daniel B, Matthias A, Alexander S. Transfer of megaplasmid pKB1 from the rubber-degrading bacterium Gordonia westfalica strain Kb1 to related bacteria and its modification. Appl Microbiol Biotechnol, 2008, 77(6): 1317-1327. |
| [15] | Chen T, Zhao QJ, Li W, et al. Mycobacterium tuberculosis PE_PGRS17 promotes the death of host cell and cytokines secretion via Erk kinase accompanying with enhanced survival of recombinant Mycobacterium smegmatis. J Interf Cytok Res, 2013, 33 (8): 452-458. |
| [16] | Wei W, Fan SY, Wang FQ, et al. A new steroid-transforming strain of Mycobacterium neoaurum and cloning of 3-ketosteroid 9α-hydroxylase in NwIB-01. Appl Biochem Biotechnol, 2010, 162(5): 1446-1456. |
| [17] | Capyk J K, Angelo I D, et al. Characterization of 3-ketosteroid- 9α-hydroxylase, a rieske oxygenase in the cholesterol degradation pathway of Mycobacterium tuberculosis. J Biol Chem, 2009, 284(15): 9937-9946. |
| [18] | Mohammad AF, Maryam A, Mojtaba TY. Metabolism of androst-4-en-3,17-dione by the filamentous fungus Neurospora crassa. Steroids, 2008, 73 (1): 13-18. |
| [19] | Horinouchi M, Hayashi T, Yamamoto T, et al. A new bacterial steroid degradation gene cluster in comamonas testosteroni TA441 which consists of aromatic-compound degradation genes for seco-steroids and 3-ketosteroid dehydrogenase genes. Appl Environ Microbiol, 2003, 69 (8): 4421-4430. |
| [20] | Wilbrink MH, Petrusma M, Dijkhuizen L, et al. FadD19 of Rhodococcus rhodochrous DSM43269, a steroid-coenzyme ligase essential for degradation of C-24 branched sterol side chains. Appl Environ Microbiol, 2011, 77(13): 4455-4464. |
 2015, Vol. 31
2015, Vol. 31