服务
文章信息
- 林兆, 董红军, 李寅
- Zhao Lin, Hongjun Dong, Yin Li
- 利用Tn5转座子介导突变提高大肠杆菌丁醇生产水平
- Improvement of butanol production by Escherichia coli via Tn5 transposon mediated mutagenesis
- 生物工程学报, 2015, 31(12): 1711-1719
- Chin J Biotech, 2015, 31(12): 1711-1719
- 10.13345/j.cjb.140646
-
文章历史
- Received: December 28, 2014
- Accepted: January 22, 2015
2. 中国科学院大学,北京 100049
2University of Chinese Academy of Sciences, Beijing 100049, China
丁醇是一种重要的化学品,可直接用作有机溶剂和合成多种酯类化合物的前体,同时它也是一种比乙醇更有优势的生物燃料[1]。生物丁醇可由一些梭菌菌株在厌氧条件下经过ABE (Acetone-butanol-ethanol) 发酵产生,至今已有超过100年的历史[2]。但是由于梭菌菌株本身是严格厌氧的革兰氏阳性细菌,遗传操作困难,并且由于本身复杂的代谢调控机制,因此很难提升梭菌发酵生产丁醇的能力,从而限制了生物丁醇市场竞争力。借助于现代生物技术,科学家开始利用易于操作的模式细菌大肠杆菌来生产丁醇,取得了显著的进展。
2008年,来自University of California,Los Angeles的Liao研究组最先报道了把一条完整的梭菌丁醇发酵途径导入到大肠杆菌中 (thl、hbd、crt、bcd、etfA、etfB、adhE2),第一次实现利用大肠杆菌生产丁醇[3]。在随后的几年里,大量的相关工作逐渐开展和发表,主要包括:1) 选择更为高效的酶。利用酶活更高的乙酰辅酶A乙酰转移酶AtoB (由大肠杆菌的atoB基因编码) 来替代硫解酶Thl (由丙酮丁醇梭菌的thl基因编码),丁醇的产量提高了3倍[3];使用可催化不可逆反应且利用NADH为辅因子的顺式酰基辅酶A还原酶Ter (由齿垢密螺旋体的ter基因编码) 替代丁酰辅酶A脱氢酶复合体BCD (由丙酮丁醇梭菌的bcd、etfAB基因编码),使丁醇产量提高了17倍[4, 5]。2) 还原力平衡设计。使用来自酵母菌的甲酸脱氢酶FDH (fdh基因编码),可把厌氧发酵时产生的甲酸转化为二氧化碳和NADH,可实现丁醇合成的还原力平衡,在丰富培养基中丁醇得率最大可达36% (W/W)[4, 5],也可以通过过表达丙酮酸脱氢酶PDH复合体 (由aceEF.lpd基因编码),增强丙酮酸到乙酰辅酶A且偶联产生NADH的能力,丁醇得率可达到28% (W/W)[4]。3) 由于大肠杆菌本身存在一些副产物途径,从而影响了丁醇的产量和得率,去除这些副产物途径中关键基因 (主要是产琥珀酸的frdABCD、产乳酸的ldhA、产乙酸的pta-ack和产乙醇的adhE),可显著提高丁醇的合成能力[4, 5],目前报道最高的大肠杆菌丁醇产量是14−15 g/L[5, 6],略低于梭菌[7]。
基于理性知识指导的生产丁醇的大肠杆菌改造已经取得了显著的进展[8, 9]。但是与梭菌相比较并没有绝对的优势,还远未达到有市场竞争力的产业化水平,仍需要更多的改进[10]。现在的菌株改造局限于有限的几个基因,然而细胞本身是一个系统性的整体,若要实现丁醇的高产,必然有基因组上其他基因的支持,但是具体哪些基因起到作用目前还难以预测。为了发现这些潜在的基因靶标,我们利用Tn5转座子[11, 12]对一株可生产丁醇的大肠杆菌工程菌株进行了基因组水平的随机插入,构建了一个突变体文库,通过对文库筛选得到了丁醇产量提高的突变体,再对转座子插入位点进行定位,找出了有效的基因靶点,为构建更高效的产丁醇大肠杆菌提供了指导。
1 材料与方法 1.1 材料 1.1.1 菌株实验所用菌株为大肠杆菌EB216菌株。EB216菌株是实验室前期根据Liao研究组[5]的工作构建,在15 mL BD小管中,利用M9培养基静置发酵能够产生1 g/L左右的丁醇。
1.1.2 试剂及引物EZ-Tn5转座子试剂盒 (EZ-Tn5™ <KAN-2> Tnp Transposome™ Kit,Cat. No. TSM99K2) 购自Epicentre公司,内含可直接用于转化的卡那霉素抗性转座子复合体及用于插入位点定位的反向PCR引物KAN-2 FP-1 Forward Primer (5ʹ-ACCTACAACAAAGCTCTCATCAACC-3ʹ) 和KAN-2 RP-1 Reverse Primer (5ʹ-GCAATGTA ACATCAGAGATTTTGAG-3ʹ);细菌基因组提取试剂盒、胶回收试剂盒购自Omega公司;限制性内 切酶SalⅠ、T4 DNA连接酶等购自NEB公司;Taq DNA聚合酶、dNTPs等购自北京全式金生物技术有限公司。
二硝基苯肼购自国药集团化学试剂有限公司,溶于2 mol/L的HCl中制备0.1%的二硝基苯肼母液,最终稀释成0.037 5%的二硝基苯肼使用;NaOH购自北京化工厂,配成浓度为 2.81 mol/L溶液备用。
1.1.3 培养基菌株发酵培养基为M9培养基,其1 L组成为:20 g葡萄糖,17.1 g Na2HPO4·12H2O,3.0 g KH2PO4,0.5 g NaCl,1.0 g NH4Cl,0.002 mol MgSO4,0.000 1 mol CaCl2,0.5 mg VB1,1 g酪蛋白氨基酸 (CA)。1 L的LB培养基组成为:10 g胰化蛋白胨,5 g酵母提取物,10 g NaCl。1 L的SOC培养基组成为:20 g胰化蛋白胨,5 g酵母提取物,0.5 g NaCl,2.5 mmol KCl,0.01 mol MgCl2,20 mmol葡萄糖。
pUC19筛选平板为含100 mg/mL氨苄青霉素的LB琼脂平板,EZ-Tn5转座复合物转化子筛选平板为含50 mg/mL卡那霉素的LB琼脂平板。
1.2 方法 1.2.1 感受态细胞制备至转化EB216菌株的电转感受态细胞制备参考《分子克隆》[13]中的甘油法,制备感受态细胞使用pUC19质粒测试转化效率,以确保达到109/µg DNA以上。电击采用Gene Pulser Xcell电穿孔系统 (Bio-Rad),使用2 mm电击杯,电击参数为:2 500 V,25 µF,200 Ω。
1.2.2 突变文库的制备利用制备好的EB216感受态细胞,电转1 µL Tn5转座子复合体。利用SOC液体培养基对电转菌株37 ℃复壮培养1 h后,涂布于含50 mg/mL卡那霉素的LB琼脂平板。
1.2.3 用二硝基苯肼法测定丙酮酸丙酮酸在酸性条件下与2,4-二硝基苯肼发生缩合反应形成丙酮酸二硝基苯腙。二硝基苯腙在碱性条件下呈现红色,可在520 nm处比色后灵敏地反映出丙酮酸的含量[14]。
以M9培养基作为溶剂,配置6.248 g/L的丙酮酸钠母液,再用M9培养基稀释成0、0.625、1.250、1.875、2.499、3.124、3.749、4.998 g/L的溶液,取80 µL不同稀释度丙酮酸钠溶液、80 µL 0.037 5%的二硝基苯肼、80 µL 2.81 mol/L NaOH混合,静置反应10 min,利用Molecular Device Spectra Max 190酶标仪在520 nm的波长下测定吸光度。每个梯度重复3次。
1.2.4 突变菌株96孔深孔板内发酵及筛选将成功插入Tn5转座子的菌株,利用灭菌的牙签接种于96孔深孔板,每个孔装有1.25 mL的M9液体培养基,37 ℃条件下厌氧培养箱(Bactron I-2,Shellab) 中培养72 h。取发酵液200 µL于96孔酶标板,以空白为对照,利用酶标仪在600 nm的波长下测定吸光度。
同时将发酵液稀释25倍后利用二硝基苯肼法测定丙酮酸含量。
1.2.5 菌株的发酵评价将最终筛选到的菌株进行小管发酵验证。首先在LB培养基里37 ℃培养过夜,转接0.5 mL的LB菌液至9.5 mL的M9培养基中,在厌氧培养箱中,利用15 mL BD试管 (非密封) 于37 ℃厌氧条件静置培养72 h。取1.5 mL发酵液12 000×g下离心1 min,将上清液过滤后利用HPLC (液相色谱仪) 检测丁醇的含量,以最终确定丁醇产量得到明显提高的菌株,对高产丁醇菌株进行3轮以上发酵重复验证,每轮3个平行,以证明其产量提高的稳定性。
1.2.6 利用HPLC对丁醇产量进行测定丁醇产量利用高压液相 (HPLC system 1260 Infinity Series,Agilent Technologies) 测定。所用分析柱为Bio-Rad (Hercules,CA) Aminex HPX-87H (7.8 mm×300 mm);流动相为5 mmol/L硫酸溶液,流速为0.5 mL/min,柱温是15 ℃;检测器为折光检测器,检测温度为30 ℃;检测时间为45 min。
1.2.7 反向PCR及测序确定Tn5插入位置提取高产丁醇突变菌株的基因组DNA。根据EZ-Tn5结构,用限制性内切酶SalⅠ酶切基因组DNA。由于EZ-Tn5转座子上含有SalⅠ酶切位点,同时基因组上其他位置同样有SalⅠ位点,当细菌基因组被切成零碎片段后,用T4 DNA连接酶保证DNA片段自连接成环状DNA。以此为模板,以EZ-Tn5反向PCR引物(KAN-2 FP-1 Forward Primer及KAN-2 RP-1 Reverse Primer) 进行反向PCR,并用琼脂糖电泳分离纯化PCR产物。反向PCR产物通过北京擎科公司测序。测序引物同样为KAN-2 FP-1 Forward Primer及KAN-2 RP-1 Reverse Primer。根据测序结果来确定转座子插入位点。
2 结果与分析 2.1 构建Tn5转座子突变文库根据试剂盒说明书,通过电击转化的方式将1 µL Tn5转座子复合体 (20 ng) 导入自行制备的40 µL电转感受态细胞EB216中,可以得到100个左右的卡那霉素抗性菌落,这远低于说明书中给出的105个突变体/µL,这可能是由于菌株差异所致。通过转化10 µL转座子复合体,我们共计得到了1 196个抗性菌落文库,用于后续筛选。
2.2 建立高通量筛选方法为了高通量筛选丁醇高产突变体菌株,需要建立有效的快速筛选方法。根据前期研究经验,我们发现作为丁醇前体的丙酮酸可以在胞外积累 (浓度在0.5−1 g/L),且其浓度与丁醇浓度呈反相关,因此我们设想产丙酮酸少的突变体具有更高产丁醇能力,而丙酮酸可以通过二硝基苯肼显色法快速测定。基于此,我们配置了含不同浓度的丙酮酸溶液,将其与二硝基苯肼进行显色反应,利用酶标仪测定520 nm处吸光度 (OD520) 时,发现酶标仪可直接测定0−100 mg/L浓度范围内的丙酮酸,并呈现出良好的线性关系,拟合线性回归方程为:y=0.028 9x,R2=0.990 7。这也说明发酵液需要稀释才能直接测定,我们把发酵液稀释25倍后,用该法测出的发酵丙酮酸浓度与HPLC测定浓度基本一致,说明稀释后的菌体及发酵液其他成分不对丙酮酸测定形成干扰。
为了测定单位菌体产丙酮酸的能力,我们设计了如下筛选流程 (图1):1) 突变体库接种至96孔深孔板培养,厌氧箱内培养3 d;2) 菌体吹吸均匀后,转移200 µL到96孔酶标板,测定菌体的光密度OD600值;3) 把菌液进行两次5倍稀释,得到25倍稀释液80 µL,先后加入80 mL 0.037 5%的二硝基苯肼和80 mL 2.81 mol/LNaOH进行显色反应,测定反应液的OD520值,根据丙酮酸标准曲线及稀释倍数算出发酵液中的丙酮酸浓度;4) 最后根据单位菌体的丙酮酸浓度 (丙酮酸浓度/OD600) 筛选出突变体菌株,用于进一步分析丁醇生产能力。

|
| 图1 高产丁醇突变菌株筛选流程图 Fig.1 The screening flowchart of high-titer butanol mutants. |
利用上述方法对1 196个Tn5突变体进行了筛选,以丙酮酸浓度/OD600值评价菌株 (图2),发现菌株分布呈现正偏态 (朝左偏) 曲线,十分陡峭,大部分丙酮酸浓度/OD600值比较接近,没有发生显著变化,平均值为2.35 g/(L·OD600),我们选择低于平均值15%的200个菌株作为潜力菌株,通过小管发酵进一步筛选获得了16个菌株,经过了再次评价,筛选出了丁醇产量得到稳定提高的3个菌株进行后续评价。

|
| 图2 突变菌株的丙酮酸浓度/OD600值 Fig.2 Pyruvate/OD600 of 1 196 mutants. |
把上述筛选得到的菌株接种于含M9培养基的试管中进行发酵评价,用HPLC对发酵产物进行分析 (表1),发现筛选得到的菌株丙酮酸含量比对照偏低,相应的丁醇产量则偏高,说明筛选策略是成功的。其中EB216-Tn5-17#、EB216-Tn5-304#、EB216-Tn5-599#丁醇产量分别提高了29%、 49%、56%。
| Strains | EB216 | EB216-Tn5-17# | EB216-Tn5-304# | EB216-Tn5-599# |
| Glucose consumed (g/L) | 6.61±1.02 | 7.64±0.62 | 7.15±0.47 | 9.00±1.46 |
| Pyruvate (g/L)* |
0.90±0.15 (0.78±0.02) |
0.88±0.10 (0.75±0.01) |
0.42±0.03 (0.34±0.01) |
0.31±0.07 (0.30±0.03) |
| Butanol (g/L) | 1.08±0.06 | 1.39±0.25 | 1.61±0.11 | 1.70±0.02 |
| Butanol yield (W/W, %) | 16 | 18 | 23 | 19 |
| OD600 | 0.75±0.04 | 0.92±0.00 | 0.93±0.03 | 1.18±0.03 |
| *: the numbers in bracket are pyruvate concentrations detected by 2,4-dinitrophenylhydrazine reaction. | ||||
通过对EB216-Tn5-17#、EB216-Tn5-304#、EB216-Tn5-599#三个菌株进行反向PCR,分别获得了1.8、2.8、1.2 kb左右的条带,经过测序,以及与大肠杆菌上的基因组进行对比,我们发现这3个菌株的插入位点分别为cadC基因 (1 539 bp) 上的124/125位点、tdk基因 (618 bp) 上的581/582位点以及pykA基因 (1 443 bp) 上的909/910位点 (表2,图3)。
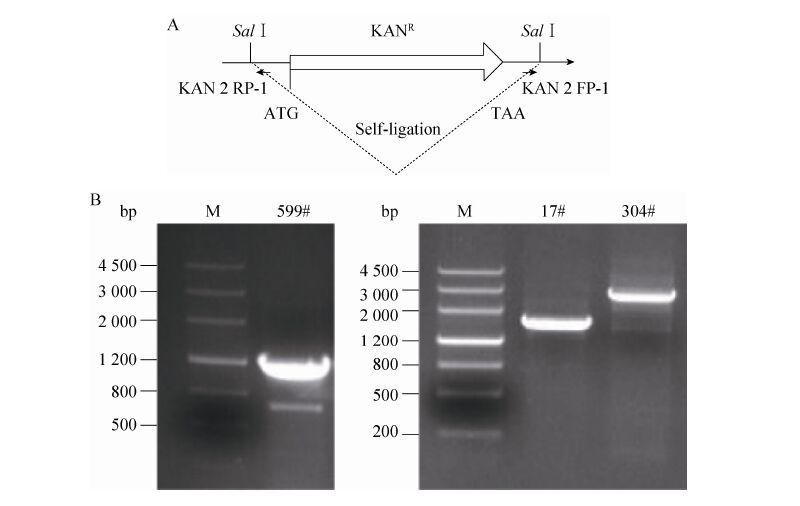
|
| 图3 利用反向PCR进行突变位点鉴定 Fig.3 Identification of insertion sites by reverse PCR. (A) Reverse PCR. (B) Reverse PCR of self-ligation. M: Maker Ⅲ. |
| Genes | pykA | cadC | tdk |
| Function | Pyruvate kinase II (glycolysis) | cadBA operon transcriptional activator (lysine metabolism) | Thymidine kinase/deoxyuridine kinase (DNA synthesis) |
生物的生理代谢是一个复杂过程,由一个整体网络中的相互作用所决定。在菌株改造实践中,除了对已知的相关基因以及代谢通路进行改造外,在基因组上同样可能存在着一些目前我们不了解以及无法预测的、却对目的产物有影响的基因。我们利用Tn5转座子对一株产丁醇的大肠杆菌工程菌株构建了一个突变体文库,对其进行基于酶标仪的快速筛选,成功获得了丁醇产量提高的突变体,基于此获得的基因靶点为构建高产菌株提供了解决方案。
本研究中发现的3个基因中,tdk基因编码胸苷激酶是DNA补救合成途径中的关键酶,又称为补救酶[15],它在Mg2+参与的情况下,催化胸苷磷酸化生成胸苷一磷酸,进而形成胸苷三磷酸,这是DNA合成中的4个必需脱氧核苷酸之一。胸苷激酶广泛存在于原核生物、真核生物以及少数病毒中[16]。已有研究发现,在经过驯化得到的生长速率加快的大肠杆菌中,hns和tdk基因之间插入了一个IS片段,转录组分析发现该菌株中tdk的转录水平显著下调[17]。这说明我们得到的tdk失活突变体菌株,可能是由于生长速率的加快导致了丁醇产量的提高。cadC是cad操纵子的转录活化子,处在cadBA的上游,负责激活cadBA编码的赖氨酸脱羧酶以及赖氨酸胺转运蛋白[18, 19]。有研究表明在沙门鼠伤寒沙门氏菌中,如果敲除cadC基因,糖酵解途径相关酶的活性会有所提高,同时cadC突变菌株更能耐酸[20]。而在大肠杆菌的耐酸进化实验 (pH 4.5−4.8) 中也发现,所有的进化种群都表现出赖氨酸脱羧酶活性的逐渐降低[21]。因此我们推测cadC突变体中的糖酵解可能是加快了,从而利用了更多的葡萄糖生产丁醇,此 外突变体的耐酸能力也有可能提升从而延长了丁醇的生产周期。pykA基因编码丙酮酸激酶A,负责催化磷酸烯醇式丙酮酸 (PEP) 形成丙酮酸以及ATP。它有一个丙酮酸激酶异构酶pykF。通常认为pykF是大肠杆菌中主要的丙酮酸激酶,而pykA所起作用相对较小[22]。然而我们通过Tn5转座子插入到pykA导致该基因的失活以及目的产物丁醇的提高,说明pykA是一个参与了重要生理过程的基因,我们推测在厌氧条件下pykA编码丙酮酸激酶是负责磷酸烯醇式丙酮酸转化为丙酮酸的关键酶,由于pykA的失活,导致了生成的丙酮酸含量降低。细胞处在相对比较“饥饿”的状态,导致细胞更多地吸收外界的葡萄糖代谢以产生足够多的丙酮酸。同时这种“饥饿感”能够保证细胞更长时间处于“兴奋”状态,以致更迟进入衰退期,于是pykA基因失活的菌株能够生成更多的丁醇。进一步生理及遗传学的研究,将可能会重新认识pykA基因的生理功能,我们也正在进行该部分工作。
Tn5转座子插入突变是一种早就认识的遗传学机制,通常被用于发现特定功能的未知基因,主要应用于微生物遗传学研究[23]。我们将这种方法应用到菌株的遗传改造过程中,为高产菌株构建提供有效的靶点。相比于理性改造,它能够提供更多、更有效的靶点,相比于传统诱变,它没有大量的无效突变背景干扰,更容易确定关键基因,因此将是代谢工程的一种有效策略。为了发挥这种策略的最大功效,我们拟继续利用Tn5转座子进行更大规模的筛选,将利用自动接种仪进行高通量培养,利用自动移液工作站对发酵液进行显色测定,通过全基因组规模的饱和突变,找出所有可以提高丁醇产量基因靶点,然后整合这些靶点到一个菌株中,构建出高效的微生物细胞工厂。
| [1] | Green EM. Fermentative production of butanol-the industrial perspective. Curr Opin Biotechnol, 2011, 22(3): 337-343. |
| [2] | Jones DT, Woods DR. Acetone-butanol fermentation revisited. Microbiol Rev, 1986, 50(4): 484-524. |
| [3] | Atsumi S, Cann AF, Connor MR, et al. Metabolic engineering of Escherichia coli for 1-butanol production. Metab Eng, 2008, 10(6): 305-311. |
| [4] | Bond-Watts BB, Bellerose RJ, Chang MCY. Enzyme mechanism as a kinetic control element for designing synthetic biofuel pathways. Nat Chem Biol, 2011, 7(4): 222-227. |
| [5] | Shen CR, Lan EI, Dekishima Y, et al. Driving forces enable high-titer anaerobic 1-butanol synthesis in Escherichia coli. Appl Environ Microbiol, 2011, 77(9): 2905-2915. |
| [6] | Dellomonaco C, Clomburg JM, Miller EN, et al. Engineered reversal of the β-oxidation cycle for the synthesis of fuels and chemicals. Nature, 2011, 476(7360): 355-359. |
| [7] | Xue C, Zhao J, Zhao JB, et al. High-titer n-butanol production by Clostridium acetobutylicum JB200 in fed-batch fermentation with intermittent gas stripping. Biotechnol Bioeng, 2012, 109(11): 2746-2756. |
| [8] | Wen M, Bond-Watts BB, Chang MCY. Production of advanced biofuels in engineered E. coli. Curr Opin Chem Biol, 2013, 17(3): 472-479. |
| [9] | Branduardi P, de Ferra F, Longo V, et al. Microbial n-butanol production from Clostridia to non-Clostridial hosts. Eng Life Sci, 2014, 14(1): 16-26. |
| [10] | Choi YJ, Lee JM, Jang YS, et al. Metabolic engineering of microorganisms for the production of higher alcohols. mBio, 2014, 5(5): e01524-14. |
| [11] | Goryshin IY, Jendrisak J, Hoffman LM, et al. Insertional transposon mutagenesis by electroporation of released Tn5 transposition complexes. Nat Biotechnol, 2000, 18(1): 97-100. |
| [12] | Reznikoff WS. Transposon Tn5. Annu Rev Genet, 2008, 42: 269-286. |
| [13] | Sambrook J, Fritsch EF, Maniatis T. Molecular Cloning: A Laboratory Manual. 2nd ed. New York: Cold Spring Harbor Laboratory Press, 1989: 119-123. |
| [14] | Eze LC, Echetebu CO. Some properties of aspartate and alanine aminotransferases from trichodermaviride. J Gen Microbiol, 1980, 120(2): 523-527. |
| [15] | Segura-Peña D, Lichter J, Trani M, et al. Quaternary structure change as a mechanism for the regulation of thymidine kinase 1-like enzymes. Structure, 2007, 15(12): 1555-1566. |
| [16] | Sandrini MPB, Clausen AR, Munch-Petersen B, et al. Thymidine kinase diversity in bacteria. Nucleos Nucleot Nucl Acides, 2006, 25(9/11): 1153-1158. |
| [17] | LaCroix RA, Sandberg TE, O'Brien EJ, et al. Use of adaptive laboratory evolution to discover key mutations enabling rapid growth of Escherichia coli K-12 MG1655 on glucose minimal medium. Appl Environ Microbiol, 2015, 81(1): 17-30. |
| [18] | Küper C, Jung K. CadC-mediated activation of the cadBA promoter in Escherichia coli. J Mol Microbiol Biotechnol, 2005, 10(1): 26-39. |
| [19] | Rhee JE, Kim KS, Choi SH. CadC activates pH-dependent expression of the Vibrio vulnificus cadBA operon at a distance through direct binding to an upstream region. J Bacteriol, 2005, 187(22): 7870-7875. |
| [20] | Lee YH, Kim BH, Kim JH, et al. CadC has a global translational effect during acid adaptation in Salmonella enterica serovar Typhimurium. J Bacteriol, 2007, 189(6): 2417-2425. |
| [21] | Harden MM, He A, Creamer K, et al. Acid-adapted strains of Escherichia coli K-12 obtained by experimental evolution. Appl Environ Microbiol, 2015, 81(6): 1932-1941. |
| [22] | Ponce E, Flores N, Martinez A, et al. Cloning of the two pyruvate kinase isoenzyme structural genes from Escherichia coli: the relative roles of these enzymes in pyruvate biosynthesis. J Bacteriol, 1995, 177(19): 5719-5722. |
| [23] | Miller WJ, Capy P. Mobile Genetic Elements. New Jersey: Humana Press, 2004: 83-96. |
 2015, Vol. 31
2015, Vol. 31




