服务
文章信息
- 李聪, 曹文广
- Cong Li, Wenguang Cao
- CRISPR/Cas9介导的基因编辑技术研究进展.
- Advances in CRISPR/Cas9-mediated gene editing
- 生物工程学报, 2015, 31(11): 1531-1542
- Chin J Biotech, 2015, 31(11): 1531-1542
- 10.13345/j.cjb.140589
-
文章历史
- Received: November 30, 2014
- Accepted:February 13, 2015
基因组靶向修饰技术是指通过某种途径对基因组DNA特定位点进行改造的一种手段。自1987年Thompsson等[1]建立起完整的ES细胞基因敲除小鼠模型至今经过20多年的发展,该技术已经建立起许多理论与方法,其中,锌指核酸酶 (Zinc-finger nucleases,ZFN) 以及类转录激活因子效应物核酸酶 (Transcription activator- like effector nucleases,TALEN) 技术的应用较为广泛。但由于这两项技术DNA结合结构域的改造较为复杂,特别是ZFN,往往需要构建庞大的锌指表达文库,筛选工作量大,成为限制其发展的瓶颈[2]。CRISPR/Cas9 (Clustered regulatory interspaced short palindromic repeats/CRISPR-associated protein 9) 技术是最近发现的一种基因组靶向修饰技术。与之前的技术相比,CRISPR/Cas9技术是通过一段RNA
来识别靶位点,因而在设计和构建上更加简单。迄今为止,CRISPR/Cas9技术已成功应用于肺炎链球菌Streptococcus pneumoniae、大肠杆菌Escherichia coli[3]、芽殖酵母Saccharomyces cerevisiae[4]、秀丽线虫Caenorhabditis elegans[5]、果蝇Drosophila melanogaster[6]、斑马鱼Danio rerio[7, 8]、小鼠Mus musculus[9]、大鼠Rattus norvegicus[10]、拟南芥[11]、玉米Zea mays[12]、水稻和小麦Triticum aestivum[13]以及包括iPS细胞在内的人体外培养细胞系[14]等多个物种内源性基因的定点修饰。本文将从CRISPR/Cas9的来源、结构以及在基因组定点修饰中的应用等方面介绍其研究进展。
1 CRISPR/Cas系统的研究历程1987年,Ishino等[15]在对大肠杆菌编码的碱性磷酸酶基因研究时发现该基因编码区下游存在一段由长度为29 bp的重复片段 (Repeats) 和32−33 bp 的非重复片段 (Spacers) 间隔相连的重复序列。之后的研究发现这种重复结构在细菌和古细菌中普遍存在[16]。随着人们对该结构的逐渐了解,2002年,这种结构被正式定义为Clustered regulatory interspaced short palindromic repeats (CRISPR)[17, 18]。生物信息序列分析发现,在人类已知物种中,有40% 的细菌和将近90%的古细菌的基因组或质粒中存在至少一个CRISPR位点[19, 20],而且其间隔序列与一些噬菌体或质粒的序列存在同源性[21],由此预测该结构可能是原核生物在进化过程中普遍存在的一种获得性免疫系统[22, 23]。随后,两个不同的研究小组分别发现该系统可以使细菌具有抵御噬菌体入侵和外源质粒转移的功 能[24, 25],揭开了探究CRISPR/Cas系统分子机制的序幕,为后续研究奠定了基础。
2 CRISPR/Cas系统的结构与分类CRISPR作为一种特殊的DNA重复序列,主要由多段长度25−50 bp的高度保守的重复片段和26−72 bp长度的间隔片段间隔串联而成。对重复片段的结构分析发现,其序列存在二重对称性,即可以形成发夹结构[26, 27]。对间隔片段的研究表明,它们可能是某些入侵遗传单位在细菌内的残留,为细菌提供一种抵抗外源DNA的特异性免疫[28]。对间隔片段的序列比对证实,约有2%的间隔片段在GenBank中能找到相匹配的质粒或噬菌体序列[29, 30] ,这可能是由于目前只有极少数噬菌体和质粒被发现并测序。在该串联结构的上游是前导序列 (Leader) 和一系列CRISPR相关蛋白基因 (CRISPR-associated genes,Cas genes) ,它们共同组成了CRISPR基因序列 (图1) 。
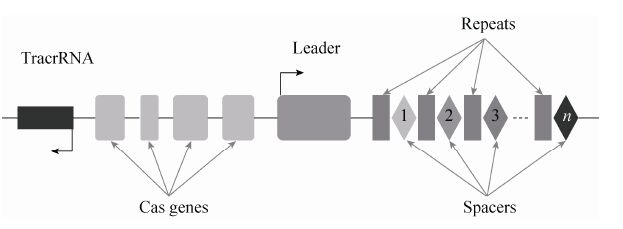
|
| 图1 CRISPR位点结构图 Fig.1 The structure of CRISPR loci. |
CRISPR/Cas系统在抵御外来噬菌体入侵时,一般经历适应、表达和干扰三个阶段[31, 32, 33]。于是,Makarova等[34, 35]根据适应阶段的高度保守性及表达和干扰阶段的差异性,将CRISPR系统分为3大类型:Ⅰ型、Ⅱ型和Ⅲ型。其中,Ⅰ型和Ⅲ型CRISPR系统介导外源DNA降 解过程中需要多种Cas蛋白参与反应,形成的切割复合体结构复杂[36, 37, 38],Ⅱ型系统组分较为简单,只需要一个Cas9蛋白来切割DNA双链。以产脓链球菌Streptococcus pyogenes为例,Cas9蛋白和两种非编码RNA:crRNA (CRISPR RNA)和tracrRNA (trans-activating chimeric RNA) 形成的复合体即可特异性降解外源DNA[39]。因此,目前以Ⅱ型CRISPR系统为基础的CRISPR/Cas9系统在基因组编辑中的应用较为广泛。
3 CRISPR/Cas9的作用机理当外源DNA入侵细菌时,其CRISPR系统会特异性地捕获一段被称为proto-spacers的外源DNA序列插入到前导序列与间隔重复序列之间,并伴随一次重复片段的复制,这样就形成了一段新的重复单元,这些重复单元就是细菌特异性免疫的结构基础。需要注意的是,外源DNA的捕获并不是完全随机的,在这些被捕获的DNA序列下游常有一段序列保守的特殊结构,被称为PAM (Proto-spacer adjacent motifs) 位点[30]。PAM位点的存在可能是CRISPR系统区分自身DNA与外源DNA而避免发生自身免疫的机制之一[40],同时也是基因编辑时靶序列选择的重要要求[31, 41, 42]。之后,形成的重复单元会被转录形成前体CRISPR RNA (Pre-crRNA),在RNase Ⅲ的作用下成熟。成熟的crRNA与tracrRNA通过碱基配对形成双链RNA结构,并进一步与Cas9蛋白结合形成靶向切割复合体对外源DNA进行特异切割,达到识别和降解外源DNA的目的。
根据上述原理,只需合成定制的crRNA,将其插入到合适的质粒中,与分别表达tracrRNA和Cas9蛋白的质粒共转染细胞,或体外转录成RNA后注射到特定细胞中,就可以建立基因敲除的细胞系或动物模型。crRNA的作用是通过碱基配对识别基因组靶序列,而不需要像ZFN和TALEN那样构建复杂的识别蛋白,极大简化了试验流程。另外,与ZFN和TALEN技术中用到的FokⅠ不同的是,Cas9蛋白存在HNH和RuvC两个活性位点[43]可以分别切割与crRNA互补的DNA链和非互补链,从而引入DSB[44],不需形成二聚体发挥作用,而且切割活性特别高。Jinek等[44]还发现,将crRNA:tracrRNA双链RNA结构改造成单链导向RNA (Single-guide RNA,sgRNA) 同样能够指导 Cas9蛋白特异切割双链DNA,这进一步提高了操作简便性 (图2)。

|
| 图2 CRISPR/Cas9介导的基因组编辑模式图 Fig.2 Schematic of genome editing by CRISPR/Cas9. |
任何一种基因编辑技术,靶位点的选择都是首要问题。前面已经提到,在CRISPR/Cas9系统中,靶序列只有在3'端存在PAM位点,它才能够被crRNA准确识别。不同来源的CRISPR/Cas9系统,PAM位点保守序列也不同,如嗜热乳链球菌Streptococcus thermophilus来源的CRISPR/Cas9系统识别的PAM位点序列为NNAGAA[45],脑膜炎奈瑟氏菌Neisseria meningitidis来源的为NNNNGATT[46],产脓链球菌来源的为NGG等。目前在各个实验室广泛应用的CRISPR/Cas9系统主要是产脓链球菌来源的,在此作重点介绍。
根据统计学意义,在生物体基因组中,应该每8 bp就会出现一个NGG PAM位点[47]。这虽然在一定程度上限制了靶位点的选择,但也提高了对靶位点的识别效率 (相当于增加了两个识别碱基)。近来,Zhang实验小组通过设计并计算大量不同sgRNA的打靶效率后,发现PAM位点序列为NAG也可被识别,但是打靶效率明显下降,仅为NGG PAM位点的1/5左 右[48]。相信随着人们对CRISPR/Cas9系统研究的深入,人们对其靶位点的选择也会有越来越清晰的认识。为了CRISPR/Cas9能够更广泛的应用,许多实验室都建立了开放的网站,免费帮助研究人员设计CRISPR/Cas9靶点。Joung团队[49]建立的ZiFiT网站 (http://zifit. partners.org/ZiFiT/),在继提供ZFN和TALEN靶点搜索之后,又增加了CRISPR/Cas9靶位点设计;Zhang研究小组[48]建立的网站 (http:// www.genome-engineering.org/) 不仅可以在用户指定的碱基序列中搜索靶位点,而且还支持人、小鼠、斑马鱼以及线虫等十几种模式生物基因组的在线比对,并列举出各个靶点潜在的脱靶位点,让用户更理性地选择靶位点。
5 CRISPR/Cas9的应用随着CRISPR/Cas9介导的基因组编辑技术的完善与发展,其在各领域的应用也在迅速展开。2012年12月,Jinek等[44]利用人工设计的crRNA形成的切割复合体,完成了对体外DNA的精确切割,是首次报道该系统可以用于目标DNA的切割。2013年初,连续有数篇研究论文报道了该方法的应用。目前,人们利用这一技术实现完成了在不同物种、多种基因的定点修饰。
5.1 在基因功能研究中的应用如何将生物体中海量的基因组信息转化成有意义的基因组功能一直是人们面临的难题之一,对于某一基因功能的验证,在个体上基因敲除是必不可少的实验环节,但是传统的基因敲除方法需要通过复杂的打靶载体构建、ES细胞筛选、嵌合体动物模型选育等一系列步骤,成功率受到多方面因素的限制。CRISPR/Cas9技术则不然,敲除基因高效快速,可在多种细胞及生物体的特定位点进行精确切割和修饰,是研究基因功能的有力工具。Shalem等[50]构建了一个含有65 000种sgRNA的文库,这些sgRNA可以靶向人类基因组中18 080种基因,几乎涵盖了每个已知的基因。他们将编码这些sgRNA的基因和编码Cas9蛋白的基因一起转运到人细胞中,从而筛选出某些有特定功能的基因。Wang等[51]更是设计了73 151种sgRNA来靶向人基因组中7 114种基因,发现被不同sgRNA靶向相同基因的细胞具有不同的存活率,并根据这些数据,推断出一些似乎控制CRISPR/Cas9系统切割效率的规则。这都证实CRISPR/Cas9极其适合作为一种在全基因组范围内探究特定基因功能的工具。
5.2 在模式生物构建中的应用ES细胞是传统方法构建生物模型的重要限制条件,而CRISPR/Cas9技术不需要经过ES细胞就可以在短时间内产生突变个体,这或许可以使得遗传学研究不再局限于具有ES细胞的有限模式生物,从而再次改变遗传工程动物模型的构建方式,甚至可能重新定义哪些物种可作为模式生物。
目前CRISPR/Cas9技术已经在微生物、植物、动物及各种细胞中实现了应用 (表1)。
| No. | Species | Genes | References |
| 1 | Homo sapiens | AASV1, EMX1, PVALB | [14] |
| 2 | Streptococcus pneumoniae | srtA, bgaA | [3] |
| 3 | Escherichia coli | rpsL | [3] |
| 4 | Saccharomyces cerevisiae | CAN1, LYP1, ADE2 | [4] |
| 5 | Toxoplasma gondii | rop18 | [52] |
| 6 | Caenorhabditis elegans | dpy, klp | [5] |
| 7 | Drosophila melanogaster | yellow, white | [6] |
| 8 | Danio rerio | drd3, fh, etsrp | [7 ] |
| 9 | Xenopus tropicalis | ptf1a/p48, hhex, pat | [53] |
| 10 | Mus musculus | Th | [9] |
| 11 | Rattus norvegicus | Mc4r, Mc4r, Tet1 | [10] |
| 12 | Macaca fascicularis | Ppar-γ, Rag1 | [54] |
| 13 | Sus scrofa | vWF | [55] |
| 14 | Bos taurus | NANOG | [56] |
| 15 | Ovis arise | MSTN | [57] |
| 16 | Oryza sativa | ROC5, CAO1, PDS | [13] |
| 17 | Triticum aestivuml | MLO | [13] |
| 18 | Arabidopsis thaliana | BRI1, JAZ1 | [11] |
| 19 | Nicotiana benthamiana | PDS | [58] |
| 20 | Zea mays | MRP4, IPK | [12] |
传统的转基因技术改造和培育新品种一般采用外源基因随机整合的方式,转基因表达的可控性差。CRISPR/Cas9技术则可以进行定点修饰,达到高效定向。Shan等[13]用CRISPR/Cas9技术敲掉了一个小麦基因,得到了耐白粉病 (Powdery mildew) 小麦新品种。该团队还利用CRISPR/Cas9技术定点突变了水稻和小麦两种作物的PDS和MLO等5个基因。原生质体中基因突变效率为14.5%−38.0%,水稻转基因植物中突变效率4.0%−9.4%,并且在T0代获得了水稻纯合PDS突变体,呈现预期的白化和矮小表型。Han等[57]利用该技术成功获得了肌肉生长抑制素 (Myostatin,MSTN) 基因敲除的绵羊。
另外,猪、牛、羊等大型家畜的基因单核苷酸多态性 (Single nucleotide polymorphism,SNP) 也能够显著影响其生产性能。例如,绵羊MSTN基因3'端非翻译区的SNP导致肌肉生长抑制因子在肌肉中的表达下降,显著促进绵羊肌肉的增长[59]。由此,我们可以利用CRISPR/Cas9技术对基因组进行精确地编辑,引入我们需要的SNP,这不仅降低了转基因的不稳定性,而且大大缩短了育种年限。有实验证明CRISPR/Cas9可以简便高效地实现多个位点切割[60],因此可以实现一次将多个SNP同时引入到种畜基因组中,从而再次提高育种效率。
目前,本课题组已经利用CRISPR/Cas9技术成功敲除了绵羊胎儿成纤维细胞中的MSTN基因,并成功建系,验证了该系统在绵羊基因编辑应用中的可行性。另外,我们还以MSTN基因缺失细胞为供体通过核移植方法生产了MSTN基因失活胚胎,并完成了相当数量的胚胎移植,预计近期可获得MSTN基因敲除的绵羊个体。考虑到转基因生物安全的问题,我们下一步还将利用体外转录获得Cas9蛋白mRNA与sgRNA一起注射到受精卵,胚胎移植后获得基因敲除绵羊,以消除转染质粒随机整合到个体基因组中的潜在风险。
5.4 在基因治疗中的应用从基因编辑技术诞生之初,人们就试图将正常的基因片段引入细胞中替代有缺陷的基因,以此来达到基因治疗的目的。但是传统方法的随机整合率高,难以精确控制。利用CRISPR/Cas9能够对特异的核苷酸序列切割,产生定点突变的性质,为人类遗传性疾病的治疗带来了新的思路。
Wu等[61]利用CRISPR/Cas9取代了引起小鼠白内障的一个单碱基对突变。他们利用该技术在受精卵突变等位基因处切割DNA,并向其提供野生型等位基因作为修复模板,从而纠正了破坏的等位基因序列。实验表明,33%注入 CRISPR/Cas9的突变体受精卵长成了无白内障小鼠。Clevers研究组[62]利用CRISPR/Cas9在人类干细胞中纠正了与囊性纤维化有关的缺陷。囊性纤维化跨膜转导受体 (Cystic fibrosis trans-membrane conductor receptor,CFTR) 基因在人体中缺失可导患者该受体蛋白错误折叠,研究人员通过CRISPR/Cas9技术及一个包含插入修补序列的供体质粒,纠正了来自囊性纤维化患者细胞样品的培育肠干细胞的遗传缺陷,这些细胞约有一半培育成了功能正常的微型肠,但是这一技术的效率还是很低,因为用于临床这一效率应该达到100%。
在感染哺乳动物细胞时,为了不被宿主免疫系统所发现,细菌会通过CRISPR系统关闭自身脂蛋白的合成[63]。因此,如何通过抑制入侵菌种的CRISPR系统并使宿主免疫系统能够识别,为相关疫苗的研发提供了一个很好的思路。2013年11月,数位CRISPR/Cas9研究领域的专家创办了一家名为Editas Medicine的公司,旨在研究直接修改与疾病有关的基因来治疗疾病的方法[64]。这是该技术在基因治疗领域发展的里程碑式的事件。
6 CRISPR/Cas9与ZFN和TALEN比较ZFN、TALEN和CRISPR/Cas9系统都可以通过用户定制而对复杂基因组进行定点编辑,这也是它们最吸引人的地方。ZFN和TALEN作为里程碑式的技术,至今仍在广泛应用。而CRISPR/Cas9作为新一代的基因编辑技术,有着ZFN和TALEN无可比拟的优势:1) 可用位置更多。理论上,在基因组中每8 bp就有一个适合CRISPR/Cas9编辑的位点,分布频率很高。而对于ZFN和TALEN,在基因组中平均分别要500 bp和125 bp才会有一个合适的编辑位点[47]。2) 可同时编辑多个位点。Wang等[60]利用CRISPR/Cas9技术一次性构建了含5个突变位点转基因鼠;Zhou团队[10]也构建了同时包含3个突变位点的基因敲除大鼠,而且3基因同时敲除的效率高达59.1%。这对于ZFN和TALEN两项技术而言是难以实现的。3) 识别域的构建更简单。在CRISPR/Cas9系统中,想要改变靶序列识别位点只需改变一段短的RNA序列即可。而ZFN和TALEN则需要根据不同的识别序列组装和构建十分复杂的蛋白识别域,费时费力,所以CRISPR/Cas9技术更容易得到推广和应用。
7 CRISPR/Cas9技术存在的问题脱靶效应是存在于所有基因组靶向修饰技术中的一道难题,它会对基因组非特性序列进行切割,造成未知突变,增加后期的鉴定工作量。CRISPR/Cas9系统中,对靶序列的识别主要是依靠一段20 bp的短RNA,但是研究表明当存在单个甚至多达5个碱基错配时,切割仍能正常发生[65]。进一步研究发现,在这20 bp中只有位于PAM位点前12 bp的种子序列对靶位点识别影响较大,即总共只有14 bp (PAM中的GG和种子序列) 是靶位点识别的关键序 列[66, 67]。这在生物体庞大的基因组中很容易出现脱靶位点,从而引入意外突变。除此之外,Vikram等[68]研究发现,高浓度的Cas9蛋白和sgRNA也会导致较高的脱靶活性,脱靶活性最高可达84%。
针对脱靶效率高的问题,Patrick等[48]构建了一个计算机模型,利用该模型可以对靶序列进行全基因组比对,筛选出特异性最高的靶序列,再结合调整sgRNA的剂量以达到高打靶、低脱靶的效果。Ran等[69]通过突变Cas9核酸酶的DNA切割结构域,将CRISPR/Cas9系统改造为切口酶,然后配对使用,能够使脱靶活性降低50−1500倍,但是打靶效率也随之降低。
8 展望与结语CRISPR/Cas9技术的出现,极大地提高了人们对基因序列修饰与编辑的能力,除此之外,该技术在其他方面也显示出很好的应用前景。Qi等[70]将Cas9蛋白的两个切割结构域都进行了突变,使其彻底失去内切酶活性,但与sgRNA形成的复合物仍具有特异结合靶位点的能力。当该复合物与靶位点结合后,虽不能切割靶位点,但却抑制了RNA聚合酶或其他转录因子的结合,在转录水平上下调了基因表达,起到类似于RNA干扰的作用。据此,我们还可以在突变的Cas9蛋白末端连接其他功能蛋白,如DNA乙酰化酶或VP64转录激活结构域,可以实现对靶位点乙酰化水平或基因表达水平的调控。当然,这能否按照我们预期的那样发挥作用还有待于进一步研究。
最近,Martin等[71]首次利用X射线晶体衍射技术成像了这种酶的精细三维结构,这对人们改良Cas9蛋白提供了巨大帮助。另外,研究人员首次发现利用专门设计的PAM递呈寡核苷酸 (PAM-presenting oligonucleotides,PAMmers),可以使Cas9蛋白特异结合或切割RNA靶点,同时避开对应的DNA序列,这一发现无疑为RNA的功能研究、分析和操控提出了新的思路,同时又进一步提高该技术的应用价值[72]。
当然,目前关于CRISPR/Cas9系统还有许多亟待解决的问题,如CRISPR/Cas9系统的基因组编辑效率是否高于ZFN和TALEN技术;Cas9蛋白作为细菌蛋白,是否会引起其他物种的免疫反应;如何更好地提高同源重组介导的基因编辑效率等。相信随着人们深入和全面的研究,CRISPR/Ca9技术将会展现出其无比强大的生命力。
| [1] | Thompsson KR, Capecchi MR. Site-directed mutagenesis by gene targeting in mouse embryo- derived stem cells. Cell, 1987, 51(3): 503-512. |
| [2] | Thomas G, Charles A, Carlos F. ZFN, TALEN, and CRISPR/Cas based methods for genome engineering. Trends Biotechnol, 2013, 31(7): 397-405. |
| [3] | Jiang W, Bikard D, Cox D. RNA-guided editing of bacterial genomes using CRISPR-Cas systems. Nat Biotechnol, 2013, 31(3): 233-239. |
| [4] | James E, Julie EN, Prashant M. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Res, 2013, 41(7): 4336-4343. |
| [5] | Friedland AE, Tzur YB, Esvelt KM, et al. Heritable genome editing in C. elegans via a CRISPR-Cas9 system. Nat Methods, 2013, 10(8): 741-743. |
| [6] | Andrew RB, Charlotte T, Chris PP. Highly efficient targeted mutagenesis of Drosophila with the CRISPR/Cas9 system. Cell Rep, 2013, 4(1): 220-228. |
| [7] | Chang N, Sun C, Gao L. Genome editing with RNA-guided Cas9 nuclease in zebrafish embryos. Cell Res, 2013, 23(4): 465-472. |
| [8] | Jao LE, Wente SR, Chen W. Efficient multiplex biallelic zebrafish genome editing using a CRISPR nuclease system. Proc Natl Acad Sci USA, 2013, 110(34): 13904-13909. |
| [9] | Shen B, Zhang J, Wu H. Generation of gene-modified mice via Cas9/RNA-mediated gene targeting. Cell Res, 2013, 23(5): 720-723. |
| [10] | Wei L, Fei T, Tianda L, et al. Simultaneous generation and germline transmission of multiple gene mutations in rat using CRISPR-Cas systems. Nat Biotechnol, 2013, 31(8): 684-686. |
| [11] | Feng ZY, Zhang BT, Ding WN. Efficient genome editing in plants using a CRISPR/Cas system. Cell Res, 2013, 23(10): 1229-1232. |
| [12] | Liang Z, Zhang K, Chen KL, et al. Targeted mutagenesis in Zea mays using TALENs and the CRISPR/Cas system. J Genet Genomics, 2014, 41(2): 63-68. |
| [13] | Shan QW, Wang Y, Li J, et al. Targeted genome modification of crop plants using a CRISPR-Cas system. Nat Biotechnol, 2013, 31(8): 686-688. |
| [14] | Mali P, Yang L, Esvelt KM, et al. RNA-guided human genome engineering via Cas9. Science, 2013, 339(6121): 823-826. |
| [15] | Ishino Y, Shinagawa H, Makino K. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli and identification of the gene product. J Bacteriol, 1987, 169(12): 5429-5433. |
| [16] | Lillestøl R, Redder P, Garrett RA. A putative viral defence mechanism in archaeal cells. Archaea, 2006, 2(1): 59-72. |
| [17] | Coffey A, Ross RP. Bacteriophage-resistance systems in dairy starter strains, molecular analysis to application. Anton Leeuw, 2002, 82(1): 303-321. |
| [18] | Jansen R, Embden JD, Gaastra W. Identification of genes that are associated with DNA repeats in prokaryotes. Mol Microbiol, 2002, 43(6): 1565-1575. |
| [19] | Grissa I, Vergnaud G, Pourcel C. The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats. BMC Bioinformatics, 2007, 8(1): 172-181. |
| [20] | Klug A. The discovery of zinc fingers and their applications in gene regulation and genome manipulation. Q Rev Biophys, 2010, 79: 213-231. |
| [21] | Boch J, Scholze H, Schornack S. Breaking the code of DNA binding specificity of TAL-type III effectors. Science, 2009, 326(5959): 1509-1512. |
| [22] | Bolotin A, Quinquis B, Sorokin A. Clustered regularly interspaced short palindrome repeats (CRISPRs) have spacers of extra-chromosomal origin. Microbiology, 2005, 151(8): 2551-2561. |
| [23] | Mojica FJ, Diez VC, Garcia MJ. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements. J Mol Evol, 2005, 60(2): 174-182. |
| [24] | Barrangou R, Fremaux C, Deveau H. CRISPR provides acquired resistance against viruses in prokaryotes. Science, 2007, 315(5819): 1709-1712. |
| [25] | Marraffini LA, Sontheimer EJ. CRISPR interference limits horizontal gene transfer in Staphylococci by targeting DNA. Science, 2008, 322(5909): 1843-1845. |
| [26] | Jansen R, Embden JD, Gaastra W. Identification of genes that are associated with DNA repeats in prokaryotes. Mol Microbiol, 2002, 43(6): 1565-1575. |
| [27] | Kunin V, Sorek R, Hugenholtz P. Evolutionary conservation of sequence and secondary structures in CRISPR repeats. Genome Biol, 2007, 8(4): R61. |
| [28] | Pouecel C, Salvignol G, Vergnaud G. CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies. Microbiology, 2005, 151(3): 653-663. |
| [29] | Mojica FJ, Ferrer C, Juez G. Long stretches of short tandem repeats are present in the largest replicons of the Archaea Halofera mediterranei and Halofera volcanii and could be involved in replicon partitioning. Mol Microbiol, 1995, 17(1): 85-93. |
| [30] | Heidelberg JF, Nelson WC, Schoenfeld T. Germ warfare in a microbial matcommunity: CRISPR provide insights into the co-evolution of host and viral genomes. PLoS ONE, 2009, 4(1): e4169. |
| [31] | Brouns SJ, Jore MM, Lundgren M. Small CRISPR RNAs guide antiviral defense in prokaryotes. Science, 2008, 321(5891): 960-964. |
| [32] | Garneau JE, Dupuis M, Villion M. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature, 2010, 468(7320): 67-71. |
| [33] | Haft DH, Selengut J, Mongodin EF, et al. A guild of 45 CRISPR-associated (Cas) protein families and multiple CRISPR/Cas subtypes exist in prokaryotic genomes. PLoS Comput Biol, 2005, 1(6): 474-483. |
| [34] | Makarova KS, Haft DH, Barrangou R. Evolution and classification of the CRISPR-Cas systems. Nat Rev Microbiol, 2011, 9(6): 467-477. |
| [35] | Makarova KS, Aravind L, Wolf YI, et al. Unification of Cas protein families and a simple scenario for the origin and evolution of CRISPR-Cas systems. Biol Direct, 2011, 6(38): 1-27. |
| [36] | Gesner EM, Schellenberg MJ, Garside EL. Recognition and maturation of effector RNAs in a CRISPR interference pathway. Nat Struct Mol Biol, 2011, 18(6): 688-692. |
| [37] | Sashital DG, Jinek M, Doudns JA. An RNA-induced conformational change required for CRISPR RNA cleavage by the endoribonuclease Cse3. Nat Struct Molr Biol, 2011, 18(6): 680-687. |
| [38] | Lintner NG, Frankel KA, Tsutakawa SE. The structure of the CRISPR-associated protein Cas3 provides insight into the regulation of the CRISPR/Cas system. J Mol Biol, 2011, 405(4): 939-955. |
| [39] | Deltcheva E, Chylinski K, Sharma CM. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III. Nature, 2011, 471(7340): 602-607. |
| [40] | Sternberg SH, Redding S, Jinek M, et al. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature, 2014, 507(7490): 62-67. |
| [41] | Beloglazova N, Brown G, Zimmerman MD. A novel family of sequence-specific endoribonucleases associated with the clustered regularly interspaced short palindromic repeats. J Biochem, 2008, 283(29): 20361-20371. |
| [42] | Semenova E, Nagornykh M, Pyatnitskiy M. Analysis of CRISPR system function in plant pathogen Xanthomonas oryzae. FEMS Microbiol Lett, 2009, 296(1): 110-116. |
| [43] | Villion M, Moineau S. The double-edged sword of CRISPR-Cas systems. Cell Res, 2013, 23(1): 15-17. |
| [44] | Jinek M, Chylinski K, Fonfara I. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337(6096): 816-821. |
| [45] | Kevin ME, Prashant M, Jonathan LB. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing. Nat Methods, 2013, 10(11): 1116-1121. |
| [46] | Hou Z, Zhang Y, Propson NE. Efficient genome engineering in human pluripotent stem cells using Cas9 from Neisseria meningitidis. Proc Natl Acad Sci USA, 2013, 110(39): 15644-15649. |
| [47] | Carlson DF, Walton MW, Fahrenkrug SC. Precision editing of large animal genomes. Adv Genet, 2012, 80: 37-97. |
| [48] | Patrick DH, David A, Joshua AW, et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol, 2013, 31(9): 827-832. |
| [49] | Hwang WY, Fu Y, Reyon D, et al. Efficient genome editing in zebrafish using a CRISPR-Cas system. Nat Biotechnol, 2013, 31(3): 227-229. |
| [50] | Shalem O, Sanjana NE, Hartenian E. Genome-scale CRISPR-Cas9 knockout screening in human cells. Science, 2014, 343(6166): 84-87. |
| [51] | Wang T, Wei JJ, Sabatini DM. Genetic screens in human cells using the CRISPR/Cas9 system. Science, 2014, 343(6166): 80-84. |
| [52] | Shen B, Brown KM, Lee TD. Efficient gene disruption in diverse strains of Toxoplasma gondii using CRISPR/Cas9. MBio, 2014, 5(3): e01114. |
| [53] | Guo X, Zhang T, Hu Z. Efficient RNA/Cas9-mediated genome editing in Xenopus tropicalis. Development, 2014, 141(3): 707-714. |
| [54] | Niu Y, Shen B, Cui YQ. Generation of gene modified cynomolgus monkey via Cas9/RNA mediated gene targeting in one-cell embryos. Cell, 2014, 156(4): 836-843. |
| [55] | Hai T, Teng F, Guo RF, et al. One-step generation of knockout pigs by zygote injection of CRISPR/Cas system. Cell Res, 2014, 24(3): 372-375. |
| [56] | Heo YT, Quan X, Xu YN, et al. CRISPR/Cas9 nuclease-mediated gene knock-in in bovine- induced pluripotent cells. Stem Cells Dev, 2015, 24(3): 393-402. |
| [57] | Han HB, Ma YH, Wang T, et al. One-step generation of myostatin gene knockout sheep via the CRISPR/Cas9 system. Front Agr Sci Eng, 2014, 1(1): 2-5. |
| [58] | Li JF, Norville JE, Aach J, et al. Multiplex and homologous recombination-mediated genome editing in Arabidopsis and Nicotiana benthamiana using guide RNA and Cas9. Nat Biotechnol, 2013, 31(8): 688-691. |
| [59] | Clop A, Marcq F, Takeda H. A mutation creating a potential illegitimate microRNA target site in the myostatin gene affects muscularity in sheep. Nat Genet, 2006, 38(7): 813-818. |
| [60] | Wang H, Yang H, Shivalila CS. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. Cell, 2013, 153(4): 910-918. |
| [61] | Wu Y, Liang D, Wang YH. Correction of a genetic disease in mouse via use of CRISPR-Cas9. Cell Stem Cell, 2013, 13(6): 659-662. |
| [62] | Schwank G, Koo BK, Sasselli V, et al. Functional repair of CFTR by CRISPR/Cas9 in intestinal stem cell organoids of cystic fibrosis patients. Cell Stem Cell, 2013, 13(6): 653-658. |
| [63] | Sampson TR, Saroj SD, Llewellyn AC. A CRISPR-CAS system mediates bacterial innate immune evasion and virulence. Nature, 2013, 497(7448): 254-257. |
| [64] | Helen S. CRISPR technology leaps from lab to industry. J Nematol, 2014, 46(2): 130-260. |
| [65] | Fu Y, Foden JA, Khayter C. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat Biotechnol, 2013, 31(9): 822-826. |
| [66] | Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems. Science, 2013, 339(6121): 819-823. |
| [67] | Cem K, Sevki A, Ritambhara S, et al. Genome-wide analysis reveals characteristics of off-target sites bound by the Cas9 endonuclease. Nat Biotechnol, 2014, 32(7): 677-683. |
| [68] | Vikram P, Steven L, John PG, et al. High- throughput profiling of off-target DNA cleavage reveals RNA-programmed Cas9 nuclease specificity. Nat Biotechnol, 2013, 31(9): 839-843. |
| [69] | Ran FA, Hsu PD, Lin CY, et al. Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity. Cell, 2013, 154(6): 1380-1389. |
| [70] | Qi LS, Larson MH, Gilbert LA, et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell, 2013, 152(5): 1173-1183. |
| [71] | Martin J, Fuguo J, David WT, et al. Structures of Cas9 endonucleases reveal RNA-mediated conformational activation. Science, 2014, 43(6176): 1247997. |
| [72] | O'Connell MR, Oakes BL, Sternberg SH, et al. Programmable RNA recognition and cleavage by CRISPR/Cas9. Nature, 2014, 516(7530): 263-266 |
 2015, Vol. 31
2015, Vol. 31




