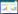服务
文章信息
- 张贤, 王建红, 喻曼, 曹凯, 庄俐, 徐昌旭, 曹卫东
- Zhang Xian, Wang Jianhong, Yu Man, Cao Kai, Zhuang Li, Xu Changxu, Cao Weidong
- 基于RNA-seq 的能源植物芒转录组分析
- Transcriptome analysis of bioenergy plant Miscanthus sinensis Anderss by RNA-Seq
- 生物工程学报, 2012, 31(10): 1437-1448
- Chin J Biotech, 2012, 31(10): 1437-1448
- 10.13345/j.cjb.150026
-
文章历史
- Received: January 16, 2015
- Accepted: March 30, 2015
2. 江西省农业科学院土壤肥料与资源环境研究所,江西 南昌 330200
3. 中国农业科学院农业资源与农业区划研究所,北京 100081
2. Institute of Soil,Fertilizer,and Environment Resource,Jiangxi Academy of Agricultural Sciences,Nanchang 330200,Jiangxi,China
3. Institute of Agricultural Resources and Regional Planning,Chinese Academy of Agricultural Sciences,Beijing 100081,China
芒 (Miscanthus sinensis Anderss),禾本科黍亚科,原产于东亚,是一种具有木质地下茎的多年生C4草本植物,自然分布从东南亚到中国、日本,直至玻利尼西亚,有一些种在非洲也有生长[1]。芒植株高大,茎秆粗壮,根茎发达,具有产量高[2],光照、水分和氮素利用率高[3],不易受病虫害侵染等特点,对不适于粮食作物生产的边际土地适应性强[4, 5],可为能量和纤维素产品生产提供高品质的木质纤维素材料,是一种理想的能源植物[6, 7]。作为一种新兴作物,芒基因资源及遗传改良的资料还非常有限[8, 9],至今芒基因组学的研究仍十分缺乏,严重阻碍了芒的遗传改良[10],对于该作物基因资源的研究也有待进一步深入。我国是世界芒的多样性中心,但大量优良的种质资源尚处于野生状态,未被驯化栽培,丰富的基因资源没有被有效利用。
转录组测序技术 (又称RNA-Seq) 可以在没有完整基因组序列的前提下,研究所有的mRNA 转录本的丰度信息,发掘新的转录本和可变剪接体[11, 12],且可以得到定量更准确、分析更可靠、重复性更高及检测范围更广的结果[13]。
选取我国野生芒种质资源,运用RNA-Seq技术,对芒花芽和叶芽转录组进行测序,测序得到的大量Unigene 进行GO、COG 和KEGG 分类统计,给出功能注释和Pathway 注释。研究旨在挖掘我国野生芒种质中的珍贵基因资源,发现芒控制优良性状的重要功能基因,为芒的基因改良提供理论依据和物质基础。
1 材料与方法 1.1 材料野生芒种质资源采集于浙江省临安市郊区,生境为林缘边际土地,于孕蕾期在其生长地直接取花芽和叶芽,分别经液氮速冻后储存于-70 ℃超低温冰箱备用。
1.2 方法 1.2.1 文库构建及测序采用通用植物总RNA 提取试剂盒提取芒花芽和叶芽总RNA,琼脂糖凝胶电泳检测RNA 完整性,Agilent 2100 Bioanalyzer检测总RNA 浓度。用带有Oligo(dT) 的磁珠富集mRNA;加入片段化缓冲液将mRNA打断成短片段,以打断后的mRNA为模板合成一链cDNA,然后加入缓冲液、dNTPs、RNase H和DNA polymeraseⅠ配制二链合成反应体系合成二链cDNA,经过PCR 扩增,建立测序文库;构建好的文库用Agilent 2100 Bioanalyzer和ABI StepOnePlus Real-Time PCR System质检合格后,使用Illumina HiSeq™ 2000进行测序。
1.2.2 数据分析对测序后得到的原始数据total Raw reads进行质量分析,去除重复、含接头、测序质量低的reads,获得Clean reads。使用短reads组装软件Trinity[10]做转录组从头组装。首先将具有一定长度重叠的reads连成更长的片段,通过reads重叠关系得到的组装片段Contig。然后,将reads比对回Contig,通过paired-end reads确定来自同一转录本的不同Contig以及这些Contig之间的距离,Trinity将这些Contig连在一起,最后得到两端不能再延长的序列,即为Unigene。
1.2.3 功能注释利用Blastx 将Unigene 序列与NR (Non-redundant Protein Sequence Database in GenBank)、Swiss-Prot (Swiss-Prot Protein Sequence Database)、KEGG (Kyoto Encyclopedia of Genes and Genomes)和COG (Cluster of Orthologous Groups of proteins) 数据库进行比对 (Evalue<1e-5),获取与Unigene 具有最高序列相似性的蛋白,从而得到该Unigene 的蛋白功能注释信息。根据NR 注释信息,使用Blast2GO 软件进行GO 注释,得到每个Unigene 的GO 信息后,用WEGO 软件进行GO 功能分类统计。
按Nr、SwissProt、KEGG、COG 的优先级顺序将Unigene 序列与以上蛋白库进行Blastx比对 (Evalue<1e-5),取比对结果中等级最高的蛋白确定该Unigene的编码区序列,然后根据标准密码子表将编码区序列翻译成氨基酸序列,从而得到该Unigene编码区的核酸序列 (序列方向5'-3') 和氨基酸序列。最后,跟以上蛋白库皆比对不上的Unigene我们用软件ESTScan预测其编码区,得到其编码区的核酸序列 (序列方向5'-3') 和氨基酸序列。
2 结果与分析 2.1 芒花芽和叶芽转录组测序产量芒花芽和叶芽文库测序得到的Raw reads 及去除杂质过滤之后的Clean reads 统计见表1,后续分析均基于Clean reads。花芽和叶芽分别生成68 136 340个和68 452 222个Clean reads,总数量均高于68 M。花芽和叶芽Q20分别为97.78%和97.62%,Q20 比例大于80%,N 均为0.01%,比例小于0.5%,GC 比例为49.73%和51.43%,在35%-65%之间,结果表明测序质量较好,满足下一步分析的要求。
| Samples | Total raw reads | Total clean reads | Total clean nucleotides (nt) | Q20 (%) | N (%) | GC (%) |
| Leaf_bud | 87 002 888 | 68 452 222 | 6 160 699 980 | 97.62 | 0.01 | 51.43 |
| Flower_bud | 83 541 566 | 68 136 340 | 6 132 270 600 | 97.78 | 0.01 | 49.73 |
经过Trinity 从头组装,芒花芽和叶芽测序文库分别获得重叠群Contig、Unigene及All-Unigene。花芽和叶芽各获得180 118个和159 514个Contig (表2),平均长度分别为314 nt和319 nt,花芽Contig个数高于叶芽;经过拼接最终获得98 326个All-Unigene,总长度 80 794 573 nt,平均长度822 nt,N50长1 337 nt。
| Sample | Total number | Total length (nt) | Mean length (nt) | N50 | Total consensus sequences | Distinct clusters | Distinct singletons | |
| Contig | Leaf_bud | 159 514 | 50 818 390 | 319 | 551 | - | - | - |
| Flower_bud | 180 118 | 56 509 785 | 314 | 510 | - | - | - | |
| Unigene | Leaf_bud | 87 257 | 58 733 614 | 673 | 1 198 | 87 257 | 31 216 | 56 041 |
| Flower_bud | 104 594 | 73 638 943 | 704 | 1 266 | 104 594 | 40 275 | 64 319 | |
| All | 98 326 | 80 794 573 | 822 | 1 337 | 98 326 | 42 810 | 55 516 |
组装序列长度是组装质量的一个评估标准。对组装出来的All-Unigene 进行长度分布特征分析 (表3)。All-Unigene 的长度均大于 200 bp,长度为100-500 bp 的Unigene 所占比例最大,约占50.67%; 长度大于1 000 bp 的Unigene比例达到28.3%。与花芽和叶芽相比,进一步组装后的All-Unigene短序列减少了,而长序列分布增多。
| Sample | Number | 100-500 nt | 500-1 000 nt | 1 000-1 500 nt | 1 500-2 000 nt | ≥2 000 nt |
| Leaf_bud | Number | 53 031 | 15 374 | 8 561 | 5 019 | 5 272 |
| % | 60.78 | 17.62 | 9.81 | 5.75 | 6.04 | |
| Flower_bud | Number | 61 630 | 18 422 | 11 021 | 6 593 | 6 928 |
| % | 58.92 | 17.61 | 10.54 | 6.30 | 6.62 | |
| All | Number | 49 818 | 20 944 | 11 998 | 7 346 | 8,220 |
| % | 50.67 | 21.30 | 12.20 | 7.47 | 8.36 |
为了预测Unigene功能,分别将Unigene与主要的生物学数据库NR、NT、SwissProt、KEGG、COG、GO 库进行比对。通过Blast 搜索比对 (表4),共有74 134 条Unigene 获得了基因注释,占All-Unigene的75.40%;有24 192 条Unigene (24.6%) 未被注释。NT数据库比对注释的信息最多,注释了70 122 条Unigene,COG 注释的信息最少,仅23 653 条Unigene得到了注释。在与蛋白数据库有同源比对信息的Unigene中,比对到同源序列比例最高的物种分别为高粱 (37 731,60.86%)、玉米 (16 258,26.22%)、水稻 (3 065,4.94%) 占所有同源序列的92.02%;其中,相似性95%以上的19 009个,80%-95%的2 577个,60%-80%的8 997个,相似性60%以上的占总注释Unigene数的85%以上。
| Sequence file | NR | NT | Swiss-Prot | KEGG | COG | GO | ALL |
| Unigene | 62 000 | 70 122 | 37 977 | 36 710 | 23 653 | 45 507 | 74 134 |
| Annotated/All annotated (%) | 83 63 | 94.59 | 51.23 | 49.52 | 31.91 | 61.38 | 100.00 |
| Annotated /All-Unigene (%) | 63.06 | 71.32 | 38.62 | 37.33 | 24.06 | 46.28 | 75.40 |
将Unigene和COG数据库比对,对其所编码的蛋白进行直系同源分类。23 653条All-Unigene被分成了25个类别 (图1)。其中,比对到一般功能预测 (General function prediction only) 的基因数量最多 (8 506,35.96%),其次是未知功能基因 (Function unknown) (6 275,26.53%),转录 (Transcription) (5 958,25.19%)及复制、重组和修复 (Replication,recombination and repair) (5 487,23.20 %);而参与核酸结构 (Nuclear structure) (10,0.04%)和细胞外结构 (Extracellular structures) (40,0.17%) 分类的基因数目较少。

|
| 图1 COG 功能分类表 Fig.1 COG classification. |
GO (Gene ontology) 是一个国际标准化的基因功能分类体系,根据NR数据库注释的信息,有45 507条All-Unigene映射到GO不同的功节点 (Term) 上,使用Blast2GO软件将这些注释的基因按照基因的分子功能 (Molecular function)、参与的生物过程 (Biological process)和所处的细胞位置 (Cellular component) 进行分类 (图2),从宏观上认识芒基因功能分布特征,全方位地注释基因信息。由于经常存在同一个转录本映射到不同节点现象,所以共有135 445条All-Unigene归入生物学过程,其中,参与细胞过程 (Cellular process) (25 147,55.26%)和代谢过程 (Metabolic process) (25 361,55.73%) 的Unigene最多,均占55%以上;有127 037条All-Unigene归入到细胞组分,细胞 (Cell) (32 798,72.07%) 和细胞构成 (Cell part) (32 798,72.07%) Unigene最多,其次是细胞器 (Organelle) (28 377,62.36%);52 742条All-Unigene归入分子功能,其中结合 (Binding) (24 070,52.89%) 和催化活性 (Catalytic activity) (21 759,47.81%) 最高,其余所占比例均在10%以下。
|
| 图2 GO 分类图 Fig.2 GO classification. |
为了系统分析测序所得到转录本在芒花和叶片形成过程中参与的代谢途径以及这些基因产物的功能,将Unigene比对到KEGG数据库,发现共有36 710个Unigene参与了128个代谢通路 (表5)。其中参与代谢途径 (Metabolic pathways) 的转录本最多 (共9 628个,占比对总数的26.23%),其次是参与RNA 转运的转录本5 066个 (13.8%),而参与mRNA surveillance 途径4 305 (11.73%) 的转录本位居三。
| Pathway | Count (36 710) | PathwayID | |
| 1 | Metabolic pathways | 9 628 | ko01100 |
| 2 | RNA transport | 5 066 | ko03013 |
| 3 | mRNA surveillance pathway | 4 305 | ko03015 |
| 4 | Biosynthesis of secondary metabolites | 3 573 | ko01110 |
| 5 | Glycerophospholipid metabolism | 2 919 | ko056 |
| 6 | Endocytosis | 2 908 | ko04144 |
| 7 | Ether lipid metabolism | 2 631 | ko00565 |
| 8 | Plant-pathogen interaction | 2 348 | ko04626 |
| 9 | Plant hormone signal transduction | 1 524 | ko04075 |
| 10 | Spliceosome | 1 505 | ko03040 |
| 11 | Purine metabolism | 1 185 | ko00230 |
| 12 | Starch and sucrose metabolism | 1 164 | ko00500 |
| 13 | Pyrimidine metabolism | 1 095 | ko00240 |
分离和鉴定芒C4核心酶基因,是深入了解和调控芒生长发育重要方法。通过同源性搜索比对,在测序结果中获得了芒基因中C4重要的酶基因。其中,功能注释为碳酸酐酶 (Carbonic anhydrase) 的Unigene 5个,磷酸烯醇丙酮酸羧激酶 (Phosphoenolpyruvate carboxylase) 10个,NADP-依赖苹果酸酶 (NADP-dependent malic enzyme) 6个,丙酮酸磷酸双激酶 (Pyruvate orthophosphate dikinas) 的基因3个 (表6)。这些Unigene 的注释信息将为进一步克隆功能基因的全长、研究其功能提供基础数据。
| geneID | Gene length | Nr-annotation | |
| CA | CL2633.Contig1_All | 1 141 | Carbonic anhydrase [Zea mays] |
| Unigene10539_All | 1 176 | Carbonic anhydrase isoform 1 [Zea mays] | |
| Unigene26976_All | 284 | Carbonic anhydrase isoform 1 [Zea mays] | |
| Unigene30101_All | 473 | Carbonic anhydrase [Zea mays] | |
| Unigene30102_All | 539 | Carbonic anhydrase [Zea mays] | |
| PEPC | Unigene28261_All | 241 | Phosphoenolpyruvate carboxylase 3 [Sorghum bicolor] |
| Unigene30692_All | 887 | Phosphoenolpyruvate carboxylase isoform 1 [Zea mays] | |
| Unigene34780_All | 324 | Phosphoenolpyruvate carboxylase kinase 1 [Zea mays] | |
| CL10194.Contig2_All | 211 | Phosphoenolpyruvate carboxylase [Panicum miliaceum] | |
| CL1659.Contig1_All | 1 585 | Phosphoenolpyruvate carboxylase 4 [Zea mays] | |
| CL1659.Contig3_All | 638 | Phosphoenolpyruvate carboxylase [Aristida rhiniochloa] | |
| CL6728.Contig1_All | 1 326 | Phosphoenolpyruvate carboxylase kinase [Sorghum bicolor] | |
| CL6728.Contig2_All | 1 302 | Phosphoenolpyruvate carboxylase kinase [Sorghum bicolor] | |
| CL6728.Contig4_All | 1 302 | Phosphoenolpyruvate carboxylase kinase [Sorghum bicolor] | |
| CL6728.Contig5_All | 523 | Phosphoenolpyruvate carboxylase kinase [Sorghum bicolor] | |
| NADP-ME | CL15033.Contig1_All | 1 138 | NADP-dependent malic enzyme [Zea mays] |
| CL15033.Contig4_All | 3 539 | NADP-dependent malic enzyme [Zea mays] | |
| CL15033.Contig5_All | 1 928 | NADP-dependent malic enzyme [Zea mays] | |
| CL15033.Contig6_All | 2 080 | NADP-dependent malic enzyme [Zea mays] | |
| CL15033.Contig8_All | 3 735 | NADP-dependent malic enzyme [Zea mays] | |
| Unigene45306_All | 352 | NADP malic enzyme4 [Zea mays] | |
| PPDK | CL12313.Contig1_All | 316 | Pyruvate orthophosphate dikinase regulatory protein [Sorghum bicolor] |
| CL12313.Contig2_All | 312 | Pyruvate orthophosphate dikinase regulatory protein [Sorghum bicolor] | |
| Unigene30741_All | 3 071 | C4-specific pyruvate orthophosphate dikinase [Miscanthus x giganteus] |
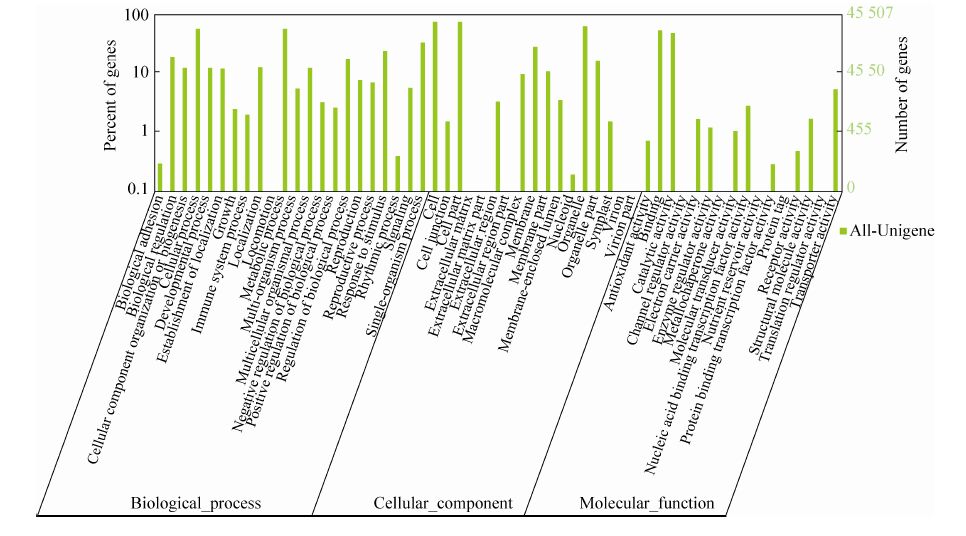
将Unigene 序列按Nr、SwissProt、KEGG 和COG数据库的优先级顺序分别进行Blastx 比对(E 值<1e-5),确定该Unigene 的编码区序列,然后根据标准密码子表将编码区序列翻译成氨基酸序列,从而得到该Unigene 编码区的核酸序列 (序列方向5′-3′) 和氨基酸序列。最后,跟以上4 个数据库皆比对不上的Unigene 用ESTscan 软件预测其编码区,得到其编码区核酸序列 (序列方向为5′-3′) 和氨基酸序列。比对上Nr、SwissProt、KEGG和COG数据库的Unigene序列,对其中的61 870个序列预测了编码蛋白框 (CDS),图3表示所预测CDS的长度
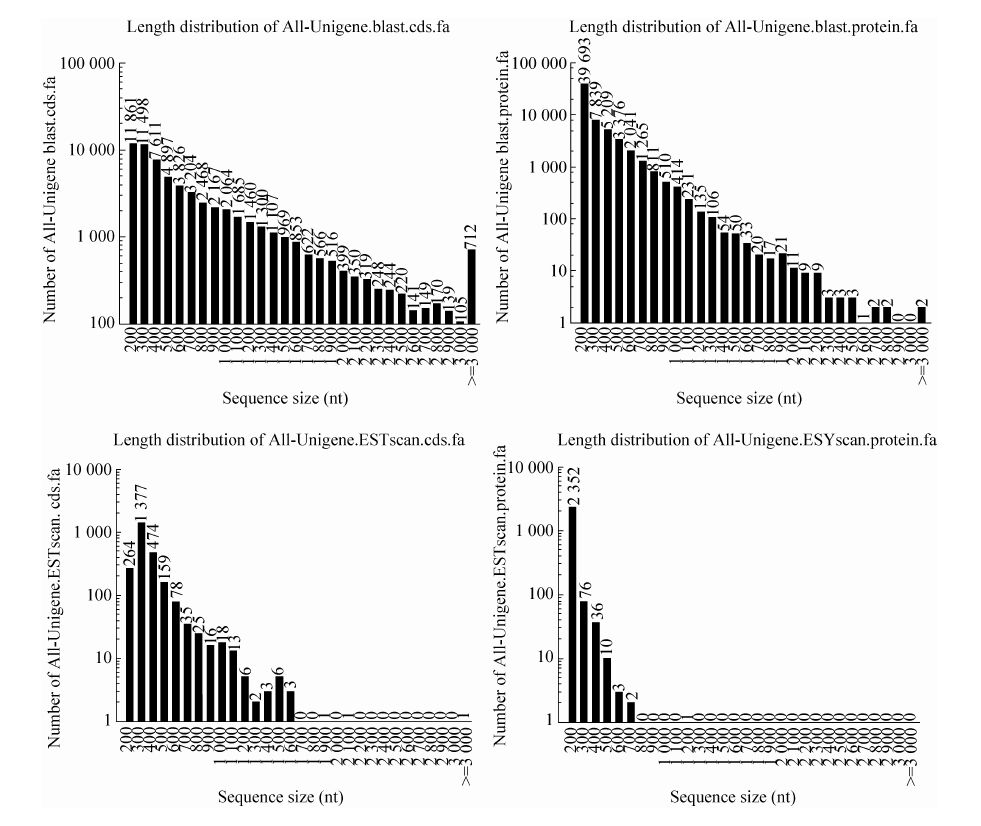
|
| 图3 CDS 的长度分布统计图 Fig.3 Length distribution of CDS. |
统计,及根据预测的CDS翻译成氨基酸后的长度统计。其中有22 175个基因预测氨基酸长度大于300,占35.84%所预测的基因,有1 124个基因预测的氨基酸长度超过1 000,占1.82%所预测的基因。另外,用ESTScan软件对未比对上数据库的2 480个基因进行了编码框的预测,有128个基因预测的氨基酸长度大于300,占所预测基因的5.16%,有1个基因预测的氨基酸长度超过1 000。
3 讨论转录组学 (Transcriptomics) 是功能基因组学研究的一个重要内容,它是从整体水平上研究细胞中基因转录的情况及其转录调控规律。基于高通量测序技术的转录组测序 (RNA-seq) 通过对组织中的RNA (包括mRNA和非编码RNA) 进行测序,能够全面快速地获得某一物种特殊组织或器官在某一特定状态下的几乎所有转录本信息,具有高准确性、高通量、高灵敏度和低运行成本等突出优势,已经广泛应用于各种生物转录组的研究[14, 15, 16]。应用Illumina高通量测序技术对芒花芽和叶芽进行转录组测序,分别获得68 136 340个和68 452 222个Clean reads,经拼接组装,花芽和叶芽分别获得180 118个和159 514个Contig,平均长度314和319 nt,最终共生成All- Unigene 98 326个。Changsoo等[17]采用454平台进行的芒 M. sinensis根茎和叶片转录组测序,叶片和根茎各获得 457 891和512 950个 reads,以及12 166 和13 170 个Contigs,平均长度970和923 nt。其所获得reads数及Contig数较低,但平均长度较高。454 平台读长长,但准确率较低。Illumina快速、高效、测序片段较短,但通过短序列有效地被组装,序列读长增加而且更精确[18, 19]。本研究采用了Illumina高通量测序,虽然测序片段较短,但是组装后得到的Unigene平均长度超过了800 bp,获得的数据产量和组装质量完全可以满足转录组分析的要求,且产生的数据量远高于Changsoo等[17]的研究。
由于芒没有全基因组数据,已知的生物信息量又很匮乏,这给转录组数据的分析带来了困难。目前对没有基因组物种的研究,主要采取将获得的数据与已知的蛋白数据库 (NR、Swiss-Prot、COG、KEGG) 进行比对,以强大的生物信息学平台作支撑,根据“基因结构相似,功能同源”的原理,对基因的功能进行注释。本研究釆用同样的方法,将获得的数据与已知的蛋白数据库进行Blast 搜索比对,共有74 134 条Unigene 获得了基因注释,占All-Unigene 的75.40%;有24 192 条Unigene (24.6%) 未被注释。对于没有得到注释的Unigene,有可能是芒特有的新基因,或由于数据库现有的基因资源有限,基因功能注释信息不丰富,从而造成部分序列暂时无法获得对应的功能注释信息。
对于有参考基因组的物种,通常选择已经公布的相同或相近物种的基因组和基因信息为参考,将所测数据映射至参考基因组的数据中,进行比对分析。Barling等[20]在对芒属Miscanthus genus植物芒和荻Miscanthus sacchariflorus的种间杂交种 Miscanthus × giganteus根茎的转录组研究中,就采用高粱Sorghum bicolor基因组作为参考序列对Miscanthus × giganteus转录组进行有参分析,63%的Miscanthus × giganteus reads 映射到高粱Sorghum bicolor基因组中。本研究中选取的植物材料芒M. sinensis,是Miscanthus × giganteus的亲本之一[20],采用无参转录组分析,通过拼接组装得到Unigene,与NT 蛋白数据库有同源比对信息的Unigene中,比对到同源序列比例最高的物种分别为高粱 (37 731,60.86%)、玉米 (16 258,26.22%)、水稻 (3 065,4.94%) 占所有同源序列的92.02%,其中芒与高粱同源序列最多高达60.86%,这与Miscanthus × giganteus有参转录组比对数据相似,芒及其与荻的种类杂交后代Miscanthus × giganteus与高粱具有较高的同源性。
芒是多年生C4草本植物,光合作用效率高,CO2 补偿点低,氮素和水分利用效率高,植株高大[21]。C4植物能通过C4途径的酶系统保持较高的光合效率。CO2被吸收后,反应过程的速率主要受C4途径中酶的数量和活性以及可利用的CO2的量的限制[22]。本研究通过NR同源性搜索比对,在测序结果中获得了芒基因中C4重要的酶基因。包括C4核心循环相关的主要酶碳酸酐酶 (Carbonic anhydrase,CA)、磷酸烯醇式丙酮酸羧化酶 (Phosphoenolpyruvate carboxylase,PEPC)、依赖NADP的苹果酸脱氢酶 (NADP-dependent malic enzyme,NADP-ME)、丙酮酸二激酶 (Pyruvate,orthophosphate dikinase,PPDK)。其中功能注释为磷酸烯醇式丙酮酸羧化酶的Unigenes 10个,数目最多。PEPC是C4光合途径关键酶之一,存在于叶肉细胞的细胞质中,形成CO2浓缩机制,为维管束鞘细胞进行的C3途径提供CO2[23]。C4植物 ppc 基因家族共有3个成员组成,其基本结构很相似,分别是:C4型 (绿叶型),主要在叶片中大量表达,并且受光照调控;根 (茎) 型,主要在根组织中特异表达;黄化叶型或 C3型,主要在黄色叶片、茎等许多部位表达[24]。Barling等[14, 20]在芒属植物根茎转录组研究中也发现了高表达的ppc基因。但本研究中通过同源比对所得到的10个PEPC相关基因,与玉米、高粱、黍表现出了高同源性,而非芒属植物。在其他C4植物中,通过序列比较发现,同一种植物来源的不同类型ppc基因同源性较小,而存在于不同植物的同一类型的pepc基因具有较高的同源性[25]。由此推测,本研究与Barling等[14, 20]所得的ppc基因可能为不同类型。这些C4代谢相关基因的注释为研究芒C4光合途径提供了重要依据。同时,了解芒光合作用相关酶基因的表达调控,对未来作物设计与改良具有重要意义。
| [1] | Lewandowski I, Clifton-Brown JC, Scurlock JMO, et al. Miscanthus: European experience with a novel energy crop. Biomass Bioenergy, 2000, 19(4): 209-227. |
| [2] | Vermerris W. Genetic Improvement of Bioenergy Crops. New York: Springer, 2008: 274-290. |
| [3] | Yan J, Chen W, Luo F, et al. Variability and adaptability of Miscanthus species evaluated for energy crop domestication. GCB Bioenergy, 2012, 4(1): 49-60. |
| [4] | Somerville C, Youngs H, Taylor C, et al. Feedstocks for lignocellulosic biofuels. Science, 2010, 329(5993): 790-792. |
| [5] | Jones MB, Mary W. Miscanthus for Energy and Fiber. London: James & James (Science Publishers), 2001: 1-10. |
| [6] | Heaton EA, Dohleman FG, Long SP. Meeting US biofuel goals with less land: the potential of Miscanthus. GCB Bioenergy, 2008, 14: 2000-2014. |
| [7] | Naidu SL, Moose SP, AL-Shoaibi AK, et al. Cold Tolerance of C4 photosynthesis in Miscanthusx giganteus: adaptation in amounts and sequence of C4 photosynthetic enzymes. Plant Physiol, 2003, 132(3): 1688-1697. |
| [8] | Heaton EA, Long SP, Voigt TB, et al. Miscanthus for renewable energy generation: European union experience and projections for Illinois. Mitig Adapt Strategy Glob Chang, 2004, 9(4): 433-451. |
| [9] | Vermerris W. Genetic Improvement of Bioenergy Crops. New York: Springer, 2008: 287. |
| [10] | Glowacka K. A review of the genetic study of the energy crop Miscanthus. Biomass Bioenergy, 2011, 35(7): 2445-2454. |
| [11] | Zhang GJ, Guo GW, Hu XD, et al. Deep RNA sequencing at single base-pair resolution reveals high complexity of the rice transcriptome. Genome Res, 2010, 20(5): 646-654. |
| [12] | Lu TT, Lu GJ, Fan DL, et al. Function annotation of the rice transcriptome at single-nucleotide resolution by RNA-seq. Genome Res, 2010, 20(9): 1238-1249. |
| [13] | Hao DC, Ge GB, Xiao PG, et al. The first insight into the tissue specific Taxus transcriptome via Illumina second generation sequencing. PLoS ONE, 2011, 6(6): e21220. |
| [14] | Grabherr MG, Haas BJ, Yassour M, et al. "Full-length transcriptome assembly from RNA-Seq data without a reference genome." Nat Biotechnol, 2011, 29(7): 644-652. |
| [15] |
Zhang CL, Qin ZJ, Wang GZ, et al. Transcriptome and RNA-Seq technology. Biotechnol Bull, 2012, (12): 51-56 (in Chinese). 张春兰, 秦孜娟, 王桂芝, 等. 转录组与RNA-Seq 技术. 生物技术通报, 2012, (12): 51-56. |
| [16] | Shendure J. The beginning of the end for microarrays? Nat Methods, 2008, 5(7): 585-587. |
| [17] | Kim C, Lee TH, Guo H, et al. Sequencing of transcriptomes from two Miscanthus species reveals functional specificity in rhizomes, and clarifies evolutionary relationships. BMC Plant Biol, 2014, 14: 134. |
| [18] | Wilhelm BT, Landry JR. RNA-Seq quantitative measurement of expression through massively parallel RNA sequencing. Methods, 2009, 48(3): 249-257. |
| [19] | Wang Z, Gerstein M, Snyder M. RNA-Seq: a revolutionary tool for transcriptomics. Nat Rev Genet, 2009, 10(1): 57-63. |
| [20] | Barling A, Kankshita S, Therese M, et al. A detailed gene expression study of the Miscanthus genus reveals changes in the transcriptome associated with the rejuvenation of spring rhizomes. BMC Genomics, 2013, 14: 864. |
| [21] | Hodkinson TR, Renvoize S. Nomenclature of Miscanthusx giganteus (Poaceae). Kew Bull, 2011, 56: 759-760. |
| [22] | Beale CV, Long SP. Can perennial C4 grasses attain high efficiencies of radiant energy conversion in cool climates? Plant Cell Environ, 1995, 18(6): 641-650. |
| [23] |
Wu H. Photosynthetic characteristic identification of Eleocharis baldwinii & the functional study of C3/C4 differentially expressed genes [D]. Wuhan: Huazhong Agricultural University, 2014 (in Chinese). 伍欢. 大莎草的光合模式鉴定及C3/C4差异表达基因的功能研究[D]. 武汉: 华中农业大学, 2014. |
| [24] |
Zhang GF. Cloning key enzyme (PEPC、PPDK) genes of C4 photosynthesis from barnyardgrass (Echinochloa) and PEPC gene transformation in rice (Oryza Sativa) and tobacco (Nicotiana tabacum) [D]. Beijing: China Agricultural University, 2005 (in Chinese). 张桂芳. 稗草C4关键酶 (PEPC、PPDK) 基因的克隆及PEPC基因对水稻和烟草的遗传转化[D]. 北京: 中国农业大学, 2005. |
| [25] | Schäffner AR, Sheen J. Maize C4 photosynthesis involves differential regulation of phosphoenolpyruvate carboxylas egenes. Plant J, 1992, 2(2): 221-232. |
 2012, Vol. 31
2012, Vol. 31