中国科学院微生物研究所,中国微生物学会
文章信息
- 闫阔诚, 李浩, 白萱洋, 韩尧, 石大伟, 贾雷立, 孙岩松. 2024
- YAN Kuocheng, LI Hao, BAI Xuanyang, HAN Yao, SHI Dawei, JIA Leili, SUN Yansong.
- 基于CRISPR/Cas13a的马尔堡病毒核酸检测方法的建立
- A CRISPR/Cas13a-based nucleic acid detection method for Marburg virus
- 微生物学报, 64(8): 3073-3085
- Acta Microbiologica Sinica, 64(8): 3073-3085
-
文章历史
- 收稿日期:2024-01-29
- 网络出版日期:2024-04-28
2. 中国人民解放军疾病预防控制中心, 北京 100071;
3. 中国食品药品检定研究院传染病诊断试剂二室, 北京 100050
2. Chinese PLA Center for Disease Control and Prevention, Beijing 100071, China;
3. Division Ⅱ of Diagnostic for Infectious Diseases, National Institutes for Food and Drug Control, Beijing 100050, China
1967年,马尔堡病毒(Marburg virus, MARV)首次发现于德国马尔堡地区,是一种丝状病毒科单股负链RNA病毒,可感染人类和非人灵长类动物[1-2]。马尔堡病毒基因组由7个单顺反子基因线性排列组成(3′-NP-VP35-VP40-GP-VP30-VP24-L-5′),分别负责编码核蛋白(nucleoprotein, NP)、病毒蛋白35 (viral protein 35, VP35)、病毒蛋白40 (viral protein 40, VP40)、糖蛋白(glycoprotein, GP)、病毒蛋白30 (viral protein 30, VP30)、病毒蛋白24 (viral protein 24, VP24)和RNA聚合酶片段(polymerase, L)[3]。NP基因编码的核蛋白是形成核衣壳的主要蛋白,不仅包绕病毒基因组RNA,保护其免受损伤,而且在病毒RNA的复制与转录中也发挥关键作用[4-5];NP基因在病毒中丰度高,存在高度保守序列[6],因此本研究选择NP基因作为检测的靶标基因。马尔堡病毒主要在非洲流行,在安哥拉、肯尼亚、乌干达等国家地区多次暴发疫情,2023年2月与3月,赤道几内亚和坦桑尼亚被证实首次暴发了马尔堡病毒,其暴发与流行对公共健康产生巨大危害[7-9]。2015年,世界卫生组织(World Health Organization, WHO)将马尔堡病毒列入可能引发大流行需要高度关注的重点传染病;在2018年,该病毒被纳入应急状态下需优先关注的重点传染病清单[7, 10]。马尔堡病毒病患者初始症状为发热、腹泻、呕吐等非特异性症状,可逐渐发展为严重的出血热,病死率高达88%[11-13]。该病致死率高,临床救治能力薄弱,无批准上市的特效药物及疫苗[4, 14-15],因此,早期快速、简便地检测马尔堡病毒对疫情的防控具有重要意义。病毒的分离和培养是马尔堡病毒检测的金标准,但其必须在生物安全四级实验室进行,不适用于快速检测[16]。目前用于马尔堡病毒检测的方法有酶联免疫吸附试验(enzyme-linked immunosorbent assay, ELISA)、实时荧光定量PCR (quantitative real-time PCR, qPCR)和基因测序等,但这些方法操作烦琐,均依赖专业的实验室和检测人员,难以在经济条件落后的地区大规模配置[17-18],因此需要开发一种更简便、快速的马尔堡病毒检测方法。
重组酶介导链替换核酸扩增(recombinase aided amplification, RAA)是一种恒温体外快速扩增核酸技术。在37−39 ℃恒温下,重组酶与引物结合形成复合体,侵入与引物互补配对的模板DNA位点辅助解链,促使模板与引物之间发生链替换,并在DNA聚合酶作用下延伸合成新的互补链,从而实现对目标序列的扩增[19]。逆转录酶重组酶介导链替换核酸扩增(reverse transcription recombinase aided amplification, RT-RAA)则在此基础上增加了逆转录步骤,先利用逆转录酶将RNA逆转录为DNA再进行RAA扩增,该技术已被应用于多种病原体的检测[20-21]。近年来,核酸检测技术快速发展,基于成簇的规则间隔短回文重复序列(clustered regularly interspaced short palindromic repeats, CRISPR)的核酸检测技术被认为是下一代分子诊断技术[22-23]。利用Cas13蛋白的“附带切割”活性,结合等温扩增技术,Gootenberg等开发了Specific High-sensitivity Enzymatic Reporter unLOCKing (SHERLOCK)核酸检测平台[24],该技术在检测病毒、细菌等多种病原体时表现出高灵敏性与高特异性[24-26]。本课题组Li等前期开发了一种基于CRISPR/Cas13a的消线法(easy-readout and sensitive enhanced, ERASE)侧流层析试纸检测方法,该方法利用RAA或RT-RAA对目标核酸进行扩增后,将DNA序列转录为可被CRISPR RNA (crRNA)特异性识别的RNA,当目标RNA与crRNA-Cas13a蛋白复合体特异性结合后,激活Cas13a蛋白的“附带切割”活性,非特异性地切割RNA,从而降解两端分别标记了FAM (fluorescein amidite)基团和生物素(biotin)基团的报告RNA;将反应后的体系滴加到侧向流层析试纸,液体流过胶体金金垫时,标记有FAM抗体的胶体金颗粒会与FAM基团结合;当存在目标核酸时,报告RNA被切割,液体流过包被有链霉亲和素的检测线(T线,test line),报告RNA的Biotin残基与链霉亲和素结合,不显示条带,胶体金颗粒与包被抗FAM抗体的质控线(C线,control line)结合显示条带;当不存在目标核酸时,结合FAM的胶体金颗粒通过完整的报告RNA与T线结合显色,从而在T线与C线都显示条带(图 1)[27]。利用该技术实现了对多种病原体的检测,包括新型冠状病毒及其变异位点[27-30]。本研究旨在利用该技术建立一种无需荧光检测仪器的马尔堡病毒核酸快速检测方法。
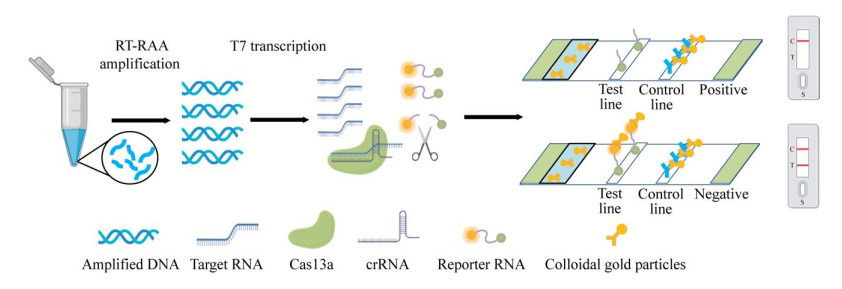
|
| 图 1 利用CRISPR-ERASE方法检测马尔堡病毒示意图 Figure 1 The schematic of CRISPR-ERASE method for Marburg virus detection. |
1 材料与方法 1.1 主要试剂和仪器
T7 Quick High Yield RNA Synthesis Kit、rNTP混合液、小鼠RNase抑制剂,New England Biolabs公司;RNaseAlertTM QC System v2、HEPES、MgCl2,ThermoFisher Scientific公司;RT-基础型核酸扩增试剂(RAA法),杭州众测生物科技有限公司;LwaCas13a蛋白,GenScript公司;2×Super Pfx Master Mix,江苏康为世纪生物科技股份有限公司;无水乙醇、三氯甲烷,国药集团药业股份有限公司;DNA提取酚试剂,北京索莱宝科技有限公司;RNAClean XP试剂盒,Beckman Coulter公司;T7 RNA聚合酶、一步法RT-qPCR染料法检测试剂盒,TaKaRa公司;RNase-free ddH2O,南京诺唯赞生物科技股份有限公司;ERASE核酸检测试纸,自研。
MiniAmpTM热循环仪,ThermoFisher Scientific公司;金属浴,COYOTE公司;实时荧光定量PCR仪,Roche公司;全自动核酸提取仪,无锡百泰克生物技术有限公司;台式小型高速离心机,Sigma-Aldrich公司;超微量分光光度计,IMPLEN公司。
1.2 引物设计和crRNA的制备马尔堡病毒基因序列下载自NCBI网站,使用MAFFT程序(https://mafft.cbrc.jp/alignment/server/)比对序列保守性,选定参考株(GenBank编号为AY430365.1)。根据保守序列设计用于RT-RAA与PCR的引物,引物核酸序列由北京天一辉远生物科技有限公司合成(表 1)。根据保守序列按碱基互补的原则设计crRNA与用于制备crRNA的序列,根据前期工作基础,按照先进行PCR扩增再进行体外转录的流程制备crRNA,PCR上游引物5′端引入T7启动子序列以便后续进行转录[31]。利用PCR扩增crRNA模板序列,用于制备crRNA的PCR引物及模板序列均由北京天一辉远生物科技有限公司合成(表 1)。PCR反应体系(50 µL):2×Super Pfx Master Mix 25 µL,上、下游引物(10 µmol/L)各2 µL,模板(crRNA-T) 2 µL,RNase-free ddH2O 19 µL。充分混匀后放入MiniAmpTM热循环仪中,设置PCR反应条件:95 ℃预变性5 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸45 s,35个循环;72 ℃终延伸10 min;4 ℃保存。PCR产物纯化后使用T7 Quick High Yield RNA Synthesis Kit进行体外转录,转录反应体系(20 μL):NTP Buffer Mix 10 μL,T7 RNA Polymerase Mix 2 μL,DNA模板1 μg,RNase-free ddH2O将体系补足至20 μL。转录反应条件:放入恒温培养箱37 ℃孵育至少12 h,孵育完成后利用RNA Clean XP试剂盒纯化crRNA。
| Name | Sequence (5′→3′) |
| RT-RAA-F1 | AATTCTAATACGACTCACTATAGGGGAAGAGATGGATTTACACAGTTTGTTGGAG |
| RT-RAA-F2 | AATTCTAATACGACTCACTATAGGGGAGATGGATTTACACAGTTTGTTGGAGTTG |
| RT-RAA-F3 | AATTCTAATACGACTCACTATAGGGGATTTACACAGTTTGTTGGAGTTGGGTAC |
| RT-RAA-R1 | TCAACACACAACGTCAGCAAACCCCCTTCT |
| RT-RAA-R2 | TCCAAGATCAATCCCTGAGTTTATTGCATC |
| RT-RAA-R3 | TAGTAATGCTCAACACACAACGTCAGCAAA |
| RT-RAA-R4 | CAACGTCAGCAAACCCCCTTCTAGGAGATC |
| T7-crRNA-F | TAATACGACTCACTATAGGGGATTTAGACTACCCCAA |
| crRNA1-R | TTCGTAATAAGAAAGTGATA |
| crRNA2-R | AAATCATCAGGTTAGTATCT |
| crRNA3-R | ATGCAATAAACTCAGGGATT |
| crRNA1-T | GGGATTTAGACTACCCCAAAAACGAAGGGGACTAAAACTCAAATAATATCACTTTCTTATTACGAA |
| crRNA2-T | GGGATTTAGACTACCCCAAAAACGAAGGGGACTAAAACCTGATTACAGATACTAACCTGATGATTT |
| crRNA3-T | GGGATTTAGACTACCCCAAAAACGAAGGGGACTAAAACCCAAGATCAATCCCTGAGTTTATTGCAT |
| PCR-R | ATTCAATATTATGGGCAAGTTCAACT |
| PCR-F | AGTCGTTGAATTCTTCGTTTACTGC |
| Reporter RNA | FAM-20U-Biotin |
1.3 RT-RAA扩增
根据RT-基础型核酸扩增试剂(RAA法)说明书,保持总体积不变的情况下调整各组分含量,优化反应效果。利用RT-RAA反应扩增模板,引物序列具体如表 1所示,RT-RAA扩增反应体系(50 µL):A缓冲液25 µL,B缓冲液2.5 µL,上、下游引物(10 µmol/L)各2 µL,模板10 µL,RNase-free ddH2O 8.5 µL。盖紧管盖后颠倒6−8次充分混合溶液,将反应体系置于金属浴中,42 ℃孵育30 min。
1.4 CRISPR荧光法检测取1.3中等温扩增产物5 µL与CRISPR/Cas13a荧光检测体系混合,放入LightCycler 96实时荧光定量PCR仪进行CRISPR荧光法检测。CRISPR荧光检测体系(25 µL):rNTP混合液(25 mmol/L) 2 µL,RNase抑制剂(40 U/µL) 1 µL,T7 RNA聚合酶(50 U/µL) 0.5 µL,HEPES (1 mol/L) 0.5 µL,MgCl2 (1 mol/L) 0.25 µL,RNaseAlertTM QC System v2 (2 nmol/mL) 2.5 µL,crRNA (100 ng/µL) 1.5 µL,Cas13a蛋白(600 nmol/L) 1 µL,RNase-free ddH2O 10.75 µL,RT-RAA扩增产物5 µL。CRISPR荧光法检测条件:37 ℃孵育60 min,选择FAM通道,激发光波长490 nm,发射光波长520 nm,每2 min采集一次荧光信号,实时监测荧光强度变化。
1.5 CRISPR-ERASE检测取1.3中等温扩增产物5 µL与CRISPR-ERASE试纸检测体系混合,在金属浴上37 ℃恒温孵育30 min。在孵育结束后,用移液枪将整个反应体系转移到ERASE核酸检测试纸上,等待3−5 min即可肉眼读取检测结果。CRISPR-ERASE试纸检测体系(50 µL):rNTP混合液(25 mmol/L) 4 µL,RNase抑制剂(40 U/µL) 2 µL,T7 RNA聚合酶(50 U/µL) 1 µL,HEPES (1 mol/L) 1 µL,MgCl2 (1 mol/L) 0.5 µL,FAM-20U-Biotin (20 nmol/L) 5 µL,Cas13a蛋白(600 nmol/L) 2 µL,crRNA (100 ng/µL) 3 µL,RNase-free ddH2O 26.5 µL,等温扩增产物5 µL。ERASE试纸结果判读方法:T线不显色、C线显色,判读为阳性;T线、C线均显色,判读为阴性;C线不显色,则结果无效。
1.6 RT-qPCR法检测使用一步法RT-qPCR染料法检测试剂盒对模板进行检测。RT-qPCR体系(25 µL):TB Green RT-PCR Buffer Ⅲ 12.5 µL,Ex Taq HS (5 U/µL) 0.5 µL,RT enzyme Mix Ⅱ 0.5 µL,上、下游引物(10 µmol/L)各0.5 µL,模板2 µL,RNase-free ddH2O 8.5 µL。颠倒6−8次充分混合溶液,将其置于实时荧光定量PCR仪中,RT-qPCR检测条件:选择SYBR通道,42 ℃逆转录5 min;95 ℃预变性10 s;95 ℃变性5 s,60 ℃退火/延伸30 s,40个循环,每个循环结束时采集一次应荧光信号。
1.7 马尔堡病毒核酸检测试剂国家参考品与核酸提取马尔堡病毒核酸检测试剂国家参考品由中国食品药品检定研究院提供(370042-201801),阳性、最低检出限和精密度参考品为人血浆稀释的包含马尔堡病毒核蛋白基因和糖蛋白基因RNA噬菌体假病毒颗粒,阴性参考品为5种灭活的虫媒病毒培养液和人血浆稀释的包含拉沙热病毒糖蛋白和膜蛋白基因、包含扎伊尔型埃博拉病毒糖蛋白和膜蛋白基因的大肠杆菌菌液制成。
阳性参考品包括:P1-1 (马尔堡病毒1980年分离株/Mt. Elgon-Musoke的全长NP基因,浓度约1×105 copies/μL)、P1-2 (马尔堡病毒1980年分离株/Mt. Elgon-Musoke的全长GP基因,浓度约1×105 copies/μL)、P2-1 (马尔堡病毒2012年分离株/Mbg-422-2012的全长NP基因,浓度约1×105 copies/μL)、P2-2 (马尔堡病毒2012年分离株/Mbg-422-2012的全长GP基因,浓度约1×105 copies/μL)、P3-1 (马尔堡病毒2014年分离株/Kampala-812601的全长NP基因,浓度约1×105 copies/μL)、P3-2 (马尔堡病毒年分离株/Kampala-812601的全长GP基因,浓度约1×105 copies/μL)。
精密度参考品:R (马尔堡病毒2012年分离株/Mbg-422-2012的全长NP和GP基因,浓度约1×105 copies/μL)。
最低检出限参考品包括:S1−S5,其中S1−S4由精密度参考品R用阴性血浆10倍梯度稀释获得,S1浓度为1×104 copies/μL,S2浓度为1×103 copies/μL,S3浓度为1×102 copies/μL,S4浓度为1×101 copies/μL,S5为阴性血浆。
阴性参考品:B1 (拉沙病毒全长NP/GP基因,NP基因浓度为5.81×106 copies/μL,GP基因浓度为5.61×107 copies/μL)、C3-1 (扎伊尔型埃博拉病毒全长NP/GP基因,NP基因浓度为1.84×106 copies/μL,GP基因浓度为2.01×105 copies/μL)、N1 (基孔肯亚病毒,浓度为5.58×108 copies/μL)、N2 (乙型脑炎病毒SA14-14-2株,滴度为0.44 lg PFU/mL)、N3 (森林脑炎病毒senzhang株,滴度为0.64 lg LD50/mL)、N4 (汉坦病毒肾综合征出血热疫苗株,滴度为0.58 lg CCID50/mL)、N5 (黄热病毒17D株,滴度为0.49 lg PFU/mL)。
使用全自动核酸提取仪(无锡百泰克生物技术有限公司),按操作说明对马尔堡病毒核酸检测试剂国家参考品进行核酸提取,每单管提取反应使用80 μL参考品样本,获得80 μL核酸提取样本。其中精密度参考品R用RNase-free ddH2O按1:10稀释后提取,其余参考品不需稀释直接提取。稀释后的精密度参考品R需平行提取10次,其余参考品需提取1次。
1.8 crRNA与RT-RAA引物的筛选将P1-1阳性参考品提取的核酸稀释至浓度为103 copies/μL,利用1.3中RT-RAA扩增的方法对该浓度样本进行扩增,crRNA按照1.2中的方法进行制备,利用1.4中CRISPR荧光检测的方法分别使用crRNA1、crRNA2、crRNA3对扩增产物进行检测,比较实验结果,选择检测荧光值最高的crRNA进行后续实验。
将P1-1阳性参考品提取的核酸稀释至浓度为101 copies/μL,选定crRNA后,将上、下游引物两两配对组合对该浓度样本进行扩增,使用选定的crRNA进行CRISPR荧光法检测,选择检测荧光值最高引物组合进行后续实验。
1.9 灵敏度评价为了评价检测方法的灵敏度,将提取的NP基因核酸10倍浓度梯度稀释成浓度为103、102、101、100、10−1 copies/μL的核酸样本作为模板,RNase-free ddH2O作为阴性对照,分别使用1.4中CRISPR荧光法检测、1.5中CRISPR-ERASE检测和1.6中RT-qPCR方法对模板进行检测,比较3种方法的检测灵敏度。同时,利用CRISPR-ERASE检测方法对参考品S1−S5进行检测。
1.10 特异性和稳定性评价利用CRISPR-ERASE检测方法检测参考品P1-1、P2-1、P3-1、P1-2、P2-2、P3-2,验证本方法的靶标基因;同时,检测参考品N1−N5、B1、C3-1,对该方法进行特异性评价。利用CRISPR-ERASE检测方法检测重复提取10次的参考品R,评价本方法的稳定性。以上实验均以RNase-free ddH2O作为阴性对照,特异性评价中以参考品P1-1提取的核酸作为阳性对照。
1.11 统计方法使用GraphPad Prism 8.0.2软件进行数据处理,比较2组间差异是否显著采用t检验,单因素多组间差异比较采用单因素方差分析进行统计学分析。为了保证检测的准确性和重复性,所有实验均设置了至少3个独立的技术重复,P<0.05认为差异具有统计学意义。
2 结果与分析 2.1 针对马尔堡病毒的特异性扩增引物与crRNA筛选在NCBI下载马尔堡病毒序列,经比对后选择保守序列NP基因设计引物与crRNA (图 2A)。为了筛选出高效的crRNA,对浓度为103 copies/μL的核酸样本进行RT-RAA扩增,使用同一批扩增产物进行crRNA筛选实验,检测实验开始后3条crRNA的CRISPR检测荧光曲线均呈上升趋势,crRNA1检测体系在30 min进入平台期(图 2B),反应开始30 min时3条crRNA中crRNA1荧光信号最高为(2.098±0.122) a.u. (图 2C),对比阴性对照组具有统计学差异(P<0.000 1),因此选用crRNA1进行后续实验。为了筛选出高效的等温扩增引物,将设计的上、下游引物两两组合进行实验,对低浓度的目标核酸(1 copy/μL)进行RT-RAA扩增,使用扩增产物进行CRISPR荧光检测。在反应进行30 min时,引物对F2R1荧光值为0.953 a.u. (图 2D),在所有12种引物组合中荧光值最高,因此选择F2R1引物对进行后续实验。初步建立了基于CRISPR/Cas13a的马尔堡病毒检测方法。
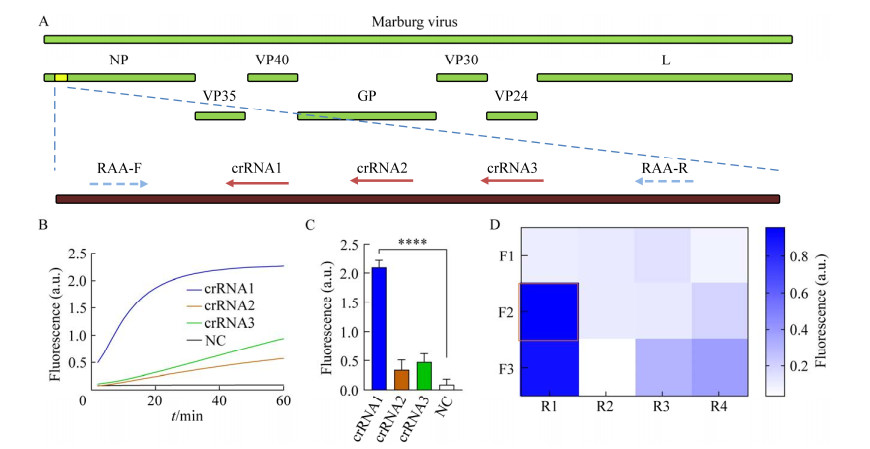
|
| 图 2 引物及crRNA的设计与筛选 Figure 2 Design and screening for efficiency primers and crRNAs. A: Design schematic of primers and crRNAs. The target gene is NP gene of Marburg virus. B: Kinetics of fluorescence signal within 60 min of three different crRNAs for CRISPR detection. NC: Negative control. C: Fluorescence values in 30 min of three different crRNAs for CRISPR detection. The data are the mean±SD, for three technical replicates; NC: Negative control (****: P<0.000 1). D: Heatmaps of different primer pairs for 30 min CRISPR fluorescence reaction. A:引物和crRNA的设计示意图,目的基因为马尔堡病毒NP基因. B:3条crRNA用于CRISPR检测60 min荧光强度曲线,NC为阴性对照. C:用于CRISPR检测的3条crRNA在反应30 min时的荧光值,每个实验组进行3个技术重复,NC为阴性对照(****: P<0.000 1). D:引物筛选实验,上、下游引物两两组合反应30 min的荧光值 |
2.2 基于CRISPR/Cas13a的马尔堡病毒检测方法灵敏度评价
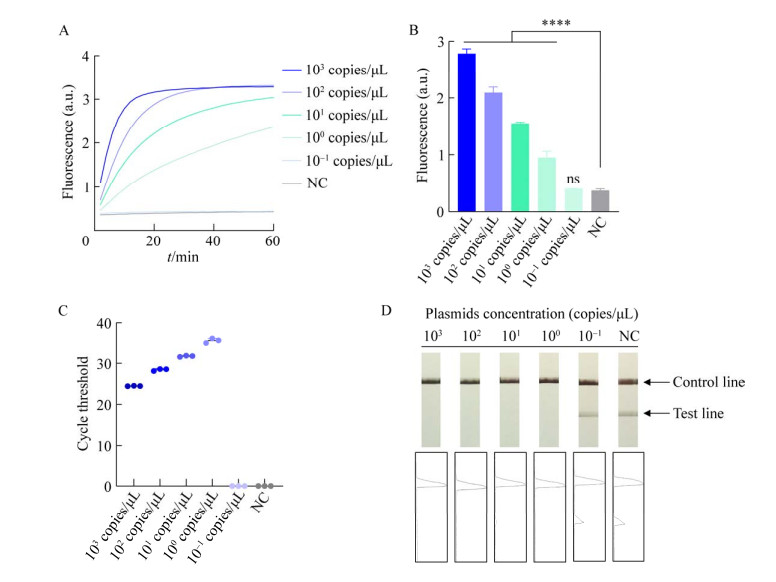
为了评价该方法的灵敏度,使用10倍浓度梯度稀释的MARV NP基因核酸作为模板进行检测实验,结果如图 3A、3B所示,检测进行10 min时,CRISPR荧光法可检出浓度最低为1 copy/μL的目标核酸,荧光值为(0.950±0.112) a.u.,与阴性对照组相比有显著差异(P<0.000 1)。此外,设计PCR引物进行RT-qPCR检测,最低可检出浓度为1 copy/μL的目标核酸(Ct值为35.69±0.55)。结合消线法侧流试纸建立马尔堡病毒的CRISPR-ERASE检测方法,结果显示该方法可检出浓度为1 copy/μL的目标核酸(图 3D)。CRISPR-ERASE方法检测MARV NP基因的灵敏度与CRISPR荧光法和RT-qPCR法一致。
|
| 图 3 基于CRISPR/Cas13a的马尔堡病毒检测方法的建立及灵敏度评价 Figure 3 Sensitivity evaluation of Marburg virus detection based on CRISPR/Cas13a. A: Kinetics of fluorescence signal within 60 min of different diluted nucleic acid for CRISPR detection. B: Fluorescence values in 10 min of different diluted nucleic acid for CRISPR detection (****: P<0.000 1; ns: P > 0.05). C: The gradient diluted target nucleic acid was detected by RT-qPCR. D: Detection results of different diluted nucleic acid using CRISPR-ERASE assay. The signal intensities of the T and C lines were generated using ImageJ software. A:CRISPR荧光法检测60 min荧光曲线,检测模板为10倍浓度梯度稀释的核酸. B:CRISPR检测进行10 min时的荧光强度(****:P<0.000 1;ns:P > 0.05). C:RT-qPCR法检测梯度稀释的目标核酸. D:CRISPR试纸法检测梯度稀释的目标核酸,灰度值的波形图使用ImageJ软件生成 |
2.3 使用国家参考品评价CRISPR-ERASE检测马尔堡病毒的特异性、最低检出限和稳定性
为了进一步评价该方法的检测性能,使用马尔堡病毒核酸检测试剂国家参考品对本方法进行评价。特异性评价实验结果如图 4A、4B所示,阳性对照(positive control, PC)组判读为阳性,阴性参考品(N1-N5、B1、C3-1)与阴性对照(negative control, NC)均判读为阴性;检测靶标序列为MARV NP基因的P1-1、P2-1、P3-1实验组均判读为阳性,检测靶标序列为MARV GP基因P1-2、P2-2、P3-2实验组与阴性对照(NC)判读为阴性。最低检出限评价结果如图 4C所示,S1−S4判读为阳性,S5 (阴性血浆)与阴性对照(NC)判读为阴性,符合参考品要求。稳定性评价结果如图 4D所示,参考品R使用RNase-freeddH2O稀释10倍后,重复进行10次病毒RNA提取,对提取的样本进行检测,10次重复结果均判读为阳性。
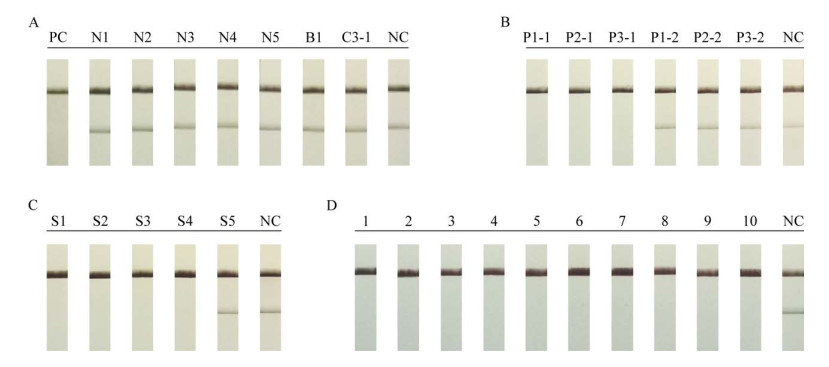
|
| 图 4 本方法的特异性、最低检出限和稳定性评价 Figure 4 Evaluation of specificity, limit of detection and stability of this method. A: Evaluation of specificity. The negative reference materials were N1−N5, B1, and C3-1; PC: Positive control; NC: Negative control. B: Evaluation of target gene. The target gene of P1-1, P2-1, and P3-1 were Marburg virus NP genes; the target gene of P1-2, P2-2, and P3-2 were Marburg virus GP genes; NC: Negative control. C: Evaluation of limit of detection. The concentration of nucleic acid in S1-S4 was decreased by 10-fold gradient; S5: Negative plasma; NC: Negative control. D: Evaluation of stability. 1−10 were the 10 test results of reference R; NC: Negative control. A:特异性评价. 阴性参考品为N1−N5、B1、C3-1;PC为阳性对照;NC为阴性对照. B:靶标基因特异性评价. P1-1、P2-1、P3-1的靶标基因为马尔堡病毒NP基因;P1-2、P2-2、P3-2的靶标基因为马尔堡病毒GP基因;NC为阴性对照. C:最低检测限评价. S1−S4中核酸浓度以10倍梯度降低,NC为阴性对照. D:稳定性评价. 1−10为参考品R的10次检测结果,NC为阴性对照 |
3 讨论与结论
马尔堡病毒是一种高致死率的烈性病原体,及时发现并阻断病毒的传播至关重要。研究者针对马尔堡病毒的检测进行了一系列研究。Boisen等利用ELISA法检测马尔堡病毒VP40抗原的检测限可以达到1 ng/mL,相较于核酸检测方法,其操作烦琐、灵敏度较低、用时较长,不利于现场快速检测[32]。Rieger等开发了检测埃博拉病毒和马尔堡病毒L基因的RT-qPCR试剂盒,使用实时荧光定量PCR仪进行检测灵敏度为95%,单反应可检测到11−67个病毒RNA拷贝,该检测依赖荧光定量PCR仪进行实验,不利于在医疗条件薄弱地区的进行筛查[33]。全基因组测序的检测方法更加准确,可区分马尔堡病毒的不同毒株,但其对样本要求较高,并且需要专业的仪器进行检测,检测在1−2 d内完成[34]。Liu等基于qPCR和微流控技术开发了TaqMan阵列卡,2.5 h内可检测6−8个样本,可检测包括马尔堡病毒在内的26种急性发热性疾病相关病原体,检测下限为在每毫升血液中可检测到104个病原体核酸拷贝,检测总体敏感性为88%,特异性为99%,但仍需专业仪器进行检测[35]。
本研究通过CRISPR荧光检测实验筛选出了针对马尔堡病毒的特异性扩增引物与crRNA,建立了基于CRISPR/Cas13a的马尔堡病毒核酸检测技术。该方法检测灵敏度达到了1 copies/μL,对比RT-qPCR检测和CRISPR荧光法具有相同的灵敏度。该方法检测国家参考品中最低检出限参考品S1−S4均检出,符合使用要求。该方法显示了良好的特异性,与拉沙病毒、埃博拉病毒、基孔肯亚病毒、乙型脑炎病毒、森林脑炎病毒、汉坦病毒、黄热病毒均无交叉反应。该方法还显示出良好的稳定性,对10倍浓度稀释的参考品R的10次检测均检出。此外,本研究结合消线法侧流试纸,阳性结果判读标准为“T线”完全消失,避免了因报告RNA自降解或“显线法”试纸的主观判读失误造成假阳性结果,并且可在1 h内完成检测,仅需一个37−42 ℃的恒温加热装置,检测结果使用试纸进行呈现,携带方便,操作简单,无需昂贵的荧光定量PCR等仪器设备,为实现马尔堡病毒的快速检测提供新的核酸检测策略和技术支撑。样本快速前处理技术是实现现场实际检测的关键,近年来,为了实现针对病毒样本的现场快速核酸提取,研究人员专门开发出了热裂解法、基于蛋白酶K的直接裂解法、基于表面活性剂病毒裂解法和基于微流控芯片和磁珠的核酸自动提取等一系列适用于现场病毒核酸快速提取的技术,实现了针对全血、尿液、粪便、唾液中病毒核酸的快速提取[36-37]。例如,Genoud等使用蛋白酶K和热裂解的方法处理鼻、咽拭子样本,通过qPCR实现了SARS-CoV-2的免核酸提取检测[38];黄媛媛等开发了一种免提取的呼吸道病毒处理试剂,可在5 min内完成包膜病毒核酸的释放,获取的样本可直接进行PCR扩增[39];Zou等开发了一种使用纤维素基纸进行核酸纯化的方法,该方法通过机械裂解或使用裂解液的方法处理植物、动物和微生物样本,使用纤维素基纸在30 s内获得可用于扩增的核酸[40]。以上方法为实现马尔堡病毒的CRISPR现场核酸快检提供了诸多候选技术,然而不同的样本处理方法与本方法的适配性和兼容性仍需进一步验证。
综上所述,本研究基于CRISPR/Cas13a,结合消线法侧流试纸建立了一种快速简便、高灵敏度和高特异性的马尔堡病毒核酸检测方法。该方法无需使用专业的荧光检测仪器,操作简单,为及时检测马尔堡病毒提供了新的技术选项。
| [1] | TIMEN A, KOOPMANS MPG, VOSSEN ACTM, van DOORNUM GJJ, GÜNTHER S, van den BERKMORTEL F, VERDUIN KM, DITTRICH S, EMMERICH P, OSTERHAUS ADME, van DISSEL JT, COUTINHO RA. Response to imported case of Marburg hemorrhagic fever, the Netherland[J]. Emerging Infectious Diseases, 2009, 15(8): 1171-1175 DOI:10.3201/eid1508.090015. |
| [2] | SLENCZKA W, KLENK HD. Forty years of Marburg virus[J]. The Journal of Infectious Diseases, 2007, 196(Supplement_2): S131-S135. |
| [3] | FELDMANN H, MÜHLBERGER E, RANDOLF A, WILL C, KILEY MP, SANCHEZ A, KLENK HD. Marburg virus, a filovirus: méssenger RNAs, gene order, and regulatory elements of the replication cycle[J]. Virus Research, 1992, 24(1): 0168170292900277. |
| [4] | BRAUBURGER K, HUME AJ, MÜHLBERGER E, OLEJNIK J. Forty-five years of Marburg virus research[J]. Viruses, 2012, 4(10): 1878-1927 DOI:10.3390/v4101878. |
| [5] | ABIR MH, RAHMAN T, DAS A, ETU SN, NAFIZ IH, RAKIB A, MITRA S, EMRAN TB, DHAMA K, ISLAM A, SIYADATPANAH A, MAHMUD S, KIM B, HASSAN MM. Pathogenicity and virulence of Marburg virus[J]. Virulence, 2022, 13(1): 609-633 DOI:10.1080/21505594.2022.2054760. |
| [6] | BHARAT TAM, RICHES JD, KOLESNIKOVA L, WELSCH S, KRÄHLING V, DAVEY N, PARSY ML, BECKER S, BRIGGS JAG. Cryo-electron tomography of Marburg virus particles and their morphogenesis within infected cells[J]. PLoS Biology, 2011, 9(11): e1001196 DOI:10.1371/journal.pbio.1001196. |
| [7] | CUOMO-DANNENBURG G, McCAIN K, McCABE R, DOOHAN P, NASH RK, HICKS JT, CHARNIGA K, GEISMAR C, LAMBERT B, NIKITIN D, SKARP J, WARDLE J, KONT M, BHATIA S, IMAI N, van ELSLAND S, CORI A, MORGENSTERN C, GROUP PER. Marburg virus disease outbreaks, mathematical models, and disease parameters: a systematic review[J]. The Lancet Infectious Diseases, 2023. DOI: S1473-S3099(23)00515-7. |
| [8] | SAMARASEKERA U. Marburg virus outbreak in Equatorial Guinea[J]. The Lancet Infectious Diseases, 2023, 23(5): 534 DOI:10.1016/S1473-3099(23)00221-9. |
| [9] | ARAF Y, MALIHA ST, ZHAI JB, ZHENG CF. Marburg virus outbreak in 2022: a public health concern[J]. The Lancet Microbe, 2023, 4(1): e9 DOI:10.1016/S2666-5247(22)00258-0. |
| [10] |
杜珊珊, 邓雪飞, 黄晓霞, 李建东. 马尔堡病毒病的流行特征及其临床表现与预防控制[J]. 疾病监测, 2022, 37(6): 832-838.
DU SS, DENG XF, HUANG XX, LI JD. Marburg virus disease: epidemiology, clinical features, and prevention[J]. Disease Surveillance, 2022, 37(6): 832-838 (in Chinese). |
| [11] | MIRE CE, GEISBERT JB, BORISEVICH V, FENTON KA, AGANS KN, FLYAK AI, DEER DJ, STEINKELLNER H, BOHOROV O, BOHOROVA N, GOODMAN C, HIATT A, KIM DH, PAULY MH, VELASCO J, WHALEY KJ, CROWE JE Jr, ZEITLIN L, GEISBERT TW. Therapeutic treatment of Marburg and Ravn virus infection in nonhuman Primates with a human monoclonal antibody[J]. Science Translational Medicine, 2017, 9(384): eaai8711 DOI:10.1126/scitranslmed.aai8711. |
| [12] | MAHANTY S, BRAY M. Pathogenesis of filoviral haemorrhagic fevers[J]. The Lancet Infectious Diseases, 2004, 4(8): 487-498 DOI:10.1016/S1473-3099(04)01103-X. |
| [13] | SRIVASTAVA S, SHARMA D, KUMAR S, SHARMA A, RIJAL R, ASIJA A, ADHIKARI S, RUSTAGI S, SAH S, AL-QAIM ZH, BASHYAL P, MOHANTY A, BARBOZA JJ, RODRIGUEZ-MORALES AJ, SAH R. Emergence of Marburg virus: a global perspective on fatal outbreaks and clinical challenges[J]. Frontiers in Microbiology, 2023, 14: 1239079 DOI:10.3389/fmicb.2023.1239079. |
| [14] | CROSS RW, MIRE CE, FELDMANN H, GEISBERT TW. Post-exposure treatments for Ebola and Marburg virus infections[J]. Nature Reviews Drug Discovery, 2018, 17: 413-434 DOI:10.1038/nrd.2017.251. |
| [15] | SUSCHAK JJ, SCHMALJOHN CS. Vaccines against Ebola virus and Marburg virus: recent advances and promising candidates[J]. Human Vaccines & Immunotherapeutics, 2019, 15(10): 2359-2377. |
| [16] | WANG YP, ZHANG XN, WEI HP. Laboratory detection and diagnosis of filoviruses[J]. Virologica Sinica, 2011, 26(2): 73-80 DOI:10.1007/s12250-011-3186-9. |
| [17] | ELSHEIKH R, MAKRAM AM, SELIM H, NGUYEN D, LE TTT, TRAN VP, ELAZIZ KHADER SA, HUY NT. Reemergence of Marburg virus disease: update on current control and prevention measures and review of the literature[J]. Reviews in Medical Virology, 2023, 33(5): e2461 DOI:10.1002/rmv.2461. |
| [18] | KORTEPETER MG, DIERBERG K, SHENOY ES, CIESLAK TJ, Medical Countermeasures Working Group of the National Ebola Training and Education Center's Special Pathogens Research Network (SPRN). Marburg virus disease: a summary for clinicians[J]. International Journal of Infectious Diseases, 2020, 99: 233-242 DOI:10.1016/j.ijid.2020.07.042. |
| [19] |
吕蓓, 程海荣, 严庆丰, 黄震巨, 沈桂芳, 张志芳, 李轶女, 邓子新, 林敏, 程奇. 用重组酶介导扩增技术快速扩增核酸[J]. 中国科学: 生命科学, 2010, 40(10): 983-988.
LÜ B, CHENG HR, YAN QF, HUANG ZJ, SHEN GF, ZHANG ZF, LI YN, DENG ZX, LIN M, CHENG Q. Recombinase-aid amplification: a novel technology of in vitro rapid nucleic acid amplification[J]. Scientia Sinica (Vitae), 2010, 40(10): 983-988 (in Chinese). |
| [20] | GUO ZH, XING GX, LI P, JIN QY, LU QX, ZHANG GP. Development and application of a recombinase-aided amplification and lateral flow assay for rapid detection of pseudorabies virus from clinical crude samples[J]. International Journal of Biological Macromolecules, 2023, 224: 646-652 DOI:10.1016/j.ijbiomac.2022.10.153. |
| [21] | WU YS, YANG Y, RU Y, QIN XD, LI MM, ZHANG ZX, ZHANG R, LI YJ, ZHANG ZD, LI YM. The development of a real-time recombinase-aid amplification assay for rapid detection of African swine fever virus[J]. Frontiers in Microbiology, 2022, 13: 846770 DOI:10.3389/fmicb.2022.846770. |
| [22] | KAMINSKI MM, ABUDAYYEH OO, GOOTENBERG JS, ZHANG F, COLLINS JJ. CRISPR-based diagnostics[J]. Nature Biomedical Engineering, 2021, 5: 643-656 DOI:10.1038/s41551-021-00760-7. |
| [23] | WANG XJ, SHANG XY, HUANG XX. Next-generation pathogen diagnosis with CRISPR/Cas-based detection methods[J]. Emerging Microbes & Infections, 2020, 9(1): 1682-1691. |
| [24] | GOOTENBERG JS, ABUDAYYEH OO, LEE JW, ESSLETZBICHLER P, DY AJ, JOUNG J, VERDINE V, DONGHIA N, DARINGER NM, FREIJE CA, MYHRVOLD C, BHATTACHARYYA RP, LIVNY J, REGEV A, KOONIN EV, HUNG DT, SABETI PC, COLLINS JJ, ZHANG F. Nucleic acid detection with CRISPR-Cas13a/C2c2[J]. Science, 2017, 356(6336): 438-442 DOI:10.1126/science.aam9321. |
| [25] | CUNNINGHAM CH, HENNELLY CM, LIN JT, UBALEE R, BOYCE RM, MULOGO EM, HATHAWAY N, THWAI KL, PHANZU F, KALONJI A, MWANDAGALIRWA K, TSHEFU A, JULIANO JJ, PARR JB. A novel CRISPR-based malaria diagnostic capable of Plasmodium detection, species differentiation, and drug-resistance genotyping[J]. eBioMedicine, 2021, 68: 103415 DOI:10.1016/j.ebiom.2021.103415. |
| [26] | ACKERMAN CM, MYHRVOLD C, THAKKU SG, FREIJE CA, METSKY HC, YANG DK, YE SH, BOEHM CK, KOSOKO-THORODDSEN TS F, KEHE J, NGUYEN TG, CARTER A, KULESA A, BARNES JR, DUGAN VG, HUNG DT, BLAINEY PC, SABETI PC. Massively multiplexed nucleic acid detection with Cas13[J]. Nature, 2020, 582: 277-282 DOI:10.1038/s41586-020-2279-8. |
| [27] | LI H, DONG X, WANG YH, YANG L, CAI K, ZHANG XL, KOU ZH, HE L, SUN SH, LI TY, NIE Y, LI XF, SUN YS. Sensitive and easy-read CRISPR strip for COVID-19 rapid point-of-care testing[J]. The CRISPR Journal, 2021, 4(3): 392-399 DOI:10.1089/crispr.2020.0138. |
| [28] | NIU MW, HAN Y, DONG X, YANG L, LI F, ZHANG YC, HU Q, XIA XS, LI H, SUN YS. Highly sensitive detection method for HV69-70del in SARS-CoV-2 alpha and Omicron variants based on CRISPR/Cas13a[J]. Frontiers in Bioengineering and Biotechnology, 2022, 10: 831332 DOI:10.3389/fbioe.2022.831332. |
| [29] |
王彦贺, 董雪, 杨明娟, 李浩, 孙岩松. 基于CRISPR的埃博拉病毒核酸检测方法的建立[J]. 中国病原生物学杂志, 2021, 16(2): 125-130.
WANG YH, DONG X, YANG MJ, LI H, SUN YS. Development of a method for CRISPR-based nucleic acid detection of the Ebola virus[J]. Journal of Pathogen Biology, 2021, 16(2): 125-130 (in Chinese). |
| [30] |
胡强, 李浩, 胡晓丰, 韩尧, 孙岩松, 柳燕. 基于CRISPR/Cas13a的金黄色葡萄球菌mecA耐药基因检测方法的建立[J]. 微生物学报, 2023, 63(9): 3628-3640.
HU Q, LI H, HU XF, HAN Y, SUN YS, LIU Y. A CRISPR/Cas13a-based detection method for the drug resistance gene mecA of Staphylococcus aureus[J]. Acta Microbiologica Sinica, 2023, 63(9): 3628-3640 (in Chinese). |
| [31] | LI H, WANG S, DONG X, LI Q, LI M, LI JF, GUO Y, JIN X, ZHOU YS, SONG HB, KOU ZH. CRISPR-Cas13a cleavage of dengue virus NS3 gene efficiently inhibits viral replication[J]. Molecular Therapy Nucleic Acids, 2020, 19: 1460-1469 DOI:10.1016/j.omtn.2020.01.028. |
| [32] | BOISEN ML, OOTTAMASATHIEN D, JONES AB, MILLETT MM, NELSON DS, BORNHOLDT ZA, FUSCO ML, ABELSON DM, ODA SI, HARTNETT JN, ROWLAND MM, HEINRICH ML, AKDAG M, GOBA A, MOMOH M, FULLAH M, BAIMBA F, GBAKIE M, SAFA S, FONNIE R, et al. Development of prototype filovirus recombinant antigen immunoassays[J]. The Journal of Infectious Diseases, 2015, 212(Suppl 2): S359-S367 DOI:10.1093/infdis/jiv353. |
| [33] | RIEGER T, KERBER R, EL HALAS H, PALLASCH E, DURAFFOUR S, GÜNTHER S, ÖLSCHLÄGER S. Evaluation of RealStar reverse transcription-polymerase chain reaction kits for filovirus detection in the laboratory and field[J]. The Journal of Infectious Diseases, 2016, 214(suppl 3): S243-S249 DOI:10.1093/infdis/jiw246. |
| [34] | MIRAGLIA CM. Marburg viruses: an update[J]. Laboratory Medicine, 2019, 50(1): 16-28 DOI:10.1093/labmed/lmy046. |
| [35] | LIU J, OCHIENG C, WIERSMA S, STRÖHER U, TOWNER JS, WHITMER S, NICHOL ST, MOORE CC, KERSH GJ, KATO C, SEXTON C, PETERSEN J, MASSUNG R, HERCIK C, CRUMP JA, KIBIKI G, MARO A, MUJAGA B, GRATZ J, JACOB ST, et al. Development of a TaqMan array card for acute-febrile-illness outbreak investigation and surveillance of emerging pathogens, including Ebola virus[J]. Journal of Clinical Microbiology, 2016, 54(1): 49-58 DOI:10.1128/JCM.02257-15. |
| [36] | SHAHI F, RASTI M, MORADI M. Overview of the different methods for RNA preparation in COVID-19 diagnosis process during the pandemic[J]. Analytical Biochemistry, 2024, 686: 115410 DOI:10.1016/j.ab.2023.115410. |
| [37] | OBINO D, VASSALLI M, FRANCESCHI A, ALESSANDRINI A, FACCI P, VITI F. An overview on microfluidic systems for nucleic acids extraction from human raw samples[J]. Sensors, 2021, 21(9): 3058 DOI:10.3390/s21093058. |
| [38] | GENOUD V, STORTZ M, WAISMAN A, BERARDINO BG, VERNERI P, DANSEY V, SALVATORI M, REMES LENICOV F, LEVI V. Extraction-free protocol combining proteinase K and heat inactivation for detection of SARS-CoV-2 by RT-qPCR[J]. PLoS One, 2021, 16(2): e0247792 DOI:10.1371/journal.pone.0247792. |
| [39] |
黄媛媛, 王宇, 周承星, 周志超, 周炳亮, 刘文宽, 周荣, 曹虹. 呼吸道病毒核酸快速检测方案的建立与应用[J]. 生物工程学报, 2023, 39(9): 3838-3848.
HUANG YY, WANG Y, ZHOU CX, ZHOU ZC, ZHOU BL, LIU WK, ZHOU R, CAO H. Development and application of a rapid scheme for detection of respiratory virus nucleic acid[J]. Chinese Journal of Biotechnology, 2023, 39(9): 3838-3848 (in Chinese). |
| [40] | ZOU YP, MASON MG, WANG YL, WEE E, TURNI C, BLACKALL PJ, TRAU M, BOTELLA JR. Nucleic acid purification from plants, animals and microbes in under 30 seconds[J]. PLoS Biology, 2017, 15(11): e2003916 DOI:10.1371/journal.pbio.2003916. |
 2024, Vol. 64
2024, Vol. 64




