中国科学院微生物研究所,中国微生物学会
文章信息
- 李昊霓, 李颖, 于思礼, 涂然, 花尔并, 刘扬, 王猛. 2024
- LI Haoni, LI Ying, YU Sili, TU Ran, HUA Erbing, LIU Yang, WANG Meng.
- 高通量构建与筛选信号肽库提高枯草芽孢杆菌外源蛋白的表达分泌
- High-throughput screening of signal peptides to improve the expression and secretion of heterologous proteins in Bacillus subtilis
- 微生物学报, 64(8): 3059-3072
- Acta Microbiologica Sinica, 64(8): 3059-3072
-
文章历史
- 收稿日期:2024-01-17
- 网络出版日期:2024-03-12
2. 中国科学院天津工业生物技术研究所, 天津 300308
2. Tianjin Institute of Industrial Biotechnology, Chinese Academy of Sciences, Tianjin 300308, China
枯草芽孢杆菌(Bacillus subtilis)是革兰氏阳性模式微生物,具有遗传背景清晰、无密码子偏好性、培养方式简单和无特殊营养需求等优点。另外,由于枯草芽孢杆菌拥有高效的蛋白分泌系统,其蛋白分泌能力强且多保持天然的构象和生物活性,生物安全性等优势,被认为是重要的分泌型酶蛋白的细胞工厂,广泛应用于工业酶的生产[1-5]。
近年来,对枯草芽孢杆菌的表达分泌元件,如不同类型的质粒、启动子、信号肽和调控因子的挖掘与表征,极大地推动了枯草芽孢杆菌重组表达系统的发展[6-10],其中信号肽能够有效提高目的蛋白的产量,受到了人们的广泛关注。目标蛋白通常以前体形式合成,其氨基末端包含一段特异的信号肽,引导其分泌表达,其中的分泌途径主要有Sec途径和Tat途径,信号肽在分泌过程中被信号肽酶识别并切除,从而释放成熟的蛋白[11-12]。由于信号肽除了被相应的蛋白转位酶定位和膜转运所必需外,还对特定目的蛋白的生物合成、折叠动力学和稳定性有影响[13]。随着研究的深入,研究人员发现信号肽与目标蛋白两者之间存在适配性的问题,同一目标蛋白在不同信号肽引导条件下,其分泌效率差异显著。Wang等[14]在枯草芽孢杆菌中使用5个信号肽分别表达聚对苯二甲酸乙二醇酯水解酶(polyethylene terephthalate hydrolase, PETase),最终信号肽SPamy联合启动子P43最大限度地分泌表达PETase;Feng等[15]在枯草芽孢杆菌WB600中使用P43启动子,联合信号肽SPamyE表达L-天冬酰胺酶,是其他信号肽的20倍,L-天冬酰胺酶酶活可达到374.9 U/mL;Fu等[16]基于PCR的克隆方法构建了一个包含173个枯草芽孢杆菌Sec型信号肽文库,以α-淀粉酶AmyS为报告基因,鉴定出15个显著提高AmyS产量的信号肽,最终AmyS的最高产量为5 086 U/mL。
综上所述,目前尚无有效的工具能够预测特定信号肽在引导目标蛋白分泌时的效率,研究人员在寻找高效信号肽以引导感兴趣蛋白分泌的过程中,仍然基于试错[17-18]。目前,信号肽文库的构建通常采用酶切连接的传统策略,这种方法存在诸多局限性,首先待筛选的信号肽序列数量多,导致可用于构建的酶切位点选择受限[19];其次信号肽酶识别位点处于信号肽的末端,而在信号肽两端人为添加酶切位点,可能会干扰信号肽酶对信号肽的识别,进而导致信号肽切割过程受阻;再则手动构建信号肽文库的方式通量低、耗时长、人力和物力成本高[20]。针对上述问题,本研究整合实验室现有的Biomek i7自动化工作站、QPix 420高通量微生物自动克隆筛选系统、KingFisher Flex磁珠纯化仪、高通量振荡摇床等自动化设备,建立了枯草芽孢杆菌酶蛋白信号肽库的高通量自动化构建和筛选平台,加快了外源蛋白的信号肽优化过程,提高外源蛋白在枯草芽孢杆菌中的表达分泌水平。本研究也为高附加值酶蛋白的工业菌种改造提升和迭代提供新的思路。
1 材料与方法 1.1 材料 1.1.1 菌株、质粒和引物本研究所用菌株和质粒由实验室储存和构建(表 1),所用引物由北京擎科生物科技股份有限公司和中国科学院天津工业生物技术研究所合成(表 2和表 3)。
| Strains and plasmids | Relevant characteristics | Sources |
| Strains | ||
| DH5α | Escherichia coli, used for gene cloning | This lab |
| DB3.1 | E. coli, gyrA462 endA1 Δ(sr1-RecA) mcrB mrr hsdS20 glnV44 (=supE44) ara14 galK2 lacY1 proA2 rpsL20 xyl-5 leuB6 mtl-1 | This lab |
| B. subtilis 168 | Bacillus subtilis, wild type | This lab |
| B. subtilis SCK6 | B. subtilis, wild type | This lab |
| Plasmids | ||
| pHP13-PgapDH-gfp | pHP13 carrying gfp gene under control of PgapDH | [21] |
| pBAC9987 | B. subtilis-E. coli shuttle vector for Cas9-based genome editing in B. subtilis, AmpR, CmR, rep pE194ts, expressing Cas9 and a ccdB cassette. | [22] |
| pHP13-PgapDH-ccdB-gfp | pHP13 carrying gfp gene under control of PgapDH, the ccdB gene was used to integrate the signal peptide fragment | This study |
| pHP13-PgapDH-SPs-gfp | pHP13 carrying gfp gene under control of PgapDH, contains different signal peptide fragments | This study |
| pMA0911-pulA | pMA0911 carrying pulA gene under control of PHapII | This lab |
| pHP13-PgapDH-pulA | pHP13 carrying pulA gene under control of PgapDH | This study |
| pMA0911-ccdB-pulA | pMA0911 carrying pulA gene under control of PHapII, the ccdB gene was used to integrate the signal peptide fragment | This study |
| pMA0911-SPs-pulA | pMA0911 carrying pulA gene under control of PHapII, contains different signal peptide fragments | This study |
| Primers name | Sequences (5′→3′) |
| gfp-ccdB-F | CGGATCCACGCGTGGTCTCCATGGGATCCATGTCGAAGGG |
| pHP13-PgapDH-ccdB-R | CTGGGGAATATAAGGTCTCGTAATATCGCCTCCTATTGTAAATTAAAATTTAATT |
| ccdB-F | CGAGACCTTATATTCCCCAGAACAT |
| ccdB-R | GGAGACCACGCGTGGATCCG |
| pHP13-pulA-F | TACAATAGGAGGCGATATTAATGCCCCCAAAACAACAGTC |
| pHP13-pulA-R | TAAAACGACGGCCAGTGAATTCAACATTGAATTAATACCCACGCACCA |
| pulA-ccdB-Bbs Ⅰ-F | GGATCCACGCGTGAAGACGAATGCCCCCAAAACAACAGTCG |
| pulA-ccdB-Bbs Ⅰ-R | GGAATATAAGAAGACGACATATGTAAATCGCTCCTTTTTAGGTGGC |
| ccdB-Bbs Ⅰ-F | ATGTCGTCTTCTTATATTCCCCAGAACATC |
| ccdB-Bbs Ⅰ-R | TCGTCTTCACGCGTGGATCC |
| Primers name | Sequences (5′→3′) |
| aspB-F | CCAGGTCTCAATTAATGAAACTGGCAAAAAGAGTATCCGC |
| citH-F | CCAGGTCTCAATTAATGGGAAATACTCGTAAAAAAGTTTCTGT |
| lytF-F | CCAGGTCTCAATTAATGAAAAAGAAATTAGCAGCAGGGC |
| rpmG-F | CCAGGTCTCAATTAATGAGAAAAAAGATTACGTTAGCATGCA |
| ykwD-F | CCAGGTCTCAATTAATGAAGAAAGCATTTATTTTATCTGCTGCC |
| aspB-R | CCAGGTCTCACCATCGCTTTCGCTGTGATTGC |
| citH-R | CCAGGTCTCACCATAACGTCTGCCAGCTCTTTTT |
| lytF-R | CCAGGTCTCACCATTGCTTCAGCTGGTGTCACTAC |
| rpmG-R | CCAGGTCTCACCATCGCTGATGCAGAGCTCTTCA |
| ykwD-R | CCAGGTCTCACCATCGCTGATGCTTGCTGTACG |
1.1.2 主要试剂
Q5 High-Fidelity DNA聚合酶、T4 DNA连接酶、各种限制性内切酶均购自纽英伦生物技术(北京)有限公司;2×ES Taq MasterMix (Dye)购自北京康为世纪生物科技有限公司;质粒小量提取试剂盒(磁珠法)、普通DNA产物纯化试剂盒(磁珠法)购自杭州倍沃医学科技有限公司;重组试剂盒ClonExpress MultiS One Step Cloning Kit购自南京诺唯赞生物科技股份有限公司;氨苄青霉素(ampicillin, Amp)、卡那霉素(kanamycin, Kan)、壮观霉素(spectinomycin, Spe)购自北京索莱宝科技有限公司。
大肠杆菌和枯草芽孢杆菌GFP表达使用LB培养基(g/L):蛋白胨10.0,酵母提取物5.0,氯化钠10.0。枯草芽孢杆菌PulA蛋白表达使用2×SR培养基(g/L):蛋白胨30.0,酵母提取物50.0,K2HPO4·3H2O 6.0。各种抗性培养基中抗生素浓度:Spe在大肠杆菌中为100 μg/mL,在枯草芽孢杆菌中为250 μg/mL;Amp在大肠杆菌中为100 μg/mL,Kan在枯草芽孢杆菌中为25 μg/mL。
1.2 枯草芽孢杆菌的转化将已活化的B. subtilis SCK6菌液按照OD600为1.0用LB液体无抗培养基稀释转接并加入20%的木糖母液,使得木糖的终浓度在2%,在37 ℃、220 r/min条件下培养2 h制备成感受态。随即转入2 μg质粒到100 μL感受态细胞中,在37 ℃、220 r/min条件下孵育3 h涂布于含有抗性的LB固体培养基上,37 ℃倒置培养过夜。
1.3 重组枯草芽孢杆菌GFP培养及荧光测定重组菌株培养:采用Biomek i7自动化工作站向96孔板中加入含抗性的LB液体培养基,利用QPix 420高通量微生物自动克隆筛选系统将含有不同信号肽的绿色荧光蛋白重组菌株单克隆接种于96孔板中,高通量振荡摇床37 ℃、800 r/min培养16 h,Biomek i7自动化工作站转接10 μL种子液至含有新鲜培养基的96孔板中,高通量振荡摇床37 ℃、800 r/min培养24 h。
荧光测定:Biomek i7自动化工作站分别转移100 μL发酵液和发酵液离心后的上清液至酶标板中,使用酶标仪测定重组菌株发酵后的绿色荧光强度以及菌株的OD600值(荧光激发波长为488 nm,吸收波长为520 nm)。目的蛋白的分泌率(%)=上清液的荧光/总荧光×100。
1.4 生产PulA重组枯草芽孢杆菌的培养及其酶活测定重组菌株培养:含有不同信号肽的PulA重组菌株单克隆接种于带有抗性的LB液体培养基中,37 ℃、220 r/min条件下培养16 h,以起始OD600为0.1转接至带有抗性的2×SR培养基中,37 ℃、220 r/min下培养48 h。
酶活测定:采用3, 5-二硝基水杨酸(3, 5-dinitrosalicylic acid, DNS)定糖法[23]。在相应的温度和pH条件下,以5%的普鲁兰糖溶液和pH为6.0的磷酸盐缓冲液按体积比1:8混合作为底物,加入50 μL预热酶液,混匀后60 ℃反应30 min,加入500 μL DNS液终止反应,沸水浴10 min,测定OD540下的光吸收值。以葡萄糖的DNS显色OD540光吸收值,绘制标准曲线进行标定。酶活定义:每分钟生成1 μmoL葡萄糖所需酶量定义为1个酶活力单位(U)。分别测定胞内和胞外的酶活,并计算分泌率。目的蛋白的分泌率(%)=胞外酶活/(胞外酶活+胞内酶活)×100。
1.5 表达产物SDS-PAGE分析收集发酵上清液与菌体沉淀,菌体沉淀用等体积磷酸缓冲液悬浮,超声破碎10 min,之后12 000 r/min离心2 min收集破碎上清与沉淀,将待测样品与蛋白上样缓冲液混匀,100 ℃金属浴加热10 min,12 000 r/min离心1 mim,取上清液点样进行SDS-PAGE分析。
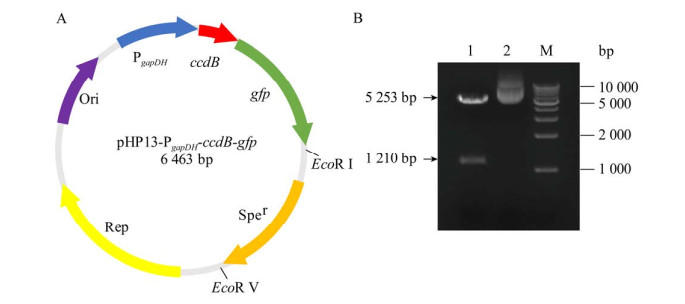
2 结果与分析 2.1 GFP信号肽筛选载体的构建以pHP13-PgapDH-gfp质粒[21]为模板使用引物对gfp-ccdB-F/pHP13-PgapDH-ccdB-R进行PCR扩增骨架片段,使用质粒pBAC9987为模板[22],用引物对ccdB-F/ccdB-R进行PCR扩增ccdB基因片段,将2个片段同源重组连接转化至大肠杆菌(Escherichia coli) DB3.1中获得重组菌,以Spe抗性筛选阳性克隆,提取质粒,使用限制性内切酶EcoR V和EcoR I进行酶切鉴定,电泳结果显示重组质粒pHP13-PgapDH-ccdB-gfp的酶切片段大小正确(图 1)。
|
| 图 1 pHP13-PgapDH-ccdB-gfp质粒谱图及酶切验证图 Figure 1 Plasmid construction and confirmation for pHP13-PgapDH-ccdB-gfp. A: Map of pHP13-PgapDH-ccdB-gfp. B: Confirmation of pHP13-PgapDH-ccdB-gfp. Lane M: DNA Molecular Weight Marker; Lane 1: pHP13-PgapDH-ccdB-gfp/EcoR V+EcoR I; Lane 2: pHP13-PgapDH-ccdB-gfp isolated from DB3.1/pHP13-PgapDH-ccdB-gfp. |
2.2 高通量构建GFP表达分泌菌株
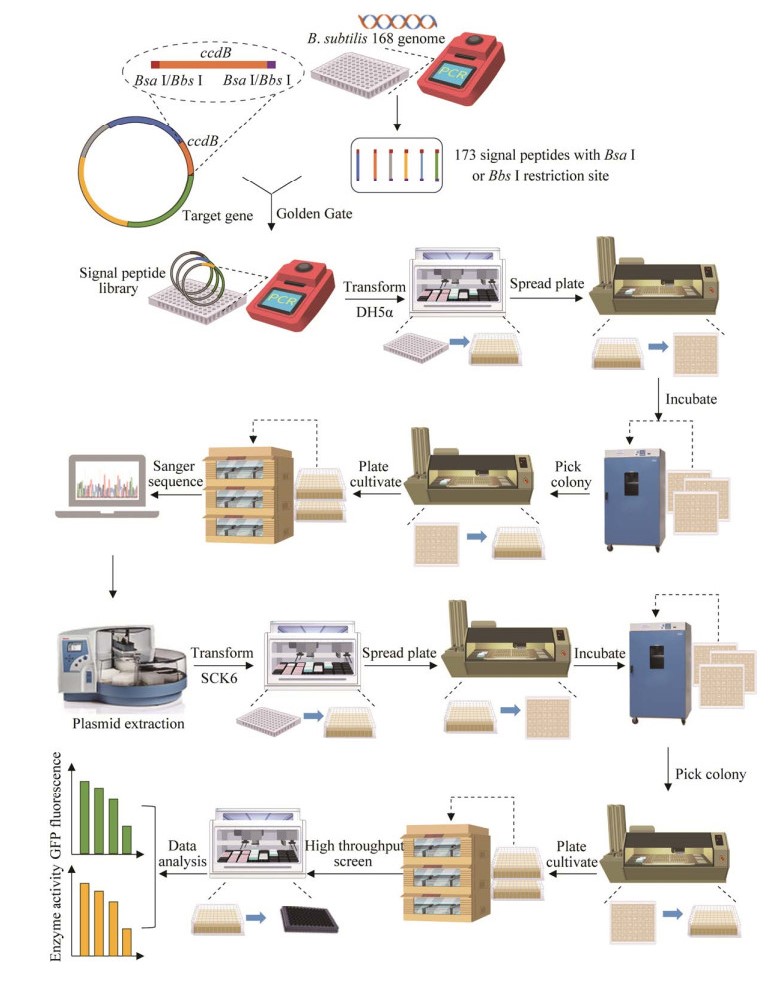
以GFP为报告基因,建立枯草芽孢杆菌GFP高分泌载体,并搭建酶蛋白信号肽库的高通量自动化构建和筛选平台。以枯草芽孢杆菌168基因组为模板分别使用不同的引物对PCR扩增173条信号肽片段(图 2),使用KingFisher Flex磁珠纯化仪高通量纯化回收173条信号肽片段,分别与pHP13-PgapDH-ccdB-gfp质粒进行Golden Gate连接,使用Biomek i7自动化工作站添加E. coli DH5α感受态至连接产物中,PCR仪42 ℃热击,高通量振荡摇床孵育1 h,QPix 420高通量微生物自动克隆筛选系统涂板,37 ℃培养箱培养过夜。经单克隆测序验证,成功构建96个重组菌株,高通量振荡摇床培养后使用KingFisher Flex磁珠纯化仪提取DNA质粒,使用Biomek i7自动化工作站添加B. subtilis SCK6感受态至2 μg质粒中,高通量振荡摇床37 ℃孵育3 h,QPix 420高通量微生物自动克隆筛选系统涂板最终获得91株重组菌株(图 2)。
|
| 图 2 高通量自动化构建和筛选信号肽库流程图 Figure 2 Flowchart of high-throughput automated construction and screening of signal peptide libraries. |
2.3 重组枯草芽孢杆菌中GFP的荧光测定
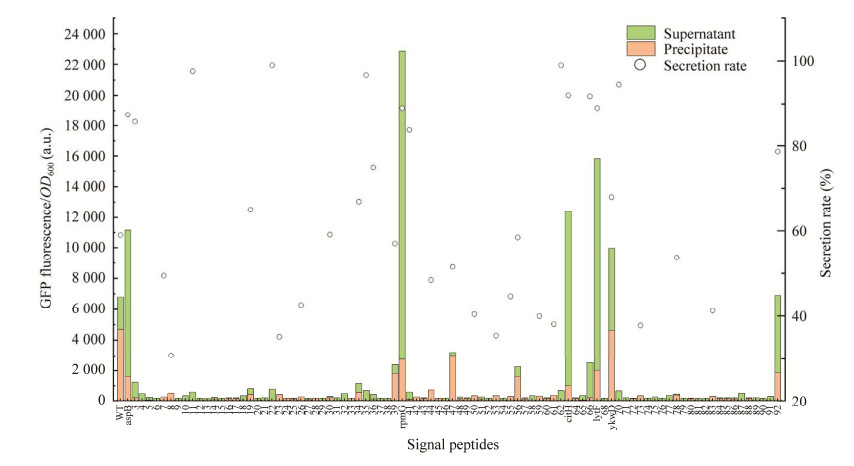
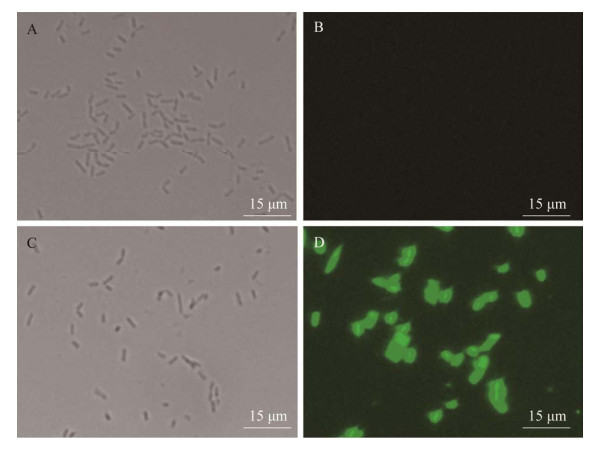
91株含不同信号肽的重组菌株经孔板发酵24 h检测荧光,结果显示含有不同信号肽重组菌株中GFP的表达分泌差异显著。其中25条信号肽(如SPykvV、SPyobB、SPyddT和SPnprB)与GFP适配性很低,重组菌的荧光值与含有空质粒的SCK6菌株相比无明显差异。SPabnA、SPywtD、SPydbK和SPywmD等61个信号肽具有一定引导GFP表达和细胞外分泌的能力,但GFP的表达较低。SPrpmG (GenBank登录号为NP_387980.1)、SPaspB (GenBank登录号为NP_390118.1)、SPcitH (GenBank登录号为NP_390790.1)、SPlytF (GenBank登录号为NP_388818.2)和SPykwD (GenBank登录号为NP_389280.2) 5个信号肽引导GFP胞外分泌的能力较强,其中SPrpmG为本次筛选得到的最优信号肽,其重组菌株SCK6/pHP13-PgapDH-SPrpmG-gfp的总荧光值达25 606 a.u.,与野生型SCK6/pHP13-PgapDH-gfp相比GFP总表达量提高了123%,其细胞外GFP荧光值可达22 854 a.u.,与野生型SCK6/pHP13-PgapDH-gfp相比细胞外GFP表达量提高了236%,为所有重组菌株的最高水平(图 3)。在荧光显微镜下观察重组菌株SCK6/pHP13-PgapDH-SPrpmG-gfp与SCK6野生型菌株的GFP荧光强度(图 4),重组菌株SCK6/pHP13-PgapDH-SPrpmG-gfp可以观察到明亮的绿色荧光。
|
| 图 3 含不同信号肽重组枯草芽孢杆菌GFP的表征 Figure 3 Characterization of recombinant Bacillus subtilis GFP containing different signal peptides. The ordinate secretion rate (%) is the GFP fluorescence in the supernatant/total GFP fluorescence. The fluorescence of LB medium for blank control was 300 a.u.; 3−39 were AmyX, LipB, LytC, PbpB, PhoA, PhrF, YbbR, YbbC, YbdN, YjfA, LytB, YlbL, WprA, NprE, YlxF, LytR, YwtD, LytR, XynA, AmyE, PbpX, YqxM, YwqC, YvbX, YpmB, YxaK, YwmB, YwfM, CotC, BglS, TyrA, YwmD, DacF, LytD, YbbE, GlpQ, TasA, respectively; 41−62 were YdjM, MotB, YlxW, YdbK, YnzA(TatAC), Pel, YobV, CccA, YoaW, MreC, YjcN, YocA, YlxY, YoqM, YhcR, YvgO, YxiT, YvpA, YpuD, YwjE, YwtF, Csn, respectively; 64−66 were YdjN, YhdC, PenP, respectively; 68 was YddT; 70−92 were WapA, SpoIIQ, Mpr, CwlD, YncM, YfkN, NprB, YbfO, YwqO, YngK, YhaK, YobB, YkvV, DltD, YvcE, YwoF, YusW, YrvJ, YqzG, YunB, YqzC, YqfZ, AbnA, respectively. |
|
| 图 4 荧光显微镜观察不同菌株表达GFP的荧光 Figure 4 Fluorescent images of Bacillus subtilis SCK6 (A and B) and Bacillus subtilis SCK6/pHP13-PgapDH-SPrpmG-gfp (C and D). A and C: Bright field images. B and D: Fluorescence images. Cells were excited at 488 nm. |
2.4 普鲁兰酶表达载体的构建及其酶活测定
针对重要的工业酶普鲁兰酶构建了其信号肽表达载体重组质粒pHP13-PgapDH-pulA。以质粒pHP13-PgapDH-gfp[21]为模板,使用引物对pHP13-PgapDH-F/pHP13-PgapDH-R进行PCR扩增骨架片段,以枯草芽孢杆菌SCK6/pMA0911-pulA为模板,使用引物对pHP13-pulA-F/pHP13-pulA-R进行PCR扩增,回收pulA片段,将2个片段同源重组连接转化E. coli DH5α中获得重组菌,以Spe抗性筛选阳性克隆,提取质粒,经测序验证,质粒构建正确(图 5)。

|
| 图 5 pHP13-PgapDH-pulA质粒谱图及酶切验证图 Figure 5 Plasmid construction and confirmation for pHP13-PgapDH-pulA. A: Map of pHP13-PgapDH-pulA. B: Confirmation of pHP13-PgapDH-pulA. Lane M: DNA Molecular Weight Marker; Lane 1: pHP13-PgapDH-pulA/EcoR V+BamH I; Lane 2: pHP13-PgapDH-pulA isolated from E. coli DH5α/pHP13-PgapDH-pulA. |
经单克隆测序验证正确后的pHP13-PgapDH-pulA质粒转入枯草芽孢杆菌SCK6后摇瓶发酵48 h,测定重组菌株中生产的普鲁兰酶的酶活(图 6A),重组菌株SCK6/pHP13-PgapDH-pulA表达PulA酶活仅为16 U/mL,说明此表达系统与普鲁兰酶的表达适配性低。因此,更换pMA0911表达载体表达普鲁兰酶,测试重组菌株SCK6/pMA0911-pulA表达PulA的酶活可达到202 U/mL,是重组菌株SCK6/pHP13-PgapDH-pulA普鲁兰酶表达量的12.6倍(图 6A)。将发酵液4 ℃离心,取上述菌株的上清液以及胞内超声破碎的上清液,采用SDS-PAGE方法进行分析。结果发现,重组菌株SCK6/pHP13-PgapDH-pulA和SCK6/pMA0911-pulA均有正确的蛋白表达条带,与文献报道[24]一致(图 6B),说明普鲁兰酶基因在2种表达系统条件下均能成功地表达,并且SCK6/pMA0911-pulA中普鲁兰酶的表达明显优于SCK6/pHP13-PgapDH-pulA。基于上述结果选择pMA0911载体用于下一步实验。

|
| 图 6 不同表达载体表达PulA酶活测定及SDS-PAGE分析 Figure 6 Enzyme activity and SDS-PAGE analysis of PulA expressed of different expression vectors. A: Enzyme activity of PulA expressed by different expression vectors. B: SDS-PAGE analysis. Lane M: Protein Molecular Weight Marker; Lane 1, 4, and 7 are the supernatant, intracellular, and inclusion bodies of pHP13-PgapDH-pulA by shake flask fermentation, respectively; Lane 2, 5, and 8 are the supernatant, intracellular, and inclusion bodies of pMA0911-pulA by shake flask fermentation, respectively; Lane 3, 6, and 9 are the supernatant, intracellular, and inclusion bodies after fermentation of pMA0911-pulA in 96 well plates, respectively. |
2.5 PulA信号肽筛选载体的构建
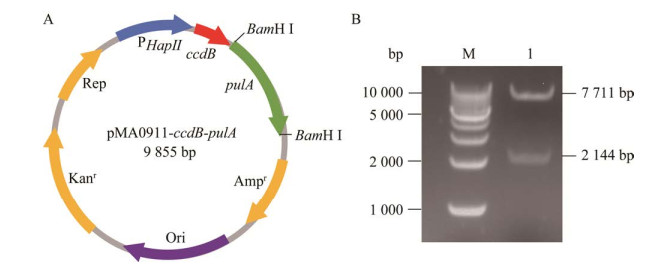
以实验室保存的pMA0911-pulA质粒为模板使用引物对pulA-ccdB-Bbs Ⅰ-F/pulA-ccdB-Bbs Ⅰ-R进行PCR扩增骨架片段,使用本实验室的质粒pBAC9987[22]为模板,用引物对ccdB-Bbs Ⅰ-F/ccdB-Bbs Ⅰ-R进行PCR扩增ccdB基因片段,同源重组连接产物转化大肠杆菌DB3.1感受态细胞,以Amp抗性筛选阳性克隆,用限制性内切酶BamH Ⅰ进行酶切鉴定,电泳结果显示重组质粒pMA0911-ccdB-pulA酶切的片段大小正确(图 7)。
|
| 图 7 pMA0911-ccdB-pulA质粒谱图及酶切验证图 Figure 7 Plasmid construction and confirmation for pMA0911-ccdB-pulA. A: Map of pMA0911-ccdB-pulA. B: Confirmation of pMA0911-ccdB-pulA. Lane M: DNA Molecular Weight Marker; Lane 1: pMA0911-ccdB-pulA/BamH I. |
2.6 高通量构建PulA表达分泌菌株
以枯草芽孢杆菌168基因组为模板分别使用不同的引物对PCR扩增,获得173条信号肽片段。与高通量构建GFP分泌载体不同的是,Golden Gate的酶切位点不同,其余构建步骤一致。经单克隆测序验证,成功构建158个重组菌株,高通量振荡摇床培养后使用KingFisher Flex磁珠纯化仪提取DNA质粒,使用Biomek i7自动化工作站添加B. subtilis SCK6感受态至2 μg质粒中,高通量振荡摇床37 ℃孵育3 h,QPix420高通量微生物自动克隆筛选系统涂板,最终获得74株正确的重组菌株(图 2)。
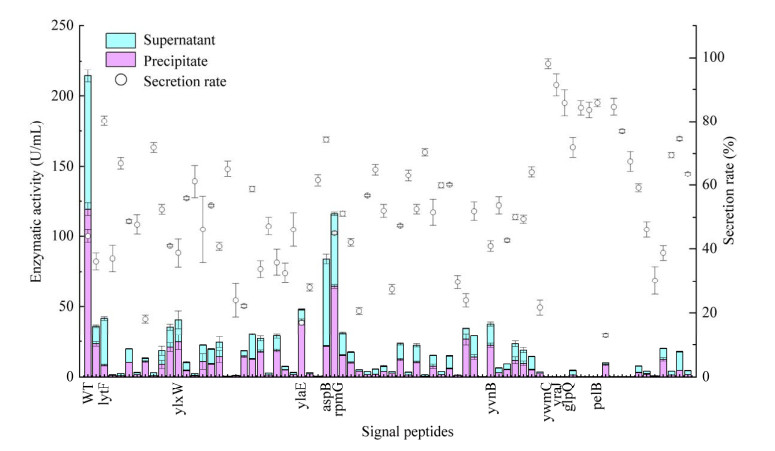
2.7 重组枯草芽孢杆菌中PulA活性测定74株重组枯草芽孢杆菌发酵48 h后检测酶活,结果显示含有不同信号肽重组菌株PulA酶活差异显著。含SPglpQ、SPywmC、SPyraJ和SPpelB等41个信号肽的重组菌株几乎检测不到普鲁兰酶酶活,说明这些信号肽与普鲁兰酶的适配性很低。含SPrpmG、SPaspB、SPylaE、SPlytF、SPylxW和SPyvnB等33个信号肽有一定的引导普鲁兰酶细胞外分泌的能力。其中SPrpmG和SPaspB为本次筛选得到的最优信号肽,重组菌株SCK6/pMA0911-SPrpmG-pulA表达普鲁兰酶酶活为所有重组菌株中的最高水平,可达116 U/mL,其表达普鲁兰酶的分泌率可达45%,与对照菌株SCK6/pMA0911-pulA相当。重组菌株SCK6/pMA0911-SPaspB-pulA表达普鲁兰酶的分泌率达到74%,与对照菌株SCK6/pMA0911-pulA相比分泌率提高了68%,其总酶活为84 U/mL,细胞外酶活为62 U/mL (图 8)。
|
| 图 8 含不同信号肽重组枯草芽孢杆菌PulA的表征 Figure 8 Characterization of recombinant Bacillus subtilis PulA containing different signal peptides. Control is the SCK6/pMA0911-pulA recombinant strain; Error bars indicate the standard deviation of three biologically parallel samples. |
3 讨论与结论
枯草芽孢杆菌是具有分泌表达外源蛋白的一类宿主。由于它具有遗传背景清晰、培养方法简单、蛋白分泌能力强和生物安全性等优点,成为工业酶表达的重要宿主之一。然而,目前除了少部分外源蛋白在枯草芽孢杆菌表达系统中实现了高效表达,大多数外源蛋白在枯草芽孢杆菌中的表达效果不理想。为了解决外源蛋白的表达分泌效果,获得更优性能的工业酶表达宿主,我们通过优化枯草芽孢杆菌中质粒构建和转化等使能技术,结合自动化实验装备,建立了枯草芽孢杆菌酶蛋白信号肽库的高通量自动化构建与筛选平台,高效筛选获得表达分泌增强的菌株以及与高表达分泌相关的元件工具,为提升枯草芽孢杆菌作为宿主底盘的表达分泌能力,解析它的高表达分泌机制奠定基础。同时,建立自动化高通量筛选平台不仅加速了枯草芽孢杆菌中外源蛋白的信号肽筛选优化过程,提升酶蛋白制造的效率,也为其他高附加值酶蛋白的工业菌种改造提升和迭代提供新的思路。
本研究以GFP绿色荧光蛋白为报告蛋白,构建91株含不同信号肽的GFP重组菌株。通过测定重组枯草芽孢杆菌中GFP的荧光,筛选出5个具有较强引导GFP胞外分泌能力的信号肽SPrpmG、SPaspB、SPcitH、SPlytF和SPykwD。其中重组菌株SCK6/pHP13-PgapDH-SPrpmG-gfp引导GFP胞外分泌效果最佳,其细胞外分泌的GFP荧光与对照菌株相比提高了236%。基于上述搭建的枯草芽孢杆菌中酶蛋白信号肽库的高通量自动化构建与筛选平台,我们进一步优化和筛选了枯草芽孢杆菌中针对普鲁兰酶分泌的信号肽,获得74株含不同信号肽的PulA重组菌株。测定重组菌株中普鲁兰酶的酶活,获得了2个具有高效引导普鲁兰酶胞外分泌能力的信号肽SPrpmG和SPaspB,其中重组菌株SCK6/pMA0911-SPrpmG-pulA的普鲁兰酶的表达量与其他重组菌株相比最高,细胞外普鲁兰酶酶活为52 U/mL,总酶活可达116 U/mL。重组菌株SCK6/pMA0911-SPaspB-pulA的普鲁兰酶分泌率可达到74%,与对照菌株SCK6/pMA0911-pulA相比分泌率提高了68%,其细胞外酶活为62 U/mL,总酶活为84 U/mL。另外,我们在研究过程中发现,构建重组质粒时有的信号肽序列极易发生突变或缺失,推测这与蛋白表达系统中所含有的强启动子有关;同时发现有的含信号肽重组质粒转入枯草芽孢杆菌时转化效率低,导致获得的重组菌株较少,推测添加信号肽序列可能对重组菌株的生长产生影响。本实验仅研究了部分枯草芽孢杆菌信号肽引导外源蛋白胞外分泌的能力,今后可以通过构建和筛选更多的含不同信号肽的重组菌株,获得更加高效生产普鲁兰酶的枯草芽孢杆菌重组菌株。
另外,通过对比不同外源蛋白GFP和PulA的信号肽库的筛选与优化结果,发现信号肽SPrpmG引导GFP和普鲁兰酶胞外分泌的能力都是最优的,信号肽SPaspB对GFP和普鲁兰酶都具有较强的引导胞外分泌的能力。可以通过进一步优化枯草芽孢杆菌中酶蛋白信号肽库的高通量自动化构建与筛选平台,获得大量有关信号肽的分析数据,采用生物信息学等方法,对所获得的信号肽序列进行归纳分析,探究信号肽与外源蛋白之间的匹配机制。
| [1] | SU Y, LIU C, FANG H, ZHANG DW. Bacillus subtilis: a universal cell factory for industry, agriculture, biomaterials and medicine[J]. Microbial Cell Factories, 2020, 19(1): 173 DOI:10.1186/s12934-020-01436-8. |
| [2] | TOLIBIA SEM, PACHECO AD, BALBUENA SYG, ROCHA J, LÓPEZ Y, LÓPEZ VE. Engineering of global transcription factors in Bacillus, a genetic tool for increasing product yields: a bioprocess overview[J]. World Journal of Microbiology and Biotechnology, 2022, 39(1): 12. |
| [3] | EJAZ S, KHAN H, SARWAR N, AQEEL SM, AL-ADEEB A, LIU S. A review on recent advancement in expression strategies used in Bacillus subtilis[J]. Protein and Peptide Letters, 2022, 29(9): 733-743 DOI:10.2174/0929866529666220803163335. |
| [4] | VOLK MJ, TRAN VG, TAN SI, MISHRA S, FATMA Z, BOOB A, LI HX, XUE P, MARTIN TA, ZHAO HM. Metabolic engineering: methodologies and applications[J]. Chemical Reviews, 2023, 123(9): 5521-5570 DOI:10.1021/acs.chemrev.2c00403. |
| [5] | STÜLKE J, GRÜPPEN A, BRAMKAMP M, PELZER S. Bacillus subtilis, a Swiss army knife in science and biotechnology[J]. Journal of Bacteriology, 2023, 205(5): e0010223 DOI:10.1128/jb.00102-23. |
| [6] | LIU YF, LIU L, LI JH, DU GC, CHEN J. Synthetic biology toolbox and chassis development in Bacillus subtilis[J]. Trends in Biotechnology, 2019, 37(5): 548-562 DOI:10.1016/j.tibtech.2018.10.005. |
| [7] |
王杰, 王晨, 杜燕, 徐晶玉, 班睿. 枯草芽孢杆菌表达和分泌异源蛋白的研究进展[J]. 微生物学通报, 2021, 48(8): 2815-2826.
WANG J, WANG C, DU Y, XU JY, BAN R. Advances in heterologous protein expression and secretion of Bacillus subtilis[J]. Microbiology China, 2021, 48(8): 2815-2826 (in Chinese). |
| [8] |
吕雪芹, 武耀康, 林璐, 徐显皓, 于文文, 崔世修, 李江华, 堵国成, 刘龙. 枯草芽孢杆菌代谢工程改造的策略与工具[J]. 生物工程学报, 2021, 37(5): 1619-1636.
LÜ XQ, WU YK, LIN L, XU XH, YU WW, CUI SX, LI JH, DU GC, LIU L. Strategies and tools for metabolic engineering in Bacillus subtilis[J]. Chinese Journal of Biotechnology, 2021, 37(5): 1619-1636 (in Chinese). |
| [9] |
康倩, 向梦洁, 张大伟. 枯草芽孢杆菌在系统与合成生物技术中研究进展及工业应用[J]. 生物工程学报, 2021, 37(3): 923-938.
KANG Q, XIANG MJ, ZHANG DW. Research progress and industrial application of Bacillus subtilis in systematic and synthetic biotechnology[J]. Chinese Journal of Biotechnology, 2021, 37(3): 923-938 (in Chinese). |
| [10] |
徐善恒, 辛瑜, 刘建民, 石贵阳, 丁重阳, 顾正华, 李由然, 张梁. 优化Bacillus subtilis对异源β-1,4-内切木聚糖酶分泌表达的研究[J]. 微生物学报, 2021, 61(10): 3222-3234.
XU SH, XIN Y, LIU JM, SHI GY, DING CY, GU ZH, LI YR, ZHANG L. Optimization of secretion of heterologous endo-β-1,4-xylanase in Bacillus subtilis[J]. Acta Microbiologica Sinica, 2021, 61(10): 3222-3234 (in Chinese). |
| [11] | GREEN ER, MECSAS J. Bacterial secretion systems: an overview[J]. Microbiology Spectrum, 2016, 4(1): 10. |
| [12] | PALMER T, BERKS BC. The twin-arginine translocation (Tat) protein export pathway[J]. Nature Reviews Microbiology, 2012, 10: 483-496 DOI:10.1038/nrmicro2814. |
| [13] | FREUDL R. Signal peptides for recombinant protein secretion in bacterial expression systems[J]. Microbial Cell Factories, 2018, 17(1): 52 DOI:10.1186/s12934-018-0901-3. |
| [14] | WANG N, GUAN F, LV X, HAN D, ZHANG Y, WU N, XIA X, TIAN J. Enhancing secretion of polyethylene terephthalate hydrolase PETase in Bacillus subtilis WB600 mediated by the SPamy signal peptide[J]. Letters in Applied Microbiology, 2020, 71(3): 235-241 DOI:10.1111/lam.13312. |
| [15] | FENG Y, LIU S, JIAO Y, WANG YL, WANG M, DU GC. Gene cloning and expression of the L-asparaginase from Bacillus cereus BDRD-ST26 in Bacillus subtilis WB600[J]. Journal of Bioscience and Bioengineering, 2019, 127(4): 418-424 DOI:10.1016/j.jbiosc.2018.09.007. |
| [16] | FU G, LIU JL, LI JS, ZHU BW, ZHANG DW. Systematic screening of optimal signal peptides for secretory production of heterologous proteins in Bacillus subtilis[J]. Journal of Agricultural and Food Chemistry, 2018, 66(50): 13141-13151 DOI:10.1021/acs.jafc.8b04183. |
| [17] | NIELSEN H, TSIRIGOS KD, BRUNAK S, von HEIJNE G. A brief history of protein sorting prediction[J]. The Protein Journal, 2019, 38(3): 200-216 DOI:10.1007/s10930-019-09838-3. |
| [18] | OWJI H, NEZAFAT N, NEGAHDARIPOUR M, HAJIEBRAHIMI A, GHASEMI Y. A comprehensive review of signal peptides: structure, roles, and applications[J]. European Journal of Cell Biology, 2018, 97(6): 422-441. |
| [19] |
李川敏, 张玮祎, 张献, 刘红星, 徐大庆. 谷氨酸棒杆菌信号肽探测载体的构建及强信号肽筛选[J]. 河北农业大学学报, 2021, 44(4): 69-75.
LI CM, ZHANG WY, ZHANG X, LIU HX, XU DQ. Construction of a signal peptide-probe vector and screening of strong signal peptides in Corynebacterium glutamicum[J]. Journal of Hebei Agricultural University, 2021, 44(4): 69-75 (in Chinese). |
| [20] |
胡哲辉, 徐娟, 卞光凯. 自动化高通量技术在天然产物生物合成中的应用[J]. 合成生物学, 2023, 4(5): 932-946.
HU ZH, XU J, BIAN GK. Application of automated high-throughput technology in natural product biosynthesis[J]. Synthetic Biology Journal, 2023, 4(5): 932-946 (in Chinese). |
| [21] | LIU Y, ZHENG H, ZHAN GH, QIN W, TIAN L, LI WL. Establishment of an efficient transformation protocol and its application in marine-derived Bacillus strain[J]. Science China Life Sciences, 2014, 57(6): 627-635. |
| [22] | LIU Y, CHENG HJ, LI HN, ZHANG YZ, WANG M. A programmable CRISPR/Cas9 toolkit improves lycopene production in Bacillus subtilis[J]. Applied and Environmental Microbiology, 2023, 89(6): e0023023. |
| [23] | MILLER GL. Use of dinitrosalicylic acid reagent for determination of reducing sugar[J]. Analytical Chemistry, 1959, 31(3): 426-428. |
| [24] |
甄杰, 胡政, 李树芳, 徐健勇, 宋诙. 一个新型耐热普鲁兰酶的结构与功能[J]. 生物工程学报, 2014, 30(1): 119-128.
ZHEN J, HU Z, LI SF, XU JY, SONG H. Structure and function of a novel thermostable pullulanase[J]. Chinese Journal of Biotechnology, 2014, 30(1): 119-128 (in Chinese). |
 2024, Vol. 64
2024, Vol. 64




