中国科学院微生物研究所,中国微生物学会
文章信息
- 何瑞鹏, 徐婉茹, 刘一萌, 张杰. 2024
- HE Ruipeng, XU Wanru, LIU Yimeng, ZHANG Jie.
- 若尔盖沙化土地治理土壤微生物群落与功能基因比较研究
- Comparison of soil microbial communities and functional genes in sandy land management of Zoige
- 微生物学报, 64(6): 2071-2090
- Acta Microbiologica Sinica, 64(6): 2071-2090
-
文章历史
- 收稿日期:2023-10-19
- 网络出版日期:2023-12-25
2. 生物资源与生态环境教育部重点实验室, 四川 成都 610064
2. Key Laboratory of Bio-resources and Eco-environment of Ministry of Education, Chengdu 610064, Sichuan, China
草原在全球范围内约占40%的陆地面积,管理的永久性牧场和草甸则占据了约25%。在青藏高原,由于高海拔、气候寒冷、空气稀薄和二氧化碳浓度低等环境因素,主要植被类型为高寒草地[1-2]。若尔盖高寒草原位于青藏高原东北部,是中国三大草原牧区之一,也是亚洲最好的天然牧场之一。近年来受全球气候变暖因素与过度放牧、采矿、湿地排水等人为因素影响,导致当地草原的净初级生产力下降[3]。由于土壤肥力降低、侵蚀加剧、沙尘增多等因素,草原上层草甸发生退化,导致脆弱的高寒草地系统生态失衡,逐渐向荒漠化发展,进而丧失草原生态服务功能。高寒草地荒漠化对当地生态环境气候、居民健康以及经济收入等方面均会造成负面影响,同样对生态可持续发展造成潜在威胁[4]。针对若尔盖高寒草地荒漠化问题,当地政府在国家政策支持下开展了一系列沙漠治理和预防工作。在政策方面实行补偿奖励措施保障当地牧民的生活收入[5];在沙漠治理与预防过程中采用围栏禁牧、人工建立草方格、布置柳沙障和补种牧草等措施[6];长期沙漠治理和预防措施有效地遏制了高寒草地荒漠化进程。
Liu等研究了毛乌素沙漠干旱植物凋落物对土壤微生物群落的影响,发现草本和灌木定植过程通过增加凋落物累积质量,降低了土壤pH,并加速了土壤胞外酶活性,从而改善了土壤微生物群落结构和共生关系[7]。土壤微生物是凋落物分解和土壤矿化等过程的重要参与者,研究表明细菌群落结构和多样性可以反映土壤生态变化,土壤微生物可以辅助植物修复来促进高陡岩质边坡生态修复[8]。在青藏高原重度退化草地建植的单播人工草地显著提高了草地地上生物量和禾本科牧草占比,能够有效恢复黑土滩退化草地[9];肖颖等在研究青藏高原高寒地区不同人工草地建立对土壤质量的影响时发现,以羊茅属为主的人工草地显著提高了土壤有机质、全氮和全磷含量,土壤酶活性以及微生物碳氮磷含量均显著提高,证明该类型人工草地的土壤改良效果最佳[10]。关于若尔盖高寒沙地的人工草方格修复过程中土壤微生物群落变化研究鲜有报道,因此,认为在高寒荒漠植被修复过程中,人工建立的草方格同样可以有效提高土壤养分含量,这对高寒草地生态系统恢复过程中土壤养分、植被定植方面具有重要作用。
近年来,随着土壤微生物研究技术在生态恢复研究过程中的发展,因此引起了广大研究者的关注。土壤微生物作为土壤生态系统中元素循环的重要参与者,在生态恢复过程中可以将土壤微生物与土壤养分结合在一起作为生态恢复的参考指标[11]。本研究主要针对若尔盖高海拔地区独特性的气候和生态环境,探讨沙地修复过程中植被恢复如何响应土壤环境中养分因素,进一步探究土壤微生物群落数量、结构组成的变化。
1 材料与方法 1.1 样点概述如图 1所示,研究样点位于四川省阿坝州若尔盖县辖曼乡(33°43′3″N, 102°29′28″E),地处若尔盖高寒草原,平均海拔3 500 m。该地属于高原寒带湿润季风气候,年平均气温在0 ℃左右,年平均降水量在500−600 mm之间,紫外线辐射强,昼夜温差大;植被生长季主要集中在每年5−9月份[12],持续161−273 d,植被生长高峰期主要集中在夏季[13];该地自1995年以来,因气候因素和过度放牧导致草地逐渐退化形成沙地[14]。研究样点中BS组为高寒草地荒漠化形成的裸沙地,属于砂质土壤;SS组和AS组分别为2017年和2011年实行围栏禁牧+人工草方格的方式进行人工植被修复,并经过长期自然恢复形成的40%−60%和80%以上植被覆盖的沙地,属于砂质土壤;GL组为围栏外的正常高寒草地,属于高寒草甸土壤。人工草方格规格为3 m×2 m,草方格中植被主要以多年生草本植物沙生苔草(Carex praeclara)和赖草(Leymus secalinus)为主,每年当地牧民进行秋冬牧场搬迁时会进行适度放牧。在长时间自然植被覆盖条件下,该地区由流动沙丘逐步演变为固定沙丘,从裸露的沙地演化为具有植被覆盖的沙地。

|
| 图 1 若尔盖高寒沙地采样点概况 Figure 1 Overview of sampling sites in the Zoige alpine sands. |
1.2 土壤样品采集
于2022年6月份前往研究区域进行土壤样品采样。选取围栏内的裸沙地BS组(小于10%植被覆盖)、半植被覆盖沙地SS组(40%−60%植被覆盖)、全植被覆盖沙地AS组(80%以上植被覆盖)和围栏外的未退化草地GL组4种不同生境样点,每个样点中随机选取3个2 m×2 m的样方(样方间距为50 m),土壤样品采集深度为5−15 cm,清除表面植物部分后,采用五点取样法使用直径为35 mm的土壤螺旋钻采集原位土壤样品,将5份样品均匀混合为一份土壤样品,重复3次,每份土壤样品分为2份,共采集24份土壤样品。1份放进液氮罐储存用于后期DNA提取,1份放进车载冰箱进行4 ℃保存运输,用于测定土壤理化性质。
1.3 土壤理化分析将所有土壤样本自然风干后通过2 mm筛子测量土壤成分含量。土壤总氮(total nitrogen content, TN)含量利用凯氏定氮法进行测定[15],全磷(total phosphorus, TP)与速效磷(available phosphorus, AP)采用钼锑抗比色法测定[16],全钾(total potassium content, TK)与速效钾(available potassium, AK)采用火焰光度法进行测定[17],土壤有机碳(soil organic carbon, SOC)含量采用K2CrO7-H2SO4法测定[18],将土壤与蒸馏水按1:2.5 (质量体积比)进行混合振荡,用pH仪测定pH值。
1.4 DNA提取和测序使用Omega E.Z.N.A. Stool DNA Kit试剂盒(Omega Bio-Tek公司)提取土壤微生物组DNA。提取后的DNA通过1%的琼脂糖凝胶电泳分析DNA质量,然后用Qubit 2.0 (ThermoFisher Scientific™公司)对DNA精确定量。将检测合格的DNA样品送往北京奥维森基因科技有限公司进行文库建立和鸟枪宏基因组测序。按照Next Ultra Ⅱ DNA Library Prep Kit for Illumina (New England BioLabs公司)试剂盒说明,在每个样品中引入索引码后,建立测序文库。为保证文库质量,文库建立后用Qubit 2.0进行初步定量并将浓度稀释至2 ng/μL,使用Agilent 2100对文库的插入片段进行检测,符合预期后用qPCR方法对文库的有效浓度进行准确定量(有效浓度 > 3 nmol/L)。文库质检合格后,利用Illumina NovaSeq PE150平台进行测序。
1.5 数据质控与基因组组装使用fastp (https://github.com/OpenGene/fastp, v0.20.0)[19]对原始数据序列进行拆分和质量剪切等操作,生成高质量的数据。然后使用MEGAHIT[20] (https://git hub.com/voutcn/megahit, v1.1.2)对优化序列进行拼接组装,根据kmer间的重叠关系,构建De-Brujin graph,获得contigs,筛选500 bp以上的contigs进行统计并用于后续分析。
使用Prodigal[21]对拼接结果中的contigs (> 500 bp)进行开放阅读框(open reading frame, ORF)预测。然后采用CD-HIT软件进行去冗余后,选取最长的序列为代表性序列来构建非冗余基因集。使用bowtie2[22]软件将每个样品的clean reads与非冗余基因集进行BLAST比对(95% identity),计算基因在对应样品中的丰度。
1.6 物种分类分析与功能注释非冗余基因集采用Diamond[23] (v0.8.35)软件将细菌、真菌、古生菌和病毒序列上的unigenes与NR数据库(version 2021.11)进行BLAST序列比对(期望值1E−5),物种注释后,使用物种对应的基因丰度总和计算该物种的丰度。将非冗余基因集的序列分别与KEGG、eggNOG和CAZy数据库进行BLAST比对,进行基因功能注释。
1.7 qPCR定量和统计分析使用Omega E.Z.N.A. Stool DNA Kit (Omega Bio-Tek公司)提取土壤基因组模板DNA。采用引物对515F (5′-GTGCCAGCMGCC GCGGTAA-3′)和802R (5′-TACNVGGGTATCTA ATCC-3′)、ITS1F (5′-GCATCGATGAAGAACGC AGC-3′)和ITS1R (5′-TCCTCCGCTTATTGATAT GC-3′)构建质粒标准品,利用已知浓度的质粒标准品计算得到目标基因的拷贝数后,用目标基因拷贝数与CT值构建标准曲线。进一步对样本中细菌16S rRNA基因片段和真菌ITS片段的总拷贝数进行qPCR定量分析,qPCR扩增使用2×Taq SYBR Green qPCR Mix (Innovagene公司)试剂,反应体系(20 μL):2×Taq SYBR Green qPCR Mix 10 μL,上、下游引物(终浓度0.4 μmol/L)各0.8 μL,Rox 0.4 μL,ddH2O 7.0 μL,模板DNA 1 μL。反应条件:在QuantStudio3仪器(ThermoFisher Scientific公司)上先94 ℃预热2 min;94 ℃ 15 s,60 ℃ 45 s,72 ℃ 15 s,进行40个循环[24]。
数据分析采用SPSS 22.0软件中的Duncan检验法进行差异性分析。通过Origin 2019和GraphPad Prism 9.0软件对数据进行统计和作图。利用R语言软件分析土壤微生物群落结构组成与环境因子进行冗余分析(redundancy analysis, RDA)。
2 结果与分析 2.1 土壤理化因子含量状况土壤理化因子含量变化如图 2所示,不同处理组间土壤养分含量存在显著性差异,人工建立草方格的沙地土壤中,随着植被修复程度的增加,土壤养分含量呈现上升趋势,其中全氮(TN)、速效磷(AP)、速效钾(AK)、土壤有机碳(SOC)的含量均随着植被修复程度的增加呈现上升趋势,即AS组 > SS组 > BS组。土壤pH随着植被修复程度的增加呈现下降趋势,表明植被修复对土壤酸碱性产生了影响。与正常草地GL组相比,BS、SS和AS组的全氮(TN)、全磷(TP)和土壤有机碳(SOC)的含量显著低于GL组,GL组的速效磷(AP)、速效钾(AK)和pH低于BS组、SS组和AS组。以上结果表明植被修复对土壤养分含量和pH值有显著影响。
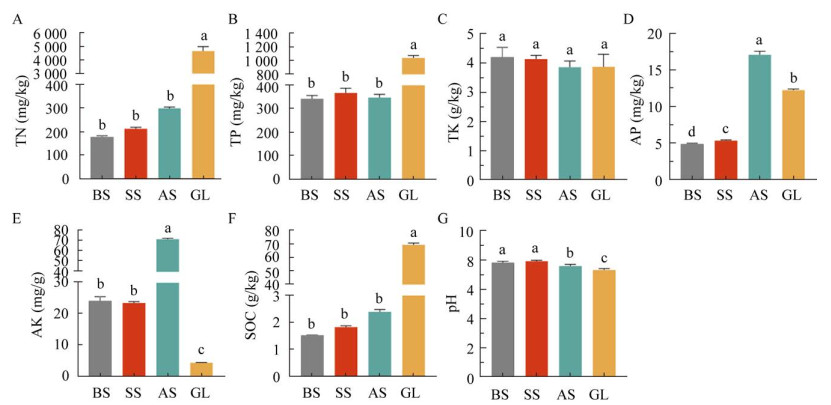
|
| 图 2 不同样点间土壤理化因子含量差异 Figure 2 Differences in soil physicochemical factors among different sites. A: Total nitrogen (TN) content. B: Total phosphorus (TP) content. C: Total potassium (TK) content. D: Instant phosphorus (AP) content. E: Instant potassium (AK) content. F: Soil organic carbon (SOC) content. G: pH value (pH). BS: Bare sand land; SS: Semi-vegetated sand land; AS: All-vegetated sand land; GL: Grassland. Data are shown as the mean of three replicates, with error bars representing the standard error of the mean might, different letters represent significant differences (P < 0.05). |
2.2 土壤微生物宏基因组测序结果
宏基因组测序数据共获得952 020 230份原始reads,平均每个样本约为79 335 019份。经过数据质控后,每个样本产生了大约78 242 259份clean reads。利用MEGAHIT进行优化序列拼接组装后,BS、SS、AS和GL不同处理组中3个生物学重复样本平均生成194 363±81 686、270 604±64 191、254 282±83 241和428 690± 54 169个contigs。对获得的contigs进行ORF预测并进行去冗余后获得非冗余基因集,经bowtie2软件与样本中的clean reads比对后,共获得2 622 735个独特的基因(unique_gene)。不同样本间的基因数目在702 837−1 105 584之间。
2.3 土壤微生物数量与组装多样性不同处理组间的土壤微生物数量存在显著差异性。通过对样本中细菌16S rRNA基因(515F-802R)和真菌ITS片段(ITS1F-ITS1R)进行实时荧光定量检测,发现细菌16S rRNA基因拷贝数在(2.46±0.57)×106–(9.94±5.40)×108 copies/g范围内,而真菌ITS片段拷贝数在(3.00±0.24)×104−(2.87±2.23)×106 copies/g范围内。结果如图 3所示,随着草方格修复程度升高,沙地土壤中的细菌16S rRNA基因拷贝数与真菌ITS拷贝数呈现上升趋势,GL组的细菌基因拷贝数和AS组的真菌基因拷贝数均显著高于其他组(P < 0.05)。
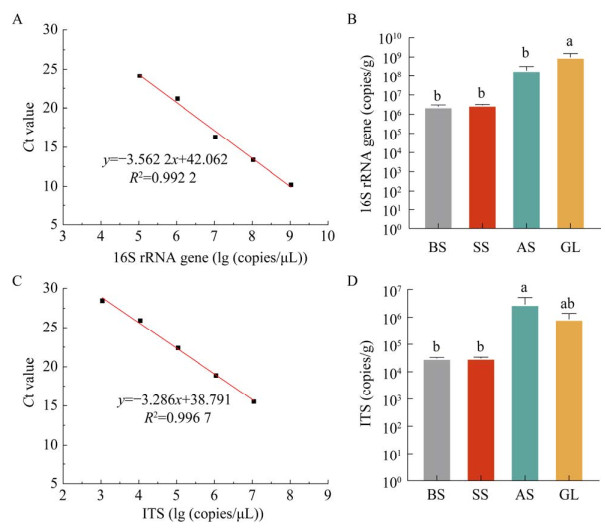
|
| 图 3 不同处理组间土壤细菌与真菌拷贝数差异分析 Figure 3 Differential analysis of copy number between soil bacterial and fungal among different treatment groups. A: 16S rRNA target gene copy number standard curve. B: Soil bacterial 16S rRNA gene copy number differences among different treatment groups. C: ITS target gene copy number standard curve. D: Soil fungal ITS gene copy number differences among different treatment groups. BS: Bare sand land; SS: Semi-vegetated sand land; AS: All-vegetated sand land; GL: Grassland. Data are shown as the mean of three replicates, with error bars representing the standard error of the mean might, different letters represent significant differences (P < 0.05). |
所有土壤样本共鉴定出41个原核生物门和5个真菌生物门。如图 4A所示,平均相对丰度≥1.00%的原核细菌门主要有变形菌门(Proteobacteria, 40.27%)、酸杆菌门(Acidobacteria, 21.88%)、放线菌门(Actinobacteria, 11.61%)、绿湾菌门(Chloroflexi, 9.34%)、芽单胞菌门(Gemmatimonadetes, 4.30%)、棒状杆菌门(Rokubacteria, 3.01%)、硝化螺旋菌门(Nitrospirae, 2.39%)和拟杆菌门(Bacteroidetes, 2.38%)。以上细菌门的相对丰度约占细菌总丰度的95.00%。在属水平上,假单胞菌属(Pseudomonas)、硝化螺菌属(Nitrospira)、慢生根瘤菌属(Bradyrhizobium)、鞘氨醇单胞菌属(Sphingomonas)和盖亚女神菌属(Gaiella)是所有样本中丰度较高的5个属(图 4B)。真菌群落中,毛霉菌门(Mucoromycota, 34.26%)、子囊菌门(Ascomycota, 30.49%)和担子菌门(Basidiomycota, 29.34%)是所有土壤样本中相对丰度较高的3个门(图 4C)。以上真菌门的丰度约占真菌总丰度的94.00%。
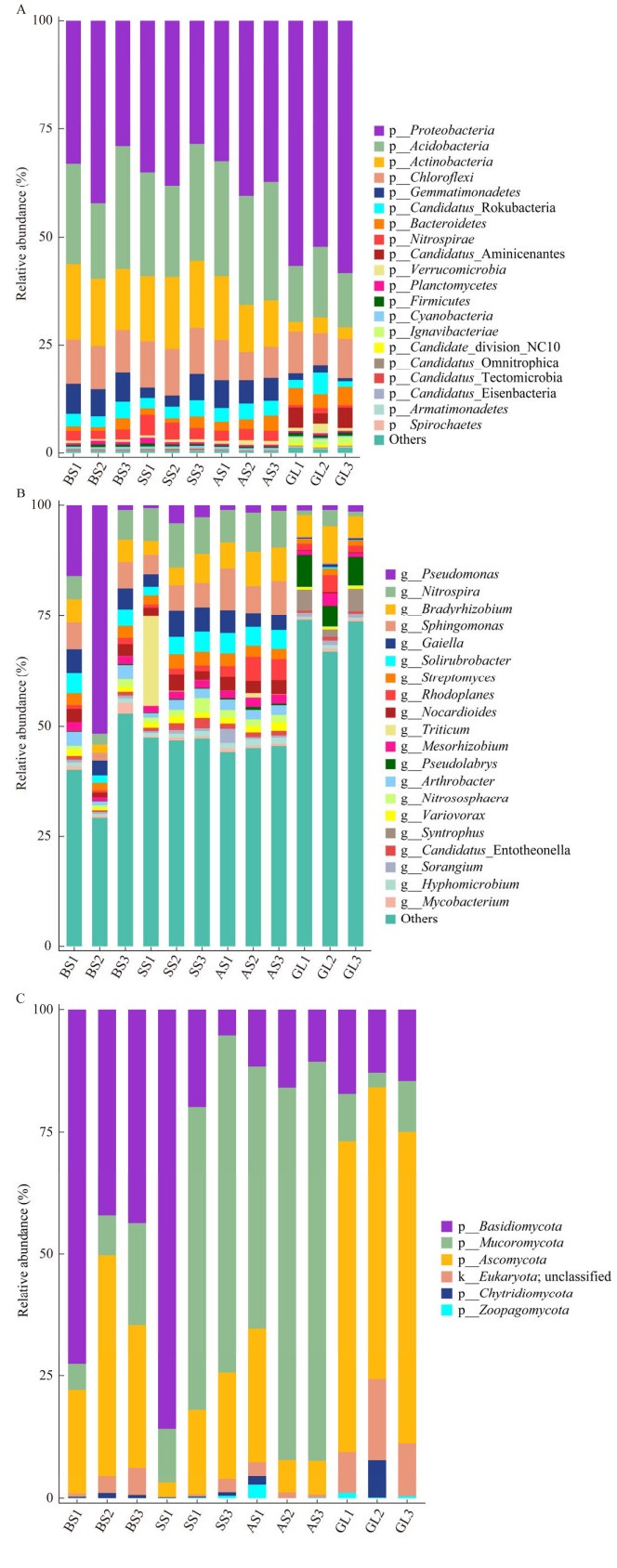
|
| 图 4 宏基因组数据注释土壤微生物群落组成图 Figure 4 Map of soil microbial community composition annotated with metagenomic data. A: Map of community composition at the level of bacterial phylum. B: Map of community composition at the level of bacterial genera. C: Map of community composition at the level of fungal phylum. BS: Bare sand land; SS: Semi-vegetated sand land; AS: All-vegetated sand land; GL: Grassland. |
通过对不同处理组间微生物的相对丰度差异性分析发现(图 5A),在细菌门分类水平上,变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、疣微菌门(Verrucomicrobia)和酸杆菌门(Acidobacteria)的相对丰度随着修复程度增加呈现上升趋势。放线菌门(Actinobacteria)的相对丰度随着植被修复程度增加而下降,而且在草地中的相对丰度最低。如图 5B所示,在细菌属水平上,随着植被修复程度的增加假单胞菌属(Pseudomonas)的相对丰度呈下降趋势,而鞘氨醇单胞菌属(Sphingomonas)、慢生根瘤菌属(Bradyrhizobium)、硝化螺旋菌属(Nitrospira)、红游动菌属(Rhodoplanes)、类诺卡氏菌属(Nocardioides)和贪噬菌属(Variovorax)的相对丰度呈上升趋势。如图 5C所示,在真菌门分类水平上,毛霉菌门(Mucoromycota)的相对丰度随着修复程度提高而显著增加,但在草地中的相对丰度最低,而子囊菌门(Ascomycota)和担子菌门(Basidiomycota)的相对丰度随着修复程度的提高呈现下降趋势,尤其是在草地中子囊菌门(Ascomycota)的相对丰度最高。
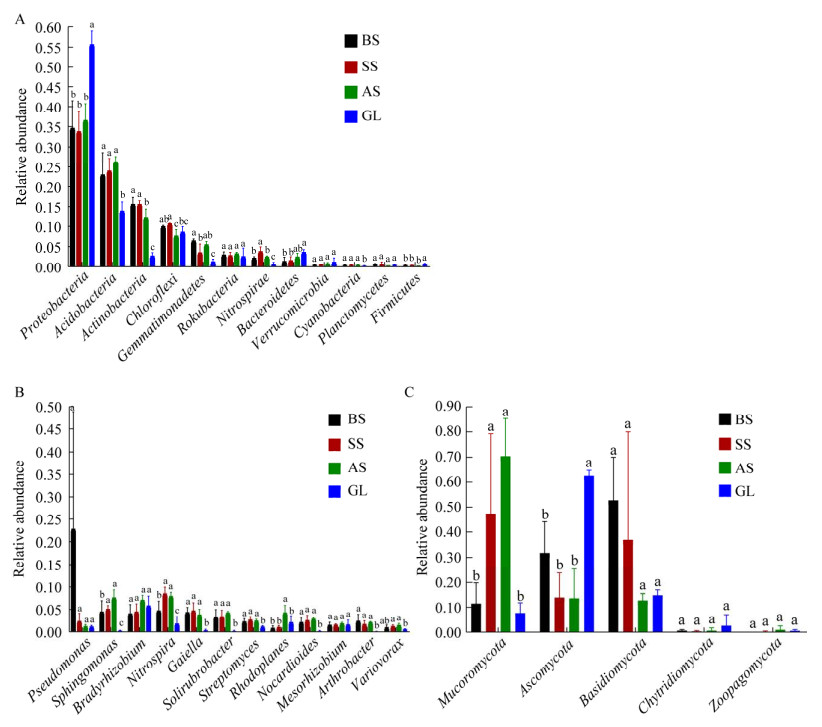
|
| 图 5 不同分类水平土壤微生物相对丰度差异性分析 Figure 5 Differential analysis of relative abundance of soil microorganisms at different taxonomic levels. A: Differential analysis of relative abundance at the level of bacterial phylum. B: Differential analysis of relative abundance at the level of bacterial genera. C: Differential analysis of relative abundance at the level of fungal phylum. BS: Bare sand land; SS: Semi-vegetated sand land; AS: All-vegetated sand land; GL: Grassland. Data are shown as the mean of three replicates, with error bars representing the standard error of the mean might, different letters represent significant differences (P < 0.05). |
2.4 土壤微生物群落功能潜力
使用BLAST工具将非冗余基因集的氨基酸序列与KEGG数据库进行比对后,获得多个KEGG二级通路,这些通路主要涵盖了氨基酸代谢、能量代谢、异源生物降解和代谢、核苷酸代谢、膜转运、多糖合成与代谢、信号转导和脂质代谢等多个功能类别。
与BS组裸沙地相比,SS组、AS组和GL组中富集的功能途径主要涉及微生物之间的相互作用、微生物和植物之间相互作用以及能量代谢(图 6)。这些功能途径包括微生物群体感应、氧化磷酸化、硫代谢,以及原核生物碳固定等;相反地,出现下降趋势的功能途径参与了碳水化合物代谢、膜转运、氨基酸代谢、聚糖生物合成与代谢、脂质代谢以及核苷酸代谢等相关途径。这些参与微生物养分循环以及微生物之间相互作用的功能途径,包括原核生物的碳固定、O-抗原核苷酸糖生物合成、磷酸戊糖途径、氧化磷酸化、硫代谢和群体感应等,反映了土壤微生物在维持土壤生态平衡和功能稳定性方面的重要作用。这些功能途径的变化与不同处理组之间土壤性质、养分含量以及植被类型的差异密切相关。
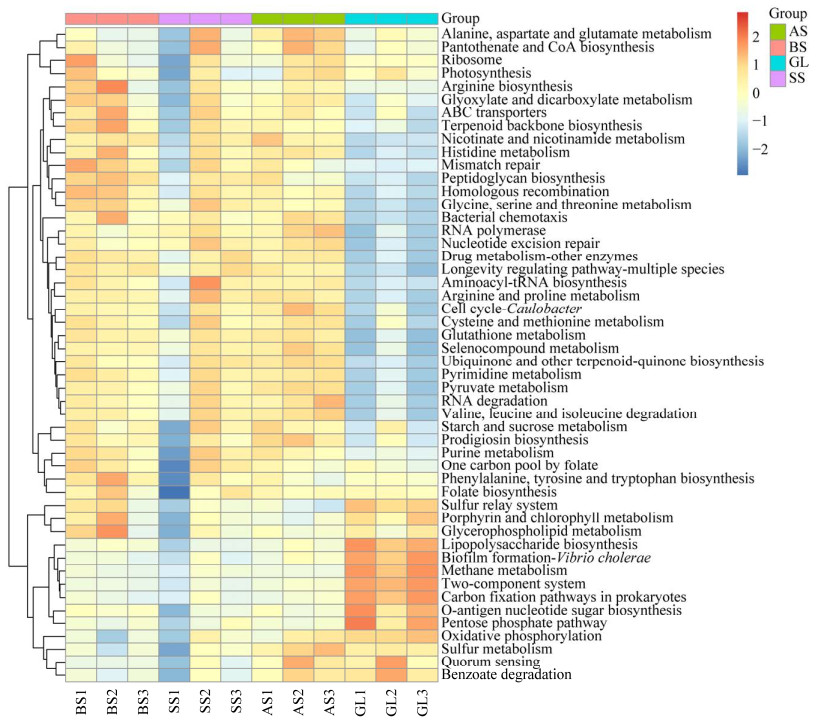
|
| 图 6 不同处理组间土壤微生物宏基因组KEGG三级功能通路分析 Figure 6 KEGG tertiary functional pathway analysis of soil microbial metagenomic among different treatment groups. BS: Bare sand land; SS: Semi-vegetated sand land; AS: All-vegetated sand land; GL: Grassland. |
2.5 土壤微生物碳、氮、磷元素循环潜力
根据土壤理化性质研究结果,进一步研究高寒沙地中土壤微生物养分循环状况。通过细菌与土壤理化因子含量的相关性分析:发现细菌门中变形菌门(Proteobacteria)、疣微菌门(Verrucomicrobia)、拟杆菌门(Bacteroidetes),细菌属中假单胞菌属(Pseudomonas)、红游动菌属(Rhodoplanes)、慢生根瘤菌属(Bradyrhizobium)均与SOC、TN、TP和AP等含量呈显著正相关。因此,随着植被修复程度的不断提升,发现沙地土壤中养分含量得到了有效改善,尤其是与碳、氮、磷元素相关的养分含量变化更加显著。
在碳循环方面(图 7),随着植被修复程度的提高,土壤有机碳氧化过程涉及纤维素降解、半纤维素脱支、几丁质降解和其他寡糖降解等通路的功能基因得到富集。说明在长期植被修复过程中,植物根系分泌物和植物残体的输入为土壤微生物提供了丰富的有机碳源,进而提高了微生物的活性,促进了土壤中元素循环。
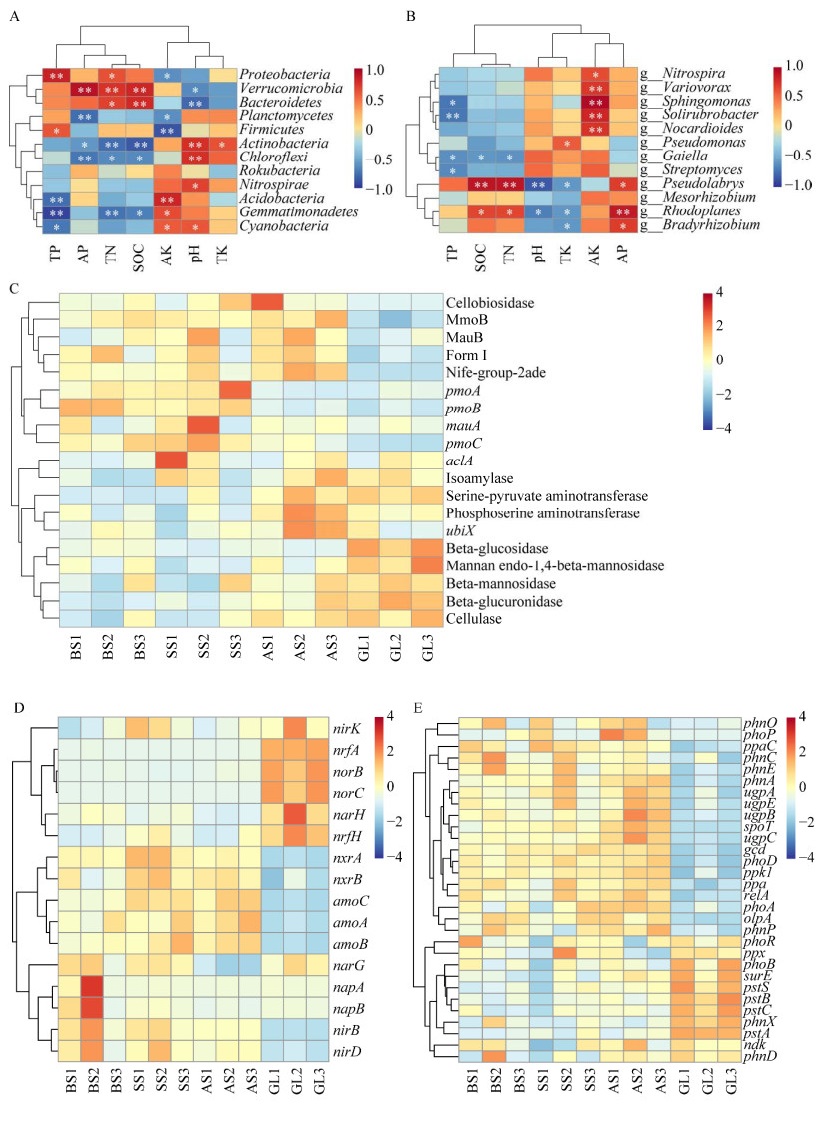
|
| 图 7 细菌与土壤理化因子相关性分析以及宏基因组碳、氮和磷元素相关的元素循环基因分析 Figure 7 Correlation analysis of bacteria with soil physicochemical factors and metagenomic carbon, nitrogen and phosphorus related elemental cycling gene analysis. A: Correlation analysis between the level of bacterial phylum and soil physicochemical factors. TN: Total nitrogen content; TP: Total phosphorus content; TK: Total potassium content; AP: Instant phosphorus content; AK: Instant potassium content; SOC: Soil organic carbon content; pH: pH value. B: Correlation analysis between the level of bacterial genera and soil physicochemical factors. C: Heat map of functional genes for carbon cycling. D: Heat map of functional genes for nitrogen cycling. E: Heat map of functional genes for phosphorus cycling. BS: Bare sand land; SS: Semi-vegetated sand land; AS: All-vegetated sand land; GL: Grassland. |
在氮循环过程中(图 7),在BS组、SS组、AS组中注释到了与氨氧化、亚硝酸盐氧化相关的基因(amoCAB、nxrAB),以上两种基因随着植被修复程度的提高呈现出明显的富集趋势。amoCAB基因簇和nxrAB基因簇的富集表明植被修复过程中土壤微生物的氨氧化作用与硝化作用增强,促进了高寒沙地土壤中氮素的循环和利用。
随着植被修复程度的提高,与其他养分含量相比,土壤中有效磷(AP)的含量变化较为显著,这表明沙地修复过程中提升了土壤磷元素循环的潜力。在磷元素循环方面(图 7),参与有机磷矿化过程的基因(phoA、phoD)、无机磷增溶过程的基因(gcd)以及参与磷转运过程的磷酸转运蛋白基因(ugpABCE)在沙地修复过程中呈显著富集趋势;在酶合成方面,参与编码膦酰基乙酸水解酶基因(phnA)和无机焦磷酸酶基因(ppa)同样呈现显著富集趋势。这些发现揭示了植被修复在改善土壤养分含量和促进土壤元素循环方面的潜力。
2.6 细菌与土壤性质RDA分析通过RDA分析方法,进一步研究了土壤养分含量对细菌门水平与属水平的影响。RDA1与RDA2分别解释了细菌门组成的71.27%和13.73%的变异。分析表明,TK、AK和AP对细菌门水平的多样性有显著影响。AK与变形菌门(Proteobacteria)、疣微菌门(Verrucomicrobia)和拟杆菌门(Bacteroidetes)之间呈显著正相关;TK和pH与棒状杆菌门(Rokubacteria)、酸杆菌门(Acidobacteria)和芽单胞菌门(Gemmatimonadetes)之间呈显著正相关;SOC、TN和TP与硝化螺旋菌门(Nitrospirae)、放线菌门(Actinobacteria)和绿湾菌门(Chloroflexi)之间呈显著正相关(图 8A)。在属水平上,RDA1与RDA2分别为47.27%和24.95%。AK和AP对细菌属水平的多样性有显著影响。AP与慢生根瘤菌属(Bradyrhizobium)、中慢生根瘤菌属(Mesorhizobium)、红游动菌属(Rhodoplanes)和链霉菌属(Streptomyces)呈显著正相关;SOC、TN和TP与硝化螺菌属(Nitrospira)、鞘氨醇单胞菌属(Sphingomonas)、类诺卡氏菌属(Nocardioides)和Solirubrobacter属呈显著正相关(图 8B)。综上所述,RDA分析揭示了土壤养分含量与细菌门和属水平多样性之间的显著关系,这些发现为更深入地理解土壤微生物群落对养分变化的响应提供了重要依据。

|
| 图 8 细菌与土壤理化因子间RDA冗余分析 Figure 8 RDA redundancy analysis between bacteria and soil physicochemical factors. BS: Bare sand land; SS: Semi-vegetated sand land; AS: All-vegetated sand land; GL: Grassland. |
3 讨论 3.1 土壤理化性质比较
高寒沙地土壤养分贫瘠致使生物多样性水平低于其他生境[25]。研究发现种植本土先锋植物(沙生苔草和赖草)可以有效改善植物根际土壤养分条件,植物修复在改善土壤养分条件方面具有重要作用,因此可以利用原生植物进行长期沙漠自然修复[26]。沙地修复过程中全氮(TN)、速效磷(AP)、速效钾(AK)和土壤有机碳(SOC)含量随着植被修复程度提高而上升,其中速效磷含量变化幅度最大。原因是先锋植物在沙漠中定植后可以调节沙地土壤微生态系统的水和碳通量[27]。植物地上部分通过光合作用固定无机碳来提高土壤有机碳储量[28]。干旱胁迫下,植物在根部形成根鞘并释放根系分泌物,这些物质包括氨基酸、糖类、羧酸盐以及次生代谢物,为土壤微生物提供有机碳源;植物根系分泌的黏液与土壤颗粒形成土壤团聚体以帮助植物稳定[29]。植物根系分泌物可以作为有机质输入来提高土壤有机碳含量,同时在植物根际富集的微生物可以溶解土壤矿物质为植物生长提供必需元素,土壤微生物在分解植物有机残体的同时释放出无机磷,进而提高土壤中速效磷含量,促进土壤养分循环潜力。
3.2 土壤微生物群落数量以及分类学变化土壤微生物群落组成和数量变化是反映土壤功能以及土壤健康状况的重要生物指标[30]。研究发现BS组裸沙地土壤中的细菌和真菌数量在4个处理组间最低,为(2.46±0.57)×106 copies/g和(3.00±0.24)×104 copies/g,但高于非洲纳米比亚西部纳米布沙漠中的土壤细菌和真菌数量(8.30±3.3)×105 copies/g和(2.20±0.8)×104 copies/g[31],这可能与若尔盖高寒沙地当地的气候条件和降水量有关。在植物修复阶段中,土壤微生物的绝对丰度和群落结构受植物发育阶段的影响,在营养限制情况下,研究发现苔草(Carex arenaria)通过根系分泌挥发性有机化合物的方式可以促进土壤中某些类群的细菌(Burkholderia、Dyella、和Pseudomonas)向根系迁移[32],即通过光合作用固定的碳以根系分泌物的形式排入土壤,根系分泌物的数量与组成随植物发育阶段、营养与环境条件,以及其他因素的变化而不同[33]。沙生赖草在高海拔环境的适应过程中与营养丰富的土壤环境相比,同样存在不同类型内生细菌群落结构[34]。本研究中,随着植被修复程度的提高,土壤微生物群落中细菌和真菌数量呈现上升趋势,SS组和AS组的细菌和真菌的数量均高于BS组,AS组中真菌的数量高于GL组,说明植物修复对沙地土壤养分条件的改善和土壤微生物群落中细菌和真菌数量的提高起到了积极作用。
在草地恢复过程中,土壤有机质(SOM)是由微生物对凋落物和死亡动物的转化形成的,土壤细菌和真菌对SOM的分解有很大的贡献,这是由元素比例(C、N、P)的平衡决定的[35]。细菌倾向于分解不稳定化合物,在分解初期主导秸秆降解,真菌主要在分解的最后阶段分解更多的研磨性物质。元素C: N: P比对细菌多样性的影响低于真菌多样性,表明真菌多样性更容易受到草地恢复的影响。原因可能是土壤真菌可以产生大多数细胞外酶来降解细菌无法获得的复杂大分子,最终导致对土壤真菌的影响高于细菌[35]。AS组中的真菌数量显著高于GL组,这可能与真菌对地上生物量以及土壤养分变化的响应有关[36]。
沙地修复过程中SS组和AS组的人工草方格与BS组裸沙地中的微生物群落组成在分类学水平上保持高度一致,这意味着土壤微生物群落中可能存在高度功能冗余。研究发现在低营养环境中,变形杆菌门中的阿尔法变形杆菌因其代谢类型多样性占主导地位[37]。如图 5B所示,人工建立草方格后,SS组和AS组中与氮循环相关的慢生根瘤菌属(Bradyrhizobium)、硝化螺菌属(Nitrospira)以及类诺卡氏菌属(Nocardioides)等细菌种类相对丰度呈现上升趋势,这可能与草方格建立后改善了土壤养分条件从而促进了微生物养分循环有关。如图 5A所示,放线菌门与土壤养分环境呈现负相关,随着植被修复程度的提高,土壤养分水平上升,营养条件变好,其相对丰度呈现下降趋势,该变化与其他文章研究放线菌门的相对丰度与养分含量呈负相关的结论一致[34, 38-39]。这可能与放线菌门因其代谢多样性在沙地生态系统中具有高度适应性有关。
3.3 土壤微生物群落功能变化潜力在植物修复过程中参与微生物养分循环以及微生物之间相互作用的功能途径得到提升,包括原核生物的碳固定、O-抗原核苷酸糖生物合成、磷酸戊糖途径、氧化磷酸化、硫代谢和群体感应等。这可能是因为植物残体的输入为土壤有机质的积累提供了物质基础,进一步为土壤微生物群落活动提供能量基础,促进微生物的生长和繁殖,土壤微生态系统中碳存储、植物养分有效性提取、土壤肥力维持以及养分循环功能等均受土壤微生物的强烈调控[40];Wang等发现在苹果园中种植三叶草作物后,土壤养分积累和微生物结构多样性变化的同时,土壤微生物表现出对碳水化合物、氨基酸和聚合物作为碳源的更高偏好,进而促进了土壤微生物的功能多样性[41]。
3.4 土壤微生物群落元素循环潜力的变化碳、氮和磷被认为是参与生物地球化学过程的重要元素,植物残体、根系分泌物和土壤有机碳为参与元素循环的微生物提供碳源[42]。土壤有机碳的形成途径(包括淋滤途径和微生物途径)以及植物凋落物与根系的空间分布差异被认为是影响土壤有机碳积累的重要因素[43]。植物的地上部分覆盖在土壤表面形成了天然的物理屏障,可以降低水分蒸发提高土壤中水的通量[44]。在土壤碳循环过程中,与纤维素降解和多糖大分子化合物降解相关的基因在植被修复过程中富集。这一现象可能是因为随着植物修复程度的提高,植物残体输入增加,从而导致土壤微生物群落数量增加。与此同时,分解植物残体的基因增加,进而促进了凋落物的分解,提高了土壤微生物碳元素循环能力[45]。总体而言,长期植物修复过程中,有效提高了沙地土壤中碳和水通量,为土壤微生物生命活动提供了基础养分并显著提高了微生物对土壤中碳的固定与分解能力。
土壤中的养分循环和生态系统服务过程很大程度上是由微生物群落驱动的,氮循环是生态系统服务的基础[46]。氮是高寒草原生物量的主要限制因素,定殖在当地的先锋植物氮素来源主要依靠大气氮沉降、动物尿源氮和微生物氮循环等途径。受气候波动因素,大气氮沉降率低,高寒沙地生态系统中由大气氮沉降来源的氮无法满足当地植被生长需求[47]。Reay等发现动物尿源氮被微生物同化到有机氮池中是氮保留的重要过程,并在之后微生物有机氮代谢过程中为植物提供可利用性氮源[48]。牲畜粪便排泄被认为是对放牧草地的一种天然施肥措施,对于养分匮乏的脆弱高寒沙地生态系统而言, 牲畜粪便养分返还是主要的氮素来源方式[49]。在围栏内进行适度控制放牧过程中,牲畜的尿液和粪便排泄物以有机氮形式进入土壤,土壤氨氧化细菌通过amoCAB基因编码氨单加氧酶(ammonia monooxygenase, AMO)将氨氧化为羟胺,羟胺进一步被羟胺氧化还原酶氧化为亚硝酸盐[50];例如,亚硝化螺菌属(Nitrosospira sp.)和维也纳亚硝化球形菌(Nitrososphaera viennensis)可以降解有机氮化合物[51]。硝化细菌通过nxrAB基因编码亚硝酸盐氧化还原酶将亚硝酸盐氧化为硝酸盐,为植物生长提供可利用性氮。近年来发现,硝化螺菌属中某些种类细菌可以进行完全氨氧化,相比之前被认为的氨氧化分两步完成,能量效率大大提高,为他们适应养分贫瘠的环境提供了能量基础[52-53];SS组和AS组中amoCAB基因簇和nxrAB基因簇的富集特征也能说明完全氨氧化存在的可能性[54]。慢生根瘤菌不仅能在豆科植物根部形成根瘤进行共生固氮,还可以通过游离固氮的方式促进非豆科植物生长,研究发现甘蔗根部的某些慢生根瘤菌具有游离固氮活性,在游离固氮期间与共生条件相比,非共生条件下nifH基因的转录水平升高[55]。如图 5B所示,慢生根瘤菌的相对丰度呈上升趋势,说明在人工草方格修复期间,慢生根瘤菌可能通过游离固氮的方式为先锋植物生长提供可利用性氮。沙地生态系统中完全氨氧化基因的存在,提示在生态治理区适度控制放牧可以通过牲畜的尿源氮和粪便排泄物进行氮素补充以外,还可以促进氮素向裸沙地的空间迁移;慢生根瘤菌属绝对丰度的显著上升,说明土壤微生物可能通过游离固氮和氨氧化等过程为植物提供可利用性氮。
磷(P)是地球上所有生命必需的大量营养元素,已被证明是植物生长和产量的重要限制性营养元素[56]。微生物在地球的磷循环中起着中心作用,既是磷源又是磷汇。Zhang等通过向辣椒盆栽中接种溶磷细菌(phosphorus-solubilizing bacteria, PSB)发现辣椒对磷的吸收量显著提高[57];这可能是溶磷细菌通过分泌有机酸将羟基和羧基与磷酸盐结合的阳离子螯合或通过释放H+,从而降低周围环境的pH,然后将固定的磷转化为植物可利用的形式;除此之外,PSB分泌的其他有机酸,如柠檬酸、琥珀酸、乙酸、草酸、丙酸、乳酸、富马酸、苹果酸、酒石酸、丙二酸、丙酮酸和草酸,也有助于磷酸盐的增溶[56]。
综上所述,人工建立草方格后,在长期植被修复过程中,植物有机残体的输入为土壤微生物的生长和繁殖提供了养分基础,为参与氮和磷元素循环的微生物提供了碳源;氮循环微生物通过分解植物残体有机氮、动物粪便尿源氮以及游离固氮等方式为植物提供可利用性氮;溶磷细菌通过分解植物有机残体和分泌有机酸等方式将土壤中固定的磷转化为植物生长可利用性磷,与植物生长建立互相反馈的关系。
4 结论通过对不同修复程度的沙化草地土壤的养分特征与微生物宏基因组的联合分析,评估了人工建立草方格措施对高寒沙地土壤恢复的影响。通过相对丰度的比较分析发现,放线菌门的相对丰度与营养条件呈负相关关系;在若尔盖高寒沙地人工建立草方格后,经过长期的植被修复,植被以其残体的形式向沙地输入有机养分,为土壤微生物的生命活动提供了有机营养,从而显著提高了土壤微生物的丰富度,增强了土壤微生物元素循环能力;适度放牧过程中动物粪便尿源氮和土壤中慢生根瘤菌属的游离固氮作用可以为沙地生态系统提供氮素来源;高寒沙地生态系统中完全氨氧化基因的存在,有助于将动物尿源氮和植物有机氮转化为植物可利用性氮,促进植物生长繁殖,提供了植物生长的边际外延可能性。以上发现不仅为生态恢复和土壤管理提供了有价值的见解,也为更好地理解植被修复对土壤微生物与养分循环之间相互关系的影响提供了重要的启示。
| [1] | ZHANG AL, LI XY, ZENG FJ, JIANG Y, WANG RZ. Variation characteristics of different plant functional groups in alpine desert steppe of the Altun Mountains, northern Qinghai-Xizang Plateau[J]. Frontiers in Plant Science, 2022, 13: 961692 DOI:10.3389/fpls.2022.961692. |
| [2] | SCHIEDUNG M, TREGURTHA CS, BEARE MH, THOMAS SM, DON A. Deep soil flipping increases carbon stocks of New Zealand grasslands[J]. Global Change Biology, 2019, 25(7): 2296-2309 DOI:10.1111/gcb.14588. |
| [3] | YAN WC, WANG YY, CHAUDHARY P, JU PJ, ZHU Q, KANG XM, CHEN H, HE YX. Effects of climate change and human activities on net primary production of wetlands on the Zoige Plateau from 1990 to 2015[J]. Global Ecology and Conservation, 2022, 35: e02052 DOI:10.1016/j.gecco.2022.e02052. |
| [4] | ZHAO Z, WANG GF, CHEN JC, WANG JY, ZHANG Y. Assessment of climate change adaptation measures on the income of herders in a pastoral region[J]. Journal of Cleaner Production, 2019, 208: 728-735 DOI:10.1016/j.jclepro.2018.10.088. |
| [5] |
文长凤, 戴睿, 尚闽, 钱晓丽, 龙瑞凤, 杨正兴. 草原生态补奖政策对若尔盖县畜牧业发展影响分析[J]. 四川环境, 2022, 41(2): 131-138.
WEN CF, DAI R, SHANG M, QIAN XL, LONG RF, YANG ZX. The analysis on the influence of grassland ecological subsidy policy on animal husbandry development in Ruoergai County[J]. Sichuan Environment, 2022, 41(2): 131-138 (in Chinese). |
| [6] | HU JJ, ZHOU QP, CAO QH, HU J. Effects of ecological restoration measures on vegetation and soil properties in semi-humid sandy land on the southeast Qinghai-Tibetan Plateau, China[J]. Global Ecology and Conservation, 2022, 33: e02000 DOI:10.1016/j.gecco.2022.e02000. |
| [7] | LIU J, WANG J, MORREALE SJ, SCHNEIDER RL, LI ZG, WU GL. Contributions of plant litter to soil microbial activity improvement and soil nutrient enhancement along with herb and shrub colonization expansions in an arid sandy land[J]. Catena, 2023, 227: 107098 DOI:10.1016/j.catena.2023.107098. |
| [8] | LIU C, ZHUANG JY, WANG J, FAN GH, FENG M, ZHANG ST. Soil bacterial communities of three types of plants from ecological restoration areas and plant-growth promotional benefits of Microbacterium invictum (strain X-18)[J]. Frontiers in Microbiology, 2022, 13: 926037 DOI:10.3389/fmicb.2022.926037. |
| [9] |
张小芳, 张春平, 杨增增, 李彩弟, 杨晓霞, 刘文亭, 俞旸, 董全民. 单播措施下三江源区高寒退化草地恢复效果评估[J]. 草地学报, 2022, 30(10): 2834-2844.
ZHANG XF, ZHANG CP, YANG ZZ, LI CD, YANG XX, LIU WT, YU Y, DONG QM. Evaluation of restoration effect in alpine degraded meadow under single sowing in the three rivers source regions[J]. Acta Agrestia Sinica, 2022, 30(10): 2834-2844 (in Chinese). |
| [10] |
肖颖, 吉使阿微, 赵文学, 田莉华. 青藏高原东缘不同人工草地土壤养分、酶活性及微生物生物量特征[J]. 中国草地学报, 2022, 44(9): 90-99.
XIAO Y, JISHI AW, ZHAO WX, TIAN LH. Soil nutrients, enzyme activities and microbial biomass characteristics of different artificial grasslands in the eastern margin of the Qinghai-Tibetan Plateau[J]. Chinese Journal of Grassland, 2022, 44(9): 90-99 (in Chinese). |
| [11] | ZHENG Y, LV GB, CHEN K, YU QB, NIU B, JIANG J, LIU GF. Impact of PaGLK transgenic poplar on microbial community and soil enzyme activity in rhizosphere soil[J]. Frontiers in Bioengineering and Biotechnology, 2022, 10: 965209 DOI:10.3389/fbioe.2022.965209. |
| [12] |
张一然, 文小航, 罗斯琼, 李茂善. 近20年若尔盖湿地植被覆盖变化与气候因子关系研究[J]. 高原气象, 2022, 41(2): 317-327.
ZHANG YR, WEN XH, LUO SQ, LI MS. Study on the relationship between vegetation cover change and climate factors in Zoige wetland in recent 20 years[J]. Plateau Meteorology, 2022, 41(2): 317-327 (in Chinese). |
| [13] | DUAN HC, XUE X, WANG T, KANG WP, LIAO J, LIU SL. Spatial and temporal differences in alpine meadow, alpine steppe and all vegetation of the Qinghai-Tibetan Plateau and their responses to climate change[J]. Remote Sensing, 2021, 13(4): 669 DOI:10.3390/rs13040669. |
| [14] |
费怡, 王继燕, 王泽根. 若尔盖高原土地沙化及其成因定量分析[J]. 干旱区资源与环境, 2019, 33(8): 146-152.
FEI Y, WANG JY, WANG ZG. Dynamic changes of land desertification in Zoige Plateau[J]. Journal of Arid Land Resources and Environment, 2019, 33(8): 146-152 (in Chinese). |
| [15] | BREMNER JM, SHAW K. Denitrification in soil. I. Methods of investigation[J]. The Journal of Agricultural Science, 1958, 51(1): 22-39 DOI:10.1017/S0021859600032767. |
| [16] | MURPHY J, RILEY JP. A modified single solution method for the determination of phosphate in natural waters[J]. Analytica Chimica Acta, 1962, 27: 31-36 DOI:10.1016/S0003-2670(00)88444-5. |
| [17] | CHEN YF, YE JR, KONG QQ. Potassium-solubilizing activity of Bacillus aryabhattai SK1-7 and its growth-promoting effect on Populus alba L.[J]. Forests, 2020, 11(12): 1348 DOI:10.3390/f11121348. |
| [18] | WALKLEY A, BLACK IA. An examination of the degtjareff method for determining soil organic matter, and a proposed modification of the chromic acid titration method[J]. Soil Science, 1934, 37(1): 29-38 DOI:10.1097/00010694-193401000-00003. |
| [19] | CHEN SF, ZHOU YQ, CHEN YR, GU J. Fastp: an ultra-fast all-in-one FASTQ preprocessor[J]. Bioinformatics, 2018, 34(17): i884-i890 DOI:10.1093/bioinformatics/bty560. |
| [20] | LI DH, LIU CM, LUO RB, SADAKANE K, LAM TW. MEGAHIT: an ultra-fast single-node solution for large and complex metagenomics assembly via succinct de Bruijn graph[J]. Bioinformatics, 2015, 31(10): 1674-1676 DOI:10.1093/bioinformatics/btv033. |
| [21] | HYATT D, CHEN GL, LOCASCIO PF, LAND ML, LARIMER FW, HAUSER LJ. Prodigal: prokaryotic gene recognition and translation initiation site identification[J]. BMC Bioinformatics, 2010, 11: 119 DOI:10.1186/1471-2105-11-119. |
| [22] | LANGMEAD B, SALZBERG SL. Fast gapped-read alignment with bowtie 2[J]. Nature Methods, 2012, 9(4): 357-359 DOI:10.1038/nmeth.1923. |
| [23] | BUCHFINK B, XIE C, HUSON DH. Fast and sensitive protein alignment using DIAMOND[J]. Nature Methods, 2015, 12(1): 59-60 DOI:10.1038/nmeth.3176. |
| [24] | QU CM, FU FY, LU K, ZHANG K, WANG R, XU XF, WANG M, LU JX, WAN HF, TANG ZL, LI JN. Differential accumulation of phenolic compounds and expression of related genes in black- and yellow-seeded Brassica napus[J]. Journal of Experimental Botany, 2013, 64(10): 2885-2898 DOI:10.1093/jxb/ert148. |
| [25] | WU SY, LIU LB, LI DL, ZHANG WT, LIU KD, SHEN JS, ZHANG LB. Global desert expansion during the 21st century: patterns, predictors and signals[J]. Land Degradation & Development, 2023, 34(2): 377-388. |
| [26] | JIAN TK, XIA Y, HE RP, ZHANG J. The influence of planting Carex praeclara and Leymus secalinus on soil properties and microbial community in a Zoige desertified alpine grassland[J]. Global Ecology and Conservation, 2022, 34: e02002 DOI:10.1016/j.gecco.2022.e02002. |
| [27] | LIU R, LI Y, WANG YG, MA J, CIERAAD E. Variation of water use efficiency across seasons and years: different role of herbaceous plants in desert ecosystem[J]. Science of the Total Environment, 2019, 647: 827-835 DOI:10.1016/j.scitotenv.2018.08.035. |
| [28] | HAPPONEN K, VIRKKALA AM, KEMPPINEN J, NIITTYNEN P, LUOTO M. Relationships between above-ground plant traits and carbon cycling in tundra plant communities[J]. Journal of Ecology, 2022, 110(3): 700-716 DOI:10.1111/1365-2745.13832. |
| [29] | CHAI YN, SCHACHTMAN DP. Root exudates impact plant performance under abiotic stress[J]. Trends in Plant Science, 2022, 27(1): 80-91 DOI:10.1016/j.tplants.2021.08.003. |
| [30] | ALI A, IMRAN GHANI M, LI YH, DING HY, MENG HW, CHENG ZH. Hiseq base molecular characterization of soil microbial community, diversity structure, and predictive functional profiling in continuous cucumber planted soil affected by diverse cropping systems in an intensive greenhouse region of northern China[J]. International Journal of Molecular Sciences, 2019, 20(11): 2619 DOI:10.3390/ijms20112619. |
| [31] | MARASCO R, MOSQUEIRA MJ, FUSI M, RAMOND JB, MERLINO G, BOOTH JM, MAGGS-KÖLLING G, COWAN DA, DAFFONCHIO D. Rhizosheath microbial community assembly of sympatric desert speargrasses is independent of the plant host[J]. Microbiome, 2018, 6(1): 1-18 DOI:10.1186/s40168-017-0383-2. |
| [32] | SCHULZ-BOHM K, GERARDS S, HUNDSCHEID M, MELENHORST J, de BOER W, GARBEVA P. Calling from distance: attraction of soil bacteria by plant root volatiles[J]. The ISME Journal, 2018, 12(5): 1252-1262 DOI:10.1038/s41396-017-0035-3. |
| [33] | ZHANG KL, MALTAIS-LANDRY G, JAMES M, MENDEZ V, WRIGHT D, GEORGE S, LIAO HL. Absolute microbiome profiling highlights the links among microbial stability, soil health, and crop productivity under long-term sod-based rotation[J]. Biology and Fertility of Soils, 2022, 58(8): 883-901 DOI:10.1007/s00374-022-01675-4. |
| [34] | XIA Y, HE RP, XU WR, ZHANG J. The Zoige pioneer plant Leymus secalinus has different endophytic bacterial community structures to adapt to environmental conditions[J]. PeerJ, 2023, 11: e15363 DOI:10.7717/peerj.15363. |
| [35] | YANG Y, LIU H, YANG X, YAO HJ, DENG XQ, WANG YQ, AN SS, KUZYAKOV YK, CHANG SX. Plant and soil elemental C: N: P ratios are linked to soil microbial diversity during grassland restoration on the Loess Plateau, China[J]. Science of the Total Environment, 2022, 806: 150557 DOI:10.1016/j.scitotenv.2021.150557. |
| [36] | WANG SK, ZUO XA, ZHAO XY, LI YQ, ZHOU X, LV P, LUO YQ, YUN JY. Responses of soil fungal community to the sandy grassland restoration in Horqin Sandy Land, northern China[J]. Environmental Monitoring and Assessment, 2016, 188(1): 21 DOI:10.1007/s10661-015-5031-3. |
| [37] | CHEN HH, MA KY, LU CY, FU Q, QIU YB, ZHAO JY, HUANG Y, YANG YC, SCHADT CW, CHEN H. Functional redundancy in soil microbial community based on metagenomics across the globe[J]. Frontiers in Microbiology, 2022, 13: 878978 DOI:10.3389/fmicb.2022.878978. |
| [38] | GAO L, WANG R, SHEN GM, ZHANG JX, MENG GX, ZHANG JG. Effects of biochar on nutrients and the microbial community structure of tobacco-planting soils[J]. Journal of Soil Science and Plant Nutrition, 2017, 17(4): 884-896 DOI:10.4067/S0718-95162017000400004. |
| [39] | de ARAUJO PEREIRA AP, MENDES LW, OLIVEIRA FAS, ANTUNES JEL, MACIEL MELO VM, ARAUJO ASF. Land degradation affects the microbial communities in the Brazilian Caatinga biome[J]. Catena, 2022, 211: 105961 DOI:10.1016/j.catena.2021.105961. |
| [40] | YOUNGQUIST CP, LIU JX, ORFE LH, JONES SS, CALL DR. Ciprofloxacin residues in municipal biosolid compost do not selectively enrich populations of resistant bacteria[J]. Applied and Environmental Microbiology, 2014, 80(24): 7521-7526 DOI:10.1128/AEM.02899-14. |
| [41] | WANG YJ, LIU L, ZHANG JT, LI DM, YU J, GAO H, LI HK, ZHAO ZY. Soil phytoremediation reveals alteration in soil microbial metabolic activities along time gradient of cover crop mulching[J]. Environmental Research, 2022, 209: 112884 DOI:10.1016/j.envres.2022.112884. |
| [42] | JING YM, LAN N, LEI L, AI YW, WANG CT, LI XZ. Total phosphorus mediates soil nitrogen cycling in alpine meadows[J]. Journal of Soils and Sediments, 2023, 23(9): 3445-3457 DOI:10.1007/s11368-023-03561-4. |
| [43] | ZHANG YX, TANG ZX, YOU YM, GUO XW, WU CJ, LIU SR, SUN OJX. Differential effects of forest-floor litter and roots on soil organic carbon formation in a temperate oak forest[J]. Soil Biology and Biochemistry, 2023, 180: 109017 DOI:10.1016/j.soilbio.2023.109017. |
| [44] | ZHENG W, GONG QL, ZHAO ZY, LIU J, ZHAI BN, WANG ZH, LI ZY. Changes in the soil bacterial community structure and enzyme activities after intercrop mulch with cover crop for eight years in an orchard[J]. European Journal of Soil Biology, 2018, 86: 34-41 DOI:10.1016/j.ejsobi.2018.01.009. |
| [45] | ZHONG YQW, YAN WM, WANG RW, WANG W, SHANGGUAN ZP. Decreased occurrence of carbon cycle functions in microbial communities along with long-term secondary succession[J]. Soil Biology and Biochemistry, 2018, 123: 207-217 DOI:10.1016/j.soilbio.2018.05.017. |
| [46] | ORTIZ M, BOSCH J, COCLET C, JOHNSON J, LEBRE P, SALAWU-ROTIMI A, VIKRAM S, MAKHALANYANE T, COWAN D. Microbial nitrogen cycling in Antarctic soils[J]. Microorganisms, 2020, 8(9): 1442 DOI:10.3390/microorganisms8091442. |
| [47] | BÁEZ S, FARGIONE J, MOORE DI, COLLINS SL, GOSZ JR. Atmospheric nitrogen deposition in the northern Chihuahuan Desert: temporal trends and potential consequences[J]. Journal of Arid Environments, 2007, 68(4): 640-651 DOI:10.1016/j.jaridenv.2006.06.011. |
| [48] | REAY MK, MARSDEN KA, POWELL S, RIVERA LM, CHADWICK DR, JONES DL, EVERSHED RP. The soil microbial community and plant biomass differentially contribute to the retention and recycling of urinary-N in grasslands[J]. Soil Biology and Biochemistry, 2023, 180: 109011 DOI:10.1016/j.soilbio.2023.109011. |
| [49] |
杜子银, 蔡延江, 王小丹, 张斌, 杜忠. 放牧牲畜粪便降解及其对草地土壤养分动态的影响研究进展[J]. 生态学报, 2019, 39(13): 4627-4637.
DU ZY, CAI YJ, WANG XD, ZHANG B, DU Z. Research progress on grazing livestock dung decomposition and its influence on the dynamics of grassland soil nutrients[J]. Acta Ecologica Sinica, 2019, 39(13): 4627-4637 (in Chinese). |
| [50] | KUYPERS MMM, MARCHANT HK, KARTAL B. The microbial nitrogen-cycling network[J]. Nature Reviews Microbiology, 2018, 16(5): 263-276 DOI:10.1038/nrmicro.2018.9. |
| [51] | TOURNA M, STIEGLMEIER M, SPANG A, KÖNNEKE M, SCHINTLMEISTER A, URICH T, ENGEL M, SCHLOTER M, WAGNER M, RICHTER A, SCHLEPER C. Nitrososphaera viennensis, an ammonia oxidizing archaeon from soil[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(20): 8420-8425. |
| [52] | DIMITRI KITS K, SEDLACEK CJ, LEBEDEVA EV, HAN P, BULAEV A, PJEVAC P, DAEBELER A, ROMANO S, ALBERTSEN M, STEIN LY, DAIMS H, WAGNER M. Kinetic analysis of a complete nitrifier reveals an oligotrophic lifestyle[J]. Nature, 2017, 549(7671): 269-272 DOI:10.1038/nature23679. |
| [53] | DAIMS H, LEBEDEVA EV, PJEVAC P, HAN P, HERBOLD C, ALBERTSEN M, JEHMLICH N, PALATINSZKY M, VIERHEILIG J, BULAEV A, KIRKEGAARD RH, von BERGEN M, RATTEI T, BENDINGER B, NIELSEN PH, WAGNER M. Complete nitrification by Nitrospira bacteria[J]. Nature, 2015, 528(7583): 504-509 DOI:10.1038/nature16461. |
| [54] | PALOMO A, JANE FOWLER S, GÜLAY A, RASMUSSEN S, SICHERITZ-PONTEN T, SMETS BF. Metagenomic analysis of rapid gravity sand filter microbial communities suggests novel physiology of Nitrospira spp.[J]. The ISME Journal, 2016, 10(11): 2569-2581 DOI:10.1038/ismej.2016.63. |
| [55] | de MATOS GF, ROUWS LFM, SIMÕES-ARAÚJO JL, BALDANI JI. Evolution and function of nitrogen fixation gene clusters in sugarcane associated Bradyrhizobium strains[J]. Environmental Microbiology, 2021, 23(10): 6148-6162 DOI:10.1111/1462-2920.15533. |
| [56] | LI HP, HAN QQ, LIU QM, GAN YN, RENSING C, RIVERA WL, ZHAO Q, ZHANG JL. Roles of phosphate-solubilizing bacteria in mediating soil legacy phosphorus availability[J]. Microbiological Research, 2023, 272: 127375 DOI:10.1016/j.micres.2023.127375. |
| [57] | ZHANG J, GUO TT, TAO Z, WANG PC, TIAN HM. Transcriptome profiling of genes involved in nutrient uptake regulated by phosphate-solubilizing bacteria in pepper (Capsicum annuum L.)[J]. Plant Physiology and Biochemistry, 2020, 156: 611-626 DOI:10.1016/j.plaphy.2020.10.003. |
 2024, Vol. 64
2024, Vol. 64




