中国科学院微生物研究所,中国微生物学会
文章信息
- 吴汉卿, 阮楚晋, 韩苗, 王钢. 2024
- WU Hanqing, RUAN Chujin, HAN Miao, WANG Gang.
- 土壤病毒之奥秘:研究进展、挑战及未来展望
- Mystery of soil viruses: advances, challenges, and perspectives
- 微生物学报, 64(6): 1824-1847
- Acta Microbiologica Sinica, 64(6): 1824-1847
-
文章历史
- 收稿日期:2023-08-29
- 网络出版日期:2023-11-17
2. 中国科学院亚热带农业生态研究所, 湖南 长沙 410125;
3. 中国农业大学国家黑土地现代农业研究院, 北京 100193
2. Institute of Subtropical Agriculture, Chinese Academy of Sciences, Changsha 410125, Hunan, China;
3. National Black Soil and Agriculture Research, China Agricultural University, Beijing 100193, China
土壤微生物(尤其是细菌)参与并驱动了不同环境条件下的元素生物地球化学循环过程[1-2],而细菌周围通常伴有高丰度的病毒存在[3]。据统计,全球病毒数量约为4.8×1031个[4],而土壤病毒丰度占全球病毒数量的10%左右,每克土壤中病毒丰度最高可达1010病毒粒子(virus-like particles, VLPs)[5]。据大量研究报道,病毒作为地球上最神秘且种类和数量繁多的生物实体,在调控微生物多样性和群落结构、元素生物地球化学循环和生物进化中起到关键作用[6-13]。一方面,土壤病毒群落能够通过感染和裂解宿主细胞对特定宿主细菌物种的丰度变化作出迅速响应[14],在此过程中释放出养分物质并影响微生物残体的生成,从而影响养分循环[4, 15-17]。另一方面,病毒能够介导宿主细胞间的水平基因转移来驱动宿主微生物进化,并通过病毒与宿主间从个体到群落水平上错综复杂的互作关系,影响微生物多样性和群落结构[15, 18]。因此,土壤病毒是土壤生态系统的重要组成部分,在维持土壤生态系统稳定和促进生物地球化学循环过程中发挥着至关重要的作用[19-23]。
土壤具有丰富的空间结构(比表面积为1.1×106−8.0×106 cm2/g),其与土壤水肥气热等要素共同塑造了土壤特有的微观异质性[5],为土壤病毒和其他微生物的活动创造了多重生态位(图 1)[24-25],但与此同时,土壤孔隙结构的异质性往往会导致土壤中微生物运动受到限制[26-27]。如土壤孔隙特征通过影响养分扩散、运移,导致不同微生境土壤中养分含量差异并影响微生物对养分的获取策略,进而影响土壤病毒与宿主细菌间的互作关系[25]。此外,病毒自身不具备自主运动能力,其主要依靠气流、水流以及其他生物的携带在环境中被动迁移,并主要通过布朗运动在土壤团聚体表面的水膜中进行扩散。在水生环境中,病毒易于运移,且与易感菌株的相遇机会较大;而在土壤环境中,由于空间异质性以及土壤胶体等物质的吸附作用会限制病毒运移,进而限制了土壤病毒-宿主微生物间的互作过程[26]。然而,自然土壤常处于非饱和水分状态,土壤部分孔隙间由于缺水,连通性受限,在土壤团粒结构中产生空间隔离[28],这也会导致土壤微生物(包括病毒)产生不同的进化轨迹,甚至使微生物种群出现平行进化,进而增加土壤微生物群落中的局部多样性并形成独特的微生物(病毒、细菌及真菌等)群落结构[27]。
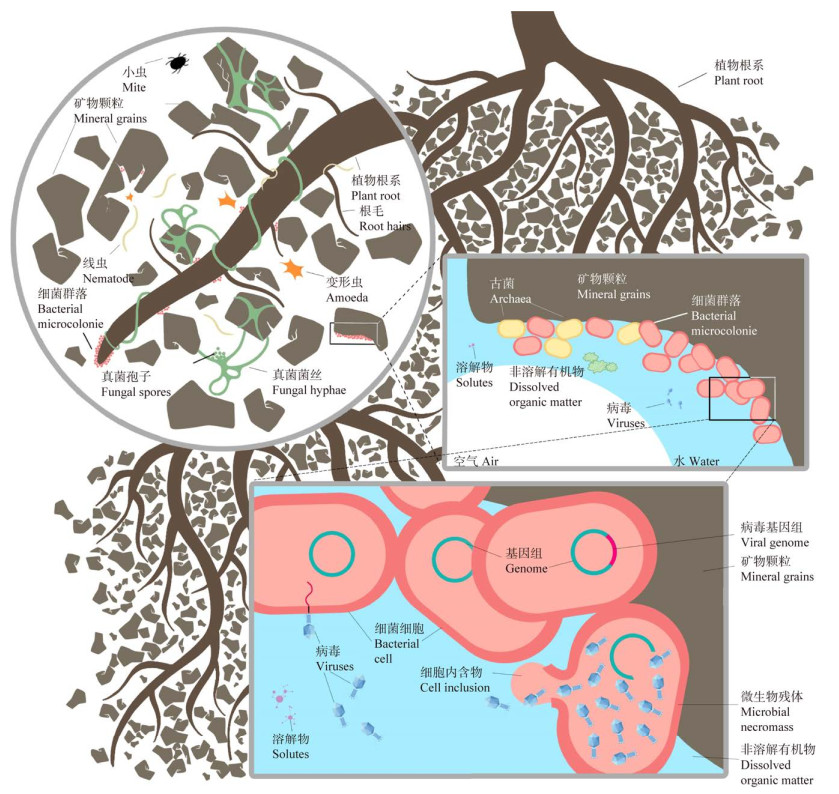
|
| 图 1 土壤团聚体微生物群落分布 Figure 1 Microbial community distribution in soil aggregates. |
近年来,土壤病毒愈发受到科研学者们的关注,了解病毒在土壤生态系统中的功能及作用机制具有重要意义。然而,由于土壤自身的复杂性及其动态变化特征,病毒在土壤生态系统中发挥的功能及其作用机理,一直是该研究领域的薄弱环节。目前,人们对土壤病毒类型、丰度、影响因素、作用机制及生态功能仍知之甚少。基于此,本文借助文献计量分析方法,系统梳理国内外关于土壤病毒相关研究进展,阐述该研究领域的现有方向及国内外研究热点,并根据当前需求,提出该研究领域的关键知识空白及未来研究展望,以期为该研究领域的探索和发展提供一定参考。
1 土壤病毒研究发展态势本研究基于Web of Science (WoS)核心合集数据库,通过主题检索,统计自1900年至今土壤病毒(包括噬菌体)研究领域的年发文量,并归纳总结出其发展态势。1936−2021年土壤病毒研究领域发展可划分为2个阶段:平稳发展阶段(1936−1990年)和迅速增长阶段(1991年至今)。1936−1990年,每年发文量不超过10篇,该时期土壤病毒研究发展缓慢;1991年至今,发文量极速增长,尤其是近几年发展更为迅速(图 2)。这说明土壤病毒研究领域当前正处于高速发展阶段,国际上对土壤病毒相关研究的关注程度越来越高,同时也证实该研究领域目前为国际土壤学研究热点,推测可能是近年来分子生物学技术的突破、生态学理论的广泛应用及新冠病毒感染疫情极大地推动了土壤病毒研究领域的发展。
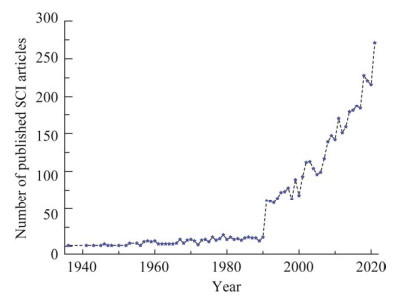
|
| 图 2 1936−2021年土壤病毒研究领域SCI发文量变化 Figure 2 Changes in the number of published SCI articles in the research field of soil viruses from 1936 to 2021. |
利用检索自WoS核心合集数据库1936−2022年发表的土壤病毒相关文献信息,进行关键词共现网络分析[29],发现土壤病毒研究领域的发展主要是围绕病毒多样性及生态学效应(主要关键词有diversity、population dynamics、communities、abundance、ecology等)、迁移传播(主要关键词有movement、transport、adsorption、porous media等)及侵染机制、抗性基因、生物进化及调控(主要关键词有infection、resistance gene、evolution、biological control等)等3个大方向(图 3)。
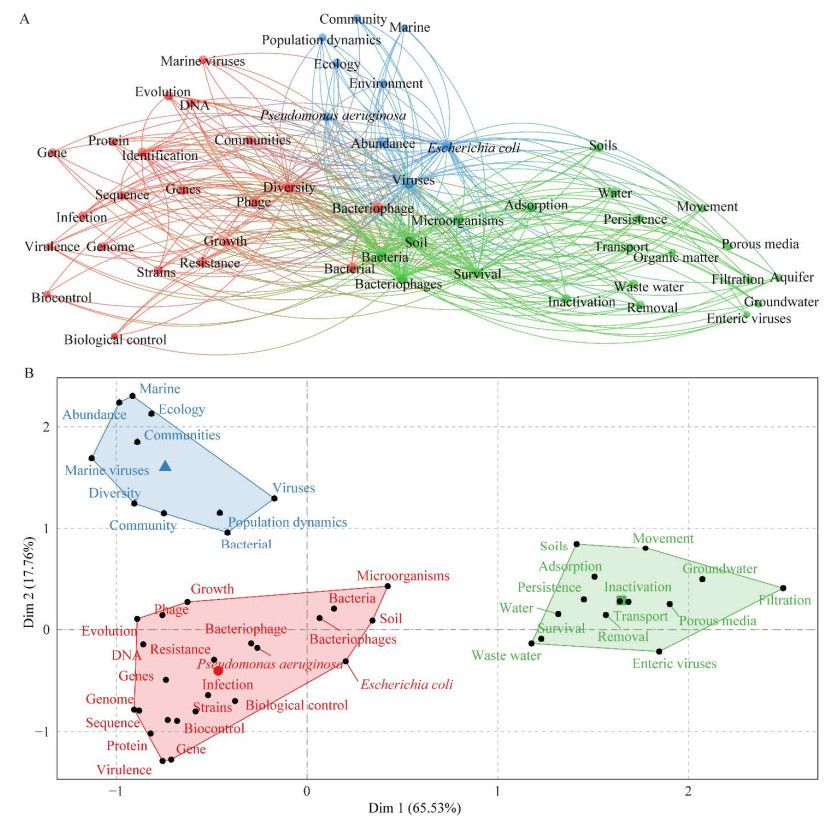
|
| 图 3 1936−2022年土壤病毒研究高频关键词共现网络图谱(A)和高频关键词聚类图谱(B) Figure 3 The co-occurrence network (A) and cluster map (B) of high-frequency keywords about soil viruses from 1936 to 2022. |
2 土壤病毒研究热点 2.1 土壤病毒类型
病毒作为宿主细胞外独立粒子,基因组物质都包裹在蛋白质结构中。土壤中大多数典型噬菌体衣壳上附着有尾丝蛋白,参与宿主识别和感染,少数有脂质包膜。Weinbauer[30]观察水生环境病毒直径为30−80 nm,土壤病毒大小也在这个范围内,典型噬菌体的衣壳最常见直径约为60 nm[5]。因此,噬菌体的等效直径一般要比细菌细胞(1−5 μm)的尺寸小很多。从噬菌体的核酸类型来看,噬菌体可以携带双链DNA (double-stranded DNA, dsDNA)、单链DNA (single-stranded DNA, ssDNA)、双链RNA (double-strandedRNA, dsRNA)、单链RNA (single-stranded RNA, ssRNA)。
常见的噬菌体分为短尾噬菌体科(Podoviridae) (尾部短,不可收缩),肌尾噬菌体科(Myoviridae) (尾部长,可收缩),长尾噬菌体科(Siphoviridae) (尾部长,不可收缩),以及一些球状、线状的病毒颗粒[5]。Williamson等[3]比较了多种不同类型土壤的病毒颗粒形态,发现多数土壤以有尾噬菌体为主(80%),仅有1种农业土壤中56%为球形病毒颗粒。Guttman等[31]估算了有尾噬菌体(Caudovirales)的质量,发现其蛋白质和核酸质量相等,而丝状病毒通常只有6%−14%的核酸。
此外有研究发现,病毒粒子大小与基因组大小存在一定相关性[32],如大肠杆菌病毒粒子MS2噬菌体直径为26 nm,单链核糖核酸基因组只有3 569个核苷酸,仅携带4个基因。而巨大芽孢杆菌(Bacillus megaterium)噬菌体的衣壳直径为160 nm,能携带细菌497 kb双链脱氧核糖核酸基因组,比一些细菌基因组还要大[30]。在土壤中,还存在着一些巨大病毒(giant viruses),如核质巨DNA病毒(nucleocytoplasmic large DNA viruses, NCLDVs),其具有至少300 kb的基因组,主要感染真核生物(尤其是原生生物和藻类)[33-35]。Schulz等[36]通过迷你宏基因组学方法(mini-metagenomics)在森林土壤中发现了16个新的巨大病毒基因组,并通过系统发育分析将它们归属于klosneuviruses、Cafeteria roenbergensis virus和tupanviruses。常见的一些土壤病毒及宿主、基因型信息如表 1所示。
| Hosts | Genome type | Typical viruses | References |
| Bacteria | dsDNA/ssDNA dsRNA/ssRNA(+) (linear and circular) |
Caudovirales (Myoviridae, Podoviridae and Siphoviridae), Inoviridae, Plasmaviridae, and Leviviridae | [38-39] |
| Archaea | dsDNA/ssDNA | Ampullaviridae, Bicaudaviridae, Spiraviridae, Fuselloviridae | [40] |
| Fungi | dsRNA (linear) ssRNA(+)/ssRNA(−) ssDNA (circular) |
Totiviridae, Chrysoviridae, Partitiviridae, Megabirnaviridae, Quadriviridae, Reoviridae, Alphaflexiviridae, Endornaviridae, Barnaviridae, Gammaflexiviridae, Hypoviridae, Narnaviridae, Mycomononegaviridae | [41] |
| Arthropods | ssRNA(+)/ssRNA(−) | Flavivirus, arboviruses, togaviruses, bunyaviruses, mesoniviruses, reoviruses, rhabdoviruses, negiviruses, birnviruses | [42] |
| Nematodes | ssRNA | Orsay virus infecting Caenorhabditis elegans | [43] |
| Protozoa | dsRNA | Trichomonas vaginalis virus, Giardia lamblia viruses | [44] |
| Plants | ssDNA/dsRNA ssRNA(+)/ssRNA(−) ssRNA(RT)/dsDNA(RT) |
Tobacco mosaic virus, tomato spotted wilt virus, tomato yellow leaf curl begomovirus | [45] |
| +: Plus-strand; –: Minus-strand; RT: Reverse transcription. | |||
2.2 土壤病毒研究常用方法
大多数土壤病毒研究的第一步工作是需要将吸附在土壤颗粒上的病毒粒子进行洗脱浸提,而10%的柠檬酸钾溶液、10%的牛肉膏、250 mmol/L的甘氨酸溶液和10 mmol/L的焦磷酸钠溶液为最常用的4种土壤病毒洗脱浸提液,其中对于农田土壤病毒洗脱提取,10%的柠檬酸钾溶液效果相对最佳[46]。Yu等[47]通过比较了3种物理分散方法(振荡、涡旋和超声处理)和10种土壤病毒洗脱浸提液,最终确定了从(亚)热带红壤中提取病毒的优化方法。此外,为得到高浓度的土壤病毒提取液,还需要对其进行土壤病毒纯化及富集[48]。目前最常用的土壤病毒纯化及富集方法是聚乙二醇(polyethylene glycol, PEG)沉淀法[49]、CsCl平衡梯度等密度离心法[50]及切向流过滤法(tangential-flow filtration, TFF)[51]。其中,对于土壤病毒粒子的分离、纯化及富集主要采用切向流过滤法,其原理是利用蠕动泵将土壤病毒洗脱液匀速流经特定孔径的过滤膜,从而实现土壤病毒提取液的纯化及浓缩。
在获取纯化及富集后的土壤病毒粒子,可依靠扫描电子显微镜(scanning electron microscope, SEM)、透射电子显微镜(transmission electron microscope, TEM)和流式细胞仪(flow cytometer, FC)等工具,对其进行计数及形态学、生物学特性分析[52-53],此外,当前宏病毒组技术也被广泛应用于土壤病毒研究,属于研究热点之一。值得注意的是,由于病毒基因组不同于原核微生物基因组,没有类似于原核生物16S rRNA基因普遍保守的系统发育标记,因此难以利用分子生物技术对土壤病毒多样性和系统发育进行综合分析[54]。当前唯有在特定病毒类群中,才可利用某些高度保守的标记基因评估某个类群中的系统发育关系(如g23[55]),但携带类似保守基因的病毒只占自然环境中所有病毒种群的极小比例,不能够完整地反映环境中病毒多样性特征。随着高通量测序和宏基因组学技术的发展,人们无须通过宿主培养及单基因标记,仅通过对整个环境中的病毒基因进行比较分析就可以研究病毒多样性[56]。宏病毒组主要有两种分析方法,一是直接收集土壤中的病毒,提取DNA并测序分析。然而,实验收集的过程会造成一定程度的病毒损失,且由于土壤病毒基因组含量少,需要通过全基因组扩增病毒基因组以达到可测序浓度,会出现偏好性扩增[9],这些因素都影响着宏基因组序列信息对土壤病毒多样性的真实反映[57];二是从细菌宏基因组中挖掘病毒序列信息[57]。然而,由于细菌基因组的干扰以及土壤病毒基因库的不完整性,导致土壤病毒的大量序列无法有效注释。由于这些技术方面的限制,目前通过宏病毒组研究土壤病毒多样性的报道较少[58-64]。此外,也有研究利用病毒指纹图谱技术来探究土壤病毒多样性,如Srinivasiah等[65]利用RAPD-PCR (random PCR amplification of polymorphic DNA)技术发现南极不同地点土壤病毒群落结构存在显著差异,且病毒群落结构随环境变化而异。
2.3 土壤理化性质对病毒的影响土壤病毒自身不具备主动运动能力,因此土壤中的病毒主要以3种状态存在:(1) 绝大部分病毒由于静电等作用吸附及固定在土壤颗粒表面;(2) 极少部分病毒游离在土壤团聚体表面的水膜中;(3) 还有一部分溶原性病毒存在于宿主细胞中[7]。而土壤中影响病毒丰度的因素也较多,如土壤水分、温度、pH、有机质及黏土矿物等[66-67]。
土壤水分对病毒丰度的影响存在一个阈值,一旦土壤含水量低于这个阈值,土壤蒸发所导致的病毒灭活作用就会被放大。Yeager和O’Brien[68]研究发现,在砂壤土中,Ⅰ型脊髓灰质炎病毒质量含水量为2.9%−18%之间时以基本相同的速率灭活,但是当质量含水量在0.6%−1.2%,病毒失活率显著增加。此外还发现在较低的土壤含水量条件下,蒸发是导致干燥土壤中Ⅰ型脊髓灰质炎病毒灭活的主要因素。因此,该团队对Ⅰ型脊髓灰质炎病毒的RNA和衣壳进行了放射性标记,发现在湿润土壤中病毒的核糖核酸和衣壳都可以进行回收,但在干燥土壤中只有RNA能有效地进行回收,这说明病毒可能与土壤矿物颗粒产生的不可逆结合,从而导致病毒灭活[68]。Straub等[69]也比较了在不同土壤含水量条件下砂壤土和黏壤土中Ⅰ型脊髓灰质炎病毒和大肠杆菌噬菌体MS2、鼠伤寒沙门氏菌噬菌体PRD-1的失活率,发现3种病毒的活性随土壤水的蒸发而迅速降低,比恒定湿度下失活更快;无论何种土壤类型,在15 ℃下蒸发7 d、27 ℃下蒸发3 d或者40 ℃下蒸发2 d均能完全灭活这3种病毒。Williamson等[3]使用荧光显微镜测定了美国特拉华州6种土壤中的病毒丰度,发现土壤病毒粒子丰度与土壤含水量之间存在显著正相关关系。王燕等[70]研究发现,水分通过调控多孔表面水膜的厚度和连通性来改变细菌在微孔尺度的运动,进而影响微生物在粗糙界面的增殖和生物膜的形成过程,以及菌落的形态特征,同时通过影响噬菌体的扩散传播来调节细菌和噬菌体之间的互作机制,即相对低水膜网络促进了细菌和噬菌体之间的空间隔离,降低了噬菌体有效侵染细菌的概率,从而有利于细菌的增殖。另外,土壤水分往往会影响土壤中养分扩散过程,导致养分有效性产生差异,进而影响噬菌体的感染机制。如Stewart和Levin[71]发现当养分有效性存在显著差异时,溶原性噬菌体比裂解性噬菌体更易于生存,且在环境中数量更为丰富,其作为水平基因转移载体和细菌遗传变异的基因来源,对微生物生态学和进化过程有着重要影响。另一方面,土壤水分分布及动态变化特征能够影响土壤微生物种群丰度与群落结构[72-75],进而影响土壤病毒的时空分布动态[15, 76-77];然而,土壤微生物与病毒之间的互作模式及相关作用机制仍不清楚[78-80]。
温度也是决定噬菌体能否存活和生长的关键因素[81]。一般而言,低温会增加病毒的生存期和潜伏期,但同时也会减少裂解量(burst size)。这可能是因为在较高的土壤温度条件下易提高土壤酶活性,而较高的蛋白酶活性会导致病毒更快失活和分解[67]。Egilmez等[82]发现,温度范围在30−50 ℃条件下分离得到的噬菌体多为裂解性噬菌体;而在温度范围为15−25 ℃条件下,则转化为溶原性噬菌体。Yeager等[68]研究了脊髓灰质炎Ⅰ型野毒株在不同温度下的生存时间,发现在37 ℃条件下,病毒最多只能生存12 d,而在4 ℃条件下,180 d仍然能够检测到存活的病毒。这一研究结果意味着不同温度对新型冠状病毒存活时间的影响不一,或许这也是之前新冠疫情期间疫情不断反复的原因之一,即其中低温冷链运输水产品等条件下病毒存活时间较长。Hurst等[83]比较了土壤温度、含水量、需氧微生物的存在、物理吸附程度、磷和铝浓度以及pH对几种典型病毒存活的影响,发现土壤温度是影响土壤病毒最为关键的因素。另外,在当前全球气候变暖背景下,温度对土壤病毒及其介导的生物地球化学循环过程和生态功能的影响机制更加值得深入探究。
土壤pH不仅能够影响宿主的生长,还影响病毒的存活率[78]。据报道,木糖氧化无色杆菌(Achromobacter xylosoxidans)噬菌体phiAxp-3在中性pH下的感染活性最高,在pH为4.0和10.0时分别下降了90.25%和75.76%[84]。Sykes等[85]试图从不同pH土壤中分离链霉菌噬菌体,尽管在这些土壤中存在嗜酸性链霉菌(Acidophilic streptomycetes),但在pH低于6.0的土壤中并未检测到噬菌体,而从中性土壤中分离的部分噬菌体却能够在pH为5.5时裂解嗜酸链霉菌。Sykes和Williams[86]发现放线菌噬菌体(actinophage)吸附在高岭石上和游离在高岭石之外两种状态下,放线菌噬菌体对宿主的感染潜力分别在pH 6.1和4.9时丧失,这可能是由于H+在高岭石表面的积累削弱了噬菌体的感染潜力。pH也会影响土壤颗粒表面对病毒的吸附和解吸行为,以间接的方式影响病毒存活。目前的研究发现,病毒吸附在固体表面是一种常见现象,其吸附特征与土壤颗粒表面的静电特性紧密相关,而土壤颗粒表面的静电特性很大程度上受到土壤pH值的影响[87]。Bales等[88]研究发现,在pH 5.0时噬菌体MS-2的吸附量比pH 7.0时高,一般认为随着pH值的升高,噬菌体在土壤颗粒上的吸附量有下降的趋势。Bales等[89]的研究表明,在有Ca2+存在时噬菌体MS-2在硅土上的吸附量是在无Ca2+存在时的10倍以上。因此,土壤pH也是土壤病毒的主要影响因子。
此外,黏土矿物能够保护噬菌体不受土壤微生物及相关因素干扰而失活,即使没有宿主细菌,病毒粒子也能长期存活[90-91]。Straub等[69]报道了不同土壤类型对土壤病毒活性的影响,结果表明病毒在黏壤土中比在砂土中存活更长时间。Kapuscinski等[87]证明病毒与胶体和颗粒物质的结合延长了存活时间。Lipson和Stotzky[92]发现高岭石增强了病毒扩散,使病毒更快地向宿主细胞表面转移发生物理接触,从而提高了病毒的存活率。黏土矿物可以保护噬菌体免受紫外线照射导致的活性下降[93]。另外,不同生态系统类型中土壤病毒丰度也存在显著差异,如森林生态系统中土壤病毒丰度约为(1.31−4.17)×109 VLPs/g,农田生态系统中约为(0.87−1.10)×109 VLPs/g,而沙漠生态系统中约为103−105 VLPs/g[3, 5]。综上所述,土壤具有众多理化性质指标,这些指标对土壤中的病毒存在产生显著的影响,但其中具体影响机制目前尚不清楚,亟待进一步研究。
2.4 土壤噬菌体感染宿主微生物的主要作用机制由于土壤生态系统中原核生物数量及多样性较高,因此土壤中病毒主要是以侵染原核生物的噬菌体(bacteriophage或phage)为主,此外土壤中一些病毒也可以侵染一些特定真菌、土壤线虫及植物根系等[5, 94]。本文主要根据噬菌体生活史的不同(图 4),将噬菌体细分为裂解性噬菌体(lytic phage)和溶原性噬菌体(lysogenic phage)[95-96]。如图 4所示,噬菌体具有不同的生活史策略,主要包括裂解循环、溶原循环、慢性循环和伪溶原循环[96]。
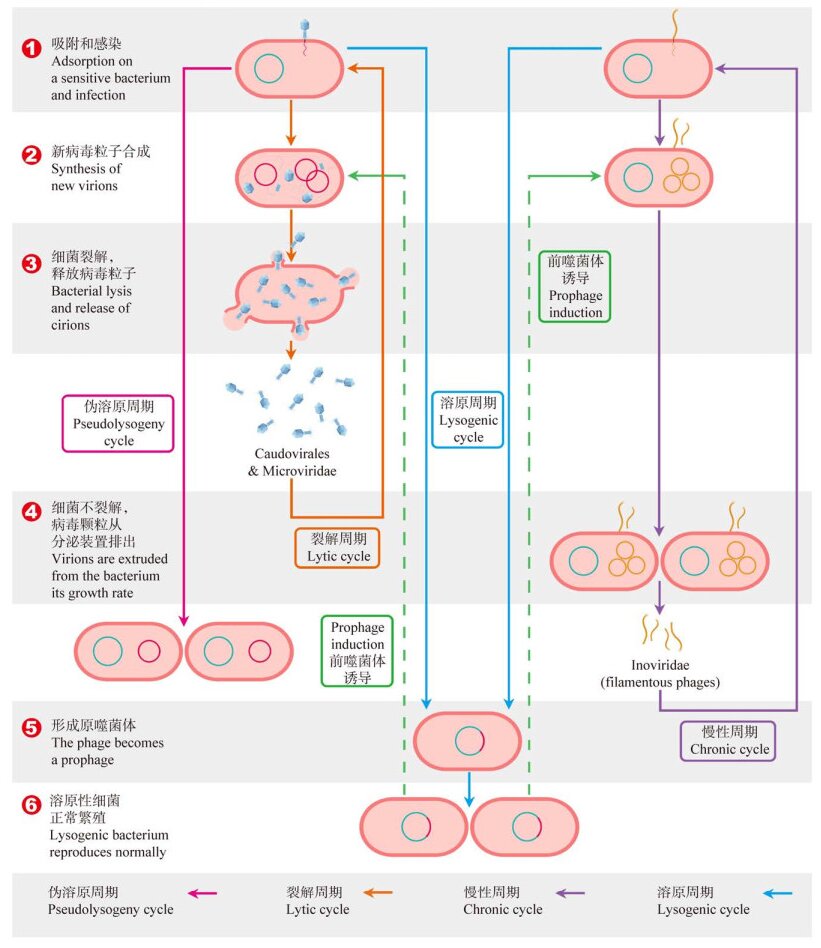
|
| 图 4 噬菌体感染宿主微生物的机制示意图 Figure 4 Infection mechanisms of phages. |
噬菌体感染宿主细菌后,快速增殖并迅速裂解宿主、释放子代的种群繁殖方式称为裂解性感染(图 4中棕色箭头)。裂解性病毒通常可以在较短时间内完成裂解循环从而实现自身增殖,而溶原性病毒则是在吸附、感染宿主细胞后,会进入溶原状态,即将自身基因组整合到宿主基因组上形成前噬菌体(prophage)并随宿主繁殖进行复制,胞内溶原性噬菌体会保持前噬菌体状态,直至遭受环境胁迫或刺激(如升温等)时方才转化为裂解性噬菌体,并在胞内增殖后裂解宿主细胞[7, 96]。由于噬菌体存在不同的生活史策略,因此噬菌体与宿主细菌群落间的互作模式较为复杂,主要包括裂解寄生及溶原共生等[30]。
土壤微环境异质性对噬菌体-宿主互作具有一定的限制,如宿主细胞的异质分布、局部寡营养条件导致宿主细胞低活性、破碎的孔隙水膜网络以及土壤颗粒表面吸附等[71]。裂解循环不利于噬菌体-宿主长期共存,因此噬菌体更偏好溶原循环这一生活史策略[97-99]。溶原噬菌体感染宿主后,将它们的基因组整合到宿主基因组中,形成前噬菌体,随着宿主细胞的生长和分裂而进行同步复制[100],而非立刻裂解宿主细菌(图 4中蓝色箭头)。然而,这种溶原性噬菌体在特定刺激下会转变为裂解性噬菌体,迅速裂解宿主细菌。这种生存策略使得病毒在面对土壤环境胁迫时,仍然能够保持种群生存能力[98, 100]。研究表明,具有溶原性的噬菌体在土壤中广泛存在,40%的陆地细菌种群含有诱导型溶原性噬菌体[71, 100]。然而,溶原性噬菌体的作用很少在土壤中得到评估。
土壤中溶原噬菌体可能根据环境条件及宿主可利用性,在裂解和溶原间转换[101]。采用前噬菌体化学诱导的方法进行实验,发现在宿主细菌数量多、活性高时,噬菌体一般采用裂解循环方式快速增殖;而当养分物质匮乏、宿主细胞的状态不佳、数量较低时,新释放的病毒粒子很难找到未感染的宿主细胞进行新一轮的感染,不能满足裂解性噬菌体快速及大量感染的需求,因此噬菌体往往采取溶原循环方式延长潜伏期,以求适应环境胁迫并存活下来。Maslov和Sneppen[98]应用博弈论建立模型研究发现,宿主细菌在环境胁迫条件下,噬菌体更倾向于进入溶原状态;而在宿主细菌处于较为适宜的环境中,并且噬菌体的扩散不受限制时,噬菌体会选择裂解途径感染宿主细菌。Liang等[99]则利用丝裂霉素C (mitomycin C)对前噬菌体进行诱导释放,完成了不同深度土壤微生物群落的溶原组分(losogenic fractions, LF)分析,发现土壤中病毒粒子和细菌的丰度均随土壤深度的增加而降低,而LF却随土壤深度的增加而增加,这可能是在深层土壤中较低的养分水平和其他环境胁迫条件诱导了更多的噬菌体以溶原的方式潜伏在宿主细胞中。
除了裂解和溶原循环模式,研究人员还发现了其他类型的噬菌体感染机制。如一些丝状噬菌体的基因组进入细菌后在细胞内进行复制增殖,组装为子代噬菌体,但子代噬菌体的释放不会裂解宿主细菌,而是以分泌的方式排出细菌,被称为慢性循环(chronic cycle) (图 4中紫色箭头)[96]。此外,还有研究发现,宿主细菌会携带噬菌体基因组在细胞中,处于携带噬菌体的载体状态(phage carrier state),称为伪溶原循环(pseudolysogeny cycle),通常被定义为噬菌体基因组既不整合到宿主基因组,也不进入裂解周期的状态,噬菌体基因组是以独特的游离形式存在(图 4中红色箭头)[96]。在这种状态下,不会引起宿主细胞裂解,游离体不能以稳定的方式复制,在子代宿主细胞之间不对称地出现,形成独特的表达谱[102]。这种状态与细胞饥饿状态有关[103],但目前关于这种感染模式仍知之甚少,其在土壤中的效应仍然未知。这些独特的感染方式都不同程度地保护了宿主细菌的生理活性,使得噬菌体和细菌在土壤中共存及发挥各自生态功能。
2.5 土壤病毒的主要生态功能 2.5.1 调控微生物群落结构及功能裂解性噬菌体对宿主菌种群的首要影响就是降低其丰度,从而影响微生物群落结构以及功能。然而,也有研究发现,噬菌体的存在维持了微生物的多样性。Zhang等[104]对中国香港近海环境中细菌的研究显示,添加噬菌体的细菌种群丰度比未添加噬菌体的对照组有所减少,而细菌多样性得到明显提高。Braga等[105]比较接种活性噬菌体和灭活噬菌体的处理,发现加入活性噬菌体会影响土壤细菌群落的组成和多样性,同时,将从一种自然土壤中分离得到的细菌悬液和噬菌体悬液(加入噬菌体分离株组成噬菌体混合剂)同时接种于无菌土壤中,发现群落α多样性增加,可能是因为噬菌体通过降低宿主菌的丰度为其他竞争对手提供了更多生态位,加速了微生物群落演替。此外也发现一些细菌进化出避免噬菌体侵染裂解的防御机制,如吸附机制、限制性修饰系统[106]、流产感染系统[107]及CRISPR相关基因系统等[108-109]。
溶原性噬菌体则可以通过溶原转化影响宿主对环境的适应能力,这种溶原转化可能是中性的,也可能对宿主生理学产生影响。溶原过程中,噬菌体携带的辅助代谢基因(auxiliary metabolic genes, AMGs)在感染期间和/或之后会影响宿主代谢,如改变宿主致病性或影响宿主生物膜的形成,即噬菌体基因组整合到宿主细胞后,会影响土壤中的微生物宿主群体代谢调节[110]。此外,病毒也可能获得特定宿主基因(即辅助代谢基因),从而提高病毒的环境适应能力。Sullivan等[111]证实了蓝藻噬菌体从蓝藻基因组上“窃取”了光合作用有关的蛋白基因,使得被入侵的蓝藻产生更多的能量,帮助噬菌体繁殖。Ghosh等[112]的研究显示阿特拉津(农药)污染土壤中的噬菌体获得了一个编码氯水解酶(分解阿特拉津所需的酶)的hetrzn基因,这可能与提高病毒环境适应能力有关。最近,Tzipilevich等[113]对枯草芽孢杆菌(Bacillus subtilis)及其裂解噬菌体SPP1的研究发现,缺乏SPP1受体的抗性菌可以被附近感染细胞释放的噬菌体裂解酶直接裂解。这些现象为土壤中病毒介导水平基因转移提供了更多的可能性。
另外,溶原性噬菌体为了不裂解宿主细胞,通常会编码抑制因子和转录调控蛋白来抑制裂解性复制相关基因的表达[86]。这些相同的调节蛋白也可以与宿主染色体中的同源序列结合,下调宿主细菌的代谢功能并降低能量消耗[103]。这种噬菌体对宿主基因的调控有助于宿主细胞在养分贫瘠的环境条件中生存。除了代谢调节之外,前噬菌体携带的基因可以保护细菌宿主免受其他类型的噬菌体感染(双重感染免疫)。双重感染免疫不仅保护前噬菌体免受竞争噬菌体的侵害,而且能够保护宿主抵抗多种病毒威胁[114]。与此同时,噬菌体也进化出多种反细菌适应性的策略,如anti-CRISPR/Cas机制和改变尾丝蛋白等策略[30, 115]。
由于土壤环境的微观异质性,再加上土壤细菌中溶原性噬菌体的普遍存在,溶原性噬菌体很可能以多种方式影响宿主种群动态,从而影响农业生产和人类健康。如通过溶原转化改变根瘤菌表型,对根瘤定殖、固氮效率和作物生产力产生正效应[116]。然而,同时也可能使无致病能力的菌株转变为致病菌,增加宿主菌株毒力,侵入生态位造成种群结构改变,对农业生产造成负效应,如β-棒状杆菌(β-corynebacterium)噬菌体的溶原转换可使无毒力的白喉棒状杆菌(Corynebacterium diphtheriae)转变为有毒力的菌株[117]。另外,也有研究表明在金黄色葡萄球菌(Staphylococcus aureus)中的毒力基因在前噬菌体的诱导下表达显著增加[118]。引入的毒力基因通过增强宿主生物的感染能力和适应能力,为宿主细菌扩大其环境生态位,而作为反馈,噬菌体增强了自身生存机会,有助于噬菌体的生存和繁殖[119]。此外,噬菌体通过溶原途径的编码产物也可以促进生物膜的发育[120],从而影响细菌对抗生素或噬菌体的抗性,增强了整个细菌群体的生存能力。例如,铜绿假单胞菌的溶原噬菌体Pf4可在宿主细胞中编码并释放一种带负电荷的长丝,将生物膜构建成液晶形式,提高生物膜的耐旱性和抗生素抗性[121]。McDougald等[122]也通过对细菌生物膜的研究发现,被溶原噬菌体Pf4感染的铜绿假单胞菌生物膜比未感染的生物更加稳定,即溶原性噬菌体引入的性状在个体水平和种群水平上影响宿主细胞。此外,噬菌体与宿主之间基于裂解与溶原互作关系还发展出“杀死优胜者(kill-the-winner)”[123]、“搭载优胜者(piggyback-the-winner)”[124]等生长模型,而噬菌体与宿主间的这种军备竞赛关系可以促进微生物群落不断进化,对微生物群落的多样性、功能稳定性和环境资源再分配均具有重要意义[125-129],但目前关于土壤噬菌体-细菌互作关系及其在土壤生态系统中的功能仍知之甚少[62, 64, 130-135]。近年来,土壤学科中涌现出一门新兴领域,即“土壤孔隙学(soilporelogy)”,将土壤微观环境与微生物群落结构的动态变化有机地融合在一起,为研究提供了新的视角。这一领域旨在从土壤物理结构的角度出发,深入剖析了微生物群落结构的变化[136]。未来,这种方法可能会成为土壤噬菌体研究的主流方向和研究热点,为更全面地理解土壤生态系统的复杂性提供有力支持。
2.5.2 参与元素生物地球化学循环有研究报道,噬菌体可以通过裂解宿主细胞释放出营养物质或调控与养分周转相关的微生物种群数量等方式影响元素生物地球化学循环[130, 137-138]。当裂解性噬菌体感染宿主细菌后,会快速增殖并迅速裂解微生物细胞,释放出多种有机化合物(细胞质、胶体碎片、大细胞壁碎片),并以可溶性有机质(dissolved organic matter, DOM)和颗粒态有机质(particulate organic matter, POM)的形式反馈给生态系统,被其他微生物重新利用,从而影响环境中元素的生物地球化学循环[139]。这些养分物质可能在数小时内就被附近其他微生物快速解聚和再利用[140-141]。裂解产物被存活的细菌利用,刺激其生长,导致病毒产生速率和细菌生长速率的耦合[142-143]。通过病毒裂解的细菌/真菌可释放一定量的可溶性碳,降低土壤碳库中矿物结合态碳等惰性碳比例,进而促进土壤碳周转,且这种由病毒介导的碳循环过程能够有效固存可溶性有机碳在土壤和水体中。土壤病毒也会在其他养分循环过程中发挥重要作用,如氮(蛋白质和核酸)、硫(蛋白质)和磷(磷酸盐、细胞膜和核酸)[7, 9]。如在农业生产中,豆科植物根瘤菌共生往往影响土壤氮通量,而能够感染各种根瘤菌菌株的噬菌体(rhizobiophages)可以通过调控根瘤菌数量[144-145],直接或者间接地影响土壤固氮量及效率,进而影响土壤供氮能力,在土壤根际生态过程中发挥重要作用。同时,宿主微生物的裂解也代表一种腐殖质的形成方式,由于微生物介导的腐殖质分解是土壤生态过程的关键一环,因此,噬菌体裂解作用可能会提高土壤生态系统的碳氮固持能力[16-17]。另外,噬菌体还能以裂解或溶原方式影响微生物多样性和群落结构[15],甚至可以调控宿主菌种群数量以及关键基因的表达来影响微生物介导的土壤养分循环周转速率[137]。对多年冻土生态系统的宏基因组分析表明,噬菌体可以感染关键的碳循环微生物,从而影响生物地球化学循环[146]。此外,Jin等[147]对中国南方红树林土壤样本中的病毒群落研究显示,病毒中有丰富的辅助碳水化合物活性酶(carbohydrate-active enzyme, CAZyme)基因,参与红树林土壤中含量丰富的复杂多糖的生物降解过程。由于土壤生态系统的复杂性,往往有多种因素影响噬菌体-宿主菌的互作过程,虽然噬菌体直接或间接地参与和影响生物地球化学的元素循环,但目前难以准确地评价病毒对土壤养分循环和土壤健康的贡献。
2.5.3 介导水平基因转移病毒作为水平基因转移(horizontal gene transfer, HGT)的媒介,可以增强宿主适应能力,促进宿主细胞的进化[148]。不需要任何媒介的裸露DNA分子与自然感受态细胞间相互作用的水平基因转移方式称为转化(transformation)。病毒引起宿主细胞裂解并释放出DNA,释放到环境中的DNA被其他微生物吸收、整合,引起微生物的遗传变异,推动微生物种群的进化。虽然噬菌体释放的细菌DNA片段可以在土壤颗粒的表面长期存在,但由于土壤环境的复杂性,细菌细胞对土壤中DNA的可利用程度仍然未知。感受态细菌和胞外游离DNA的存在是发生转化的先决条件,目前研究认为,不是每种细菌都能够进入感受态,因此噬菌体裂解释放宿主DNA推动水平基因转移具有特异性和局限性[148]。转导(transduction)是土壤中病毒介导水平基因转移的主要方式。正常情况下,病毒仅利用宿主细胞体内机制来复制它们自己的遗传物质。然而,子代噬菌体在细菌胞内进行装配时,可能将宿主细菌的DNA误认作噬菌体本身的DNA组装在头部蛋白质衣壳内,当进行新一轮感染时,这些遗传物质会进入同种或异种的受体细菌内,实现水平基因转移[148]。转导是细菌间噬菌体介导的基因转移机制,包含广义转导和特殊转导两种机制。在广义转导中,任何单个或一组细菌基因都可能被噬菌体包装[149];而特异性转导是噬菌体裂解复制时,只转移靠近前噬菌体整合位点的基因[150]。
另外,病毒也是抗生素抗性基因(antibiotic resistance genes, ARGs)的主要传播者。Subirats等[151]通过宏基因组学分析发现,病毒是环境中抗生素抗性基因的主要储存库。相关研究表明,噬菌体DNA片段中ARGs的相对丰度(0.26%)高于细菌DNA片段(0.18%)[152]。研究发现,通过噬菌体的转导可以转移抗生素抗性基因,从而增加供体菌的耐药性[153],如噬菌体对基因的转导增加了链球菌对四环素的抗性以及对氯霉素、大环内酯类抗生素、林可霉素的多重耐药性[154]。目前,已经在不同环境介质中发现噬菌体携带β-内酰胺类抗生素抗性基因的报道[155]。然而,由于噬菌体的特异性及其传播的DNA量较少,抗药性的转导现象仅能发生在同种细菌内,且通常仅能传递对一种抗生素的耐药性。在土壤中,尤其是黏土和有机质含量较多的土壤中,土壤颗粒具有极高的比表面积,为噬菌体和宿主的吸附和互作提供了许多潜在的位点。然而,由于检测技术的局限性,目前还没有明确证据表明土壤细菌中存在着原位转导现象。
然而关于土壤噬菌体介导的水平基因转移研究目前仍是土壤病毒研究的一个难点。绝大多数土壤噬菌体受到宿主细菌的选择性限制,不同类型的噬菌体具有不同的宿主范围,大部分噬菌体只能感染特定种类的细菌。这就意味着,在这种情况下,只有与噬菌体宿主细菌相容的细菌才能在转移中参与,某种程度上限制了基因流动的广度。其次,环境条件对土壤噬菌体介导的基因转移也具有一定影响。例如,温度、湿度、营养物质的可用性等因素都可能影响噬菌体和细菌的生存能力和活性,从而间接地影响水平基因转移的频率和效率。此外,土壤中存在的DNA分解酶和其他降解因子可能会迅速降解游离的DNA,从而限制基因片段在环境中的存活时间。这使得基因片段更难以在环境中保持稳定,并减少了水平基因转移的机会。最后,基因转移的成功还取决于水平基因转移所涉及的细菌是否能够有效地整合新的基因片段到其基因组中。这个过程并不是绝对高效的,很多转移的基因可能会因为宿主细菌的防御机制或其他原因而无法被整合。总体而言,尽管土壤噬菌体介导的水平基因转移是微生物界中重要的基因传递方式,但其在自然界中受到诸多限制,尤其是在具有高度异质性的土壤环境中。
3 土壤噬菌体疗法在农业生产中的应用前景每年全球有大量粮食产量因植物病害而损失,传统化学农药和熏蒸等方法虽然短期内见效快,但也具有靶向性不强、破坏土壤正常微生物群落等缺点,影响土壤微生物发挥其生态功能。另外近几十年来,抗生素发展迅速,越来越多的抗生素进入环境中,诱导出大量抗生素抗性细菌/ 基因,给土壤和农业生产带来了较大的安全隐患[156]。基于此,目前“农业噬菌体疗法”已被开发用于灭活土壤-植物系统中的病原菌[157-158]。相比于其他方法,噬菌体疗法突出优点是特异性裂解某个宿主细菌,而不攻击整个细菌群体。此外,当宿主细胞减少时,噬菌体数量也减少,产生一定的比例关系,维持了微生物的稳定性和多样性。甚至噬菌体裂解病原菌的同时可以为其他微生物群落提供更多的生态位和养分、恢复群落多样性以及降低病原菌与其他菌群的竞争能力,这可能导致病原体丰度的进一步下降。
近年来,噬菌体治疗法逐渐应用于农业生产中,并取得了一定的成效。Askora等[159]利用青枯病菌(Ralstonia solanacearum)噬菌体原液接种土壤,显著降低了番茄和烟草植物中青枯病的发病率。Wang等[157]利用从土壤中分离到的4株高效裂解的青枯病菌的噬菌体,通过温室和大田研究发现噬菌体疗法能够显著降低青枯病的发生。Fujiwara等[160]报道了番茄种子浸泡在噬菌体φRSL1溶液中能抑制青枯病菌在番茄根系的定殖。Frampton等[161]从果园土壤中分离获得了275种噬菌体(属于Caudovirales),发现其中大部分噬菌体能够裂解植物病原细菌丁香假单胞菌(Pseudomonas syringae)。此外,Chae等[162]用脱脂奶粉保护噬菌体活性,制成噬菌体药剂并应用在水稻上,成功控制了Xanthomonas菌对水稻的感染。Wang等[157]揭示,噬菌体不仅可以“专性猎杀”和“精准靶向”土传青枯病的病原菌,降低其生存竞争能力,还能重新调整根际土壤菌群结构、恢复群落多样性并增加群落中有益菌丰度。
噬菌体疗法应用于土壤-植物系统中时,由于细菌会对噬菌体产生抗性突变,单宿主特异性噬菌体的应用效率有限。因此,未来可能需要开发多价噬菌体(polyvalent phages)或多种噬菌体混合物(phage cocktails),以提高病原菌灭活效率。Yu等[163]发现通过实验室选择,某些宿主特异性噬菌体可以进化成能够感染一种以上宿主细菌的多价噬菌体。Ye等[164]也利用一种可以感染大肠杆菌和铜绿假单胞菌的多价噬菌体探索了噬菌体对土壤中宿主细菌的灭活效果,在土壤培养63 d后,大肠杆菌和铜绿假单胞菌的丰度不仅显著降低,还相应减少了土壤中抗生素抗性基因数量。另外,相关研究发现多价噬菌体可以降低莴苣中寄生细菌及其抗生素抗性基因数量[165]。Zhao等[166]研究了多种噬菌体混合剂、多价噬菌体、单宿主特异噬菌体对土壤中大肠杆菌和铜绿假单胞菌的灭杀率,研究表明灭杀率依次表现为多种噬菌体混合剂 > 多价噬菌体 > 单宿主特异噬菌体。因此,研究开发多价噬菌体同时结合多种噬菌体制成噬菌体药剂治疗土传病害是未来农业噬菌体疗法主要的发展方向。
4 研究展望综上所述,土壤微生物学是地球科学、农业科学和生命科学的交叉学科前沿和热点。未来土壤病毒领域的研究将会是土壤学、生态学、微生物学、环境科学、信息学和计算机科学等多学科和多尺度交叉的结果。由于土壤自身的复杂特性以及其动态变化特征,人们对土壤中病毒类型、丰度、分布特征与功能、生态环境效应、病毒-宿主微生物互作过程及作用机理仍知之甚少,相关研究一直是微生物学研究领域的薄弱环节,也是当前微生态学和环境科学等领域的研究热点和前沿。
基于此,未来研究可能需要更多地集中于土壤病毒分离纯化研究、土壤病毒多样性及生态学效应研究、土壤病毒迁移传播机制研究、土壤病毒-宿主互作机制研究和土壤病毒应用研究5个方面。
(1) 土壤病毒分离纯化研究:不断优化提取、分离并纯化土壤病毒方法,并开展土壤病毒(包括烈性噬菌体和温和噬菌体等)的生物学特性鉴定研究,建立完善的病毒资源信息库,是研究土壤病毒的基础,可以促进我们对土壤病毒有充分认知及了解,目前将噬菌体作为指示病毒的研究和应用也因此受到大量关注。
(2) 土壤病毒多样性及生态学效应研究:通过高通量组学技术及宏基因组、宏转录组、宏蛋白质组、宏代谢组等先进分子生物学技术,获取土壤病毒完整基因组信息,深入探究不同土壤类型中土壤病毒的分布特征、多样性、基因组结构、组成及变异、基因功能、基因演化以及宿主特性、侵染宿主范围等,以期进一步揭示土壤病毒在生态系统中的作用,为农业生产、环境保护和生态平衡的维护提供一定科学依据。
(3) 土壤病毒迁移传播机制研究:土壤特有的微观孔隙特征和水分分布的异质性及动态变化特性定义了微生物特殊的微生境条件,因此,从孔隙结构特征和个体细胞等微观尺度及区域、全国甚至全球等宏观尺度上剖析土壤病毒活动轨迹及病毒在土壤中迁移传播的时空模式,是深入了解土壤病毒生命形态及生态过程的关键。
(4) 土壤病毒-宿主互作机制研究:通过细胞生物学、生物化学和生物物理学等方面技术,深入了解土壤病毒的生命周期、侵染能力和致病性等重要特征,揭示土壤病毒与宿主之间的相互作用方式和信号传递途径及其对土壤养分循环等复杂过程的作用机制,了解土壤病毒的侵染机制和宿主耐受机制,从生物进化的角度加深对土壤病毒的了解。值得注意的是,利用土壤病毒生物地球化学互作过程计算机仿真模型,一定程度上可以从机理层面探究土壤病毒和宿主微生物在动态变化环境系统中的生命轨迹过程以及环境因子对微生物个体能动性、种群扩张和互作的影响机制,对深入探究土壤病毒-微生物种群互作过程以及微生物多样性维持机制具有重要推动作用。
(5) 土壤病毒应用研究:结合基础研究的理论积累,通过从分子、细胞、菌落和环境生境等不同尺度明确土壤病毒的生理生态学特征以及其与其他生命形态和在不同界面间的互作模式与响应机制,提升对土壤病毒存在机制与生态功能的认知,构建土壤病毒的生物地球化学过程模式,以对土壤病毒及宿主微生物进行合理利用与调控。通过生物工程、生物防治和环境工程等技术,开发新型的针对不同病原菌的土壤病毒生物制剂,并大量开展长期培养、盆栽或大田试验,探究利用土壤病毒(如噬菌体)来靶向调控细菌、真菌及古菌等宿主微生物数量和群落结构进而达到控制土壤中的有害生物和污染源、保障生态环境安全这一目标的可行性和安全性,提高土壤质量和生产力,充分发挥噬菌体疗法在农业中的应用价值。
| [1] | KUYPERS MMM, MARCHANT HK, KARTAL B. The microbial nitrogen-cycling network[J]. Nature Reviews Microbiology, 2018, 16(5): 263-276 DOI:10.1038/nrmicro.2018.9. |
| [2] | TRUBL G, JANG HB, ROUX S, EMERSON JB, SOLONENKO N, VIK DR, SOLDEN L, ELLENBOGEN J, RUNYON AT, BOLDUC B, WOODCROFT BJ, SALESKA SR, TYSON GW, WRIGHTON KC, SULLIVAN MB, RICH VI. Soil viruses are underexplored players in ecosystem carbon processing[J]. mSystems, 2018, 3(5): e00076-18. |
| [3] | WILLIAMSON KE, RADOSEVICH M, WOMMACK KE. Abundance and diversity of viruses in six Delaware soils[J]. Applied and Environmental Microbiology, 2005, 71(6): 3119-3125 DOI:10.1128/AEM.71.6.3119-3125.2005. |
| [4] | SUTTLE CA. Marine viruses—major players in the global ecosystem[J]. Nature Reviews Microbiology, 2007, 5(10): 801-812 DOI:10.1038/nrmicro1750. |
| [5] | WILLIAMSON KE, FUHRMANN JJ, WOMMACK KE, RADOSEVICH M. Viruses in soil ecosystems: an unknown quantity within an unexplored territory[J]. Annual Review of Virology, 2017, 4(1): 201-219 DOI:10.1146/annurev-virology-101416-041639. |
| [6] | BI L, YU DT, HAN LL, DU S, YUAN CY, HE JZ, HU HW. Unravelling the ecological complexity of soil viromes: challenges and opportunities[J]. Science of the Total Environment, 2022, 812: 152217 DOI:10.1016/j.scitotenv.2021.152217. |
| [7] | KUZYAKOV Y, MASON-JONES K. Viruses in soil: nano-scale undead drivers of microbial life, biogeochemical turnover and ecosystem functions[J]. Soil Biology and Biochemistry, 2018, 127: 305-317 DOI:10.1016/j.soilbio.2018.09.032. |
| [8] | PIEL D, BRUTO M, LABREUCHE Y, BLANQUART F, GOUDENÈGE D, BARCIA-CRUZ R, CHENIVESSE S, le PANSE S, JAMES A, DUBERT J, PETTON B, LIEBERMAN E, WEGNER KM, HUSSAIN FA, KAUFFMAN KM, POLZ MF, BIKARD D, GANDON S, ROCHA EPC, le ROUX F. Phage-host coevolution in natural populations[J]. Nature Microbiology, 2022, 7(7): 1075-1086 DOI:10.1038/s41564-022-01157-1. |
| [9] |
王光华, 刘俊杰, 朱冬, 叶茂, 朱永官. 土壤病毒的研究进展与挑战[J]. 土壤学报, 2020, 57(6): 1319-1332.
WANG GH, LIU JH, ZHU D, YE M, ZHU YG. A review of researches on viruses in soil-advancement and challenges[J]. Acta Pedologica Sinica, 2020, 57(6): 1319-1332 (in Chinese). |
| [10] | PRATAMA AA, DIRK van ELSAS J. The 'neglected' soil virome-potential role and impact[J]. Trends in Microbiology, 2018, 26(8): 649-662 DOI:10.1016/j.tim.2017.12.004. |
| [11] | STARR EP, NUCCIO EE, PETT-RIDGE J, BANFIELD JF, FIRESTONE MK. Metatranscriptomic reconstruction reveals RNA viruses with the potential to shape carbon cycling in soil[J]. Proceedings of the National Academy of Sciences of the United States of America, 2019, 116(51): 25900-25908. |
| [12] | WANG S, YU SX, ZHAO XY, ZHAO XL, MASON-JONES K, ZHU ZK, REDMILE-GORDON M, LI Y, CHEN JP, KUZYAKOV Y, GE TD. Experimental evidence for the impact of phages on mineralization of soil-derived dissolved organic matter under different temperature regimes[J]. Science of the Total Environment, 2022, 846: 157517 DOI:10.1016/j.scitotenv.2022.157517. |
| [13] | WEI XM, GE TD, WU CF, WANG S, MASON-JONES K, LI Y, ZHU ZK, HU YJ, LIANG C, SHEN JL, WU JS, KUZYAKOV Y. T4-like phages reveal the potential role of viruses in soil organic matter mineralization[J]. Environmental Science & Technology, 2021, 55(9): 6440-6448. |
| [14] | SRINIVASIAH S, LOVETT J, GHOSH D, ROY K, FUHRMANN JJ, RADOSEVICH M, WOMMACK KE. Dynamics of autochthonous soil viral communities parallels dynamics of host communities under nutrient stimulation[J]. FEMS Microbiology Ecology, 2015, 91(7): fiv063 DOI:10.1093/femsec/fiv063. |
| [15] | CHEVALLEREAU A, PONS BJ, van HOUTE S, WESTRA ER. Interactions between bacterial and phage communities in natural environments[J]. Nature Reviews Microbiology, 2022, 20(1): 49-62 DOI:10.1038/s41579-021-00602-y. |
| [16] | WU HQ, WAN SC, RUAN CJ, WAN W, HAN M, CHEN GW, LIU Y, ZHU K, LIANG C, WANG G. Soil microbial necromass: the state-of-the-art, knowledge gaps, and future perspectives[J]. European Journal of Soil Biology, 2023, 115: 103472 DOI:10.1016/j.ejsobi.2023.103472. |
| [17] | WU HQ, WAN SC, RUAN CJ, NIU XY, CHEN GW, LIU Y, ZHU K, SCHULIN R, WANG G. Phage‐bacterium interactions and nutrient availability can shape C and N retention in microbial biomass[J]. European Journal of Soil Science, 2022, 73(4): e13296 DOI:10.1111/ejss.13296. |
| [18] | EMERSON JB. Soil viruses: a new hope[J]. mSystems, 2019, 4(3): e00120-19. |
| [19] | SOKOL NW, SLESSAREV E, MARSCHMANN GL, NICOLAS A, BLAZEWICZ J, BRODIE EL, FIRESTONE MK, FOLEY MM, HESTRIN R, HUNGATE BA, KOCH BJ, STONE BW, SULLIVAN MB, ZABLOCKI O, SOIL MICROBIOME CONSORTIUM LL, PETT-RIDGE J. Life and death in the soil microbiome: how ecological processes influence biogeochemistry[J]. Nature Reviews Microbiology, 2022, 20(7): 415-430 DOI:10.1038/s41579-022-00695-z. |
| [20] | VASAR M, DAVISON J, SEPP SK, MUCINA L, OJA J, AL-QURAISHY S, ANSLAN S, BAHRAM M, BUENO CG, CANTERO JJ, DECOCQ G, FRASER L, HIIESALU I, HOZZEIN WN, KOOREM K, MENG YM, MOORA M, ONIPCHENKO V, ÖPIK M, PÄRTEL M, et al. Global soil microbiomes: a new frontline of biome‐ecology research[J]. Global Ecology and Biogeography, 2022, 31(6): 1120-1132 DOI:10.1111/geb.13487. |
| [21] | AL-SHAYEB B, SACHDEVA R, CHEN LX, WARD F, MUNK P, DEVOTO A, CASTELLE CJ, OLM MR, BOUMA-GREGSON K, AMANO Y, HE C, MÉHEUST R, BROOKS B, THOMAS A, LAVY A, MATHEUS-CARNEVALI P, SUN C, GOLTSMAN DSA, BORTON MA, SHARRAR A, et al. Clades of huge phages from across Earth's ecosystems[J]. Nature, 2020, 578(7795): 425-431 DOI:10.1038/s41586-020-2007-4. |
| [22] | SCHULZ F, ROUX S, PAEZ-ESPINO D, JUNGBLUTH S, WALSH DA, DENEF VJ, MCMAHON KD, KONSTANTINIDIS KT, ELOE-FADROSH EA, KYRPIDES NC, WOYKE T. Giant virus diversity and host interactions through global metagenomics[J]. Nature, 2020, 578(7795): 432-436 DOI:10.1038/s41586-020-1957-x. |
| [23] | LIANG XL, RADOSEVICH M, DeBRUYN JM, WILHELM SW, McDEARIS R, ZHUANG J. Incorporating viruses into soil ecology: a new dimension to understand biogeochemical cycling[J]. Critical Reviews in Environmental Science and Technology, 2024, 54(2): 117-137 DOI:10.1080/10643389.2023.2223123. |
| [24] | NUCCIO EE, STARR E, KARAOZ U, BRODIE EL, ZHOU JZ, TRINGE SG, MALMSTROM RR, WOYKE T, BANFIELD JF, FIRESTONE MK, PETT-RIDGE J. Niche differentiation is spatially and temporally regulated in the rhizosphere[J]. The ISME Journal, 2020, 14(4): 999-1014 DOI:10.1038/s41396-019-0582-x. |
| [25] | WILPISZESKI RL, AUFRECHT JA, RETTERER ST, SULLIVAN MB, GRAHAM DE, PIERCE EM, ZABLOCKI OD, PALUMBO AV, ELIAS DA. Soil aggregate microbial communities: towards understanding microbiome interactions at biologically relevant scales[J]. Applied and Environmental Microbiology, 2019, 85(14): e00324-19. |
| [26] | WANG G, OR D. Aqueous films limit bacterial cell motility and colony expansion on partially saturated rough surfaces[J]. Environmental Microbiology, 2010, 12(5): 1363-1373 DOI:10.1111/j.1462-2920.2010.02180.x. |
| [27] | VOS M, WOLF AB, JENNINGS SJ, KOWALCHUK GA. Micro-scale determinants of bacterial diversity in soil[J]. FEMS Microbiology Reviews, 2013, 37(6): 936-954 DOI:10.1111/1574-6976.12023. |
| [28] |
韩苗, 阮楚晋, 韩峥, 魏芯蕊, 刘莹, 朱堃, 王钢. 空间组织模式: 微生物群落组装的"游戏"规则[J]. 微生物学通报, 2022, 49(10): 4391-4411.
HAN M, RUAN CJ, HAN Z, WEI XR, LIU Y, ZHU K, WANG G. Spatial organization patterns: rules of "game" for microbial community assembly[J]. Microbiology China, 2022, 49(10): 4391-4411 (in Chinese). |
| [29] |
吴汉卿, 阮楚晋, 万炜, 李胜龙, 裴丁仪, 韩苗, 陈国炜, 刘莹, 朱堃, 王钢. 基于知识图谱分析的土壤氮循环功能基因研究进展[J]. 土壤学报, 2023, 60(1): 7-22.
WU HQ, RUAN CJ, WAN W, LI SL, PEI DY, HAN M, CHEN GW, LIU Y, ZHU K, WANG G. Progress of functional genes related to soil nitrogen cycling based on knowledge mapping[J]. Acta Pedologica Sinica, 2023, 60(1): 7-22 (in Chinese). |
| [30] | WEINBAUER MG. Ecology of prokaryotic viruses[J]. FEMS Microbiology Reviews, 2004, 28(2): 127-181 DOI:10.1016/j.femsre.2003.08.001. |
| [31] | GUTTMAN B, RAYA R, KUTTER E. Basic phage biology[M]//Bacteriophages: Biology and Applications. Boca Raton: CRC Press, 2004. |
| [32] | YUAN YH, GAO MY. Jumbo bacteriophages: an overview[J]. Frontiers in Microbiology, 2017, 8: 403. |
| [33] | ABERGEL C, LEGENDRE M, CLAVERIE JM. The rapidly expanding universe of giant viruses: mimivirus, pandoravirus, pithovirus and mollivirus[J]. FEMS Microbiology Reviews, 2015, 39(6): 779-796 DOI:10.1093/femsre/fuv037. |
| [34] | ABRAHÃO J, SILVA L, SILVA L SANTOS, BOU KHALIL JY, RODRIGUES R, ARANTES T, ASSIS F, BORATTO P, ANDRADE M, KROON EG, RIBEIRO B, BERGIER I, SELIGMANN H, GHIGO E, COLSON P, LEVASSEUR A, KROEMER G, RAOULT D, la SCOLA B. Tailed giant tupanvirus possesses the most complete translational apparatus of the known virosphere[J]. Nature Communications, 2018, 9: 749 DOI:10.1038/s41467-018-03168-1. |
| [35] | FISCHER MG. Giant viruses come of age[J]. Current Opinion in Microbiology, 2016, 31(4): 50-57. |
| [36] | SCHULZ F, ALTEIO L, GOUDEAU D, RYAN EM, YU FB, MALMSTROM RR, BLANCHARD J, WOYKE T. Hidden diversity of soil giant viruses[J]. Nature Communications, 2018, 9: 4881 DOI:10.1038/s41467-018-07335-2. |
| [37] | DIRK van ELSAS J, TREVORS JT, ROSADO AS, NANNIPIERI P. Modern Soil Microbiology[M]. Boca Raton: CRC Press, 2019. |
| [38] | ASHELFORD KE, DAY MJ, FRY JC. Elevated abundance of bacteriophage infecting bacteria in soil[J]. Applied and Environmental Microbiology, 2003, 69(1): 285-289 DOI:10.1128/AEM.69.1.285-289.2003. |
| [39] | SALMOND GPC, FINERAN PC. A century of the phage: past, present and future[J]. Nature Reviews Microbiology, 2015, 13(12): 777-786 DOI:10.1038/nrmicro3564. |
| [40] | PRANGISHVILI D, FORTERRE P, GARRETT RA. Viruses of the archaea: a unifying view[J]. Nature Reviews Microbiology, 2006, 4(11): 837-848 DOI:10.1038/nrmicro1527. |
| [41] | GHABRIAL SA, CASTÓN JR, JIANG DH, NIBERT ML, SUZUKI N. 50-plus years of fungal viruses[J]. Virology, 2015, 479/480: 356-368 DOI:10.1016/j.virol.2015.02.034. |
| [42] | LI CX, SHI M, TIAN JH, LIN XD, KANG YJ, CHEN LJ, QIN XC, XU JG, HOLMES EC, ZHANG YZ. Unprecedented genomic diversity of RNA viruses in arthropods reveals the ancestry of negative-sense RNA viruses[J]. eLife, 2015, 4: e05378 DOI:10.7554/eLife.05378. |
| [43] | GUO YR, HRYC CF, JAKANA J, JIANG HB, WANG D, CHIU W, ZHONG WW, TAO YJ. Crystal structure of a nematode-infecting virus[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(35): 12781-12786. |
| [44] | WANG AL, WANG CC. Viruses of the protozoa[J]. Annual Review of Microbiology, 1991, 45(1): 251-263 DOI:10.1146/annurev.mi.45.100191.001343. |
| [45] | RYBICKI EP. A top ten list for economically important plant viruses[J]. Archives of Virology, 2015, 160(1): 17-20 DOI:10.1007/s00705-014-2295-9. |
| [46] | WILLIAMSON KE, WOMMACK KE, RADOSEVICH M. Sampling natural viral communities from soil for culture-independent analyses[J]. Applied and Environmental Microbiology, 2003, 69(11): 6628-6633 DOI:10.1128/AEM.69.11.6628-6633.2003. |
| [47] | YU SX, WANG S, ZHAO XL, HU C, WEI L, ZHU ZK, LI Y, KUZYAKOV Y, CHEN JP, GE TD. Extraction optimisation to measure viral abundance in red soils[J]. Biology and Fertility of Soils, 2023, 59: 927-937 DOI:10.1007/s00374-023-01765-x. |
| [48] | PEDUZZI P, AGIS M, LUEF B. Evaluation of confocal laser scanning microscopy for enumeration of virus-like particles in aquatic systems[J]. Environmental Monitoring and Assessment, 2013, 185(7): 5411-5418 DOI:10.1007/s10661-012-2955-8. |
| [49] |
江艳华, 李风铃, 王联珠, 翟毓秀, 姚琳. 一株沙门氏菌裂解性噬菌体的分离鉴定及生物学特性[J]. 微生物学通报, 2015, 42(3): 534-542.
JIANG YH, LI FL, WANG LZ, ZHAI YX, YAO L. Isolation, identification and biological properties of a lytic phage against Salmonella[J]. Microbiology China, 2015, 42(3): 534-542 (in Chinese). |
| [50] | THURBER RV, HAYNES M, BREITBART M, WEGLEY L, ROHWER F. Laboratory procedures to generate viral metagenomes[J]. Nature Protocols, 2009, 4(4): 470-483 DOI:10.1038/nprot.2009.10. |
| [51] | CASTRO-MEJÍA JL, MUHAMMED MK, KOT W, NEVE H, FRANZ CMAP, HANSEN LH, VOGENSEN FK, NIELSEN DS. Optimizing protocols for extraction of bacteriophages prior to metagenomic analyses of phage communities in the human gut[J]. Microbiome, 2015, 3: 64 DOI:10.1186/s40168-015-0131-4. |
| [52] | RUAN CJ, NIU XY, XIONG GZ, CHEN GW, WU HQ, MA ZC, ZHU K, LIU Y, WANG G. Phenotypic and genotypic characterization of the new Bacillus cereus phage SWEP1[J]. Archives of Virology, 2021, 166(11): 3183-3188 DOI:10.1007/s00705-021-05222-8. |
| [53] | HAN M, NIU XY, XIONG GZ, RUAN CJ, CHEN GW, WU HQ, LIU Y, ZHU K, WANG G. Isolation, characterization and genomic analysis of a novel Arthrobacter sp. phage SWEP2[J]. Archives of Virology, 2023, 168(11): 276 DOI:10.1007/s00705-023-05898-0. |
| [54] | PAUL JH, SULLIVAN MB, SEGALL AM, ROHWER F. Marine phage genomics[J]. Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology, 2002, 133(4): 463-476 DOI:10.1016/S1096-4959(02)00168-9. |
| [55] | WANG GH, HAYASHI M, SAITO M, TSUCHIYA K, ASAKAWA S, KIMURA M. Survey of major capsid genes (g23) of T4-type bacteriophages in Japanese paddy field soils[J]. Soil Biology and Biochemistry, 2009, 41(1): 13-20 DOI:10.1016/j.soilbio.2008.07.008. |
| [56] | YU DT, HE JZ, ZHANG LM, HAN LL. Viral metagenomics analysis and eight novel viral genomes identified from the Dushanzi mud volcanic soil in Xinjiang, China[J]. Journal of Soils and Sediments, 2019, 19(1): 81-90 DOI:10.1007/s11368-018-2045-9. |
| [57] | SCOLA V, RAMOND JB, FROSSARD A, ZABLOCKI O, ADRIAENSSENS EM, JOHNSON RM, SEELY M, COWAN DA. Namib desert soil microbial community diversity, assembly, and function along a natural xeric gradient[J]. Microbial Ecology, 2018, 75(1): 193-203 DOI:10.1007/s00248-017-1009-8. |
| [58] |
阮楚晋, 熊广州, 牛欣尧, 陈国炜, 吴汉卿, 马泽超, 朱堃, 刘莹, 王钢. 一个东北农田黑土样品宏病毒组的初步分析[J]. 土壤学报, 2022, 59(5): 1447-1456.
RUAN CJ, XIONG GZ, NIU XY, CHEN GW, WU HQ, MA ZC, ZHU K, LIU Y, WANG G. Preliminary analysis of the virus metagenome in a farmland black soil of northeast China[J]. Acta Pedologica Sinica, 2022, 59(5): 1447-1456 (in Chinese). |
| [59] | DU SC, TONG XZ, LAI ACK, CHAN CK, MASON CE, LEE PKH. Highly host-linked viromes in the built environment possess habitat-dependent diversity and functions for potential virus-host coevolution[J]. Nature Communications, 2023, 14: 2676 DOI:10.1038/s41467-023-38400-0. |
| [60] | CAO MM, LIU SY, BI L, CHEN SJ, WU HY, GE Y, HAN B, ZHANG LM, HE JZ, HAN LL. Distribution characteristics of soil viruses under different precipitation gradients on the Qinghai-Xizang Plateau[J]. Frontiers in Microbiology, 2022, 13: 848305 DOI:10.3389/fmicb.2022.848305. |
| [61] | LIAO H, LI H, DUAN CS, ZHOU XY, AN XL, ZHU YG, SU JQ. Metagenomic and viromic analysis reveal the anthropogenic impacts on the plasmid and phage borne transferable resistome in soil[J]. Environment International, 2022, 170: 107595 DOI:10.1016/j.envint.2022.107595. |
| [62] | LIAO H, LI H, DUAN CS, ZHOU XY, LUO QP, AN XL, ZHU YG, SU JQ. Response of soil viral communities to land use changes[J]. Nature Communications, 2022, 13: 6027 DOI:10.1038/s41467-022-33771-2. |
| [63] | ZHENG XX, JAHN MT, SUN MM, FRIMAN VP, BALCAZAR JL, WANG JF, SHI Y, GONG X, HU F, ZHU YG. Organochlorine contamination enriches virus-encoded metabolism and pesticide degradation associated auxiliary genes in soil microbiomes[J]. The ISME Journal, 2022, 16(5): 1397-1408 DOI:10.1038/s41396-022-01188-w. |
| [64] |
祁慧鹓, 郑晓璇, 孙明明, 王金锋, 马迎飞, 朱冬, 王风贺, 蒋新, 叶茂. 土壤宏病毒组的研究方法与进展[J]. 土壤学报, 2021, 58(3): 568-577.
QI HY, ZHENG XX, SUN MM, WANG JF, MA YF, ZHU D, WANG FH, JIANG X, YE M. Review in the soil virus metagenome analytical methods and progress[J]. Acta Pedologica Sinica, 2021, 58(3): 568-577 (in Chinese). |
| [65] | SRINIVASIAH S, LOVETT J, POLSON S, BHAVSAR J, GHOSH D, ROY K, FUHRMANN JJ, RADOSEVICH M, WOMMACK KE. Direct assessment of viral diversity in soils by random PCR amplification of polymorphic DNA[J]. Applied and Environmental Microbiology, 2013, 79(18): 5450-5457 DOI:10.1128/AEM.00268-13. |
| [66] | JOŃCZYK-MATYSIAK E, ŁODEJ N, KULA D, OWCZAREK B, ORWAT F, MIĘDZYBRODZKI R, NEUBERG J, BAGIŃSKA N, WEBER-DĄBROWSKA B, GÓRSKI A. Factors determining phage stability/activity: challenges in practical phage application[J]. Expert Review of Anti-infective Therapy, 2019, 17(8): 583-606 DOI:10.1080/14787210.2019.1646126. |
| [67] | KIMURA M, JIA ZJ, NAKAYAMA N, ASAKAWA S. Ecology of viruses in soils: past, present and future perspectives[J]. Soil Science and Plant Nutrition, 2008, 54(1): 1-32 DOI:10.1111/j.1747-0765.2007.00197.x. |
| [68] | YEAGER JG, O'BRIEN RT. Enterovirus inactivation in soil[J]. Applied and Environmental Microbiology, 1979, 38(4): 694-701 DOI:10.1128/aem.38.4.694-701.1979. |
| [69] | STRAUB TM, PEPPER IL, GERBA CP. Persistence of viruses in desert soils amended with anaerobically digested sewage sludge[J]. Applied and Environmental Microbiology, 1992, 58(2): 636-641 DOI:10.1128/aem.58.2.636-641.1992. |
| [70] |
王燕, 阮楚晋, 谢文琳, 朱堃, 陈国炜, 刘莹, 王钢. 微观水分和养分条件对铜绿假单胞菌噬菌体裂解宿主过程的影响[J]. 土壤学报, 2023, 60(3): 835-845.
WANG Y, RUAN CJ, XIE WL, ZHU K, CHEN GW, LIU Y, WANG G. Effects of microscopic water and nutrient conditions on the host lysis process of Pseudomonas aeruginosa phage[J]. Acta Pedologica Sinica, 2023, 60(3): 835-845 (in Chinese). |
| [71] | STEWART FM, LEVIN BR. The population biology of bacterial viruses: why be temperate[J]. Theoretical Population Biology, 1984, 26(1): 93-117 DOI:10.1016/0040-5809(84)90026-1. |
| [72] | WANG G, OR D. A hydration-based biophysical index for the onset of soil microbial coexistence[J]. Scientific Reports, 2012, 2: 881 DOI:10.1038/srep00881. |
| [73] | WANG G, OR D. Hydration dynamics promote bacterial coexistence on rough surfaces[J]. The ISME Journal, 2013, 7(2): 395-404 DOI:10.1038/ismej.2012.115. |
| [74] | RUAN CJ, BORER B, RAMONEDA J, WANG G, JOHNSON DR. Evaporation-induced hydrodynamics control plasmid transfer during surface-associated microbial growth[J]. npj Biofilms and Microbiomes, 2023, 9: 58 DOI:10.1038/s41522-023-00428-x. |
| [75] | RUAN CJ, RAMONEDA J, CHEN GW, JOHNSON DR, WANG G. Evaporation-induced hydrodynamics promote conjugation-mediated plasmid transfer in microbial populations[J]. ISME Communications, 2021, 1: 54 DOI:10.1038/s43705-021-00057-5. |
| [76] | KOSKELLA B, BROCKHURST MA. Bacteria-phage coevolution as a driver of ecological and evolutionary processes in microbial communities[J]. FEMS Microbiology Reviews, 2014, 38(5): 916-931 DOI:10.1111/1574-6976.12072. |
| [77] | KRUPOVIC M, DOLJA VV, KOONIN EV. Origin of viruses: primordial replicators recruiting capsids from hosts[J]. Nature Reviews Microbiology, 2019, 17(7): 449-458 DOI:10.1038/s41579-019-0205-6. |
| [78] | PAEZ-ESPINO D, ELOE-FADROSH EA, PAVLOPOULOS GA, THOMAS AD, HUNTEMANN M, MIKHAILOVA N, RUBIN E, IVANOVA NN, KYRPIDES NC. Uncovering Earth's virome[J]. Nature, 2016, 536(7617): 425-430 DOI:10.1038/nature19094. |
| [79] | JANSSON JK, WU RN. Soil viral diversity, ecology and climate change[J]. Nature Reviews Microbiology, 2023, 21(5): 296-311 DOI:10.1038/s41579-022-00811-z. |
| [80] | WU RN, DAVISON MR, GAO YQ, NICORA CD, MCDERMOTT JE, BURNUM-JOHNSON KE, HOFMOCKEL KS, JANSSON JK. Moisture modulates soil reservoirs of active DNA and RNA viruses[J]. Communications Biology, 2021, 4: 992 DOI:10.1038/s42003-021-02514-2. |
| [81] | LEON-VELARDE CG, HAPPONEN L, PAJUNEN M, LESKINEN K, KROPINSKI AM, MATTINEN L, RAJTOR M, ZUR J, SMITH D, CHEN S, NAWAZ A, JOHNSON RP, ODUMERU JA, GRIFFITHS MW, SKURNIK M. Yersinia enterocolitica-specific infection by bacteriophages TG1 and ϕR1-RT is dependent on temperature-regulated expression of the phage host receptor OmpF[J]. Applied and Environmental Microbiology, 2016, 82(17): 5340-5353 DOI:10.1128/AEM.01594-16. |
| [82] | EGILMEZ HI, MOROZOV AY, CLOKIE MRJ, SHAN JY, LETAROV A, GALYOV EE. Temperature- dependent virus lifecycle choices may reveal and predict facets of the biology of opportunistic pathogenic bacteria[J]. Scientific Reports, 2018, 8: 9642 DOI:10.1038/s41598-018-27716-3. |
| [83] | HURST CJ, GERBA CP, CECH I. Effects of environmental variables and soil characteristics on virus survival in soil[J]. Applied and Environmental Microbiology, 1980, 40(6): 1067-1079 DOI:10.1128/aem.40.6.1067-1079.1980. |
| [84] | MA YY, LI EN, QI ZZ, LI H, WEI X, LIN WS, ZHAO RX, JIANG AM, YANG HY, YIN Z, YUAN J, ZHAO XN. Isolation and molecular characterisation of Achromobacter phage phiAxp-3, an N4-like bacteriophage[J]. Scientific Reports, 2016, 6: 24776 DOI:10.1038/srep24776. |
| [85] | SYKES IK, LANNING S, WILLIAMS ST. The effect of pH on soil actinophage[J]. Microbiology, 1981, 122(2): 271-280 DOI:10.1099/00221287-122-2-271. |
| [86] | SYKES IK, WILLIAMS ST. Interactions of actinophage and clays[J]. Journal of General Microbiology, 1978, 108(1): 97-102 DOI:10.1099/00221287-108-1-97. |
| [87] | KAPUSCINSKI RB, MITCHELL R. Processes controlling virus inactivation in coastal waters[J]. Water Research, 1980, 14(4): 363-371 DOI:10.1016/0043-1354(80)90084-6. |
| [88] | BALES RC, LI SM, MAGUIRE KM, YAHYA MT, GERBA CP. MS-2 and poliovirus transport in porous media: hydrophobic effects and chemical perturbations[J]. Water Resources Research, 1993, 29(4): 957-963 DOI:10.1029/92WR02986. |
| [89] | BALES RC, HINKLE SR, KROEGER TW, STOCKING K, GERBA CP. Bacteriophage adsorption during transport through porous media: chemical perturbations and reversibility[J]. Environmental Science & Technology, 1991, 25(12): 2088-2095. |
| [90] | BELLOU MI, SYNGOUNA VI, TSELEPI MA, KOKKINOS PA, PAPARRODOPOULOS SC, VANTARAKIS A, CHRYSIKOPOULOS CV. Interaction of human adenoviruses and coliphages with kaolinite and bentonite[J]. Science of the Total Environment, 2015, 517: 86-95 DOI:10.1016/j.scitotenv.2015.02.036. |
| [91] | KATZ A, PEÑA S, ALIMOVA A, GOTTLIEB P, XU M, BLOCK KA. Heteroaggregation of an enveloped bacteriophage with colloidal sediments and effect on virus viability[J]. Science of the Total Environment, 2018, 637/638: 104-111 DOI:10.1016/j.scitotenv.2018.04.425. |
| [92] | LIPSON SM, STOTZKY G. Effect of kaolinite on the specific infectivity of reovirus[J]. FEMS Microbiology Letters, 1986, 37(1): 83-88 DOI:10.1111/j.1574-6968.1986.tb01771.x. |
| [93] | VETTORI C, GALLORI E, STOTZKY G. Clay minerals protect bacteriophage PBS1 of Bacillus subtilis against inactivation and loss of transducing ability by UV radiation[J]. Canadian Journal of Microbiology, 2000, 46(8): 770-773 DOI:10.1139/w00-055. |
| [94] |
王光华. 掀开土壤生物"暗物质": 土壤病毒的神秘面纱[J]. 中国科学院院刊, 2017, 32(6): 575-584.
WANG GH. Lift mysterious veil of soil virus: 'dark matter' of soil biota[J]. Bulletin of Chinese Academy of Sciences, 2017, 32(6): 575-584 (in Chinese). |
| [95] | CLOKIE MRJ, MILLARD AD, LETAROV AV, HEAPHY S. Phages in nature[J]. Bacteriophage, 2011, 1(1): 31-45 DOI:10.4161/bact.1.1.14942. |
| [96] | SAUSSET R, PETIT MA, GABORIAU-ROUTHIAU V, de PAEPE M. New insights into intestinal phages[J]. Mucosal Immunology, 2020, 13(2): 205-215 DOI:10.1038/s41385-019-0250-5. |
| [97] | HERSKOWITZ I, HAGEN D. The lysis-lysogeny decision of phage λ: explicit programming and responsiveness[J]. Annual Review of Genetics, 1980, 14(1): 399-445 DOI:10.1146/annurev.ge.14.120180.002151. |
| [98] | MASLOV S, SNEPPEN K. Well-temperate phage: optimal bet-hedging against local environmental collapses[J]. Scientific Reports, 2015, 5: 10523 DOI:10.1038/srep10523. |
| [99] | LIANG XL, ZHANG YY, WOMMACK KE, WILHELM SW, DeBRUYN JM, SHERFY AC, ZHUANG J, RADOSEVICH M. Lysogenic reproductive strategies of viral communities vary with soil depth and are correlated with bacterial diversity[J]. Soil Biology and Biochemistry, 2020, 144: 107767 DOI:10.1016/j.soilbio.2020.107767. |
| [100] | WILLIAMSON KE, RADOSEVICH M, SMITH DW, WOMMACK KE. Incidence of lysogeny within temperate and extreme soil environments[J]. Environmental Microbiology, 2007, 9(10): 2563-2574 DOI:10.1111/j.1462-2920.2007.01374.x. |
| [101] | GHOSH D, ROY K, WILLIAMSON KE, SRINIVASIAH S, WOMMACK KE, RADOSEVICH M. Acyl-homoserine lactones can induce virus production in lysogenic bacteria: an alternative paradigm for prophage induction[J]. Applied and Environmental Microbiology, 2009, 75(22): 7142-7152 DOI:10.1128/AEM.00950-09. |
| [102] | CENENS W, MAKUMI A, GOVERS SK, LAVIGNE R, AERTSEN A. Viral transmission dynamics at single-cell resolution reveal transiently immune subpopulations caused by a carrier state association[J]. PLoS Genetics, 2015, 11(12): e1005770 DOI:10.1371/journal.pgen.1005770. |
| [103] | FEINER R, ARGOV T, RABINOVICH L, SIGAL N, BOROVOK I, HERSKOVITS AA. A new perspective on lysogeny: prophages as active regulatory switches of bacteria[J]. Nature Reviews Microbiology, 2015, 13(10): 641-650 DOI:10.1038/nrmicro3527. |
| [104] | ZHANG R, WEINBAUER MG, QIAN PY. Viruses and flagellates sustain apparent richness and reduce biomass accumulation of bacterioplankton in coastal marine waters[J]. Environmental Microbiology, 2007, 9(12): 3008-3018 DOI:10.1111/j.1462-2920.2007.01410.x. |
| [105] | BRAGA LPP, SPOR A, KOT W, BREUIL MC, HANSEN LH, SETUBAL JC, PHILIPPOT L. Impact of phages on soil bacterial communities and nitrogen availability under different assembly scenarios[J]. Microbiome, 2020, 8: 52 DOI:10.1186/s40168-020-00822-z. |
| [106] | PLEŠKA M, GUET CC. Effects of mutations in phage restriction sites during escape from restriction-modification[J]. Biology Letters, 2017, 13(12): 20170646 DOI:10.1098/rsbl.2017.0646. |
| [107] | FINERAN PC, BLOWER TR, FOULDS IJ, HUMPHREYS DP, LILLEY KS, SALMOND GP. The phage abortive infection system, ToxIN, functions as a protein-RNA toxin-antitoxin pair[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(3): 894-899. |
| [108] | DUPUIS M, VILLION M, MAGADÁN AH, MOINEAU S. CRISPR-Cas and restriction-modification systems are compatible and increase phage resistance[J]. Nature Communications, 2013, 4: 2087 DOI:10.1038/ncomms3087. |
| [109] | OFIR G, SOREK R. Contemporary phage biology: from classic models to new insights[J]. Cell, 2018, 172(6): 1260-1270 DOI:10.1016/j.cell.2017.10.045. |
| [110] | SECOR PR, SWEERE JM, MICHAELS LA, MALKOVSKIY AV, LAZZARESCHI D, KATZNELSON E, RAJADAS J, BIRNBAUM ME, ARRIGONI A, BRAUN KR, EVANKO SP, STEVENS DA, KAMINSKY W, SINGH PK, PARKS WC, BOLLYKY PL. Filamentous bacteriophage promote biofilm assembly and function[J]. Cell Host & Microbe, 2015, 18(5): 549-559. |
| [111] | SULLIVAN MB, LINDELL D, LEE JA, THOMPSON LR, BIELAWSKI JP, CHISHOLM SW. Prevalence and evolution of core photosystem II genes in marine cyanobacterial viruses and their hosts[J]. PLoS Biology, 2006, 4(8): e234 DOI:10.1371/journal.pbio.0040234. |
| [112] | GHOSH D, ROY K, WILLIAMSON KE, WHITE DC, WOMMACK KE, SUBLETTE KL, RADOSEVICH M. Prevalence of lysogeny among soil bacteria and presence of 16S rRNA and trzN genes in viral-community DNA[J]. Applied and Environmental Microbiology, 2008, 74(2): 495-502 DOI:10.1128/AEM.01435-07. |
| [113] | TZIPILEVICH E, HABUSHA M, BEN-YEHUDA S. Acquisition of phage sensitivity by bacteria through exchange of phage receptors[J]. Cell, 2017, 168(1/2): 186-199.e12. |
| [114] | DEDRICK RM, JACOBS-SERA D, BUSTAMANTE CAG, GARLENA RA, MAVRICH TN, POPE WH, REYES JCC, RUSSELL DA, ADAIR T, ALVEY R, ALFRED BONILLA J, BRICKER JS, BROWN BR, BYRNES D, CRESAWN SG, DAVIS WB, DICKSON LA, EDGINGTON NP, FINDLEY AM, GOLEBIEWSKA U, et al. Prophage-mediated defence against viral attack and viral counter-defence[J]. Nature Microbiology, 2017, 2(3): 16251 DOI:10.1038/nmicrobiol.2016.251. |
| [115] | WANG JY, MA J, CHENG Z, MENG X, YOU LL, WANG M, ZHANG XZ, WANG YL. A CRISPR evolutionary arms race: structural insights into viral anti-CRISPR/Cas responses[J]. Cell Research, 2016, 26(10): 1165-1168 DOI:10.1038/cr.2016.103. |
| [116] | ABEBE HM, SADOWSKY MJ, KINKLE BK, SCHMIDT EL. Lysogeny in Bradyrhizobium japonicum and its effect on soybean nodulation[J]. Applied and Environmental Microbiology, 1992, 58(10): 3360-3366 DOI:10.1128/aem.58.10.3360-3366.1992. |
| [117] | BOYD EF. Bacteriophage-encoded bacterial virulence factors and phage-pathogenicity island interactions//Bacteriophages, Part A[J]. Amsterdam: Elsevier, 2012: 91-118. |
| [118] | van HOUTE S, BUCKLING A, WESTRA ER. Evolutionary ecology of prokaryotic immune mechanisms[J]. Microbiology and Molecular Biology Reviews, 2016, 80(3): 745-763 DOI:10.1128/MMBR.00011-16. |
| [119] | ABEDON ST, LeJEUNE JT. Why bacteriophage encode exotoxins and other virulence factors[J]. Evolutionary Bioinformatics Online, 2007, 1: 97-110. |
| [120] | HOSSEINIDOUST Z, TUFENKJI N, van de VEN TGM. Formation of biofilms under phage predation: considerations concerning a biofilm increase[J]. Biofouling, 2013, 29(4): 457-468 DOI:10.1080/08927014.2013.779370. |
| [121] | SIMMONS EL, BOND MC, KOSKELLA B, DRESCHER K, BUCCI V, NADELL CD. Biofilm structure promotes coexistence of phage-resistant and phage-susceptible bacteria[J]. mSystems, 2020, 5(3): e00877-19. |
| [122] | McDOUGALD D, RICE SA, BARRAUD N, STEINBERG PD, KJELLEBERG S. Should we stay or should we go: mechanisms and ecological consequences for biofilm dispersal[J]. Nature Reviews Microbiology, 2012, 10(1): 39-50 DOI:10.1038/nrmicro2695. |
| [123] | MASLOV S, SNEPPEN K. Population cycles and species diversity in dynamic kill-the-winner model of microbial ecosystems[J]. Scientific Reports, 2017, 7: 39642 DOI:10.1038/srep39642. |
| [124] | SILVEIRA CB, ROHWER FL. Piggyback-the-winner in host-associated microbial communities[J]. npj Biofilms and Microbiomes, 2016, 2: 16010 DOI:10.1038/npjbiofilms.2016.10. |
| [125] | SCANLAN PD, HALL AR, BLACKSHIELDS G, FRIMAN VP, DAVIS MR, GOLDBERG JB, BUCKLING A. Coevolution with bacteriophages drives genome-wide host evolution and constrains the acquisition of abiotic-beneficial mutations[J]. Molecular Biology and Evolution, 2015, 32(6): 1425-1435 DOI:10.1093/molbev/msv032. |
| [126] | DEIRDRE SCANLAN P, BUCKLING A. Co-evolution with lytic phage selects for the mucoid phenotype of Pseudomonas fluorescens SBW25[J]. The ISME Journal, 2012, 6(6): 1148-1158 DOI:10.1038/ismej.2011.174. |
| [127] | LEAVITT A, YIRMIYA E, AMITAI G, LU A, GARB J, HERBST E, MOREHOUSE BR, HOBBS SJ, ANTINE SP, SUN ZJ, KRANZUSCH PJ, SOREK R. Viruses inhibit TIR gcADPR signalling to overcome bacterial defence[J]. Nature, 2022, 611(7935): 326-331 DOI:10.1038/s41586-022-05375-9. |
| [128] | AZAM AH, TANJI Y. Bacteriophage-host arm race: an update on the mechanism of phage resistance in bacteria and revenge of the phage with the perspective for phage therapy[J]. Applied Microbiology and Biotechnology, 2019, 103(5): 2121-2131 DOI:10.1007/s00253-019-09629-x. |
| [129] | HAMPTON HG, WATSON BNJ, FINERAN PC. The arms race between bacteria and their phage foes[J]. Nature, 2020, 577(7790): 327-336 DOI:10.1038/s41586-019-1894-8. |
| [130] |
夏蓉, 郑晓璇, 叶茂, 朱冬, 张辉, 朱春梧, 胡锋, 孙明明. 噬菌体对土壤碳氮元素循环转化影响的研究进展[J]. 土壤, 2021, 53(4): 661-671.
XIA R, ZHENG XX, YE M, ZHU D, ZHANG H, ZHU CW, HU F, SUN MM. Advances in effects of bacteriophages on transformation of carbon and nitrogen in soil[J]. Soils, 2021, 53(4): 661-671 (in Chinese). |
| [131] |
孙廷丽, 韩玉茹, 王青柏, 黎玉莲, 谢小保. 噬菌体作为指示病毒的应用研究进展[J]. 微生物学报, 2023, 63(7): 2511-2522.
SUN TL, HAN YR, WANG QB, LI YL, XIE XB. Research progress in the application of bacteriophages as indicator viruses[J]. Acta Microbiologica Sinica, 2023, 63(7): 2511-2522 (in Chinese). |
| [132] |
张晓文, 刘焱, 郭东毅, 董海良, 王海纳. 黏土矿物对病毒生物地球化学作用的影响探讨与展望[J]. 科学通报, 2023, 68(1): 87-99.
DOI:10.3969/j.issn.2096-1693.2023.01.006 ZHANG XW, LIU Y, GUO DY, DONG HL, WANG HN. Effects of clay minerals on biogeochemical significance of viruses: a review[J]. Chinese Science Bulletin, 2023, 68(1): 87-99 (in Chinese). |
| [133] |
高瑞川, 胡敏, 李芳柏, 陈冠虹, 方利平. 土壤噬菌体的研究进展及生态功能解析[J]. 华南农业大学学报, 2022, 43(5): 1-11.
GAO RC, HU M, LI FB, CHEN GH, FANG LP. Research progress and ecological function of phages in soil[J]. Journal of South China Agricultural University, 2022, 43(5): 1-11 (in Chinese). |
| [134] |
韩丽丽, 曹苗苗, 毕丽, 张丽梅, 贺纪正. 土壤病毒的研究进展与应用前景[J]. 科技导报, 2022, 40(3): 75-86.
HAN LL, CAO MM, BI L, ZHANG LM, HE JZ. Research advances and application prospect of soil viruses[J]. Science & Technology Review, 2022, 40(3): 75-86 (in Chinese). |
| [135] | HAN LL, YU DT, BI L, DU S, SILVEIRA C, COBIÁN GÜEMES AG, ZHANG LM, HE JZ, ROHWER F. Distribution of soil viruses across China and their potential role in phosphorous metabolism[J]. Environmental Microbiome, 2022, 17(1): 1-11 DOI:10.1186/s40793-021-00396-9. |
| [136] |
李保国, 周虎, 王钢, 刘刚, 高伟达, 朱堃, 陈冲. 探索"透明"土壤体: 土壤孔隙学的时代已经启航[J]. 土壤学报, 2023, 60(5): 1221-1230.
LI BG, ZHOU H, WANG G, LIU G, GAO WD, ZHU K, CHEN C. Explore the "transparent" soils: soilporelogy has sailed[J]. Acta Pedologica Sinica, 2023, 60(5): 1221-1230 (in Chinese). |
| [137] | ALLEN B, WILLNER D, OECHEL WC, LIPSON D. Top-down control of microbial activity and biomass in an Arctic soil ecosystem[J]. Environmental Microbiology, 2010, 12(3): 642-648 DOI:10.1111/j.1462-2920.2009.02104.x. |
| [138] | ALBRIGHT MBN, GALLEGOS-GRAVES LV, FEESER KL, MONTOYA K, EMERSON JB, SHAKYA M, DUNBAR J. Experimental evidence for the impact of soil viruses on carbon cycling during surface plant litter decomposition[J]. ISME Communications, 2022, 2: 24 DOI:10.1038/s43705-022-00109-4. |
| [139] | MITARAI N, BROWN S, SNEPPEN K. Population dynamics of phage and bacteria in spatially structured habitats using phage λ and Escherichia coli[J]. Journal of Bacteriology, 2016, 198(12): 1783-1793 DOI:10.1128/JB.00965-15. |
| [140] | FUHRMAN JA. Marine viruses and their biogeochemical and ecological effects[J]. Nature, 1999, 399(6736): 541-548 DOI:10.1038/21119. |
| [141] | GUNINA A, KUZYAKOV Y. Sugars in soil and sweets for microorganisms: review of origin, content, composition and fate[J]. Soil Biology and Biochemistry, 2015, 90: 87-100 DOI:10.1016/j.soilbio.2015.07.021. |
| [142] | MATTESON AR, LOAR SN, PICKMERE S, DEBRUYN JM, ELLWOOD MJ, BOYD PW, HUTCHINS DA, WILHELM SW. Production of viruses during a spring phytoplankton bloom in the south Pacific Ocean near of New Zealand[J]. FEMS Microbiology Ecology, 2012, 79(3): 709-719 DOI:10.1111/j.1574-6941.2011.01251.x. |
| [143] | WINTER C, BOUVIER T, WEINBAUER MG, THINGSTAD TF. Trade-offs between competition and defense specialists among unicellular planktonic organisms: the "killing the winner" hypothesis revisited[J]. Microbiology and Molecular Biology Reviews, 2010, 74(1): 42-57 DOI:10.1128/MMBR.00034-09. |
| [144] | ALI FS, LOYNACHAN TE, HAMMAD AMM, AHARCHI Y. Polyvirulent rhizobiophage from a soybean rhizosphere soil[J]. Soil Biology and Biochemistry, 1998, 30(14): 2171-2175 DOI:10.1016/S0038-0717(98)00048-0. |
| [145] | SHARMA RS, MOHMMED A, BABU CR. Diversity among rhizobiophages from rhizospheres of legumes inhabiting three ecogeographical regions of India[J]. Soil Biology and Biochemistry, 2002, 34(7): 965-973 DOI:10.1016/S0038-0717(02)00030-5. |
| [146] | EMERSON JB, ROUX S, BRUM JR, BOLDUC B, WOODCROFT BJ, BIN JANG H, SINGLETON CM, SOLDEN LM, NAAS AE, BOYD JA, HODGKINS SB, WILSON RM, TRUBL G, LI CS, FROLKING S, POPE PB, WRIGHTON KC, CRILL PM, CHANTON JP, SALESKA SR, et al. Host-linked soil viral ecology along a permafrost thaw gradient[J]. Nature Microbiology, 2018, 3(8): 870-880 DOI:10.1038/s41564-018-0190-y. |
| [147] | JIN M, GUO X, ZHANG R, QU W, GAO BL, ZENG RY. Diversities and potential biogeochemical impacts of mangrove soil viruses[J]. Microbiome, 2019, 7: 1-15 DOI:10.1186/s40168-018-0604-3. |
| [148] | DOMINGUES S, NIELSEN KM. Horizontal gene transfer: uptake of extracellular DNA by bacteria[M]//Reference Module in Biomedical Sciences. Amsterdam: Elsevier, 2016. |
| [149] | PENADÉS JR, CHEN J, QUILES-PUCHALT N, CARPENA N, NOVICK RP. Bacteriophage-mediated spread of bacterial virulence genes[J]. Current Opinion in Microbiology, 2015, 23(4): 171-178. |
| [150] | YIN XM, STOTZKY G. Gene transfer among bacteria in natural environments[M]//Advances in Applied Microbiology. Amsterdam: Elsevier, 1997: 153-212. |
| [151] | SUBIRATS J, SÀNCHEZ-MELSIÓ A, BORREGO CM, BALCÁZAR JL, SIMONET P. Metagenomic analysis reveals that bacteriophages are reservoirs of antibiotic resistance genes[J]. International Journal of Antimicrobial Agents, 2016, 48(2): 163-167 DOI:10.1016/j.ijantimicag.2016.04.028. |
| [152] | DEBROAS D, SIGURET C. Viruses as key reservoirs of antibiotic resistance genes in the environment[J]. The ISME Journal, 2019, 13(11): 2856-2867 DOI:10.1038/s41396-019-0478-9. |
| [153] | TOUCHON M, MOURA DE SOUSA JA, ROCHA EP. Embracing the enemy: the diversification of microbial gene repertoires by phage-mediated horizontal gene transfer[J]. Current Opinion in Microbiology, 2017, 38: 66-73 DOI:10.1016/j.mib.2017.04.010. |
| [154] | TAYLOR VL, FITZPATRICK AD, ISLAM Z, MAXWELL KL. The diverse impacts of phage morons on bacterial fitness and virulence[J]. Advances in Virus Research, 2019, 103: 1-31. |
| [155] | JIANG SC, PAUL JH. Gene transfer by transduction in the marine environment[J]. Applied and Environmental Microbiology, 1998, 64(8): 2780-2787 DOI:10.1128/AEM.64.8.2780-2787.1998. |
| [156] | LIU K, SUN MM, YE M, CHAO HZ, ZHAO YC, XIA B, JIAO WT, FENG YF, ZHENG XX, LIU MQ, JIAO JG, HU F. Coexistence and association between heavy metals, tetracycline and corresponding resistance genes in vermicomposts originating from different substrates[J]. Environmental Pollution, 2019, 244: 28-37 DOI:10.1016/j.envpol.2018.10.022. |
| [157] | WANG XF, WEI Z, YANG KM, WANG JN, JOUSSET A, XU YC, SHEN QR, FRIMAN VP. Phage combination therapies for bacterial wilt disease in tomato[J]. Nature Biotechnology, 2019, 37(12): 1513-1520 DOI:10.1038/s41587-019-0328-3. |
| [158] |
韦中, 王孝芳, 马迎飞, 叶茂, 徐阳春, 沈其荣, 朱永官. 噬菌体微生态疗法与一体化健康: 现状、挑战与机遇[J]. 中国科学(生命科学), 2023, 53(5): 698-710.
WEI Z, WANG XF, MA YF, YE M, XU YC, SHEN QR, ZHU YG. Phage therapy for One Health approach: current status, challenges and opportunities[J]. Scientia Sinica (Vitae), 2023, 53(5): 698-710 (in Chinese). |
| [159] | ASKORA A, KAWASAKI T, FUJIE M, YAMADA T. Insights into the diversity of φRSM phages infecting strains of the phytopathogen Ralstonia solanacearum complex: regulation and evolution[J]. Molecular Genetics and Genomics, 2014, 289(4): 589-598 DOI:10.1007/s00438-014-0835-3. |
| [160] | FUJIWARA A, FUJISAWA M, HAMASAKI R, KAWASAKI T, FUJIE M, YAMADA T. Biocontrol of Ralstonia solanacearum by treatment with lytic bacteriophages[J]. Applied and Environmental Microbiology, 2011, 77(12): 4155-4162 DOI:10.1128/AEM.02847-10. |
| [161] | FRAMPTON RA, TAYLOR C, HOLGUÍN MORENO AV, VISNOVSKY SB, PETTY NK, PITMAN AR, FINERAN PC. Identification of bacteriophages for biocontrol of the kiwifruit canker phytopathogen Pseudomonas syringae pv. actinidiae[J]. Applied and Environmental Microbiology, 2014, 80(7): 2216-2228 DOI:10.1128/AEM.00062-14. |
| [162] | CHAE JC, HUNG NB, YU SM, LEE HK, LEE YH. Diversity of bacteriophages infecting Xanthomonas oryzae pv. oryzae in paddy fields and its potential to control bacterial leaf blight of rice[J]. Journal of Microbiology and Biotechnology, 2014, 24(6): 740-747 DOI:10.4014/jmb.1402.02013. |
| [163] | YU PF, MATHIEU J, LU GW, GABIATTI N, ALVAREZ PJ. Control of antibiotic-resistant bacteria in activated sludge using polyvalent phages in conjunction with a production host[J]. Environmental Science & Technology Letters, 2017, 4(4): 137-142 DOI:10.3969/j.issn.1673-288X.2017.04.046. |
| [164] | YE M, SUN MM, ZHAO YC, JIAO WT, XIA B, LIU MQ, FENG YF, ZHANG ZY, HUANG D, HUANG R, WAN JZ, DU RJ, JIANG X, HU F. Targeted inactivation of antibiotic-resistant Escherichia coli and Pseudomonas aeruginosa in a soil-lettuce system by combined polyvalent bacteriophage and biochar treatment[J]. Environmental Pollution, 2018, 241: 978-987 DOI:10.1016/j.envpol.2018.04.070. |
| [165] | YE M, SUN MM, HUANG D, ZHANG ZY, ZHANG H, ZHANG ST, HU F, JIANG X, JIAO WT. A review of bacteriophage therapy for pathogenic bacteria inactivation in the soil environment[J]. Environment International, 2019, 129: 488-496 DOI:10.1016/j.envint.2019.05.062. |
| [166] | ZHAO YC, YE M, ZHANG XT, SUN MM, ZHANG ZY, CHAO HZ, HUANG D, WAN JZ, ZHANG ST, JIANG X, SUN DW, YUAN YL, HU F. Comparing polyvalent bacteriophage and bacteriophage cocktails for controlling antibiotic-resistant bacteria in soil-plant system[J]. Science of the Total Environment, 2019, 657: 918-925 DOI:10.1016/j.scitotenv.2018.11.457. |
 2024, Vol. 64
2024, Vol. 64




