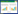中国科学院微生物研究所,中国微生物学会
文章信息
- 金佳凡, 舒国靖, 朱四东, 杨季芳, 陈吉刚. 2024
- JIN Jiafan, SHU Guojing, ZHU Sidong, YANG Jifang, CHEN Jigang.
- 阿拉伯海假交替单胞菌N1230-9两个甲基受体趋化蛋白的功能鉴定
- Functions of two methyl-accepting chemotaxis proteins of Pseudoalteromonas arabiensis N1230-9
- 微生物学报, 64(5): 1641-1653
- Acta Microbiologica Sinica, 64(5): 1641-1653
-
文章历史
- 收稿日期:2023-12-19
- 网络出版日期:2024-02-02
细菌趋化性(chemotaxis)是一种感应环境中化学物质浓度变化而调控自身运动的基本生理机制,其在细菌趋近有利环境而避离不利环境过程中发挥关键作用[1]。趋化响应受化学感觉信号通路控制,该通路广泛分布于细菌和古生菌中[2]。趋化信号通路,始于化学效应物即配体与化学受体的特异性结合,化学受体在趋化信号转导系统中,又称甲基受体趋化蛋白(methyl-accepting chemotaxis protein, MCP)。经典的MCP由配体结合结构域(ligand binding domain, LBD)和组氨酸激酶、腺苷酸环化酶、甲基接受趋化蛋白、磷酸酶(histidine kinases, adenylyl cyclases, methyl-accepting chemotaxis proteins, phosphatases, HAMP)及胞质信号转换结构域(cytoplasmic signaling domain, SD)组成[3]。化学效应物与LBD结合,引起LBD构象改变并产生信号刺激,这种刺激通过细胞质HAMP结构域传递给偶联蛋白CheW和组氨酸激酶CheA,并改变CheA的自磷酸化活性;磷酸化的CheA将其磷酸基团传递给响应调节蛋白CheY,磷酸化的CheY与鞭毛马达蛋白结合并调节鞭毛马达的运动,最终调节细菌的运动方向导致趋化响应[4-5]。除了介导趋化,化学感觉信号通路还参与多种生物过程的调控,如生物膜的形成、胞外多糖产生、鞭毛生物合成和致病性等[6-7]。
由于海洋细菌的趋化性微观行为并不立即显现出来,因此海洋细菌的趋化性长期被研究者所忽视。细菌趋化性不仅在海洋环境中广泛存在,而且在海洋生态学和生物地球化学中发挥着非常关键的作用[8-10]。海洋细菌的进化发生于复杂而多变且营养匮乏的异质环境,因此它们所呈现出的趋化性可能明显不同于处于特定生态位的肠道细菌或陆源细菌。海洋环境拥有多样的微尺度化学生态位,其中的大分子和营养物质可通过聚集成颗粒的形式进行积累,导致营养丰富的颗粒或点源微环境在营养缺乏的微环境中不断形成[11-14]。然而,由于上述微环境通常是短暂且容易耗尽的,因此海洋细菌对营养源的有效趋化对于它们的生存至关重要[8, 15]。
作为海洋环境的特有种群,假交替单胞菌(Pseudoalteromonas)的适应机制和存活策略具有多样性和有效性,这使得它们能够在各种海洋环境中生存[16-20]。对已测序的假交替单胞菌基因组分析表明,该属成员普遍拥有数量众多的MCP编码基因,这表明假交替单胞菌可能响应环境中的多种信号分子。然而,目前尚不清楚假交替单胞菌所拥有的MCP具体响应哪些化学效应物。为此,本研究选择一株阿拉伯海假交替单胞菌(Pseudoalteromonas arabiensis) N1230-9作为代表菌株,通过实验深入探讨2个具有同一结构域的甲基受体趋化蛋白与菌株碳源趋化间的关联。
1 材料与方法 1.1 材料 1.1.1 菌株和质粒阿拉伯海假交替单胞菌(Pseudoalteromonas arabiensis) N1230-9为本实验室成员在参加大洋54航次科考期间,从太平洋表层海水(178.71°W, 12.51°N)中分离获得的一株具有几丁质降解能力的菌株[21]。该菌株以甘油菌的形式保存于实验室−80 ℃超低温冰箱。本研究所使用的其他菌株和质粒如表 1所示。
| Strains and plasmids | Genotype/Relevant characteristics | Source |
| Escherichia coli strains | ||
| DH5α | F−Φ80lacZΔM15Δ(lacZYA-argF) U169 recA1 endA1 hsdR17 (rK−, mK+) phoA supE44 λ− thi-1 gyrA96 relA1 | Invitrogen |
| WM3064 | Donor strain for conjugation, diaminopimelate (DAP) auxotrophic | [22] |
| Pseudoalteromonas arabiensis strains | ||
| N1230-9 | Wild-type strain isolated from pacific seawater | This study |
| Δwoc28264 | N1230-9 with the chromosomal woc28264 gene deleted | This study |
| Δwoc27036 | N1230-9 with the chromosomal woc27036 gene deleted | This study |
| Δwoc28264-C | woc28264 cloned into Δwoc28264 | This study |
| Δwoc27036-C | woc27036 cloned into Δwoc27036 | This study |
| Plasmids | ||
| pK18mobsacB-Ery | KanR, EryR, suicide vector for gene knockout | [22] |
| pBBR1MCS-Ery | EryR, broad-host-range mobilizable plasmid | [22] |
| pK18mobSacB-Ery-woc28264 | pK18mobsacb-Ery with 679 bp upstream fragment and 646 bp downstream fragment of woc28264 | This study |
| pK18mobSacB-Ery-woc27036 | pK18mobsacb-Ery with 713 bp upstream fragment and 712 bp downstream fragment of woc27036 | This study |
| pBBR1MCS-Ery-woc28264 | EryR, KanR; pBBR1Ery containing woc28264 and its own promoter | This study |
| pBBR1MCS-Ery-woc27036 | EryR, KanR; pBBR1Ery containing woc27036 and its own promoter | This study |
| KanR: Kanamycin resistance; EryR: Erythromycin resistance. | ||
1.1.2 引物
用于菌株N1230-9基因缺失突变体构建和DNA测序的引物如表 2所示。
| Primer names | Direction (5′→3′) | Restriction enzyme cutting site |
| woc27036-up-F | CGCGTCGACGTTGGTGAGCGTAGTAGTGATTGGT | Sal I |
| woc27036-up-R | AGGTTGGTCTTACTGGCATCATCGAGCGATAACCGTGATACTTCATCATTACC | |
| woc27036-dn-F | GGTAATGATGAAGTATCACGGTTATCGCTCGATGATGCCAGTAAGACCAACCT | |
| woc27036-dn-R | CGCGGATCCTTGAGTATTTGATAGGGATTGCTCTGTT | BamH I |
| woc27036-wS | CCTATGAGCATGACGAAGGAACATACAT | |
| woc27036-wA | GCTGTAGCCACTTGAGTTGAACTGT | |
| woc27036-dS | GGAGAAGGTGACTTAACTCAGCAACT | |
| woc27036-dA | CGAGCTTGAAGCGGTTTACAACATC | |
| woc28264-up-F | AACTGCAGCTTCTTGCCCTTCTTCCACCTTTA | Pst I |
| woc28264-up-R | CTTCCATTGACGCTGCCGACATCGGACTTGTGATACTGCGAGATACC | |
| woc28264-dn-F | GGTATCTCGCAGTATCACAAGTCCGATGTCGGCAGCGTCAATGGAAG | |
| woc28264-dn-R | CGCGGATCCGCATATAAGAGGTTGCTACATCAGTATCAC | BamH I |
| woc28264-wS | AGGCGAATTTGATGCGACTGTGTAT | |
| woc28264-wA | CTTCCATTGACGCTGCCGACAT | |
| woc28264-dS | TTGCCTCGGTGCTTGCTGTTG | |
| woc28264-dA | AAGTGTGACCTAACCTAAGCCTTATAACG | |
| woc28264C-F | CCGCTCGAGATGGCGCTATCAATTCAGCAA | Xho I |
| woc28264C-R | CCGGAATTCCCTAACCTAAGCCTTATAACGGCT | EcoR I |
| woc27036C-F | CCGCTCGAGGAGCATGACGAAGGAACATAC | Xho I |
| woc27036C-R | CCGGAATTCTTGTCGTTAGTCGAGCTTGAAGCG | EcoR I |
| pK18-F | ATTCCGCTGGCAGCTTAAG | |
| PK18-R | GGTAACGCCAGGGTTTTCC | |
| pBBR1MCS-F | TCGTTAAATAGCCGCTTATG | |
| pBBR1MCS-R | AATTTCACACAGGAAACAGC | |
| F indicates the forward primer and R indicates the reverse primer. Underlined and italicized sequences indicate restriction enzymes. | ||
1.1.3 培养基、试剂和菌株培养条件
基础培养基(minimal medium, MM):取适量体积的人工海水(0.4 mol/L NaCl、0.1 mol/L MgSO4·7H2O、0.02 mol/L KCl和0.02 mol/L CaCl2·2H2O),加入等体积的蒸馏水,1×105 Pa高压灭菌处理后,加入经过滤除菌的50 mmol/L HEPES缓冲液(pH 7.5)、19 mmol/L NH4Cl、0.33 mmol/L K2HPO4·3H2O、0.01 mmol/L FeSO4·7H2O和适宜浓度的无菌碳源。如配制MM软琼脂培养基则在MM培养基中加入0.25%终浓度琼脂粉。
PCR扩增试剂盒、DNA回收试剂盒,康为世纪生物科技股份有限公司;T4 DNA连接酶、BamH Ⅰ、EcoR I、Pst Ⅰ、Sal Ⅰ和Xho I,宝生物工程(大连)有限公司。
E. coli采用LB培养基培养,培养温度为37 ℃;P. arabiensis N1230-9及其基因缺失突变体采用Marine Broth/Agar 2216培养基(BD Difco公司,以下简称2216培养基)培养,培养温度为25 ℃;E. coli WM3064使用含0.3 mmol/L终浓度的2,6-二氨基庚二酸(2,6-diamino-pimelicacid, DAP) LB (LB-DAP)培养基[22]培养,培养温度为37 ℃;菌株接合实验使用1/2 SW-LB-DAP固体培养基[22]。二次同源重组菌株筛选使用含15%终浓度蔗糖的2216-蔗糖培养基[22]。根据实验需求,培养基中添加15 μg/mL的红霉素。
1.2 MCP编码基因及其结构域分析菌株P. arabiensis N1230-9全基因组已完成测序(GenBank登录号:CP090419.1 & CP090420.1)[21]。基于NCBI和JGI数据库[23]的基因组注释结果,获得菌株N1230-9的MCP编码基因。利用InterPro数据库(https://www.ebi.ac.uk/interpro/)[24]在线预测MCP结构域。使用MAFFT软件(http://mafft.cbrc.jp/alignment/server)进行结构域氨基酸序列同源比对。
1.3 野生菌株碳源利用能力检测挑取单菌落接种至2216培养基中,25 ℃、180 r/min培养6−8 h至细菌对数生长期中期(OD600约为0.5)。取细菌培养液,4 500 r/min离心5 min收集菌体。菌体用无菌人工海水离心洗涤2次后,再用无菌人工海水将细胞浓度调整至OD600为0.5。取菌液按1:100 (体积比)比例接种至新鲜MM培养基中振荡培养,并在培养过程中定时取样,用分光光度法测定培养物的OD600值。每个实验样本均设置3个生物学重复。MM培养基中添加的碳源为L-阿拉伯糖、海藻糖、麦芽糖、乳糖、蔗糖、D-果糖、甘露糖、甘露醇、D-葡萄糖、N-乙酰氨基葡萄糖、L-苹果酸、D-半乳糖、N-乙酰氨基半乳糖、D-山梨醇、L-鼠李糖、L-岩藻糖、D-核糖、D-木糖、柠檬酸、琥珀酸、乙酸钠、丙酸钠和丙酮酸钠这23种碳源中的任意一种,添加终浓度均为10 mmol/L。
1.4 野生菌株碳源趋化能力检测挑取单菌落接种于2216培养基中,25 ℃、180 r/min培养12−16 h。次日,取细菌培养液按1:100 (体积比)比例接种至新鲜2216培养基中,继续培养6−8 h至细菌对数生长期中期,3 800 r/min离心5 min收集菌体。菌体用无菌人工海水离心洗涤2次后,用适量无菌人工海水将细胞浓度调整至OD600为0.6。取5 μL菌液接种至含有不同碳源的MM软琼脂平板中央,置于25 ℃恒温培养箱中培养,并在培养后的第18小时测量趋化圈直径。MM软琼脂培养基的碳源添加量均为0.5 mmol/L,以不添加任何碳源的MM软琼脂培养基作为阴性对照组。每个实验样本均设置3个生物学重复。
1.5 基因缺失突变体及回补菌株的构建基因敲除实验使用Wang等[22]建立的假交替单胞菌基因敲除系统。以woc27036基因敲除为例,简要描述基因敲除步骤。使用两对引物(woc27036-up-F/woc27036-up-R和woc27036-dn-F/woc27036-dn-R)扩增靶区的上游片段(713 bp)和下游片段(712 bp),继而利用引物对(woc27036-up-F和woc27036-dn-R)进行融合PCR,获得上游片段和下游片段的融合片段。将融合片段用Sal I/BamH I双酶切后亚克隆至自杀质粒pK18mobsacB-Ery中获得重组质粒pK18mobsacB-Ery-woc27036。将重组质粒转化至E. coli WM3064中,并利用引物对pK18-F/pK18-R进行DNA测序验证。通过属间接合方式将重组自杀质粒从WM3064转移至菌株N1230-9中,基于同源重组的两次交换原理,实现靶基因的无痕敲除[22]。以第1次交换成功菌株染色体中带有的Ery抗性基因筛选标识,利用含Ery的2216培养基筛选发生一次交换的克隆,并用woc27036-dS/woc27036-wA引物对筛选的克隆进行PCR验证。以第2次交换成功菌株对蔗糖的敏感性作为筛选标识,利用2216-蔗糖培养基筛选基因缺失菌株,并利用3对引物对(woc27036-dS/woc27036-wA、woc27036-wS/woc27036-wA和woc27036-dS/woc27036-dA)对筛选的基因缺失突变体进行PCR验证。
基因回补实验使用宽宿主范围质粒pBBR1MCS-Ery[22]。以菌株N1230-9基因组DNA为模板,分别利用引物对woc28264C-F/woc28264C-R和woc27036C-F/woc27036C-R进行PCR扩增,获得含有自身启动子的woc28264基因片段和woc27036基因片段,并分别将其亚克隆至质粒pBBR1MCS-Ery中,获得重组质粒pBBR1MCS-Ery-woc28264和pBBR1MCS-Ery-woc27036。将2个重组质粒分别转入WM3064中,继而使用引物对pBBR1MCS-F/pBBR1MCS-R进行DNA测序验证后,通过接合方式[22]分别将其转移至Δwoc28264和Δwoc27036中,获得基因回补菌株Δwoc28264-C和Δwoc27036-C。
1.6 基因缺失突变体碳源趋化能力分析按照1.4中的实验步骤,测定基因缺失突变体和基因回补菌株的碳源趋化能力。以野生菌株为阳性对照菌株,且每个实验组至少设置3个生物学重复。趋化圈记录时间为培养后的第18小时。以基因缺失突变体或基因回补菌株的趋化圈直径与野生菌株的趋化圈直径的百分比为纵坐标作图。
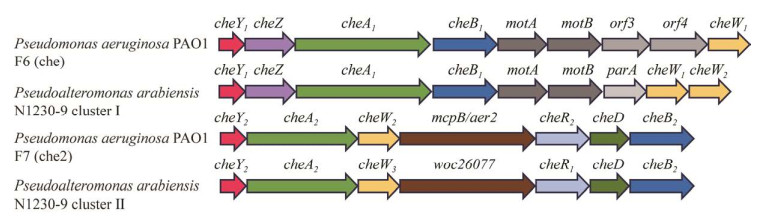
2 结果与分析 2.1 菌株N1230-9趋化受体编码基因菌株N1230-9基因组大小4 627 470 bp,G+C含量为40.85%,共编码4 202个蛋白[21]。基因组注释结果显示,菌株N1230-9基因组共编码28个MCP (图 1)。InterPro数据库预测结果显示,28个MCP呈现出3种结构特征,包括有跨膜区和LBD、无跨膜区但有LBD,以及有跨膜区但无LBD (图 1)。此外,LBD结构域类型也呈现出多样性,不但包括可被InterPro数据库预测的8个类型结构域(4HB、HBM、sCache、dCache、Tar、PAS、PDC1和PDC2),还包括未能被数据库预测的潜在LBD结构域(“?”) (图 1)。在上述含有LBD结构域的MCP中,6个MCP因缺失HAMP结构域(图 1),无法将感应信号传递给下游的信号转导蛋白,因此推测这6个MCP是“无功能性”甲基受体趋化蛋白。
|
| 图 1 菌株Pseudoalteromonas arabiensis N1230-9化学感受器的结构域展示 Figure 1 Domains display of chemoreceptor of Pseudoalteromonas arabiensis N1230-9. Cache: Calcium channels and chemotaxis receptors; HBM: Helical bimodular; 4HB: Four helix bundle; PDC: PhoQ/DcuS/CitA sensor domains; PAS: Per-Arnt-Sim. Chemoreceptors are present in higher oligomeric states in vivo but are shown as monomers for simplicity. WOC numbers mean GenBank protein ID. |
2.2 菌株N1230-9信号传导蛋白编码基因
菌株N1230-9含有19个信号传导蛋白编码基因(che),包括1个cheD、2个cheR、2个cheB、1个cheA、3个cheW、2个cheV、6个cheY、1个cheZ和1个cheX。这19个che基因中,12个che基因形成2个基因簇(cluster Ⅰ、cluster Ⅱ,图 2),而其他che基因则分散在基因组中。进一步比较分析发现,菌株N1230-9的2个che基因簇与铜绿假单胞菌(Pseudomonas aeruginosa) PAO1的2个che基因簇中的che基因组成和排布方式非常相似[25] (图 2)。目前,已经证实P. aeruginosa PAO1的cluster I和cluster V形成具有趋化功能的F6型通路(che),而cluster Ⅱ形成的F7型(che2)通路的功能尚不清楚[25]。以此推断,菌株N1230-9中与菌株P. aeruginosa PAO1的2个cluster (Ⅰ和Ⅱ)相对应的cluster (Ⅰ和Ⅱ)也发挥类似功能。此外,cluster Ⅱ中的woc26077所处位置对应P. aeruginosa PAO1 cluster Ⅱ中的趋氧受体编码基因mcpB/aer2。同源性比较分析发现,WOC26077是菌株所有编码蛋白中与McpB氨基酸序列一致性最高的蛋白(56%),因此推测WOC26077可能是菌株1230-9的趋氧受体。
2.3 菌株N1230-9对23种碳源的趋化性
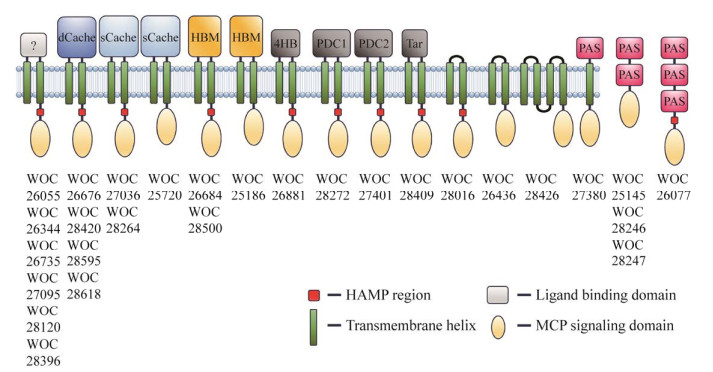
为了分析菌株N1230-9的碳源利用能力与碳源趋化性之间的联系,本研究首先测定了该菌株对23种碳源的利用能力。碳源利用能力实验结果显示,菌株N1230-9能以L-阿拉伯糖、L-岩藻糖、海藻糖、麦芽糖、蔗糖、琥珀酸、D-葡萄糖、N-乙酰氨基葡萄糖、L-苹果酸、N-乙酰氨基半乳糖、乙酸钠、丙酸钠或丙酮酸钠为唯一碳源生长,而不能以甘露糖、甘露醇、乳糖、D-果糖、D-半乳糖、D-山梨醇、L-鼠李糖、D-核糖、D-木糖或柠檬酸为唯一碳源生长。趋化实验结果显示,菌株N1230-9不但对海藻糖、麦芽糖、蔗糖、N-乙酰氨基葡萄糖、L-苹果酸、琥珀酸、乙酸钠、丙酸钠和丙酮酸钠9种可作为唯一碳源支持细菌生长的碳源具有趋化性,且对柠檬酸这1种不能作为唯一碳源支持菌株生长的碳源具有趋化能力,而对其他13种碳源均无趋化能力(图 3)。此外,趋化实验结果还显示,菌株N1230-9对柠檬酸和琥珀酸的趋化能力最强,而对丙酮酸钠的趋化能力最弱(图 3)。
|
| 图 3 泳动平板法检测Pseudoalteromonas arabiensis N1230-9对23种碳源的趋化性 Figure 3 The chemotaxis phenotype of Pseudoalteromonas arabiensis N1230-9 to 23 carbon sources in swimming plate assay. L-Mla: L-malic acid; Mal: Maltose; Sua: Succinic acid; Ca: Citric acid; Tre: Trehalose; Scr: Sucrose; GlcNAc: N-acetylglucosamine; Sp: Sodium propionate; Soa: Sodium acetate; Sop: Sodium pyruvate; Man: Mannose; Mtl: Mannitol; GalNAc: N-acetylgalactosamine; Las: Lactose; D-Glu: D-gluconate; D-Fru: D-fructose; D-Gal: D-galactose; D-Sor: D-sorbitol; D-Rbs: D-ribose; D-Xyl: D-xylose; L-Rha: L-rhamnose; L-Fuc: L-fucose; L-Ara: L-arabinose; NC: Negative control. |
2.4 基因缺失突变体对碳源的趋化响应
已有结果表明,某些细菌主要利用含sCache型LBD的MCP感应环境中的碳源[26]。基于此,本研究以含有sCache型LBD,同时具有HAMP结构域的2个MCP编码基因为靶标基因,利用同源重组技术分别构建了woc28264缺失突变体Δwoc28264和woc27036缺失突变体Δwoc27036 (图 4A、4B),继而检测了这2个突变体的碳源趋化能力。趋化实验结果显示,2个突变体对各种碳源的趋化能力既存在相同点也存在不同之处。相同的是,2个突变体对乙酸钠、丙酸钠、丙酮酸钠和麦芽糖的趋化能力相对于野生菌株均没有发生变化,且2个突变体对N-乙酰氨基葡萄糖和海藻糖的趋化能力均较野生菌株明显减弱(图 5A)。不同的是,Δwoc28264对L-苹果酸和蔗糖的趋化能力较野生菌株明显减弱,而Δwoc27036对这两种碳源的趋化能力与野生菌株的没有明显差异(图 5A)。此外,Δwoc27036对柠檬酸和琥珀酸的趋化能力较野生菌株明显减弱,而菌株Δwoc28264对柠檬酸的趋化能力则强于野生菌株,且对琥珀酸的趋化能力与野生菌株的基本相同(图 5A)。对基因回补菌株的趋化性检测结果显示,Δwoc28264-C和Δwoc27036-C对10种碳源的趋化能力与野生菌株无明显差异(图 5B),说明靶基因的敲除没有产生极性效应。
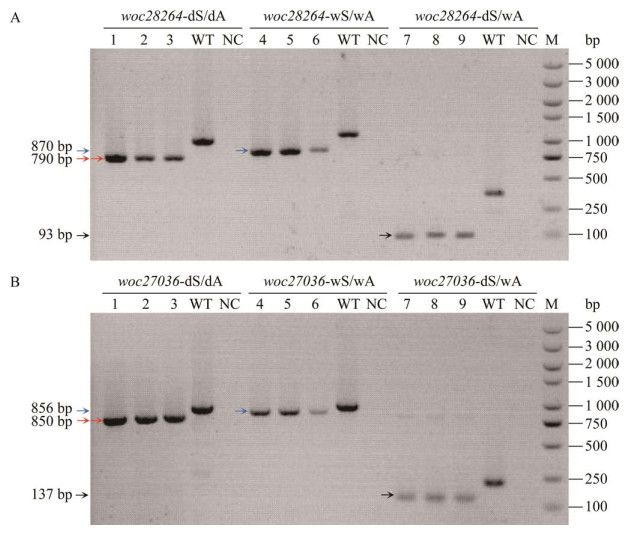
|
| 图 4 缺失突变株的菌落PCR验证 Figure 4 Identification of the mutants by colony PCR. A: PCR detection of Δwoc28264 double crossover mutants using three primer pairs. B: PCR detection of Δwoc27036 double crossover mutants using three primer pairs. M: DNA marker. PCR templates used are: 1–9, nine independent colonies that can grow on the selective plate after mating. WT: N1230-9 wild-type strain. NC: Negative control. |
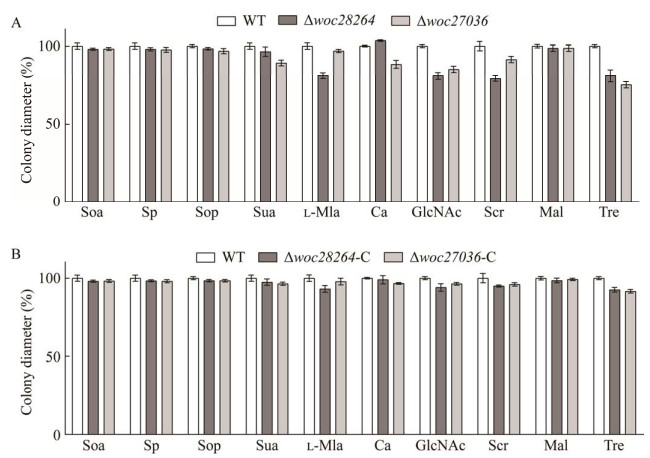
|
| 图 5 基因缺失突变体及基因回补菌株对10种碳源的趋化表型 Figure 5 Participation of woc28264, woc27036, woc28264-C and woc27036-C in the chemotactic responses to 10 carbon sources demonstrated in swimming plate assays. A: Chemotactic responses to 10 carbon sources with WT and two mutants (Δwoc28264 and Δwoc27036). B: Chemotactic responses to 10 carbon sources with WT and two complement mutants (Δwoc28264-C and Δwoc27036-C). |
3 讨论与结论
大多数细菌已经进化出检测各种环境信号的能力,以便在快速变化的条件下生存和繁殖。控制运动的趋化性网络是最突出的原核感应系统之一[27]。虽然信号通路的核心部件在细菌中是保守的,但化学受体的数量和特异性是高度可变的,并且是菌株特异性的[3]。不同于E. coli所拥有的4个趋化受体仅参与一种化学感应途径,类球红细菌(Rhodobacter sphaeroides)[28]、霍乱弧菌(Vibrio cholerae)[29]、P. aeruginosa[30]、黄色黏球菌(Myxococcus xanthus)[31]和伯氏疏螺旋体(Borrelia burgdorferi)[32]等多种细菌不仅具有数量众多的化学受体,还具有多个化学感应途径。确定哪种化学受体进入哪种途径是复杂化学感受系统研究的核心问题。多项研究已证实P. aeruginosa PAO1的WSP和CHP途径分别由单个化学受体McpB/Aer2、WspA和PilJ刺激,而分散在基因组中的化学受体被预测为进入介导趋化性的F6途径[33-36]。比较分析发现,菌株P. arabiensis N1230-9具有类似于铜绿假单胞菌F6和F7途径相关基因簇,而不具有其他类别的化学感受途径相关基因簇,因此推断菌株N1230-9的28个化学受体只能进入F6型和F7型途径,其中位于cluster Ⅱ中的WOC26077负责F7途径的激活,而其他散在分布在基因组中的27个MCP可能进入F6途径。不同于目前报道的趋氧受体通常只具有一个PAS结构域[25],潜在的趋氧受体WOC26077具有3个PAS结构域,因此不排除该化学受体除了作为O2受体外,还可能在感应胞内外其他刺激信号中发挥重要作用。
综合菌株N1230-9对23种碳源的利用能力和趋化性结果,该菌株并非对其能代谢利用的碳源均有趋化性,也并非对不能利用的碳源均不具有趋化能力。此外,趋化实验结果还表明,菌株N1230-9对同一检测浓度下的不同碳源的趋化能力也存在较大差异。上述实验结果说明,有效的趋化因子并不一定是菌株N1230-9优先代谢的或其用作碳源时生长滞后时间最短的化合物。为了证明具有sCache结构域的MCP是否参与了菌株N1230-9对碳源的趋化,本研究通过软琼脂平板法分析了2个基因缺失突变体和基因回补菌株对10种碳源的趋化能力,得到了3个主要实验结论:(1) WOC28264和WOC27036所结合的化学效应因子存在重叠,如这两个化学受体均是N-乙酰氨基葡萄糖和海藻糖的趋化受体;(2) WOC28264和WOC27036具有化学效应因子的特异性,前者是L-苹果酸和蔗糖的特异性受体,而后者则是柠檬酸和琥珀酸的趋化受体;(3) WOC28264和WOC27036均不是乙酸钠、丙酸钠、丙酮酸钠和麦芽糖的趋化受体。相对于其他检测碳源,N-乙酰氨基葡萄糖和海藻糖在菌株生存环境中出现的频率高,因此不难理解菌株N1230-9为何拥有多个响应这两种碳源的化学受体。突变菌株趋化实验还显示了有趣的实验现象,即woc28264缺失突变体对柠檬酸的趋化性增强。多项研究结果表明,细菌趋化受体与趋化效应因子的结合需要周质结合蛋白的参与[37]。同源比对结果显示,WOC27036的LBD结构域与WOC28264的LBD结构域间的氨基酸序列一致性最高(29%)。因此,我们推测WOC27036与柠檬酸和琥珀酸的结合可能也需要某种周质结合蛋白的参与,以及同样具有sCache型LBD的WOC28264可与WOC27036竞争结合这一周质结合蛋白。
致谢
感谢中国科学院南海海洋研究所王晓雪研究员团队提供菌株和质粒。
| [1] | LARSEN SH, READER RW, KORT EN, TSO WW, ADLER J. Change in direction of flagellar rotation is the basis of the chemotactic response in Escherichia coli[J]. Nature, 1974, 249(452): 74-77. |
| [2] | SZURMANT H, ORDAL GW. Diversity in chemotaxis mechanisms among the bacteria and archaea[J]. Microbiology and Molecular Biology Reviews: MMBR, 2004, 68(2): 301-319 DOI:10.1128/MMBR.68.2.301-319.2004. |
| [3] | SALAH UD-DIN AIM, ROUJEINIKOVA A. Methyl-accepting chemotaxis proteins: a core sensing element in prokaryotes and archaea[J]. Cellular and Molecular Life Sciences, 2017, 74(18): 3293-3303 DOI:10.1007/s00018-017-2514-0. |
| [4] | BI S, LAI L. Bacterial chemoreceptors and chemoeffectors[J]. Cellular and Molecular Life Sciences, 2015, 72(4): 691-708 DOI:10.1007/s00018-014-1770-5. |
| [5] | HUANG Z, PAN X, XU N, GUO M. Bacterial chemotaxis coupling protein: structure, function and diversity[J]. Microbiological Research, 2019, 219: 40-48 DOI:10.1016/j.micres.2018.11.001. |
| [6] | HUANG Z, WANG YH, ZHU HZ, ANDRIANOVA EP, JIANG CY, LI D, MA L, FENG J, LIU ZP, XIANG H, ZHULIN IB, LIU SJ. Cross talk between chemosensory pathways that modulate chemotaxis and biofilm formation[J]. mBio, 2019, 10(1): e02876-18. |
| [7] | KEEGSTRA JM, CARRARA F, STOCKER R. The ecological roles of bacterial chemotaxis[J]. Nature Reviews Microbiology, 2022, 20(8): 491-504 DOI:10.1038/s41579-022-00709-w. |
| [8] | STOCKER R, SEYMOUR JR. Ecology and physics of bacterial chemotaxis in the ocean[J]. Microbiology and Molecular Biology Reviews: MMBR, 2012, 76(4): 792-812 DOI:10.1128/MMBR.00029-12. |
| [9] | SMRIGA S, FERNANDEZ Ⅵ, MITCHELL JG, STOCKER R. Chemotaxis toward phytoplankton drives organic matter partitioning among marine bacteria[J]. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(6): 1576-1581. |
| [10] | STOCKER R, SEYMOUR JR, SAMADANI A, HUNT DE, POLZ MF. Rapid chemotactic response enables marine bacteria to exploit ephemeral microscale nutrient patches[J]. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(11): 4209-4214. |
| [11] | AZAM F. Microbial control of oceanic carbon flux: the plot thickens[J]. Science, 1998, 280(5364): 694-696 DOI:10.1126/science.280.5364.694. |
| [12] | BLACKBURN N, FENCHEL T, MITCHELL J. Microscale nutrient patches in planktonic habitats shown by chemotactic bacteria[J]. Science, 1998, 282(5397): 2254-2256 DOI:10.1126/science.282.5397.2254. |
| [13] | FENCHEL T. Microbial behavior in a heterogeneous world[J]. Science, 2002, 296(5570): 1068-1071 DOI:10.1126/science.1070118. |
| [14] | MITCHELL JG, OKUBO A, FUHRMAN JA. Microzones surrounding phytoplankton form the basis for a stratified marine microbial ecosystem[J]. Nature, 1985, 316(6023): 58-59 DOI:10.1038/316058a0. |
| [15] | YAWATA Y, CARRARA F, MENOLASCINA F, STOCKER R. Constrained optimal foraging by marine bacterioplankton on particulate organic matter[J]. Proceedings of the National Academy of Sciences of the United States of America, 2020, 117(41): 25571-25579. |
| [16] | ZHENG K, DONG Y, LIANG Y, LIU Y, ZHANG X, ZHANG W, WANG Z, SHAO H, SUNG YY, MOK WJ, WONG LL, McMINN A, WANG M. Genomic diversity and ecological distribution of marine Pseudoalteromonas phages[J]. Marine Life Science & Technology, 2023, 5(2): 271-285. |
| [17] | WEI W, WANG L, FANG J, LIU R. Population structure, activity potential and ecotype partitioning of Pseudoalteromonas along the vertical water column of the New Britain Trench[J]. FEMS Microbiology Letters, 2021, 368(13): fnab078 DOI:10.1093/femsle/fnab078. |
| [18] | DUHAIME MB, SOLONENKO N, ROUX S, VERBERKMOES NC, WICHELS A, SULLIVAN MB. Comparative omics and trait analyses of marine Pseudoalteromonas phages advance the phage OTU concept[J]. Frontiers in Microbiology, 2017, 8: 1241 DOI:10.3389/fmicb.2017.01241. |
| [19] | LIU J, ZHENG Y, LIN H, WANG X, LI M, LIU Y, YU M, ZHAO M, PEDENTCHOUK N, LEA-SMITH DJ, TODD JD, MAGILL CR, ZHANG WJ, ZHOU S, SONG D, ZHONG H, XIN Y, YU M, TIAN J, ZHANG XH. Proliferation of hydrocarbon-degrading microbes at the bottom of the Mariana Trench[J]. Microbiome, 2019, 7(1): 47 DOI:10.1186/s40168-019-0652-3. |
| [20] | MÉDIGUE C, KRIN E, PASCAL G, BARBE V, BERNSEL A, BERTIN PN, CHEUNG F, CRUVEILLER S, D'AMICO S, DUILIO A, FANG G, FELLER G, HO C, MANGENOT S, MARINO G, NILSSON J, PARRILLI E, ROCHA EP, ROUY Z, SEKOWSKA A, et al. Coping with cold: the genome of the versatile marine Antarctica bacterium Pseudoalteromonas haloplanktis TAC125[J]. Genome Research, 2005, 15(10): 1325-1335 DOI:10.1101/gr.4126905. |
| [21] |
徐莹, 兰晓敏, 周敏婕, 陈秀暖, 金佳凡, 朱四东, 杨季芳, 陈吉刚 . 一株太平洋表层海水来源阿拉伯海假交替单胞菌N1230-9的全基因组测序与比较基因组学分析[J]. 微生物学报, 2024.
DOI:10.13343/j.cnki.wsxb.20230669 XU Y, LAN XM, ZHOU MJ, CHEN XN, JIN JF, ZHU SD, YANG JF, CHEN JG. Genome sequencing and comparative genomic analysis of Pseudoalteromonas arabiensis N1230-9, isolated from Pacific Ocean surface-seawater[J]. Acta Microbiologica Sinica, 2024 (in Chinese). |
| [22] | WANG P, YU Z, LI B, CAI X, ZENG Z, CHEN X, WANG X. Development of an efficient conjugation-based genetic manipulation system for Pseudoalteromonas[J]. Microbial Cell Factories, 2015, 14: 11 DOI:10.1186/s12934-015-0194-8. |
| [23] | NORDBERG H, CANTOR M, DUSHEYKO S, HUA S, POLIAKOV A, SHABALOV I, SMIRNOVA T, GRIGORIEV Ⅵ, DUBCHAK I. The genome portal of the Department of Energy Joint Genome Institute: 2014 updates[J]. Nucleic Acids Research, 2014, 42(D1): D26-D31 DOI:10.1093/nar/gkt1069. |
| [24] | PAYSAN-LAFOSSE T, BLUM M, CHUGURANSKY S, GREGO T, PINTO BL, SALAZAR GA, BILESCHI ML, BORK P, BRIDGE A, COLWELL L, GOUGH J, HAFT DH, LETUNIĆ I, MARCHLER-BAUER A, MI H, NATALE DA, ORENGO CA, PANDURANGAN AP, RIVOIRE C, SIGRIST CJA, et al. InterPro in 2022[J]. Nucleic Acids Research, 2023, 51(D1): D418-D427 DOI:10.1093/nar/gkac993. |
| [25] | MATILLA MA, MARTÍN-MORA D, GAVIRA JA, KRELL T. Pseudomonas aeruginosa as a model to study chemosensory pathway signaling[J]. Microbiology and Molecular Biology Reviews: MMBR, 2021, 85(1): e00151-20. |
| [26] | ALVAREZ-ORTEGA C, HARWOOD CS. Identification of a malate chemoreceptor in Pseudomonas aeruginosa by screening for chemotaxis defects in an energy taxis-deficient mutant[J]. Applied and Environmental Microbiology, 2007, 73(23): 7793-7795 DOI:10.1128/AEM.01898-07. |
| [27] | BI S, SOURJIK V. Stimulus sensing and signal processing in bacterial chemotaxis[J]. Current Opinion in Microbiology, 2018, 45: 22-29 DOI:10.1016/j.mib.2018.02.002. |
| [28] | MARTIN AC, WADHAMS GH, ARMITAGE JP. The roles of the multiple CheW and CheA homologues in chemotaxis and in chemoreceptor localization in Rhodobacter sphaeroides[J]. Molecular Microbiology, 2001, 40(6): 1261-1272 DOI:10.1046/j.1365-2958.2001.02468.x. |
| [29] | GOSINK KK, KOBAYASHI R, KAWAGISHI I, HÄSE CC. Analyses of the roles of the three cheA homologs in chemotaxis of Vibrio cholerae[J]. Journal of Bacteriology, 2002, 184(6): 1767-1771 DOI:10.1128/JB.184.6.1767-1771.2002. |
| [30] | STOVER CK, PHAM XQ, ERWIN AL, MIZOGUCHI SD, WARRENER P, HICKEY MJ, BRINKMAN FS, HUFNAGLE WO, KOWALIK DJ, LAGROU M, GARBER RL, GOLTRY L, TOLENTINO E, WESTBROCK-WADMAN S, YUAN Y, BRODY LL, COULTER SN, FOLGER KR, KAS A, LARBIG K, et al. Complete genome sequence of Pseudomonas aeruginosa PAO1, an opportunistic pathogen[J]. Nature, 2000, 406(6799): 959-964. |
| [31] | YANG Z, GENG Y, XU D, KAPLAN HB, SHI W. A new set of chemotaxis homologues is essential for Myxococcus xanthus social motility[J]. Molecular Microbiology, 1998, 30(5): 1123-1130. |
| [32] | FRASER CM, CASJENS S, HUANG WM, SUTTON GG, CLAYTON R, LATHIGRA R, WHITE O, KETCHUM KA, DODSON R, HICKEY EK, GWINN M, DOUGHERTY B, TOMB JF, FLEISCHMANN RD, RICHARDSON D, PETERSON J, KERLAVAGE AR, QUACKENBUSH J, SALZBERG S, HANSON M, et al. Genomic sequence of a Lyme disease spirochaete, Borrelia burgdorferi[J]. Nature, 1997, 390(6660): 580-586. |
| [33] | ORTEGA DR, FLEETWOOD AD, KRELL T, HARWOOD CS, JENSEN GJ, ZHULIN IB. Assigning chemoreceptors to chemosensory pathways in Pseudomonas aeruginosa[J]. Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(48): 12809-12814. |
| [34] | GARCÍA-FONTANA C, CORRAL LUGO A, KRELL T. Specificity of the CheR2 methyltransferase in Pseudomonas aeruginosa is directed by a C-terminal pentapeptide in the McpB chemoreceptor[J]. Science Signaling, 2014, 7(320): ra34. |
| [35] | GARCÍA-FONTANA C, REYES-DARIAS JA, MUÑOZ-MARTÍNEZ F, ALFONSO C, MOREL B, RAMOS JL, KRELL T. High specificity in CheR methyltransferase function: CheR2 of Pseudomonas putida is essential for chemotaxis, whereas cher1 is involved in biofilm formation[J]. The Journal of Biological Chemistry, 2013, 288(26): 18987-18999. |
| [36] | COVARRUBIAS PC, MOYA-BELTRÁN A, ATAVALES J, MOYA-FLORES F, TAPIA PS, ACUÑA LG, SPINELLI S, QUATRINI R. Occurrence, integrity and functionality of AcaML1-like viruses infecting extreme acidophiles of the Acidithiobacillus species complex[J]. Research in Microbiology, 2018, 169(10): 628-637. |
| [37] | WADHAMS GH, ARMITAGE JP. Making sense of it all: bacterial chemotaxis[J]. Nature Reviews Molecular Cell Biology, 2004, 5(12): 1024-1037. |
 2024, Vol. 64
2024, Vol. 64