中国科学院微生物研究所,中国微生物学会
文章信息
- 王梦竹, 赵梦诗, 马晓丹, 曾玲霜, 杨秋娥, 邓辉. 2024
- WANG Mengzhu, ZHAO Mengshi, MA Xiaodan, ZENG Lingshuang, YANG Qiu'e, DENG Hui.
- 驯化噬菌体提高噬菌体对碳青霉烯类耐药肺炎克雷伯菌的杀菌能力
- Phage training improves the bactericidal ability against carbapenem-resistant Klebsiella pneumoniae
- 微生物学报, 64(1): 254-267
- Acta Microbiologica Sinica, 64(1): 254-267
-
文章历史
- 收稿日期:2023-06-14
- 网络出版日期:2023-08-29
2. 福建省土壤环境健康与调控重点实验室, 福建 福州 350002
2. Fujian Provincial Key Laboratory of Soil Environmental Health and Regulation, Fuzhou 350002, Fujian, China
抗生素的广泛使用在抑制细菌生长的同时,还对细菌施加了选择压力,促进了耐药性的出现和传播,致使抗生素治疗失败[1]。一旦病原菌获得抗生素耐药性(antimicrobial resistance, AMR),将会导致多重耐药菌株的出现,甚至进化出“超级细菌”。其中,治疗携带碳青霉烯酶编码基因的碳青霉烯类耐药肺炎克雷伯菌(carbapenem-resistant Klebsiella pneumoniae, CRKp)感染已经成为了一个世界性的问题[2]。在我国,院内CRKp感染又以高传播性、高致病性及高耐药性的ST11型为主,因此解决ST11型CRKp感染问题已迫在眉睫。
针对ST11型CRKp耐药菌感染,噬菌体(bacteriophage, phage)有望成为替代或补充传统抗生素的候选物[3]。噬菌体是具有多种抗菌效应的靶向细菌病毒,可以减少人体内的多重耐药菌感染[4]。与此同时,噬菌体具有宿主特异性高的显著优点,这使得机体微生物群落不受噬菌体疗法的影响[5]。近年来,噬菌体疗法在治疗分枝杆菌[6]、肺炎克雷伯菌、霍乱杆菌[7]等细菌感染等疾病方面均取得进展。
细菌和噬菌体在共同进化过程中相互施加强大的选择压力,如果这种相互作用是长期的,将可能引起快速的进化,从而导致双方抗性和传染性的增加[8]。细菌的噬菌体抗性进化对噬菌体治疗的有效性构成了不可避免的威胁,并且针对单一噬菌体的抗性已被证明在体外很容易出现[9]。另一方面,有文献指出,经过驯化的噬菌体在侵染宿主菌时可降低宿主菌抗性的产生[10],从而改善治疗结果。
本研究以前期临床分离的CRKp (Kp2092)为宿主菌,从福州市浮村污水处理厂和福建省星源中德牧业有限公司采集的污水样品中筛选出9株肺炎克雷伯菌噬菌体。其中噬菌体P55anc侵染能力最强且裂解谱最广,故以该噬菌体及其宿主菌Kp2092为研究对象进行噬菌体驯化,以得到侵染和杀菌能力更强且宿主菌不易产生抗性的噬菌体进化株,为增强噬菌体杀菌能力并避免抗噬菌体细菌的出现提供理论依据。
1 材料与方法 1.1 材料 1.1.1 菌株及样品本研究所使用的宿主细菌为一株碳青霉烯类耐药肺炎克雷伯菌Kp2092,该细菌分离自临床病人痰液样品,并通过药敏试验测定最小抑菌浓度(minimum inhibitory concentration, MIC)来评估其耐药情况。噬菌体分离所需污水样品于2022年5月采集自福州市浮村污水处理厂和福建省星源中德牧业有限公司。
取保存于−80 ℃的肺炎克雷伯菌20 μL加入2 mL无菌LB肉汤中,振荡培养(37 ℃、200 r/min,下同) 2−3 h至指数生长期,置于4 ℃备用。
1.1.2 主要试剂和仪器实验过程中所使用的LB琼脂培养基、LB肉汤购自广东省环凯微生物科技有限公司;尿道定位显色培养基(urine orientation chromogenic medium, UTI)购自青岛高科技工业园海博生物技术有限公司;噬菌体DNA提取试剂盒DNeasy® Blood & Tissue Kit购自QIAGEN GmbH;DNase Ⅰ和RNase-free购自南京诺唯赞生物科技股份有限公司;RNase A (1 000 mg/mL)购自翌圣生物科技(上海)股份有限公司;Qubit Flex购自Thermo Fisher Scientific;Qubit工作液dsDNA快速定量试剂盒购自生工生物工程(上海)股份有限公司;SM缓冲液购自上海源叶生物科技有限公司;Speactra Max® iD3多功能酶标仪购自Molecular Devices。
1.2 噬菌体的筛选和分离纯化噬菌体的分离纯化方法参照文献[11]:将污水样品12 000 r/min离心3 min,采用0.22 μm滤膜过滤上清液3遍后收集滤液备用。取滤液1 mL分别加至60株ST11型肺炎克雷伯菌的1 mL指数期菌液中,再加入2 mL LB肉汤,37 ℃振荡培养过夜后离心过滤,收集上清液即为噬菌体原液。
将噬菌体原液梯度稀释至10−8,取各梯度稀释液100 μL至100 μL对应宿主菌液中混匀,静置15 min后加入3.5 mL 0.6%半固体琼脂,倒入下层琼脂(1.5%琼脂培养基)中制备双层琼脂平板。37 ℃倒置过夜培养后观察有无噬菌斑。
用枪头挑取单个噬菌斑加入500 μL宿主菌液中,并加入2 mL LB肉汤后再次37 ℃培养,离心取上清过滤,滤液梯度稀释后制备双层琼脂进行培养以纯化噬菌体,重复上述步骤3次后得到纯化的噬菌体。将纯化的噬菌体滤液与30%甘油肉汤按1:1 (体积比)混合后保存于−80 ℃冰箱。
1.3 噬菌体裂解谱分析根据已有文献报道[11-12]修改,本研究分析了9株噬菌体分离株对60株ST11型CRKp的裂解谱。将60株CRKp培养至指数期,各取菌液100 μL加入10 mL LB琼脂中制备双层琼脂平板。将9株噬菌体与其宿主菌共培养8−12 h后离心过滤,各取其噬菌体滤液5 μL滴于含不同Kp的双层琼脂平板上,待滤液被吸收后倒置平板,37 ℃过夜培养后观察有无噬菌斑产生以及噬菌斑形态,以附图S1 [已提交至国家微生物科学数据中心NMDC (http://nmdc.cn),No. NMDCX0000222]所示为标准(1和0.75为透明噬菌斑,0.5和0.25为模糊噬菌斑)进行分类统计。根据噬菌体裂解谱结果,选择裂解谱广、侵染能力强的噬菌体P55anc进行后续噬菌体生理特征及进化分析。
1.4 噬菌体P55anc形态观察参考文畅等[13]的方法观察噬菌体形态:取100 μL P55anc滤液滴于含有宿主菌Kp2092的双层琼脂平板上过夜培养,取100 μL无菌水反复轻轻吹打噬菌斑后进行电镜拍摄。取20 μL噬菌体液滴到200目铜网上,静置5 min后用滤纸吸掉多余液体,干燥1 min。向铜网加入1滴1%磷钨酸,室温静置10 min,置于日立透射电子显微镜下观察噬菌体形态。
1.5 噬菌体基因组分析提取噬菌体DNA过程中,由于不同的噬菌体对不同的物质敏感性不同,故本研究利用DNeasy® Blood & Tissue Kit试剂盒提取噬菌体DNA。具体步骤参照文献[14]:将5 mL SM缓冲液加入到有噬菌斑的平板中,于4 ℃冰箱放置12 h后离心过滤。吸取滤液450 μL至2 mL离心管中,加入50 μL DNaseⅠ 10×buffer,1 μL DNaseⅠ (1 U/μL)和1 μL RNase A (1 000 mg/mL),37 ℃加热1.5 h后加入20 μL 0.5 mol/L EDTA。加入1.25 μL proteinase K (20 mg/mL),56 ℃加热1.5 h。后续操作按照DNeasy® Blood & Tissue Kit试剂盒说明书进行。纯化后的DNA送至福州生工生物工程股份有限公司进行Illumina测序,通过CLC Genomics Workbench软件去除宿主DNA序列,并利用SPAdes从头拼接噬菌体基因组,利用Prokka和NCBI注释该基因组,最后利用Snap Gene® 6.0.2绘制噬菌体P55anc的基因图谱[11, 13, 15]。将P55anc的基因组序列上传至NCBI的GenBank数据库,登录号为OR387886。
1.6 P55anc系统发育树分析为了说明P55anc与其他同源噬菌体之间的系统进化关系,利用P55anc全基因组序列进行NCBI检索,获得4株同源噬菌体序列,并与其他5种不同属的噬菌体序列通过Molecular Evolutionary Genetics Analysis (version 11.0)软件[16]绘制系统发育树。
1.7 噬菌体P55anc及其宿主菌共进化根据已有报道[17]修改,将Kp2092培养至指数期,取180 μL加入96孔板中,再加入20 μL噬菌体P55anc培养液,共培养液37 ℃振荡培养24 h后按1:200接入新的LB肉汤后继续振荡培养,连续传代培养9 d,共设置30个重复。在第1、3、5、7、9天分离每个样品中的噬菌体,具体方法参照1.3。将5 μL噬菌体滤液滴加于含宿主菌Kp2092的双层琼脂平板上,37 ℃培养并观察噬菌斑产生情况。如有较为透明的噬菌斑,则表明该样品中仍有可感染Kp2092的噬菌体,并计算可感染Kp2092的噬菌体所占30个重复样品的比率(%)。在共进化实验第9天,仅3个重复的噬菌体样品可产生透明噬菌斑,挑取单个噬菌斑培养后进行保存,3株噬菌体进化株分别命名为P55evo-1、P55evo-2、P55evo-3,并以其为研究对象进行后续实验。参照1.5的方法对噬菌体进化株P55evo-1基因组测序并进行单核苷酸多态性(single nucleotide polymorphism, SNP)分析,以确定P55evo-1突变位点。
1.8 进化后噬菌体杀菌能力测定挑取Kp2092单菌落加入1 mL LB肉汤,混匀后取180 μL加入96孔板中,再加入20 μL噬菌体滤液混匀后,置于37 ℃培养。使用Speactra Max® iD3多功能酶标仪每小时取点测定OD600,并利用GraphPad prism (version 8.3.0)软件绘制24 h生长曲线。
进化过程中分离一株噬菌体耐受细菌,将其命名为Kp2092evo,验证3株进化噬菌体对该耐受细菌的杀菌能力。实验步骤及方法同上。
1.9 进化后噬菌体裂解谱分析参照1.3的方法分析进化后噬菌体裂解谱,并与P55anc裂解谱进行对比。
1.10 进化后噬菌体MOI及一步生长曲线测定根据王九儒等[11]的方法测定最佳感染复数(multiplicity of infection, MOI)及一步生长曲线[18]。挑取Kp2092单菌落于LB肉汤培养1.5 h,用磷酸缓冲盐溶液(phosphate-buffered saline, PBS)梯度稀释,各梯度稀释液取10 μL滴于LB平板并十字晃开,37 ℃过夜培养后计算菌液浓度(CFU/mL)。将培养好的噬菌体通过双层琼脂平板法计算噬菌体滴度(PFU/mL)。按照噬菌体/细菌浓度为1、0.1、0.01、0.001、0.000 1、0.000 01和0.000 001的比例混合噬菌体与菌液,使混合液体积为200 μL,再加至2 mL LB肉汤中,37 ℃振荡培养5 h,共培养液经离心后取上清过滤,滤液梯度稀释后,各稀释液利用双层琼脂平板法测定噬菌体滴度。重复3次,产生最高滴度的噬菌体/细菌比例即为最佳MOI。
按最佳MOI混合噬菌体及其宿主菌共400 μL,再加至20 mL LB肉汤中,37 ℃培养5 min。共培养液经离心后弃上清,加入5 mL LB肉汤重悬沉淀,再次离心弃上清液,重新加入20 mL LB肉汤37 ℃振荡培养。每10 min取样1 mL,通过双层琼脂平板法测定噬菌体滴度,重复3次。绘制噬菌体一步生长曲线,分析噬菌体侵染宿主菌的潜伏期、裂解期和平台期并计算裂解量。裂解量=裂解末期噬菌体滴度/感染初期细菌浓度[11]。
1.11 进化后噬菌体稳定性测定热稳定性[16]:取1 mL噬菌体滤液在4、30、37、50、60、70 ℃和80 ℃条件下培养2 h后梯度稀释,运用双层琼脂平板法计算噬菌体滴度。
紫外稳定性:取1 mL噬菌体滤液置于无菌培养皿中,在距离紫外灯30 cm处静置照射0、5、10、15、20、25、30 min后取样20 μL梯度稀释,通过双层琼脂平板法测定噬菌体滴度。
pH敏感性:取100 μL噬菌体滤液加入900 μL pH值为3、4、5、6、7、8、9、10的无菌LB肉汤,37 ℃培养2 h后梯度稀释,通过双层琼脂平板法测定噬菌体滴度。
根据报道实验方法[1]进行改进:将血清分为2份,一份放4 ℃保存,另一份56 ℃灭活30 min为对照。将PFU为106的500 μL噬菌体滤液分别加入到新鲜和灭活的血清中,混匀后于37 ℃条件下振荡培养,分别于第1、2、4、6、8小时取样,样品通过双层琼脂平板法测定噬菌体滴度。
2 结果与分析 2.1 Kp2092 MIC测定及噬菌体分离纯化通过测定Kp2092对14种抗生素的MIC值可知Kp2092呈现多重耐药,对美罗培南、头孢喹肟、环丙沙星等抗生素耐药(MIC值≥64),而仅对替加环素、多粘菌素敏感(表 1)。细菌Kp2092的基因组多位点序列分型(multilocus sequence typing, MLST)为ST11型,是我国院内肺炎克雷伯菌感染较流行的ST亚型[2]。以该菌株为宿主菌分离噬菌体,将能更好地筛选出针对多重耐药菌的噬菌体。本研究以污水为样品,共分离纯化得到9株可侵染Kp2092的噬菌体。
| Antibiotics | MIC (μg/mL) | Sensitivity |
| Mero | ≥64 | R |
| IMP | ≥64 | R |
| AMP | ≥32 | R |
| AMC | ≥32 | R |
| TZP | ≥128 | R |
| CZ | ≥64 | R |
| CTX | ≥64 | R |
| CRO | ≥64 | R |
| FEP | ≥64 | R |
| ATM | ≥64 | R |
| CIP | ≥64 | R |
| LEV | ≥8 | R |
| TGC | 2 | S |
| CS | 2 | S |
| S: Sensitive; R: Resistant; Mero: Meropenem; IMP: Imipenem; AMP: Ampicillin; AMC: Amoxicillin/Potassium clavulanate; TZP: Tazobactam/Piperacillin; CZ: Cefazolin; CTX: Cefquinome; CRO: Ceftriaxone; FEP: Cefepime; ATM: Aztreonam; CIP: Ciprofloxacinhydrochloride; LEV: Levofloxacin tablets; TIG: Tigecycline; CS: Colistinsulfate. | ||
2.2 噬菌体裂解谱分析
将分离得到的9株噬菌体侵染60株ST11型CRKp,评估其侵染能力。结果显示不同噬菌体具有不同的裂解谱和裂解能力(图 1)。9株噬菌体均可侵染Kp2092,但仅有P55anc可产生透明噬菌斑,其他噬菌体产生的噬菌斑较模糊。并且P55anc可侵染最多宿主菌(25/60),其中侵染20株菌时可产生明显噬菌斑。因此,基于裂解能力强,裂解谱广的噬菌体P55anc进行后续驯化实验。

|
| 图 1 肺炎克雷伯菌噬菌体裂解谱 Figure 1 Lytic spectrum of phage to Klebsiella pneumoniae. The ordinate and abscissa are the name of phage (phage ID) and bacteria (bacteria ID), respectively; The deeper color represents the stronger infection ability. |
2.3 噬菌体P55anc形态
噬菌体P55anc在其宿主菌平板上形成的噬菌斑边缘光滑、有透亮圆形的晕环(图 2A)。电镜结果(图 2B)显示,P55anc是头部直径约63 nm和尾部直径约11 nm的短尾噬菌体。
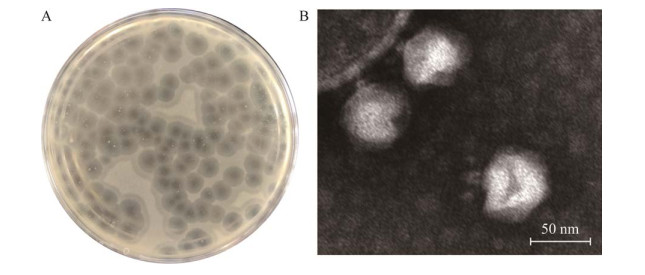
|
| 图 2 噬菌体P55anc平板和电镜形态 Figure 2 Plaque and morphology of phage P55anc. A: Phage plaque of P55anc on agar plate. B: Microscopic graph of P55anc. |
2.4 噬菌体基因组分析
噬菌体基因组分析能够提高对噬菌体基因组的理解,并提高噬菌体应用于临床治疗的可靠性、安全性。基因组分析发现噬菌体P55anc基因组全长40 301 bp,包含51个编码序列(coding sequences, CDSs),其中27个编码已知功能的蛋白,其余为假设蛋白,未发现与耐药或毒力相关的基因(图 3)。
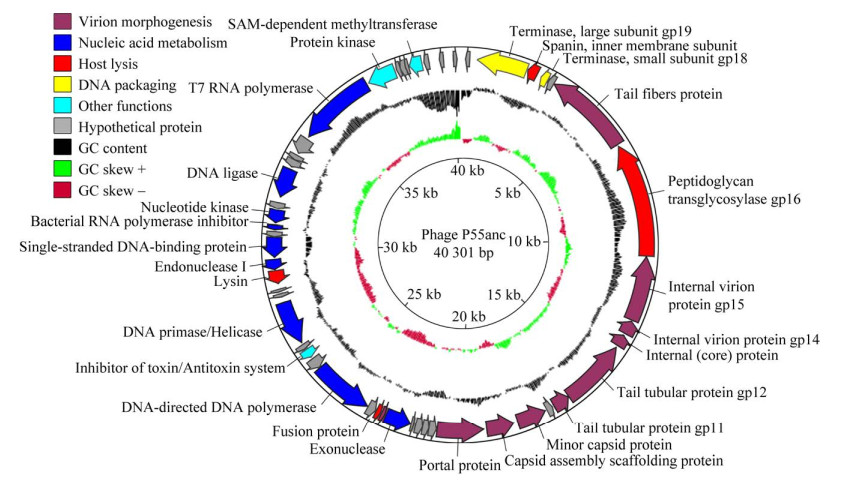
|
| 图 3 噬菌体P55anc全基因组图 Figure 3 Genomic map of phage P55anc. |
依据功能进行分类,27个编码已知功能蛋白的CDSs中,9个编码噬菌体结构蛋白:尾纤维蛋白(ORF 6)、内部病毒体蛋白(ORF 8、9、10)、尾管蛋白(ORF 11、12)、次要衣壳蛋白(ORF 14)、衣壳装配支架蛋白(ORF 15)和门户蛋白(ORF 16);9个CDSs涉及核酸代谢:外切核酸酶(ORF 21)、DNA聚合酶(ORF 25)、DNA引物酶/解旋酶(ORF 29)、核酸内切酶Ⅰ (ORF 33)、单链DNA结合蛋白(ORF 34)、细菌RNA聚合酶抑制剂(ORF 36)、核苷酸激酶(ORF 37)、DNA连接酶(ORF 39)和T7 RNA聚合酶(ORF 43);4个CDSs参与细胞裂解:膜融合蛋白(ORF 3)、葡聚糖转糖基酶(ORF 7)、融合蛋白(ORF 23)和内溶菌素(ORF 32);而终止酶大亚基(ORF 2)和终止酶小亚基(ORF 4)参与噬菌体DNA包装。另外3个其他功能的蛋白为毒素/抗毒素系统抑制剂(ORF 27)、蛋白激酶(ORF 44)和SAM依赖性甲基转移酶(ORF 48)。详情见表S1 (NMDC编号No. NMDCX0000221)。
2.5 系统发育树分析通过NCBI噬菌体数据库比对分析,P55anc与已知肺炎克雷伯菌噬菌体基因组序列Klebsiella phage K5-4 (NC 047799)、Klebsiella phage cp29 (OX 335401)、Klebsiella phage Kp11 (ON 148528)及大肠杆菌噬菌体基因组Enterobacteria phage K30 (NC 015719)的匹配度较高(覆盖率>85%,一致性>90%),且以上已知噬菌体均为Przondovirus属病毒。而P55anc与其他5种不同属的噬菌体基因组覆盖率低(<60%)。基于噬菌体全基因组序列绘制的系统发育树,如图 4所示,P55anc与肺炎克雷伯菌噬菌体(Klebsiella phage) K5-4 (NC 047799)位于同一分支,自展值为81,进一步说明P55anc属于Przondovirus属病毒。
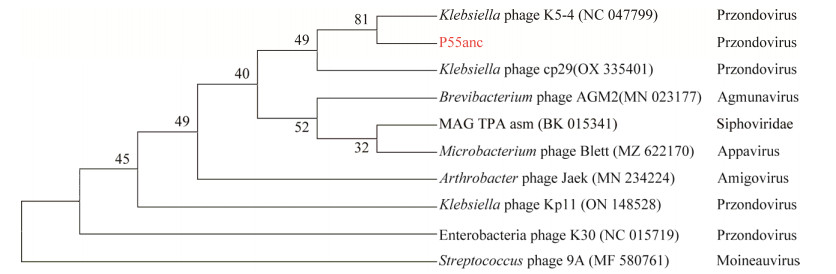
|
| 图 4 P55anc系统发育树分析 Figure 4 Phylogenetic tree analysis of phage P55anc. |
2.6 噬菌体P55anc及其宿主菌共进化
对宿主-噬菌体混合培养液进行连续传代培养,并验证共进化过程中噬菌体对原宿主菌的侵染能力。结果显示在为期9 d的进化实验中,随着时间的推移,可侵染Kp2092并产生透明噬菌斑的噬菌体样品个数不断下降(图 5A)。到第9天时,在总共30个重复中,只有3个重复的噬菌体仍保持对原宿主菌的强裂解能力,占比从100%下降到10%。3个重复中分离纯化的噬菌体进化株(P55evo-1、P55evo-2、P55evo-3)的噬菌斑形态如图 5B所示,中间为圆形透明斑且周围有光滑圆形晕环。

|
| 图 5 噬菌体驯化过程中噬菌体侵染能力的变化 Figure 5 The dynamics of infection ability of phage during 9-day training. A: Infection rate of the phage to Kp2092 during the co-evolutionary experiment. B: The plaque of three evolved phages at day 9. |
SNP分析结果显示,噬菌体进化株P55evo-1有3个突变位点,分别位于尾管蛋白(ORF 6)、尾纤维蛋白(ORF 11)和假定蛋白(ORF 24)中,见附表S2 (NMDC No. NMDCX0000222)。关于噬菌体受体研究将在今后的研究中进一步深入探究。
2.7 进化后噬菌体杀菌能力测定初始噬菌体P55anc及3株进化噬菌体对Kp2092的杀菌曲线如图 6A所示。由图可知,噬菌体P55anc侵染的Kp2092在6 h时OD600值开始上升,表明Kp2092对P55anc产生了抗性;而进化株P55evo-1或P55evo-3侵染的Kp2092在24 h呈现出很好的杀菌效果,即OD600无明显变化,始终保持在0.1,表明Kp2092未对噬菌体产生噬菌体抗性。P55evo-2侵染的Kp2092在19 h时呈现缓慢上升趋势,到24 h时OD600达到0.2左右,此时依然具有杀菌能力。结果表明,噬菌体进化株对细菌Kp2092有明显杀菌效果且Kp2092不易对噬菌体进化株产生抗性。P55anc与3株进化株的曲线下面积(area under curve, AUC)均有显著差异(P<0.000 1),3株进化株中两两比较也各有差异性。

|
| 图 6 进化后噬菌体杀菌能力比较 Figure 6 Determination of bactericidal ability of evolved phages. A: Comparison of the bactericidal ability of evolved phages against Kp2092. B: Their comparisons by calculating the area under curve (AUC). C: Comparison of the bactericidal ability of evolved phages against Kp2092evo. ****: Very significant difference (P < 0.000 1); ***: Very significant difference (P < 0.001); **: Significant difference (P < 0.01). |
与此同时,分离一株对P55anc耐受的细菌Kp2092evo,即P55anc对该细菌没有杀菌或抑菌作用(图 6C)。然而,3株噬菌体进化株在10 h内对该耐受细菌保持良好杀菌能力,OD600保持在0.2左右,进一步证明了进化后的噬菌体获得了比原始噬菌体更强的裂解能力。
2.8 进化后噬菌体裂解谱分析对P55anc与3株进化后噬菌体的裂解谱进行比较分析。如图 7所示,与原始噬菌体相比,进化后噬菌体增加了对2株ST11型CRKp (Kp2219和Kp2300)的裂解能力,使其裂菌谱增加至27株ST11型CRKp。与此同时,进化后噬菌体对其他细菌宿主的裂解能力也有所提升,例如P55anc侵染Kp1426时产生模糊噬菌斑,而进化株可产生透亮的噬菌斑,表明进化噬菌体对该菌的裂解能力增强。
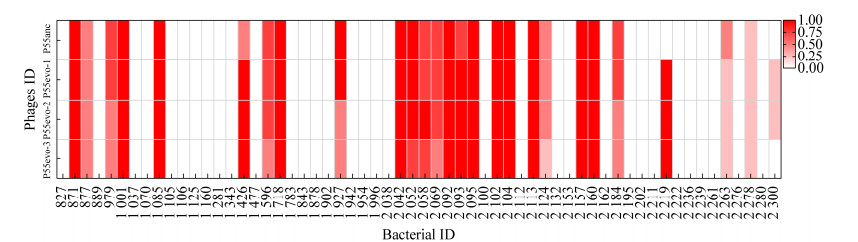
|
| 图 7 进化后噬菌体的裂解谱 Figure 7 Lytic spectrum of evolved-phages. |
2.9 进化后噬菌体MOI及一步生长曲线测定
进化前后噬菌体的MOI如图 8A所示,当MOI为0.000 01时,噬菌体P55anc感染宿主后释放的子代噬菌体数量最多,即P55anc的最佳MOI为0.000 01。而3株噬菌体进化株P55evo-1、P55evo-2和P55evo-3的最佳MOI分别为0.001、0.000 1和0.001。噬菌体进化株相较于原噬菌体P55anc的最佳感染复数变大。

|
| 图 8 进化后噬菌体MOI、一步生长曲线和暴发量的变化 Figure 8 Changes in phage MOI and one-step growth curve of evolved phages. A: Multiplicity of infection (MOI), it is calculated by dividing the phage PFU by bacterial density (CFU/mL). B: One-step growth curve. C: The comparison of phage burst sizes between parental and evolved phages. **: Significant difference (P < 0.01); *: Significant difference (P < 0.05). |
噬菌体P55anc及其3株进化株的一步生长曲线如图 8B所示。噬菌体P55anc在10 min时开始暴发,到120 min时到达平台期,此时滴度为1.01×1010,经计算P55anc裂解量为16.8。P55evo-1在0−20 min时处于潜伏期,从20 min开始暴发,到70 min时到达平台期,此时滴度为1.95×1010,裂解量为32.5。P55evo-2在10 min时处于潜伏期,从20 min开始暴发,到100 min时到达平台期,此时滴度为1.3×1011,裂解量为216。P55evo-3在10 min时处于潜伏期,从20 min开始暴发,到100 min时到达平台期,此时效价为1.5×1010,裂解量为25。总体而言,进化株的MOI增大且一步生长曲线提前20−50 min进入暴发期。将4株噬菌体的裂解量进行t检验分析(图 8C),结果显示3株进化株的裂解量均显著高于P55anc,其中P55evo-2裂解量约为P55anc的13倍。
2.10 进化后噬菌体稳定性测定通过不同温度、pH、紫外和血清的稳定性实验,评估进化后噬菌体的稳定性。如图 9A热稳定性结果所示,噬菌体P55anc和3株噬菌体进化株(P55evo-1、P55evo-2、P55evo-3)在低于50 ℃时均能维持较稳定的滴度,当温度高于50 ℃时噬菌体滴度急剧下降。pH稳定性实验结果(图 9B)显示,各噬菌体在pH值5−8的环境中可稳定存在,pH值大于8时滴度缓慢下降,而pH值小于4时滴度急剧下降,至pH值等于3时为0。进化前后噬菌体在紫外暴露下的稳定性基本一致,且均对紫外有较高的耐受性。在血清8 h处理下噬菌体稳定性高,其各噬菌体滴度基本保持一致,表明该噬菌体可应用于体内环境且保持较好的杀菌能力。

|
| 图 9 不同处理下噬菌体稳定性的比较 Figure 9 Comparison of bacteriophage stability under different treatments. A: Thermal stability comparison. B: Comparison of pH stability. C: Comparison of ultraviolet stability. D: Comparison of serum stability. |
3 讨论与结论
肺炎克雷伯菌是临床上常见的一种条件致病菌,可引起肺炎、脑膜炎、尿路感染与败血症等多种疾病[19]。肺炎克雷伯菌的常规治疗方式是采用碳青霉烯类抗生素,例如亚胺培南、美罗培南等[20],但由于抗生素的不规范使用,导致临床上肺炎克雷伯菌出现多重耐药菌株[21],尤其是ST11型CRKp的出现给疾病的治疗带来巨大挑战。
迄今为止,肺炎克雷伯菌耐药问题依旧严峻,在全国细菌耐药监测网发布的2021年全国细菌耐药检测报告中指出,肺炎克雷伯菌对碳青霉烯类药物的耐药率平均为10.9%,且总体耐药率仍然呈缓慢上升趋势。多项研究表明噬菌体疗法在治疗肺炎克雷伯菌感染治疗中的有效性,并已然成为抗生素的有效代替物之一[22-23]。噬菌体广泛存在于自然环境中且易于分离,本研究从2个不同地点收集污水样品,通过双层琼脂平板法筛选出一株具有广谱型的碳青霉烯类耐药肺炎克雷伯菌噬菌体(P55anc),该噬菌体可侵染25/60株ST11型CRKp,且其中20株产生完全透明噬菌斑,是侵染能力强、裂解谱广的噬菌体。通过电镜形态观察结合全基因组系统发育树分析可知,该噬菌体为Przondovirus属短尾噬菌体,其基因组全长40 301 bp,包含51个编码序列,其中未发现耐药或毒力相关基因,表明P55anc不存在耐药和毒力扩散的问题,可应用于临床治疗。
虽然噬菌体能很好地应用于肺炎克雷伯菌感染且对机体无害,但噬菌体疗法因细菌对噬菌体耐受性的频繁出现而受到挑战[24]。细菌和噬菌体在相互作用过程中共进化是推动微生物生物多样性的重要力量之一。在宿主和噬菌体都具有高的抗性/传染性遗传率的前提下,宿主-噬菌体相互作用可以驱动快速的、相互的遗传变化[25]。在与噬菌体共进化过程中,细菌已经发展出多种方法来抵抗噬菌体的侵染[26]。由此宿主菌在被噬菌体侵染过程中易产生抗性,该缺陷可能导致噬菌体疗法失败。
本研究中,噬菌体P55anc侵染宿主菌Kp2092过程中,前期P55anc可以完全抑制Kp2092的生长,体现出良好的抑菌效果。但随着时间延长,Kp2092逐渐产生对P55anc的抗性并逐步恢复生长活性,直至P55anc无任何抑菌能力。通过共进化实验共获得3株进化株,24 h杀菌曲线表明3株进化株始终保持对Kp2092的侵染能力,长时间内可很好地抑制细菌生长,表明噬菌体进化株的抑菌能力更强且细菌不易产生噬菌体抗性。经计算,噬菌体进化株裂解量均显著高于原噬菌体,这可能是进化株抑菌能力更强的主要原因之一。与此同时,噬菌体进化株的裂解谱与P55anc相比,其侵染CRKp宿主菌的范围略有增加,且总体依旧保持强裂解能力。与原噬菌体相比,进化株的噬菌斑、MOI、一步生长曲线、热稳定性、pH稳定性、紫外稳定性及人血清稳定性等生理特性无显著变化。基于P55evo进化株的特性与优势,驯化后的噬菌体可在一定程度上解决临床上肺炎克雷伯菌的噬菌体耐受性问题,对肺炎克雷伯菌感染疾病的治疗具有重要意义,噬菌体进化具有提高噬菌体治疗效果方法的前景。
| [1] | GORDILLO ALTAMIRANO FL, KOSTOULIAS X, SUBEDI D, KORNEEV D, PELEG AY, BARR JJ. Phage-antibiotic combination is a superior treatment against Acinetobacter baumannii in a preclinical study[J]. EBioMedicine, 2022, 80: 104045 DOI:10.1016/j.ebiom.2022.104045. |
| [2] | YANG XM, SUN QL, LI JP, JIANG Y, LI Y, LIN JP, CHEN KC, CHAN EWC, ZHANG R, CHEN S. Molecular epidemiology of carbapenem-resistant hypervirulent Klebsiella pneumoniae in China[J]. Emerging Microbes & Infections, 2022, 11(1): 841-849. |
| [3] | ZURABOV F, ZHILENKOV E. Characterization of four virulent Klebsiella pneumoniae bacteriophages, and evaluation of their potential use in complex phage preparation[J]. Virology Journal, 2021, 18(1): 1-20 DOI:10.1186/s12985-020-01472-1. |
| [4] | LUONG T, SALABARRIA AC, ROACH DR. Phage therapy in the resistance era: where do we stand and where are we going?[J]. Clinical Therapeutics, 2020, 42(9): 1659-1680 DOI:10.1016/j.clinthera.2020.07.014. |
| [5] | GOLKAR Z, BAGASRA O, PACE DG. Bacteriophage therapy: a potential solution for the antibiotic resistance crisis[J]. Journal of Infection in Developing Countries, 2014, 8(2): 129-136 DOI:10.3855/jidc.3573. |
| [6] | HATFULL GF. Mycobacteriophages: windows into tuberculosis[J]. PLoS Pathogens, 2014, 10(3): e1003953 DOI:10.1371/journal.ppat.1003953. |
| [7] | BHANDARE S, COLOM J, BAIG A, RITCHIE JM, BUKHARI H, SHAH MA, SARKAR BL, SU JL, WREN B, BARROW P, ATTERBURY RJ. Reviving phage therapy for the treatment of cholera[J]. The Journal of Infectious Diseases, 2019, 219(5): 786-794 DOI:10.1093/infdis/jiy563. |
| [8] | HALL AR, SCANLAN PD, BUCKLING A. Bacteria-phage coevolution and the emergence of generalist pathogens[J]. The American Naturalist, 2011, 177(1): 44-53 DOI:10.1086/657441. |
| [9] | HESSE S, RAJAURE M, WALL E, JOHNSON J, BLISKOVSKY V, GOTTESMAN S, ADHYA S. Phage resistance in multidrug-resistant Klebsiella pneumoniae ST258 evolves via diverse mutations that culminate in impaired adsorption[J]. mBio, 2020, 11(1): e02530-e02519. |
| [10] | BORIN JM, AVRANI S, BARRICK JE, PETRIE KL, MEYER JR. Coevolutionary phage training leads to greater bacterial suppression and delays the evolution of phage resistance[J]. Proceedings of the National Academy of Sciences of the United States of America, 2021, 118(23): e2104592118. |
| [11] |
王九儒, 赵飞扬, 李曼莉, 裴广倩, 范航, 张湘莉兰, 米志强, 童贻刚. 一株肺炎克雷伯菌噬菌体的生物学特性及全基因组分析[J]. 微生物学通报, 2019, 46(12): 3402-3413.
WANG JR, ZHAO FY, LI ML, PEI GQ, FAN H, ZHANG XLL, MI ZQ, TONG YG. Characterization and genome analysis of a novel Klebsiella pneumoniae phage[J]. Microbiology China, 2019, 46(12): 3402-3413 (in Chinese). |
| [12] | SOFY AR, EL-DOUGDOUG NK, REFAEY EE, DAWOUD RA, HMED AA. Characterization and full genome sequence of novel KPP-5 lytic phage against Klebsiella pneumoniae responsible for recalcitrant infection[J]. Biomedicines, 2021, 9(4): 342 DOI:10.3390/biomedicines9040342. |
| [13] |
文畅, 刘晨, 卢诗韵, 许忠兵, 艾超凡, 廖汉鹏, 周顺桂. 一株新的多重耐药福氏志贺菌噬菌体生物学特性及基因组分析[J]. 生物技术通报, 2022(9): 127-135.
WEN C, LIU C, LU SY, XU ZB, AI CF, LIAO HP, ZHOU SG. Biological characteristics and genome analysis of a novel multidrugresistant Shigella flexneri phage[J]. Biotechnology Bulletin, 2022(9): 127-135 (in Chinese). |
| [14] | JAKOČIŪNĖ D, MOODLEY A. A rapid bacteriophage DNA extraction method[J]. Methods and Protocols, 2018, 1(3): 27 DOI:10.3390/mps1030027. |
| [15] |
高明明, 王灿, 李璞媛, 刘慧莹, 裴广倩, 范航, 张湘莉兰, 米志强, 童贻刚, 柏长青. 一株新型裂解K63荚膜型肺炎克雷伯菌的噬菌体分离鉴定和生物学特性研究及全基因组分析[J]. 微生物学通报, 2020, 47(1): 210-221.
GAO MM, WANG C, LI PY, LIU HY, PEI GQ, FANG H, ZHANG XLL, MI ZQ, TONG YG, BAI CQ. Biological characteristics and complete genomic analysis of a novel virulent bacteriophage that infects Klebsiella pneumoniae capsular type K63[J]. Microbiology China, 2020, 47(1): 210-221 (in Chinese). |
| [16] |
王猛, 王雪, 赵佳男, 曾君, 左君豪, 郭志良, 季芳, 邵建立, 张立敏, 王承民, 秦建华. 五株克雷伯氏菌噬菌体的生物学特性及比较基因组学研究[J]. 微生物学报, 2023, 63(8): 3110-3128.
WANG M, WANG X, ZHAO JN, ZENG J, ZOU JH, GUO ZL, JI F, SHAO JL, ZHANG LM, Wang CM, QIN JH. Biological characteristics and comparative genomics of five Klebsiella phage isolates[J]. Acta Microbiologica Sinica, 2023, 63(8): 3110-3128 (in Chinese). |
| [17] | YUAN YH, PENG Q, ZHANG SW, LIU TT, YANG S, YU QH, WU Y, GAO MY. Phage reduce stability for regaining infectivity during antagonistic coevolution with host bacterium[J]. Viruses, 2019, 11(2): 118 DOI:10.3390/v11020118. |
| [18] | LI CC, SHI TM, SUN YC, ZHANG YY. A novel method to create efficient phage cocktails via use of phage-resistant bacteria[J]. Applied and Environmental Microbiology, 2022, 88(6): e0232321 DOI:10.1128/aem.02323-21. |
| [19] |
杨帆, 魏纪东, 李敏, 赵永新. 动物源肺炎克雷伯菌耐药性及MLST分析[J]. 中国预防兽医学报, 2016, 38(10): 776-780.
YANG F, WEI JD, LI M, ZHAO YX. Antibiotic resistance and multilocus sequence typing anlysis of Klebsiella pneumonia isolated from animals[J]. Chinese Journal of Preventive Veterinary Medicine, 2016, 38(10): 776-780 (in Chinese). |
| [20] |
刘兰, 刘潺, 加明明, 万璐, 吴小军. 肺炎克雷伯菌ATCC700603裂解性噬菌体的分离鉴定和基因组学研究[J]. 中国抗生素杂志, 2022, 47(8): 786-792.
LIU L, LIU C, JIA MM, WAN L, WU XJ. Isolation, identification, and complete genomic analysis of a novel lytic bacteriophage that infects Klebsiella pneumoniae ATCC700603[J]. Chinese Journal of Antibiotics, 2022, 47(8): 786-792 (in Chinese). |
| [21] | CHEN IL, LEE CH, SU LH, TANG YF, CHANG SJ, LIU JW. Antibiotic consumption and healthcare-associated infections caused by multidrug-resistant Gram-negative bacilli at a large medical center in Taiwan from 2002 to 2009: implicating the importance of antibiotic stewardship[J]. PLoS One, 2013, 8(5): e65621 DOI:10.1371/journal.pone.0065621. |
| [22] | CANO EJ, CAFLISCH KM, BOLLYKY PL, van BELLEGHEM JD, PATEL R, FACKLER J, BROWNSTEIN MJ, HORNE B, BISWAS B, HENRY M, MALAGON F, LEWALLEN DG, SUH GA. Phage therapy for limb-threatening prosthetic knee Klebsiella pneumoniae infection: case report and in vitro characterization of anti-biofilm activity[J]. Clinical Infectious Diseases, 2021, 73(1): e144-e151 DOI:10.1093/cid/ciaa705. |
| [23] | ANAND T, VIRMANI N, KUMAR S, MOHANTY AK, PAVULRAJ S, BERA BC, VAID RK, AHLAWAT U, TRIPATHI BN. Phage therapy for treatment of virulent Klebsiella pneumoniae infection in a mouse model[J]. Journal of Global Antimicrobial Resistance, 2020, 21: 34-41. |
| [24] | XUAN GH, DOU Q, KONG JN, LIN H, WANG JX. Pseudomonas aeruginosa resists phage infection via eavesdropping on indole signaling[J]. Microbiology Spectrum, 2023, 11(1): e0391122. |
| [25] | MORRAN LT, PARRISH RC Ⅱ, GELARDEN IA, ALLEN MB, LIVELY CM. Experimental coevolution: rapid local adaptation by parasites depends on host mating system[J]. The American Naturalist, 2014, 184(S1): S91-S100. |
| [26] | POURCEL C, MIDOUX C, VERGNAUD G, LATINO L. The basis for natural multiresistance to phage in Pseudomonas aeruginosa[J]. Antibiotics, 2020, 9(6): 339. |
 2024, Vol. 64
2024, Vol. 64




