中国科学院微生物研究所,中国微生物学会
文章信息
- 姜忠俊, 李小波. 2022
- JIANG Zhongjun, LI Xiaobo.
- 宏基因组重叠群分箱方法研究综述
- Methods for binning metagenomic contigs
- 微生物学报, 62(8): 2954-2968
- Acta Microbiologica Sinica, 62(8): 2954-2968
-
文章历史
- 收稿日期:2021-12-17
- 修回日期:2022-03-29
- 网络出版日期:2022-04-15
2. 宁波大学信息科学与工程学院, 浙江 宁波 315211
2. Faculty of Electrical Engineering and Computer Science, Ningbo University, Ningbo 315211, Zhejiang, China
越来越多的研究表明,生存在人类肠道、口腔、皮肤以及泌尿生殖道上的微生物群落对于人类的健康具有至关重要的作用[1–2]。这些微生物群落构成脆弱的生态系统,当它们被破坏时,可能会导致很多的疑难杂症,例如哮喘[3]、过敏[4]、肥胖[5]、自身免疫性疾病[6]、糖尿病[7]和自闭症[8]。因此,对微生物群落的认知对人类疾病的诊断以及治疗具有重要价值。
此外,人们对微生物生态系统的理解在海洋研究[9]、农业研究[10]、生物威胁检测[11]、全球变暖[12]以及生物燃料[13]的研究等方面具有重要意义。土壤、海洋中都蕴含着大量目前为止未知的微生物种类,对未知的微生物进行研究,对于土壤和海洋污染的治理、生命科学的认识及生物制药等方面具有重要作用。对于微生物群落的传统研究方法只是将微生物群落分离,然后将分离出的单一微生物在实验室培养皿上纯培养。这不仅需要科研人员对每一种微生物培养所需的营养素和温度等条件了如指掌,同时还需要昂贵并且高维护费用的机器。这就导致人们对于微生物系统多样性的研究较少,更不用说理解生态系统中各微生物之间的相互关系了,而且环境中绝大多数微生物对于人类而言都是未知的[14],很难对其进行分离和纯培养。
随着第二代测序技术的发展,宏基因组学应运而生,它可以避开传统微生物培养方法遇到的困难,提供了一套对微生物群落研究的新方法和流程[15]。宏基因组学技术可以对环境中的所有微生物进行测序,并得到其所有的遗传物质。环境中所有微生物遗传物质的总和被称为宏基因组,而宏基因组学是一种以基因组为对象,以功能基因的筛选和测序分析为研究手段,以微生物的多样性、种群结构特征、进化关系等为研究目的的技术[16]。宏基因组学技术已经广泛应用于上述提到的各个领域,并取得了很好的结果。
宏基因组学分析的一般流程如图 1所示。宏基因组学可以对第二代测序得到的海量宏基因组样本进行质量检测、组装、分箱以及进行注释,来研究环境中微生物群落内的代谢关系,其对于人类认识环境中微生物群落多样性以及了解微生物生态系统具有划时代的意义。宏基因组学的研究热点和难点在于如何将经过一系列处理后得到的reads片段在较低分类水平上进行分类,即分箱(binning)。优秀的宏基因组分箱方法对于从复杂宏基因组环境中重建出较为完整的微生物基因具有重要意义。只有重建出较为完整的微生物基因才能有助于下一步的微生物物种注释以及功能注释等相关研究,这对研究微生物群落多样性以及微生物生态系统有着关键作用。
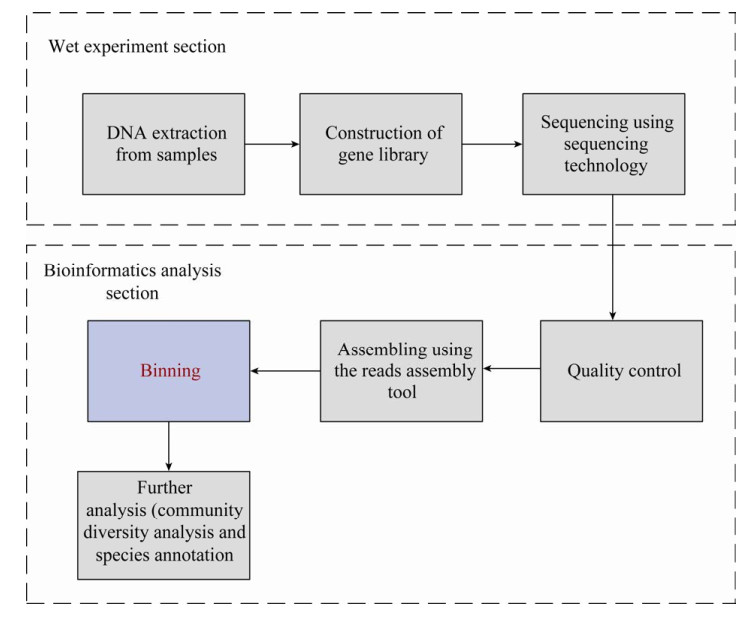
|
| 图 1 宏基因组学分析的一般流程 Figure 1 The general pipeline of metagenomics analysis. |
宏基因组学技术可以从环境中获取所有微生物的遗传物质并测序,然后得到被打断后大小不一、数量众多的DNA片段,这些混合在一起的基因片段就像是打乱后的拼图零件,宏基因组分箱要做的就是将来自相同基因的零件划分到同一分箱中去。由于宏基因组数据量大,并且样本中包含的物种数量众多且丰度不一,现有的宏基因组分箱方法很难在较低分类水平上进行分箱,并且当宏基因组样本较为复杂时,分箱表现不好。我们关注并研究了宏基因组分箱方法存在的主要问题,并对国内外宏基因组分箱方法和未来的展望进行了阐述。
1 宏基因组学相关概念与工具 1.1 宏基因组相关概念第二代测序会将样本中的微生物基因打断并测序,得到长度在100 bp左右的DNA序列即为reads片段,而重叠群片段即contigs片段是通过reads组装工具将reads片段进行组装而形成长度通常在1 000 bp左右的DNA序列。宏基因组分箱是指将宏基因组样本中来自同一微生物基因的序列放到一个簇(即分箱)中,宏基因组分类不仅把宏基因组样本中来自同一微生物基因的序列放到同一分箱中,并且还对每个分箱进行生物水平上的注释,通常使用比对和有监督机器学习算法来进行分类。而宏基因组聚类则只是将来自同一微生物基因的序列放到同一分箱中,并不对分箱进行注释,其通常使用无监督聚类算法进行分箱。
1.2 宏基因组组装工具第二代测序技术获得的宏基因组reads片段,通常大小只有50碱基对到几百碱基对之间,携带遗传信息较少。如果直接对reads片段进行聚类,则效果十分有限,在面对复杂的宏基因组数据集时尤为如此。随着宏基因组组装工具的发展,我们可以将来自同一物种甚至同一菌株的reads片段组装起来形成更长的基因片段,称之为重叠群。常用的宏基因组组装工具包括Megahit[17]、Ray Meta[18]、MetaSPAde[19]等。由于宏基因组样本中绝大多数微生物都是未知的,不能通过参考序列进行比对引导拼接,因此通常采用重新拼接也称作从头拼接方法(de novo)进行宏基因组组装,主要是基于greedy算法、Overlap-Layout-Consensus以及de Bruijn graph算法等[20]。目前的宏基因组聚类方法大多数是先将reads片段组装为重叠群片段,然后进行后续的聚类分析。因此重叠群是宏基因组分箱主要研究的数据对象,对重叠群进行分箱可以得到更多高质量的基因。同时基于宏基因组重叠群的分箱方法也有其局限性,因为reads组装工具会造成序列来自同一物种但包含不同菌株嵌合体的情况,组装出的重叠群片段为嵌合体,会对后续的分箱分析产生影响。
1.3 宏基因组分箱质量评估工具宏基因组分箱质量检测工具有MetaQuast[21]、BUSCO[22]、CheckM[23]等。其中CheckM是评估效果最好的宏基因组分箱质量检测工具之一。CheckM可以对宏基因组聚类后的分箱结果进行完整度和污染度的估算,得到分箱的质量信息。CheckM使用了参考基因组树来推断特定于某基因谱系中的标志基因,根据其标志基因的完整性和重复量推断该基因的完整度和污染度。一些研究会针对某些微生物具体的门类进行重新细化[24–25],当谱系发生改变后,其选择的标志基因集合需要随之改变。想要进一步提高CheckM的评估效果,可以使其包含来自其他谱系的额外参考基因。当待评估的宏基因组样本可能来自包含真菌和其他真核微生物的环境时,把真核微生物基因组整合到CheckM所使用的参考基因树中将会对基因评估有实质性的帮助。进一步探索CheckM的参数空间也会提高评估效果[23]。
宏基因组分箱方法的基准测试通常会在宏基因组模拟数据集以及真实数据集上进行。针对宏基因组模拟数据集评估分箱结果时,通常不会用CheckM等分箱质量评估工具进行分箱质量评估,这是因为在宏基因组模拟数据集中通常每个重叠群都有其对应的分类标签。根据预先知道的分类标签就可以对聚类后的分箱结果进行准确率和召回率的计算。在面对真实数据集时,由于事先不知道数据集中每个重叠群的分类标签,因而无法用在模拟数据集中采用的质量评估方法去计算真实数据集中分箱结果的准确率和召回率。这时通常会使用宏基因组分箱质量评估工具如CheckM来进行分箱质量的评估,它会针对每个分箱生成完整度和污染度信息,来近似表示分箱的准确率和召回率,从而完成对聚类结果的评估。
2 宏基因组数据分箱方法针对宏基因组分箱问题,国内外学者已经做了不少的工作。现有的宏基因组分箱方法根据数据对象的类别不同分为基于标志基因(如16S rRNA)序列[26–28]和基于全基因组序列的宏基因组样本的分箱[29–31]。基于标志基因序列的分箱方法只能对含有标志基因的序列进行分箱,常见的标志基因有16S rRNA、18S rRNA等。此类方法只能针对含有标志基因的序列进行分箱,而对于没有标志基因的序列则无能为力。而基于全基因组序列进行分箱可以对微生物的全部基因组序列进行分箱。相比基于标志基因序列的分类方法,这种方法的数据对象更全面,适用范围更广,同时也携带着更多关于微生物本身的信息。因此本综述主要针对基于全基因组序列的宏基因组分箱方法进行阐述。
如图 2所示,基于全基因组序列的宏基因组分箱方法可以划分为宏基因组数据分类方法以及宏基因组数据聚类方法。两类方法最大的区别有2个:(1) 宏基因组分类方法通常使用参考数据库进行分箱,而宏基因组数据聚类方法则较少使用;(2) 分类方法可以对宏基因组数据进行生物分类水平上的注释,而宏基因组聚类方法仅仅是将宏基因组数据进行分箱。在早期的宏基因组分箱研究中数据对象通常为reads片段,随着reads组装工具的发展,reads片段能被较为准确地组装成重叠群片段,对重叠群数据的分箱可以极大提高分箱表现。
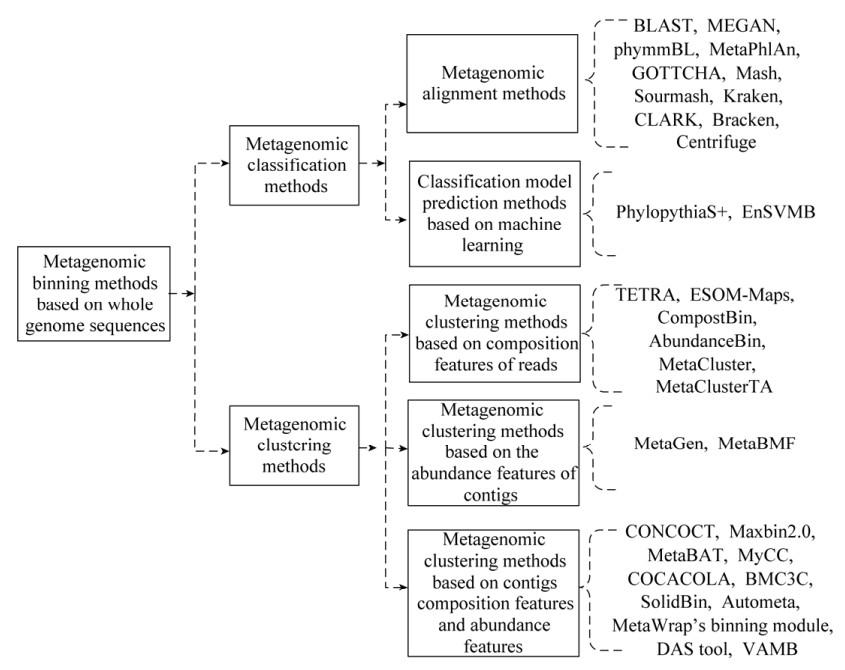
|
| 图 2 基于全基因组序列的宏基因组分箱方法概述 Figure 2 Overview of methods for metagenomic binning based on whole gene sequences. |
2.1 宏基因组数据分类方法
宏基因组数据分类主要依赖于参考数据库,主要分为比对方法以及构建分类模型预测方法[32–33]。
2.1.1 宏基因组数据比对方法在早期宏基因组分类时,一般采用BLAST工具[34]去比较宏基因组样本中的每个序列(通常为reads数据或者为组装后的重叠群数据)和GenBank数据库中的所有序列去得到宏基因组样本中每个序列的分类标签。基于BLAST方法并结合机器学习算法又产生出一些用于宏基因组数据分类的方法,如MEGAN[35]、PhymmBL[36]等。MEGAN使用BLAST方法将待预测序列与多个数据库进行比对,每个数据库最佳匹配序列的最小公共祖先即为待预测序列的分类标签。PhymmBL结合BLAST方法查询的结果以及用插值马尔可夫模型产生的分数,可以取得比单独使用BLAST方法更高的准确率。
随着新一代测序技术的逐渐发展,从环境中得到的宏基因组样本以及可用的参考数据库越来越大,使用BLAST进行一个个比对计算代价过于大,甚至不可能实现。于是出现了一大批基于标记基因的宏基因组数据分析方法,其相比BLAST这种传统比对方法速度要快且占用内存小。该种方法构建参考数据库时,没有像之前的比对方法去构建所有基因的数据库,而仅仅只是构建标志基因,如单一拷贝基因以及被发现对某种进化分支具有特异性的基因,因此构建数据库所需要存储空间少,比对时间较快。同时由于该数据库只包含标志基因以及一些具有特异性的基因,它们仅仅是基因的一小部分,因此宏基因组样本待预测DNA序列中,仅仅只有一小部分可以被分类。通常该类方法用于进行宏基因组样本的丰度分析,来表征宏基因组样本中的微生物分布情况,其无法对每一个read序列进行生物水平上的分类。
在人类基因工程的初始分析时,用MetaPhlAn[37]方法分析了从数百个人类样本中收集到的万亿碱基宏基因组序列。GOTTCHA[38]基于独特的24个碱基对片段,生成了包含独特基因标签的数据库,根据基因标签的覆盖度可以进行二元分类以及分类注释。以上这些分类方法虽然不能对宏基因组样本中每一个read或重叠群序列在生物分类水平上进行注释,但它们提供了宏基因组样本的丰度分析。Mash[39]和Sourmash[40]方法都使用了序列中的MinHash标签以分析序列之间的相似性,这种MinHash标签搜索方法,占用数据库空间小,可以很快地构建并搜索,使搜索者可以在笔记本电脑上对整个GenBank数据库进行搜索。
Kraken[41]是第一个为宏基因组样本中所有reads数据提供快速识别并注释的分箱方法。不同微生物基因的k-mer是不同的(k-mer即k个寡核苷酸组成的序列),长k-mer具有很强的物种特异性。基于此,Kraken构建了一个存储每个基因k-mer特征以及对应分类标签的数据库,通过比对宏基因组样本序列的k-mers特征与数据库的各个基因k-mers特征来得到每个宏基因组样本序列的分类标签。Kraken方法被提出之后,出现了一些与它相似的宏基因组样本分类方法以及Kraken方法的一些拓展,如CLARK[42],Bracken[43]等。CLARK与Kraken方法一样,利用k-mers进行序列的特征比对,其中k值的选取对比对结果有很大的影响,默认值为k=31。与Kraken不同的是,CLARK构建参考数据库时只对物种水平或者属水平上的k-mers进行分类标签的构建,丢弃了匹配到更高分类水平上的k-mers序列。使用CLARK方法对宏基因组样本进行分类时,可以将宏基因组序列分配到更低的分类水平上,以便于后续的微生物代谢以及群落分析。
Bracken方法是Kraken方法的扩展,它基于贝叶斯概率算法,可以估计宏基因组样本物种或者属水平上的丰度。这些方法可以对宏基因组样本中所有数据进行分类注释,相比于传统比对方法如BLAST,它们的索引速度更快同时所需存储空间较小。随着测序技术的发展以及参考数据库的逐渐庞大,基于k-mers比对查询方法的改进趋向于对数据库索引结构的改进。Centrifuge[44]利用Burrows-Wheeler转换和FM索引,以存储和索引基因数据库,使其可以快速准确地进行索引。对于同一数据库,Centrifuges使用的索引空间是Kraken的十分之一,大大减少了存储空间并且提升了索引速度,并且它使用MUMmer[45]可对来自密切相关基因中共享的序列去冗余,这进一步减少了索引数据结构的大小。
基于比对方法主要问题在于如果参考数据库里没有要查询的read序列或重叠群序列的信息时,那么就无法对其进行有效地分类。
2.1.2 基于机器学习的分类模型预测方法由于基于参考数据库比对的方法所需的计算代价和空间代价非常大,于是产生了基于机器学习的宏基因组数据分类预测方法。此类方法主要通过构建机器学习模型,用预先准备好的训练数据进行训练,其可以对宏基因组样本中的reads序列或组装后的重叠群序列进行分类预测。基于机器学习的分类模型预测方法与基于序列比对的分类方法的区别在于,基于机器学习的分类模型预测方法是免比对的,在对样本中的序列进行预测前,需要用提前准备好的训练数据进行模型训练。训练数据和所用模型的好坏直接影响预测效果。该方法一旦构建好分类模型,其宏基因组样本数据分类效率远高于比对方法并且所占用存储空间较小。
PhyloPythiaS+[46]方法可以从宏基因组样本中直接获取训练样本和标签,然后构建集成SVM分类器并进行训练,从而对宏基因组样本中的重叠群序列进行预测。EnSVMB[47]方法运用了集成学习的方法,构建了多个线性SVM分类器,将每个分类器的结果通过投票的方式整合在一起作为最终结果,然后对结果中无法分类的重叠群序列进行BLAST比对,从而提高分类注释效果。此类方法在特定环境下,比如人类肠道菌群,由于其更容易得到用于分类模型训练的训练数据及标签,效果会更好。在宏基因组数据分类中,集成SVM分类器往往比单一的SVM分类器有更好的分类效果。
深度学习算法近年来兴起并迅速发展,并已广泛应用于健康信息学等领域。深度学习算法的应用主要体现在对于疾病的预测,如基于宏基因组数据的结直肠癌疾病预测等[48]和基于标志基因的宏基因组分箱[49],而较少用于基于全基因组的宏基因组分箱。宏基因组样本数据包含大多数未知的微生物物种,缺少训练数据和标签,因此有监督机器学习方法和深度学习算法较少用于重叠群分箱。此外特定环境如人类肠道菌群,已经被人类广泛地进行研究,有较为完善的参考数据库,从参考数据库中可以获得较为可靠的训练数据和标签,用传统的机器学习方法便可以获得很好的分箱效果,而深度学习方法在基于宏基因组数据的疾病预测等问题上优势更加明显。
基于机器学习的分类模型预测方法主要问题在于,构建分类模型所需的训练数据和标签难以获得。当进行生态学研究时,由于来自环境中的宏基因组样本包含大量未知的基因,训练数据无从获得。
2.2 宏基因组数据聚类方法宏基因组数据聚类方法则可以一定程度上避免宏基因组分类方法的弊端,其可以在物种水平甚至是菌株水平上进行分箱。现有的宏基因组聚类方法根据宏基因组数据特征的不同,大体上分为3类:基于宏基因组reads组成特征的聚类方法、基于重叠群覆盖度特征的聚类方法和基于重叠群组成特征和覆盖度特征的聚类方法。
2.2.1 基于宏基因组reads组成特征的聚类方法在研究早期,对宏基因组数据聚类方法的研究主要是针对reads数据,即测序得到的原生数据(raw reads)经过质检筛选得到的短DNA片段,通常大小只有100 bp左右。早期的宏基因组聚类方法大多是基于宏基因组序列组成特征的聚类方法,如TETRA[50]、ESOM-Maps[51]、CompostBin[52]、AbundanceBin[53]、MetaCluster[54]等方法。其中,TETRA采用方法较为简单,它是通过计算reads的四联寡核苷酸频率并计算reads片段之间的距离,根据此信息将reads数据进行分箱。ESOM-Maps方法使用了自组织映射的方法进行聚类,采用的特征同样为reads的四联寡核苷酸频率。与TETRA、ESOM-Maps方法只采用reads序列的组成特征(四联寡核苷酸频率)不同,CompostBin采用了序列的不同长度寡核苷酸频率,然后使用带权重的基于PCA降维的策略对序列组成特征空间进行降维。通常情况下,宏基因组样本中包含微生物物种的丰度差异较大,像TETRA的宏基因组聚类方法,它们会将具有较大丰度的微生物物种聚类成多个分箱。AbundanceBin利用分离泊松分布对来自不同物种的reads数量进行建模,可以将具有相似丰度的reads数据分箱在一起,该方法采用的特征依然是序列的四联寡核苷酸频率。
MetaCluster有很多版本,从版本1.0到版本5.0等,到出现MetaClusterTA[55],它可以对宏基因组reads数据进行聚类并注释,是基于reads数据聚类方法中表现较好的宏基因组聚类注释工具。然而当宏基因组样本中基因数量超过10之后,它的聚类效果会明显下降。这些方法都使用宏基因组序列的寡核苷酸频率作为reads数据的组成特征来进行聚类,通常会使用四联寡核苷酸频率作为序列的组成特征。并且它们都基于来自同一基因的微生物DNA序列的寡核苷酸频率相似,而来自不同基因的微生物DNA序列的寡核苷酸频率差异较大这一观点进行特征选择。由于基因片段较短(100 bp左右),其包含遗传信息较少,因此这些方法的聚类效果极其有限,只能应用在简单的宏基因组数据集。
宏基因组组装工具的发展则打破了这一限制,从最初的组装工具如IDBA_UD[56]、Megahit[17]、Ray Meta[18]、MetaSPAdes[19]到最新的组装工具Strainberry[57]等,它们可以将来自同一基因的reads片段组装成较长的重叠群片段(contigs),长度可以达500 bp以上。重叠群数据的长度增加也就意味着其携带的遗传信息更多,用于聚类的效果也就会更好。聚类效果好的前提是需要对reads数据进行准确率较高的组装操作,这样生成的重叠群片段中出现嵌合体的概率也就越小。
在早期,由于宏基因组组装工具不能很好地对测序得到的reads数据进行组装,准确率较低,因此大多数宏基因组分箱方法的数据对象是reads数据,这就造成了上述所述的很多局限。随着宏基因组组装工具的准确率上升,准确率可以达到97%以上,并且最新的组装工具Strainberry甚至可以在菌株水平上进行组装。因此对组装后的重叠群数据进行分箱已经是宏基因组分箱的趋势,越来越多的宏基因组分箱工具采取对重叠群数据进行分箱,出现了基于宏基因组重叠群覆盖度特征的聚类方法以及基于宏基因组重叠群组成特征和覆盖度特征的聚类方法。
2.2.2 基于重叠群覆盖度特征的聚类方法最新的基于宏基因组重叠群覆盖度特征的聚类方法是MetaGen[58]和MetaBMF[2]。已经有研究表明[30],当有较大宏基因组样本可用时,基因的联合丰度特征即覆盖度特征(经比对后不同样本中覆盖重叠群序列每个碱基对的短reads的平均碱基数量)可以非常有效地进行不同基因的区分。MetaGen利用来自多个样本的相对丰度信息(即覆盖度信息),使用EM算法将重叠群聚类到不同的分箱中,并依靠贝叶斯信息准则(BIC)来确定样本中的基因数量。由于MetaGen仅使用跨样本的丰度模式进行分箱而不使用寡核苷酸分布模式,所以用于聚类的宏基因样本数量应大于10个。相对于大多数无监督宏基因组聚类方法而言,MetaGen不仅可以为低覆盖率的样本准确地聚类短重叠群,而且还具有区分拥有较高序列相似性物种的能力,同时该方法具有很高计算和内存花费。
为了将MetaGen应用到大规模宏基因组应用中,Ma等[2]提出了MetaBMF。MetaBMF计算每个重叠群上跨不同样本映射的reads片段的数量,并将其作为输入矩阵,提出了可扩展的分层角度回归算法。该算法可以将计数矩阵分解为二进制矩阵和非负矩阵的乘积,二进制矩阵可以用来分离微生物物种,非负矩阵可量化不同样本中的物种分布。该方法具有较高的分箱准确率以及快速计算的能力,可以在较短的时间内得到分箱结果。由于MetaBMF只使用重叠群序列的覆盖度特征,因此它的序列向量特征化时需要大量的宏基因组样本参与。与MetaGen局限性相同,如果2个物种在所有样本中的丰度几乎成比例,那么它们对应的重叠群序列将倾向于具有高度相似的样本特征描述,这使得此类算法难以区分这2个物种。并且该类方法需要大量宏基因组样本进行联合组装,菌株的相似性较高会使得组装的质量下降。
2.2.3 基于重叠群组成特征和覆盖度特征的聚类方法该类聚类方法是通过提取宏基因组重叠群的组成特征以及覆盖度特征来作为聚类使用的数据特征,然后通过机器学习算法进行聚类。目前此类方法主要包括CONCOCT[59]、Maxbin2.0[29]、MetaBAT[30]、MyCC[31]、COCACOLA[60]、BMC3C[61]、SolidBin[62]、Autometa[63]、metaWRAP分箱模块[64]、DAS工具[65]以及VAMB方法[66]。此类方法结合了2种特征的优点,被广泛应用在宏基因组研究的各个领域。为了使得这些宏基因组聚类方法的分箱效果有一个统一的评价标准,CAMI模拟数据集被提出[67],其可以作为宏基因组聚类方法的基准测试数据集。CAMI模拟数据集分为低复杂度数据集(40个基因20个圆形元素)、中等复杂度数据集(132个基因和100个圆形元素)以及高复杂度数据集(596个基因和478个圆形元素)。这些数据集来自700个分离的微生物和600个不同于公共基因组里的菌株、物种、属、科的圆形元素。它们充分模拟了真实环境下的情形,其中包括大量彼此密切相关的菌株、质粒以及病毒序列和真实的丰度描述。鉴于有一些研究将数量众多的宏基因组聚类方法放在CAMI数据集上进行比较,本节对宏基因组聚类方法的评价和比较在CAMI数据集上进行介绍[67–69]。
CONCOCT首次结合序列的组成特征(四联寡核苷酸频率)和联合丰度特征将序列特征向量化,然后利用主成分分析的方法进行降维。它使用高斯混合模型并结合EM算法进行聚类。该方法在CAMI低复杂度数据集上表现很好,但是在CAMI中等复杂度和高复杂度数据集上表现较差。Maxbin 2.0和MetaBAT都结合了序列的组成特征以及联合丰度,并通过预先训练的概率模型来计算每条序列到聚类中心点的概率,然后分别用改进后的最大期望值算法和k-medoid算法进行聚类。其中Maxbin2.0在CAMI中等复杂度数据集上表现很好,但是在高复杂度数据集上重建高质量基因数量会降低,同时无法应用到超高复杂度数据集,如含有人类肠道菌群的1 704个样本的数据集[70]。MetaBAT是专门为高复杂度数据集设计的算法,可以在超高复杂度数据集上取得很好的效果[30]。该方法的缺点是算法参数太多,针对不同的数据集要调参否则无法达到预期的效果。
MyCC结合了基因组特征,标志基因以及重叠群的覆盖度特征作为聚类使用的数据特征。该方法在CAMI低和中等复杂度数据集上表现得很好,但是在CAMI高复杂度数据集上表现大大下降。COCACOLA中相似性度量没有使用欧式距离而是使用L1距离,稀疏正则化的同时,结合了硬聚类和软聚类的优点,并且它还包含了先验知识来提高聚类准确率,和大多数方法一样无法在CAMI高复杂度数据集上获得很好的表现。Yu等[61]开发的BMC3C是一种集成聚类的方法,在选择数据特征时,除了利用DNA序列的组成特征、联合覆盖度特征,还包括密码子特征。通过多次采用不同的初始聚类中心进行多次的k-means聚类,然后从这些聚类簇中得到它们的权重图(每个节点代表一个重叠群),如果2个重叠群被频繁分箱到同一个簇中,那它们的权重就越高,否则就越低。最后采用图分割技术将权重图分割成不同的子图,每个子图代表一个基因分箱。这是宏基因组重叠群分箱第一次使用密码子特征以及集成聚类方法。
上面所述的重叠群聚类方法很少使用已知的一些重叠群序列之间的关系以及分类标识等额外的生物信息,仅仅使用了重叠群序列的覆盖度和序列组成信息。而SolidBin则是一种半监督的谱聚类方法。它构建了2种类型的先验信息:必须链接约束和不能链接约束。必须链接约束意味着某对重叠群序列应该被聚类到同一分箱,而不能链接的约束意味着某对重叠群序列应该被聚类到不同分箱。这些约束被整合到经典的谱聚类方法(归一化切割)去提升其聚类效果。当宏基因组样本复杂度较高且先验信息较少时,SolidBin的分箱表现不好[68]。
现有的重叠群聚类方法都存在真核生物污染等问题以及不能应对高复杂度单个宏基因组数据集。为了解决以上问题,提出了Autometa方法,它整合了序列同源性、核苷酸组成、覆盖度以及单拷贝标志基因等特征,将微生物基因和不需要进行建模的宿主基因以及其他真核生物污染区分开。其聚类过程中簇数个数的选取对该方法的效果有较大影响,并且该方法难以区分重叠群序列来自同一微生物物种但不同菌株的这一情况。
由于没有一种宏基因组分箱工具可以在所有的宏基因组样本分箱中表现都较好,所以便出现了集成的分箱工具如MetaWRAP中的分箱模块以及DAS工具等,它们采用了一种整合和去冗余的策略可以将不同分箱工具分箱后得到的结果进行整合,使其准确率和召回率更高,然而其性能主要取决于它们所包含子分箱工具的性能。
在VAMB方法中,使用了变分自编码器并结合序列的覆盖度特征和k-mer组成特征进行聚类。在VAMB方法出现之前,重叠群聚类方法中没有使用过深度学习方法。该方法在CAMI数据集上表现较好,同时可以分离相似的菌株。将VAMB方法作为DAS工具中一个子分箱工具,可以提高整体的分箱效果[66]。在做宏基因组数据分析时,可以选用MetaWRAP工具,该工具包含了宏基因组数据分析的整套流程,包括基因序列的质控、拼接组装、分箱以及注释等。在实现宏基因组数据聚类任务时,也可以选用DAS工具,DAS工具可以将其中包含的子分箱方法的结果进行整合,来提高分箱效果。在应用时可以将Maxbin 2.0、MetaBAT以及VAMB方法作为DAS工具的子分箱方法,对分箱结果进行集成从而提高分箱表现。
宏基因组数据聚类方法主要问题如下:(1) 对宏基因组序列来自同一物种但不同菌株的情况分箱效果不好;(2) 宏基因组样本中基因的个数难以确定,而这个参数对绝大多数宏基因组聚类方法而言极为关键;(3) 难以从宏基因组数据集中获得更多高质量的分箱。
3 宏基因组重叠群分箱方法存在的问题和挑战尽管宏基因组重叠群分箱方法研究取得了一系列的进展,但该方面仍存在着诸多的问题和挑战,主要包含宏基因组重叠群分类问题以及宏基因组重叠群聚类问题。其中,宏基因组重叠群分类问题主要包括:
(1) 当宏基因组样本数据量较大时,对其进行拼接,服务器内存的上限可能会成为瓶颈从而导致拼接无法完成。
(2) 当宏基因组数据集包含大量参考数据库没有的微生物遗传信息时,只基于参考数据库是无法进行准确研究的,如生态学等[71–72]。这方面的研究可以基于扩增子测序和分析以及宏基因组重叠群聚类等分析来进行研究。医学等方面则可以基于参考数据库进行非常精确的研究[73–74],这是由于肠道菌群的基因组被研究得较多,有较为完整的参考数据库。
宏基因组重叠群聚类问题主要包括:
(1) 宏基因组重叠群聚类方法很少使用已经构建的参考数据库,造成了一定程度的资源浪费,同时受限于方法自身所采用的序列特征以及聚类算法,聚类效果有限。目前尚没有任何一种宏基因组重叠群聚类方法可以在所有环境宏基因组数据集上表现都好,集成学习可以将不同的分箱方法集成在一起,互相取长补短从而提高分箱效果[65]。使用参考数据库中包含的信息,并将其应用于聚类过程中可以提高其分箱表现[62]。
(2) 同一物种但不同菌株的序列相似度很高,因此对于重叠群来自同一物种但不同菌株的情况分箱效果不理想。一种结合单细胞基因组学和宏基因组学的框架可以从微生物群落中重建出菌株水平的基因,该框架称为SMAGLinker[75]。该框架使用了微流体技术产生的单细胞扩增基因作为分箱指南,将其与宏基因组组装数据进行结合,从而提高分箱效果。
(3) 在CAMI高复杂度数据集上,大多数分箱方法的分箱表现并不好。通过使用集成学习方法以及深度学习方法可以提高分箱效果,如DAS工具[65]和VAMB方法[66]。我们通过结合分箱质量评估工具CheckM,提出了一种用于宏基因组重叠群数据的无监督聚类方法MetaCRS[69],其中采用的递归策略可以不断降低宏基因组样本的复杂度,进而提高聚类效果。同时还提出了一种新的宏基因组样本中选择簇个数的方法,可以将其应用到该聚类方法的初始化。
(4) 宏基因组样本中微生物基因的个数难以确定,而这个参数对绝大多数宏基因组聚类方法而言极为关键。建议可以使用qPCR方法对样本中基因个数进行分析。
4 总结与展望宏基因组分箱方法在宏基因组分析流程中有着承上启下的作用,对分箱方法的改进有助于进一步的数据挖掘和生命现象揭示。现有的宏基因组分箱方法存在着一些问题,亟待进一步改进。
未来宏基因组重叠群分箱方法的发展方向包括:
(1) 如何设计出对于宏基因分箱更具有分辨性的序列特征,可以使重叠群序列在较低生物水平上进行分箱(如菌株水平)。
(2) 使用集成学习和深度学习方法,并将参考数据库等信息应用到宏基因组重叠群分箱中去。
(3) 随着第三代测序技术的发展,有希望在未来能够快速且较为便宜地得到大量较长的reads片段。直接对长reads片段进行聚类或者有监督分类可以得到更多高质量的基因。
(4) 由于大量宏基因组样本数据的涌现,如何快速且准确地将宏基因组样本数据进行分箱是研究的热点和难点。
(5) 基于密度的聚类方法不需要事先指定宏基因组样本中基因的个数,可能对宏基因组数据分箱起到更好的效果。
| [1] | Gerritsen J, Smidt H, Rijkers GT, De Vos WM. Intestinal microbiota in human health and disease: the impact of probiotics. Genes & Nutrition, 2011, 6(3): 209-240. |
| [2] | Ma T, Xiao D, Xing X. MetaBMF: a scalable binning algorithm for large-scale reference-free metagenomic studies. Bioinformatics, 2019, 36(2): 356-363. |
| [3] | Huang YJ, Boushey HA. The microbiome in asthma. The Journal of Allergy and Clinical Immunology, 2015, 135(1): 25-30. DOI:10.1016/j.jaci.2014.11.011 |
| [4] | Huang YJ, Marsland BJ, Bunyavanich S, O'Mahony L, Leung DYM, Muraro A, Fleisher TA. The microbiome in allergic disease: current understanding and future opportunities—2017 PRACTALL document of the American Academy of Allergy, Asthma & Immunology and the European Academy of Allergy and Clinical Immunology. Journal of Allergy and Clinical Immunology, 2017, 139(4): 1099-1110. DOI:10.1016/j.jaci.2017.02.007 |
| [5] | Turnbaugh PJ, Ley RE, Mahowald MA, Magrini V, Mardis ER, Gordon JI. An obesity-associated gut microbiome with increased capacity for energy harvest. Nature, 2006, 444(7122): 1027-1031. DOI:10.1038/nature05414 |
| [6] | Severance EG, Yolken RH, Eaton WW. Autoimmune diseases, gastrointestinal disorders and the microbiome in schizophrenia: more than a gut feeling. Schizophrenia Research, 2016, 176(1): 23-35. DOI:10.1016/j.schres.2014.06.027 |
| [7] | Brown CT, Davis-Richardson AG, Giongo A, Gano KA, Crabb DB, Mukherjee N, Casella G, Drew JC, Ilonen J, Knip M, Hyöty H, Veijola R, Simell T, Simell O, Neu J, Wasserfall CH, Schatz D, Atkinson MA, Triplett EW. Gut microbiome metagenomics analysis suggests a functional model for the development of autoimmunity for type 1 diabetes. PLoS One, 2011, 6(10): e25792. DOI:10.1371/journal.pone.0025792 |
| [8] | Clemente JC, Ursell LK, Parfrey LW, Knight R. The impact of the gut microbiota on human health: an integrative view. Cell, 2012, 148(6): 1258-1270. DOI:10.1016/j.cell.2012.01.035 |
| [9] | Hentschel U, Piel J, Degnan SM, Taylor MW. Genomic insights into the marine sponge microbiome. Nature Reviews Microbiology, 2012, 10(9): 641-654. DOI:10.1038/nrmicro2839 |
| [10] |
Ding R, Chen XH, Li BX. Research advances on phytase and prospect of applying soil phytase. Biotechnology Bulletin, 2019, 35(7): 190-195.
(in Chinese) 丁锐, 陈旭辉, 李炳学. 植酸酶研究进展及土壤植酸酶应用展望. 生物技术通报, 2019, 35(7): 190-195. |
| [11] | Gardner SN, Frey KG, Redden CL, Thissen JB, Allen JE, Allred AF, Dyer MD, Mokashi VP, Slezak TR. Targeted amplification for enhanced detection of biothreat agents by next-generation sequencing. BMC Research Notes, 2015, 8: 682. DOI:10.1186/s13104-015-1530-0 |
| [12] | Zhou J, Xue K, Xie J, Deng Y, Wu L, Cheng X, Fei S, Deng S, He Z, Van Nostrand JD, Luo Y. Microbial mediation of carbon-cycle feedbacks to climate warming. Nature Climate Change, 2012, 2(2): 106-110. DOI:10.1038/nclimate1331 |
| [13] | Xing MN, Zhang XZ, Huang H. Application of metagenomic techniques in mining enzymes from microbial communities for biofuel synthesis. Biotechnology Advances, 2012, 30(4): 920-929. DOI:10.1016/j.biotechadv.2012.01.021 |
| [14] | Kellenberger E. Exploring the unknown.The silent revolution of microbiology. EMBO Reports, 2001, 2(1): 5-7. DOI:10.1093/embo-reports/kve014 |
| [15] | Riesenfeld CS, Schloss PD, Handelsman J. Metagenomics: genomic analysis of microbial communities. Annual Review of Genetics, 2004, 38: 525-552. DOI:10.1146/annurev.genet.38.072902.091216 |
| [16] | 梁艺馨. 基于改进密度峰值的宏基因组重叠群聚类算法研究. 吉林大学硕士学位论文, 2020. |
| [17] | Li DH, Liu CM, Luo RB, Sadakane K, Lam TW. MEGAHIT: an ultra-fast single-node solution for large and complex metagenomics assembly via succinct de Bruijn graph. Bioinformatics, 2015, 31(10): 1674-1676. DOI:10.1093/bioinformatics/btv033 |
| [18] | Boisvert S, Raymond F, Godzaridis E, Laviolette F, Corbeil J. Ray meta: scalable de novo metagenome assembly and profiling. Genome Biology, 2012, 13(12): R122. DOI:10.1186/gb-2012-13-12-r122 |
| [19] | Nurk S, Meleshko D, Korobeynikov A, Pevzner PA. metaSPAdes: a new versatile metagenomic assembler. Genome Research, 2017, 27(5): 824-834. DOI:10.1101/gr.213959.116 |
| [20] | 张安琪. 面向宏基因组数据的拼接算法研究. 哈尔滨工业大学硕士学位论文, 2018. |
| [21] | Mikheenko A, Saveliev V, Gurevich A. MetaQUAST: evaluation of metagenome assemblies. Bioinformatics, 2015, 32(7): 1088-1090. |
| [22] | Seppey M, Manni M, Zdobnov EM. BUSCO: assessing genome assembly and annotation completeness. Methods in Molecular Biology: Clifton, NJ, 2019, 1962: 227-245. |
| [23] | Parks DH, Imelfort M, Skennerton CT, Hugenholtz P, Tyson GW. CheckM: assessing the quality of microbial genomes recovered from isolates, single cells, and metagenomes. Genome Research, 2015, 25(7): 1043-1055. DOI:10.1101/gr.186072.114 |
| [24] | Rinke C, Rubino F, Messer LF, Youssef N, Parks DH, Chuvochina M, Brown M, Jeffries T, Tyson GW, Seymour JR, Hugenholtz P. A phylogenomic and ecological analysis of the globally abundant marine group Ⅱ archaea (Ca. Poseidoniales ord. nov.). The ISME Journal, 2019, 13(3): 663-675. DOI:10.1038/s41396-018-0282-y |
| [25] | Liu Y, Makarova KS, Huang WC, Wolf YI, Nikolskaya AN, Zhang X, Cai M, Zhang CJ, Xu W, Luo Z, Cheng L, Koonin EV, Li M. Expanded diversity of Asgard archaea and their relationships with eukaryotes. Nature, 2021, 593(7860): 553-557. DOI:10.1038/s41586-021-03494-3 |
| [26] | Callahan BJ, McMurdie PJ, Rosen MJ, Han AW, Johnson AJA, Holmes SP. DADA2: high-resolution sample inference from Illumina amplicon data. Nature Methods, 2016, 13(7): 581-583. DOI:10.1038/nmeth.3869 |
| [27] | Amir A, McDonald D, Navas-Molina JA, Kopylova E, Morton JT, Xu ZZ, Kightley EP, Thompson LR, Hyde ER, Gonzalez A, Knight R. Deblur rapidly resolves single-nucleotide community sequence patterns. mSystems, 2017, 2(2): e00191-e00116. |
| [28] | Edgar RC. UPARSE: highly accurate OTU sequences from microbial amplicon reads. Nature Methods, 2013, 10(10): 996-998. DOI:10.1038/nmeth.2604 |
| [29] | Wu YW, Simmons BA, Singer SW. MaxBin 2.0:an automated binning algorithm to recover genomes from multiple metagenomic datasets. Bioinformatics, 2015, 32(4): 605-607. |
| [30] | Kang DD, Froula J, Egan R, Wang Z. MetaBAT, an efficient tool for accurately reconstructing single genomes from complex microbial communities. PeerJ, 2015, 3: e1165. DOI:10.7717/peerj.1165 |
| [31] | Lin HH, Liao YC. Accurate binning of metagenomic contigs via automated clustering sequences using information of genomic signatures and marker genes. Scientific Reports, 2016, 6: 24175. DOI:10.1038/srep24175 |
| [32] | Breitwieser FP, Lu J, Salzberg SL. A review of methods and databases for metagenomic classification and assembly. Briefings in Bioinformatics, 2017, 20(4): 1125-1136. |
| [33] | Mande SS, Mohammed MH, Ghosh TS. Classification of metagenomic sequences: methods and challenges. Briefings in Bioinformatics, 2012, 13(6): 669-681. DOI:10.1093/bib/bbs054 |
| [34] | Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ. Basic local alignment search tool. Journal of Molecular Biology, 1990, 215(3): 403-410. DOI:10.1016/S0022-2836(05)80360-2 |
| [35] | Huson DH, Auch AF, Qi J, Schuster SC. MEGAN analysis of metagenomic data. Genome Research, 2007, 17(3): 377-386. DOI:10.1101/gr.5969107 |
| [36] | Brady A, Salzberg SL. Phymm and PhymmBL: metagenomic phylogenetic classification with interpolated Markov models. Nature Methods, 2009, 6(9): 673-676. DOI:10.1038/nmeth.1358 |
| [37] | Liu B, Gibbons T, Ghodsi M, Treangen T, Pop M. Accurate and fast estimation of taxonomic profiles from metagenomic shotgun sequences. BMC Genomics, 2011, 12(S2): S4. DOI:10.1186/1471-2164-12-S2-S4 |
| [38] | Freitas TAK, Li PE, Scholz MB, Chain PSG. Accurate read-based metagenome characterization using a hierarchical suite of unique signatures. Nucleic Acids Research, 2015, 43(10): e69. DOI:10.1093/nar/gkv180 |
| [39] | Ondov BD, Treangen TJ, Melsted P, Mallonee AB, Bergman NH, Koren S, Phillippy AM. Mash: fast genome and metagenome distance estimation using MinHash. Genome Biology, 2016, 17(1): 132. DOI:10.1186/s13059-016-0997-x |
| [40] | Pierce NT, Irber L, Reiter T, Brooks P, Brown CT. Large-scale sequence comparisons with sourmash. F1000Research, 2019, 8: 1006. DOI:10.12688/f1000research.19675.1 |
| [41] | Davis MPA, Van Dongen S, Abreu-Goodger C, Bartonicek N, Enright AJ. Kraken: a set of tools for quality control and analysis of high-throughput sequence data. Methods, 2013, 63(1): 41-49. DOI:10.1016/j.ymeth.2013.06.027 |
| [42] | Ounit R, Wanamaker S, Close TJ, Lonardi S. CLARK: fast and accurate classification of metagenomic and genomic sequences using discriminative k-mers. BMC Genomics, 2015, 16(1): 236. DOI:10.1186/s12864-015-1419-2 |
| [43] | Lu J, Breitwieser FP, Thielen P, Salzberg SL. Bracken: estimating species abundance in metagenomics data. PeerJ Computer Science, 2017, 3: e104. DOI:10.7717/peerj-cs.104 |
| [44] | Kim D, Song L, Breitwieser FP, Salzberg SL. Centrifuge: rapid and sensitive classification of metagenomic sequences. Genome Research, 2016, 26(12): 1721-1729. DOI:10.1101/gr.210641.116 |
| [45] | Delcher AL, Phillippy A, Carlton J, Salzberg SL. Fast algorithms for large-scale genome alignment and comparison. Nucleic Acids Research, 2002, 30(11): 2478-2483. DOI:10.1093/nar/30.11.2478 |
| [46] | Gregor I, Dröge J, Schirmer M, Quince C, McHardy AC. PhyloPythiaS+: a self-training method for the rapid reconstruction of low-ranking taxonomic bins from metagenomes. PeerJ, 2016, 4: e1603. DOI:10.7717/peerj.1603 |
| [47] | Jiang Y, Wang J, Xia D, Yu G. EnSVMB: metagenomics fragments classification using ensemble SVM and BLAST. Scientific Reports, 2017, 7: 9440. DOI:10.1038/s41598-017-09947-y |
| [48] |
Li Q, Yi Y, Wu ZD, Ding T. Review of gut microbiome analysis prediction models and algorithms. Microbiology China, 2021, 48(1): 180-196.
(in Chinese) 李强, 衣杨, 吴忠道, 丁涛. 基于机器学习的肠道菌群数据建模与分析研究综述. 微生物学通报, 2021, 48(1): 180-196. |
| [49] | Fiannaca A, La Paglia L, La Rosa M, Lo Bosco G, Renda G, Rizzo R, Gaglio S, Urso A. Deep learning models for bacteria taxonomic classification of metagenomic data. BMC Bioinformatics, 2018, 19(S7): 198. DOI:10.1186/s12859-018-2182-6 |
| [50] | Teeling H, Waldmann J, Lombardot T, Bauer M, Glöckner FO. TETRA: a web-service and a stand-alone program for the analysis and comparison of tetranucleotide usage patterns in DNA sequences. BMC Bioinformatics, 2004, 5: 163. DOI:10.1186/1471-2105-5-163 |
| [51] | Ultsch A, Morchen F. ESOM-Maps: tools for clustering, visualization, and classification with Emergent SOM. Technical Report. Vol. 46. Germany: Department of Mathematics and Computer Science, University of Marburg, 2005. |
| [52] | Chatterji S, Yamazaki I, Bai ZJ, Eisen JA. CompostBin: a DNA composition-based algorithm for binning environmental shotgun reads. Lecture notes in computer science. Berlin, Heidelberg: Springer Berlin Heidelberg, 2008: 17-28. |
| [53] | Wu YW, Ye YZ. A novel abundance-based algorithm for binning metagenomic sequences using l-tuples. Journal of Computational Biology: a Journal of Computational Molecular Cell Biology, 2011, 18(3): 523-534. DOI:10.1089/cmb.2010.0245 |
| [54] | Leung HCM, Yiu SM, Yang B, Peng Y, Wang Y, Liu ZH, Chen JC, Qin JJ, Li RQ, Chin FYL. A robust and accurate binning algorithm for metagenomic sequences with arbitrary species abundance ratio. Bioinformatics, 2011, 27(11): 1489-1495. DOI:10.1093/bioinformatics/btr186 |
| [55] | Wang Y, Leung H, Yiu S, Chin F. MetaCluster-TA: taxonomic annotation for metagenomic data based on assembly-assisted binning. BMC Genomics, 2014, 15(Suppl 1): S12. |
| [56] | Peng Y, Leung HCM, Yiu SM, Chin FYL. IDBA-UD: a de novo assembler for single-cell and metagenomic sequencing data with highly uneven depth. Bioinformatics, 2012, 28(11): 1420-1428. DOI:10.1093/bioinformatics/bts174 |
| [57] | Vicedomini R, Quince C, Darling AE, Chikhi R. Strainberry: automated strain separation in low-complexity metagenomes using long reads. Nature Communications, 2021, 12: 4485. DOI:10.1038/s41467-021-24515-9 |
| [58] | Xing X, Liu JS, Zhong WX. MetaGen: reference-free learning with multiple metagenomic samples. Genome Biology, 2017, 18(1): 187. DOI:10.1186/s13059-017-1323-y |
| [59] | Alneberg J, Bjarnason BS, De Bruijn I, Schirmer M, Quick J, Ijaz UZ, Lahti L, Loman NJ, Andersson AF, Quince C. Binning metagenomic contigs by coverage and composition. Nature Methods, 2014, 11(11): 1144-1146. DOI:10.1038/nmeth.3103 |
| [60] | Lu YY, Chen T, Fuhrman JA, Sun FZ. COCACOLA: binning metagenomic contigs using sequence composition, read coverage, co-alignment and paired-end read linkage. Bioinformatics, 2016, 33(6): 791-798. |
| [61] | Yu GX, Jiang Y, Wang J, Zhang H, Luo HW. BMC3C: binning metagenomic contigs using codon usage, sequence composition and read coverage. Bioinformatics, 2018, 34(24): 4172-4179. |
| [62] | Wang ZY, Wang ZY, Lu YY, Sun FZ, Zhu SF. SolidBin: improving metagenome binning with semi-supervised normalized cut. Bioinformatics, 2019, 35(21): 4229-4238. DOI:10.1093/bioinformatics/btz253 |
| [63] | Miller IJ, Rees ER, Ross J, Miller I, Baxa J, Lopera J, Kerby RL, Rey FE, Kwan JC. Autometa: automated extraction of microbial genomes from individual shotgun metagenomes. Nucleic Acids Research, 2019, 47(10): e57. DOI:10.1093/nar/gkz148 |
| [64] | Uritskiy GV, DiRuggiero J, Taylor J. MetaWRAP-a flexible pipeline for genome-resolved metagenomic data analysis. Microbiome, 2018, 6(1): 158. DOI:10.1186/s40168-018-0541-1 |
| [65] | Sieber CMK, Probst AJ, Sharrar A, Thomas BC, Hess M, Tringe SG, Banfield JF. Recovery of genomes from metagenomes via a dereplication, aggregation and scoring strategy. Nature Microbiology, 2018, 3(7): 836-843. DOI:10.1038/s41564-018-0171-1 |
| [66] | Nissen JN, Johansen J, Allesøe RL, Sønderby CK, Armenteros JJA, Grønbech CH, Jensen LJ, Nielsen HB, Petersen TN, Winther O, Rasmussen S. Improved metagenome binning and assembly using deep variational autoencoders. Nature Biotechnology, 2021, 39(5): 555-560. DOI:10.1038/s41587-020-00777-4 |
| [67] | Sczyrba A, Hofmann P, Belmann P, Koslicki D, Janssen S, Dröge J, Gregor I, Majda S, Fiedler J, Dahms E, Bremges A, Fritz A, Garrido-Oter R, Jørgensen TS, Shapiro N, Blood PD, Gurevich A, Bai Y, Turaev D, DeMaere MZ, Chikhi R, Nagarajan N, Quince C, Meyer F, Balvočiūtė M, Hansen LH, Sørensen SJ, Chia BKH, Denis B, Froula JL, Wang Z, Egan R, Don Kang D, Cook JJ, Deltel C, Beckstette M, Lemaitre C, Peterlongo P, Rizk G, Lavenier D, Wu YW, Singer SW, Jain C, Strous M, Klingenberg H, Meinicke P, Barton MD, Lingner T, Lin HH, Liao YC, Silva GGZ, Cuevas DA, Edwards RA, Saha S, Piro VC, Renard BY, Pop M, Klenk HP, Göker M, Kyrpides NC, Woyke T, Vorholt JA, Schulze-Lefert P, Rubin EM, Darling AE, Rattei T, McHardy AC. Critical assessment of metagenome interpretation—a benchmark of metagenomics software. Nature Methods, 2017, 14(11): 1063-1071. DOI:10.1038/nmeth.4458 |
| [68] | Yue Y, Huang H, Qi Z, Dou HM, Liu XY, Han TF, Chen Y, Song XJ, Zhang YH, Tu J. Evaluating metagenomics tools for genome binning with real metagenomic datasets and CAMI datasets. BMC Bioinformatics, 2020, 21(1): 334. DOI:10.1186/s12859-020-03667-3 |
| [69] | Jiang ZJ, Li XB, Guo LJ. MetaCRS: unsupervised clustering of contigs with the recursive strategy of reducing metagenomic dataset's complexity. BMC Bioinformatics, 2022, 22(Suppl 12): 315. |
| [70] | Nielsen HB, Almeida M, Juncker AS, Rasmussen S, Li J, Sunagawa S, Plichta DR, Gautier L, Pedersen AG, Le Chatelier E, Pelletier E, Bonde I, Nielsen T, Manichanh C, Arumugam M, Batto JM, Quintanilha Dos Santos MB, Blom N, Borruel N, Burgdorf KS, Boumezbeur F, Casellas F, Doré J, Dworzynski P, Guarner F, Hansen T, Hildebrand F, Kaas RS, Kennedy S, Kristiansen K, Kultima JR, Léonard P, Levenez F, Lund O, Moumen B, Le Paslier D, Pons N, Pedersen O, Prifti E, Qin J, Raes J, Sørensen S, Tap J, Tims S, Ussery DW, Yamada T, Renault P, Sicheritz-Ponten T, Bork P, Wang J, Brunak S, Ehrlich SD. Identification and assembly of genomes and genetic elements in complex metagenomic samples without using reference genomes. Nature Biotechnology, 2014, 32(8): 822-828. DOI:10.1038/nbt.2939 |
| [71] | Namirimu T, Kim YJ, Park MJ, Lim D, Lee JH, Kwon KK. Microbial community structure and functional potential of deep-sea sediments on low activity hydrothermal area in the central Indian ridge. Frontiers in Marine Science, 2022, 9: 784807. DOI:10.3389/fmars.2022.784807 |
| [72] | Valadez-Cano C, Hawkes K, Calvaruso R, Reyes-Prieto A, Lawrence J. Amplicon-based and metagenomic approaches provide insights into toxigenic potential in understudied Atlantic Canadian lakes. FACETS, 2022, 7: 194-214. DOI:10.1139/facets-2021-0109 |
| [73] |
Chen X, Xue Y, Song XF, Zhu BL. Gut microbiota in diabetic patients and diabetic patients with cardiovascular complications. Acta Microbiologica Sinica, 2019, 59(9): 1660-1673.
(in Chinese) 陈茜, 薛勇, 宋晓峰, 朱宝利. 糖尿病及糖尿病心血管并发症患者肠道菌群的特征. 微生物学报, 2019, 59(9): 1660-1673. |
| [74] |
Wang W, Shang XX, Zeng QY, Liu DD, Li BB, Zhang JQ, Yang HY, Huang YL, Hu W, Fu JJ, Xu JH. Characteristics of intestinal microflora and biochemical immune molecules in patients with polycystic ovarian syndrome. Acta Microbiologica Sinica, 2021, 61(2): 452-468.
(in Chinese) 汪湾, 尚潇潇, 曾秋耀, 刘冬冬, 李贝贝, 张嘉琪, 杨洪艳, 黄译乐, 胡薇, 傅锦坚, 徐建华. 多囊卵巢综合征患者肠道菌群和生化免疫分子特征. 微生物学报, 2021, 61(2): 452-468. |
| [75] | Arikawa K, Ide K, Kogawa M, Saeki T, Yoda T, Endoh T, Matsuhashi A, Takeyama H, Hosokawa M. Recovery of strain-resolved genomes from human microbiome through an integration framework of single-cell genomics and metagenomics. Microbiome, 2021, 9(1): 202. |
 2022, Vol. 62
2022, Vol. 62