中国科学院微生物研究所,中国微生物学会
文章信息
- 王加龙, 刘驰, 雷丽, 李香真, 姚敏杰. 2022
- WANG Jialong, LIU Chi, LEI Li, LI Xiangzhen, YAO Minjie.
- 非共生固氮菌及其固氮作用
- Asymbiotic nitrogen-fixing bacteria and their nitrogen fixation potential
- 微生物学报, 62(8): 2861-2878
- Acta Microbiologica Sinica, 62(8): 2861-2878
-
文章历史
- 收稿日期:2021-11-14
- 修回日期:2022-01-27
- 网络出版日期:2022-03-18
2. 中国科学院成都生物研究所,环境与应用微生物重点实验室,环境微生物四川省重点实验室,四川 成都 610041
2. Key Laboratory of Environmental and Applied Microbiology, Sichuan Key Laboratory of Environmental Microbiology, Chengdu Institute of Biology, Chinese Academy of Sciences, Chengdu 610041, Sichuan, China
氮素在植物生长过程中起着重要的作用,植物可以利用某些原核微生物来获取大气中的分子氮。这些微生物通过固氮酶的作用,将大气氮转化成植物可以直接利用的铵态氮。根据固氮微生物的特点及其与植物的关系,通常把固氮微生物分为共生固氮菌、自生固氮菌和联合固氮菌,后两者统称为非共生固氮菌。虽然在大多数生态系统中共生固氮菌的固氮量要大于非共生固氮菌固氮量,但近年来的研究表明, 非共生固氮对自然界的氮素循环也发挥着很大的作用,每年每公顷固氮量在0.01–60.00 kg范围内,甚至在温带草地和热带常绿森林生态系统中,非共生固氮的作用也占主导地位[1–2]。全球陆地生态系统每年生物固氮量约110 Tg,其中非共生固氮量约占陆地生物固氮量的一半[3]。非共生固氮微生物固氮速率虽然不高,但其分布广泛,各生态系统中均存在种类众多的非共生固氮菌群落[4]。随着测序技术的发展和检测固氮酶活性方法的改进,非共生固氮的研究得到广泛关注。
非共生固氮在全球各生态系统氮素循环过程中发挥着重要作用,影响非共生固氮作用的因素众多,如气候因素、地理位置、土壤性质、植物种类、植物不同部位、农业措施、养分添加等[5–6]。在生态系统的不断演替中,驱动非共生固氮菌群变化的关键因子具有不确定性,单一因素对非共生固氮菌群的影响是暂时的,应综合考虑气候因子和各种环境参数等对非共生固氮作用的综合影响。
对于非共生固氮微生物,不同文献有着不同的定义。本文将非共生固氮微生物定义为:除与豆科植物共生固氮的根瘤菌外,具有独立或半独立固氮能力的微生物,包括土壤中自生或联合固氮微生物、具有固氮能力的藻类生物[7]。本文将总结非共生固氮微生物的分类、系统发育及其群落构建机制,同时对不同生态系统、植物不同部位和土壤中的非共生固氮微生物群落的组成和固氮量进行比较,并归纳了影响非共生固氮微生物群落组成和固氮潜势的影响因素,列举了测定固氮菌和固氮潜势的常用分析方法,最后对非共生固氮菌研究中存在的问题及发展趋势进行展望,目的是对非共生固氮菌的研究现状和发展有个比较全面系统的认识。
1 非共生固氮菌的分类 1.1 自生固氮自生固氮菌是指各种自由生活、能独立固定大气氮的原核生物。自从微生物学家巴斯德从土壤中分离出第一株自生固氮菌—巴斯德梭菌属(Pasteurella)后,人们在土壤、植物根际、叶际和根内等分离到多种非共生固氮菌属[5]。常见的自生固氮菌有固氮菌属(Azotobacter)、梭菌属(Clostridium)、拜叶林克氏菌属(Beijerinckia)、具有异形胞的蓝藻门(Cyanobacteria)中的念珠藻属(Nostoc)和颤藻属(Oscillaria)[8]等。按照代谢类型可分为三类[9–12]:
(1) 光能自养固氮菌:以二氧化碳为碳源进行光合作用、以光合产物为能源进行固氮作用的微生物。代表种类有念珠藻属、鱼腥藻属(Anabaena)和绿硫菌属(Clathrochloris)等。
(2) 化能自养固氮菌:有些化能自养微生物,如氧化亚铁硫杆菌(Thiobacillus ferrooxidans)、硫氧化细菌(Azospirillum thiophilum)和盐生硫杆菌属(Halothiobacillus sp.)等,利用氧化还原反应所提供的能量进行固氮作用。
(3) 异养固氮菌:以适宜的有机碳化合物为碳源和能源,满足自身生活和固氮需要。异养固氮菌种类众多,如芽孢杆菌属(Bacillus)、兼性厌氧性的克雷伯氏菌属(Klebsiella)和红螺菌属(Rhodospirillum)、好氧性的固氮菌属、专性厌氧性的巴氏梭菌(Clostridium pasteurianum)等。
1.2 联合固氮联合固氮是指固氮微生物在植物根表或根皮层处进行固氮,但不会像根瘤菌与豆科植物形成共生结构的固氮方式。1976年,Baldani等通过比较禾本植物与豆科植物的固氮方式发现,有一类固氮微生物大多生活在植物根表,与植物关系密切,互相利用,但不与宿主植物形成特异分化结构,因此提出“联合固氮”的概念[13]。我们讨论的联合固氮菌包括定殖于植物根表的附生固氮菌(Epiphytic diazotrophs)和植物体内的内生固氮菌(Endophytic diazotrophs)。常见的附生固氮菌有鱼腥藻属、类芽孢杆菌属(Paenibacillus)、施氏假单胞菌(Pseudomonas stutzeri)等,常见的内生固氮菌有草螺菌属(Herbaspirillum)和雀稗属(Paspalum)等[14–15]。玉米的气生根中含有丰富的联合固氮菌,如伯克氏菌科(Burkholderiaceae)、假单胞菌科(Pseudomonadaceae)和固氮螺菌属(Azospirillum)等。一种墨西哥品种玉米的生物固氮量大于植物总吸收氮的80%[16],这主要由于玉米的气生根在下雨后可分泌大量粘液,这些粘液富含多糖(如木糖、半乳糖、阿拉伯糖和海藻糖等),给固氮菌提供了一个很好的富碳、低氧和低氮环境来发挥固氮作用[17]。大多数联合固氮作用发生在海洋、森林、农田和草原等生态系统中。
1.3 系统发育固氮酶是生物固氮的催化剂,由固氮基因nif所编码,是微生物固氮的遗传基础。2012年的一项研究通过对当时已有基因组测序的原核微生物(共1 002个,包括细菌和古菌)进行分析,发现有大约15%的种(共149个)有相对完整的nif基因簇[18]。与固氮酶有关的基因有:nifHDK、nifS、nifU、nifB、nifE、nifN、nifV、nifZ、nifQ、nifW和nifJ等,6个保守基因:nifH、nifD、nifK、nifE、nifN和nifB是固氮所必需的[19–20]。nifH是基因编码氮固定过程中的关键酶—二氮酶还原酶,而且具有高度保守性、分布广泛等特点,所以nifH基因可作为分子标记物检测固氮微生物的多样性和系统发育[21–22]。图 1展示的系统发育树是由NifH和NifD蛋白及其同源物序列构建而成,同时也支持替代固氮酶(钒固氮酶和铁固氮酶)最早由钼固氮酶进化而来[2, 23]。Ⅰ–Ⅲ组都是功能性固氮酶,其中Ⅰ组为典型的Mo-Fe固氮酶,主要由变形菌门(Proteobacteria)和蓝藻门组成,由于Mo酶能量利用率高,被认为是自然界中固氮效率最高的同工酶;Ⅱ组为厌氧Mo-Fe固氮酶,包括梭状芽孢杆菌属(Clostridia)、醋酸菌属(Acetobacter)和几种产甲烷菌(Methanogens);Ⅲ组为替代固氮酶(V-Fe固氮酶、Fe-Fe固氮酶),包括棕色固氮菌(Azotobacter vinelandii)、产酸克雷伯氏杆菌(Klebsiella oxytoca)等。Mo酶的含量在自然生态系统土壤中非常低,因此替代固氮酶在生态系统中是同等重要的;Ⅳ组由未被鉴定nifH和nifD基因编码的固氮酶同系物组成;Ⅴ组由与光合色素生物合成有关酶的亚基组成[6, 24]。
2 非共生固氮菌的群落构建
在宏观生态学理论中,群落的构建机制是生物多样性产生和维持的核心研究内容,研究非共生固氮菌的群落构建对揭示非共生固氮作用过程、固氮量的多少及动态变化是非常重要的。常用生态位理论和中性理论来解释群落的构建过程,生态位理论强调确定性过程(如环境过滤、生物之间的捕食、竞争和共存等)在群落构建过程中的作用,而中性理论主要强调随机性过程(如扩散限制、迁入和迁出概率、生态漂变等)的作用[25–26]。生态位和中性理论争论的核心问题是生态位分化和随机作用在群落构建中的相对贡献大小的问题[27]。研究表明,非共生固氮微生物群落多样性在不同区域不同条件下受随机性过程和确定性过程的影响不同[28]。Wang等对青藏高原高寒草甸土壤非共生固氮菌群落多样性和组成进行了研究,发现土壤pH是影响高寒草甸土壤非共生固氮菌群多样性的主要因素,不同的固氮菌群对pH的响应模式是不同的,其中由pH过滤的确定性过程在非共生固氮菌的群落构建过程中发挥着主要作用[29]。但对内蒙古草原土壤非共生固氮菌的研究发现,扩散限制和异质性选择(变量选择)在非共生固氮菌群落构建过程中起着主要作用[30]。Fan等对华北平原小麦土壤中固氮菌群的研究发现,在中性pH (6.5–7.5)范围内,小麦根际土的非共生固氮菌的群落构建由随机性过程占主导,而在酸性pH (4.5–6.5)或碱性pH (7.5–8.5)条件下,非共生固氮菌的群落构建由确定性过程占主导[31]。Wang等研究发现,我国草原土壤中固氮微生物β多样性具有明显的空间分布差异,其驱动因素为区域物种库和局域生态构建过程。首先草原土壤固氮微生物的群落构建受其区域物种库的限制,进一步经过环境异质性选择和扩散限制等的筛选,形成当地特有的微生物群落,最后物种间相互作用(如竞争等)也调控着固氮微生物的群落构建过程[32]。在垂直梯度带上,海拔对微生物群落构建过程也发挥着重要作用,研究表明,随着贡嘎山海拔梯度的变化,确定性和随机性过程对非共生固氮菌群落构建的相对贡献也是变化的。其中,随着海拔升高,确定性过程对非共生固氮菌群落组成的作用是先降低后升高,随机性过程在中海拔地区起主要作用[33]。研究生态位理论和中性理论,有助于我们理解确定性过程和随机性过程在非共生固氮菌的群落构建和维持生物多样性中的重要作用。
3 不同生态系统非共生固氮菌及固氮潜力气候、植被和土壤性质等的特异性导致不同生态系统拥有其独特的固氮微生物群落。草地类型决定着土壤固氮微生物群落组成,在中国呼伦贝尔草地生态系统中,克氏针茅草地土壤中非共生固氮菌群占优势,主要有红假单胞菌属(Rhodopseudomonas)、固氮螺菌属和伪枝藻菌属(Scytonema)等,而共生固氮菌为线叶菊草原、羊草草原和大针茅草原优势菌群[34]。据估算,内蒙古退化草地中生物土壤结皮(以蓝藻和地衣为主)每年固氮量约在13.0–129.9 kg N/hm2 [35]。非共生固氮是森林生态系统氮素的重要来源之一,土壤、森林凋落物、植物表面、苔藓和地衣等活跃着大量的非共生固氮菌群,如念珠藻属和单歧藻属(Tolypothrix)等[36]。据估算美国波多黎各低海拔森林(260 m)每年的非共生固氮量为(12.3±2.7) kg N/hm2,高海拔森林(640 m)每年的非共生固氮量为(8.4±1.4) kg N/hm2[37]。在海洋生态系统中,大西洋每年固氮量约为3.3 kg N/hm2,全球海洋每年固氮量约为6.0 kg N/hm2,其中蓝藻被认为是最主要的固氮菌群,利用分子生物学和基因组学方法证实,在海洋中存在一类特殊的固氮蓝藻—UCYN-A (Candidatus Atelocyanobacterium thalassa),它不能像蓝藻一样进行光合作用,但也能固氮[38]。海洋中除了蓝藻门含有大量的固氮菌属,如束毛藻属(Trichodesmium),在变形菌门、厚壁菌门(Firmicutes)和浮霉菌门(Planctomycetes)中也存在着固氮菌属,如脱硫弧菌属(Desulfovibrio)等[39]。农田生态系统中非共生固氮更加普遍,1961–2010年间,全球谷类作物氮的24% (共370 Tg)来自非共生固氮,其中种植小麦、玉米和水稻的土壤每年的非共生固氮量约为13、13和22 kg N/hm2 [40]。农田中最为普遍的非共生固氮微生物多为藻类,其中蓝藻门中的颤藻目(Oscillatoriales)和念珠藻目是水稻田中重要的固氮者[41]。本文整理了重要的生态系统和生态位的非共生固氮菌的组成及固氮潜势(表 1)。总之,生物固氮在各个生态系统中普遍存在,非共生固氮在维持不同生态系统稳定性等方面起着重要作用。
| Ecosystem/Niche | Asymbiotic nitrogen-fixing bacteria | Nitrogen-fixing potential/(kg N/hm2 year) | References |
| Steppe | Rhodopseudomonas, Azospirillum, Scytonema | 13.0–129.9 (Inner Mongolia degraded steppe) | [34–35] |
| Tropical forest | Nostoc, Tolypothrix | 8.4–12.3 (Puerto Rico, USA) | [36–37] |
| Ocean | UCYN-A, Trichodesmium, Desulfovibrio | 3.3–6.0 (Atlantic and Global) | [38] |
| Farmland | Oscillatoriales, Nostocales | 13.0–22.0 (Global wheat, maize and rice) | [40] |
| Forest soil | Rhodopseudomonas, Rhodomicrobium, Burkholderia | 0.8±0.4 (French Guiana rainforest) | [45, 47] |
| Forest bryophyte | Nostoc, Nodularia | 0.4±0.2 (French Guiana rainforest) | [46–47] |
| Forest litter | Burkholderia, Pseudomonas | 0.2±0.09 (French Guiana rainforest) | [46–47] |
4 植物不同部位和土壤中非共生固氮菌群落的组成及固氮潜力
植物不同部位存在着不同的固氮菌群和固氮活性。作物根际主要的非共生固氮菌属有芽孢杆菌属、类芽孢杆菌属和节杆菌属(Arthrobacters)等[42]。在玉米、甘蔗和小麦等作物中发现了种类众多的根内生固氮微生物,如克雷伯菌属、固氮螺菌属、类芽孢杆菌属和假单胞菌属(Pseudomonas)等[43]。植物表面也是固氮微生物的理想栖息地,利用nifH基因测序发现,植株叶际具有高度多样性的固氮微生物群落,如芽孢杆菌属、泛菌属(Pantoea)、念珠藻属和伪枝藻属(Scytonema)等[44]。中国热带森林土壤中的非共生固氮菌主要有红假单胞菌属(Rhodopseudomonas)、红微菌属(Rhodomicrobium)和伯克氏菌属(Burkholderia)等[45]。芬兰北方森林苔藓中固氮菌主要为蓝藻,主要优势属有念珠藻属和节球藻属(Nodularia)等,捷克西部森林凋落物中常见的非共生固氮菌属有伯克氏菌属、假单胞菌属等[46]。在法属圭亚那热带森林生态系统中,林冠苔藓有着最高的固氮酶活性[(1.264±0.407) nmol C2H4/(g·h)],每年非共生固氮量约为(0.388±0.182) kg N/hm2;由于土壤磷含量限制,导致森林土壤的固氮酶活性最低[(0.022±0.009) nmol C2H4/(g·h)],但土壤面积大,每年非共生固氮量可达到(0.806±0.353) kg N/hm2,土壤苔藓和森林凋落物次之[47]。在同一生态系统中,由于养分、光照和温度等条件限制,其优势固氮菌属也有所差别,进而导致非共生固氮作用的大小不同。
5 影响非共生固氮菌群落组成和固氮潜力的因素 5.1 土壤含水量土壤水分含量直接影响固氮微生物的数量和活性,适宜的土壤水分有助于非共生固氮,最佳降雨量下固氮酶活性要显著大于干旱气候下的固氮酶活性。降雨量直接影响土壤水分含量,与典型草原和温带草原相比,荒漠草原中红螺菌目(Rhodospirillales)和伯克氏菌目(Burkholderiales)的相对丰度是最高的,而念珠藻目的含量最低[48]。Zhao等发现,年均降雨量显著影响了中国森林土壤固氮菌多样性及群落结构,土壤固氮菌的α多样性指数与年均降雨量呈显著正相关,在降雨量多的热带森林土壤中慢生根瘤菌属(Bradyrhizobium)和红假单胞菌属的相对丰度较高,在降雨量较低的冷温带森林土壤中固氮螺菌属和草螺菌属(Herbaspirillum)的相对丰度较高[49]。在夏季干燥的澳大利亚西部和南部,由于土壤湿度的限制,1–6月份的非共生固氮量仅为10–15 kg N/hm2。而在夏季降雨频繁的澳大利亚北部,温暖潮湿的条件使得1–6月份的非共生固氮量约在32–38 kg N/hm2 [50]。据报道,哥斯达黎加热带森林生态系统中非共生固氮量与降雨量呈正相关,森林叶片凋落物和土壤表现出一致的规律性,如旱季时叶片凋落物每年的非共生固氮量为0.36 kg N/hm2,雨季时为5.48 kg N/hm2;同样地,旱季时土壤每年的非共生固氮量最低为0.26 kg N/hm2,雨季时最高为2.71 kg N/hm2 [51]。冯帅研究固氮施氏假单胞菌-玉米根际联合固氮作用时发现,土壤水分显著影响了施氏假单胞菌A1501的固氮贡献量,水分充足条件下的固氮贡献率为31.6%,而干旱条件下为20.1%[52]。
5.2 温度固氮菌群落组成、多样性和固氮酶活性与温度密切相关。Rui等发现高寒草甸土壤中共生固氮菌群,如中慢生根瘤菌属(Mesorhizobium),和非共生固氮菌群,如红假单胞菌属、叶杆菌属(Phyllobacterium)的相对丰度与土壤温度呈正相关,土壤温度变化能够显著改变固氮菌的群落结构[53]。在美国明尼苏达州的森林泥炭沼泽地中,土壤温度的变化显著改变了土壤固氮菌多样性及群落结构,其中拜叶林克氏菌属(Beijerinckia)、念珠藻属和红假单胞菌属的丰度随着土壤温度升高而增加;真枝藻属(Stigonema)和费氏藻属(Fischerella)的丰度随着土壤温度升高而减少[54]。在宁夏沙坡头荒漠生态系统中,张鹏等发现藻类、苔藓和地衣的固氮酶活性与温度(20–40 ℃)均呈极显著的负相关关系,其中藻类和苔藓结皮在25–30 ℃具有最高的固氮酶活性,地衣结皮在20–30 ℃具有最高的固氮酶活性[55]。Rousk等也证明苔藓在20–30 ℃具有最高的固氮酶活性[56]。对瑞典北方森林土壤中的小羽藓固氮酶活性的研究发现,低温(16.3 ℃)时固氮酶活性较低,随着温度升到中温(22.0 ℃)时,固氮酶活性达到最大,而温度继续升高(30.3 ℃)则会降低固氮酶活性[57]。在中国广西喀斯特森林中发现,随着温度逐步升高(15–25–35 ℃),森林土壤和凋落物的固氮酶活性逐渐变高,而对森林苔藓的固氮酶活性却无显著影响[58]。
5.3 土壤pH土壤pH对非共生固氮菌多样性、群落组成和固氮酶活性有着重要的影响。中国红壤耕地中优势非共生固氮菌群有甲基杆菌属(Methylobacterium)和脱硫弧菌属(Desulfovibrio)等,而固氮螺菌属和斯克尔曼氏菌属(Skermanella)在潮土耕地中相对丰度较高,粘球菌属(Myxobacter)和固氮螺菌属是黑土耕地中主要的固氮菌属,这主要是因为土壤pH驱动了固氮菌群落组成的变化[59]。Lin等通过对比无机肥、有机肥、石灰施用和秸秆还田等多种施肥方式,发现土壤pH是导致固氮菌群落结构变化的主要因子,土壤pH的升高(由原来的4.96升高到6.59)使得优势共生固氮菌属慢生根瘤菌属(Bradyrhizobium)演替为非共生固氮菌属,固氮螺菌属和氮氢单胞菌(Azohydromonas)[60]。同样地,在旱地农田土壤中,土壤pH升高会导致nifH基因拷贝数减少,主要是由于优势固氮菌属慢生根瘤菌属相对丰度减少,而非共生固氮菌如红假单胞菌属和固氮螺菌属等相对丰度增加导致的[60–61]。草原生态系统中,土壤pH是草甸土壤非共生固氮菌群多样性及组成变化的最佳预测因子,固氮螺菌属、伯克氏菌属的相对丰度随着土壤pH的增加而减少[29]。而土壤pH对于土壤固氮酶活性的影响因生态系统的不同而有所差异。在农田土壤中,土壤pH的升高能够显著提升土壤固氮酶活性,这主要得益于非共生固氮菌群的贡献[59, 62]。在森林生态系统中,土壤pH通过改变土壤中非共生固氮菌群落结构,间接影响固氮酶活性,其中在酸性热带森林土壤中,非共生固氮菌的固氮速率与土壤pH显著正相关[63–65]。
5.4 外源物质添加外源物质的添加(如化学肥料施用等)能够改变土壤性质,影响固氮菌的生存环境,进而影响固氮酶活性。在亚热带森林土壤中,外源氮添加显著降低了土壤固氮微生物如慢生根瘤菌属和固氮螺菌属的丰富度,显著改变了固氮菌的群落结构,使得固氮酶活性显著降低;而外源磷的添加则会增强土壤的固氮作用[66]。但也有研究表明,在亚热带森林土壤中,外源氮、磷的添加并未显著改变非共生固氮菌群的群落结构[67]。固氮酶由钼蛋白和铁蛋白组成,在热带森林土壤中,施用钼肥使得土壤非共生固氮量急剧增加,而无钼处理的土壤非共生固氮酶活性无显著变化,说明钼是限制热带森林非共生固氮最重要的元素[68]。在氮、磷和钼元素的有效性对热带森林和温带森林非共生固氮的影响研究中,发现施氮能显著抑制非共生固氮作用,施钼能显著促进非共生固氮作用,施磷能促进热带森林的非共生固氮作用,而抑制温带森林的非共生固氮作用,说明外援物质的添加会影响土壤固氮作用,但对不同生态系统土壤固氮作用的影响不同[69]。另外通过实验室模拟实验发现,砷污染物会限制农业土壤中自生固氮菌的活性[70]。
5.5 农业生产措施农业生产措施会直接或间接地改变固氮微生物群落,从而使得生物固氮量发生改变。在旱地农田中,长期单施化学肥料显著降低了土壤中nifH基因丰度、固氮菌多样性、群落结构和固氮潜力;当配施有机肥时,显著提高了土壤固氮菌多样性,并通过富集一些固氮菌属如Pseudacidovorax、固氮螺菌属、红假单胞菌属等来提高土壤固氮潜力和固氮菌抗逆性[71]。在水稻田中,与单施NPK无机肥相比,有机肥配施无机肥能够显著提高土壤中nifH基因丰度、固氮菌多样性和固氮潜力,这是因为有机肥可以有效改善土壤营养状况,为非共生固氮菌如蓝藻提供更好的生存环境[72]。
在中国黄土丘陵地区,退耕还林对恢复当地生物多样性有着重要意义,一方面能够显著提高土壤固氮微生物多样性,另一方面显著改变了固氮菌群落组成及结构。与农田土壤相比,林地土壤富集了较多的非共生固氮菌群,如红假单胞菌属、假黄色单胞菌属(Pseudoxanthomonas)和固氮菌属等,由于林地恢复时,高投入的氮磷肥更利于非共生固氮菌群的生长[73]。另外,在热带森林生态系统中,土地利用方式的转变(原始森林—农田、原始森林—草原、原始森林—伐木道路)使得土壤中固氮菌多样性及群落结构发生改变,在伐木道路的土壤中固氮螺菌属和慢生根瘤菌属的相对丰度最高,而原始森林土壤中含有较多的伯克氏菌和硝化螺旋菌属(Nitrospira)等。这主要由于人为措施导致土壤性质转变,固氮微生物需要调整群落结构来适应土壤扰动带来的影响[74]。随着森林转变为牧场,由于植物对氮素需求量的增加,导致土壤中自生固氮菌群落结构显著变化,nifH基因丰度增加10倍[75]。
6 非共生固氮菌的研究方法随着微生物学研究方法的不断突破和改进,非共生固氮菌的研究方法也愈加多样化。近年来,随着荧光定量PCR、高通量测序和同位素标记等技术手段的发展,非共生固氮菌丰度、多样性、群落结构和物种组成等研究得到了快速发展。
6.1 荧光定量PCR利用荧光定量PCR (qPCR)技术对固氮菌nifH基因进行定量分析,确定其在环境中的丰度。在qPCR体系中,荧光染料或荧光探针可与扩增产物结合发出荧光,荧光强度与扩增产物量成正比,所以,通过检测荧光量就能够确定样品中nifH基因的表达量。如通过qPCR技术,发现束毛藻属(Trichodesmium)和单细胞蓝藻UCYN–A和UCYN–B的nifH基因拷贝数与海水温度显著相关,当海水温度达到28 ℃,这3种固氮菌属有着最大的基因拷贝数[76]。但利用nifH基因丰度预估微生物固氮量也并不是非常可靠,如qPCR技术存在引物特异性不同,导致不同模板扩增效率存在显著差异,从而对nifH基因拷贝数的估算产生影响[77]。
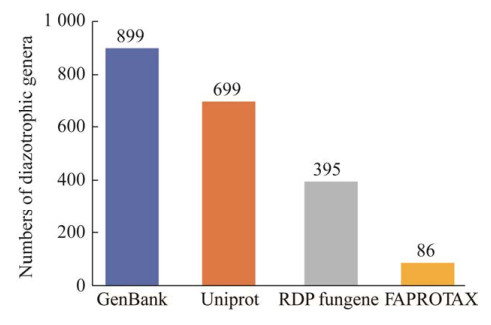
6.2 高通量测序高通量测序技术已经成为研究微生物群落结构的重要技术手段,主要利用扩增子测序方法研究固氮菌,即利用二代高通量测序技术对nifH基因的扩增片段进行测序和分析。常见的通用引物对有PolF/PolR、F2/R6、IGK3/DVV、nifHF/nifHR等(表 2),扩增片段从310 bp到470 bp不等[78]。固氮菌nifH基因的高通量测序数据主要用于分析固氮微生物的多样性和群落组成,研究固氮微生物群落在生态系统中的生态过程和功能等。扩增子测序的流程包括DNA提取、nifH基因的PCR扩增、建库、测序和生物信息学分析。其中生物信息学分析主要包括数据过滤、降噪/聚类(产生ASV/OTU)、进化树重构建和代表性序列的比对注释等。如果基于蛋白序列进行注释,需要进行移码突变检测[79]。代表性序列的比对注释依赖于nifH数据库的整理。我们统计了GenBank、Uniprot和RDP Fungene数据库中nifH序列的基本信息(截至2021年11月)。GenBank目前覆盖了超过890个原核微生物的属,Uniprot覆盖了超过690个属,而Fungene数据库覆盖了390多个属(图 2),这也说明了不同的数据库的nifH序列的涵盖范围有较大差别。同时各个数据库中大量的序列都是未培养微生物的,说明依然有大量的未知的固氮菌存在。除了基于nifH基因的扩增子测序外,基于16S rRNA基因扩增子测序结果的注释分类信息也能筛选出部分固氮菌,如使用FAPROTAX数据库对每个ASV/OTU进行预测提取,目前可使用基于R语言的microeco包中的trans_func进行分析[80–82]。不过,目前FAPROTAX只涉及到了86个属,存在大量的属未被收录的问题。
| Primer set (F/R)a | Length/bpb | Tm/℃c | Sequence (5′→3′) | Coverage/%d | References |
| PolF/PolR | 360 | 55 | TGCGAYCCSAARGCBGACTC | 39 | [83] |
| ATSGCCATCATYTCRCCGGA | 35 | ||||
| F2/R6 | 360 | 52 | TGYGAYCCIAAIGCIGA | 96 | [84] |
| TCIGGIGARATGATGGC | 96 | ||||
| KAD3/DVV | 310 | 58 | ATHGTIGGITGYGAYCCIAARGCIGA | 70 | [85] |
| ATIGCRAAICCICCRCAIACIACRTC | 94 | ||||
| nifHF/nifHR | 400 | 53 | AAAGGYGGWATCGGYAARTCCACCAC | 16 | [86] |
| TTGTTSGCSGCRTACATSGCCATCAT | 49 | ||||
| nifH1/nifH2 | 360 | 50 | ADNGCCATCATYTCNCC | 94 | [87] |
| TGYGAYCCNAARGCNGA | 95 | ||||
| nifH3/nifH4 | 470 | 55 | ATRTTRTTNGCNGCRTA | 94 | [88] |
| TTYTAYGGNAARGGNGG | 52 | ||||
| Ueda19F/407R | 390 | 50 | GCIWTYTAYGGIAARGGIGG | 93 | [89] |
| AAICCRCCRCAIACIACRTC | 91 | ||||
| a: Forward (F) and Reverse (R); b: Length expected for PCR amplicon; c: PCR primer annealing temperature; d: Data indicate primer binding to all nifH sequences in the database with 0 mismatches allowed. Adapted from Reference[78]. | |||||
|
| 图 2 几个基于nifH的固氮基因数据库所覆盖的固氮菌属个数 Figure 2 Numbers of diazotrophic genera covered by different nifH based nitrogen-fixing gene database. |
6.3 同位素标记法
同位素标记可用来研究活跃的固氮微生物。13C-SIP技术从2000年建立以来已取得一些进展,但15N-DNA(RNA)-SIP技术进展缓慢。DNA (RNA)中N较C要少,这就需要更多的DNA (RNA)。纳米二次离子质谱(nanoscale secondary ion mass spectrometry,NanoSIMS)实现了成像与单细胞稳定性同位素测定的有机结合。NanoSIMS技术可以满足DNA密度梯度离心后各层中极微量DNA-15N的测定[90]。除此之外,研究RNA中的nifH基因也被认为是可行的研究活性固氮微生物的方法[91]。
7 固氮潜力的测定方法在小尺度上,非共生固氮量的测定主要有乙炔还原法(ARA)、15N同位素稀释法、15N自然丰度法、全氮差值法和酰脲估测法等;而在大尺度上主要靠模型估算。
7.1 乙炔还原法乙炔还原法主要是通过测定固氮酶活性来估算非共生固氮量,具有操作简便、费用较低、试验周期短等特点。基本原理为,固氮酶可以催化乙炔还原为乙烯,而这一反应中气体的微量变化可以用气相色谱仪检测出来,根据乙烯的变化量计算固氮酶活性,再根据乙烯与氢的换算比例估算微生物固氮量。Zhao等通过乙炔还原法发现,添加秸秆生物碳能够显著提升果园土壤中非共生固氮菌的固氮酶活性,这主要由于生物碳的增加显著提高了土壤中可利用碳氮及钼的含量,进而为非共生固氮菌的活动提供丰富能源[92]。乙炔还原法也有一些缺点。Saiz等使用15N标记技术和乙炔还原法同时测定固氮酶活性,发现乙炔还原法实际测得的转换系数远低于理论的3:1[93];另外,该方法不宜在田间试验中使用,只能短时间内测定固氮酶活性等。将光腔衰荡激光吸收光谱技术(cavity ring-down spectroscopy,CRDS)与乙炔还原法相结合,能够短时间内连续、快速、可重复、实时测定固氮酶活性,避免因乙炔对植物功能的影响而造成的测量偏差[94]。
7.2 15N同位素稀释法相比于乙炔还原法,15N同位素稀释法具有灵敏度高、准确度高、可靠性强、直接等特点。该法可测出植株在田间等综合条件下的固氮量,而且能够区分出植株从大气、土壤和肥料中的氮吸收量。主要原理是,参考植株(非固氮植株)和测定植株(固氮植株)生长在15N标记肥料的土壤中,参考植株仅利用肥料和土壤中的氮进行生长,而测定植株还需要利用大气中的氮,然后根据植物体内各形态N含量变化,即可计算出测定植株固氮速率和固氮量[95]。但此法也有缺点,15N价格昂贵,测定手段繁琐,需选定参考植物等。农倩等将属于伯克氏菌属的甘蔗内生固氮菌GXS16接种到甘蔗根部,采用15N同位素稀释法检测菌株固氮效率,发现接种后植株能够显著从空气中固定氮气,证实菌株GXS16是一株高效甘蔗内生固氮菌[96]。
7.3 15N自然丰度法植株体内存在同位素分馏效应,导致植株体内同位素自然丰度的差异。15N自然丰度法基于此原理,选择同一生存条件下的非固氮植株和固氮植株,相较于固氮植株,非固氮植株能从土壤中吸收较高的15N,使得植株间体内的15N同位素丰度产生差异,利用同位素质谱仪测定δ15N值,从而得到植株固氮量[97–98]。该方法便于野外操作,不需要添加带15N标记的肥料,可以长时间内测定植株的固氮量,对于较大型植株如木本植株等的固氮量测定有着独特优势,可以连续多年测定植株固氮量,不会干扰植株的生长及土壤生态系统。但同时也存在不足之处,如参照植株的选择在很大程度上决定着测定数值的准确性、植株不同部位δ15N值存在差异[99–101]。Russow等通过15N自然丰度法对沙漠中地衣固氮量进行了估算,选择非固氮地衣鳞茶渍(Squamarina)作为参照,得出内盖夫沙漠地衣每年的生物固氮量约为10–41 kg N/hm2 [102]。
7.4 酰脲估测法和全氮量差法酰脲估测法和全氮量差法主要采用化学实验方法测定植物和对照植株全氮、铵态氮和硝态氮含量的变化,进而估算植株的固氮量,与以上方法相比,具有简单、易行、价格低廉等特点,但存在实验误差大、测定不准确、需要采集完整的植株样品等缺点。20世纪后很少使用酰脲估测法和全氮量差法进行植株固氮量的计算[103]。
7.5 模型估算法如何在全球大尺度范围内估算非共生固氮量是我们当前需要解决的一个问题。在大范围内估算非共生固氮量,常用的方法是模型估算法。例如对于全球玉米、水稻和小麦非共生固氮量的估算,Ladha等指出,基于氮平衡原理的差分法可以相对准确地计算出植物体内的非共生固氮量[40]。Xu等通过使用全球植被动态模型(DyN-LPJ)估算,全球非农陆地生态系统中每年所需的氮投入大约为340 Tg,其中生物固氮是主要来源[104]。Herridge等将世界农业生产数据库(FAOSTAT)中的谷物面积和产量与已公布和未公布的生物固氮量相结合,进一步估算全球农业生态系统中生物固氮量约在50–70 Tg[105]。Staccone等通过林业部门查询主要固氮树种及分布丰度,进一步通过文献查询相关树种的固氮速率和固氮树种在生长周期内对氮的需求,估算了美国森林生态系统每年的非共生固氮量约为0.37 Tg[106]。在测定和估算生态系统中的自生固氮量时,需要针对不同生态系统的特点设计方法,并着重考虑空间尺度和影响因素等主要因子进行统计建模,在规范化的尺度上进行非共生固氮作用的估算[107]。
8 展望目前非共生固氮菌群和固氮潜势的研究多集中于某一特定的生态系统,众多研究结果均表明了非共生固氮作用的重要性。然而,固氮菌的多样性、群落结构和构建机制对固氮潜势的影响还未系统地阐明。针对土壤-植物系统研究非共生固氮菌群落构建机制、固氮潜势及其相互作用关系,可以揭示非共生固氮在植物和土壤相关的N循环过程中的贡献,更好地理解非共生固氮在生物地球化学循环中发挥的作用及其在生态系统中参与的功能。当前非共生固氮量的测定方法多样,没有统一的标准,因此固氮潜势的方法学研究也是一个研究重点。由于大量未知固氮菌的存在,宏基因组测序就凸显出了其价值,同时宏基因组测序还能探讨固氮基因与其他代谢通路间的关系等。
应加强难培养非共生固氮微生物分离方法的探索,以获取更多的固氮菌资源。在分子水平上研究固氮菌株基因改造,以提高固氮菌的固氮能力及非固氮菌株向固氮菌株的转化等。从作物与固氮菌互作角度思考,揭示非共生固氮菌株与植物的相互作用及固氮作用发生的机理,可以为植物育种提供新方向。应该大力探索非共生固氮微生物在维持土壤肥力、提高植物抗逆性及作物产量方面的作用机制,以其在农业生产中能够调控非共生固氮菌群的构成及活性,如优化农业生产的管理措施及施肥方式,平衡微生物固氮作用与施肥措施的高效协同作用,实现化学肥料的少投入,作物的高产出。
| [1] |
Sun XB, Zhang QS, Xiao KC, Li DJ. Variation of asymbiotic nitrogen fixation with post-agricultural succession in a Karst region of Northwest Guangxi. Research of Agricultural Modernization, 2020, 41(4): 709-717.
(in Chinese) 孙锡斌, 张青山, 肖孔操, 李德军. 桂西北喀斯特山区非共生固氮速率随退耕后生态演替变化特征. 农业现代化研究, 2020, 41(4): 709-717. |
| [2] | Reed SC, Cleveland CC, Townsend AR. Functional ecology of free-living nitrogen fixation: a contemporary perspective. Annual Review of Ecology, Evolution and Systematics, 2011, 42: 489-512. DOI:10.1146/annurev-ecolsys-102710-145034 |
| [3] | Kuypers MMM, Marchant HK, Kartal B. The microbial nitrogen-cycling network. Nature Reviews Microbiology, 2018, 16(5): 263-276. DOI:10.1038/nrmicro.2018.9 |
| [4] | Elbert W, Weber B, Burrows S, Steinkamp J, Büdel B, Andreae MO, Pöschl U. Contribution of cryptogamic covers to the global cycles of carbon and nitrogen. Nature Geoscience, 2012, 5(7): 459-62. DOI:10.1038/ngeo1486 |
| [5] |
Xu PX, Han LL, He JZ, Luo F, Zhang LM. Research advance on molecular ecology of asymbiotic nitrogen fixation microbes. Chinese Journal of Applied Ecology, 2017, 28(10): 3440-3450.
(in Chinese) 徐鹏霞, 韩丽丽, 贺纪正, 罗锋, 张丽梅. 非共生生物固氮微生物分子生态学研究进展. 应用生态学报, 2017, 28(10): 3440-3450. |
| [6] | Bellenger JP, Darnajoux R, Zhang X, Kraepiel AML. Biological nitrogen fixation by alternative nitrogenases in terrestrial ecosystems: a review. Biogeochemistry, 2020, 149(1): 53-73. DOI:10.1007/s10533-020-00666-7 |
| [7] | Vitousek PM, Cassman K, Cleveland C, Crews T, Field CB, Grimm NB, Howarth RW, Marino R, Martinelli L, Rastetter EB, Sprent JI. Towards an ecological understanding of biological nitrogen fixation. The Nitrogen Cycle at Regional to Global Scales. Dordrecht: Springer Netherlands, 2002: 1-45. |
| [8] | Bottomley PJ, Myearold DD. Chapter 15 - Biological N Inputs//Paul EA. Soil Microbiology, Ecology and Biochemistry (Fourth Edition). Boston: Academic Press, 2015. |
| [9] |
Zhang W, Yang L, Wang ZJ. Advance and development trend of biological nitrogen fixation research. Journal of Yunnan Agricultural University: Natural Science, 2015, 30(5): 810-821.
(in Chinese) 张武, 杨琳, 王紫娟. 生物固氮的研究进展及发展趋势. 云南农业大学学报: 自然科学, 2015, 30(5): 810-821. |
| [10] | Santi C, Bogusz D, Franche C. Biological nitrogen fixation in non-legume plants. Annals of Botany, 2013, 111(5): 743-767. DOI:10.1093/aob/mct048 |
| [11] |
Zhou F, Fang XT, Gao H. Advances on the heterocysts of the nitrogen-fixing Cyanobacteria. Journal of Anhui Agricultural Sciences, 2013, 41(2): 524-527.
(in Chinese) 周芳, 方仙桃, 高宏. 固氮蓝藻的异形胞研究进展. 安徽农业科学, 2013, 41(2): 524-527. DOI:10.3969/j.issn.0517-6611.2013.02.022 |
| [12] | 韩佳悯. 化能自养细菌Halothiobacillus sp. LS2硫氧化和固氮作用的研究. 华南理工大学硕士学位论文, 2019. |
| [13] | Baldani J, Caruso L, Baldani VLD, Goi SR, Döbereiner J. Recent advances in BNF with non-legume plants. Soil Biology and Biochemistry, 1997, 29(5/6): 911-922. |
| [14] |
Lin L, Li YR, An QL. Biological nitrogen fixation in association with sugarcane: retrospect and prospect. Biotechnology Bulletin, 2019, 35(10): 46-56.
(in Chinese) 林丽, 李杨瑞, 安千里. 甘蔗联合固氮的回顾与展望. 生物技术通报, 2019, 35(10): 46-56. |
| [15] |
Jing XS, Ding Y, Han XM, Wang Z, Gao DY. Advances in synthetic biology of associated nitrogen-fixation bacteria. Acta Microbiologica Sinica, 2021, 61(10): 3026-3034.
(in Chinese) 荆晓姝, 丁燕, 韩晓梅, 王哲, 高德艳. 联合固氮菌的合成生物学研究进展. 微生物学报, 2021, 61(10): 3026-3034. |
| [16] | Van Deynze A, Zamora P, Delaux PM, Heitmann C, Jayaraman D, Rajasekar S, Graham D, Maeda J, Gibson D, Schwartz KD, Berry AM, Bhatnagar S, Jospin G, Darling A, Jeannotte R, Lopez J, Weimer BC, Eisen JA, Shapiro HY, Ané JM, Bennett AB. Nitrogen fixation in a Landrace of maize is supported by a mucilage-associated diazotrophic microbiota. PLoS Biology, 2018, 16(8): e2006352. DOI:10.1371/journal.pbio.2006352 |
| [17] | Bennett AB, Pankievicz VCS, Ané JM. A model for nitrogen fixation in cereal crops. Trends in Plant Science, 2020, 25(3): 226-235. DOI:10.1016/j.tplants.2019.12.004 |
| [18] | Dos Santos PC, Fang Z, Mason SW, Setubal JC, Dixon R. Distribution of nitrogen fixation and nitrogenaselike sequences amongst microbial genomes. BMC Genomics, 2012, 13: 162. DOI:10.1186/1471-2164-13-162 |
| [19] | Addo MA, Dos Santos PC. Distribution of nitrogen-fixation genes in prokaryotes containing alternative nitrogenases. ChemBioChem, 2020, 21(12): 1749-1759. DOI:10.1002/cbic.202000022 |
| [20] | Harwood CS. Iron-only and vanadium nitrogenases: fail-safe enzymes or something more?. Annual Review of Microbiology, 2020, 74: 247-266. DOI:10.1146/annurev-micro-022620-014338 |
| [21] | Hamelin J, Fromin N, Tarnawski S, Teyssier-Cuvelle S, Aragno M. NifH gene diversity in the bacterial community associated with the rhizosphere of Molinia coerulea, an oligonitrophilic perennial grass. Environmental Microbiology, 2002, 4(8): 477-481. DOI:10.1046/j.1462-2920.2002.00319.x |
| [22] | Ryu MH, Zhang J, Toth T, Khokhani D, Geddes BA, Mus F, Garcia-Costas A, Peters JW, Poole PS, Ané JM, Voigt CA. Control of nitrogen fixation in bacteria that associate with cereals. Nature Microbiology, 2020, 5(2): 314-330. DOI:10.1038/s41564-019-0631-2 |
| [23] | Raymond J, Siefert JL, Staples CR, Blankenship RE. The natural history of nitrogen fixation. Molecular Biology and Evolution, 2004, 21(3): 541-554. DOI:10.1093/molbev/msh047 |
| [24] | Harris DF, Lukoyanov DA, Kallas H, Trncik C, Yang ZY, Compton P, Kelleher N, Einsle O, Dean DR, Hoffman BM, Seefeldt LC. Mo-, V-, and Fe-nitrogenases use a universal eight-electron reductive-elimination mechanism to achieve N2 reduction. Biochemistry, 2019, 58(30): 3293-3301. DOI:10.1021/acs.biochem.9b00468 |
| [25] | Gravel D, Canham CD, Beaudet M, Messier C. Reconciling niche and neutrality: the continuum hypothesis. Ecology Letters, 2006, 9(4): 399-409. DOI:10.1111/j.1461-0248.2006.00884.x |
| [26] | Zhou JZ, Ning DL. Stochastic community assembly: does it matter in microbial ecology?. Microbiology and Molecular Biology Reviews, 2017, 81(4): e00002-17. DOI:10.1128/MMBR.00002-17 |
| [27] |
Chai YF, Yue M. Research advances in plant community assembly mechanisms. Acta Ecologica Sinica, 2016, 36(15): 4557-4572.
(in Chinese) 柴永福, 岳明. 植物群落构建机制研究进展. 生态学报, 2016, 36(15): 4557-4572. |
| [28] | Prado VHM, Rossa-Feres DCC. The influence of niche and neutral processes on a neotropical anuran metacommunity. Austral Ecology, 2014, 39(5): 540-547. DOI:10.1111/aec.12114 |
| [29] | Wang YS, Li CN, Kou YP, Wang JJ, Tu B, Li H, Li XZ, Wang CT, Yao MJ. Soil pH is a major driver of soil diazotrophic community assembly in Qinghai-Tibet alpine meadows. Soil Biology and Biochemistry, 2017, 115: 547-555. DOI:10.1016/j.soilbio.2017.09.024 |
| [30] | Wang YS, Kou YP, Li CN, Tu B, Wang JM, Yao MJ, Li XZ. Contrasting responses of diazotrophic specialists, opportunists, and generalists to steppe types in Inner Mongolia. CATENA, 2019, 182: 104168. DOI:10.1016/j.catena.2019.104168 |
| [31] | Fan KK, Weisenhorn P, Gilbert JA, Shi Y, Bai Y, Chu HY. Soil pH correlates with the co-occurrence and assemblage process of diazotrophic communities in rhizosphere and bulk soils of wheat fields. Soil Biology and Biochemistry, 2018, 121: 185-192. DOI:10.1016/j.soilbio.2018.03.017 |
| [32] | Wang YS, Li CN, Tu B, Kou YP, Li XZ. Species pool and local ecological assembly processes shape the β-diversity of diazotrophs in grassland soils. Soil Biology and Biochemistry, 2021, 160: 108338. DOI:10.1016/j.soilbio.2021.108338 |
| [33] | Wang YS, Li CN, Shen ZH, Rui JP, Jin DC, Li JB, Li XZ. Community assemblage of free-living diazotrophs along the elevational gradient of Mount Gongga. Soil Ecology Letters, 2019, 1(3/4): 136-146. |
| [34] |
Wendurile, Li G, Yang DL, Zhang JN, Yi J. NifH gene diversity and community structure of soil nitrogen-fixing bacteria in Hulunbeier grassland Inner Mongolia. Chinese Journal of Ecology, 2011, 30(4): 790-797.
(in Chinese) 文都日乐, 李刚, 杨殿林, 张静妮, 易津. 呼伦贝尔草原土壤固氮微生物nifH基因多样性与群落结构. 生态学杂志, 2011, 30(4): 790-797. |
| [35] | 刘华杰. 内蒙古草原中生物土壤结皮的生态学研究. 中国科学院植物研究所博士后论文, 2007. |
| [36] | Fürnkranz M, Wanek W, Richter A, Abell G, Rasche F, Sessitsch A. Nitrogen fixation by phyllosphere bacteria associated with higher plants and their colonizing epiphytes of a tropical lowland rainforest of Costa Rica. The ISME Journal, 2008, 2(5): 561-570. DOI:10.1038/ismej.2008.14 |
| [37] | Cusack DF, Silver W, McDowell WH. Biological nitrogen fixation in two tropical forests: ecosystem-level patterns and effects of nitrogen fertilization. Ecosystems, 2009, 12(8): 1299-1315. DOI:10.1007/s10021-009-9290-0 |
| [38] | Deutsch C, Sarmiento JL, Sigman DM, Gruber N, Dunne JP. Spatial coupling of nitrogen inputs and losses in the ocean. Nature, 2007, 445(7124): 163-167. DOI:10.1038/nature05392 |
| [39] | Zehr JP, Capone DG. Changing perspectives in marine nitrogen fixation. Science, 2020, 368(6492): eaay9514. DOI:10.1126/science.aay9514 |
| [40] | Ladha JK, Tirol-Padre A, Reddy CK, Cassman KG, Verma S, Powlson DS, van Kessel C, de B Richter D, Chakraborty D, Pathak H. Global nitrogen budgets in cereals: a 50-year assessment for maize, rice and wheat production systems. Scientific Reports, 2016, 6: 19355. DOI:10.1038/srep19355 |
| [41] | Vijayan D, Ray JG. Ecology and diversity of Cyanobacteria in Kuttanadu paddy wetlands, Kerala, India. American Journal of Plant Sciences, 2015, 6(18): 2924-2938. DOI:10.4236/ajps.2015.618288 |
| [42] |
Sun JG, Xu J, Hu HY, Zhang YC, Liu J, Wang WB, Sun YH. Collection and investigation on asymbiotic nitrogen-fixing microbial resources from 13 provinces over China. Journal of Plant Nutrition and Fertilizer, 2009, 15(6): 1450-1465.
(in Chinese) 孙建光, 徐晶, 胡海燕, 张燕春, 刘君, 王文博, 孙燕华. 中国十三省市土壤中非共生固氮微生物菌种资源研究. 植物营养与肥料学报, 2009, 15(6): 1450-1465. DOI:10.3321/j.issn:1008-505X.2009.06.030 |
| [43] | Liu HW, Zhang L, Meng AH, Zhang JB, Xie MM, Qin YH, Faulk DC, Zhang BH, Yang SS, Qiu L. Isolation and molecular identification of endophytic diazotrophs from seeds and stems of three cereal crops. PLoS One, 2017, 12(10): e0187383. DOI:10.1371/journal.pone.0187383 |
| [44] | Abadi VAJM, Sepehri M, Rahmani HA, Dolatabad HK, Shamshiripour M, Khatabi B. Diversity and abundance of culturable nitrogen-fixing bacteria in the phyllosphere of maize. Journal of Applied Microbiology, 2021, 131(2): 898-912. DOI:10.1111/jam.14975 |
| [45] | Zhang J, Zheng MH, Zhang YJ, Wang J, Shen H, Lin YB, Tang XL, Hui DF, Lambers H, Sardans J, Peñuelas J, Liu ZF. Soil phosphorus availability affects diazotroph communities during vegetation succession in lowland subtropical forests. Applied Soil Ecology, 2021, 166: 104009. DOI:10.1016/j.apsoil.2021.104009 |
| [46] | Urbanová M, Šnajdr J, Baldrian P. Composition of fungal and bacterial communities in forest litter and soil is largely determined by dominant trees. Soil Biology and Biochemistry, 2015, 84: 53-64. DOI:10.1016/j.soilbio.2015.02.011 |
| [47] | Van Langenhove L, Depaepe T, Verryckt LT, Fuchslueger L, Donald J, Leroy C, Krishna Moorthy SM, Gargallo-Garriga A, Ellwood MDF, Verbeeck H, Van Der Straeten D, Peñuelas J, Janssens IA. Comparable canopy and soil free-living nitrogen fixation rates in a lowland tropical forest. Science of the Total Environment, 2021, 754: 142202. DOI:10.1016/j.scitotenv.2020.142202 |
| [48] | Sheng R, Li K, Zhang WZ, Wang H, Liu HL, Zhu XY, Wu HX, Zhang XQ, Lin QM, Sun XC, Tang YF, A L, Wei WX. Differentiations of determinants for the community compositions of bacteria, fungi, and nitrogen fixers in various steppes. Ecology and Evolution, 2019, 9(6): 3239-3250. DOI:10.1002/ece3.4940 |
| [49] | Zhao WQ, Kou YP, Wang XH, Wu YH, Bing HJ, Liu Q. Broad-scale distribution of diazotrophic communities is driven more by aridity index and temperature than by soil properties across various forests. Global Ecology and Biogeography, 2020, 29(12): 2119-2130. DOI:10.1111/geb.13178 |
| [50] | Gupta VVSR, Roper MM, Roget DK. Potential for non-symbiotic N2-fixation in different agroecological zones of southern Australia. Soil Research, 2006, 44(4): 343-354. DOI:10.1071/SR05122 |
| [51] | Reed SC, Cleveland CC, Townsend AR. Controls over leaf litter and soil nitrogen fixation in two lowland tropical rain forests. Biotropica, 2007, 39(5): 585-592. DOI:10.1111/j.1744-7429.2007.00310.x |
| [52] | 冯帅. 固氮施氏假单胞菌A1501—玉米根际联合固氮作用及其微生物群落分析. 西南科技大学硕士学位论文, 2017. |
| [53] | Rui JP, Li JB, Wang SP, An JX, Liu WT, Lin QY, Yang YF, He ZL, Li XZ. Responses of bacterial communities to simulated climate changes in alpine meadow soil of the Qinghai-Tibet plateau. Applied and Environmental Microbiology, 2015, 81(17): 6070-6077. DOI:10.1128/AEM.00557-15 |
| [54] | Carrell AA, Kolton M, Glass JB, Pelletier DA, Warren MJ, Kostka JE, Iversen CM, Hanson PJ, Weston DJ. Experimental warming alters the community composition, diversity, and N2 fixation activity of peat moss (Sphagnum fallax) microbiomes. Global Change Biology, 2019, 25(9): 2993-3004. DOI:10.1111/gcb.14715 |
| [55] |
Zhang P, Li XR, Jia RL, Hu YG, Huang L. Nitrogenase activity of biological soil crusts and its response to hydrothermic factors in the Shapotou region of Northern China. Chinese Journal of Plant Ecology, 2011, 35(9): 906-913.
(in Chinese) 张鹏, 李新荣, 贾荣亮, 胡宜刚, 黄磊. 沙坡头地区生物土壤结皮的固氮活性及其对水热因子的响应. 植物生态学报, 2011, 35(9): 906-913. |
| [56] | Rousk K, Pedersen PA, Dyearnum K, Michelsen A. The interactive effects of temperature and moisture on nitrogen fixation in two temperate-Arctic mosses. Theoretical and Experimental Plant Physiology, 2017, 29(1): 25-36. DOI:10.1007/s40626-016-0079-1 |
| [57] | Gundale MJ, Nilsson M, Bansal S, Jäderlund A. The interactive effects of temperature and light on biological nitrogen fixation in boreal forests. New Phytologist, 2012, 194(2): 453-463. DOI:10.1111/j.1469-8137.2012.04071.x |
| [58] | Li DJ, Zhang QS, Xiao KC, Wang ZC, Wang KL. Divergent responses of biological nitrogen fixation in soil, litter and moss to temperature and moisture in a Karst forest, southwest China. Soil Biology and Biochemistry, 2018, 118: 1-7. DOI:10.1016/j.soilbio.2017.11.026 |
| [59] | Han LL, Wang Q, Shen JP, Di HJ, Wang JT, Wei WX, Fang YT, Zhang LM, He JZ. Multiple factors drive the abundance and diversity of the diazotrophic community in typical farmland soils of China. FEMS Microbiology Ecology, 2019, 95(8): fiz113. DOI:10.1093/femsec/fiz113 |
| [60] | Lin YX, Ye GP, Liu DY, Ledgard S, Luo JF, Fan JB, Yuan JJ, Chen ZM, Ding WX. Long-term application of lime or pig manure rather than plant residues suppressed diazotroph abundance and diversity and altered community structure in an acidic Ultisol. Soil Biology and Biochemistry, 2018, 123: 218-228. DOI:10.1016/j.soilbio.2018.05.018 |
| [61] | Wang C, Zheng MM, Song WF, Wen SL, Wang BR, Zhu CQ, Shen RF. Impact of 25 years of inorganic fertilization on diazotrophic abundance and community structure in an acidic soil in Southern China. Soil Biology and Biochemistry, 2017, 113: 240-249. DOI:10.1016/j.soilbio.2017.06.019 |
| [62] | Wu CF, Wei XM, Hu ZY, Liu Y, Hu YJ, Qin HL, Chen XB, Wu JS, Ge TD, Zhran M, Su YR. Diazotrophic community variation underlies differences in nitrogen fixation potential in paddy soils across a climatic gradient in China. Microbial Ecology, 2021, 81(2): 425-436. DOI:10.1007/s00248-020-01591-w |
| [63] | Tang YQ, Tian J, Li XZ, Yao MJ, Wang SQ, Kuzyakov Y, Dungait JAJ. Higher free-living N2 fixation at rock-soil interfaces than topsoils during vegetation recovery in Karst soils. Soil Biology and Biochemistry, 2021, 159: 108286. DOI:10.1016/j.soilbio.2021.108286 |
| [64] | Winbourne JB, Brewer SW, Houlton BZ. Iron controls over di-nitrogen fixation in Karst tropical forest. Ecology, 2017, 98(3): 773-781. DOI:10.1002/ecy.1700 |
| [65] | Van Langenhove L, Depaepe T, Vicca S, van den Berge J, Stahl C, Courtois E, Weedon J, Urbina I, Grau O, Asensio D, Peñuelas J, Boeckx P, Richter A, van der Straeten D, Janssens IA. Regulation of nitrogen fixation from free-living organisms in soil and leaf litter of two tropical forests of the Guiana shield. Plant and Soil, 2020, 450(1): 93-110. |
| [66] | Wang Q, Wang JL, Li YZ, Chen DW, Ao JH, Zhou WL, Shen DC, Li QW, Huang ZR, Jiang Y. Influence of nitrogen and phosphorus additions on N2-fixation activity, abundance, and composition of diazotrophic communities in a Chinese fir plantation. Science of the Total Environment, 2018, 619/620: 1530-1537. DOI:10.1016/j.scitotenv.2017.10.064 |
| [67] | Tang YQ, Yu GR, Zhang XY, Wang QF, Tian J, Niu SL, Tian DS, Ge JP. Different strategies for regulating free-living N2 fixation in nutrient-amended subtropical and temperate forest soils. Applied Soil Ecology, 2019, 136: 21-29. DOI:10.1016/j.apsoil.2018.12.014 |
| [68] | Barron AR, Wurzburger N, Bellenger JP, Wright SJ, Kraepiel AML, Hedin LO. Molybdenum limitation of asymbiotic nitrogen fixation in tropical forest soils. Nature Geoscience, 2009, 2(1): 42-45. DOI:10.1038/ngeo366 |
| [69] | Dynarski KA, Houlton BZ. Nutrient limitation of terrestrial free-living nitrogen fixation. New Phytologist, 2018, 217(3): 1050-1061. DOI:10.1111/nph.14905 |
| [70] | Chakraborty A, Bhakat K, Islam E. Arsenic contamination in agricultural soil reduces metabolic activity of total and free-living nitrogen-fixing bacteria as revealed by real-time qPCR. Soil and Sediment Contamination: an International Journal, 2017, 26(7/8): 736-748. |
| [71] | Shi W, Zhao HY, Chen Y, Wang JS, Han B, Li CP, Lu JY, Zhang LM. Organic manure rather than phosphorus fertilization primarily determined asymbiotic nitrogen fixation rate and the stability of diazotrophic community in an upland red soil. Agriculture, Ecosystems & Environment, 2021, 319: 107535. |
| [72] | Liao HK, Li YY, Yao HY. Fertilization with inorganic and organic nutrients changes diazotroph community composition and N-fixation rates. Journal of Soils and Sediments, 2018, 18(3): 1076-1086. DOI:10.1007/s11368-017-1836-8 |
| [73] | Xu YD, Zhang W, Zhong ZK, Guo SJ, Han XH, Yang GH, Ren CJ, Chen ZX, Dai YY, Qiao WJ. Vegetation restoration alters the diversity and community composition of soil nitrogen-fixing microorganisms in the loess hilly region of China. Soil Science Society of America Journal, 2019, 83(5): 1378-1386. DOI:10.2136/sssaj2019.03.0066 |
| [74] | Eaton WD, McGee KM, Larimer M, Hoke E, Karas O, Hernandez B, Wayland NA. Changes in soil bacterial communities, and carbon and nitrogen metrics as potential indicators of land use effects in a humid tropical forest. Pedobiologia, 2021, 85/86: 150730. DOI:10.1016/j.pedobi.2021.150730 |
| [75] | Mirza BS, Potisap C, Nüsslein K, Bohannan BJM, Rodrigues JLM. Response of free-living nitrogen-fixing microorganisms to land use change in the Amazon rainforest. Applied and Environmental Microbiology, 2014, 80(1): 281-288. DOI:10.1128/AEM.02362-13 |
| [76] | Shiozaki T, Chen YLL, Lin YH, Taniuchi Y, Sheu DS, Furuya K, Chen HY. Seasonal variations of unicellular diazotroph groups A and B, and Trichodesmium in the northern South China Sea and neighboring upstream Kuroshio Current. Continental Shelf Research, 2014, 80: 20-31. DOI:10.1016/j.csr.2014.02.015 |
| [77] | Gaby JC, Buckley DH. The use of degenerate primers in qPCR analysis of functional genes can cause dramatic quantification bias as revealed by investigation of nifH primer performance. Microbial Ecology, 2017, 74(3): 701-708. DOI:10.1007/s00248-017-0968-0 |
| [78] | Gaby JC, Buckley DH. A comprehensive evaluation of PCR primers to amplify the nifH gene of nitrogenase. PLoS One, 2012, 7(7): e42149. DOI:10.1371/journal.pone.0042149 |
| [79] | Wang Q, Quensen JF 3rd, Fish JA, Lee TK, Sun YN, Tiedje JM, Cole JR. Ecological patterns of nifH genes in four terrestrial climatic zones explored with targeted metagenomics using FrameBot, a new informatics tool. mBio, 2013, 4(5): e00592-e00513. |
| [80] |
Liu C, Li JB, Rui JP, An JX, Li XZ. The applications of the 16S rRNA gene in microbial ecology: current situation and problems. Acta Ecologica Sinica, 2015, 35(9): 2769-2788.
(in Chinese) 刘驰, 李家宝, 芮俊鹏, 安家兴, 李香真. 16S rRNA基因在微生物生态学中的应用. 生态学报, 2015, 35(9): 2769-2788. |
| [81] | Liu C, Cui YM, Li XZ, Yao MJ. Microeco: an R package for data mining in microbial community ecology. FEMS Microbiology Ecology, 2020, 97(2): fiaa255. |
| [82] | Louca S, Parfrey LW, Doebeli M. Decoupling function and taxonomy in the global ocean microbiome. Science, 2016, 353(6305): 1272-1277. DOI:10.1126/science.aaf4507 |
| [83] | Poly F, Monrozier LJ, Bally R. Improvement in the RFLP procedure for studying the diversity of nifH genes in communities of nitrogen fixers in soil. Research in Microbiology, 2001, 152(1): 95-103. DOI:10.1016/S0923-2508(00)01172-4 |
| [84] | Marusina AI, Boulygina ES, Kuznetsov BB, Tourova TP, Kravchenko IK, Gal'chenko VF. A system of oligonucleotide primers for the amplification of nifH genes of different taxonomic groups of prokaryotes. Microbiology, 2001, 70(1): 73-78. DOI:10.1023/A:1004849022417 |
| [85] | Ando S, Goto M, Meunchang S, Thongra-ar P, Fujiwara T, Hayashi H, Yoneyama T. Detection of nifH sequences in sugarcane (Saccharum officinarum L.) and pineapple (Ananas comosus[L.]Merr.). Soil Science and Plant Nutrition, 2005, 51(2): 303-308. DOI:10.1111/j.1747-0765.2005.tb00034.x |
| [86] | Rösch C, Mergel A, Bothe H. Biodiversity of denitrifying and dinitrogen-fixing bacteria in an acid forest soil. Applied and Environmental Microbiology, 2002, 68(8): 3818-3829. DOI:10.1128/AEM.68.8.3818-3829.2002 |
| [87] | Zehr JP, McReynolds LA. Use of degenerate oligonucleotides for amplification of the nifH gene from the marine cyanobacterium Trichodesmium thiebautii. Applied and Environmental Microbiology, 1989, 55(10): 2522-2526. DOI:10.1128/aem.55.10.2522-2526.1989 |
| [88] | Zani S, Mellon MT, Collier JL, Zehr JP. Expression of nifH genes in natural microbial assemblages in Lake George, New York, detected by reverse transcriptase PCR. Applied and Environmental Microbiology, 2000, 66(7): 3119-3124. DOI:10.1128/AEM.66.7.3119-3124.2000 |
| [89] | Ueda T, Suga Y, Yahiro N, Matsuguchi T. Remarkable N2-fixing bacterial diversity detected in rice roots by molecular evolutionary analysis of nifH gene sequences. Journal of Bacteriology, 1995, 177(5): 1414-1417. DOI:10.1128/jb.177.5.1414-1417.1995 |
| [90] |
Xie ZB, Zhang YH, Wang H. Advances and perspectives in paddy biological nitrogen fixation. Acta Pedologica Sinica, 2020, 57(3): 540-546.
(in Chinese) 谢祖彬, 张燕辉, 王慧. 稻田生物固氮研究进展及方向. 土壤学报, 2020, 57(3): 540-546. |
| [91] | Tang YF, Zhang MM, Chen AL, Zhang WZ, Wei WX, Sheng R. Impact of fertilization regimes on diazotroph community compositions and N2-fixation activity in paddy soil. Agriculture, Ecosystems & Environment, 2017, 247: 1-8. |
| [92] | Zhao JW, Tao Q, Li B, Luo JP, Zhang HY, Lu CL, Li QQ, Xu Q, Huang R, Li HX, Li B, Chen YL, Wang CQ. Low-pyrolysis-temperature biochar promoted free-living N2-fixation in calcareous purple soil by affecting diazotrophic composition. Geoderma, 2021, 388: 114969. DOI:10.1016/j.geoderma.2021.114969 |
| [93] | Saiz E, Sgouridis F, Drijfhout FP, Ullah S. Biological nitrogen fixation in peatlands: comparison between acetylene reduction assay and 15N2 assimilation methods. Soil Biology and Biochemistry, 2019, 131: 157-165. DOI:10.1016/j.soilbio.2019.01.011 |
| [94] | Bytnerowicz TA, Min E, Griffin KL, Menge DNL. Repeatable, continuous and real-time estimates of coupled nitrogenase activity and carbon exchange at the whole-plant scale. Methods in Ecology and Evolution, 2019, 10(7): 960-970. DOI:10.1111/2041-210X.13186 |
| [95] |
Li XZ, Chen Q. 15N isotopic dilution method to determine the biological nitrogen fixation. He Nong Xue Tong Bao, 1997, 18(6): 42-44.
(in Chinese) 李香真, 陈清. 15N同位素稀释法测定生物固氮量. 核农学通报, 1997, 18(6): 42-44. |
| [96] |
Nong Q, Lin L, Xie JL, Mo ZH, Huang X, Li CN. Characters identification and sugarcane growth promotion analysis of an endophytic nitrogen fixing bacteria GXS16. Microbiology China, 2021, 48(12): 4710-4718.
(in Chinese) 农倩, 林丽, 谢金兰, 莫璋红, 黄杏, 李长宁. 一株高效甘蔗内生固氮细菌GXS16的鉴定及其对甘蔗的促生长作用. 微生物学通报, 2021, 48(12): 4710-4718. |
| [97] |
Ma CM, Wang J, Xia X, Wang C, Lyu XC, Li S, Cheng J, Gong ZP. Study on absorption and distribution characteristics of nitrogen in soybeans with dual root systems based on 15N tracing technique. Journal of Plant Nutrition and Fertilizers, 2019, 25(11): 1909-1919.
(in Chinese) 马春梅, 王晶, 夏玄, 王畅, 吕晓晨, 李莎, 程娟, 龚振平. 基于15N示踪法的双根大豆系统氮素吸收和分配特性研究. 植物营养与肥料学报, 2019, 25(11): 1909-1919. DOI:10.11674/zwyf.18469 |
| [98] | Chalk PM. The strategic role of 15N in quantifying the contribution of endophytic N2 fixation to the N nutrition of non-legumes. Symbiosis, 2016, 69(2): 63-80. DOI:10.1007/s13199-016-0397-8 |
| [99] | 杨子文. 应用15N自然丰度技术量化陇东苜蓿生物固氮的研究. 兰州大学硕士学位论文, 2010. |
| [100] | Conrad KA, Dalal RC, Dalzell SA, Allen DE, Fujinuma R, Menzies NW. Soil nitrogen status and turnover in subtropical Leucaena-grass pastures as quantified by δ15N natural abundance. Geoderma, 2018, 313: 126-134. DOI:10.1016/j.geoderma.2017.10.029 |
| [101] | Chalk PM, Inácio CT, Chen DL. An overview of contemporary advances in the usage of 15N natural abundance (δ15N) as a tracer of agro-ecosystem N cycle processes that impact the environment. Agriculture, Ecosystems & Environment, 2019, 283: 106570. |
| [102] | Russow R, Veste M, Böhme F. A natural 15N approach to determine the biological fixation of atmospheric nitrogen by biological soil crusts of the Negev Desert. Rapid Communications in Mass Spectrometry, 2005, 19(23): 3451-3456. DOI:10.1002/rcm.2214 |
| [103] |
Chen (C/Z)X, Xi LQ, Yao T, Zhang H, Zhang DG. Research progress of biological nitrogen fixation methods. Grassland and Turf, 2005, 25(2): 24-26.
(in Chinese) 陈朝勋, 席琳乔, 姚拓, 张虎, 张德罡. 生物固氮测定方法研究进展. 草原与草坪, 2005, 25(2): 24-26. DOI:10.3969/j.issn.1009-5500.2005.02.006 |
| [104] | Xu R, Prentice IC. Modelling the demand for new nitrogen fixation by terrestrial ecosystems. Biogeosciences, 2017, 14(7): 2003-2017. DOI:10.5194/bg-14-2003-2017 |
| [105] | Herridge DF, Peoples MB, Boddey RM. Global inputs of biological nitrogen fixation in agricultural systems. Plant and Soil, 2008, 311(1/2): 1-18. |
| [106] | Staccone A, Liao WY, Perakis S, Compton J, Clark CM, Menge DC. A spatially explicit, empirical estimate of tree-based biological nitrogen fixation in forests of the United States. Global Biogeochemical Cycles, 2020, 34(2): e2019GB006241. |
| [107] | Soper FM, Taylor BN, Winbourne JB, Wong MY, Dynarski KA, Reis CRG, Peoples MB, Cleveland CC, Reed SC, Menge DNL, Perakis SS. A roadmap for sampling and scaling biological nitrogen fixation in terrestrial ecosystems. Methods in Ecology and Evolution, 2021, 12(6): 1122-1137. DOI:10.1111/2041-210X.13586 |
 2022, Vol. 62
2022, Vol. 62