中国科学院微生物研究所,中国微生物学会
文章信息
- 卞永霞, 剧柠, 郭蓉, 苟萌, 王媛媛. 2022
- BIAN Yongxia, JU Ning, GUO Rong, GOU Meng, WANG Yuanyuan.
- 原料乳冷藏过程中微生物细胞的宏蛋白组学分析
- Metaproteomic analysis of microbial cells during cold storage of raw milk
- 微生物学报, 62(7): 2782-2795
- Acta Microbiologica Sinica, 62(7): 2782-2795
-
文章历史
- 收稿日期:2021-11-17
- 修回日期:2022-04-07
- 网络出版日期:2022-04-20
原料乳在投入生产前通常需4 ℃冷藏一段时间(1–4 d)[1],而冷藏无法完全抑制原料乳中微生物的生长,其中嗜冷和适冷微生物所产生的一系列酶对原料乳及乳制品会造成负面作用[2–5]。近年来,研究者针对原料乳中的微生物多样性进行了分析,发现虽受地理区域、季节、储藏温度等因素的影响,但其中的微生物大多以假单胞菌属、黄杆菌属、沙雷氏菌属、不动杆菌属、明串珠菌属、乳球菌属、链球菌属、芽孢杆菌属为主[6–9]。同时研究者针对乳中的主要致腐微生物假单胞菌属的特性及检测方法进行了重点研究[10–11]。然而,整个原料乳冷藏过程是一个微生物与乳环境之间动态交互的过程,乳中微生物的基因表达与代谢物之间相互影响密不可分。只有了解冷藏过程中微生物与乳环境如何相互影响,才能解释原料乳最终造成品质劣变直至腐败的根本原因。目前关于这方面的研究较少。宏蛋白组学是研究环境中微生物蛋白质的集合[12],对微生物蛋白水平的研究能够提供宏基因组无法获取的信息[13–14]。该技术通过质谱法采集特定环境条件下全体微生物蛋白表达信息,探索微生物组成以及代谢方式,揭示微生物与环境之间的相互关系。随着质谱技术的发展及数据库信息的完善,宏蛋白组学已成为研究微生物群落时空表征的有效方法之一[15]。本研究利用非标记(label-free)宏蛋白组学策略,采用LC-MS/MS的方法对原料乳4 ℃冷藏6 d期间的微生物菌体蛋白质进行相对定量分析,从分子水平上表征原料乳冷藏过程中品质劣变的本质。
1 材料与方法 1.1 原料乳的收集及预处理采集宁夏银川春天然乳业乳罐原料乳。经检测,该牧场的原料乳体细胞数 < 2.0×105个/mL,菌落总数 < 1.0×105 CFU/mL,蛋白质含量 > 3.0 g/100 mL。严格遵循无菌操作采样,采样后装入灭菌后的样品瓶中,并立即置于放有冰袋的隔热背包中,2 h内运回实验室,置于4 ℃下冷藏。依据前期Illumina Hiseq宏基因组测序平台的测序结果,取门水平物种丰度有显著转折的时间点,即冷藏0、1、3、4、6 d时间点的原料乳样品,并进行原料乳微生物菌体蛋白质组学分析。每个时间点取3个平行样本。
1.2 原料乳中微生物菌体蛋白质的提取及肽段酶解每次取37 mL原料乳样品120 000×g超速离心30 min,沉淀加入31 mL预冷的PBS缓冲液,重悬后再次120 000×g超速离心30 min,重复洗涤3次,以去除牛乳蛋白,按照100 µg沉淀添加200 μL的比例加入SDT裂解液,以裂解菌体释放蛋白质,SDT裂解液由4%十二烷基硫酸钠(SDS),100 mmol/L Tris/HCl (三氨基甲烷盐酸盐,pH=7.6),0.1 mol/L二硫苏糖醇(DTT)制成;裂解后的蛋白粗提取液用ProteoMinerTM Protein Enrichment Small-Capacity Kit富集低丰度蛋白,以消除牛奶中高丰度蛋白的影响。采用BCA法进行蛋白质定量检测后,将富集的低丰度蛋白采用filter aided sample preparation (FASP)方法进行胰蛋白酶酶解。取150 µg蛋白溶液,按照FASP方法进行处理后,加入40 µL胰蛋白酶缓冲液(Trypsin buffer) (0.665 4 µg胰蛋白酶in 40 µL 100 mmol/L NH4HCO3)进行酶解。酶解结束后,采用C18 Cartridge对肽段进行脱盐,肽段冻干后加入40 μL 0.1%甲酸溶液复溶,利用紫外光吸收法进行肽段定量(OD280)。
1.3 LC-MS/MS数据采集样品采用纳升流速的HPLC液相系统Easy nLC进行分离。洗脱液A液为0.1%甲酸水溶液,B液为0.1%甲酸乙腈水溶液(乙腈为84%)。色谱柱以95%的A液平衡,样品由自动进样器上样到上样柱(Thermo Scientific Acclaim PepMap100,100 μm×2 cm,nanoViper C18),经过分析柱(Thermo Science Easy column,75 μm×10 cm,C18-A2)分离,流速为300 nL/min。色谱分离后用Q-Exactive质谱仪对样品进行质谱分析。
1.4 原料乳微生物菌体蛋白质的鉴定和定量分析获得质谱原始数据之后,采用三步法查库策略,依照前期宏基因组实验得到的物种列表构建的NR数据库进行分析,首先使用X!Tandem 2017.2.1.4软件对数据库先后进行两次串联检索,每次通过过滤的蛋白质整合成新的数据库,作为下一轮搜索用数据库,最后使用MaxQuant1.6.0.16进行数据检索,设定参数:enzyme为trypsin;max missed cleavage设为2;固定修饰设定Carbamidomethy C;可变修饰设定oxidation M。Protein FDR < 0.01,peptides FDR < 0.01,main search设为6 mg/L,first search设为20 mg/L。用无标记定量(label-free quantitative,LFQ)值表征蛋白质丰度。
1.5 原料乳微生物菌体蛋白质的物种来源分析利用前期宏基因组实验得到的物种列表建成的数据库,对样本中蛋白质的物种组成进行注释,去除非微生物蛋白。将注释到同一物种条目下的所有唯一物种肽段的定量值之和作为该物种的定量值,计算在各样本中该物种占所有物种的相对丰度,以展现各时间点样本主要的菌群及其占比和菌群结构变化。
1.6 原料乳不同储存时间下的微生物菌体差异蛋白质分析将原料乳冷藏期间微生物菌体蛋白质进行前后两个时间节点差异蛋白变化比较,以差异表达倍数大于1.2或小于0.833,且P < 0.05为标准,筛选差异蛋白质,并对其进行KEGG代谢通路富集。
1.7 数据分析提取目标蛋白对应的COG (cluster of orthologous groups of proteins)数据库(http://www.ncbi.nlm.nih.gov/COG/)注释信息,按照COG功能分类划分为26个COG功能类别,分别统计每个COG功能类别注释到的目标蛋白表达量强度总和,绘制堆积柱状图展示。采用京都基因和基因组百科全书(KEGG)数据库(http://www.genome.jp/kegg/pathway.html)对所有鉴定到的微生物蛋白及差异蛋白进行鉴定,注释蛋白质参与的通路信息,对目标蛋白质集合进行KEGG通路注释的富集分析。
2 结果与分析 2.1 原料乳中微生物菌体蛋白质肽段及蛋白质的鉴定非标记(label-free)法是利用蛋白丰度和峰面积或MS/MS质谱数的相关性来定量的一种方法[16],本研究利用非标记定量蛋白质组技术从原料乳各个时间采样点获取的微生物蛋白中共鉴定出590个肽段,利用这些肽段鉴定出冷藏过程中对应的341个蛋白质。由Venn图(图 1)可知,5个时间点样本中检测到的共同蛋白质为56个。各时间点特有的蛋白质数量分别为1、7、5、21、184个,冷藏后期微生物变化较大,4 d (含4 d)后样本中的蛋白质占所有检出蛋白质的60.12%。主成分分析图(图 2)的横纵坐标分别表示变量与第一主成分、第二主成分的相关系数。如图显示,样本组间差异大于组内差异,说明实验稳定性较好。所有样本中,0 d和1 d的样本较为接近,样本相似性较高,1 d和4 d组间平行样本分离趋势较其他组大。
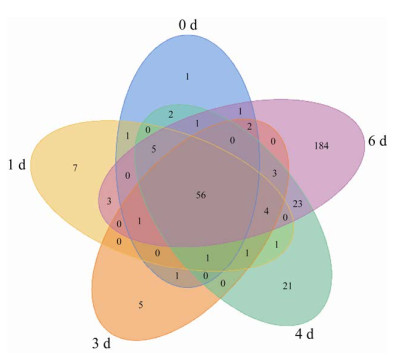
|
| 图 1 冷藏期间原料乳各取样时间点微生物蛋白数量Venn图 Figure 1 Venn diagram of microbial protein quantity at each sampling time point of raw milk during refrigeration. |
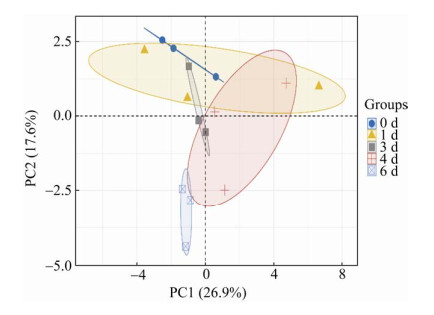
|
| 图 2 冷藏期间原料乳各取样时间点微生物蛋白样本主成分分析图 Figure 2 Principal component analysis of microbial protein samples at each sampling time point of raw milk during refrigeration. |
2.2 原料乳微生物菌体蛋白质物种来源分析
对所有样本中鉴定到的微生物菌体蛋白质进行物种注释,结果如图 3所示。原料乳中鉴定到的微生物菌体蛋白质按占比高低依次来源于假单胞菌属(Pseudomonas)、梭菌属(Clostridium)、乳球菌属(Lactococcus)、不动杆菌属(Acinetobacter)、分枝杆菌属(Mycobacterium)、乳杆菌属(Lactobacillus)、黄杆菌属(Flavobacterium)、肠球菌属(Enterococcus)、链球菌属(Streptococcus)、环丝菌属(Brochothrix)和无浆体属(Anaplasma)。

|
| 图 3 冷藏期间原料乳各取样时间点微生物菌体蛋白注释结果图 Figure 3 Graph of microbial protein annotation results for each sampling time point of raw milk during refrigeration. |
随时间变化来源于不同菌属的蛋白质相对丰度的变化如图 4所示。整个冷藏过程中相对丰度较高的蛋白质主要来源于假单胞菌属(Pseudomonas)、梭菌属(Clostridium)、黄杆菌属(Flavobacterium)以及环丝菌属(Brochothrix)。假单胞菌属蛋白质相对丰度在整个过程中始终占据主要地位(38.39%–42.41%),冷藏储存4 d时达到最高,为42.41%。来源于梭菌属的蛋白质相对丰度在冷藏过程中占比幅度为19.61%–27.57%,其中冷藏1 d时相对丰度最低为19.61%。黄杆菌属和环丝菌属表达的蛋白质相对丰度总体呈下降趋势,分别从冷藏初期的15.91%和9.20%降至冷藏末期的10.98%和6.15%,特别是,黄杆菌属表达的蛋白质在4 d时有明显的下降,从16.07%降至11.32%。另外,初始阶段含量极少的乳球菌属与链球菌属表达的蛋白质在冷藏末期(6 d)有一定幅度的提升,分别从0.02%和0.64%升至3.22%和2.74%。
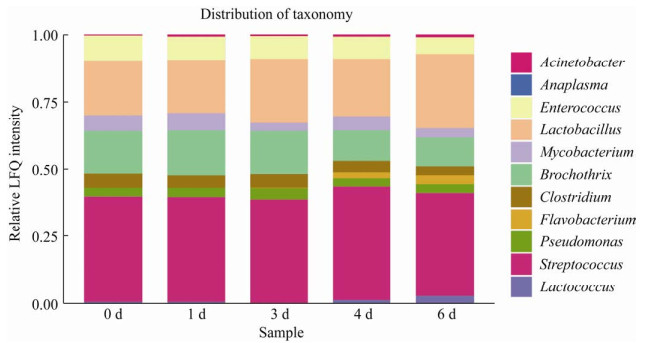
|
| 图 4 各时间点样本中主要的蛋白质物种菌群及其占比图 Figure 4 Main protein species and their proportion in the samples at each time point. |
2.3 原料乳微生物菌体蛋白质的COG分析
将鉴定得到的蛋白质和COG数据库进行比对分析,如图 5所示。所有样本共注释到22个条目,其中,对应着未知功能(function unknown)的S条目蛋白质数目最多,5个时间点分别有:35、36、32、42和38种蛋白质,这意味着原料乳中大部分微生物菌体蛋白质的功能是未知的。除此之外,蛋白质数目前五名的功能条目为L、G、K、J、E,分别对应着复制(replication),重组和修复(replication,recombination and repair)、碳水化合物的运输和代谢(carbohydrate transport and metabolism)、转录(transcription)、翻译,核糖体结构和生物发生(translation, ribosomal structure and biogenesis)、氨基酸转运和代谢(amino acid transport and metabolism),即冷藏过程中的微生物蛋白主要执行着信息存储与处理和代谢相关的功能。大多数条目下的蛋白质在冷藏过程中变化不大,而执行氨基酸转运和代谢功能、翻译,核糖体结构和生物发生这两种功能的蛋白质数量分别从3 d的13、14个增加至4 d的21、25个,上升幅度较大,说明冷藏4 d乳体系中微生物的基因表达发生了较大改变。
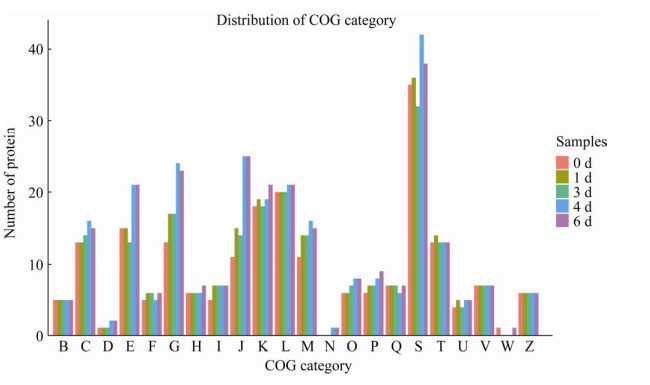
|
| 图 5 原料乳微生物菌体蛋白质COG功能分类图 Figure 5 Functional classification map of raw milk microbial protein COG. B: chromatin structure and dynamics; C: energy production and conversion; D: cell cycle control, cell division, chromosome partitioning; E: amino acid transport and metabolism; F: nucleotide transport and metabolism; G: carbohydrate transport and metabolism; H: coenzyme transport and metabolism; I: lipid transport and metabolism; J: translation, ribosomal structure and biogenesis; K: transcription; L: replication, recombination and repair; M: cell wall/membrane/envelope biogenesis; N: cell motility; O: posttranslational modification, protein turnover, chaperones; P: inorganic ion transport and metabolism; Q: secondary metabolites biosynthesis, transport and catabolism; S: function unknown; T: signal transduction mechanisms; U: intracellular trafficking, secretion, and vesicular transport; V: defense mechanisms; W: extracellular structures; Z: cytoskeleton. |
参与不同功能条目的蛋白质相对丰度如图 6所示,在原料乳整个冷藏过程中,执行M、P、K、S、E这些功能的微生物蛋白质始终保持较高的相对丰度。这些条目分别对应着细胞壁/膜/包膜生物发生(cell wall/membrane/envelope biogenesis)、无机离子的运输与代谢(inorganic ion transport and metabolism)、转录(transcription)、未知功能(function unknown)和氨基酸转运和代谢(amino acid transport and metabolism)。其中,参与细胞壁/膜/包膜生物发生、氨基酸转运和代谢及功能未知的微生物蛋白质在冷藏中始终保持着较多数目和较高丰度。这说明,乳环境中微生物群落的抗逆性能在不断增加快速适应变换的环境[17]。值得关注的是,冷藏4 d,参与细胞壁/膜/包膜生物发生功能的蛋白质相对丰度有明显的降低(30.48%–24.61%);参与未知功能的蛋白质相对丰度总体保持上升趋势,在冷藏4 d有明显的跃增(10.18%–14.05%)。
2.4 原料乳微生物菌体蛋白质KEGG通路分析
为了探究微生物基因表达对原料乳的影响,对所检测到的微生物菌体蛋白质进行KEGG通路注释。这些微生物蛋白质参与的通路主要有核糖体(ribosome)、糖酵解/糖异生(glycolysis/gluconeogenesis)、群体感应(quorum sensing)、双组分系统(two-component system)、HIF-1信号通路(HIF-1 signaling pathway)和ABC转运蛋白(ABC transporters)等,在冷藏各阶段蛋白质数目变化如图 7所示,许多代谢通路在冷藏4 d时,参与的微生物蛋白数目有明显上升,与群体感应(quorum sensing)通路相关的蛋白质在冷藏4 d出现,数目从4 d时的7个最终上升至6 d时的15个。ABC转运体代谢通路与HIF-1信号通路的蛋白质数目也呈上升趋势,在4 d时上升幅度较大,分别从3 d的3个、2个升至4 d的8个、10个。参与这些通路的蛋白质数目的明显变化说明冷藏4 d有可能是原料乳中微生物作用的一个关键时间节点。
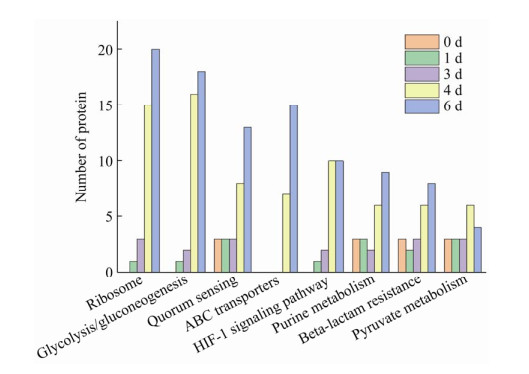
|
| 图 7 原料乳冷藏过程中微生物菌体蛋白质参与KEGG通路数目变化分析图 Figure 7 Analysis of changes in the number of microbial proteins involved in the KEGG pathway during the cold storage of raw milk. |
对所有定量到的微生物蛋白质相对丰度进行KEGG注释分析如图 8所示,相对丰度排名前三的代谢通路为氨基糖和核苷酸糖代谢、ABC转运蛋白和双组分系统。其中参与氨基糖和核苷酸糖代谢通路的蛋白质相对丰度前3天变化并不显著,第四天出现大幅度下降(30.04%降至23.89%);参与ABC转运蛋白通路的蛋白质相对丰度在第三天有小幅度的增加(7.89%升至8.58%);参与双组分系统代谢通路的蛋白质相对丰度在冷藏过程中持续波动,在冷藏4 d时最高占8.58%。淀粉和蔗糖代谢(starch and sucrose metabolism)、碳水化合物的消化吸收代谢通路(carbohydrate digestion and absorption)在原料乳冷藏过程中也占有重要作用,且这两条代谢通路相对丰度总体呈下降趋势,从冷藏初期的4.95%和4.96%降至冷藏末期的3.64%和3.62%。

|
| 图 8 原料乳冷藏过程中微生物菌体蛋白质KEGG Pathway分布比例图 Figure 8 Distribution ratio of microbial protein KEGG Pathway during raw milk refrigeration. |
2.5 原料乳冷藏过程中微生物差异表达蛋白质功能的通路富集
为探究原料乳冷藏过程中差异蛋白质的动态变化,将原料乳冷藏时间进行前后2个时间节点差异蛋白变化比较,以差异表达倍数大于1.2或小于0.833,且P < 0.05为标准,筛选差异蛋白质。结果如表 1所示,随着冷藏时间延长,与前一时间点相比,各时间点差异蛋白质数目逐渐增多,差异蛋白质富集在不同的通路中。
| Comparison group | Protein IDs | Difference protein description | Change | Species origin of protein | KEGG enrichment pathway |
| 1 d/0 d | WP_058790836.1 | Tyrosine-tRNA ligase | Up | Pseudomonas psychrotolerans | Aminoacyl-tRNA biosynthesis |
| WP_133442427.1 | Septum formation initiator family protein | Up | Lactobacillus sp. HSLZ-75 | ||
| 3 d/1 d | WP_026727825.1 | Pyridoxal phosphate-dependent aminotransferase | Up | Flavobacterium denitrificans | |
| WP_080062998.1 | Chemotaxis protein CheA | Up | Clostridium hungatei | ||
| WP_087674308.1 | Translation elongation factor EF-1 subunit alpha | Up | Pseudomonas syringae | ||
| 4 d/3 d | WP_079505296.1 | Elongation factor Tu | Up | Lactococcus piscium | Quorum sensing |
| WP_053579372.1 | MULTISPECIES: 50S ribosomal protein L7/L12 | Up | Acinetobacter | Galactose metabolism | |
| WP_103230885.1 | actin, cytoplasmic 2 | Down | Clostridium sp. chh4-2 | ||
| 6 d/4 d | WP_003138831.1 | MULTISPECIES: cold-shock protein | Up | Lactococcus | Viral carcinogenesis |
| WP_103230885.1 | Actin, cytoplasmic 2 | Up | Clostridium sp. chh4-2 | Alcoholism | |
| WP_079864572.1 | Hypothetical protein | Up | Acinetobacter baumannii | ||
| WP_019781240.1 | Galactose-6-phosphate isomerase subunit LacB, partial | Down | Streptococcus sobrinus | ||
| WP_129751688.1 | Hypothetical protein | Down | Flavobacterium beibuense | ||
| WP_133442427.1 | Septum formation initiator family protein | Down | Lactobacillus sp. HSLZ-75 | ||
| WP_029161742.1 | Transporter | Down | Clostridium scatologenes | ||
| WP_058790836.1 | Tyrosine-tRNA ligase | Down | Pseudomonas psychrotolerans | ||
| PZQ37592.1 | RNA polymerase subunit sigma | Down | Pseudomonas putida | ||
| EUJ34615.1 | Monovalent cation/H+ antiporter subunit A | Down | Brochothrix thermosphacta | ||
| WP_109833933.1 | TypeⅠglyceraldehyde- 3-phosphate dehydrogenase | Down | Lactococcus piscium | ||
| WP_057740323.1 | Glyoxalase | Down | Lactobacillus alimentarius | ||
| WP_079505296.1 | Elongation factor Tu | Down | Lactococcus piscium |
对原料乳冷藏过程中4个比较组的差异表达蛋白进行了综合分析,结果表明,根据筛选标准,共产生了21个差异蛋白质,大部分差异蛋白质在冷藏4 d后产生,变化多样且大多数功能未知。已知的差异蛋白富集在氨酰基-tRNA生物合成、群体感应、半乳糖代谢、病毒致癌作用和酒精中毒等通路。其中,冷藏0–1 d的差异蛋白富集在氨基酰-tRNA的生物合成通路,氨基酰-tRNA是蛋白质生物合成的中间转运产物,为蛋白质生物合成提供原料[18];冷藏3–4 d时差异蛋白通路富集在群体感应和半乳糖代谢上;冷藏6 d后原料乳的差异蛋白检出最多,这些蛋白富集通路集中在一些与人类疾病相关的微生物蛋白,如病毒致癌作用和酒精中毒。值得注意的是,所检出的21个差异蛋白中有9个未检测到KEGG富集通路,这说明这些蛋白所参与的具体通路尚不清晰,有待进一步研究。
3 讨论原料乳在加工前的冷藏期间其中的微生物多样性是使乳中代谢物和蛋白组成变化的重要原因,这些变化影响着原料乳的理化性质。微生物产生的蛋白质(酶)在原料乳冷藏过程中发挥重要作用,因此,明确原料乳微生物结构,了解冷藏过程中涉及的微生物群和蛋白质的作用,对乳制品的生产具有重要的指导意义。本实验室前期针对冷藏过程中的原料乳微生物多样性进行了研究,然而这些微生物在冷藏各个阶段的基因表达情况及所发挥的作用尚不明晰。宏基因组学研究能够发现微生物群落中潜在基因的功能[19],宏转录组学可对基因表达和活性的程度进行较为深入的了解[20],但其细胞定位与活动调节是发生在蛋白质水平上的,因而,转录组与蛋白质组得到的信息往往大不相同[21]。
本研究采用的非标记(label-free)定量法测定了原料乳冷藏过程中乳环境的微生物菌体蛋白质,在微生物的蛋白提取方法上,首先对原料乳样品进行了超速离心,且对裂解后的蛋白粗提取液用ProteoMinerTM Protein Enrichment Small-Capacity Kit富集低丰度蛋白(ProteoMinerTM技术是一种样品制备工具,通过使用一个高度多样化的基于珠子的组合肽配体文库来完成,当复杂的生物样品应用到珠上时,高丰度的蛋白质饱和其高亲和力的配体,多余的蛋白质被冲走),在后期数据分析时,以宏基因实验结果为参考,针对已测定微生物的基因组数据进行检索,这种操作方法基本保证所分析的蛋白是来自原料乳中的微生物蛋白。研究结果表明,原料乳在4 ℃冷藏6 d的过程中,60.12%微生物细胞的菌体蛋白在冷藏的4 d及4 d后产生;COG分析发现,与氨基酸转运和代谢功能、翻译,核糖体结构和生物发生功能相关的蛋白质在4 d时数量显著增加;在KEGG分析中发现,参与糖酵解/糖异生、ABC转运蛋白、氨基糖和核苷酸糖代谢等通路的蛋白数量在冷藏4 d后均出现了显著变化,因此,4 d或可作为原料乳冷藏的关键时间点。
本实验前期进行的宏基因组研究结果表明,原料乳在4 ℃冷藏期间,其中的优势微生物为不动杆菌属、链球菌属、无浆体属、假单胞菌属等[22],然而,通过对在整个原料乳冷藏过程中作为这些微生物活动的主要体现者——蛋白质(酶)的分析显示,除假单胞菌属外,其余优势微生物却始终保持较低丰度(< 3%),这也从侧面反映了微生物群落中虽然某些种类的微生物占据数量优势,但其参与代谢活动的基因表达量并不一定占据优势,该结果与吴重德等的[23]观点一致,即使是同一生物因受环境等因素的影响,其所表达的蛋白质也可能有所不同。在通常情况下,生物的基因组是少量表达的,而且因受到生物生存环境及内在状态变化的影响,表达的基因类型及表达程度会表现出较大的差别,并且这种差别存在着严格调控的时空特异性[24–25]。
对原料乳冷藏过程中的微生物菌体蛋白质进行COG功能分析发现,冷藏4 d时,执行氨基酸转运和代谢的微生物蛋白数量从3 d的13个增加至21个。结合宏基因检测结果分析[26],此时乳球菌属的相对丰度从3 d的4.54%增加至10.67%,成为优势菌属。有研究表明乳球菌属可参与蛋白质水解,将氨基酸转化为风味化合物[27],因此,氨基酸转运和代谢功能的提升可能是乳球菌属适应乳环境并在原料乳中分泌了一些蛋白质,参与分解代谢乳中蛋白质的表现。宏基因组分析结果显示从4 d起,假单胞菌属不再是优势菌属(从3 d的13.57%降至3.97%),但冷藏整个阶段所检测到的假单胞菌属的蛋白质占比却一直很高(图 4),说明这类嗜冷微生物在冷藏过程中其代谢活动一直表现活跃。除此之外,分析显示与细菌细胞壁/膜/包膜生物发生、转录等微生物生存所必需功能相关的蛋白质在冷藏过程数目也相对较多,相对丰度也较高。有研究表明,微生物细胞壁、膜的产生与变化能够感知冷刺激,使微生物更好的适应环境[28–29]。这说明原料乳中的微生物在储存器的低温环境下也在不断努力适应着低温的环境。
对冷藏过程中的微生物菌体蛋白质进行KEGG通路分析显示,参与群体感应通路的蛋白数量在4 d时显著增加。群体感应是细菌细胞间通过对自诱导物浓度的感知,调节细菌行为和基因表达的一种现象[30],并在调控细菌生物被膜的形成过程中起重要作用。而在有关原料乳嗜冷菌致腐潜力的研究中发现细胞生物被膜的形成是菌群数量增加的主要原因[6, 31],因此可以推断群体感应调控生物被膜形成对此阶段嗜冷微生物的迅速增长发挥了重要作用。此外,本研究中发现,原料乳冷藏4 d时参与ABC转运蛋白数目增加。ABC转运蛋白是一类跨膜运输蛋白家族,在生物体内参与多种物质的转运积累、有害物质解毒等生理活动[32]。有研究指出ABC转运蛋白能够促进氨基酸、糖类等营养物质的跨膜运输,同时也可将不利于细胞生长的物质排出胞外,进而减轻细胞生长负荷,促进细胞生长[33]。因此,冷藏原料乳中微生物ABC转运蛋白的增加有助于调节细胞膜的通透性,提高微生物对原料乳底物的利用程度,增强对乳环境的适应性。另外,在原料乳冷藏的整个过程中,与双组分系统相关的蛋白数量保持稳定且具有较高的相对丰度。双组分系统是细菌中广泛存在的信号转导途径,并能与组氨酸激酶协同以调控细菌生物被膜的形成,其在适应环境变化方面起着重要作用[34]。总体来看,虽然冷藏过程中微生物种类和数量发生着变化,但通过群体感应、ABC转运蛋白及双组分系统来适应乳环境的变化,增强其抗逆性,是造成冷藏原料乳复杂体系中微生物存在并不断演替的重要原因。
宏蛋白组学技术已经应用于多种环境微生物群落的研究中,如人体肠道[35]、发酵食品[36–37]、土壤[38]、海洋[39]中微生物区系变化。在宏蛋白质组学研究中一个相当大的挑战是蛋白质推断,它直接影响分类和功能注释,最好是通过彻底的宏基因组测序和注释获得的参考蛋白质组来分析和解释所获得的质谱数据。本研究虽然是在前期宏基因组学的研究基础上开展[22, 26],但无论是对所检测到的蛋白质还是各阶段差异蛋白质所参与的KEGG通路的富集,仍有相当大部分信息是未知的。这与宏蛋白组学应用于乳制品加工的研究缺乏,数据库中可参考信息有限有关,本实验采用的label-free定量方法通常依赖于每个蛋白质光谱匹配的数量,肽检测的缺乏也确实影响了定量工作,这也是检测方法的局限性对实验结果的影响。另外,受到微生物样本的异质性和复杂性以及缺乏标准化蛋白质样本的影响,使微生物样本中细微变化的量化变得复杂[40–41]。本文只是对原料乳中微生物菌体细胞的蛋白质组进行了分析,但对于原料乳在储存期间原料乳中的微生物所分泌的蛋白的宏蛋白质组的分析,目前还缺少有效的方法,还难以进行分析,这也为探索原料乳这种高丰度蛋白存在下的低丰度微生物蛋白提供了相关研究思路。
| [1] | Li L, Renye JA Jr, Feng L, Zeng QK, Tang Y, Huang L, Ren DX, Yang P. Characterization of the indigenous microflora in raw and pasteurized buffalo milk during storage at refrigeration temperature by high-throughput sequencing. Journal of Dairy Science, 2016, 99(9): 7016-7024. DOI:10.3168/jds.2016-11041 |
| [2] | Von Neubeck M, Baur C, Krewinkel M, Stoeckel M, Kranz B, Stressler T, Fischer L, Hinrichs J, Scherer S, Wenning M. Biodiversity of refrigerated raw milk microbiota and their enzymatic spoilage potential. International Journal of Food Microbiology, 2015, 211: 57-65. DOI:10.1016/j.ijfoodmicro.2015.07.001 |
| [3] | Salgado CA, De Almeida FA, Barros E, Baracat- Pereira MC, Baglinière F, Vanetti MCD. Identification and characterization of a polyurethanase with lipase activity from Serratia liquefaciens isolated from cold raw cow's milk. Food Chemistry, 2021, 337: 127954. DOI:10.1016/j.foodchem.2020.127954 |
| [4] | Bekker A, Jooste P, Steyn L, Bothma C, Hugo A, Hugo C. Lipid breakdown and sensory analysis of milk inoculated with Chryseobacterium joostei or Pseudomonas fluorescens. International Dairy Journal, 2016, 52: 101-106. DOI:10.1016/j.idairyj.2015.09.003 |
| [5] | Zhang D, Palmer J, Teh KH, Calinisan MMA, Flint S. Milk fat influences proteolytic enzyme activity of dairy Pseudomonas species. International Journal of Food Microbiology, 2020, 320: 108543. DOI:10.1016/j.ijfoodmicro.2020.108543 |
| [6] | Hahne J, Isele D, Berning J, Lipski A. The contribution of fast growing, psychrotrophic microorganisms on biodiversity of refrigerated raw cow's milk with high bacterial counts and their food spoilage potential. Food Microbiology, 2019, 79: 11-19. DOI:10.1016/j.fm.2018.10.019 |
| [7] | Porcellato D, Aspholm M, Skeie SB, Monshaugen M, Brendehaug J, Mellegård H. Microbial diversity of consumption milk during processing and storage. International Journal of Food Microbiology, 2018, 266: 21-30. DOI:10.1016/j.ijfoodmicro.2017.11.004 |
| [8] | Ribeiro JC Jr, De Oliveira AM, De G Silva F, Tamanini R, De Oliveira ALM, Beloti V. The main spoilage-related psychrotrophic bacteria in refrigerated raw milk. Journal of Dairy Science, 2018, 101(1): 75-83. DOI:10.3168/jds.2017-13069 |
| [9] |
Zhang SF, Xue YL, Cui Y, Han JH, Zhu H, Wang SJ. Research progress of psychrophilic bacteria and thermostable enzymes in milk. China Dairy Industry, 2020, 48(11): 35-38.
(in Chinese) 张树飞, 薛玉玲, 崔玥, 韩俊华, 朱宏, 王世杰. 牛乳中嗜冷菌及其耐热酶研究进展. 中国乳品工业, 2020, 48(11): 35-38. |
| [10] | Schlusselhuber M, Girard L, Cousin FJ, Lood C, De Mot R, Goux D, Desmasures N. Pseudomonas crudilactis sp. nov., isolated from raw milk in France. Antonie Van Leeuwenhoek, 2021, 114(6): 719-730. DOI:10.1007/s10482-021-01552-4 |
| [11] | Maier C, Hofmann K, Huptas C, Scherer S, Wenning M, Lücking G. Simultaneous quantification of the most common and proteolytic Pseudomonas species in raw milk by multiplex qPCR. Applied Microbiology and Biotechnology, 2021, 105(4): 1693-1708. DOI:10.1007/s00253-021-11109-0 |
| [12] | Rodríguez-Valera F. Environmental genomics, the big picture?. FEMS Microbiology Letters, 2004, 231(2): 153-158. DOI:10.1016/S0378-1097(04)00006-0 |
| [13] | Hettich RL, Pan CL, Chourey K, Giannone RJ. Metaproteomics: harnessing the power of high performance mass spectrometry to identify the suite of proteins that control metabolic activities in microbial communities. Analytical Chemistry, 2013, 85(9): 4203-4214. DOI:10.1021/ac303053e |
| [14] | Kowalchuk GA, Heidelberg JF, Bailey MJ. The ISME journal: multidisciplinary journal of microbial ecology. The ISME Journal, 2007, 1(1): 1-3. DOI:10.1038/ismej.2007.13 |
| [15] | Kleiner M. Metaproteomics: much more than measuring gene expression in microbial communities. mSystems, 2019, 4(3): e00115-e00119. |
| [16] | Neilson KA, Ali NA, Muralidharan S, Mirzaei M, Mariani M, Assadourian G, Lee A, Van Sluyter SC, Haynes PA. Less label, more free: approaches in label-free quantitative mass spectrometry. Proteomics, 2011, 11(4): 535-553. DOI:10.1002/pmic.201000553 |
| [17] | Zhao CC, Eun JB. Shotgun metagenomics approach reveals the bacterial community and metabolic pathways in commercial Hongeo product, a traditional Korean fermented skate product. Food Research International, 2020, 131: 109030. DOI:10.1016/j.foodres.2020.109030 |
| [18] | Perona JJ, Hadd A. Structural diversity and protein engineering of the aminoacyl-tRNA synthetases. Biochemistry, 2012, 51(44): 8705-8729. DOI:10.1021/bi301180x |
| [19] |
Feng MQ. Research progress of metagenomics. Journal of Anhui Agricultural Sciences, 2008, 36(2): 415-416.
(in Chinese) 冯美琴. 宏基因组学的研究进展. 安徽农业科学, 2008, 36(2): 415-416. DOI:10.3969/j.issn.0517-6611.2008.02.013 |
| [20] | Song ZW, Du H, Zhang Y, Xu Y. Unraveling core functional microbiota in traditional solid-state fermentation by high-throughput amplicons and metatranscriptomics sequencing. Frontiers in Microbiology, 2017, 8: 1294. DOI:10.3389/fmicb.2017.01294 |
| [21] |
Ma HX, Zhang LL, Sun XM, Zhang HQ, He MX, Chen GJ, Wang LS. Understanding microbial communities and their functions by meta-omics approaches. Microbiology China, 2015, 42(5): 902-912.
(in Chinese) 马海霞, 张丽丽, 孙晓萌, 张怀强, 何明雄, 陈冠军, 王禄山. 基于宏组学方法认识微生物群落及其功能. 微生物学通报, 2015, 42(5): 902-912. |
| [22] |
Wang YY, Ju N, Gou M, Luo YL, Li PY. Metagenomics approach to explore the changes of bacterial community in raw milk during refrigerated storage. Transactions of the Chinese Society of Agricultural Engineering, 2020, 36(22): 333-340.
(in Chinese) 王媛媛, 剧柠, 苟萌, 罗玉龙, 李璞钰. 宏基因组学探究原料乳冷藏过程菌群变化规律. 农业工程学报, 2020, 36(22): 333-340. DOI:10.11975/j.issn.1002-6819.2020.22.037 |
| [23] |
Wu CD, Huang J, Zhou RQ. Recent advance and application of metaproteomics. Food and Fermentation Industries, 2016, 42(5): 259-263.
(in Chinese) 吴重德, 黄钧, 周荣清. 宏蛋白质组学研究进展及应用. 食品与发酵工业, 2016, 42(5): 259-263. |
| [24] | Maron PA, Ranjard L, Mougel C, Lemanceau P. Metaproteomics: a new approach for studying functional microbial ecology. Microbial Ecology, 2007, 53(3): 486-493. DOI:10.1007/s00248-006-9196-8 |
| [25] |
Long J, Yang XH, Yu GB. Development and prospect of microbial proteomics. Biotechnology Bulletin, 2006(S1): 91-94.
(in Chinese) 龙娟, 杨晓红, 于桂宝. 微生物蛋白组学的发展及前景. 生物技术通报, 2006(S1): 91-94. |
| [26] | 王媛媛. 冷藏原料乳品质变化过程中微生物与乳代谢物关系探究. 宁夏大学硕士学位论文, 2021. |
| [27] | Smit G, Smit BA, Engels WJM. Flavour formation by lactic acid bacteria and biochemical flavour profiling of cheese products. FEMS Microbiology Reviews, 2005, 29(3): 591-610. DOI:10.1016/j.fmrre.2005.04.002 |
| [28] | Lamendella R, Domingo JWS, Ghosh S, Martinson J, Oerther DB. Comparative fecal metagenomics unveils unique functional capacity of the swine gut. BMC Microbiology, 2011, 11: 103. DOI:10.1186/1471-2180-11-103 |
| [29] | Erickson AR, Cantarel BL, Lamendella R, Darzi Y, Mongodin EF, Pan CL, Shah M, Halfvarson J, Tysk C, Henrissat B, Raes J, Verberkmoes NC, Fraser CM, Hettich RL, Jansson JK. Integrated metagenomics/ metaproteomics reveals human host-microbiota signatures of Crohn՚s disease. PLoS One, 2012, 7(11): e49138. DOI:10.1371/journal.pone.0049138 |
| [30] | Geethanjali, Dinesh Kumar V, Raghu N, Gopenath TS, Veerana Gowda S, Ong KW, Ranjith MS, Gnanasekaran A, Karthikeyan M, Roy B, Pugazhandhi B, Pradeep P, Balasubramanian S, Basalingappa KM. Quorum sensing: a molecular cell communication in bacterial cells. Journal of Biomedical Sciences, 2019, 5(2): 23-34. DOI:10.3126/jbs.v5i2.23635 |
| [31] | Oliveira GS, Lopes DRG, Andre C, Silva CC, Baglinière F, Vanetti MCD. Multi species biofilm formation by the contaminating microbiota in raw milk. Biofouling, 2019, 35(8): 819-831. DOI:10.1080/08927014.2019.1666267 |
| [32] |
Cai FY, Zhao YC, Li Y, Zhao DG. Identification and analysis of ABC transporters family from Eucommia ulmoides. Acta Agriculturae Zhejiangensis, 2021, 33(9): 1581-1591.
(in Chinese) 蔡方阳, 赵懿琛, 李义, 赵德刚. 杜仲ABC转运蛋白基因家族成员鉴定与分析. 浙江农业学报, 2021, 33(9): 1581-1591. DOI:10.3969/j.issn.1004-1524.2021.09.02 |
| [33] | Chen D, Li XY, Zhao X, Qin YS, Zhang XX, Li J, Wang JM, Wang CF. Proteomics and microstructure profiling of goat milk protein after homogenization. Journal of Dairy Science, 2019, 102(5): 3839-3850. DOI:10.3168/jds.2018-15363 |
| [34] | Capra EJ, Laub MT. Evolution of two-component signal transduction systems. Annual Review of Microbiology, 2012, 66: 325-347. DOI:10.1146/annurev-micro-092611-150039 |
| [35] | Issa IN, Philippe D, Nicholas A, Raoult D, Eric C. Metaproteomics of the human gut microbiota: challenges and contributions to other OMICS. Clinical Mass Spectrometry, 2019, 14: 18-30. DOI:10.1016/j.clinms.2019.06.001 |
| [36] | Okeke ES, Ita RE, Egong EJ, Udofia LE, Mgbechidinma CL, Akan OD. Metaproteomics insights into fermented fish and vegetable products and associated microbes. Food Chemistry: Molecular Sciences, 2021, 3: 100045. DOI:10.1016/j.fochms.2021.100045 |
| [37] | Xie MX, Wu JR, An FY, Yue XQ, Tao DB, Wu RN, Lee YK. An integrated metagenomic/metaproteomic investigation of microbiota in dajiang-meju, a traditional fermented soybean product in Northeast China. Food Research International, 2019, 115: 414-424. DOI:10.1016/j.foodres.2018.10.076 |
| [38] | Starke R, Jehmlich N, Bastida F. Using proteins to study how microbes contribute to soil ecosystem services: the current state and future perspectives of soil metaproteomics. Journal of Proteomics, 2019, 198: 50-58. DOI:10.1016/j.jprot.2018.11.011 |
| [39] | Xie ZX, He YB, Wang MH, Zhang SF, Kong LF, Lin L, Liu SQ, Wang DZ. Dissecting microbial community structure and metabolic activities at an oceanic deep chlorophyll maximum layer by size-fractionated metaproteomics. Progress in Oceanography, 2020, 188: 102439. DOI:10.1016/j.pocean.2020.102439 |
| [40] | Tang J, Fu JB, Wang YX, Li B, Li YH, Yang QX, Cui XJ, Hong JJ, Li XF, Chen YZ, Xue WW, Zhu F. ANPELA: analysis and performance assessment of the label-free quantification workflow for metaproteomic studies. Briefings in Bioinformatics, 2020, 21(2): 621-636. DOI:10.1093/bib/bby127 |
| [41] | Heyer R, Schallert K, Zoun R, Becher B, Saake G, Benndorf D. Challenges and perspectives of metaproteomic data analysis. Journal of Biotechnology, 2017, 261: 24-36. DOI:10.1016/j.jbiotec.2017.06.1201 |
 2022, Vol. 62
2022, Vol. 62





