中国科学院微生物研究所,中国微生物学会
文章信息
- 卜旭莹, 任敏, 万传星, 张利莉. 2022
- BU Xuying, REN Min, WAN Chuanxing, ZHANG Lili.
- 帕米尔高原可培养需氧冷适应细菌及古菌多样性
- Diversity of aerobic cold-adapted bacteria and archaea isolated from the Pamir Plateau
- 微生物学报, 62(7): 2568-2581
- Acta Microbiologica Sinica, 62(7): 2568-2581
-
文章历史
- 收稿日期:2021-10-19
- 修回日期:2022-01-25
- 网络出版日期:2022-03-12
冷适应微生物包括耐冷菌和嗜冷菌两种类群,其中耐冷菌能够在0–5 ℃生长,最适生长温度在15 ℃以上,嗜冷菌能够在0 ℃及0 ℃以下生长,最适生长温度在15 ℃[1]。冷适应微生物可在高山、极地、深海、洞穴以及大气上层等[2–6]永久寒冷的环境中生存。冷适应细菌可产生低温蛋白酶、脂肪酶和几丁质酶等多种胞外酶,并已广泛用于食品、洗涤剂、饲料以及环境生物治理等领域[7–13]。常见的冷适应细菌有假单胞菌属(Pseudomonas)、节杆菌属(Arthrobacter)、黄杆菌属(Flavobacterium)、冷杆菌属(Cryobacterium)等[14]。冷适应古菌也是冷适应微生物中的一个重要分支,其中嗜盐古菌作为可培养古菌,可产生多种胞外酶和胞外多糖[15–16],而且还具有产生可再生燃料及化学品的潜力[17–18]。因此,冷适应微生物为工业发展及人类生活做出了巨大贡献。
中国帕米尔高原地处新疆西南部,由天山、昆仑山、喀喇昆仑山和兴都库什山等交汇而成,是世界十大高原之一,约80%的面积分布在海拔3 300–6 000 m之间,1月份平均气温–15 ℃,7月份平均气温25 ℃,年平均气温3.2 ℃,呈独特的高寒干旱气候,分布有火山、湿地和湖泊等多种地貌[19],是天然的冷环境宝库,适合冷适应微生物的生长,且Khomutovska等[20]对帕米尔高原4 000–4 500 m岩石缝隙中的微生物多样性进行了分析,李明源等[21]对帕米尔高原慕士塔格峰洋布拉克冰川的雪冰及融水中可培养细菌多样性进行了初步研究,已发现帕米尔高原存在丰富的冷适应微生物资源[20–21],故帕米尔高原是选择性分离冷适应微生物的理想区域。
分离培养技术是获得纯培养物的关键技术,虽然宏基因组技术已然成为探究微生物物种多样性的重要手段[22],但只有分离获得纯培养物才能更好地实现微生物资源的充分开发及利用。另外,针对性地按照目标微生物的生长习性和营养需求来选择培养条件,是高效分离目标微生物的必要手段。经前人研究发现,TSA和R2A培养基是选择性分离冷适应微生物的有效培养基[23],NOM和F6培养基是选择性分离可培养嗜盐古菌的培养基[24–25]。
本研究以自帕米尔高原1 000–5 000 m采集的72份土壤样品为研究对象,采用选择性分离的培养方法,利用TSA、R2A、NOM和F6四种分离培养基,选择4 ℃和15 ℃两个培养温度。期望分离得到特殊生境下有应用潜力的冷适应微生物资源,并揭示帕米尔高原不同培养条件以及不同海拔梯度的可培养冷适应微生物多样性。
1 材料与方法 1.1 样品采集本研究的72份土壤样品于2019年9月和2020年10月采集自新疆帕米尔高原(表 1),自帕米尔高原1 760–4 539 m沿河流和植被进行采样,采集地表以下0–20 cm土壤。样品采集完成后,迅速将其运送至实验室进行实验操作,剩余样品放置于4 ℃冷库保藏,备用。
| Altitude ranges /m | Longitude ranges | Latitude ranges | Number of samples |
| 1 000–2 000 | 75°31′22″–76°12′15″ | 38°58′54″–37°40′44″ | 10 |
| 2 000–3 000 | 76°2′57″–75°24′33″ | 37°50′39″–37°52′4″ | 16 |
| 3 000–4 000 | 75°17′25″–74°56′10″ | 37°51′59″–38°17′46″ | 40 |
| 4 000–5 000 | 74°58′15″–75°25′46″ | 38°41′19″–36°51′3″ | 6 |
1.2 冷适应细菌及古菌分离培养 1.2.1 土样分类
为探究帕米尔高原以海拔1 000 m为间隔的土壤样品中冷适应细菌及古菌的多样性,将其以低海拔(1 000–2 000 m;low altitude)、中海拔(2 000–3 000 m;intermediate altitude)、中高海拔(3 000–4 000 m;middle altitude)和高海拔(4 000–5 000 m;high altitude)为分类依据合并成4份样品。
1.2.2 分离培养本研究使用TSA和R2A培养基用于冷适应细菌的分离,NOM和F6培养基用于冷适应古菌的分离。
(1) TSA培养基(g/L)[26]:TSA 25.0,NaCl 20.0,0.1 mol/L蔗糖,琼脂15.0,蒸馏水1 L,pH 7.0。
(2) R2A培养基(g/L)[27]:胰蛋白胨0.25,蛋白胨0.25,酸水解酪蛋白0.5,酵母浸粉0.5,葡萄糖0.5,可溶性淀粉0.5,丙酮酸钠0.3,MgSO4·7H2O 0.1,K2HPO4 0.3,琼脂15.0,蒸馏水1 L,pH 7.0。
(3) NOM培养基(g/L)[24]:酵母提取物0.05,鱼蛋白胨0.25,丙酮酸钠1.0,KCl 5.4,K2HPO4 0.36,CaCl2 0.25,NH4Cl 0.25,MgSO4·7H2O 0.25,MgCl2·6H2O 23.0,NaCl 184.0,琼脂20.0,pH 7.0。
(4) 壳聚糖-天冬酰胺培养基(F6)(g/L)[25]:酪素水解蛋白2.0,天冬酰胺1.0,壳聚糖4.0,MgSO4 6.0,K2HPO4 0.36,NaCl 150.0,琼脂16.0,1 mL制霉菌素(50 mg/mL),1 mL四环素(50 mg/mL),1 mL维生素溶液,pH 7.0。
维生素溶液(mg/L):烟酸10.0,生物素4.0,泛酸10.0,硫辛酸10.0,叶酸4.0,对氨基苯甲酸10.0,维生素B1 10.0,维生素B2 10.0,维生素B6 10.0,维生素B12 10.0,蒸馏水1 L (0.45 μm微孔滤膜过滤除菌)。
以上培养基均以100倍稀释的土壤浸提液代替蒸馏水配制。
采用稀释涂布法进行冷适应古菌及细菌的分离培养。首先取不同海拔梯度的2 g土样加入装有18 mL无菌生理盐水的锥形瓶中,放置于4 ℃摇床振荡180 r/min悬浮30 min,制备成土壤悬液,倍性稀释至10–4,最后将10–3和10–4的土壤稀释液分别涂布于培养基平板上,采用连续划线法对形态不同的菌落进行分离纯化。鉴于古菌的生长周期较长,冷适应微生物的适宜生长温度在15 ℃左右,因此将NOM及F6平板分别放置于4 ℃和15 ℃培养,TSA及R2A平板放置于4 ℃培养,所得菌株分离纯化后置于15 ℃培养。同时对15 ℃获得的古菌菌株于4 ℃培养,进行生长温度验证,判断其是否为冷适应古菌。所有纯化的菌株均用40% (V/V)甘油于–80 ℃冻存管保藏。
1.3 菌株16S rRNA基因鉴定使用天根生化科技有限公司细菌全基因组DNA提取试剂盒进行菌株DNA的提取,细菌16S rRNA基因扩增引物为27F (5ʹ-AGAGTTTG ATCCTGGCTCAG-3ʹ)和1492R (5ʹ-GGTTACCT T-GTTACGACTT-3ʹ),古菌16S rRNA基因扩增引物为0018F (5ʹ-ATTCCGGTTGATCCTGCC-3ʹ)和1518R (5ʹ-AGGAGG- TGATCCAGCCGC-3ʹ)。PCR反应条件为:94 ℃ 5 min;94 ℃ 30 s,54 ℃ 30 s,72 ℃ 90 s,30个循环;72 ℃ 10 min,4 ℃保存。采用1%琼脂糖凝胶检测扩增产物,将出现目的条带的PCR扩增产物送至上海生物工程有限公司进行双向测序。
1.4 菌株系统发育树构建采用Seqman软件对测序所得序列双向末端的杂峰进行适当裁剪和序列拼接,将处理好的16S rRNA基因序列上传至EzBioCloud (https://www.ezbiocloud.net/identify)[28]进行序列比对,确认分类地位,当与最相似菌株的序列相似度<98.65%时判断为潜在新物种。使用MEGA 7[29]软件以邻接法构建菌株系统发育树,重复计算次数设置为1 000次,并用iTOL (https://itol.embl.de/)[30]网站进一步完善美化进化树。将无重复序列菌株的16S rRNA基因序列上传至GenBank数据库,登录号为OK298973–OK299070。
2 结果与分析 2.1 不同海拔可培养需氧冷适应细菌及古菌多样性本研究通过选择培养温度4 ℃对帕米尔高原冷适应细菌,以及于4 ℃和15 ℃对帕米尔高原冷适应古菌进行分离培养。结果显示,4 ℃时未分离得到冷适应古菌,15 ℃时分离得到古菌25株,但在4 ℃生长温度验证时并未生长,表明非冷适应古菌。
另外,经4 ℃分离培养获得了自帕米尔高原低海拔(25株,3种)的古菌以及低海拔(81株,40种)、中海拔(60株,28种)、中高海拔(206株,72种)和高海拔(47株,21种)的冷适应细菌类群(图 1A)。冷适应细菌广泛分布于所有海拔中,在中高海拔土壤样品中,冷适应细菌的种类最为丰富,高海拔土壤样品中的物种群落组成略低于低海拔区域。帕米尔高原冷适应细菌整体海拔分布结果显示,随海拔升高,其数量呈现高-低-高-低的变化趋势。而古菌域的钠线菌属和盐陆生菌属仅在低海拔被分离得到。

|
| 图 1 不同海拔可培养需氧冷适应细菌多样性 Figure 1 Diversity of aerobic cold-adapted bacteria isolated from the samples of different altitudes. A: occurrence at different taxonomic levels; B: occurrence at the phylum level; C: occurrence at the class level; D: relative abundance at the genus level (top 20); E: Venn diagram of the genus level abundance. Low: 1 000–2 000 m; intermediate: 2 000–3 000 m; middle: 3 000–4 000 m; high: 4 000–5 000 m. |
基于门和纲水平(图 1B和1C),低、中和中高海拔冷适应细菌的优势菌群均为变形菌门的γ-变形菌纲,生长范围广泛,中高海拔分布最多。随海拔升高,放线菌门的放线菌纲物种数量逐渐增加,且为高海拔地区的主导菌群,表明该物种更适应高海拔环境。厚壁菌门杆菌纲(Bacilli)的物种主要分布在低海拔梯度,表明高海拔环境不适宜其生长繁殖。此外,不同海拔梯度同时存在特有菌群,鞘脂杆菌纲(Sphingobacteriia)为中高海拔的特有纲,拟杆菌门的黄杆菌纲(Flavobacteriia)存在于低海拔至中高海拔之间。
基于属水平(图 1D),假单胞菌属为4个海拔的优势属,该属在帕米尔高原分布最广泛,尤其在中高海拔区域最为显著,而动性球菌属(Planococcus)在低海拔具有显著地位,黄杆菌属为中海拔区域第二优势菌群,节杆菌属为中高海拔区域优势菌群,链霉菌属(Streptomyces)为高海拔的优势菌群。动性球菌属、节杆菌属、假单胞菌属、假节杆菌属(Pseudarthrobacter)、细杆菌属(Microbacterium)、棒形杆菌属(Clavibacter)和类节杆菌属(Paenarthrobacter)属7个属为4个海拔的共有属(图 1E)。另外,部分属随海拔梯度变化分布随之改变:黄杆菌属、红球菌属(Rhodococcus)和冷杆菌属为1 000– 4 000 m特有属,沙雷氏菌属(Serratia)为2 000– 4 000 m特有属,Mycetocola属为2 000–5 000 m特有属,Peribacillus属和叶杆菌属(Phyllobacterium)为3 000–5 000 m特有属。此外,每个海拔梯度还拥有各自的特有属(表 2)。
| Altitude/m | Genus |
| 1 000–2 000 | Acidovorax, Dietzia, Haloterrigena, Hydrogenophaga, Natrinema, Paracoccus, Psychrobacter, Saccharibacillus |
| 2 000–3 000 | Nesterenkonia, Rhizobium, Stenotrophomonas |
| 3 000–4 000 | Aeromicrobium, Aeromonas, Agrococcus, Brevibacterium, Brevundimonas, Caulobacter, Exiguobacterium, Knoellia, Neorhizobium, Nocardioides, Paenibacillus, Pseudoxanthomonas, Rahnella, Sphingobacterium, Sphingobium, Zhihengliuella |
| 4 000–5 000 | Conyzicola, Paeniglutamicibacter, Promicromonospora |
分离培养结果表明,低海拔地区适合冷适应细菌中动性球菌属以及古菌域钠线菌属和盐陆生菌属的生存,中海拔及中高海拔地区适合冷适应黄杆菌属及节杆菌属的生存,链霉菌属是高海拔菌属。帕米尔高原不同海拔均存在大量可培养冷适应微生物资源,且不同海拔间物种有明显差异,说明海拔变化会导致微生物群落组成的变化。
2.2 不同培养基需氧冷适应细菌及古菌多样性本研究采用TSA和R2A以及NOM和F6四种培养基,分别对帕米尔高原不同海拔梯度土壤样品中的冷适应细菌及古菌进行分离培养,TSA和R2A培养基分别获得275株86个种和119株47个种的冷适应细菌,NOM和F6培养基分别获得13株2个种和12株3个种的古菌,结果表明TSA培养基对冷适应细菌的分离效果最好,而NOM培养基对古菌的分离效果较好。
培养结果显示,基于门和纲水平(图 2A和2B),变形菌门的γ-变形菌纲在TSA和R2A培养基所分离的冷适应细菌菌株中占优势地位,但在TSA培养基(132株)中的菌株数量高于R2A培养基(60株)。TSA培养基对放线菌门的放线菌纲选择性分离效果最好。NOM和F6培养基分离的古菌菌株均隶属于广古菌门的盐杆菌纲。
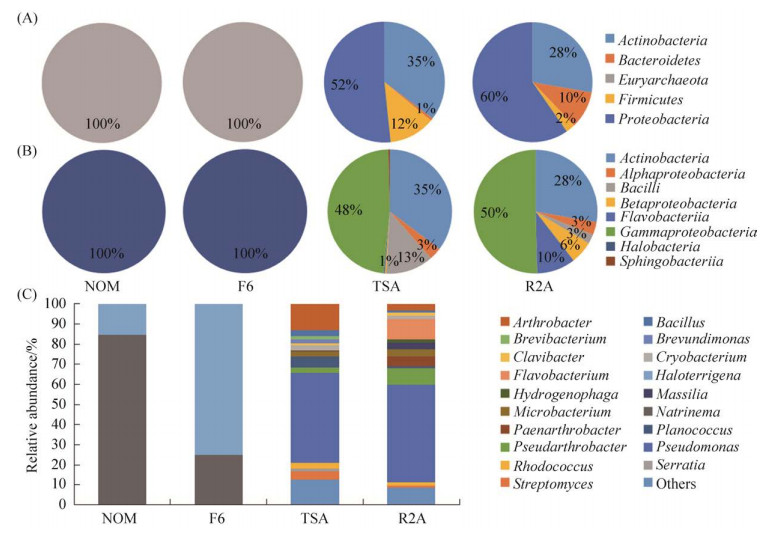
|
| 图 2 不同培养基需氧冷适应细菌及古菌组成 Figure 2 Composition of aerobic cold-adapted bacteria and archaea isolated with four different culture media. A: sorted at the phylum level. B: sorted at the class level. The percentage in pie charts represents the number of isolates. C: relative abundance at the genus level. |
基于属水平(图 2C),TSA培养基(37属)分离获得属的多样性最丰富,该培养基对假单胞菌属(123株)的选择性分离效果最为显著,R2A培养基(23属)中假单胞菌属(58株)同样占优势地位,表明两种培养基对假单胞菌属都具有选择性分离效果,但两种培养基中第二优势属存在差异,分别为节杆菌属和黄杆菌属。NOM培养基(2属)和F6培养基(2属)均仅获得钠线菌属和盐陆生菌属的古菌。
以上结果表明,节杆菌属、芽孢杆菌属(Bacillus)、棒形杆菌属、冷杆菌属、黄杆菌属、马赛菌属(Massilia)、细杆菌属、叶杆菌属、动性球菌属、假节杆菌属、假单胞菌属、红球菌属和链霉菌属13个属为2种培养基的共有属,故两种培养基均适用于以上冷适应细菌属的分离。但相较R2A培养基,TSA培养基中节杆菌属、假单胞菌属、棒形杆菌、冷杆菌属、细杆菌属、动性球菌属、假单胞菌属、红球菌属及链霉菌属数量较多,故TSA培养基可选择性分离以上属的物种,而R2A培养基较适用于分离黄杆菌属、马赛菌属及假节杆菌属菌株。此外,由于培养基组分存在差异,TSA培养基及R2A培养基还拥有各自的特有属(表 3)。NOM培养基中钠线菌属的数量占优势,而F6培养基中盐陆生菌属占比较大,说明鱼蛋白胨是钠线菌属适宜生长的碳氮源,而当培养基中添加壳聚糖、酸水解酪蛋白和天冬酰胺时,则可用于选择性分离盐陆生菌属的嗜盐古菌。
| Culture media | Genus |
| TSA | Aeromicrobium, Aeromonas, Agrococcus, Brevibacterium, Brevundimonas, Caulobacter, Dietzia, Exiguobacterium, Knoellia, Mycetocola, Neorhizobium, Nesterenkonia, Nocardioides, Paeniglutamicibacter, Paenisporosarcina, Pantoea, Peribacillus, Psychrobacter, Rahnella, Saccharibacillus, Serratia, Sphingobacterium, Sphingobium, Zhihengliuella |
| R2A | Acidovorax, Conyzicola, Hydrogenophaga, Paenarthrobacter, Paenibacillus, Paracoccus, Promicromonospora, Pseudoxanthomonas, Rhizobium, Stenotrophomonas |
2.3 帕米尔高原可培养需氧冷适应细菌及古菌多样性
本研究自帕米尔高原采集的土壤样品中共分离得到419株需氧原核微生物,包括394株冷适应细菌,25株古菌(图 3A)。经16S rRNA基因测序,结果表明以上菌株隶属于2个域、5个门、8个纲、18个目、28个科、49个属及118个种。
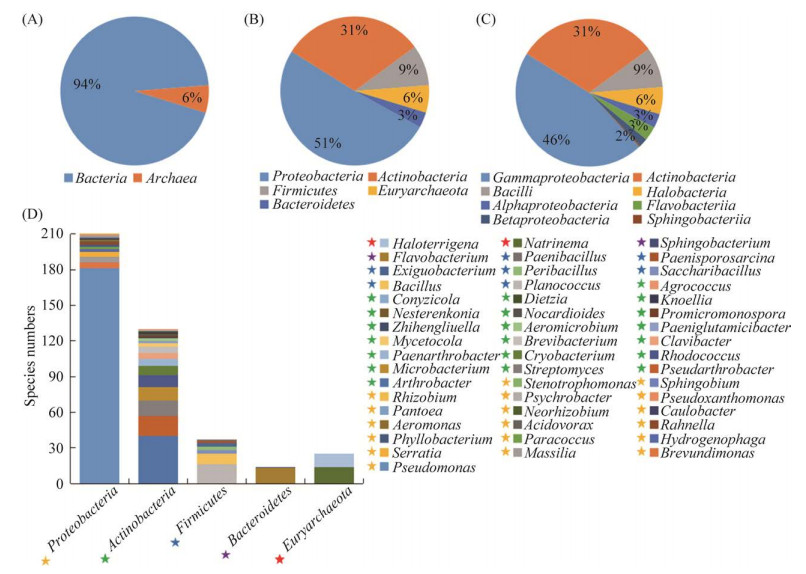
|
| 图 3 帕米尔高原可培养需氧冷适应细菌及古菌群落组成 Figure 3 Composition of aerobic cold-adapted bacteria and archaea in the Pamir Plateau revealed by a cultivation-dependent approach. A: at the domain level; B: at the phylum level; C: at the class level; D: at the genus level. |
基于门水平(图 3B),冷适应细菌中变形菌门(18属213株)为第一主导门,放线菌门(20属130株)为第二优势门,此外依次为厚壁菌门(7属37株)和拟杆菌门(2属14株),古菌域仅含广古菌门(2属25株)一个门。
基于纲水平(图 3C),冷适应细菌中γ-变形菌纲(8属18种192株)分离菌株数量最多,放线菌纲(20属52种130株)排名第二,其余为杆菌纲(7属21种37株)、α-变形杆菌纲(Alphaproteobacteria,7属13种13株)、黄杆菌纲(1属7种13株)、β-变形菌纲(Betaproteobacteria,3属6种8株)和鞘脂杆菌纲(1属1种1株),古菌均隶属于盐杆菌纲(2属3种25株)。
基于属水平(图 3D),冷适应细菌变形菌门中假单胞菌属(10种181株)菌株数量占绝对优势,占总分离菌株数量的45.9%,放线菌门的节杆菌属(14种40株)分离菌株数量较多,占总分离菌株数量的10.2%。另外假节杆菌属(4种17株)、动性球菌属(8种16株)、链霉菌属(4种13株)、黄杆菌属(7种13株)、细杆菌属(4种11株)、红球菌属(5种10株)、芽孢杆菌属(5种9株)、冷杆菌属(4种8株)和类节杆菌属(2种6株)占比较少。其他属还有马赛菌属及短波单胞菌属(Brevundimonas)等4个属均5株,沙雷氏菌属4株,微小杆菌属(Exiguobacterium) 及Peribacillus属等4个属均3株,气微菌属(Aeromicrobium)及嗜氢菌属(Hydrogenophaga)等7个属均2株,嗜酸菌属(Acidovorax)及气单胞菌属(Aeromonas)等20个属均仅1株。此外,古菌域广古菌门中仅含钠线菌属(1种14株)及盐陆生菌属(2种11株)两个属,且钠线菌属数量较多。
以上分离培养结果表明,帕米尔高原物种多样性较为丰富,存在可培养冷适应细菌及古菌资源。
2.4 帕米尔高原可培养微生物潜在新物种本研究通过将分离获得菌株的16S rRNA基因测序结果上传至Ezbiocloud进行比对,发现相似度小于98.65%的潜在新物种共23株(图 4),包括冷适应细菌21株,古菌2株,占总菌株数的5.49%。其中,相似度介于96.00%−97.00%间的潜在物种3株(TRM85882、TRM86049、TRM85033),介于97.00%−98.00%间的潜在新物种8株(TRM86076、TRM86002、TRM86096、TRM86207、TRM86202、TRM86038、TRM86107、TRM85034),介于98.00%−98.65%间潜在新物种12株(TRM86091、TRM86001、TRM86048、TRM86005、TRM86077、TRM86180、TRM86094、TRM86156、TRM85870、TRM86067、TRM85914、TRM85147),无潜在新属。潜在新物种中2株隶属于放线菌门,8株隶属于变形菌门,9株隶属于拟杆菌门,2株隶属于厚壁菌门,2株隶属于广古菌门。1 000−5 000 m四个海拔梯度由低至高分别分离得到7株、9株、4株和3株潜在新物种。在培养温度为4 ℃,分离培养基为R2A培养基时,中海拔梯度的新物种数量最多,共分离得到9株疑似新种。此外,分别在TSA、R2A和F6培养基中分别分离得到5株、16株和2株潜在新物种,未在NOM培养基中分离得到潜在新物种。另外,黄杆菌属疑似新种共8株,占比最大。上述研究结果表明,帕米尔高原存在丰富的微生物潜在新物种资源,有待挖掘、开发及利用。

|
| 图 4 帕米尔高原可培养潜在新物种分布 Figure 4 Overview of the predicted novel taxa of cold-adapted bacteria and archaea isolated from the Pamir Plateau. |
3 讨论
土壤微生物群落丰富多样,在自然界生物地球化学转化中发挥着重要作用,对生态系统有着不可忽视的影响,是研究环境生态学的理想材料[31]。组学技术已成为研究微生物多样性及其生态适应性的有力工具[22],然而剧增的基因组数据除了使我们认识到微生物的多样性及在地球元素循环的功能之外,也与我们不能获得其中许多新类群形成鲜明对比。但冷适应细菌、嗜盐古菌本身具备多种生物学功能,已广泛应用到多种工农业生产中[32–35],为人类生产、生活做出了一定贡献,因而获得可培养微生物资源对于人类发展至关重要。
本研究为探究帕米尔高原需氧冷适应细菌及古菌群落的多样性,采用低温分离培养的方法,对帕米尔高原海拔1 000–5 000 m四个海拔梯度土壤样品中的需氧冷适应细菌及古菌进行研究,使用4种培养基及2种培养温度,共分离得到394株冷适应细菌和25株古菌,隶属于49个属118个种,结果表明帕米尔高原存在丰富多样的冷适应细菌及古菌资源。本研究获得了变形菌门、放线菌门、厚壁菌门和拟杆菌门4个门的冷适应细菌以及广古菌门的古菌,其中冷适应细菌分离结果与前人研究一致[21]。冷适应细菌中变形菌门的γ-变形菌纲为帕米尔高原的主导类群,放线菌门的放线菌纲和厚壁菌门的杆菌纲同样占有一定比例,拟杆菌门整体占比较小。假单胞菌属被报道为较高海拔环境冷适应微生物中的优势属,在帕米尔高原同样印证了这一事实[36–37]。古菌类群为广古菌门,这与青藏高原[24]、南极[38]、北极[4]及阿尔卑斯山[5]土壤菌群组成类似。本研究自4 ℃共分离得到冷适应细菌394株,可见帕米尔高原是冷适应细菌良好的生存场所。
海拔和土壤品质及环境温度息息相关[39],而这些要素与微生物的生长之间同样存在密切联系。本研究结果表明,在4 000–5 000 m高海拔地区随温度的降低,变形菌门数量明显减少,主导菌群变为放线菌门,其中链霉菌为主导属,与青藏高原[27]、西藏高原[38]和南极[4]高海拔冻土区域群落组成相同。阿尔卑斯山和南极冻土中拟杆菌门在冷适应环境中数量增加[6],但本研究并未发现此规律,可能与采样地点和培养基不适合于拟杆菌门的生长相关。因帕米尔高原不同海拔间的物种存在差异,且假单胞菌属、黄杆菌属和马赛菌属等微生物与元素循环之间存在密切联系[40–41],故本研究揭示帕米尔高原不同海拔间物种差异的同时也为生态学研究提供了理论支持。此外,因帕米尔高原中高海拔地区地势地貌最为多样,为展现该海拔区间帕米尔高原更为全面的不同生境的物种多样性,在此区域内采集样品数量最多,因此各海拔区间的特有属可能存在局限性。
多种微生物的培养严格依赖于环境条件和不同的生长因子(比如维生素、氨基酸、核苷酸以及腐殖酸或其他外部的电子穿梭载体),因此在实验室条件下模拟原位环境进行培养也是一种挑战[42–43]。本研究配制培养基时选用帕米尔高原不同海拔土壤浸提液替代蒸馏水,获得118种冷适应细菌和古菌,物种种类较丰富,可能与模拟原位环境的培养方法相关。采用TSA和R2A两种适合需氧冷适应细菌生长的培养基,NOM和F6两种适合需氧古菌生长的培养基,对帕米尔高原冷适应细菌及古菌进行分离培养。结果表明,假单胞菌属和黄杆菌属分别为TSA和R2A两种培养基中优势属,钠线菌属和盐陆生菌属分别为NOM和F6培养基中优势属,与先前研究相似[44–46]。本研究采用TSA培养基得到了大量冷适应细菌,且物种丰富,则相较R2A培养基,该培养基更适用于冷适应细菌的选择性分离。R2A培养基分离冷适应细菌菌株总数不高,但菌株种类丰富,可能与其寡营养的特性相关,故该培养基可适用于黄杆菌属及假节杆菌属等多种冷适应细菌的培养。此外,本研究所使用的壳聚糖、酸水解酪蛋白、天冬酰胺和鱼蛋白胨等营养物质有利于嗜盐古菌的生长,培养时间达半年以上,但仅在15 ℃的培养条件下,得到钠线菌属和盐陆生菌属的古菌,且4 ℃没有得到古菌。多数古菌是不可培养的,Imachi团队通过共培养的方式获得阿斯加德古菌门(Asgard),为真核生物的起源提供了非常重要的证据,但富集分离培养时间长达10年之久[47],也许我们在冷适应古菌的分离过程中受到了时间的限制。嗜盐古菌是高盐环境的“定居者”[46],且存在冷适应嗜盐古菌类群[48],本研究自帕米尔高原土壤样品中获得嗜盐古菌种类较少,且未得到冷适应嗜盐古菌,猜测原因为培养基的选择对帕米尔高原土壤中古菌种类针对性存在偏差导致。
本研究为帕米尔高原的冷适应微生物群落分布及多样性提供了新的见解,获得了宝贵的冷适应微生物资源,且可能有助于预测在气候变暖背景下帕米尔高原的微生物群落和生物地球化学循环的未来。
| [1] | Morita RY. Psychrophilic bacteria. Bacteriological Reviews, 1975, 39(2): 144-167. DOI:10.1128/br.39.2.144-167.1975 |
| [2] | Liu KS, Yao TD, Pearce DA, Jiao NZ, Zeng YH, Guo BX, Liu YQ. Bacteria in the lakes of the Tibetan Plateau and polar regions. Science of the Total Environment, 2021, 754: 142248. DOI:10.1016/j.scitotenv.2020.142248 |
| [3] |
Wang YF, Wei SP, Cui HP, Su X, Zhu YH, Lu ZQ, Hu F, Li LP, Zhang S, Liu H. Distribution and diversity of microbial community along a vertical permafrost profile, Qinghai-Tibetan Plateau. Microbiology China, 2016, 43(9): 1902-1917.
(in Chinese) 王艳发, 魏士平, 崔鸿鹏, 苏新, 祝有海, 卢振权, 胡非, 李来鹏, 张帅, 刘晖. 青藏高原冻土区土壤垂直剖面中微生物的分布与多样性. 微生物学通报, 2016, 43(9): 1902-1917. |
| [4] | Signori CN, Thomas F, Enrich-Prast A, Pollery RCG, Sievert SM. Microbial diversity and community structure across environmental gradients in Bransfield Strait, Western Antarctic Peninsula. Frontiers in Microbiology, 2014, 5: 647. |
| [5] | Nemergut DR, Costello EK, Meyer AF, Pescador MY, Weintraub MN, Schmidt SK. Structure and function of alpine and Arctic soil microbial communities. Research in Microbiology, 2005, 156(7): 775-784. DOI:10.1016/j.resmic.2005.03.004 |
| [6] | Wunderlin T, Ferrari B, Power M. Global and local-scale variation in bacterial community structure of snow from the Swiss and Australian Alps. FEMS Microbiology Ecology, 2016, 92(9): fiw132. DOI:10.1093/femsec/fiw132 |
| [7] | Óskarsson KR, Kristjánsson MM. Improved expression, purification and characterization of VPR, a cold active subtilisin-like serine proteinase and the effects of calcium on expression and stability. Biochimica et Biophysica Acta: BBA - Proteins and Proteomics, 2019, 1867(2): 152-162. DOI:10.1016/j.bbapap.2018.11.010 |
| [8] | Perfumo A, Von Sass GJF, Nordmann EL, Budisa N, Wagner D. Discovery and characterization of a new cold-active protease from an extremophilic bacterium via comparative genome analysis and in vitro expression. Frontiers in Microbiology, 2020, 11: 881. DOI:10.3389/fmicb.2020.00881 |
| [9] | Maharana AK, Singh SM. A cold and organic solvent tolerant lipase produced by Antarctic strain Rhodotorula sp. Y-23. Journal of Basic Microbiology, 2018, 58(4): 331-342. DOI:10.1002/jobm.201700638 |
| [10] | Guo CC, Zheng RK, Cai RN, Sun CM, Wu SM. Characterization of two unique cold-active lipases derived from a novel deep-sea cold seep bacterium. Microorganisms, 2021, 9(4): 802. DOI:10.3390/microorganisms9040802 |
| [11] | Santa-Cruz Vasquez YM, Gomes MB, E Silva TR, Duarte AWF, Rosa LH, De Oliveira VM. Cold-adapted chitinases from Antarctic bacteria: taxonomic assessment and enzyme production optimization. Biocatalysis and Agricultural Biotechnology, 2021, 34: 102029. DOI:10.1016/j.bcab.2021.102029 |
| [12] | Han ZG, Shang-Guan F, Yang JK. Characterization of a novel cold-active xylanase from Luteimonas species. World Journal of Microbiology & Biotechnology, 2018, 34(8): 123. |
| [13] | Al-Ghanayem AA, Joseph B. Current prospective in using cold-active enzymes as eco-friendly detergent additive. Applied Microbiology and Biotechnology, 2020, 104(7): 2871-2882. DOI:10.1007/s00253-020-10429-x |
| [14] |
Liu Q, Yang LL, Zhou YG, Xin YH, Dong XZ. Research progress on the diversity of glacial bacteria Cryobacterium. Acta Microbiologica Sinica, 2021, 61(4): 807-815.
(in Chinese) 刘庆, 杨蕾蕾, 周宇光, 辛玉华, 东秀珠. 冰川细菌冷杆菌属的多样性研究进展. 微生物学报, 2021, 61(4): 807-815. |
| [15] | Menasria T, Aguilera M, Hocine H, Benammar L, Ayachi A, Si Bachir A, Dekak A, Monteoliva-Sánchez M. Diversity and bioprospecting of extremely halophilic archaea isolated from Algerian arid and semi-arid wetland ecosystems for halophilic-active hydrolytic enzymes. Microbiological Research, 2018, 207: 289-298. DOI:10.1016/j.micres.2017.12.011 |
| [16] | Biswas J, Saha P, Ganguly J, Paul AK. Production and characterization of a bioactive extracellular homopolysaccharide produced by Haloferax sp. BKW301. Journal of Basic Microbiology, 2020, 60(11/12): 938-949. |
| [17] | Kasirajan L, Maupin-Furlow JA. Halophilic archaea and their potential to generate renewable fuels and chemicals. Biotechnology and Bioengineering, 2021, 118(3): 1066-1090. DOI:10.1002/bit.27639 |
| [18] | Dayakar B, Xavier KAM, Das O, Porayil L, Balange AK, Nayak BB. Application of extreme halophilic archaea as biocatalyst for chitin isolation from shrimp shell waste. Carbohydrate Polymer Technologies and Applications, 2021, 2: 100093. DOI:10.1016/j.carpta.2021.100093 |
| [19] |
Yang SP, Jiang J, Yan P. Eco-geographic distribution of rare and endangered plants and endemism in the Pamirs Region of China. Journal of Arid Land Resources and Environment, 2018, 32(1): 115-120.
(in Chinese) 杨淑萍, 姜洁, 阎平. 中国帕米尔高原珍稀濒危植物及特有种的生态地理分布. 干旱区资源与环境, 2018, 32(1): 115-120. |
| [20] | Khomutovska N, Jerzak M, Kostrzewska-Szlakowska I, Kwiatowski J, Suska-Malawska M, Syczewski M, Jasser I. Life in extreme habitats: diversity of endolithic microorganisms from cold desert ecosystems of eastern Pamir. Polish Journal of Ecology, 2017, 65(4): 303-319. DOI:10.3161/15052249PJE2017.65.4.001 |
| [21] |
Li MY, Wang JL, Sawuti G. Culturable bacterial diversity in snow, ice and meltwater of the Yangbark Glacier, Muztag Ata. Journal of Glaciology and Geocryology, 2015, 37(6): 1634-1641.
(in Chinese) 李明源, 王继莲, 古丽巴哈尔·萨吾提. 新疆东帕米尔高原慕士塔格峰洋布拉克冰川雪冰及融水中可培养细菌多样性分析. 冰川冻土, 2015, 37(6): 1634-1641. |
| [22] | Valentine G, Prince A, Aagaard KM. The neonatal microbiome and metagenomics: what do we know and what is the future?. NeoReviews, 2019, 20(5): e258-e271. DOI:10.1542/neo.20-5-e258 |
| [23] |
Wu FX, Zhang SB, Ying MZ, Liu JB. Effects of nutrient medium concentrations on isolation of cold adapted microorganisms. Journal of Shandong Agricultural University: Natural Science Edition, 2019, 50(2): 315-318.
(in Chinese) 武凤霞, 张淑彬, 应梦真, 刘建斌. 不同培养基浓度对土壤耐低温微生物分离效果的影响. 山东农业大学学报: 自然科学版, 2019, 50(2): 315-318. DOI:10.3969/j.issn.1000-2324.2019.02.030 |
| [24] | Cui HL, Gao X, Yang X, Xu XW. Halorussus rarus gen. nov., sp. nov., a new member of the family Halobacteriaceae isolated from a marine solar saltern. Extremophiles: Life Under Extreme Conditions, 2010, 14(6): 493-499. DOI:10.1007/s00792-010-0329-0 |
| [25] | 刘琴. 罗布泊地区嗜盐古菌多样性分析及其多相分类. 塔里木大学硕士学位论文, 2014. |
| [26] | Vishnivetskaya T, Kathariou S, McGrath J, Gilichinsky D, Tiedje JM. Low-temperature recovery strategies for the isolation of bacteria from ancient permafrost sediments. Extremophiles: Life Under Extreme Conditions, 2000, 4(3): 165-173. DOI:10.1007/s007920070031 |
| [27] |
Zhang BG, Liu XJ, Liu M, Zhang W, Zhang GS, Wu XK, Chen T, Liu GX. Characteristics of culturable bacteria diversity among different permafrost types in the upstream regions of the Shule River Basin, Qinghai-Tibet Plateau. Journal of Glaciology and Geocryology, 2018, 40(1): 156-165.
(in Chinese) 张宝贵, 刘晓娇, 刘敏, 张威, 章高森, 伍修琨, 陈拓, 刘光琇. 青藏高原疏勒河上游不同类型冻土可培养细菌多样性特征研究. 冰川冻土, 2018, 40(1): 156-165. |
| [28] | Yoon SH, Ha SM, Kwon S, Lim J, Kim Y, Seo H, Chun J. Introducing EzBioCloud: a taxonomically united database of 16S rRNA gene sequences and whole-genome assemblies. International Journal of Systematic and Evolutionary Microbiology, 2017, 67(5): 1613-1617. DOI:10.1099/ijsem.0.001755 |
| [29] | Kumar S, Stecher G, Tamura K. MEGA 7: molecular evolutionary genetics analysis version 7.0 for bigger datasets. Molecular Biology and Evolution, 2016, 33(7): 1870-1874. DOI:10.1093/molbev/msw054 |
| [30] | Letunic I, Bork P. Interactive tree of life (iTOL) v5: an online tool for phylogenetic tree display and annotation. Nucleic Acids Research, 2021, 49(W1): W293-W296. DOI:10.1093/nar/gkab301 |
| [31] | Traulsen A, Sieber M. Evolutionary ecology theory—microbial population structure. Current Opinion in Microbiology, 2021, 63: 216-220. DOI:10.1016/j.mib.2021.08.001 |
| [32] | Lou HH, Hu LF, Lu HY, Wei TY, Chen QH. Metabolic engineering of microbial cell factories for biosynthesis of flavonoids: a review. Molecules: Basel, Switzerland, 2021, 26(15): 4522. DOI:10.3390/molecules26154522 |
| [33] | Zhao XH, Niu YN, Mi CH, Gong HL, Yang XY, Cheng JSY, Zhou ZQ, Liu JX, Peng XL, Wei DX. Electrospinning nanofibers of microbial polyhydroxyalkanoates for applications in medical tissue engineering. Journal of Polymer Science, 2021, 59(18): 1994-2013. DOI:10.1002/pol.20210418 |
| [34] | Ranjbaran M, Carciofi BAM, Datta AK. Engineering modeling frameworks for microbial food safety at various scales. Comprehensive Reviews in Food Science and Food Safety, 2021, 20(5): 4213-4249. DOI:10.1111/1541-4337.12818 |
| [35] | Yaguchi AL, Lee SJ, Blenner MA. Synthetic biology towards engineering microbial lignin biotransformation. Trends in Biotechnology, 2021, 39(10): 1037-1064. DOI:10.1016/j.tibtech.2021.02.003 |
| [36] | 章高森. 青藏高原多年冻土区微生物多样性及其潜在应用的研究. 兰州大学博士学位论文, 2007. |
| [37] |
Peng YL, Cai XB, Yu BZ. Distribution and change of soil microorganism in alpine meadow under different states. Southwest China Journal of Agricultural Sciences, 2018, 31(6): 1241-1245.
(in Chinese) 彭岳林, 蔡晓布, 于宝政. 不同状态高寒草甸土壤微生物分布及其变化. 西南农业学报, 2018, 31(6): 1241-1245. |
| [38] |
Li GX, Song YY, Zhao X, Wang GL. High-throughput sequencing analysis of soil microbial diversity from Antarctic Fildes Peninsula. Journal of Fisheries Research, 2020, 42(3): 195-204.
(in Chinese) 李桂秀, 宋易洋, 赵芯, 王国良. 基于高通量测序对南极菲尔德斯半岛土壤微生物多样性的初步研究. 渔业研究, 2020, 42(3): 195-204. |
| [39] | Hoffmann T, Bremer E. Protection of Bacillus subtilis against cold stress via compatible-solute acquisition. Journal of Bacteriology, 2011, 193(7): 1552-1562. DOI:10.1128/JB.01319-10 |
| [40] | Guo HL, Chen C, Lee DJ. Nitrogen and sulfur metabolisms of Pseudomonas sp. C27 under mixotrophic growth condition. Bioresource Technology, 2019, 293: 122169. DOI:10.1016/j.biortech.2019.122169 |
| [41] | Li P, Jiang DW, Li B, Dai XY, Wang YH, Jiang Z, Wang YX. Comparative survey of bacterial and archaeal communities in high arsenic shallow aquifers using 454 pyrosequencing and traditional methods. Ecotoxicology: London, England, 2014, 23(10): 1878-1889. DOI:10.1007/s10646-014-1316-5 |
| [42] | Lewis WH, Tahon G, Geesink P, Sousa DZ, Ettema TJG. Innovations to culturing the uncultured microbial majority. Nature Reviews Microbiology, 2021, 19(4): 225-240. DOI:10.1038/s41579-020-00458-8 |
| [43] | Sun YH, Liu Y, Pan J, Wang FP, Li M. Perspectives on cultivation strategies of archaea. Microbial Ecology, 2020, 79(3): 770-784. DOI:10.1007/s00248-019-01422-7 |
| [44] | Klarenberg IJ, Keuschnig C, Warshan D, Jónsdóttir IS, Vilhelmsson O. The total and active bacterial community of the chlorolichen Cetraria islandica and its response to long-term warming in sub-Arctic tundra. Frontiers in Microbiology, 2020, 11: 540404. DOI:10.3389/fmicb.2020.540404 |
| [45] | Choe YH, Kim M, Lee YK. Distinct microbial communities in adjacent rock and soil substrates on a high Arctic polar desert. Frontiers in Microbiology, 2021, 11: 607396. DOI:10.3389/fmicb.2020.607396 |
| [46] | Cui HL, Dyall-Smith ML. Cultivation of halophilic archaea (class Halobacteria) from thalassohaline and athalassohaline environments. Marine Life Science & Technology, 2021, 3(2): 243-251. |
| [47] | Imachi H, Nobu MK, Nakahara N, Morono Y, Ogawara M, Takaki Y, Takano Y, Uematsu K, Ikuta T, Ito M, Matsui Y, Miyazaki M, Murata K, Saito Y, Sakai S, Song C, Tasumi E, Yamanaka Y, Yamaguchi T, Kamagata Y, Tamaki H, Takai K. Isolation of an archaeon at the prokaryote–eukaryote interface. Nature, 2020, 577(7791): 519-525. DOI:10.1038/s41586-019-1916-6 |
| [48] | Williams TJ, Liao Y, Ye J, Kuchel RP, Poljak A, Raftery MJ, Cavicchioli R. Cold adaptation of the Antarctic haloarchaea Halohasta litchfieldiae and Halorubrum lacusprofundi. Environmental Microbiology, 2017, 19(6): 2210-2227. DOI:10.1111/1462-2920.13705 |
 2022, Vol. 62
2022, Vol. 62




