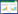中国科学院微生物研究所,中国微生物学会
文章信息
- 聂小琴, 董发勤, 刘明学, 李小安, 黄文波, 王成林, 逯小静. 2022
- NIE Xiaoqin, DONG Faqin, LIU Mingxue, LI Xiaoan, HUANG Wenbo, WANG Chenglin, LU Xiaojing.
- 生物含磷大分子与铀酰相互作用模拟计算研究
- Microbial mechanism on distribution, renewal, and maintenance of soil organic carbon pool in karst area
- 微生物学报, 62(6): 2198-2211
- Acta Microbiologica Sinica, 62(6): 2198-2211
-
文章历史
- 收稿日期:2022-03-25
- 修回日期:2022-04-25
- 网络出版日期:2022-05-05
2. 西南科技大学固体废物处理与资源化教育部重点实验室,四川 绵阳 621010;
3. 西南科技大学生命科学与工程学院,四川 绵阳 621010;
4. 绵阳市中心医院国家卫健委核技术医学转化重点实验室,四川 绵阳 621000
2. Key Laboratory of Solid Waste Treatment and Resource Recycle, Ministry of Education, Southwest University of Science and Technology, Mianyang 621010, Sichuan, China;
3. School of Life Science and Engineering, Southwest University of Science and Technology, Mianyang 621010, Sichuan, China;
4. NHC Key Laboratory of Nuclear Technology Medical Transformation, Mianyang Central Hospital, Mianyang 621000, Sichuan, China
铀在环境介质中的迁移及其生物地球化学行为是核环境研究中最受关注的前沿问题之一[1]。微生物在铀的地球化学循环中起着十分重要的作用,凭借界膜作用、新陈代谢改变微环境以影响铀在环境中的分布、循环、富集和迁移[2]。卢龙等[3]发现,作为花岗岩型的下庄铀矿,矿田中铀酰磷酸矿物是非常普遍的,推测在铀矿成矿和风化的过程中,磷除了来源于地下水,极有可能有细菌细胞磷的参与和贡献。笔者课题组近期研究发现,铀在活体生物细胞表面主要以铀的磷酸盐矿物形式稳定存在,磷来自微生物壁膜结构及代谢活动,然而其代谢调控过程和分子作用机制仍不清楚[4]。为了更好地理解铀在生物矿化过程中生物来源的含磷大分子的具体贡献与作用方式,一些科研工作者通过现场实地修复、模拟实验和计算的方法,从宏观到微观不同层面对生物矿化过程进行了探索[5-9]。微生物及酶可以分解环境中的植酸,得到自由磷酸配体从而生成铀磷酸盐矿化体,在具有自由PO43-环境下,微生物还原菌还原得到的U(IV)并不形成二氧化铀矿物(UO2),而是与磷酸盐形成U(IV)-磷酸盐复合物[10]。李福春等[10]研究表明,微生物表面的带负电基团可通过静电吸引菌体周围的金属阳离子,而重金属阳离子再通过“阳离子桥”吸引来自菌体内部以及溶液中的磷酸根基团,形成磷酸铀矿,完成生物矿化过程。生物矿化不仅仅只通过生物活性调节,另外一种比较重要的生物矿化方式-无机矿化,即生物大分子诱导重金属离子矿化,其主要机制是矿物前体分子和细胞壁表面有机分子间的分子识别作用为矿化结构的控制提供了较大的机会。为更好地理解生物分子参与铀-固定的微观过程,近年来利用基于量子理论的计算化学开展分子模拟取得了重要进展。一些学者[11-12]采用密度泛函理论(density functional theory,DFT)和分子动力学(molecular dynamics,MD)方法,从分子、原子和电子结构层面模拟计算了在理想条件下,铀与一些生物大分子的络合配位信息。如Lin等[11]采用MD模拟了铀酰与甘油磷酸盐的作用效应,结果表明,在相互作用过程中,UO22+使甘油磷酸盐的sn-1和sn-2链间的平均角度变大,同时与H2O、Cl-、磷酸基团配位,导致细胞膜流动性降低。Benavides-Maria等[12]采用DFT计算了UO22+与转铁蛋白(transferrin,Tf)相互作用以及Tf-UO22+络合物的结构特征,计算红外光谱结果发现,UO22+主要与Tf中结合位点处的2个酪氨酸,一个天冬氨酸,一个碳酸根离子配位,但是组氨酸并未参与配位。Samir等[13]建立骨桥蛋白中与金属结合位点处的磷酸化多肽片段,利用密度泛函理论计算,表明UO22+与磷酸基团中的一个氧原子、羧酸基团中的2个氧原子以及2个水分子进行络合配位。结合上述研究成果,U与P元素之间存在着密切的联系,UO22+与磷酸化多肽、蛋白质中氨基酸的相互作用也研究比较广泛,但是由于在水溶液中UO22+随着pH不同而络合种态存在差异,因此针对真实环境体系下,应考虑共存离子及常见介质的影响,需要在各种可能的结构中搜寻最稳定的配位方式和聚集体[14]。鉴于在铀矿山环境中,大多数铀污染水体pH基本上在5-9之间[1-2],弱酸性条件下,铀在溶液中主要以UO22+形式为主,随着pH增加,UO22+在水溶液中逐渐与常见的碳酸根离子形成稳定可溶的络阴离子[4, 7],因此本文利用Materials Studio软件进行模拟计算,主要研究在弱酸性(纯铀酰离子)、中性至弱碱性(碳酸铀酰离子)两种体系下,铀酰与4种代表性生物细胞壁膜及代谢含磷大分子(如脂多糖、磷壁酸、植酸、磷脂)的相互作用微观信息,明确相互结合的活性位点、作用能、作用力以及距离等参数,旨在为深入认识铀的生物矿化过程与机制提供理论参考。
1 模拟计算方法 1.1 理论建模方法简介根据文献报道及笔者前期研究发现,生物体与铀酰离子之间的相互作用,主要取决于生物壁膜分子中的关键基团; 由此,可通过建立铀酰离子模型与生物含磷大分子的简化模型,更加直观地研究二者的关系。主要步骤包括:通过Material Studio软件中的Visualizer窗口进行4种含磷大分子的简易分子模型的搭建; 用Forcite tool模块对搭建好的模型进行分子结构力学优化; 通过Amorphous将优化后的模型进行晶胞处理。蒙特卡罗数据采用模块进行计算,其计算模型作为分子动力学弛豫运算基础。
1.2 蒙特卡罗计算方法细节(MC)计算前,利用Amorphous Cell tool模块将分子模型进行装盒处理,每个盒子里装设一个生物含磷大分子模型,密度为0.1 g/cm3。在Adsorption Locator tool模块中,每个含磷大分子模型中加入3个铀酰离子(模拟弱酸性条件)或者3个铀酰离子以及1个碳酸根离子(模拟中性至弱碱性条件)。计算参数:计算立场采用Universal (UFF)普适立场,利用Metropolis计算法,以使体系能量逐渐降低、趋于稳定状态。计算精度为中等(medium),温度设置为298 K; 步均衡步数为1 000 000,计算10个循环,共计10 000 000步; 初始电荷为各原子的设置电荷,收敛精度为5.0×10-4。在求和方法中,范德华力采用原子算法,截断距离:9 Å,样条曲线宽度:1 Å,缓冲宽度:0.5 Å。Ewald求和法用于计算库伦静电作用力,缓冲宽度:0.5 Å,精度控制为1.0×10-5 kcal/mol[15]。
1.3 分子动力学计算方法细节(MD)分子动力学弛豫采用蒙特卡罗计算模拟后的模型,利用Forcite中的Dynamics模式进行。该部分的计算内容分两步进行,第一步进行正则系综(canonical ensemble,NVT)计算,第二部分进行微正则系综(microcanonical ensemble,NVE)计算。计算细节:计算立场同样采用Universal (UFF)普适立场,计算精度为medium,压力为1 atm,压力由Berendsen恒压器控制,时间为1 000 ps,温度设置为298 K,其控制为Nose控制(Q ratio值为1.0);在求和方法中,范德华力采用原子算法,截断距离:9 Å,样条曲线宽度:1 Å,缓冲宽度:0.5 Å。Ewald求和法计算用于计算库伦静电作用力,缓冲宽度:0.5 Å,精度控制为1.0×10-5 kcal/mol[16]。
2 结果与讨论 2.1 分子模型构建及优化 2.1.1 生物含磷大分子模型构建脂多糖是革兰氏阴性菌细胞壁的主要组成部分,主要由三部分构成,分别是核心多糖、特异性多糖和类脂A。其中,类脂A作为脂多糖的主要成分,作为生物致病性的关键因素。因此,本文主要采用类脂A的简化模型进行模拟计算,分子结构式为C96H182O25N2P2,其结构如图 1A所示。磷壁酸是革兰氏阳性菌细胞壁上特有的酸性多糖物质,其在细胞壁中按结合位置可分为两种。一种是膜磷壁酸,其贯穿细胞壁中的整个肽聚糖层,与细胞膜相连。另一种是壁磷壁酸,主要是末端以磷酸二酯键与肽聚糖的N-乙酰胞壁酸残基相连接,其含量最高时可占细胞壁的50%。因此,本研究采用简化壁磷壁酸的结构进行建模计算,其分子结构式为C5H13O8P,结构如图 1B所示。磷脂是细胞中磷脂双分子层的主要成分,对细胞中物质的进出以及形态起着控制作用,可分为甘油磷脂和鞘磷脂。甘油磷脂是细胞壁膜的主要成分,由此本文采用简化甘油磷脂后的磷酸化甘油磷脂进行模拟计算,其分子结构式为C19H37O7P,结构如图 1C所示。植酸是许多植物组织中作为磷存储的物质,其由来是肌醇的6个羟基均被磷酸酯化后生成的肌醇衍生物,其在植物中的胚芽、种子、茎和芽中广泛存在。本课题组前期研究表明,植酸是铀胁迫下浮萍分泌的重要有机酸,且对铀具有极强的络合能力,其分子模型如图 1D所示,其分子式为C6H18O24P6。
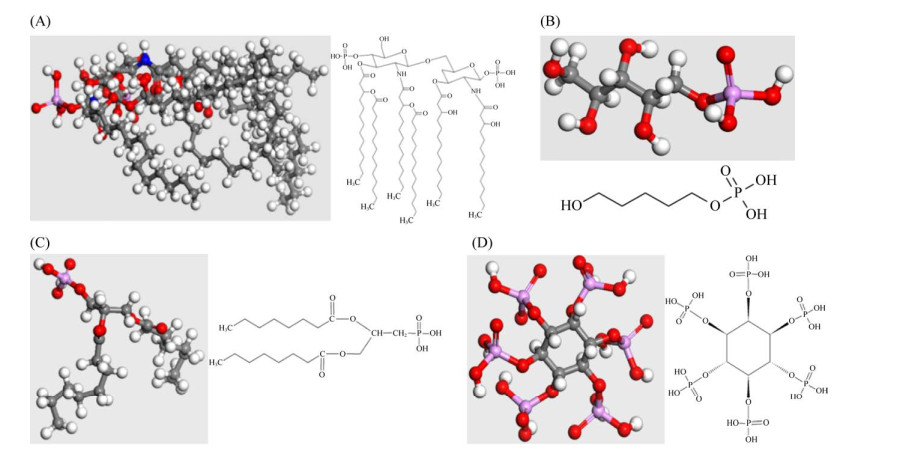
|
| 图 1 脂多糖(A)、磷壁酸(B)、磷脂(C)和植酸(D)简化模型 Figure 1 The simplified models of lipopolysaccharide (A), teichoic acid (B), phospholipid (C) and phytic acid (D). |
2.1.2 铀酰及碳酸根离子模型
铀酰离子在弱酸性水溶液中,主要呈现为UO22+,根据文献报道及实验数据[8],其模型建立参数设置为:U-O键长为1.77 Å,键角为180度,铀原子价态为+6价,氧原子价态为-2价,分子模型如图 2A所示。在生物介导的铀-磷生物矿化的有效pH区间为3-6,当pH > 5时,UO22+逐渐与溶液的CO32-形成稳定可溶的碳酸铀酰络合物[5],碳酸根离子在水溶液中呈现为三角锥,在本文中的参数设置为C-O键长为1.36 Å,键角为109.47度,碳原子价态为+4价,氧原子价态为-2价,分子模型如图 2B所示。
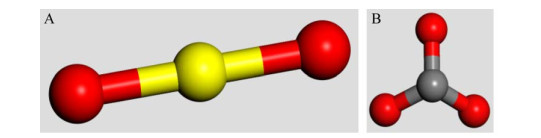
|
| 图 2 铀酰离子(A)和碳酸根离子(B)模型 Figure 2 The models of uranyl ion (A) and carbonate ion (B). |
2.1.3 分子模型结构优化
对建立的磷壁酸、脂多糖、磷脂、植酸简化模型进行分子力学结构优化,优化结果如表 1所示。相应计算能量按照公式(1-3)进行。
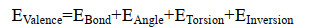
|
公式(1) |
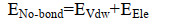
|
公式(2) |
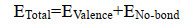
|
公式(3) |
| Structural model | Optimized conditions | Bond valence energy/(kcal/mol) | Non-bond valence energy/(kcal/mol) | Total energy/ (kcal/mol) |
|||||
| Bond | Angle | Torsion | Inversion | Vdw | Ele | ||||
| Teichoic acid | Before optimization | 149.58 | 82.26 | 1.00 | 0.00 | 8.64 | 0.00 | 241.49 | |
| After optimization | 2.16 | 76.38 | 0.36 | 0.00 | 10.77 | 0.00 | 89.66 | ||
| Lipopolysaccharide | Before optimization | 856.11 | 185.23 | 20.75 | 0.31 | 4 291.09 | -284.47 | 5 069.01 | |
| After optimization | 27.39 | 218.34 | 32.93 | 0.81 | 79.48 | -473.64 | -114.69 | ||
| Phospholipid | Before optimization | 66.75 | 208.26 | 10.86 | 0.25 | 249.08 | -134.16 | 401.05 | |
| After optimization | 3.37 | 84.57 | 2.84 | 0.01 | 17.59 | -115.95 | -7.58 | ||
| Phytic acid | Before optimization | 522.61 | 507.07 | 2.15 | 0.00 | 59.58 | 0.00 | 1 091.41 | |
| After optimization | 6.89 | 472.54 | 2.29 | 0.00 | 15.35 | 0.00 | 497.07 | ||
4种含磷大分子模型的结构优化后,自身体系能量都减小,体系趋于更加稳定。磷壁酸优化前,主要能量项为键伸缩能(149.58 kcal/mol)。优化后,其能量约为原始能量的三分之一,其主要能量项变为键角面内弯曲能,能量为76.38 kcal/mol。植酸的优化结果类似于磷壁酸,其主要能量项也为键角面内弯曲能,其能量总量由1 091.41 kcal/mol降至497.07 kcal/mol。脂多糖结构优化后,其能量大约减少至原来的五十分之一,主要能量供应项为静电作用力,静电力上升约1.5倍,范德华力下降了2个数量级,优化后的二面角扭转能高于优化前,其最终总能量为-114.69 kcal/mol。磷脂优化结果类同与脂多糖,主要能量项也为静电作用力,其最终总能量也由正值变为负值(-115.95 kcal/mol)。
2.2 蒙特卡罗分析(MC)4种生物含磷大分子对铀酰离子的吸附作用力可以从吸附能的大小判断,吸附能为负值,则表明反应自发进行,并释放热量,反之亦然; 吸附能绝对值越大,作用效果越强烈。吸附能的计算见公式(4)。
|
公式(4) |
其中,EAdsorption energy表示体系总吸附能; ERigid adsorption energy为刚性吸附能,表示未弛豫的被吸附物在吸附剂表面发生吸附作用的吸附能; EDeformation energy表示被吸附的物质在吸附剂表面发生弛豫变形时释放的能量[17]。
弱酸性(纯铀酰离子)体系下,脂多糖、磷壁酸、植酸、磷脂与铀酰离子相互作用的吸附能大小以及吸附概率如图 3A所示,4种含磷大分子对铀酰离子的吸附能均为负值,表明吸附过程中都存在能量的释放,其吸附能量主要集中在-109 kcal/mol至-114 kcal/mol。综合考虑吸附能和吸附概率,吸附效果最佳为脂多糖,其次分别是磷脂、植酸和磷壁酸。脂多糖的吸附能覆盖-109 kcal/mol至-114 kcal/mol范围,磷脂吸附能范围主要为-109 kcal/mol至-112.5 kcal/mol,而-109 kcal/mol至-111.5 kcal/mol则为磷壁酸和植酸的吸附能覆盖范围。
|
| 图 3 4种含磷大分子在纯铀酰体系(A)和碳酸铀酰体系(B)下对铀酰的吸附能及概率 Figure 3 Adsorption energy and probability of four macromolecules with phosphorus for uranyl in pure uranyl system (A) and uranyl carbonate system (B). |
中性及弱碱性(碳酸根与铀酰离子)体系条件下,4种含磷大分子吸附铀酰离子的吸附能大小及吸附概率如图 3B所示,从吸附能角度考虑,吸附效果顺序类同于纯铀酰离子体系。而从最大吸附概率排序,则是脂多糖 > 磷壁酸 > 植酸 > 磷脂。但是,该体系与纯铀酰离子体系相比,其整体吸附概率都存在下降,可能是体系中的碳酸根离子对铀酰离子存在静电相互作用,牵制铀酰离子附着于含磷大分子。
纯铀酰离子体系下,4种含磷大分子对铀酰离子的吸附能如表 2所示,吸附性能最佳为脂多糖,平均吸附能为-337.31 kcal/mol,刚性吸附能为-9.679 kcal/mol,弛豫形变的吸附能为-327.63 kcal/mol。其次顺序为磷脂 > 植酸 > 磷壁酸。结合能的大小取决于向心配合体的稳定性[10],也就是本文中的含磷大分子; 由于脂多糖结构比较稳定,因此其结合能较大。值得注意的是,4种含磷分子弛豫形变的吸附能相同,这是因为铀酰离子本身结构相对简单,其空间变化比较单一,导致EDefor相同。
| Energy/(kcal/mol) | EAd | ERigid-ad | EDefor |
| Teichoic acid | -332.09 | -4.46 | -327.63 |
| Lipopolysaccharide | -337.31 | -9.68 | -327.63 |
| Phospholipid | -334.30 | -6.67 | -327.63 |
| Phytic acid | -332.42 | -4.79 | -327.63 |
在碳酸根+铀酰离子体系下,4种含磷大分子对铀酰离子的吸附能如表 3所示,磷壁酸、脂多糖、磷脂、植酸的平均吸附能分别为-332.09、-337.31、-334.30、-332.42 kcal/mol,顺序类似于纯铀酰离子体系; 但其吸附能绝对值和刚性吸附能绝对值都略高于纯铀酰离子体系,吸附能绝对值大约高出4-7 kcal/mol,刚性吸附能绝对值高出约为2-5 kcal/mol。这可能是由于碳酸根离子的存在,铀酰离子和碳酸根离子体系作为吸附质被吸附在作为吸附剂的生物含磷大分子上,导致ERigid adsorption energy和EDeformation energy的绝对值略大于纯铀酰离子体系。该体系下的所有弛豫形变的吸附能也都相同,均为-329.48 kcal/mol。
| Energy/(kcal/mol) | EAd | ERigid-ad | EDefor |
| Teichoic acid | -336.40 | -6.92 | -329.48 |
| Lipopolysaccharide | -344.34 | -14.85 | -329.48 |
| Phospholipid | -339.71 | -10.22 | -329.48 |
| Phytic acid | -337.56 | -8.07 | -329.48 |
2.3 分子动力学分析(MD) 2.3.1 磷壁酸与铀酰离子的分子动力学分析
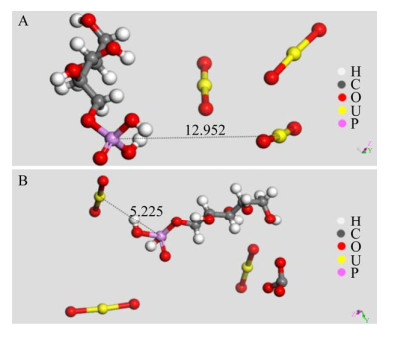
磷壁酸与铀酰离子的模拟计算中,在纯铀体系下,整个计算体系密度由0.1 g/cm3变为0.449 g/cm3,密度大幅上升。铀酰离子中的铀原子与磷壁酸中的磷原子最近为12.952 Å (图 4A)。而碳酸根和铀酰离子体系下,经过分子动力学弛豫后密度变为0.475 g/cm3,大于纯铀酰离子体系。铀原子与磷原子的最近距离为5.225 Å,这是因为碳酸根位于最佳P-U键的相反侧,并与其中一个铀酰离子存在静电作用,导致其对磷酸根与铀酰离子之间的相互作用影响很小。碳酸根与磷壁酸的尾端羟基比较亲和,其存在时磷壁酸结构趋近于线性结构(图 4B),而纯铀酰离子体系中的磷壁酸略有弯曲,倾向于折叠。此外,铀原子对碳酸根中的3个氧原子存在耦合作用,加上碳链上的氧原子,在其赤道面形成四氧原子或六氧原子配位体系,有助于铀原子往体系稳定方向调和[18]。
|
| 图 4 在纯铀酰体系(A)和碳酸铀酰体系(B)中磷壁酸与铀酰离子相互作用图 Figure 4 Interaction between teichoic acid and uranyl ion in pure uranyl system (A) and uranyl carbonate system (B). |
2.3.2 脂多糖与铀酰离子的分子动力学分析
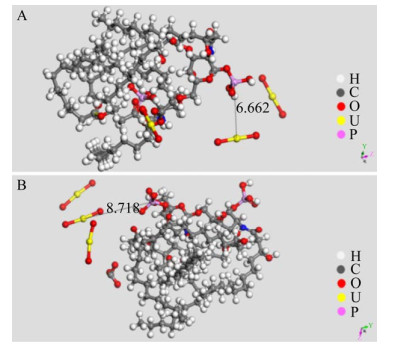
脂多糖的模拟计算中,纯铀酰离子体系条件下P-U距离更短(6.662 Å),体系密度相比较初始密度没有太大变化(0.1 g/cm3),也较为相近(0.140-0.150 g/cm3)。3个铀酰离子均出现在磷酸基团附近,表明含磷基团对其存在吸引作用,这是因为脂多糖结构不对称以及表面存在静电位梯度,其对带电离子和两性物质存在良好的亲和[19]。而碳酸铀酰离子体系中,P-U距离为8.718 Å,略大于纯铀酰离子体系下的P-U键距离,根据文献报道,脂多糖对阴性离子也存在亲和作用,但作用强度较弱,这可能干扰磷酸基团对铀酰离子的亲和,导致P-U间距变大[20] (图 5)。3个铀酰离子聚集在磷酸基团与碳酸根离子之间,可能是碳酸根离子带负电,铀酰离子带正电,碳酸根对磷酸基团吸引铀酰离子存在牵制作用。同时,碳酸根对脂多糖中的碳链十分亲和,其氧原子与碳链中的氢原子趋于接近,有助于形成氢键,稳定体系结构。
|
| 图 5 在纯铀酰体系(A)和碳酸铀酰体系(B)中脂多糖与铀酰离子相互作用图 Figure 5 Interaction between lipopolysaccharides and uranyl ion in pure uranyl system (A) and uranyl carbonate system (B). |
2.3.3 磷脂与铀酰离子的分子动力学分析
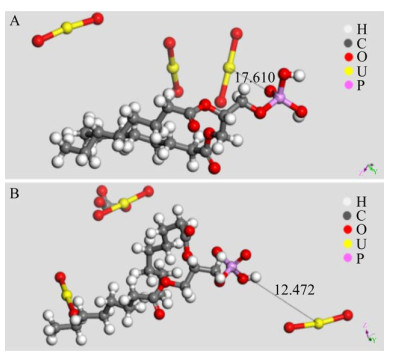
磷脂与铀酰离子的模拟计算结果表明:在纯铀酰离子体系下,其密度增大至0.291 g/cm3,体系变得较为紧致。磷脂中的两条碳链并行排布,3个铀酰离子趋近于包裹磷脂分子,铀酰离子对碳链中衔接的氧原子十分亲和[21]; 但U-P间距较远(17.610 Å)。而在碳酸根和铀酰离子体系中,体系密度增大至0.305 g/cm3; U-P间距略小(12.472 Å),其情况与磷壁酸的实验结果类似。碳酸根的存在使磷脂中碳链发生重排,表明溶剂化离子对磷脂的空间结构存在较大影响(图 6)。
|
| 图 6 在纯铀酰体系(A)和碳酸铀酰体系(B)中磷脂与铀酰离子相互作用图 Figure 6 Interaction between phospholipid and uranyl ion in pure uranyl system (A) and uranyl carbonate system (B). |
2.3.4 植酸与铀酰离子的分子动力学分析
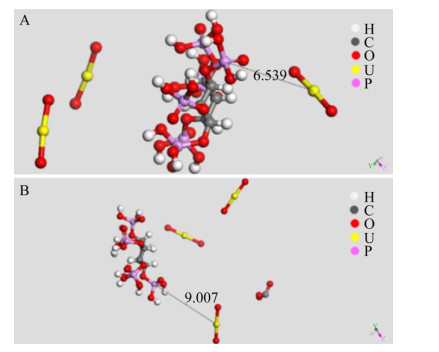
植酸与铀酰离子的模拟计算结果表明:两种体系的密度比较相近(0.220-0.230 g/cm3),纯铀酰离子体系下,铀酰离子分布在植酸碳环两侧,U-P距离为6.539 Å。此外,通过铀原子还通过静电力吸引以及耦合磷酸基团中的氧原子,导致磷酸基团中的2个羟基氧往U元素一侧分布[11]。而在碳酸根+铀酰离子体系中,铀酰离子主要集中在碳环一侧,最里层为铀酰离子,最外围为碳酸根离子。U-P键距离变大(9.007 Å),碳酸根离子位于3个铀酰离子中央,由此推断静电吸引可能是U-P键距离变大的原因之一(图 7)。
|
| 图 7 在纯铀酰体系(A)和碳酸铀酰体系(B)中植酸与铀酰离子相互作用图 Figure 7 Interaction between phytic acid and uranyl ion in pure uranyl system (A) and uranyl carbonate system (B). |
2.3.5 相互作用前后的能量变化
蒙特卡罗模拟计算后的分子模型进行分子动力学弛豫,先进行NVT系综,后进行NVE系综,得到生物含磷分子对铀酰的最佳吸附模型。
其中,纯铀酰离子体系下其能量变化计算公式(5-8)。
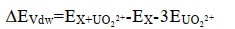
|
公式(5) |
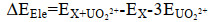
|
公式(6) |
碳酸根+铀酰体系其能量变化计算公式如下:
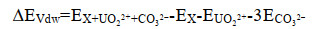
|
公式(7) |
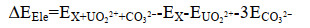
|
公式(8) |
其中,EX+UO22+表示纯铀酰离子体系下,分子动力学弛豫后的体系总能量; EX表示单个含磷大分子体系的总能量; EUO22+表示铀酰离子的总能量; EX+UO22++CO32-表示碳酸根离子和铀酰离子体系下,分子动力学弛豫后的体系总能量; ECO32-表示碳酸根离子的总能量; ∆EVdw表示体系之间的范德华力之差; ∆EVdw表示体系之间的静电力之差。
在纯铀酰体系和碳酸铀酰体系下,4种含磷大分子模型与铀酰作用前后的能量变化如表 4所示。正值表示排斥能,负值表示吸引能。分子动力学弛豫后,在2种体系下,4种含磷分子与铀酰的体系能量供应项都变为由键角面内弯曲能占主导。分析认为,生物含磷大分子在与铀酰离子相互作用前,铀酰离子与生物含磷大分子的各项能量值相加,其结构并不稳定; 当完成分子动力学弛豫后,体系中的各个子体在立场、温度和压力等参数条件的驱动下,体系趋于稳定,能量趋向于最低; 这个过程中主要通过改变原有分子模型中的键角来实现,同时也改变了体系中的非键合力(范德华力和静电作用力),由此键角面内弯曲能成为主要能量供应项[22]。在纯铀酰体系下,磷壁酸和植酸与铀酰离子作用力主要是范德华力占主导,二者都是排斥能。而脂多糖与磷脂则是静电力占主导,脂多糖对铀酰表现为静电吸附,而磷脂则是静电排斥。在碳酸铀酰体系下,磷壁酸和植酸对铀酰均是范德华力; 磷脂对铀酰作用中,范德华力略大于静电力,且都为斥力。脂多糖对铀酰的静电斥力远大于范德华引力。
| Structural models | Bond valence energy/(kcal/mol) | Non-bond valence energy/(kcal/mol) | Total energy/ (kcal/mol) |
|||||||
| Bond | Angle | Torsion | Inversion | Vdw | ∆EVdw | Ele | ∆EEle | |||
| UO22+ | 109.21 | - | - | - | 0.00 | - | 0.00 | - | 109.21 | |
| CO32- | 1.85 | 0.00 | 0.00 | 0.00 | 0.00 | - | 0.00 | - | 1.85 | |
| Teichoic acid | 2.16 | 76.38 | 0.36 | 0.00 | 10.77 | - | 0.00 | - | 89.66 | |
| Teichoic acid+UO22+ | 12.31 | 90.68 | 0.77 | 0.00 | 9.67 | -1.094 | 0.00 | 0.00 | 113.43 | |
| Teichoic acid+UO22++CO32- | 10.79 | 89.34 | 1.01 | 0.00 | 11.38 | 0.610 | 0.00 | 0.00 | 112.52 | |
| Lipopolysaccharide | 27.39 | 218.34 | 32.93 | 0.81 | 79.47 | - | -473.64 | - | -114.69 | |
| Lipopolysaccharide+UO22+ | 133.10 | 329.80 | 30.62 | 0.32 | 35.26 | -44.220 | -346.39 | 127.25 | 182.70 | |
| Lipopolysaccharide+UO22++CO32- | 110.53 | 352.97 | 30.95 | 2.11 | 47.77 | -31.710 | -340.97 | 132.68 | 203.36 | |
| Phospholipid | 3.37 | 84.57 | 2.84 | 0.00 | 17.59 | - | -115.9 | - | -7.58 | |
| Phospholipid+UO22+ | 25.04 | 106.5 | 4.04 | 0.01 | 14.80 | -2.790 | -112.6 | 3.31 | 37.71 | |
| Phospholipid+UO22++CO32- | 23.51 | 112.54 | 7.54 | 0.66 | 16.59 | -1.000 | -115.4 | 0.54 | 45.42 | |
| Phytic acid | 6.893 | 472.54 | 2.29 | 0.00 | 15.35 | - | 0.00 | - | 497.07 | |
| Phytic acid+UO22+ | 28.89 | 495.70 | 2.76 | 0.00 | 21.17 | 5.823 | 0.00 | 0.00 | 548.52 | |
| Phytic acid+UO22++CO32- | 25.45 | 502.66 | 4.75 | 0.00 | 15.16 | -0.186 | 0.00 | 0.00 | 548.01 | |
| -: not detected. | ||||||||||
2.3.6 含磷大分子与铀的作用模式推测
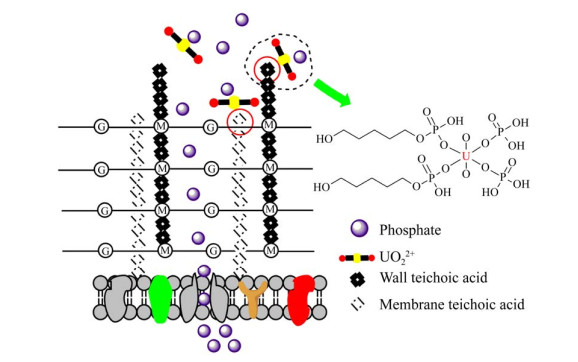
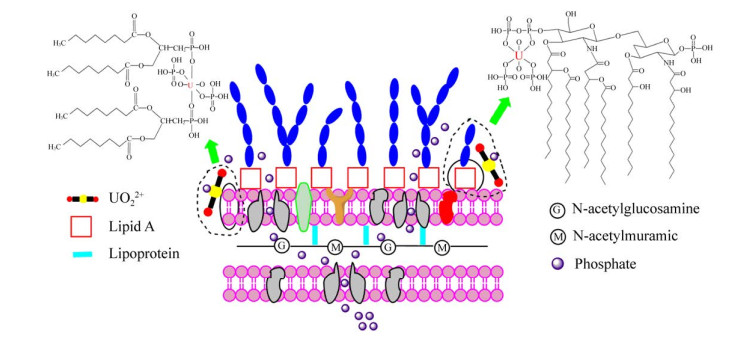
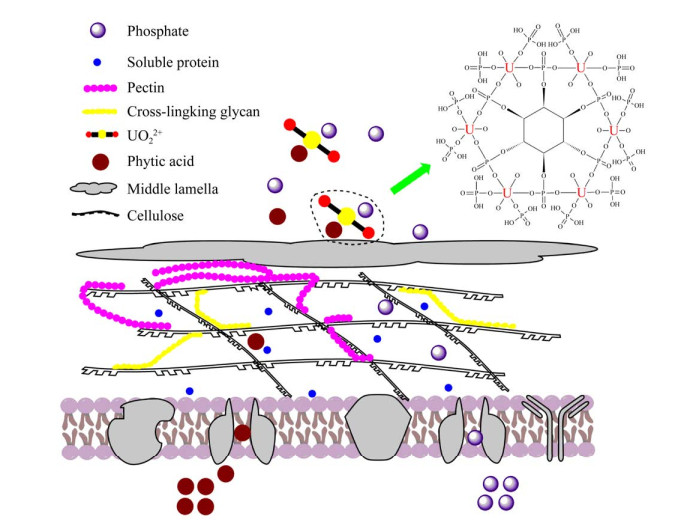
根据理论计算数据,4种含磷大分子的含P基团对UO22+都存在较高的亲和作用。结合课题组前期研究基础[23-26],对生物含磷大分子介导P-U生物矿化机制做如下推断:对于革兰氏阳性菌,细胞壁特有的成分是磷壁酸,其处于细胞壁外侧,推测革兰氏阳性菌与UO22+的相互作用过程中,磷铀矿化体或无定型磷铀络合物的形成过程如图 8所示,铀酰离子刚开始与革兰氏阳性菌细胞接触时,向磷壁酸靠近,优先与其中的磷酸基团络合,成核后菌体因铀酰离子的胁迫,释放的无机磷酸根与磷壁酸上的铀酰相结合,由此逐步堆积,促进矿化体生长。对于革兰氏阴性菌,如图 9所示,细胞壁外的脂多糖存在天然的负电性梯度,成为与UO22+接触的活跃成分,而细胞膜中的磷脂分子主要结合被转运或游离靠近的UO22+相互作用。两种成分对铀酰离子的吸附主要是优先形成成核位点,矿化体的生长需要菌体释放的磷酸物质进行周期性延展。对于植物细胞,如图 10所示,在UO22+的胁迫作用下,其特有的生理代谢物植酸(肌醇六磷酸)含有大量的磷酸基团,其释放需要穿越植物细胞的细胞膜、果胶与纤维素交杂的细胞壁,到达胞间层,与铀酰离子络合,而后续生长的矿化体则主要着落于胞间层[27]。
|
| 图 8 革兰氏阳性菌与铀酰离子相互作用的示意图 Figure 8 Schematic diagram of Gram-staining positive bacteria interacting with uranyl ions. |
|
| 图 9 革兰氏阴性菌与铀酰离子相互作用的示意图 Figure 9 Schematic diagram of Gram-staining negative bacteria interacting with uranyl ions. |
|
| 图 10 植物与铀酰离子相互作用的示意图 Figure 10 Schematic diagram of plant interaction with uranyl ions. |
3 结论
本论文主要研究了纯铀酰体系与碳酸铀酰体系下,代表性生物壁膜及代谢含磷大分子与铀酰离子的蒙特卡罗与分子动力学模拟分析。结果表明:(1) 磷壁酸、脂多糖、磷脂和植酸分子力学结构优化后,体系能量都大幅减少,结构都趋于稳定。4种含磷大分子在纯UO22+、CO32-与UO22+共存的两种条件下,其吸附能的范围主要分布在-109 kcal/mol至-114 kcal/mol之间,吸附均容易发生,吸附的主要能量以弛豫形变能量为主。(2) 分子动力学弛豫结果从原子层面证实铀酰与磷酸基团确实存在相互作用,范德华力与静电力在铀酰与磷酸基团相互作用过程中扮演重要推动力。其中,在纯铀酰体系下,U-P键作用距离最短的是脂多糖和植酸; 而在碳酸铀酰体系中,U-P键作用距离最短的是磷壁酸和磷脂; 这与分子结构与离子种态有关。在两种体系下,脂多糖对UO22+的主导作用力是静电力; 磷壁酸和植酸对UO22+的主导作用力是范德华力,磷脂对UO22+表现为范德华力与静电力相当。(3) 4种生物含磷大分子与铀酰离子相互作用过程中,磷酸基团中的氧原子对铀酰中的氧原子存在强亲和力。推测生物矿化体的生成过程中,静电力与范德华力推动铀酰离子与磷酸基团靠近,形成成核位点,为矿化体的生长形成基本的环境,而生物自身新陈代谢,释放含磷物质,促进矿化体生长。
| [1] | Newsome L, Morris K, Trivedi D, Bewsher A, Lloyd JR. Biostimulation by glycerol phosphate to precipitate recalcitrant uranium(IV) phosphate. Environmental Science & Technology, 2015, 49(18): 11070-11078. |
| [2] | Salome KR, Beazley MJ, Webb SM, Sobecky PA, Taillefert M. Biomineralization of U(VI) phosphate promoted by microbially-mediated phytate hydrolysis in contaminated soils. Geochimica et Cosmochimica Acta, 2017, 197: 27-42. DOI:10.1016/j.gca.2016.10.008 |
| [3] |
Lu L, Chen FR, Wang RC. The compositional characteristic of uranyl minerals in Xiazhuang uranium ore field: insight for the oxidation behavior of UO2 in nuclear waste repository. Geological Review, 2006, 52(4): 562-569.
(in Chinese) 卢龙, 陈繁荣, 王汝成. 粤北下庄铀矿田中铀酰矿物的成分特征——对核废料处置库中UO2氧化行为的启示. 地质论评, 2006, 52(4): 562-569. DOI:10.3321/j.issn:0371-5736.2006.04.016 |
| [4] | Huang WB, Cheng WC, Nie XQ, Dong FQ, Ding CC, Liu MX, Li Z, Hayat T, Alharbi NS. Microscopic and spectroscopic insights into uranium phosphate mineral precipitated by Bacillus mucilaginosus. ACS Earth and Space Chemistry, 2017, 1(8): 483-492. DOI:10.1021/acsearthspacechem.7b00060 |
| [5] | MacAskie LE, Empson RM, Cheetham AK, Grey CP, Skarnulis AJ. Uranium bioaccumulation by a Citrobacter sp. as a result of enzymically mediated growth of polycrystalline HUO2PO4. Science, 1992, 257(5071): 782-784. DOI:10.1126/science.1496397 |
| [6] | Stetten L, Blanchart P, Mangeret A, Lefebvre P, Le Pape P, Brest J, Merrot P, Julien A, Proux O, Webb SM, Bargar JR, Cazala C, Morin G. Redox fluctuations and organic complexation govern uranium redistribution from U(IV)-phosphate minerals in a mining-polluted wetland soil, Brittany, France. Environmental Science & Technology, 2018, 52(22): 13099-13109. |
| [7] | Tu H, Lan T, Yuan GY, Zhao CS, Liu J, Li FZ, Yang JJ, Liao JL, Yang YY, Wang DQ, Liu N. The influence of humic substances on uranium biomineralization induced by Bacillus sp. dwc-. Journal of Environmental Radioactivity, 2019, 197: 23-29. DOI:10.1016/j.jenvrad.2018.11.010 |
| [8] | Alessi DS, Lezama-Pacheco JS, Stubbs JE, Janousch M, Bargar JR, Persson P, Bernier-Latmani R. The product of microbial uranium reduction includes multiple species with U(IV)-phosphate coordination. Geochimica et Cosmochimica Acta, 2014, 131: 115-127. DOI:10.1016/j.gca.2014.01.005 |
| [9] | Rui X, Kwon MJ, O'Loughlin EJ, Dunham-Cheatham S, Fein JB, Bunker B, Kemner KM, Boyanov MI. Bioreduction of hydrogen uranyl phosphate: mechanisms and U(IV) products. Environmental Science & Technology, 2013, 47(11): 5668-5678. |
| [10] |
Li FC, Ma H, Su N, Wang JP, Liu MY, Wang J, Teng F. Clostridium sp. controlled morphology of Mg-bearing calcite and its implication for possible mechanism. Geological Journal of China Universities, 2011, 17(1): 13-20.
(in Chinese) 李福春, 马恒, 苏宁, 王金平, 刘铭艳, 汪君, 滕飞. 梭菌对含镁方解石形态的控制及其可能机理. 高校地质学报, 2011, 17(1): 13-20. DOI:10.3969/j.issn.1006-7493.2011.01.002 |
| [11] | Lin YW, Liao LF. Probing interactions between uranyl ions and lipid membrane by molecular dynamics simulation. Computational and Theoretical Chemistry, 2011, 976(1): 130-134. |
| [12] | Benavides-Garcia MG, Krishnan B. Structural insights into the binding of uranyl with human serum protein apotransferrin structure and spectra of protein-uranyl interactions. Chemical Research in Toxicology, 2009, 22(9): 1613-21. DOI:10.1021/tx900184r |
| [13] | Samir S, Gaëlle C, Aurélie J, Lei Q, Christian B, Jérome R, Lorenzo SP, Eric S, Claude V, Christophe DA. Osteopontin: a uranium phosphorylated binding-site characterization. Chemistry: Weinheim an Der Bergstrasse, Germany, 2013, 19(34): 11261-9. |
| [14] | Chen X, Zhao YF, Zhang YY, Li J. TGMin: an efficient global minimum searching program for free and surface-supported clusters. Journal of Computational Chemistry, 2019, 40(10): 1105-1112. |
| [15] | Prachi J, Vasundhara S, Ravindra P, Virendra S, Saber H, Karna SP. Site specific interaction between ZnO nanoparticles and tryptophan: a first principles quantum mechanical study. Physical Chemistry Chemical Physics: PCCP, 2011, 13(2): 476-9. DOI:10.1039/C0CP01466D |
| [16] | Almora-Barrios N, Austen KF, De Leeuw NH. Density functional theory study of the binding of glycine, proline, and hydroxyproline to the hydroxyapatite (0001) and (0110) surfaces. Langmuir, 2009, 25(9): 5018-5025. DOI:10.1021/la803842g |
| [17] | 周青. 矿物粉尘与微生物壁膜结构分子相互作用模拟研究. 西南科技大学硕士学位论文, 2016. |
| [18] | Kelly SD, Kemner KM, Fein JB, Fowle DA, Boyanov MI, Bunker BA, Yee N. X-ray absorption fine structure determination of pH-dependent U-bacterial cell wall interactions. Geochimica et Cosmochimica Acta, 2002, 66(22): 3855-3871. DOI:10.1016/S0016-7037(02)00947-X |
| [19] | Lins RD, Vorpagel ER, Guglielmi M, Straatsma TP. Computer simulation of uranyl uptake by the rough lipopolysaccharide membrane of Pseudomonas aeruginosa. Biomacromolecules, 2008, 9(1): 29-35. DOI:10.1021/bm700609r |
| [20] | Vazquez GJ, Dodge CJ, Francis AJ. Bioreduction of U(VI)-phthalate to a polymeric U(IV)-phthalate colloid. Inorganic Chemistry, 2009, 48(19): 9485-9490. DOI:10.1021/ic900694k |
| [21] | Chi FT, Li P, Xiong J, Hu S, Gao T, Xia XL, Wang XL. Density functional study of uranyl (VI) amidoxime complexes. Chinese Physics B, 2012, 21(9): 179-186. |
| [22] | 赵二正. 有机物插层氧化石墨的分子动力学计算研究. 西南科技大学硕士学位论文, 2015. |
| [23] | Liu MX, Dong FQ, Yan XY, Zeng WM, Hou LY, Pang XF. Biosorption of uranium by Saccharomyces cerevisiae and surface interactions under culture conditions. Bioresource Technology, 2010, 101(22): 8573-8580. DOI:10.1016/j.biortech.2010.06.063 |
| [24] | Nie XQ, Dong FQ, Liu MX, He HC, Sun SY, Bian L, Yang G, Zhang W, Qin YL, Huang R, Li Z, Ren W, Wang L. Microbially mediated stable uranium phosphate nano-biominerals. Journal of Nanoscience and Nanotechnology, 2017, 17(9): 6771-6780. DOI:10.1166/jnn.2017.14463 |
| [25] | Wang YQ, Nie XQ, Cheng WC, Dong FQ, Zhang YY, Ding CC, Liu MX, Asiri AM, Marwani HM. A synergistic biosorption and biomineralization strategy for Kocuria sp. to immobilizing U(VI) from aqueous solution. Journal of Molecular Liquids, 2019, 275: 215-220. DOI:10.1016/j.molliq.2018.11.079 |
| [26] | Xiang SH, Nie XQ, Dong FQ, Cheng WC, Liu MX, Ding CC, Bian L, Sun SY, Li HL. Spectroscopic and theoretical calculation insight into interaction mechanism between U(VI) and phospholipid under carbonate environment. Journal of Molecular Liquids, 2020, 305(prepublish): 112852. |
| [27] | Nie XQ, Dong FQ, Bian L, Liu MX, Ding CC, He HC, Yang G, Sun SY, Qin YL, Huang R, Li Z, Ren W, Wang L. Uranium binding on Landoltia punctata as a result of formation of insoluble nano-U(VI) and U(IV) phosphate minerals. ACS Sustainable Chemistry & Engineering, 2017, 5(2): 1494-1502. |
 2022, Vol. 62
2022, Vol. 62