中国科学院微生物研究所,中国微生物学会
文章信息
- 郑雪芳, 陈燕萍, 肖荣凤, 刘欣, 江城, 王阶平, 刘波. 2022
- ZHENG Xuefang, CHEN Yanping, XIAO Rongfeng, LIU Xin, JIANG Cheng, WANG Jieping, LIU Bo.
- 水肥菌一体化番茄基质栽培系统青枯病病株和健株根际微生物群落结构的差异
- Difference of rhizobacterial community structure between bacterial wilt infected and healthy tomato plants in a substrate culture system
- 微生物学报, 62(4): 1524-1535
- Acta Microbiologica Sinica, 62(4): 1524-1535
-
文章历史
- 收稿日期:2021-08-20
- 修回日期:2021-11-01
- 网络出版日期:2021-12-14
设施土壤栽培,由于多年连作,土壤板结、土传病害发生严重,如番茄的连茬种植使得青枯病原菌在土壤中的数量不断积累,导致青枯病的大面积爆发[1]。连作障碍的防控技术主要包括轮作换茬、伴生、土壤消毒、土壤改良等[2-3],这些方法取得了一定成效,但不能从根本上解决连作障碍。无土栽培是解决土壤连作障碍最有效的方法之一[4]。水肥菌一体化基质栽培系统是利用物联网技术设计的水肥菌一体化装备,根据地理气候、作物种类和生长阶段进行施肥灌溉,获得作物高产,已成为我国现代农业发展的趋势[5]。
番茄青枯病是由青枯雷尔氏菌(Ralstonia solanacearum)引起的一种毁灭性的土传病害,该病害堪称“植物癌症”,一旦发病就难以控制[6]。病原青枯雷尔氏菌主要通过土壤侵染作物根系,其在土壤中存活能力强,因此防治极为困难。目前,对该病害的一些防治措施主要包括农业防治(如嫁接、抗病育种、土壤改良、轮作套种等)、化学防治、生物防治(如芽胞杆菌、链霉菌等生防菌的利用)等[7]。其中,抗病育种是防治番茄青枯病的理想途径,但由于青枯病原菌寄主范围广,国内外育种专家虽做了大量工作,但能供生产上使用的抗病品种不多[8]; 化学防治见效快,但污染环境且危害人类健康,因此,有必要开辟更多的防治途径。
根际是受植物根部分泌物影响、紧靠根部的狭小土壤区域,是土壤微生态系统的重要组成部分[9]。根际庞大而复杂的微生物群落被称为作物的第二基因组,主要包括细菌、真菌、卵菌、线虫、原生动物、藻类、病毒、古生菌、节肢动物等[10]。作为植物-土壤互作的热点区域,根际栖息着较土体土壤更丰富的微生物群落,是土传病原物入侵作物根系的必经之路[11]。近年来,随着根际微生物组的深入研究,科学家提出通过调节土壤原著微生物群落增强土壤自身的免疫能力,以实现对土传病害的控制[11-12]。
本研究调查水肥菌一体化基质栽培系统番茄的生长状况,采集感染青枯病和健康的番茄植株根际基质土,采用Illumina MiSeq高通量测序技术,比较分析病株和健株根际基质细菌群落结构组成及多样性,为番茄青枯病的有效控制提供参考依据。
1 材料与方法 1.1 供试材料水肥菌一体化装备由福建大丰收灌溉科技有限公司生产,包括了增压装备、过滤装备、控制装备、五桶菌肥装备等; 番茄(Solanum lycopersicum L. cv. Beiying)种苗购自厦门如意种苗高科技股份有限公司; 栽培基质购自厦门江平生物基质技术股份有限公司。盛绿佳农微生物菌肥购自河北田佳力有限公司。
青枯雷尔氏菌TTC (2, 3, 5-氯化三苯基四氮唑)固体培养基[13]:10 g蛋白胨,1 g酪朊水解物,5 g葡萄糖,17 g琼脂,1 000 mL蒸馏水,pH 7.4,121 ℃灭菌20 min。灭菌后冷却至55 ℃左右加入已灭菌的1%的TTC水溶液,其终浓度为0.005%。青枯雷尔氏菌SP (sucrose peptone)液体培养基:5 g蛋白胨,20 g蔗糖,0.5 g KH2PO4,0.25 g MgSO4,1 000 mL蒸馏水,pH 7.2,121 ℃灭菌20 min。
1.2 试验地点田间试验选址在福建福州宦溪镇降虎村(北纬25°15′,东经118°08′,海拔600 m)属南亚热带季风性湿润气候,年平均温度19 ℃,年平均降雨量达1 500 mm。
1.3 试验设计设计了水肥菌一体化基质栽培系统,包括了设施大棚、水肥菌一体化装备、栽培架设施等。大棚面积266.67 m2,选取相邻3个大棚(编号1#、2#、3#)为试验田块,2021年5月15日种植,采用基质栽培模式,即将10 kg栽培基质装置于50 cm (长)×35 cm (宽)×27 cm (高)的塑料框中,每框种植2株番茄苗,每大棚种植400株。移栽后,每周施肥配方如下:营养元素和微生物菌剂的五桶配制系统(每个桶200 kg水):A桶:硝酸钙7.0 kg,硝酸铵3.9 kg; B桶:磷酸二氢钾1.9 kg; C桶:硫酸镁4.5 kg,硼酸45.76 g,钼酸铵0.32 g; D桶:螯合铁320 g,螯合锰25.83 g,螯合锌3.5 g,螯合铜1.28 g; E桶:盛绿佳农微生物菌肥2.0 kg; 通过水肥菌一体机滴灌施肥水。
1.4 田间病害调查及样本采集分别于2021年6月15日(营养生长期)和2021年7月15日(生殖生长期)调查3个试验大棚青枯病发病情况,根据病情程度可分为5个级别[14]:健株(0级); 植株有1-2个枝条出现萎蔫症状(1级); 植株有3-5个枝条出现萎蔫症状(2级); 植株整体出现枯萎状态,植株中1-2个叶片干枯、退绿、变黄(3级); 植株全部叶片萎蔫、退绿,茎秆干枯退绿,植株茎部无法正常挺直(4级)。统计青枯病发病率(disease incidence,DI)和病情指数(disease severity index,DSI) (公式1-2)。

|
公式(1) |

|
公式(2) |
采集生殖生长期番茄根际基质样本,每个试验大棚随机选取不同发病级别植株各5株,将整株挖出,抖去松散基质,收集附着根上的基质为小样,再将相同大棚采集相同发病等级植株根际基质小样分别混合、拌匀、去砂砾和植物残体,过2 mm筛后,于4 ℃冰箱保存,用于青枯病原菌分离鉴定; 另将病株(1、2、3、4发病等级)根际土样混合,3个大棚病株根际基质样本分别编号D1#、D2#和D3#,健株(0级)根际基质样本编号H1#、H2#和H3#,用于高通量测序。
1.5 番茄根际基质青枯病病原菌分离与鉴定称取10 g基质样本,用无菌水进行10×梯度稀释后,取200 μL稀释液涂布于TTC培养基上进行青枯病原菌的分离。每个处理重复3次,置于(30±1) ℃人工气候箱内培养48 h后,观察菌落的形态,计算活菌数及每克基质青枯雷尔氏菌数量。
分离的青枯雷尔氏菌采用水煮法提取DNA[15]。采用青枯雷尔氏菌的特异性检测引物pehA#6和pehA#3对分离的菌株进行分子检测,引物设计参见文献[16],以GMI1000为参比菌株(阳性对照)。25 μL的PCR反应体系中包括:10×Buffer 2.5 μL,10 mmol/L dNTPs 0.5 μL,ddH2O 18.7 μL和10 mmol/L引物对各1 μL,2.5 U Taq酶,25 ng DNA模板。PCR反应程序为:96 ℃预变性1 min; 96 ℃变性30 s,70 ℃退火30 s,72 ℃复性1 min,重复2个循环; 94 ℃变性30 s、70 ℃退火30 s、72 ℃复性1 min,重复33个循环; 最后72 ℃延伸5 min。PCR产物的检测:PCR产物点样于1.5%的琼脂糖凝胶,以100 bp DNA Ladder Marker作为标准分子量,80 V电压、1倍TAE缓冲液中电泳1 h,EB染色后,在自动凝胶成像分析系统上观察、拍照。
1.6 16S rRNA基因高通量测序供试基质微生物多样性检测,由北京奥维森基因科技有限公司完成。选取细菌16S rRNA基因V3-V4区,利用Illumina MiSeq PE300高通量测序平台测序,16S rRNA基因引物为520 F (5′-AYTGGGYDTAAAGNG-3′)和806 R (5′-TAC NVG GGTATCTAATCC-3′)。PCR反应体系为(25 μL):12.5 μL KAPA 2G Robust Hot Start Ready Mix,上下游引物(5 µmol/L)各1 μL,2 μL模板DNA和8.5 μL无菌水。PCR反应程序为95 ℃预变性5 min; 95 ℃变性45 s,55 ℃退火50 s,72 ℃复性45 s,28个循环; 最后72 ℃延伸10 min。
1.7 数据处理通过Illumina MiSeq平台进行paired-end测序,下机数据经过QIIME (v1.8.0)软件过滤、拼接、去除嵌合体,去除打分低于20、碱基模糊、引物错配或测序长度小于150 bp的序列。根据barcodes归类各处理组序列信息聚类为用于物种分类的OTU (operational taxonomic units),OTU相似性设置为97%。对比silva数据库,得到每个OTU对应的物种分类信息。再利用Mothur软件(v1.31.2)进行α多样分析(包括Shannon、Simpson和Chao1等)。使用Canoco 5.0软件进行冗余分析(redundancy analysis,RDA),用于辨别番茄根际微生物群落组成与青枯雷尔氏菌分布数量的相关性。试验数据采用Excel 2007和DPS 7.05软件进行系统处理和统计分析,LSD法检验P=0.05水平的差异显著性。
2 结果与分析 2.1 番茄田间青枯病发病率与病情指数分析番茄田间青枯病病情调查结果如表 1所示,营养生长期植株的发病率为2.50%,病情指数是1.25,生殖生长期植株的发病率为6.17%,病情指数为5.11,随着番茄的生长发病率逐渐提高; 番茄植株在生殖生长期的发病率和病情指数均大于营养生长期,且3级和4级的发病植株数量分别是营养生长期的4.55倍和14.00倍。
| Growth stage | Percentage of plants with different disease grades (average±SD)/% | DI/% | DSI | ||||
| 0 grade | 1st grade | 2nd grade | 3rd grade | 4th grade | |||
| Vegetative stage | 97.50±0.90 | 1.00±0.87 | 0.92±0.29 | 0.33±0.14 | 0.25±0.01 | 2.50±0.90 | 1.25±0.23 |
| Reproductive stage | 93.83±0.58 | 0.42±0.14 | 0.75±0.25 | 1.50±0.50 | 3.50±0.87 | 6.17±0.58 | 5.11±0.60 |
2.2 番茄青枯病病株与健株根际基质青枯雷尔氏菌的分布特征 2.2.1 青枯雷尔氏菌的鉴定
从番茄青枯病病株与健株根际基质共分离到44株经形态鉴定为青枯雷尔氏菌的菌株,且均为强致病力菌株的形态特征,即菌落呈不规则形状或近圆形,流动性强,中间为粉红色,白边较宽,菌落表面湿润(图 1A); 进一步进行分子鉴定,43株供试菌株与阳性对照GMI1000经青枯雷尔氏菌特异检测引物pehA#6/pehA#3的PCR扩增出一条约500 bp的特异性靶带,1株供试菌株与阴性对照(不加DNA样品)没有扩增出相应条带(图 1B),表明44株分离菌株中有43株确认为青枯雷尔氏菌,占比97.73%。
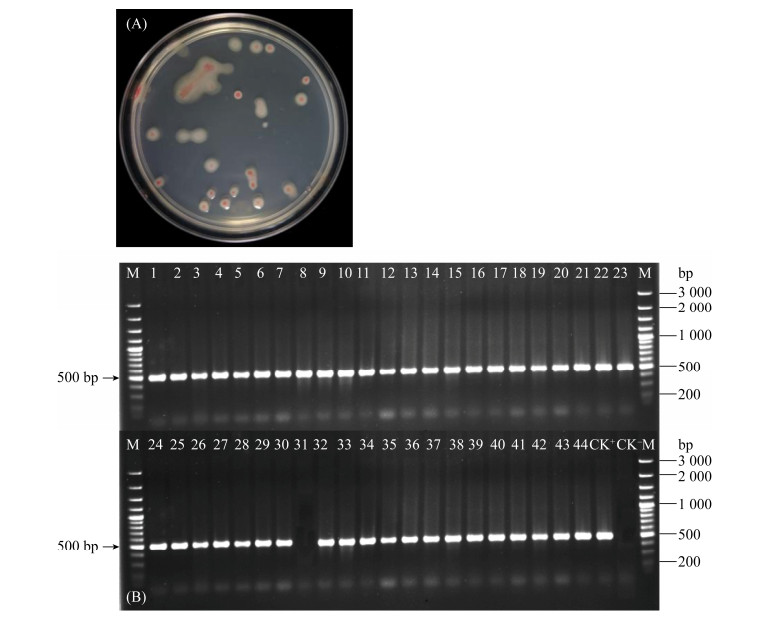
|
| 图 1 青枯雷尔氏菌的形态鉴定(A)和分子鉴定(B) Figure 1 Morphological (A) and molecular identification (B) of Ralstonia solanacearum. M: DNA marker; lane 1-44: the tested isolates; CK+: positive control GMI1000 template; CK-: negative control distilled water. |
2.2.2 青枯雷尔氏菌分布特征
青枯雷尔氏菌在不同发病级别番茄植株根际基质的分布数量不同(图 2)。不同发病级别植株根际基质均有青枯雷尔氏菌的分布,分布数量为6.67×103-7.13×106 CFU/g,发病级别2的番茄根际基质分布的青枯雷尔氏菌最多,为7.13×106 CFU/g,显著大于其他发病级别和健株的青枯雷尔氏菌分布数量(P < 0.05)。3级和4级植株根际基质青枯雷尔氏菌分布数量相当,显著低于1级和2级发病植株根际基质(P < 0.05)。健株根际基质青枯雷尔氏菌分布数量最低,为6.67×103 CFU/g,比其他发病级别植株根际基质土降低约1 000倍。表明健株根际基质青枯雷尔氏菌数量低于104 CFU/g,病株根际基质青枯雷尔氏菌数量高于106 CFU/g,且随着病情发展,1级到2级青枯雷尔氏菌数量增加,2级到3-4级数量下降。
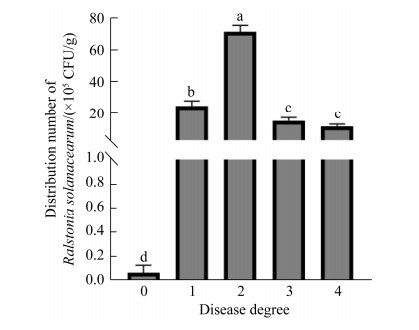
|
| 图 2 青枯病不同发病级别植株根际基质青枯雷尔氏菌的分布数量 Figure 2 Distribution number of R. solanacearum in the rhizosphere substrate of tomato plants with different disease degree. Different letters above each bar indicate significant difference at P < 0.05 level by LSD test. |
2.3 番茄青枯病病株与健株根际基质细菌群落丰富度及多样性指数差异
稀释曲线能反映样品文库测序数据量的合理性。供试6个基质样品的稀释曲线均平缓,表明当前测序深度已足够反映样本所包含微生物多样性。Chao 1指数能够体现微生物的群落丰富度,而Shannon和Simpson指数主要体现微生物的群落多样性。病株根际基质Chao 1指数(1 954.82)和Shannon (8.72)指数均显著低于健株根际(分别为2 181.67和9.36),而Simpson在病株和健株基质样本中差异不显著(表 2),表明番茄植株感染青枯病后,根际微生物丰富度(Chao 1)和多样性指数(Shannon)均降低,微生物丰富度(Chao 1)的病健比达0.98 (Chao 1病/Chao 1健),微生物多样性指数(Shannon)病健比达0.93,番茄发病。
| Samples | Chao1 | Simpson | Shannon |
| Rhizosphere substrate of diseased tomato plants | 1 954.82±96.55 b | 0.99±0.00 b | 8.72±0.14 b |
| Rhizosphere substrate of healthy tomato plants | 2 181.67±85.83 a | 0.10±0.01 a | 9.36±0.21 a |
| Data are mean±SD. Different letters in the same column indicate significant difference at P < 0.05 level by LSD test. | |||
2.4 番茄青枯病病株与健株根际基质细菌群落组成结构的差异
番茄青枯病病株与健株根际基质相对含量高的前5个细菌门均为变形菌门(Proteobacteria)、拟杆菌门(Bacteroidota)、酸杆菌门(Acidobacteriota)、放线菌门(Actinobacteriota)和疣微菌门(Verrucomicrobiota),这5个门在病株和健株根际基质的相对总含量分别为83.77%和81.86% (图 3)。变形菌门在病株根际基质的相对含量(56.89%)极显著高于其在健株根际基质的相对含量(47.64%) (P < 0.01),放线菌门和蓝细菌门(Cyanobacteria)在病株根际基质的相对含量(3.29%和0.93%)极显著低于其在健株根际基质的相对含量(5.53%和4.10%) (P < 0.01);表明青枯病的发生与根际优势微生物含量变化相关,与变形菌门含量成正比,与放线菌门和蓝细菌门含量成反比。
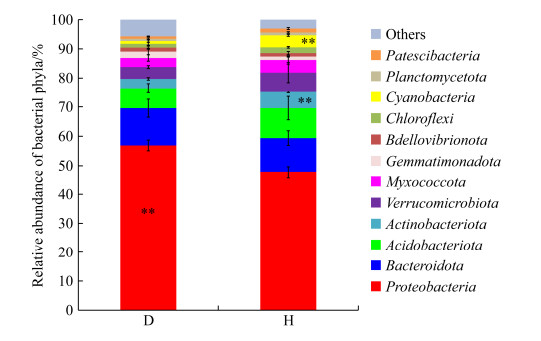
|
| 图 3 番茄青枯病病株与健株根际基质细菌门的相对丰度 Figure 3 Relative abundances of bacterial phyla in the rhizosphere substrate of bacterial wilt infected and healthy tomato plants. D: rhizosphere substrate sample from bacterial wilt infected tomato plant; H: rhizosphere substrate sample from healthy tomato plant. ** P < 0.01. |
番茄健株根际基质共检测到448个属,病株根际基质共检测到398个属,其中有392个属共同存在于病株和健株根际基质中。在共有的相对丰度最高的前20个属中,罗纳杆菌属(Rhodanobacter)在病株根际基质的相对含量显著高于健株(P < 0.05),海管菌属(Haliangium)、德沃斯氏菌属(Devosia)和慢生根瘤菌属(Bradyrhizobium)在病株根际基质的相对含量显著低于健株(P < 0.05),粒状胞菌属(Granulicella)在病株根际基质的相对含量极显著低于健株(P < 0.01) (图 4)。此外,青枯病原菌所在属Ralstonia在病株根际基质的相对含量为0.3%,极显著高于其在健株根际基质的相对含量(0.004%) (P < 0.01)。

|
| 图 4 番茄青枯病病株与健株根际基质差异显著关键属的相对丰度 Figure 4 Relative abundance of some key bacterial genera with significant difference in rhizosphere substrate from bacterial wilt infected and healthy tomato plants. D: rhizosphere substrate sample from bacterial wilt infected tomato plant; H: rhizosphere substrate sample from healthy tomato plant. * P < 0.05 and ** P < 0.01. |
土源杆菌属(Chthonobacter)、沙单胞菌属(Arenimonas)、食螯合剂菌属(Chelativorans)、粉色单胞菌属(Roseomonas)、候选属UBA6140、根瘤菌科未分类的1属uncultured Rhizobiaceae bacterium、鼎湖山杆菌属(Dinghuibacter)、金色线菌属(Chryseolinea)、特吕珀菌属(Truepera)、脂环酸芽胞杆菌属(Alicyclobacillus)为病株根际基质特有的细菌属,这10个属中6个归类于变形菌门(Proteobacteria)、2个归类于拟杆菌门(Bacteroidota)、1个归类于异常球菌门(Deinococcota)和1个归类于厚壁菌门(Firmicutes); 候选属Candidatus Captivus、候选属Candidatus Omnitrophus和未培养的厌氧氨氧化细菌属uncultured anaerobic ammoniumoxidizing bacterium为健株根际基质特有的细菌属,这3个属分别归类于变形菌门(Proteobacteria)、疣微菌门(Verrucomicrobiota)和绿弯菌门(Chloroflexi)。
2.5 番茄根际细菌群落与青枯雷尔氏菌分布的关系通过对番茄根际细菌群落与青枯雷尔氏菌分布数量进行冗余分析(redundancy analysis,RDA)可以看出(图 5),第一主成分和第二主成分对细菌群落结构变化总解释率为69.79%。罗纳杆菌属(Rhodanobacter)和雷尔氏菌属(Ralstonia)的相对含量与青枯雷尔氏菌分布数量呈较大正相关,相关系数(r2)分别为0.77和0.84,表明这2个属的细菌相对含量增加会促进青枯雷尔氏菌的生长。德沃斯氏菌属(Devosia)、甲基芽胞杆菌属(Methylobacillus)、假双头斧形菌属(Pseudolabrys)、鲍尔德氏菌属(Bauldia)和海管菌属(Haliangium)呈较大负相关,r2值分别为-0.26、-0.37、-0.71、-0.88和-0.94,表明这些属的菌相对含量增加会抑制青枯雷尔氏菌的生长。
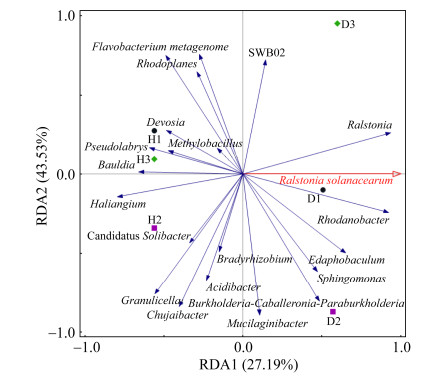
|
| 图 5 番茄根际基质细菌群落组成(属水平)与青枯雷尔氏菌分布数量的冗余分析 Figure 5 Redundancy analysis (RDA) of bacterial community composition (at the genus level) and the distribution number of R. solanacearum. |
3 讨论与结论
本研究发现,水肥菌一体化基质栽培系统中番茄仍有一些植株感染青枯病(营养生长期和生殖生长期发病率分别为2.50%和6.17%),推测可能种苗带有青枯病原菌。研究发现,番茄青枯病发病植株,不同发病等级的番茄植株根际基质土均含有一定数量的青枯雷尔氏菌,且均为强致病力菌株。健株根际基质也检测到青枯雷尔氏菌,但分布量低(6.67×103 CFU/g),其数量可能尚未达到青枯病症状出现的阈值; 葛慈斌等[17]认为,青枯雷尔氏菌浓度对青枯病病程的潜伏期有一定的影响,浓度越低潜伏期越长。2级病株根际基质土中青枯雷尔氏菌分布数量最多,其次是1级植株根际。随着病情加重(3级、4级),植株根际基质土的病原菌数量反而降低,说明番茄植株根际基质病原菌的数量和病害严重程度并非成正相关性,此结果与刘波等[18]和Zheng等[19]的研究结果一致,刘波等比较了青枯雷尔氏菌在不同寄主、不同发病状态以及不同生育期植株体内的分布,结果发现青枯雷尔氏菌在寄主植株体内呈现出明显的生态位分化的特征,随着病害严重程度的增加,青枯雷尔菌的数量下降,但同时致病力加强。
许多研究表明,植物根际土壤群落结构的alpha多样性与植株健康呈正相关[20-21],alpha多样性高的土壤比多样性低的土壤更能抵御病原菌的侵染[22-23],健株比病株根际土壤具有更高的微生物丰度和多样性[24-25]。Garbeva等[26]指出,根际土壤细菌多样性高能抑制土传病害的发生,本研究同样发现基质栽培系统的番茄健株根际基质的Chao1和Shannon多样性指数显著高于感染青枯病植株的根际基质,证实了细菌多样性与生态系统生产力之间存在的正相关关系[25]。
土传病害的发生与土壤环境,特别是微生物群落结构密切相关[27]。Raaijmakers等[12]指出,土壤微生物群落结构及组成对于番茄抵御青枯病菌的侵染有重要影响。沈宗专等[25]研究表明,与健康番茄根际土壤相比,病株番茄根际土壤变形菌门的相对丰度显著增高而拟杆菌门相对丰度显著降低。向立刚等[28]研究表明,感染青枯病烟株根际土壤较健康烟株根际土壤变形菌门增加4.47%、酸杆菌门增加1.48%、放线菌门降低6.42%,感染青枯病茎杆较健康烟株茎杆中变形菌门增加28.60%、蓝细菌门降低34.49%。本研究结果与前人研究结果相一致,感染青枯病植株的根际基质土中变形菌门相对丰度极显著高于健株,而放线菌门和蓝细菌门则极显著低于健株。变形菌门细菌是最为普遍的细菌种类[29],在碳、氮、硫循环中起重要作用[30]。本研究中感染青枯病和健康的番茄根际基质土中变形菌门均为优势细菌门,病株根际基质土变形菌门相对丰度增加可能与青枯雷尔氏菌的侵染有关[28]。放线菌门和蓝细菌门细菌含量通常代表土壤的健康状况[31],本研究证实健康的番茄根际放线菌门和蓝细菌门相对含量显著高于感染青枯病番茄植株的根际基质土。
本研究发现,与健株根际相比,病株根际罗纳杆菌属(Rhodanobacter)相对含量显著增高,而慢生根瘤菌属(Bradyrhizobium)、粒状胞菌属(Granulicella)、海管菌属(Haliangium)和德沃斯氏菌属(Devosia)含量显著降低。RDA显示,罗纳杆菌属相对含量与番茄根际青枯雷尔氏菌分布数量呈正相关,慢生根瘤菌属、粒状胞菌属、海管菌属和德沃斯氏菌属相对含量均与番茄根际青枯雷尔氏菌分布数量呈负相关。罗纳杆菌属具有氨氧化、亚硝酸氧化和反硝化功能[32],是土壤氮转化的主要参与者[33]。赖宝春等[34]研究发现患枯萎病辣椒植株根际产黄杆菌属的相对丰度比健株根际增加3.56%。根瘤菌和寄主植株产生互利共生关系,具有固氮作用,能有效促进植物生长[35]; 根瘤菌分类上目前有17个属,其中慢生根瘤菌属细菌是常见的土壤微生物,因其缓慢的生长速度而得名[36]。粒状胞菌属和嗜盐囊菌属都是土壤中有益细菌,在健株根际基质土中的相对含量高于病株[37-38]。此外,本研究中青枯病原菌所在雷尔氏菌属(Ralstonia)在病株根际基质的相对含量极显著高于健株,与向立刚等[28]、沈宗专等[25]的研究结果相吻合。
本研究只是对生殖生长期感染青枯病番茄植株与健株根际微生物群落组成及多样性进行了初步比较分析。然而,番茄根际微生物的群落结构与植株健康状况、土壤类型、根际养分和作物生长发育时期等密切相关[26, 39]。因此,在后续研究中将对不同生长期、不同发病程度及不同基质类型栽培番茄根际微生物区系展开研究,以期更深入揭示番茄青枯病害发生与微生物区系之间的关系。
| [1] | Yadessa GB, Van Bruggen AHC, Ocho FL. Effects of different soil amendments on bacterial wilt caused by Ralstonia solanacearum and on the yield of tomato. Journal of Plant Pathology, 2010, 92(2): 439-450. |
| [2] | Renaud M, Chelinho S, Alvarenga P, Mourinha C, Palma P, Sousa JP, Natal-da-Luz T. Organic wastes as soil amendments-effects assessment towards soil invertebrates. Journal of Hazardous Materials, 2017, 330: 149-156. DOI:10.1016/j.jhazmat.2017.01.052 |
| [3] |
Meng TZ, Guo DJ, Wang GF, Ma Y. Effect of combination of soil disinfestation and bioorganic fertilizer application on improving watermelon diseased soil. Soils, 2020, 52(3): 494-502.
(in Chinese) 孟天竹, 郭德杰, 王光飞, 马艳. 土壤消毒结合施用生物有机肥对西瓜病土改良效果的影响. 土壤, 2020, 52(3): 494-502. |
| [4] |
Mao LP, Zhao J, Yi ZH. Simplified cultivation of greenhouse tomato using substrates. Xiandai Horticulture, 2019, 42(17): 65-66.
(in Chinese) 毛丽萍, 赵婧, 仪泽会. 设施番茄简易基质栽培技术. 现代园艺, 2019, 42(17): 65-66. DOI:10.3969/j.issn.1006-4958.2019.17.034 |
| [5] |
Cao SN, Li JS, Gao YM, Wu SP, Liu MJ, Li J. Effects of embedding position of moisture sensors on growth characteristics of tomato in substrate culture in greenhouse. Acta Agriculturae Zhejiangensis, 2017, 29(6): 933-942.
(in Chinese) 曹少娜, 李建设, 高艳明, 吴素萍, 刘梦锦, 李娟. 水分传感器埋设位置对温室基质栽培番茄生长特性的影响. 浙江农业学报, 2017, 29(6): 933-942. DOI:10.3969/j.issn.1004-1524.2017.06.11 |
| [6] | Hayward AC. Biology and epidemiology of bacterial wilt caused by Pseudomonas solanacearum. Annual Review of Phytopathology, 1991, 29: 65-87. DOI:10.1146/annurev.py.29.090191.000433 |
| [7] | Pradhanang PM, Momol MT, Olson SM, Jones JB. Effects of plant essential oils on Ralstonia solanacearum population density and bacterial wilt incidence in tomato. Plant Disease, 2003, 87(4): 423-427. DOI:10.1094/PDIS.2003.87.4.423 |
| [8] |
Wang GP, Lin MB, Wu DH. Classical and molecular genetics of bacterial wilt resistance in tomato. Acta Horticulturae Sinica, 2004, 31(3): 403-407.
(in Chinese) 汪国平, 林明宝, 吴定华. 番茄青枯病抗性遗传研究进展. 园艺学报, 2004, 31(3): 403-407. DOI:10.3321/j.issn:0513-353X.2004.03.033 |
| [9] | Philippot L, Raaijmakers JM, Lemanceau P, Van Der Putten WH. Going back to the roots: the microbial ecology of the rhizosphere. Nature Reviews Microbiology, 2013, 11(11): 789-799. DOI:10.1038/nrmicro3109 |
| [10] | Mendes R, Garbeva P, Raaijmakers JM. The rhizosphere microbiome: significance of plant beneficial, plant pathogenic, and human pathogenic microorganisms. FEMS Microbiology Reviews, 2013, 37(5): 634-663. DOI:10.1111/1574-6976.12028 |
| [11] |
Wei Z, Shen ZZ, Yang TJ, Wang XF, Li R, Xu YC, Shen QR. From suppressive soil to rhizosphere immunity: towards an ecosystem thinking for soil-borne pathogen control. Acta Pedologica Sinica, 2021, 58(4): 814-824.
(in Chinese) 韦中, 沈宗专, 杨天杰, 王孝芳, 李荣, 徐阳春, 沈其荣. 从抑病土壤到根际免疫: 概念提出与发展思考. 土壤学报, 2021, 58(4): 814-824. |
| [12] | Raaijmakers JM, Paulitz TC, Steinberg C, Alabouvette C, Moënne-Loccoz Y. The rhizosphere: a playground and battlefield for soilborne pathogens and beneficial microorganisms. Plant and Soil, 2009, 321(1/2): 341-361. |
| [13] | Kelman A. The relationship of pathogenicity in Pseudomonas solanacearum to colony appearance on a tetrazolium medium. Phytopathology, 1954, 44: 693-695. |
| [14] | Roberts DP, Denny TP, Schell MA. Cloning of the egl gene of Pseudomonas solanacearum and analysis of its role in phytopathogenicity. Journal of Bacteriology, 1988, 170(4): 1445-1451. DOI:10.1128/jb.170.4.1445-1451.1988 |
| [15] |
Cheng BL, Che JM, Liu B. Construction of avirulent mutants of Ralstonia solanacearum by Tn5 transposon and analysis of its biological characteristics. Journal of Agricultural Biotechnology, 2011, 19(1): 26-35.
(in Chinese) 程本亮, 车建美, 刘波. 青枯雷尔氏菌Tn5转座子无致病力突变株构建及其生物学特性. 农业生物技术学报, 2011, 19(1): 26-35. DOI:10.3969/j.issn.1674-7968.2011.01.004 |
| [16] | Gillings M, Fahy P, Davies C. Restriction analysis of an amplified polygalacturonase gene fragment differentiates strains of the phytopathogenic bacterium Pseudomonas solanacearum. Letters in Applied Microbiology, 1993, 17(1): 44-48. DOI:10.1111/j.1472-765X.1993.tb01432.x |
| [17] |
Ge CB, Liu B, Zhu YJ, Lin YZ, Xiao RF. Study on the methods on identifying the virulence of Ralstonia solanacearum using tissue cultured tomatoes. Wuyi Science Journal, 2004, 20: 13-16.
(in Chinese) 葛慈斌, 刘波, 朱育菁, 林营志, 肖荣凤. 青枯雷尔氏菌致病力的番茄组培苗鉴定方法研究. 武夷科学, 2004, 20: 13-16. |
| [18] |
Liu B, Zhu YJ, Lin KM, Xiao RF, Ge CB, Lan JL, Mao NH. Study on numerical and pathogenic variations of Ralstonia solanacearum distributed within the tissue of host plants. Scientia Agricultura Sinica, 2007, 40(7): 1559-1566.
(in Chinese) 刘波, 朱育菁, 林抗美, 肖荣凤, 葛慈斌, 蓝江林, 冒乃和. 青枯雷尔氏菌在植株体内分布及其致病力的异质性研究. 中国农业科学, 2007, 40(7): 1559-1566. DOI:10.3321/j.issn:0578-1752.2007.07.034 |
| [19] | Zheng XF, Zhu YJ, Liu B, Zhou Y, Che JM, Lin NQ. Relationship between Ralstonia solanacearum diversity and severity of bacterial wilt disease in tomato fields in China. Journal of Phytopathology, 2014, 162(9): 607-616. DOI:10.1111/jph.12234 |
| [20] | Luan FG, Zhang LL, Lou YY, Wang L, Liu YN, Zhang HY. Analysis of microbial diversity and niche in rhizosphere soil of healthy and diseased cotton at the flowering stage in southern Xinjiang. Genetics and Molecular Research, 2015, 14(1): 1602-1611. DOI:10.4238/2015.March.6.7 |
| [21] | Poudel R, Jumpponen A, Schlatter DC, Paulitz TC, Gardener BBM, Kinkel LL, Garrett KA. Microbiome networks: a systems framework for identifying candidate microbial assemblages for disease management. Phytopathology, 2016, 106(10): 1083-1096. DOI:10.1094/PHYTO-02-16-0058-FI |
| [22] | Fargione JE, Tilman D. Diversity decreases invasion via both sampling and complementarity effects. Ecology Letters, 2005, 8(6): 604-611. DOI:10.1111/j.1461-0248.2005.00753.x |
| [23] | Van Elsas JD, Chiurazzi M, Mallon CA, Elhottova D, Kristufek V, Salles JF. Microbial diversity determines the invasion of soil by a bacterial pathogen. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(4): 1159-1164. DOI:10.1073/pnas.1109326109 |
| [24] | Zheng XF, Liu B, Zhu YJ, Wang JP, Zhang HF, Wang ZR. Bacterial community diversity associated with the severity of bacterial wilt disease in tomato fields in southeast China. Canadian Journal of Microbiology, 2019, 65(7): 538-549. DOI:10.1139/cjm-2018-0637 |
| [25] |
Shen ZZ, Huang Y, Cao YF, Wang DS, Liu HJ, Li R, Shen QR. Comparison of bacterial communities in bulk and rhizosphere soils of healthy and diseased tomato infected by bacterial wilt. Soils, 2021, 53(1): 5-12.
(in Chinese) 沈宗专, 黄炎, 操一凡, 王东升, 刘红军, 李荣, 沈其荣. 健康与罹患青枯病的番茄土壤细菌群落特征比较. 土壤, 2021, 53(1): 5-12. |
| [26] | Garbeva P, Van Veen JA, Van Elsas JD. Microbial diversity in soil: selection microbial populations by plant and soil type and implications for disease suppressiveness. Annual Review of Phytopathology, 2004, 42: 243-270. DOI:10.1146/annurev.phyto.42.012604.135455 |
| [27] |
Shi PY, Yang CC, Jia M, Bai YX, Liu Q, Zhu XQ, Cheng YD, Zhang JF, Wang RL, Tan XB, Yang HW, Wang G. Comparison of soil enzyme activity and bacterial community structure in tobacco-growing rhizosphere under different incidence of black shank disease. Soil and Fertilizer Sciences in China, 2020(1): 179-187.
(in Chinese) 史普酉, 杨成翠, 贾孟, 白羽祥, 刘棋, 朱宣全, 程亚东, 张金峰, 王润玲, 谭小兵, 杨焕文, 王戈. 不同黑胫病发病程度下植烟根际土壤酶活性及细菌群落结构差异比较. 中国土壤与肥料, 2020(1): 179-187. |
| [28] |
Xiang LG, Zhou H, Wang HC, Li Z, Chen QL, Yu ZH. Bacterial community structure and diversity of rhizosphere soil and stem of healthy and bacterial wilt tobacco plants. Acta Micorbiologica Sinica, 2019, 59(10): 1984-1999.
(in Chinese) 向立刚, 周浩, 汪汉成, 李震, 陈乾丽, 余知和. 健康与感染青枯病烟株根际土壤与茎秆细菌群落结构与多样性. 微生物学报, 2019, 59(10): 1984-1999. |
| [29] | Spain AM, Krumholz LR, Elshahed MS. Abundance, composition, diversity and novelty of soil Proteobacteria. The ISME Journal, 2009, 3(8): 992-1000. DOI:10.1038/ismej.2009.43 |
| [30] | Rampelotto PH, De Siqueira Ferreira A, Barboza ADM, Roesch LFW. Changes in diversity, abundance, and structure of soil bacterial communities in Brazilian savanna under different land use systems. Microbial Ecology, 2013, 66(3): 593-607. DOI:10.1007/s00248-013-0235-y |
| [31] | Bhatti AA, Haq S, Bhat RA. Actinomycetes benefaction role in soil and plant health. Microbial Pathogenesis, 2017, 111: 458-467. DOI:10.1016/j.micpath.2017.09.036 |
| [32] | Gao Y, Xie YW, Zhang Q, Wang AL, Yu YX, Yang LY. Intensified nitrate and phosphorus removal in an electrolysis-integrated horizontal subsurface-flow constructed wetland. Water Research, 2017, 108: 39-45. DOI:10.1016/j.watres.2016.10.033 |
| [33] |
Li WP, Zhang SK, Li XG, Wang F, Li GW, Hao Y, Zhang LY. Restoration of black-odorous sediment by low-dosage calcium nitrate combined with hypoxic aeration. Research of Environmental Sciences, 2020, 33(4): 940-948.
(in Chinese) 李卫平, 张少康, 李晓光, 王凡, 李国文, 郝禹, 张列宇. 低剂量硝酸钙联合低氧曝气对黑臭底泥的修复探究. 环境科学研究, 2020, 33(4): 940-948. |
| [34] |
Lai BC, Dai RQ, Wu ZQ, Li F, Lin DF, Wang JR. Bacterial diversities in rhizosphere soils at sites of healthy and Fusarium wilt infected Chili plants. Fujian Journal of Agricultural Sciences, 2019, 34(9): 1073-1080.
(in Chinese) 赖宝春, 戴瑞卿, 吴振强, 李丰, 林德峰, 王家瑞. 辣椒健康植株与患枯萎病植株根际土壤细菌群落多样性的比较研究. 福建农业学报, 2019, 34(9): 1073-1080. |
| [35] |
Zhang W, Zhang LP, Yang RL, Lü ZT. Nitrogen fixation activity of Frankia sp. strains. Journal of Hebei University: Natural Science Edition, 2006, 26(1): 21-24.
(in Chinese) 张维, 张利平, 杨润蕾, 吕志堂. 几株弗兰克氏菌的固氮活性. 河北大学学报: 自然科学版, 2006, 26(1): 21-24. DOI:10.3969/j.issn.1000-1565.2006.01.006 |
| [36] | Menna P, Barcellos FG, Hungria M. Phylogeny and taxonomy of a diverse collection of Bradyrhizobium strains based on multilocus sequence analysis of the 16S rRNA gene, ITS region and glnⅡ, recA, atpD and dnaK genes. International Journal of Systematic and Evolutionary Microbiology, 2009, 59(Pt 12): 2934-2950. |
| [37] |
Xiong DW, Jin DF, Gu BT, Huang GC, Huang XP, Liu L. Endophytic bacterial community in Gannan navel orange affected by huanglongbing. Microbiology China, 2017, 44(3): 631-638.
(in Chinese) 熊大维, 金丹凤, 顾斌涛, 黄国昌, 黄筱萍, 刘兰. 柑橘黄龙病赣南脐橙内生菌种群结构分析. 微生物学通报, 2017, 44(3): 631-638. |
| [38] | Kwak MJ, Kong HG, Choi K, Kwon SK, Song JY, Lee J, Lee PA, Choi SY, Seo M, Lee HJ, Jung EJ, Park H, Roy N, Kim H, Lee MM, Rubin EM, Lee SW, Kim JF. Rhizosphere microbiome structure alters to enable wilt resistance in tomato. Nature Biotechnology, 2018, 36(11): 1100-1109. DOI:10.1038/nbt.4232 |
| [39] |
Wu FZ, An MJ. Effects of watermelon cultivars with different resistances to Fusarium oxysporum f. sp. niveum and grafting on rhizosphere soil microorganism population and community structure. Scientia Agricultura Sinica, 2011, 44(22): 4636-4644.
(in Chinese) 吴凤芝, 安美君. 西瓜枯萎病抗性及其嫁接对根际土壤微生物数量及群落结构的影响. 中国农业科学, 2011, 44(22): 4636-4644. DOI:10.3864/j.issn.0578-1752.2011.22.010 |
 2022, Vol. 62
2022, Vol. 62




