中国科学院微生物研究所,中国微生物学会
文章信息
- 张庆芳, 王严, 郭星, 杨超, 迟乃玉, 姜南. 2022
- ZHANG Qingfang, WANG Yan, GUO Xing, YANG Chao, CHI Naiyu, JIANG Nan.
- Akkermansia与高原牛肺水肿病相关性研究
- Correlation between Akkermania and pulmonary edema of plateau cattle
- 微生物学报, 62(11): 4367-4384
- Acta Microbiologica Sinica, 62(11): 4367-4384
-
文章历史
- 收稿日期:2022-01-27
- 修回日期:2022-04-25
- 网络出版日期:2022-05-30
2. 辽宁省海洋微生物工程技术研究中心, 辽宁 大连 116622;
3. 上海美吉生物医药科技有限公司, 上海 201318
2. Liaoning Marine Microbial Engineering and Technology Center, Dalian 116622, Liaoning, China;
3. Shanghai Majorbio Bio-Pharm Technology Co. Ltd., Shanghai 201318, China
Akkermansia属疣微菌门(Verrucomicrobia)的黏液蛋白降解菌[1‒2]。其作为一种严格厌氧,能够耐受低氧浓度的革兰氏阴性菌,居住在人类肠道黏液层,占健康个体微生物群落的0.5%‒5.0%[3],于2004年才被发现[4]。Akkermansia不仅在各种临床情况下具生理益处,而且是表征人类生理的重要生物标志物。它的丰度与几种疾病状态呈负相关,如在糖尿病[5‒6]、肝脏疾病[5‒6]、心血管疾病[5‒6]、炎症性肠病[5‒6]、阑尾炎[5‒6]、渐冻症[7]、自闭症[8]、高血压[8]等疾病中都发现其丰度降低。Akkermansia将粘蛋白作为碳和氮元素的唯一来源,其可能提供对致病性黏液降解菌的竞争性排斥,并可能具有门控和信号传递功能[9]。通过将粘蛋白转化为有益的副产物来维持宿主肠道微生物的平衡,同时黏蛋白降解物可通过肿瘤坏死因子α (TNF-a)、干扰素γ (INF-c)、白介素10 (IL-10)和IL-4等信号调节宿主免疫系统[10]。尽管Akkermansia利用黏液蛋白作为营养来源,但适量的Akkermansia实际上与人类和动物的黏液厚度和肠道屏障完整性呈正相关,而且能通过降解黏液蛋白制造短链脂肪酸来刺激黏液周转率[11],以此来增强肠道上皮细胞的完整性和黏液层的厚度,从而增加肠道屏障功能[10, 12]。只有当肠道内Akkermansia大量生长,丰度显著升高时,高丰度嗜粘蛋白Akkermansia大量降解黏蛋白,导致肠道通透性增加,黏液保护功能受损、细菌粘附性和炎症性微生物群增加[13‒14],即高丰度的艾克曼菌可能存在潜在的致病性。
Akkermansia是一种极具潜力的益生菌,在预防与治疗肥胖和代谢紊乱疾病以及增加癌症免疫治疗效果方面具有广阔的应用前景,对未来的研究和开发具有重要意义[1],近年来逐步成为肠道微生态和益生菌研究热点[11]。由于Akkermansia生长特性,其不易分离培养[10];因此寻找Akkermansia作为优势菌群的资源非常重要。
反刍动物牛与单胃动物不同,有瘤胃、网胃、瓣胃和皱胃4个胃室,瘤胃微生物中细菌的数量和种类最多,瘤胃液中细菌数量为1010–1011 CFU/mL[15]。牛的肠道拥有一个复杂的生态系统,该系统主要由微生物组成,在牛肠道中有大量的共生微生物,肠道菌群的数量远远高于牛自身的体细胞数,粪便中细菌数量超过1011 CFU/g,并且种类繁多[16]。早在20世纪70年代,西藏自治区就成为我国五大牧区之一,西藏地区2/3区域为草原,总面积0.8亿hm2左右,大部分分布在海拔4 500 m以上的地区[17]。西藏拉萨海拔3 594 m,地处高原,氧浓度为20%左右[18]。由于高原极端的条件(低氧、低温、高海拔),引进牛易患肺水肿病,而肺水肿是由于肺通气与换气功能严重障碍所导致,在临床上表现为极度的呼吸困难,更进一步造成肠胃缺氧状态,更有利严格厌氧的Akkermansia的生长。
基于高通量测序优势[19‒21],本研究通过Illumina MiSeq高通量测序技术分析高原健康牛与高原病牛肠道菌群组成及丰度的差异,在寻找具益生菌特性的Akkermansia资源的同时,也从微生物学角度分析其与高原牛肺水肿病的关系。
1 材料与方法 1.1 材料 1.1.1 实验动物试验动物为辽宁沈阳辉山娟姗奶牛场(N42°17′14.81″,E123°07′40.62″)的健康娟姗牛(syjs,CK)以及西藏拉萨曲水县净土奶牛养殖有限公司(N29°38′44.53″,E91°15′49.30″)的拉萨本地黄牛(xzh)和娟姗牛[引入拉萨半年的健康娟姗牛(xzjs)和引进入拉萨半年患肺水肿病的娟姗牛(xzjspe)]。不同类型各3头,均为3.5年龄牛,沈阳地区的娟姗牛作为原产地奶牛,拉萨地区娟姗牛为从沈阳地区引进品种至少生活半年,与西藏黄牛为同一养殖地点。
1.1.2 饲养管理沈阳地区奶牛的试验日粮与拉萨地区奶牛的试验日粮相同,表 1为精饲料组成及营养水平,表 2为玉米稻秆青贮饲料的化学成分。奶牛精饲料量按奶牛体重的1.8%饲喂,青贮饲料不限量,每天饲喂2次(7:30和17:30)。且不同类别的牛分开饲养,同组牛单独饲养。
| Feed | Proportion/% | Nutritional levels | Content |
| Maize | 47.6 | Dry matter DM/% | 89.56 |
| Soybean meal | 14.0 | Crude protein CP/% | 19.35 |
| Cottonseed meal | 6.0 | NDF/% | 20.27 |
| Bran | 15.0 | ADF/% | 12.38 |
| Corn lees protein feed | 12.0 | Ca/% | 0.83 |
| Ca(HCO3)2 | 1.5 | P/% | 1.12 |
| NaHCO3 | 0.5 | Net lactation energy/(MJ/kg) | 6.12 |
| Powdered food | 0.8 | ||
| Salt | 1.2 | ||
| Premixes | 1.4 | ||
| Total | 100.0 | ||
| Premixes can be supplied with: vitamin A 2 900 IU, vitamin D 1 100 IU, vitamin E 30 IU, niacin 3 mg, I 0.25 mg, Zn 20 mg, Cu 6 mg, Fe 20 mg, Mn 30 mg, Co 0.1 mg, Se 0.6 mg/kg of full price feed. | |||
| Measurement items | Test group |
| Dry matter/(g/kg FW) | 227.03 |
| pH value | 4.15 |
| Lactic acid/(g/kg DM) | 35.87 |
| Acetic acid/(g/kg DM) | 30.83 |
| Lactic/acetic acid | 1.17 |
| Propionic acid/(g/kg DM) | 0.86 |
| Total volatile fatty acids/(g/kg DM) | 31.69 |
| Crude protein/(g/kg DM) | 45.18 |
| Ammoniacal nitrogen/total nitrogen/(g/kg TN) | 299.78 |
| Water-soluble carbohydrates/(g/kg DM) | 8.69 |
| Neutral detergent fibre/(g/kg DM) | 666.22 |
| Acid detergent fibre/(g/kg DM) | 371.79 |
| Crude ash/(g/kg DM) | 66.97 |
| Total energy/(MJ/kg DM) | 17.06 |
1.1.3 样品采集
粪便微生物与胃肠道微生物关系密切,消化代谢系统的终产物为粪便,再加之样品易于采集,因此研究胃肠道微生物,选择粪便微生物表征其特点。本研究样品的采集时间在2017年12月,采集地点为辽宁沈阳辉山娟姗奶牛场(N42°17′14.81″,E123°07′40.62″),当地海拔为68 m;西藏拉萨曲水县净土奶牛养殖有限公司(N29°38′44.53″,E91°15′49.30″),当地海拔为3 594 m。分别采集syjs (CK)、xzjs、xzh和xzjspe在1 d中所有排出的新鲜粪便进行混合,装入标记好的离心管后立刻盖严,做好标记和记录,立即投入干冰中冻存运输,及时带回实验室于‒80 ℃冰箱中保存。
1.2 主要仪器设备和试剂 1.2.1 主要仪器设备主要仪器设备见表 3。
| Name of instrumentation | Model | Manufacturers |
| ‒80 ℃ Laboratory Refrigerator (ultra low temperature refrigerator) | ULUF-86 | Arctiko |
| Electronic scales | BSA224S-CW | Sartorius |
| Ultra-frozen centrifuges | JE-80K | Gilson |
| Constant temperature water baths | HH.S11-2-S | Prius |
| Horizontal electrophoresis | Wide Format | Scie-Plas |
| Pipette guns | SL-2XLS+ | Ruining |
| Vortex oscillators | XH-D | Jerean |
| Gel imager | SmartGelTM 6000 | Sage |
| Gradient PCR instrument | Veriti 96-Well Thermal Cycler | Thermofisher |
| Ultra clean bench | SCV-4A1 | ESCO |
1.2.2 主要试剂
溶菌酶、氯化钠、乙二胺四乙酸(EDTA)、蛋白酶K、十二烷基硫酸钠(SDS)、三羟甲基氨基甲烷-盐酸(Tris-HCl)缓冲液、磷酸盐缓冲液、苯酚、氯仿、异戊醇、异丙醇、乙醇、TE缓冲液、粪便DNA试剂盒、Axyprep DNA凝胶回收试剂盒等。
1.3 肠道细菌基因组DNA提取细菌基因组DNA提取按照Lamontagne等方法进行[22]。
1.4 16S rRNA基因扩增与文库构建其中细菌多样性研究主要是基于16S rDNA (V3–V4)区。以提取的细菌基因组DNA为模板,合成细菌V3–V4区带有barcode的16S rDNA基因,特异性引物:515F (5′-GTGCCAGC MGCCGCGGTA-3′)和806R (5′-GGACTACHVG GGTWTCTAAT-3′)。进行PCR扩增并对其产物进行纯化、定量和均一化形成测序文库。
PCR反应体系(50 μL):细菌DNA (5 ng/μL) 5 μL,上游引物(1 μmol/L) 10 μL,下游引物(1 μmol/L) 10 μL,2×KAPA HiFi HotStart ReadyMixTotal 25 μL;扩增程序如下:96 ℃预变性5 min;90 ℃变性45 s,54 ℃退火32 s,72 ℃延伸1 min,25个循环,72 ℃延伸10 min。用琼脂糖凝胶电泳检测PCR产物,将检测合格的样品送至华大基因公司进行16S rRNA高通量测序。
1.5 16S rRNA基因序列处理分析下机后将FASTQ数据利用重叠关系,将双末端测序reads组装成一条序列得到Tags,拼接条件如下:最小匹配长度15 bp;重叠区域允许错配率为0.1;去除没有重叠关系的reads。
得到的拼接序列原始数据(raw tags);使用Trimmomatic v0.33软件,对拼接得到的raw tags进行过滤,得到高质量的tags数据(clean tags);使用UCHIME软件,对质检序列进行反向互补,筛选嵌合序列,得到有效数据(effective tags)[22]。具体步骤如下:采取按窗口去低质量的方法,设置25 bp为窗口长度,如果窗口平均质量值低于20,从窗口开始截除read末端序列,移除最终read长度低于原始read长度75%的reads;去除接头污染reads、含N的reads和低复杂度reads。
使用QIIME Ⅱ中的UCLUST[21]对tags在97%的相似度水平下进行聚类、获得操作分类单元(operational taxonomic unit,OTU),并通过Silva (细菌)分类学数据库比对,进行OTU分类学注释;使用Mothur (versionv.1.30)软件[23],分析样品α多样性指数;利用QIIME Ⅱ生成各分类水平的物种丰度表,再利用R语言绘制成样品各分类水平下的群落结构图及组间物种差异分析图;使用PICRUSt软件对微生物的功能基因进行预测与分析。
数据采用Microsoft Excel软件数据分析进行方差分析。
2 结果与分析 2.1 16S rRNA高通量测序结果16S rRNA高通量测序结果,见表 4、表 5。syjs (CK)和xzjs样品共得到262 115条raw tags,质检处理后得到249 719条tags,平均每个样品41 620条。聚类OTU的丰度初步代表样品的物种丰度。样品中每个样品OTU统计结果见表 4。选取OTUs进行物种注释,样品物种注释见表 5。
| Sample | OTU |
| syjs1 | 560 |
| syjs2 | 455 |
| syjs3 | 590 |
| xzjs1 | 784 |
| xzjs2 | 821 |
| xzjs3 | 782 |
| xzh1 | 836 |
| xzh2 | 837 |
| xzh3 | 864 |
| xzjspe1 | 653 |
| xzjspe2 | 692 |
| xzjspe3 | 712 |
| Item No. | Phylum | Class | Order | Family | Genus |
| syjs | 13 | 24 | 30 | 52 | 93 |
| xzjs | 15 | 29 | 37 | 61 | 103 |
| xzh | 15 | 30 | 41 | 65 | 114 |
| xzjspe | 16 | 30 | 37 | 63 | 109 |
xzjs和xzh样品共得到260 505条raw tags,质检处理后得到250 047条tags,平均每个样品41 675条。样品中每个样品OTU统计结果见表 4。选取OTUs进行物种注释,样品物种注释见表 5。
xzjs和xzjspe样品共得到261 202条raw tags,质检处理后得到250 667条tags,平均每个样品41 778条。样品中每个样品OTU统计结果见表 4。选取OTUs进行物种注释,样品物种注释见表 5。
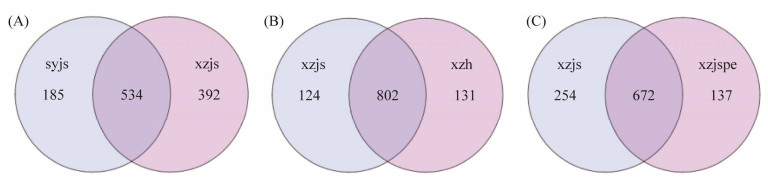
2.2 基于OTU的Venn图分析OTU的Venn图分析见图 1,OTU显示在syjs (CK)与xzjs个体的分布,2组样品共获得1 111个OTUs,其中syjs (CK)与xzjs个体间存在共有的核心OTUs为534个,其中xzjs独有的OTUs为392个,syjs独有的OTUs为185个;OTU显示在xzjs与xzh个体的分布,2组样品共获得1 057个OTUs,其中xzjs与xzh个体间存在共有的核心OTUs为802个,其中xzjs独有的OTUs为124个,xzh独有的OTUs为131个;OTU显示在xzjs与xzjspe个体的分布,2组样品共获得1 063个OTUs,其中xzjs与xzjspe个体间存在共有的核心OTUs为672个,其中xzjs独有的OTUs为254个,xzjspe独有的OTUs为137个。
|
| 图 1 syjs、xzjs、xzh、xzjspe样本中OTU的Venn图 Figure 1 Venn diagram of OTU in syjs, xzjs, xzh and xzjspe samples. A: Venn diagram of OTUs in the syjs and xzjs samples; B: Venn diagram of OTUs in the xzjs and xzh samples; C: Venn diagram of OTUs in the xzjs and xzjspe samples. |
2.3 α多样性分析
在OTU水平,syjs (CK) 3个样品的平均丰富度指数Chao1指数、Ace指数分别为594、589,α多样性指数Shannon指数、Simpson指数分别为4.38、0.039 5,Goods coverage均在99%以上;xzjs 3个样品的平均丰富度指数Chao1指数、Ace指数分别为855、858,α多样性指数Shannon指数、Simpson指数分别为5.28、0.013 8;xzh 3个样品的平均丰富度指数Chao1指数、Ace指数分别为916、916,α多样性指数Shannon指数、Simpson指数分别为5.14、0.016 1;Goods coverage均在99%以上;xzjspe 3个样品的平均丰富度指数Chao1指数、Ace指数分别为774、776,α多样性指数Shannon指数、Simpson指数分别为4.52、0.034 0。
α多样性指数表明,xzjs肠道菌群的丰富度指数Chao1指数和Ace指数均显著高于syjs (CK)、xzjspe (P < 0.05),且多样性指数Shannon指数显著高于syjs (CK)、xzjspe (P < 0.05);xzh肠道菌群的丰富度指数Chao1指数和Ace指数均显著高于xzjs (P < 0.05),多样性指数Shannon指数和Simpson指数均无显著性差异。
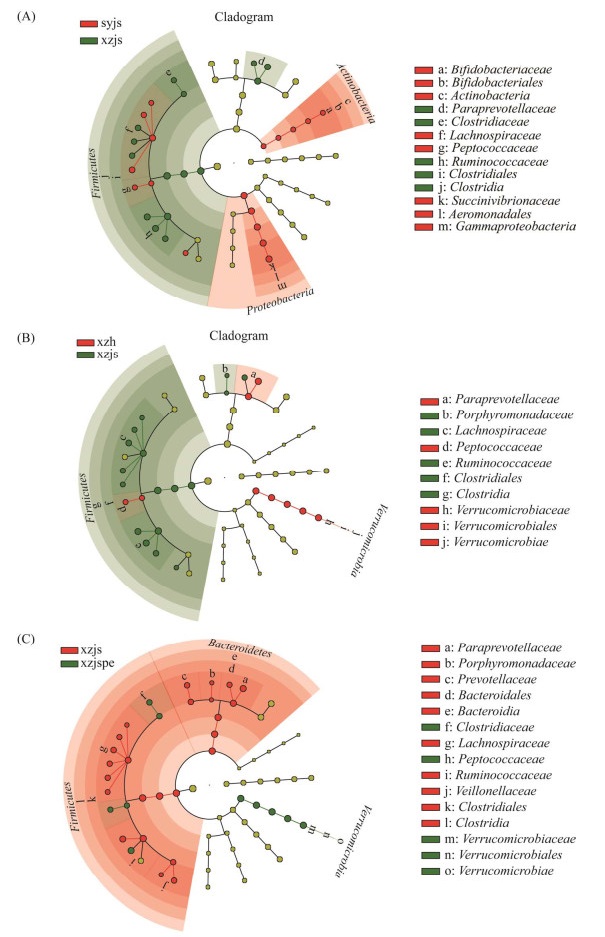
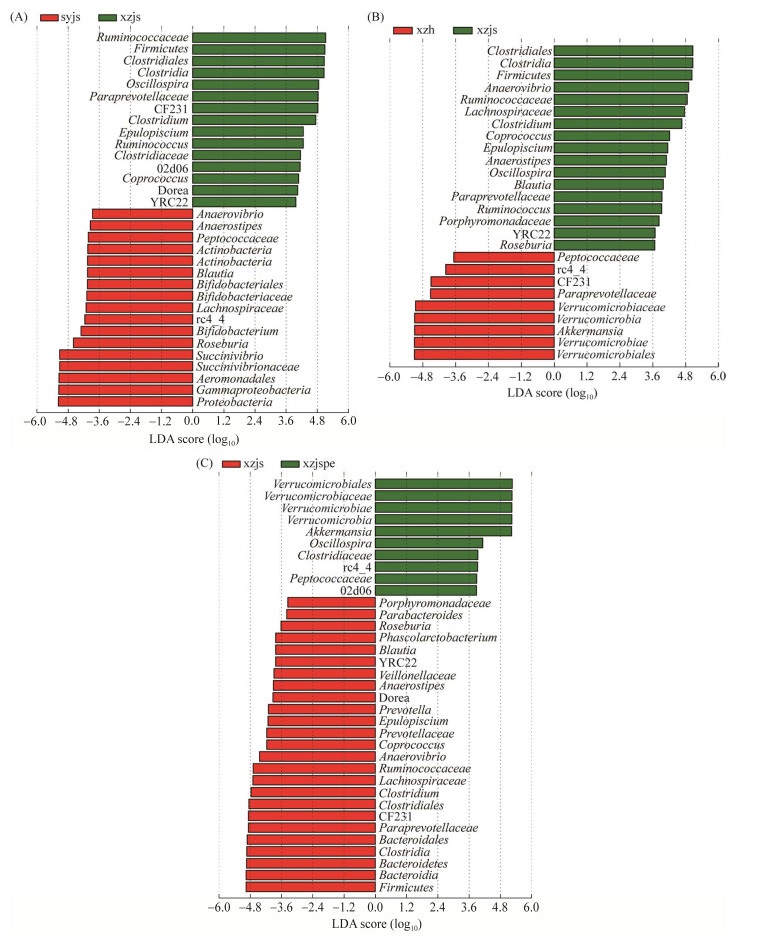
2.4 菌群组成差异各组肠道菌群LEFSe分析环形树状图和LEFSe分析柱状图如图 2和图 3所示,在xzjs和syjs (CK) 2组样品比较中,厚壁菌门(Firmicutes)、梭菌纲(Clostridia)、梭菌目(Clostridiales)、瘤胃菌科(Ruminococcaceae)在xzjs的胃肠道中的含量显著高于syjs (CK) (P < 0.05);变形菌门(Proteobacteria)、γ-变形菌纲(Gammaproteobacteria)、气单胞菌目(Aeromonadales)、琥珀酸弧菌科(Succinivibrionaceae)、琥珀酸弧菌属(Succinivibrio)在xzjs胃肠道中的含量显著低于syjs (CK) (P < 0.05)。在xzh和xzjs 2组样品比较中,疣微菌门(Verrucomicrobia)中的艾克曼菌属(Akkermansia)在xzh胃肠道中的含量显著高于xzjs (P < 0.05);厚壁菌门(Firmicutes)、梭菌纲(Clostridia)、梭菌目(Clostridiales)、瘤胃菌科(Ruminococcaceae)、厌氧弧菌属(Anaerovibrio)在xzh胃肠道中的含量显著低于xzjs (P < 0.05)。在xzjspe和xzjs 2组样品比较中,疣微菌门(Verrucomicrobia)中的艾克曼菌属(Akkermansia)在xzjspe肠道中的含量显著高于xzjs (P < 0.05);厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、梭菌纲(Clostridia)、拟杆菌纲(Bacteroidia)、梭菌目(Clostridiales)、CF231菌属、拟杆菌目(Bacteroidales)、普雷沃氏菌科(Paraprevotellaceae)在xzjspe胃肠道中的含量显著低于xzjs (P < 0.05)。
|
| 图 2 syjs、xzjs、xzh、xzjspe样本肠道菌群LEfSe分析环形树状图 Figure 2 A circular dendrogram of the LEfSe analysis of the intestinal microbiota of syjs, xzjs, xzh and xzjspe samples. A: a circular dendrogram of the LEfSe analysis of the intestinal microbiota in syjs and xzjs samples; B: a circular dendrogram of the LEfSe analysis of the intestinal microbiota in xzjs and xzh samples; C: a circular dendrogram of the LEfSe analysis of the intestinal microbiota in xzjs and xzjspe samples. |
|
| 图 3 syjs、xzjs、xzh、xzjspe样本肠道菌群LEfSe分析柱状图 Figure 3 LEfSe analysis histogram of the intestinal microbiota in syjs, xzjs, xzh and xzjspe samples. A: LEfSe analysis histogram of the intestinal microbiota in syjs and xzjs samples; B: LEfSe analysis histogram of the intestinal microbiota in xzjs and xzh samples; C: LEfSe analysis histogram of the intestinal microbiota in xzjs and xzjspe samples. |
通过肠道菌群LEFSe分析可知, 疣微菌门(Verrucomicrobia)中的艾克曼菌属(Akkermansia)在xzh胃肠道中的含量显著高于xzjs (P < 0.05),在xzjspe胃肠道中的含量显著高于xzjs (P < 0.05)。
2.5 菌群结构组成分析 2.5.1 门水平菌群结构组成分析由图 4可知,syjs (CK)肠道菌群优势菌门为厚壁菌门(Firmicutes) (48.40%)、拟杆菌门(Bacteroidetes) (33.12%)、变形菌门(Proteobacteria) (14.53%)、螺旋体门(Spirochaetes) (1.51%)和柔壁菌门(Tenericutes) (1.12%),占相对丰度的98%以上;xzjs肠道菌群优势菌门为厚壁菌门(Firmicutes) (56.82%)、拟杆菌门(Bacteroidetes) (37.55%)、柔壁菌门(Tenericutes) (1.21%)和螺旋体门(Spirochaetes) (1.04%),占相对丰度的96%以上;xzh肠道菌群优势菌门为拟杆菌门(Bacteroidete) (45.12%)、厚壁菌门(Firmicutes) (42.60%)、疣微菌门(Verrucomic) (7.12%)、柔壁菌门(Tenericutes) (1.68%)、变形菌门(Proteobacteria) (1.20%),占相对丰度的97%以上;xzjspe肠道菌群优势菌门为厚壁菌门(Firmicutes) (56.67%)、拟杆菌门(Bacteroidetes) (25.10%)、疣微菌门(Verrucomicrobia) (12.09%)、柔壁菌门(Tenericutes) (1.35%)、浮霉菌门(Planctomycetes) (1.31%)和广古菌门(Euryarchaeota) (1.22%),占相对丰度的97% 以上。
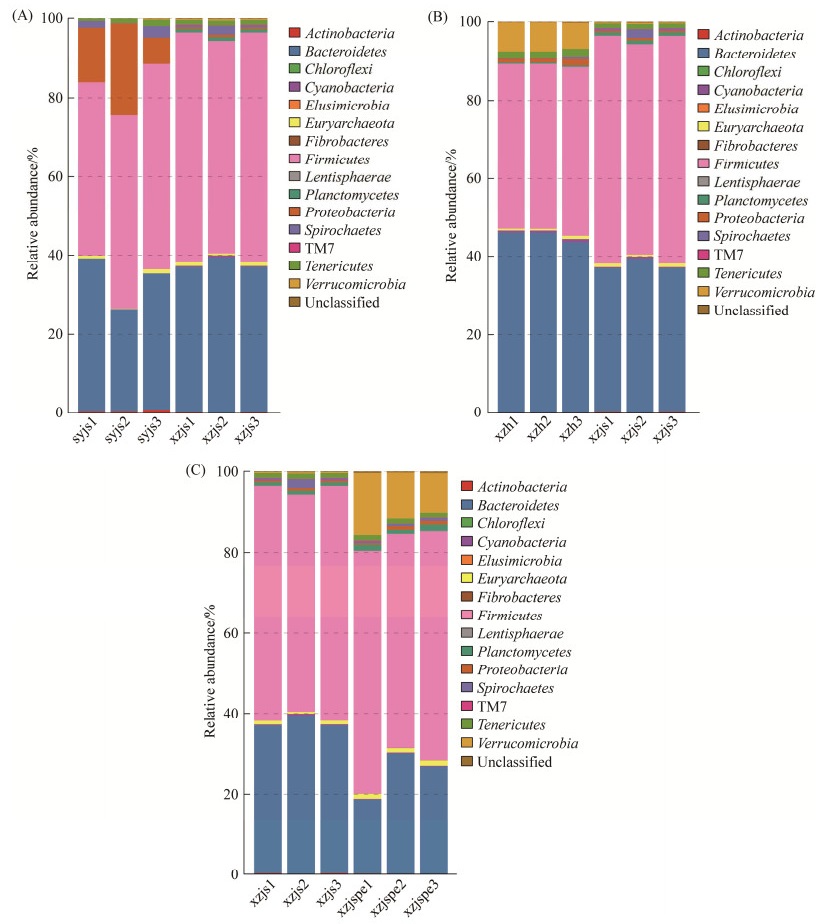
|
| 图 4 基于门水平syjs、xzjs、xzh、xzjspe样本肠道菌群结构分析 Figure 4 Structural analysis of intestinal microbiota based on phylum level syjs, xzjs, xzh and xzjspe samples. A: structural analysis of intestinal microbiota based on phylum level syjs vs. xzjs samples; B: structural analysis of intestinal microbiota based on phylum level xzjs vs. xzh samples; C: structural analysis of intestinal microbiota based on phylum level xzjs vs. xzjspe samples. |
xzjs肠道菌群在门水平上较syjs (CK)均无显著性差异;xzh与xzjs肠道菌群相比疣微菌门(Verrucomicrobia)丰度极显著升高(P < 0.01),拟杆菌门(Bacteroidetes)丰度显著升高(P < 0.05),厚壁菌门(Firmicutes)、浮霉菌门(Planctomycetes)丰度极显著降低(P < 0.01);xzjspe与xzjs肠道菌群相比广古菌门(Euryarchaeota)和疣微菌门(Verrucomicrobia)丰度显著升高(P < 0.05),拟杆菌门(Bacteroidetes)丰度显著降低(P < 0.05)。
2.5.2 属水平菌群结构组成分析由图 5可知,syjs (CK)胃肠道菌群丰度大于1%的菌属为琥珀酸弧菌属(Succinivibrio) (14.01%)、普雷沃菌属(Prevotella) (9.31%)、5-7N15菌属(4.77%)、罗氏菌属(Roseburia) (4.53%)、CF231菌属(3.76%)、梭菌属(Clostridium) (2.75%)、粪球菌属(Coprococcus) (1.83%)、考拉杆菌属(Phascolarctobacterium) (1.61%)、密螺旋体属(Treponema) (1.51%)、布劳特氏菌属(Blautia) (1.24%)、颤螺菌属(Oscillospira) (1.16%),除此之外还有艾克曼菌属(Akkermansia) (0.07%);xzjs肠道菌群丰度大于1%的菌属为CF231菌属(5.19%)、颤螺菌属(Oscillospira) (4.06%)、梭菌属(Clostridium) (4.05%)、5-7N15菌属(3.69%)、粪球菌属(Coprococcus) (1.36%)、瘤胃菌属(Ruminococcus) (1.19%)、普雷沃菌属(Prevotella) (1.07%)、密螺旋体属(Treponema) (1.01%),除此之外还有艾克曼菌属(Akkermansia) (0.09%);xzh肠道菌群丰度大于1%的菌属为CF231菌属(6.71%)、艾克曼菌属(Akkermansia) (6.62%)、颤螺菌属(Oscillospira) (3.28%)、5-7N15菌属(2.90%)、梭菌属(Clostridium) (1.59%);xzjspe肠道菌群丰度大于1%的菌属为Akkermansia (11.85%)、颤螺菌属(Oscillospira) (6.32%)、5-7N15菌属(5.06%)、CF231菌属(1.96%)、瘤胃菌属(Ruminococcus) (1.34%)、02d06菌属(1.09%)、梭菌属(Clostridium) (1.07%)。

|
| 图 5 基于属水平syjs、xzjs、xzh、xzjspe样本肠道菌群结构分析 Figure 5 Structural analysis of intestinal microbiota based on genus level syjs, xzjs, xzh and xzjspe samples. A: structural analysis of intestinal microbiota based on genus level syjs vs. xzjs samples; B: structural analysis of intestinal microbiota based on genus level xzjs vs. xzh samples; C: structural analysis of intestinal microbiota based on genus level xzjs vs. xzjspe samples. |
xzjs与syjs (CK)胃肠道菌群相比,罗氏菌属(Roseburia)丰度极显著降低(P < 0.01),考拉杆菌属(Phascolarctobacterium)、布劳特氏菌属(Blautia)丰度显著降低(P < 0.05),颤螺菌属(Oscillospira)丰度极显著升高(P < 0.01);xzh与xzjs肠道菌群相比,艾克曼菌属(Akkermansia)丰度极显著升高(P < 0.01),梭菌属(Clostridium)丰度极显著降低(P < 0.01),布劳特氏菌属(Blautia)、粪球菌属(Coprococcus)和瘤胃菌属(Ruminococcus)丰度显著降低(P < 0.05);xzjspe与xzjs肠道菌群相比,02d06菌属丰度显著升高(P < 0.05),艾克曼菌属(Akkermansia)、颤螺菌属(Oscillospira)丰度显著升高(P < 0.05),梭菌属(Clostridium)丰度极显著降低(P < 0.01),CF231菌属、粪球菌属(Coprococcus)丰度显著降低(P < 0.05)。
2.6 基于16S rRNA基因的菌株功能预测和分析基于16S rRNA基因的菌株功能预测和分析如图 6,使用PICRUSt软件通过比对16S rRNA基因测序数据获得的物种组成信息、代谢途径的差异分析,推测样品中的功能基因组成及在代谢途径上的差异和变化。如图 6所示,xzh和xzjs功能差异性显著所注释的类别达到7种;xzjspe和xzjs功能差异性显著所注释的类别达到10余种;xzjs和syjs (CK)功能差异性显著所注释的类别高达76种。
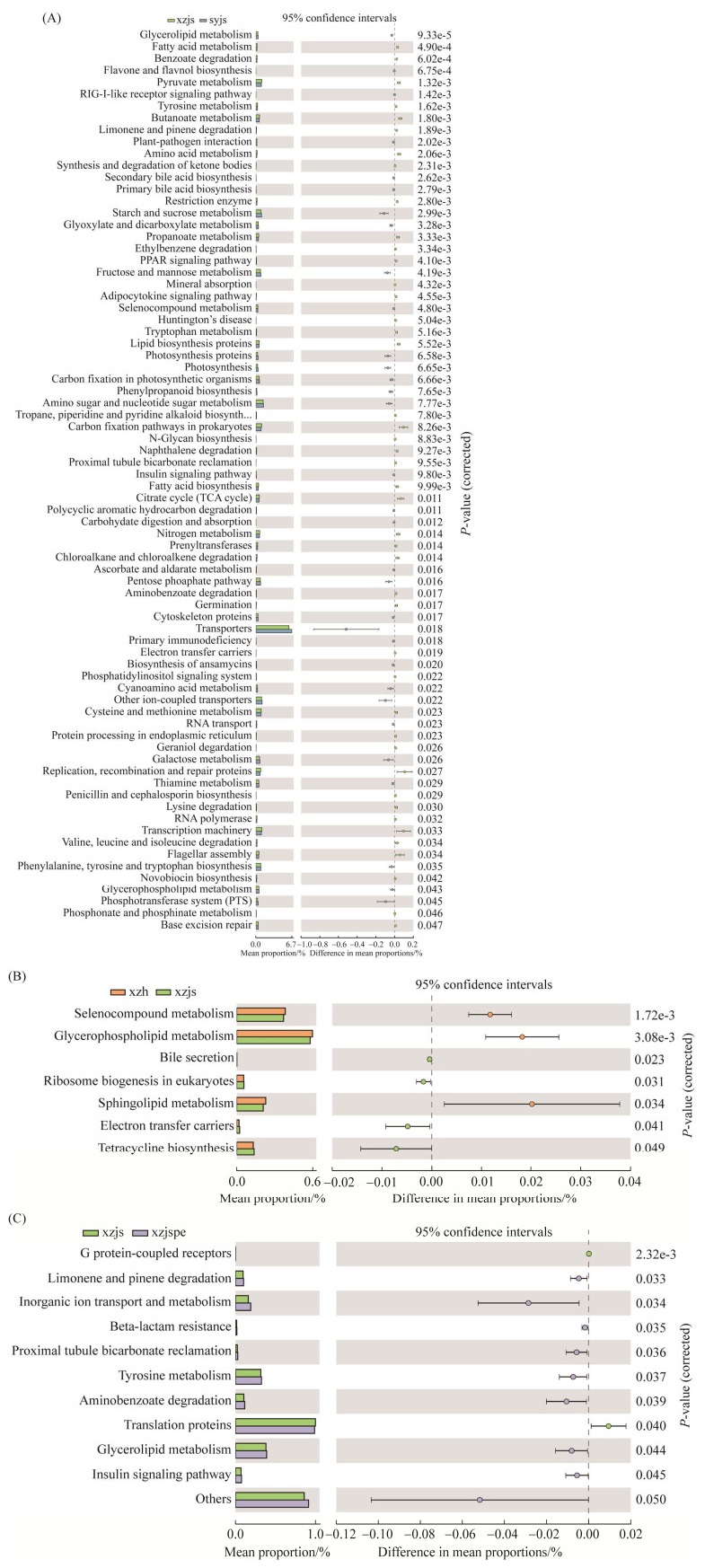
|
| 图 6 syjs、xzjs、xzh、xzjspe样本肠道菌群PICRUSt功能预测分析图 Figure 6 Predictive analysis graph of PICRUSt function of intestinal microbiota in syjs, xzjs, xzh and xzjspe samples. A: predictive analysis graph of PICRUSt function of intestinal microbiota in syjs and xzjs samples; B: predictive analysis graph of PICRUSt function of intestinal microbiota in xzjs and xzh samples; C: predictive analysis graph of PICRUSt function of intestinal microbiota in xzjs and xzjspe samples. |
3 讨论与结论
Akkermansia存在于成人、婴儿和动物肠道中[9]。Akkermansia作为肠道中的益生菌,近年来逐步引起研究人员的关注,成为肠道微生态和益生菌研究热点[11]。Routy等发表于《科学》杂志上的一篇研究论文,证明Akkermansia能显著增加癌症免疫治疗效果[23];且Akkermansia在预防和治疗肥胖[24]、自闭症[8]、渐冻症[7]和代谢紊乱疾病等方面具有广阔的应用前景,对未来的研究和开发具有重要意义。
本研究结果表明,在属水平上,沈阳地区健康娟姗牛(对照)肠道菌群中Akkermansia占比为0.07%,占比相对较低;拉萨本地黄牛肠道菌群中Akkermansia占比为6.62%,丰度极显著高于引进入拉萨半年的健康娟姗牛Akkermansia (0.09%) (P < 0.01);引进入拉萨半年患肺水肿娟姗牛肠道菌群中Akkermansia占比为11.85%,且成为第一优势菌属。
黏蛋白在动物的胃肠道表面有大量分布,Akkermansia作为肠道中益生菌之一,可以利用宿主分泌的粘蛋白,通过竞争性排斥的方式在肠道内定居并保护肠道免受病原体的侵害[25]。在本研究中Akkermansia在拉萨本地黄牛中的丰度较高,该菌属的抗炎、增强肠道屏障等益生功能使得拉萨本地黄牛抗病能力更优于引进入拉萨半年的健康娟姗牛。
本研究结果说明,Akkermansia大量存在于牛的胃肠道中与该菌生长特性、牛肠胃结构、高原缺氧环境等综合因素有关。从本研究中可以看出高原环境可导致刚引进半年的娟姗牛胃肠道菌群紊乱[26],免疫力降低,使其患病风险升高。同时本研究16S rRNA功能基因预测分析结果(图 6)显示,高原环境对引进半年的娟姗牛的生理代谢影响是最显著的。
厚壁菌门(Firmicutes)和拟杆菌门(Bacteroidetes)的比值被认为是肠道菌群组成的重要指标[27]。拟杆菌门具有降解碳水化合物以及蛋白质的功能,并且能够补充宿主基因组,参与降解具有抗性的纤维素、果胶和木聚糖等食物[28]。Van Der Waaij等[29]用抗生素治疗小鼠消化道定殖抗性研究表明,拟杆菌门的丰度还能反映肠道微生物的定殖抗力,在xzh (拉萨本地黄牛)肠道菌群中厚壁菌门(Firmicutes)和拟杆菌门(Bacteroidetes)的比值仅为0.94,syjs (沈阳健康娟姗牛)、xzjs (引进入拉萨半年的健康娟姗牛)、xzjspe (引进入拉萨半年患肺水肿病的牛)中二者的比值分别为1.46、1.51、2.26。4种牛中二者比值大小排序为xzh > syjs > xzjs > xzjspe;4种牛肠胃中拟杆菌门的丰度大小排序为xzh (45.12%) > xzjs (37.55%) > syjs (33.12%) > szjspe (25.10%)。说明高原牛的肠道菌群定殖能力及稳定性的强弱顺序为xzh > xzjs > xzjspe,这可能是拉萨本地黄牛具有抗病力强、适应性好[30]的原因之一,也是引进入拉萨半年的娟姗牛患肺水肿病的原因之一。
xzjs与syjs (CK)肠道菌群相比,考拉杆菌属(Phascolarctobacterium)、罗氏菌属(Roseburia)、布劳特氏菌属(Blautia)丰度极显著降低。布劳特氏菌属(Blautia)具有抗炎症的特性[31];考拉杆菌属(Phascolarctobacterium)产短链脂肪酸(short-chain fatty acid)[31];罗氏菌属(Roseburia)产丁酸盐[32]。机体免疫反应可通过免疫细胞(如Treg细胞)和肠道菌群产生的SCFA (如醋酸盐、丙酸盐和丁酸盐)来调节[33],免疫细胞的生成又可被SCFA调节,通过调节还可促进T细胞分化成Treg细胞。因此有益菌考拉杆菌属(Phascolarctobacterium)、罗氏菌属(Roseburia)、布劳特氏菌属(Blautia)丰度的降低会影响肠道稳态、肠道损伤恢复以及肠道健康。
xzjspe与xzjs肠道菌群相比梭菌属(Clostridium)丰度极显著降低(P < 0.01),粪球菌属(Coprococcus)丰度显著降低(P < 0.05)。粪球菌属[34] (Coprococcus)和梭菌属(Clostridium)可以抑制有害菌生长,梭菌属(Clostridium)还可产生维生素。由于粪球菌属和梭菌属等有益菌的减少,机体抑制有害菌生长的能力减弱,导致胃肠道菌群紊乱,胃肠道机能下降、机体出现炎症可能性升高,从而影响到机体健康水平和免疫力的下降。
有一些刊物报道了高丰度的艾克曼菌可能存在潜在的致病性。Chassaing等[35]观察到,Akkermansia增加了IL10‒/‒小鼠的肠道通透性,导致黏液保护功能的受损、细菌粘附性和炎症性微生物群的增加。Seregin等[36]发现,反复灌胃艾克曼菌可导致SPFIL10‒/‒小鼠结肠炎严重程度的增加。在严重肺部疾病中,肠道细菌在肺部被检测到,这被认为是由屏障完整性受损造成的[37]。通过LEFSe分析,Akkermansia在患肺水肿病牛肠道中的含量高于拉萨本地黄牛,显著高于健康娟姗牛,拉萨本地黄牛肠道中Akkermansia含量显著高于健康娟姗牛(图 2和图 3),但拉萨本地黄牛并未患肺水肿病。因此可推测,适量的Akkermansia并不会导致高原牛患肺水肿病(本研究中对本地黄牛来讲,Akkermansia丰度6.62%为适量),而较高丰度的Akkermansia可能通过导致肠道通透性增加,黏液保护功能受损,细菌粘附性和炎症性微生物群增加,从而使高原牛肺水肿病情加重(对娟姗牛来讲,Akkermansia丰度11.85%为高丰度,且为第一优势菌属)。
综上所述,本研究首次从微生物学角度分析Akkermansia与高原牛肺水肿病的关系:当牛适应高原环境时,肠道菌群未发生紊乱,Akkermansia生长正常,适量的Akkermansia可发挥有益作用,如可增加肠道屏障功能,于高原牛生长有益,如拉萨本地黄牛肠道菌群中Akkermansia占比6.62%;而当刚引进娟姗牛无法适应高原环境时,高原环境可导致其胃肠道菌群失调、免疫力下降、患病风险升高;加之牛所处缺氧环境,牛肠胃中也缺少氧气,较利于Akkermansia的生长。高原肺水肿病是高原常见病,由于肺水肿病造成肺通气与换气严重障碍,更进一步造成肠胃缺氧状态,更有利严格厌氧的Akkermansia的生长。当高原牛患肺水肿病时,Akkermansia则大量生长,丰度大大升高,由于其以黏蛋白为唯一氮源的特性,可能通过导致肠道通透性增加、黏液保护功能受损、细菌粘附性和炎症性微生物群增加,从而使高原牛肺水肿病情恶化。外部所处低压环境,水分易扩散,肠道菌群及其代谢产物更易于扩散到肺部中,从而造成恶性循环。
本文通过对高原牛胃肠道菌群结构组成的分析发现,高原本地牛和高原肺水肿病牛胃肠道中存在大量Akkermansia,为新的Akkermansia资源来源,可为后期Akkermansia资源的挖掘与应用研究提供方向;同时探讨了Akkermansia与高原牛肺水肿病的关系,为将Akkermansia丰度作为诊断肺水肿病的监测指标提供了参考,但具体丰度值还有待进一步研究。
4 展望Akkermansia是严格的厌氧菌,以粘蛋白作为碳、氮的唯一来源生长,不易培养,本研究结果表明,高原患有肺水肿病牛的肠胃中存在大量的Akkermansia,推测可以仿生高原患有肺水肿病牛的肠胃环境作为孵化器用于Akkermansia的大规模培养,为后期Akkermansia资源的挖掘与其益生特性应用研究提供菌种来源,且本研究中Akkermansia与高原肺水肿病之间的作用机理还有待进一步研究。
| [1] | Xu Y, Wang N, Tan HY, Li S, Zhang C, Feng Y. Function of Akkermansia muciniphila in obesity: interactions with lipid metabolism, immune response and gut systems. Frontiers in Microbiology, 2020, 11: 219. DOI:10.3389/fmicb.2020.00219 |
| [2] | Naito Y, Uchiyama K, Takagi T. A next-generation beneficial microbe: Akkermansia muciniphila. Journal of Clinical Biochemistry and Nutrition, 2018, 63(1): 33-35. DOI:10.3164/jcbn.18-57 |
| [3] | Arumugam M, Raes J, Pelletier E, Le Paslier D, Yamada T, Mende DR, Fernandes GR, Tap J, Bruls T, Batto JM, Bertalan M, Borruel N, Casellas F, Fernandez L, Gautier L, Hansen T, Hattori M, Hayashi T, Kleerebezem M, Kurokawa K, Leclerc M, Levenez F, Manichanh C, Nielsen HB, Nielsen T, Pons N, Poulain J, Qin JJ, Sicheritz-Ponten T, Tims S, Torrents D, Ugarte E, Zoetendal EG, Wang J, Guarner F, Pedersen O, De Vos WM, Brunak S, Doré J, Consortium M, Antolín M, Artiguenave F, Blottiere HM, Almeida M, Brechot C, Cara C, Chervaux C, Cultrone A, Delorme C, Denariaz G, Dervyn R, Foerstner KU, Friss C, Van De Guchte M, Guedon E, Haimet F, Huber W, Van Hylckama-Vlieg J, Jamet A, Juste C, Kaci G, Knol J, Lakhdari O, Layec S, Le Roux K, Maguin E, Mérieux A, Melo Minardi R, M'rini C, Muller J, Oozeer R, Parkhill J, Renault P, Rescigno M, Sanchez N, Sunagawa S, Torrejon A, Turner K, Vandemeulebrouck G, Varela E, Winogradsky Y, Zeller G, Weissenbach J, Ehrlich SD, Bork P. Enterotypes of the human gut microbiome. Nature, 2011, 473(7346): 174-180. DOI:10.1038/nature09944 |
| [4] | Cheng D, Xie MZ. A review of a potential and promising probiotic candidate-Akkermansia muciniphila. Journal of Applied Microbiology, 2021, 130(6): 1813-1822. DOI:10.1111/jam.14911 |
| [5] | Derrien M, Aughan EEV, Plugge CM, De Vos WM. Akkermansia muciniphila gen. nov., sp. nov., a human intestinal mucin-degrading bacterium. International Journal of Systematic and Evolutionary Microbiology, 2004, 54(5): 1469-1476. |
| [6] | Ottman N, Geerlings SY, Aalvink S, De Vos WM, Belzer C. Action and function of Akkermansia muciniphila in microbiome ecology, health and disease. Best Practice & Research Clinical Gastroenterology, 2017, 31(6): 638-640. |
| [7] | Blacher E, Bashiardes S, Shapiro H, Rothschild D, Mor U, Dori-Bachash M, Kleimeyer C, Moresi C, Harnik Y, Zur M, Zabari M, Brik RBZ, Kviatcovsky D, Zmora N, Cohen Y, Bar N, Levi I, Amar N, Mehlman T, Brandis A, Biton I, Kuperman Y, Tsoory M, Alfahel L, Harmelin A, Schwartz M, Israelson A, Arike L, Johansson MEV, Hansson GC, Gotkine M, Segal E, Elinav E. Potential roles of gut microbiome and metabolites in modulating ALS in mice. Nature, 2019, 572(7770): 474-480. DOI:10.1038/s41586-019-1443-5 |
| [8] | Li J, Zhao FQ, Wang YD, Chen JR, Tao J, Tian G, Wu SL, Liu WB, Cui QH, Geng B, Zhang WL, Weldon R, Auguste K, Yang L, Liu XY, Chen L, Yang XC, Zhu Bl, Cai J. Gut microbiota dysbiosis contributes to the development of hypertension. Microbiome, 2017, 5(1): 1-19. DOI:10.1186/s40168-016-0209-7 |
| [9] | Gómez-Gallego C, Pohl S, Salminen S, De Vos WM, Kneifel W. Akkermansia muciniphila: a novel functional microbe with probiotic properties. Beneficial Microbes, 2016, 7(4): 571-584. DOI:10.3920/BM2016.0009 |
| [10] | Zhang T, Li QQ, Cheng L, Buch H, Zhang FM. Akkermansia muciniphila is a promising probiotic. Microbial Biotechnology, 2019, 12(6): 1109-1125. DOI:10.1111/1751-7915.13410 |
| [11] | Zhou KQ. Strategies to promote abundance of Akkermansia muciniphila, an emerging probiotics in the gut, evidence from dietary intervention studies. Journal of Functional Foods, 2017, 33: 194-201. DOI:10.1016/j.jff.2017.03.045 |
| [12] | Geerlings SY, Ioannis K, De Vos WM, Clara B. Akkermansia muciniphila in the human gastrointestinal tract: when, where, and how?. Microorganisms, 2018, 6(6): 1-13. |
| [13] | Lu ZY, Guo XJ, Li H, Huang ZZ, Lin, KF, Liu YD. High-throughput screening for a moderately halophilic phenol-degrading strain and its salt tolerance response. International Journal of Molecular Sciences, 2015, 16(6): 11834-11848. |
| [14] | Reon BJ, Dutta A. Biological processes discovered by high-throughput sequencing. The American Journal of Pathology, 2016, 186(4): 722-732. DOI:10.1016/j.ajpath.2015.10.033 |
| [15] | Wang ZR, Yang GJ, Yi SH, Wu Z, Guan JY, He XB, Ye BS. Different response of vegetation to permafrost change in semi-arid and semi-humid regions in Qinghai-Tibetan Plateau. Environmental Earth Sciences, 2012, 66(3): 985-991. DOI:10.1007/s12665-011-1405-1 |
| [16] | Shi PJ, Chen YQ, Zhang GF, Tang HP, Chen Z, Yu DY, Yang J, Ye T, Wang JA, Liang SL, Ma YG, Wu JD, Gong P. Factors contributing to spatial-temporal variations of observed oxygen concentration over the Qinghai-Tibetan Plateau. Scientific Reports, 2021, 11(1): 17338. DOI:10.1038/s41598-021-96741-6 |
| [17] | He Y, Zhao YC, Zhou GM, Huang MS. Evaluation of extraction and purification methods for obtaining PCR-amplifiable DNA from aged refuse for microbial community analysis. World Journal of Microbiology and Biotechnology, 2009, 25(11): 2043-2051. DOI:10.1007/s11274-009-0106-3 |
| [18] | Lawley B, Tannock GW. Analysis of 16S rRNA gene amplicon sequences using the QIIME software package. Oral Biology, 2017, 1537: 153-163. |
| [19] | Sarnaik A, Liu A, Nielsen D, Varman AM. High-throughput screening for efficient microbial biotechnology. Current Opinion in Biotechnology, 2020, 64: 141-150. DOI:10.1016/j.copbio.2020.02.019 |
| [20] |
Si LW, Han HY. Cattle and sheep՚s rumen microbial diversity and the influencing factors. China Feed, 2020(21): 8-14.
(in Chinese) 司丽炜, 韩红燕. 牛羊瘤胃微生物多样性及其影响因素. 中国饲料, 2020(21): 8-14. DOI:10.15906/j.cnki.cn11-2975/s.20202102 |
| [21] | Wolcott RD, Hanson JD, Rees EJ, Koenig LD, Phillips CD, Wolcott RA, Cox SB, White JS. Analysis of the chronic wound microbiota of 2 963 patients by 16S rDNA pyrosequencing. Wound Repair and Regeneration: Official Publication of the Wound Healing Society and the European Tissue Repair Society, 2016, 24(1): 164-172. |
| [22] | Schloss PD, Westcott SL, Ryabin T, Hall JR, Hartmann M, Hollister EB, Lesniewski RA, Oakley BB, Parks DH, Robinson CJ, Sahl JW, Stres B, Thallinger GG, Van Horn DJ, Weber CF. Introducing mothur: open-source, platform-independent, community- supported software for describing and comparing microbial communities. Applied and Environmental Microbiology, 2009, 75(23): 7537-7541. DOI:10.1128/AEM.01541-09 |
| [23] | Routy B, Le Chatelier E, Derosa L, Duong CPM, Alou MT, Daillère R, Fluckiger A, Messaoudene M, Rauber C, Roberti MP, Fidelle M, Flament C, Poirier-Colame V, Opolon P, Klein C, Iribarren K, Mondragón L, Jacquelot N, Qu B, Ferrere G, Clémenson C, Mezquita L, Masip JR, Naltet C, Brosseau S, Kaderbhai C, Richard C, Rizvi H, Levenez F, Galleron N, Quinquis B, Pons N, Ryffel B, Minard-Colin V, Gonin P, Soria JC, Deutsch E, Loriot Y, Ghiringhelli F, Zalcman G, Goldwasser F, Escudier B, Hellmann MD, Eggermont A, Raoult D, Albiges L, Kroemer G, Zitvogel L. Gut microbiome influences efficacy of PD-1-based immunotherapy against epithelial tumors. Science, 2018, 359(6371): 91-97. DOI:10.1126/science.aan3706 |
| [24] | Yang YJ, Zhong ZQ, Wang BJ, Xia XW, Yao WY, Huang L, Wang YL, Ding WJ. Early-life high-fat diet-induced obesity programs hippocampal development and cognitive functions via regulation of gut commensal Akkermansia muciniphila. Neuropsychopharmacology, 2019, 44(12): 2054-2064. DOI:10.1038/s41386-019-0437-1 |
| [25] | Roshanravan N, Bastani S, Tutunchi H, Kafil B, Nikpayam O, Mesri Alamdari N, Hadi A, Sotoudeh S, Ghaffari S, Ostadrahimi A. A comprehensive systematic review of the effectiveness of Akkermansia muciniphila, a member of the gut microbiome, for the management of obesity and associated metabolic disorders. Archives of Physiology and Biochemistry, 2021. DOI:10.1080/13813455.2021.1871760 |
| [26] |
Zhang CG, Gong WJ, Li ZH, Gao DW, Gao Y. Gut flora-centric theory of evolution: a new model for understanding the evolution of animals. Chinese Journal of Bioinformatics, 2018, 16(4): 203-213.
(in Chinese) 张成岗, 巩文静, 李志慧, 高大文, 高艳. 菌心进化论: 一种对于动物进化的新理解. 生物信息学, 2018, 16(4): 203-213. |
| [27] | Mariat D, Firmesse O, Levenez F, Guimarăes V, Sokol H, Doré J, Corthier G, Furet JP. The Firmicutes/Bacteroidetes ratio of the human microbiota changes with age. BMC Microbiology, 2009, 9: 123. DOI:10.1186/1471-2180-9-123 |
| [28] | Thoetkiattikul H, Mhuantong W, Laothanachareon T, Tangphatsornruang S, Pattarajinda V, Eurwilaichitr L, Champreda V. Comparative analysis of microbial profiles in cow rumen fed with different dietary fiber by tagged 16S rRNA gene pyrosequencing. Current Microbiology, 2013, 67(2): 130-137. DOI:10.1007/s00284-013-0336-3 |
| [29] | Huijbregts DHH, Van Der Waaij LA, Harmsen HJM, Boersma BJ, Smith AE, Welling GW. Evaluation of small bowel bacterial overgrowth in human duodenal biopsies by FISH. European Journal of Gastroenterology & Hepatology, 2006, 18(1): A64. |
| [30] | Petersson J, Schreiber O, Hansson GC, Gendler SJ, Velcich A, Lundberg JO, Roos S, Holm L, Phillipson M. Importance and regulation of the colonic mucus barrier in a mouse model of colitis. AJP Gastrointestinal and Liver Physiology, 2011, 300(2): G327. DOI:10.1152/ajpgi.00422.2010 |
| [31] | Luedde M, Winkler T, Heinsen FA, Rühlemann MC, Spehlmann ME, Bajrovic A, Lieb W, Franke A, Ott SJ, Frey N. Heart failure is associated with depletion of core intestinal microbiota. ESC Heart Failure, 2017, 4(3): 282-290. DOI:10.1002/ehf2.12155 |
| [32] | Morgan XC, Tickle TL, Sokol H, Gevers D, Devaney KL, Ward DV, Reyes JA, Shah SA, LeLeiko N, Snapper SB, Bousvaros A, Korzenik J, Sands BE, Xavier RJ, Huttenhower C. Dysfunction of the intestinal microbiome in inflammatory bowel disease and treatment. Genome Biology, 2012, 13(9): R79. DOI:10.1186/gb-2012-13-9-r79 |
| [33] | Chung KF. Airway microbial dysbiosis in asthmatic patients: a target for prevention and treatment?. The Journal of Allergy and Clinical Immunology, 2017, 139(4): 1071-1081. DOI:10.1016/j.jaci.2017.02.004 |
| [34] |
Zhao R, Shan CQ, Song F, Song HN, Xu FL, Liu Y. Inhibition of 4 strains of probiotics on 6 strains of pathogenic bacteria. Heilongjiang Animal Science and Veterinary Medicine, 2020(21): 104-108.
(in Chinese) 赵荣, 单春乔, 宋凡, 宋蕙男, 徐福利, 刘艳. 4株益生菌对6株致病菌的抑制作用研究. 黑龙江畜牧兽医, 2020(21): 104-108. |
| [35] | Chassaing B, Koren O, Goodrich JK, Poole AC, Srinivasan S, Ley RE, Gewirtz AT. Dietary emulsifiers impact the mouse gut microbiota promoting colitis and metabolic syndrome. Nature, 2015, 519(7541): 92-96. DOI:10.1038/nature14232 |
| [36] | Seregin SS, Golovchenko N, Schaf B, Chen JC, Pudlo NA, Mitchell J, Baxter NT, Zhao LL, Schloss PD, Martens EC, Eaton KA, Chen GY. NLRP6 protects Il10‒/‒ mice from colitis by limiting colonization of Akkermansia muciniphila. Cell Reports, 2017, 19(4): 733-745. DOI:10.1016/j.celrep.2017.03.080 |
| [37] | Cruz-Aguliar RM, Wantia N, Clavel T, Vehreschild MJGT, Buch T, Bajbouj M, Haller D, Busch D, Schmid RM, Stein-Thoeringer CK. An open-labeled study on fecal microbiota transfer in irritable bowel syndrome patients reveals improvement in abdominal pain associated with the relative abundance of Akkermansia muciniphila. Digestion, 2019, 100(2): 127-138. DOI:10.1159/000494252 |
 2022, Vol. 62
2022, Vol. 62