中国科学院微生物研究所,中国微生物学会
文章信息
- 李娜, 袁晓鸣, 王涓, 吴清平, 丁郁. 2022
- LI Na, YUAN Xiaoming, WANG Juan, WU Qingping, DING Yu.
- 短尾噬菌体识别宿主机制的研究进展
- Progress in the host recognition mechanisms of Podoviridae phages
- 微生物学报, 62(11): 4324-4335
- Acta Microbiologica Sinica, 62(11): 4324-4335
-
文章历史
- 收稿日期:2022-03-15
- 修回日期:2022-05-24
- 网络出版日期:2022-06-13
2. 暨南大学食品科学与工程系, 广东 广州 510632;
3. 华南农业大学食品学院, 广东 广州 510642
2. Department of Food Science and Engineering, Jinan University, Guangzhou 510632, Guangdong, China;
3. College of Food Science, South China Agricultural University, Guangzhou 510642, Guangdong, China
噬菌体是能感染细菌、真菌、放线菌或螺旋体的病毒,其结构简单,主要由蛋白质外壳和遗传物质核酸组成[1]。绝大多数噬菌体具有尾部结构,根据其尾部形态的差异又可分为三大类:短尾噬菌体、肌尾噬菌体和长尾噬菌体[2]。长尾噬菌体是公共数据库中数量最多的有尾噬菌体(66%),其次是肌尾噬菌体(20%)和短尾噬菌体(14%)[3]。肌尾噬菌体基因组较大,平均约为(133±80) kb,通常由较大的头部(约150 nm)和可收缩的尾部组成;长尾噬菌体基因组大小平均约为(51±23) kb,由相对较小的衣壳蛋白(约50‒60 nm)和长的、但不可收缩的尾组成;短尾噬菌体基因组较小,平均约为(49±20) kb,由较小的头部和不可收缩的短尾组成[4‒5]。不同形态的噬菌体感染宿主的能力也不同。通常,肌尾噬菌体有较强的裂解能力且宿主范围较广;短尾噬菌体通常也具有较强的裂解能力,但宿主范围较窄,对宿主感染具有特异性;长尾噬菌体裂解能力通常较弱,宿主范围介于短尾噬菌体和肌尾噬菌体之间[6]。宿主范围的差异,很大程度上是由感染过程中噬菌体受体结合蛋白(receptor binding protein,RBP)对宿主的不同识别作用决定的[7]。Nobrega等[8]系统阐述了有尾噬菌体靶向宿主的不同机制。Goulet等[3]也针对长尾噬菌体的RBP与宿主受体分子进行了综述。不同的短尾噬菌体虽然有相似的结构特征,但其尾部结构和识别宿主的机制仍存在明显差异。目前有关短尾噬菌体识别机制的系统总结较为缺乏,因此,本文以短尾噬菌体为对象,概述了不同短尾噬菌体的尾部结构特征及识别机制,旨在增强对噬菌体和宿主共进化关系的认识,期待未来实现短尾噬菌体的有效改造,以达到更好地利用噬菌体防控病原微生物的目的。
1 短尾噬菌体的分类和特点传统的病毒分类学依据噬菌体的形态差异进行分类。有尾噬菌体由头-尾结构组成,通常为双链DNA病毒,能够感染细菌和古菌。根据国际病毒分类委员会(International Committee on Taxonomy of Viruses,ICTV)第9次报告[9],将有尾噬菌体归为有尾噬菌体目(Caudalovirales),并依据不同的尾部形态,分为肌尾噬菌体科(Myoviridae)、长尾噬菌体科(Siphoviridae)和短尾噬菌体科(Podoviridae) 3个科。虽然在2021年最新的ICTV报告中,从基因组角度对所有噬菌体进行了重新分类,但本文为清晰起见,仍沿用基于形态的分类方式,探讨短尾噬菌体科中不同类型噬菌体的识别宿主机制。
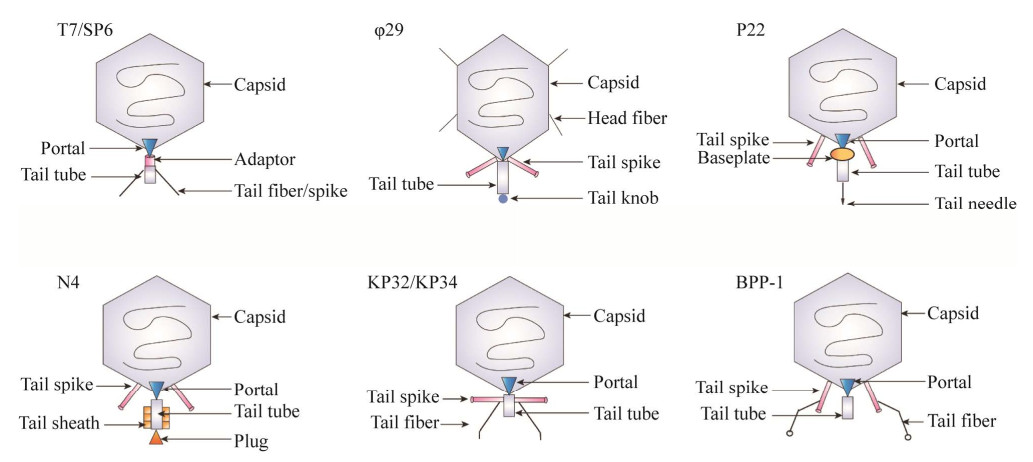
根据属内(40%)和属间(20%)同源蛋白的一致性,短尾噬菌体科分为Autographivirinae和Picovirinae两个亚科,共11个属[9]。这些噬菌体的头部为等距或拉长的二十面体,由支架蛋白和衣壳蛋白组装而成,少数噬菌体含有头部纤维,有利于噬菌体附着于细菌[10]。噬菌体尾部与头部的一个顶点相连,通常由尾钉、尾纤维和尾管等结构组成。尽管不同类型的短尾噬菌体其尾部结构和组成存在差异(图 1),但都包含由特定蛋白亚基组成的RBP,负责与宿主表面受体结合[10]。
2 宿主表面受体结构
噬菌体主要通过RBP识别宿主表面的受体,而受体的多样性是由宿主细胞的表层结构决定的[11]。革兰氏阴性菌的表面存在一层细胞外膜,包括蛋白和脂多糖分子(LPS)。脂多糖分子由极性磷脂A (lipid A)、核心寡聚糖以及O抗原(O-antigen)三部分组成(图 2A)[12],而O抗原则由低聚糖重复单位(O单元)构成,通常含有2-8种不同类型的糖残基。不同菌株的O抗原在糖残基的类型、O单元内部残基排列顺序、以及糖环在单元内部和单元之间的连接上存在差异,使其成为最多样化的细胞成分之一[16]。噬菌体识别革兰氏阴性菌时通常以O抗原作为受体。依据O抗原糖基化的不同,革兰氏阴性菌分为不同血清型,同时抗原的不同结构也影响着噬菌体的识别。例如,噬菌体P22能够特异性识别O4抗原,而不能识别包含1-6连接葡萄糖单元的O1抗原[17]。在大肠杆菌中,O抗原的O-乙酰基化对于噬菌体的识别也至关重要。噬菌体G7C能够特异性识别大肠杆菌4s表面的乙酰基化O抗原,而O-乙酰化的缺失可导致G7C对宿主菌的敏感性发生改变[18]。此外,革兰氏阴性菌的外膜蛋白也可以作为一些噬菌体识别宿主的受体[19]。
革兰氏阳性菌的细胞壁在结构和化学成分上与革兰氏阴性菌存在明显差异,其肽聚糖层厚,且没有细胞外膜的覆盖。壁磷壁酸(wall teichoic acid,WTA)和脂磷壁酸(lipoteichoic acid,LTA)是革兰氏阳性菌细胞壁的重要组成成分(图 2B)[20]。同时,磷壁酸也是革兰氏阳性菌细胞壁中最多样化的表面分子,大部分短尾噬菌体可利用磷壁酸识别革兰氏阳性菌宿主[21]。例如噬菌体φ29主要通过尾钉蛋白识别枯草芽胞杆菌的磷壁酸[22]。同样地,在我们前期的研究中也发现,φ29类噬菌体—蜡样芽胞杆菌噬菌体DLc1利用宿主细胞壁上的多糖结构作为识别受体[23]。在不同物种或菌株间,磷壁酸有着不同的残基修饰情况[24]。通常引起磷壁酸糖苷键酰基化的酶为α-糖基转移酶(TarM)和β-糖基转移酶(TarS)。研究发现短尾噬菌体(φ44AHJD、φ66、φP68)能够识别金黄色葡萄球菌TarS介导的酰基化磷壁酸,而不能识别TarM介导的酰基化磷壁酸,表明磷壁酸酰基化的形式对于噬菌体的识别具有特异性[25]。对于枯草芽胞杆菌而言,不同菌株的磷壁酸由不同形式的葡萄糖修饰。例如枯草芽胞杆菌168的磷壁酸由α-连接葡萄糖(α-Glc)修饰,而菌株W23的磷壁酸则由β-Glc修饰。在噬菌体感染枯草芽胞杆菌168时,需要识别α-Glc糖基化的磷壁酸才能够感染宿主[26]。
3 短尾噬菌体的识别宿主机制由于尾部结构的差异,不同类型短尾噬菌体识别宿主的方式也各不相同(表 1)。大部分短尾噬菌体利用尾纤维蛋白(tail fiber protein,TFP)或尾钉蛋白(tail spike protein,TSP)识别宿主表面的受体,还有少部分噬菌体也会利用尾管蛋白(tail tube protein,TTP)、尾鞘蛋白(tail sheath protein,TSHP)或尾针旋钮(tail needle knob,TNK)识别宿主。
| Phage | Binding protein | Bacterial host | Receptor | References |
| T7 | TFP | Escherichia coli | LPS | [27] |
| SP6 | TSP | Salmonella enterica | O antigen | [28] |
| φ29 | TSP | Bacillus subtilis | WTA | [21] |
| P22 | TSP | Salmonella enterica | O antigen | [29] |
| HK620 | TSP | Escherichia coli H TD2158 | LPS | [30] |
| Sf6 | TSP/TNK | Shigella flexneri | LPS, OmpA/OmpC | [31] |
| N4 | TSP/TSHP | Escherichia coli K-12 | NfrA | [32] |
| G7C | TSP | Escherichia coli 4s | O antigen | [33] |
| KP32 | TSP/TTP | Klebsiella pneumoniae | EPS | [34] |
| KP34 | TSP/TTP | Klebsiella pneumoniae | CPS | [34] |
| BPP-1 | TFP | Bordetella | Pertactin | [35] |
| EP335 | TFP | Escherichia coli | O antigen | [36] |
3.1 利用TFP识别宿主
TFP是一类长而具有柔性的尾部蛋白。大肠杆菌噬菌体T7是Autographivirinae亚科的典型噬菌体,其头部形态是二十面体,尾部由门户(portal)、适配器(adaptor)、管口(nozzle)和尾纤维(tail fiber) 4种蛋白组成[37] (图 1)。在感染过程中,管口的尾尖端区域朝向细菌外膜,同时,尾部的TFP识别宿主细胞表面的脂多糖,并与之发生相互作用[27]。通常,噬菌体的TFP中N端负责连接尾纤维与病毒粒子,C端结构域负责结合宿主受体[10]。噬菌体T7的尾纤维蛋白gp17包含一个N端结构域,与病毒粒子相连;与之相邻的是一个杆状结构域(长度为16 nm,宽度为2 nm),连接了N端与C端部分;C端结构域则由4个线性排列的、包含多个β-折叠结构的模块组成[38]。当噬菌体T7在多重宿主中发生进化时,多数突变位点集中在尾纤维蛋白的C端末梢,说明噬菌体主要通过该区域识别宿主;但也有大量突变发生在杆状结构域,说明该结构域可能也在病毒感染的某个阶段与细菌受体发生作用。此外,杆状结构域的突变可能改变C端结构域末梢的取向,间接调节RBP与细菌表面脂多糖的相互作用[38]。
博代氏杆菌噬菌体BPP-1也利用TFP识别宿主。与很多短尾噬菌体相比,BPP-1具有特殊的尾部结构[39]。该噬菌体由圆柱状中心尾管、6个尾钉蛋白及6个长且具有柔性的尾纤维组成,每个尾纤维末梢呈球形结构[35] (图 1)。噬菌体的尾钉蛋白附着在衣壳上,且表现出向中心轻微倾斜的扭曲模式,尾纤维则附着在尾钉上。BPP-1这种长而柔性的尾纤维结构在长尾噬菌体中较为常见,在短尾噬菌体中少见。BPP-1的尾纤维末梢球形结构上的Mtd蛋白是噬菌体的受体结合蛋白,可以识别细胞表面的受体pertactin从而启动感染[40]。当噬菌体颗粒接触宿主细菌时,6个尾纤维之一末梢上的Mtd分子首先结合细胞膜受体,并使噬菌体颗粒停留在细胞表面附近[35]。Mtd分子的初始结合增加了其他尾纤维上Mtd分子与受体结合的概率,从而增加了噬菌体颗粒在细胞表面的附着强度;多个尾纤维与细胞膜受体的结合也使噬菌体颗粒更朝向细胞表面。此外,与Mtd结合后,受体的横向运动也可能会将噬菌体颗粒拉向细胞表面,为尾管的插入提供机械支撑。在此过程中,尾钉会进行构象变化以减小空间位阻[35]。
一些短尾噬菌体也被发现同时具有多种尾纤维结构,如噬菌体EP335中含有2种尾纤维(gp12和gp13),其中尾纤维蛋白gp13可以通过识别O抗原结合大肠杆菌O157和O26菌株,而尾纤维蛋白gp12未发现有结合能力[36]。EP335的尾蛋白gp12和gp13与噬菌体phiEco32的尾蛋白gp14和gp15具有高度同源性,说明EP335的尾纤维也可能在基板上形成分支状网络结构[36]。
3.2 利用TSP识别宿主相比于TFP,尾钉蛋白TSP较短且更具刚性。大多数P22类噬菌体采用尾钉蛋白识别宿主[41]。沙门氏菌噬菌体P22具有等距的二十面体头部,尾部由门户蛋白(gp1)、基板蛋白(gp4和gp10)、尾针蛋白(gp26)和6个尾钉蛋白(gp9)组成[42]。噬菌体P22的尾钉蛋白可以与O抗原及核心脂多糖(core-LPS)发生作用,通过含有水解酶活性的位点识别细胞壁的多糖,进而降解细胞壁[43]。P22颗粒最初以倾斜角度与细胞表面结合,6个尾钉蛋白中的任意2个与尾针蛋白共同推动噬菌体P22与细胞表面的O抗原接触[44];随后,由于离细胞最近的2个尾钉蛋白与O抗原的结合位点距离core-LPS约8 nm,是O抗原重复单元长度的5倍,因此吸附的噬菌体必须水解几个O抗原重复单元,或借助尾钉蛋白的若干次快速结合和释放以接近LPS[42]。大肠杆菌噬菌体HK620在形态上与P22噬菌体类似[45],其尾钉蛋白是一种内N-乙酰氨基葡萄糖酶,能够特异性识别和水解大肠杆菌O18A1型的脂多糖[45],还可以区分大肠杆菌中同时存在的O18A1抗原和O18A2抗原[46]。值得注意的是,噬菌体HK620尾钉蛋白中单个氨基酸的变化可提高其与脂多糖的亲和力[47]。在其他噬菌体中也存在类似现象,单个氨基酸的变化甚至可以将RBP的识别位点由蛋白变成糖配体[48]。
以尾钉蛋白作为RBP的还有Picovirinae亚科噬菌体,以噬菌体φ29为典型代表,能够特异性侵染枯草芽胞杆菌、蜡样芽胞杆菌等芽胞杆菌。这一类型的噬菌体尾部有12个尾钉蛋白,一些噬菌体还含有头部纤维蛋白[49]。Xu等[49]对噬菌体φ29的尾部结构进行了详细的解析,表明其尾钉蛋白分为3个结构部分,包括N端、臂茎连接单元和尾穗末端。通过对尾穗取向的观察,发现尾钉蛋白可分成两组,第一组蛋白垂直于尾轴,而第二组蛋白大致平行于尾轴。与其他短尾噬菌体一样,这类尾钉蛋白的尾穗末端结构域可能具有酶活性,并在感染期间参与结合和消化细胞壁,促使病毒尾部接近细胞膜[50]。噬菌体φ29也采用与P22类似的倾斜吸附方式,在感染枯草芽胞杆菌时,最初通过12个尾钉蛋白中的1个接触细胞壁,进而水解细胞壁的磷壁酸并使噬菌体更接近细胞。在此过程中,头部纤维蛋白与细胞表面的弱结合力也可以为尾钉蛋白识别磷壁酸提供额外的机会[21]。除芽胞杆菌短尾噬菌体外,Picovirinae亚科内的链球菌噬菌体C1也含有12个与噬菌体φ29类似结构的尾钉蛋白。同样的,每个尾钉蛋白包含3个结构域:1个与病毒结合的近端结构域、1个长臂结构域和1个末端结构域。12个尾钉蛋白都参与了噬菌体对宿主的识别,其中1个尾钉蛋白与受体的结合增加了其他11个尾钉蛋白的吸附概率,从而增加了噬菌体与受体的结合力[50]。
与T7同属Autographivirinae亚科的噬菌体SP6,头部形态也为二十面体,但其不具有类似噬菌体T7的尾纤维,取而代之的是两组不同的尾钉蛋白,分别为TSP-α和TSP-β[28],之间由一个适配器蛋白连接(图 1)。噬菌体SP6能够感染不同血清型的沙门氏菌,即鼠伤寒沙门氏菌(B血清型)和纽波特沙门氏菌(C2血清型)[28],其中B血清型的O抗原由四糖(阿比可糖、半乳糖、甘露糖、鼠李糖)组成,而C2血清型的O抗原由六糖(阿比可糖、半乳糖、葡萄糖、2个甘露糖和鼠李糖)组成[51]。不同尾钉蛋白对宿主表面O抗原的识别,赋予了噬菌体SP6双重的宿主特异性。当SP6识别不同的宿主时,其TSP会发生构象变化:以中间的适配器为支点,当识别鼠伤寒沙门氏菌时,TSP-α构象旋转至更靠近细胞膜的方向;当识别纽波特沙门氏菌时,TSP-β则更靠近细胞膜[28]。于此同时,Tu等[28]也提出,虽然TSP与O抗原的结合明显促进了噬菌体SP6对宿主细胞的吸附,但这种特异性识别对于感染并不是必不可少的,在此感染过程中,噬菌体尾部的其他部分可能也发挥了重要的作用。
3.3 利用多种尾部蛋白识别宿主除TFP和TSP外,其他尾部结构也可能参与到噬菌体对宿主的识别过程中。噬菌体N4由直径为70 nm的二十面体头部、尾鞘蛋白(gp65)包裹的尾管、以及12个尾钉蛋白(gp66)等部分组成[32]。与其他类型噬菌体不同,为了将衣壳蛋白内部的病毒RNA聚合酶连同噬菌体DNA一并注射入宿主体内,N4的2个尾蛋白(gp65和gp66)在识别宿主和吸附中均起到重要作用。吸附开始阶段,尾钉蛋白gp66与宿主脂多糖或抗原发生可逆吸附,并使噬菌体靠近细胞外膜;随后,尾鞘蛋白gp65再与外膜蛋白受体NfrA发生不可逆结合[32]。类似地,志贺菌噬菌体Sf6对宿主菌的识别也分为2个阶段,首先利用TSP识别宿主脂多糖,随后利用其尾针蛋白末梢特殊的旋钮结构TNK与外膜蛋白OmpA和OmpC相互作用,将其作为第二受体,促进噬菌体的感染[52]。
由于肺炎克雷伯菌具有一层较厚的荚膜多糖,许多克雷伯菌噬菌体的RBP具有多糖解聚酶功能,能够以荚膜多糖作为受体[12]。肺炎克雷伯菌噬菌体KP32和KP34都是短尾噬菌体[53],其中KP32能够特异性裂解血清型为K3和K21的菌株,而KP34只能裂解血清型为K63的菌株,这一宿主范围是由噬菌体的RBP决定的。Pyra等[34]发现KP32的尾纤维蛋白gp37和尾钉蛋白gp38都具有荚膜多糖解聚酶功能,能够作为噬菌体的RBP,高度特异性地结合K3和K21型荚膜多糖;而在噬菌体KP34中,尾钉蛋白gp57只能结合K63型荚膜多糖[54]。此外,研究发现噬菌体KP32的尾部还存在另外2个与细菌感染相关的尾蛋白:尾管蛋白A (tail tubular protein A,TTPA)和尾管蛋白B (tail tubular protein B,TTPB)。其中TTPA是一种位于噬菌体尾部中心的结构蛋白,同时还包含2个凝集素样结构域,具有多糖解聚酶的功能,可以结合并降解胞外多糖(extracellular polysaccharide,EPS),使噬菌体到达细胞表面,与外膜受体结合[55]。噬菌体KP34编码的尾管蛋白(gp44)也具有类似的功能,对荚膜多糖具有水解活性[56]。
4 噬菌体与细菌的共进化噬菌体与细菌之间存在复杂的共进化关系。在长期共存中,噬菌体和细菌构成不断变化的网络。在噬菌体感染细菌的过程中,细菌基因组的多个可移动防御基因会在细菌间进行快速交换,从而抵抗噬菌体的感染[57];而噬菌体也会进化以应对细菌的防御,二者处于动态平衡[58]。细菌对噬菌体产生的抗性往往与细菌表面受体的自发突变和适应有关,细菌可以通过更改自身表面的受体以阻止噬菌体的识别[59]。例如在N4类短尾噬菌体Ab09感染铜绿假单胞杆菌时,一些染色体的突变和缺失赋予了该菌对于噬菌体的抗性,而这些突变的基因都与细菌表面脂多糖的合成有关[60]。一些细菌也会对表面受体进行修饰,从而阻止噬菌体的识别。在一些短尾噬菌体感染金黄色葡萄球菌过程中,TarM介导的磷壁酸α-O-氨基葡萄糖乙酰化可保护金黄色葡萄球菌免受短尾噬菌体攻击[24],而这种修饰不会阻止一些肌尾噬菌体的感染。T7类噬菌体具有较强的宿主特异性,只感染1‒2个近缘的宿主[61]。细菌对T7类噬菌体产生抗性的频率较高,通常也通过阻止噬菌体对细菌表面受体的识别和吸附来实现[62]。
与此同时,噬菌体尾纤维的突变也使其能够感染抗性菌并获得更高的裂解能力。在N4类噬菌体LUZ7与铜绿假单胞菌共同培养时,噬菌体尾纤维中单个氨基酸的替换提高了其对不敏感或较不敏感菌株的感染性,这种突变可以扩大宿主范围,有更好的应用价值[60]。同时,Schwartz等[63]也发现,T7类噬菌体也会通过突变尾部基因从而感染具有抗性的宿主。
对宿主细菌的识别是噬菌体感染过程中关键的第一步,而大多数噬菌体利用尾部蛋白作为RBP来识别宿主。通过研究噬菌体在进化过程中尾部结构发生的变化,可以对噬菌体尾部进行工程化改造,以解决细菌对噬菌体产生的抗性[64‒65]。由此可见,了解噬菌体识别宿主的机制,对于解决细菌对噬菌体的抗性问题至关重要,也将极大推动噬菌体的改造与实际应用。
5 总结和展望噬菌体的吸附过程具有很高的特异性,使得噬菌体疗法更加安全,但这种特异性也限制了噬菌体的广泛应用。因此,了解噬菌体识别宿主的机制对于推动噬菌体的实际应用至关重要。在噬菌体识别宿主的过程中,RBP对于宿主表面受体的识别尤为关键。短尾噬菌体除了利用尾纤维蛋白识别宿主受体外,还可利用尾钉蛋白、尾管蛋白等不同结构作为RBP来识别其对应宿主的不同部位。通过分析RBP的结构及功能,研究RBP与对应宿主受体的识别机制,能够增强对噬菌体与其宿主相互作用的了解。在噬菌体识别宿主的过程中,细菌通过受体的突变来抵抗噬菌体的感染,而噬菌体也会发生相应变化来应对细菌的防御。通过对噬菌体与宿主相互作用的深入研究,有望更好地对噬菌体进行工程化改造,进而将噬菌体作为有效抗菌剂更加广泛地应用在医药、食品、农业等不同领域。
| [1] | Ge HJ, Hu MZ, Zhao G, Du Y, Xu NN, Chen X, Jiao XA. The "fighting wisdom and bravery" of tailed phage and host in the process of adsorption. Microbiological Research, 2020, 230: 126344. DOI:10.1016/j.micres.2019.126344 |
| [2] | Kizziah JL, Manning KA, Dearborn AD, Dokland T. Structure of the host cell recognition and penetration machinery of a Staphylococcus aureus bacteriophage. PLoS Pathogens, 2020, 16(2): e1008314. DOI:10.1371/journal.ppat.1008314 |
| [3] | Goulet A, Spinelli S, Mahony J, Cambillau C. Conserved and diverse traits of adhesion devices from Siphoviridae recognizing proteinaceous or saccharidic receptors. Viruses, 2020, 12(5): 512. DOI:10.3390/v12050512 |
| [4] | Royer S, Morais AP, Da Fonseca Batistão DW. Phage therapy as strategy to face post-antibiotic era: a guide to beginners and experts. Archives of Microbiology, 2021, 203(4): 1271-1279. DOI:10.1007/s00203-020-02167-5 |
| [5] | Zrelovs N, Dislers A, Kazaks A. Motley crew: overview of the currently available phage diversity. Frontiers in Microbiology, 2020, 11: 579452. DOI:10.3389/fmicb.2020.579452 |
| [6] |
Zhang YU, Huang CX, Yang J, Jiao NZ. Interactions between marine microorganisms and their phages. Chinese Science Bulletin, 2011, 56(14): 1071-1079.
(in Chinese) 张永雨, 黄春晓, 杨军, 焦念志. 海洋微生物与噬菌体间的相互关系. 科学通报, 2011, 56(14): 1071-1079. |
| [7] | Olszak T, Latka A, Roszniowski B, Valvano MA, Drulis-Kawa Z. Phage life cycles behind bacterial biodiversity. Current Medicinal Chemistry, 2017, 24(36): 3987-4001. |
| [8] | Nobrega FL, Vlot M, De Jonge PA, Dreesens LL, Beaumont HJE, Lavigne R, Dutilh BE, Brouns SJJ. Targeting mechanisms of tailed bacteriophages. Nature Reviews Microbiology, 2018, 16(12): 760-773. DOI:10.1038/s41579-018-0070-8 |
| [9] | King AM, Lefkowitz E, Adams MJ, Carstens EB. Virus Taxonomy: Ninth Report of the International Committee on Taxonomy of Viruses. St Louis: Elsevier, 2011. |
| [10] | Hrebík D, Štveráková D, Škubník K, Füzik T, Pantůček R, Plevka P. Structure and genome ejection mechanism of Staphylococcus aureus phage P68. Science Advances, 2019, 5(10): eaaw7414. DOI:10.1126/sciadv.aaw7414 |
| [11] | Letarov AV, Kulikov EE. Adsorption of bacteriophages on bacterial cells. Biochemistry Biokhimiia, 2017, 82(13): 1632-1658. DOI:10.1134/S0006297917130053 |
| [12] | Knirel YA, Valvano MA. Bacterial Lipopolysaccharides: Structure, Chemical Synthesis, Biogenesis and Interaction with Host Cells. Vienna: Springer-Verlag, 2011. |
| [13] | Casjens SR, Molineux IJ. Short noncontractile tail machines: adsorption and DNA delivery by podoviruses. Viral Molecular Machines, 2012: 143-179. |
| [14] | Latka A, Leiman PG, Drulis-Kawa Z, Briers Y. Modeling the architecture of depolymerase-containing receptor binding proteins in Klebsiella phages. Frontiers in Microbiology, 2019, 10: 2649. DOI:10.3389/fmicb.2019.02649 |
| [15] | Brown L, Wolf JM, Prados-Rosales R, Casadevall A. Through the wall: extracellular vesicles in Gram-positive bacteria, mycobacteria and fungi. Nature Reviews Microbiology, 2015, 13(10): 620-630. DOI:10.1038/nrmicro3480 |
| [16] | Wang L, Wang Q, Reeves PR. Endotoxins: Structure, Function and Recognition. Dordrecht: Springer, 2010. |
| [17] | Schmidt A, Rabsch W, Broeker NK, Barbirz S. Bacteriophage tailspike protein based assay to monitor phase variable glucosylations in Salmonella O-antigens. BMC Microbiology, 2016, 16(1): 1-11. DOI:10.1186/s12866-015-0617-z |
| [18] | Knirel YA, Prokhorov NS, Shashkov AS, Ovchinnikova OG, Zdorovenko EL, Liu B, Kostryukova ES, Larin AK, Golomidova AK, Letarov AV. Variations in O-antigen biosynthesis and O-acetylation associated with altered phage sensitivity in Escherichia coli 4s. Journal of Bacteriology, 2015, 197(5): 905-912. DOI:10.1128/JB.02398-14 |
| [19] | Bohm K, Porwollik S, Chu WP, Dover JA, Gilcrease EB, Casjens SR, McClelland M, Parent KN. Genes affecting progression of bacteriophage P22 infection in Salmonella identified by transposon and single gene deletion screens. Molecular Microbiology, 2018, 108(3): 288-305. DOI:10.1111/mmi.13936 |
| [20] | Mistou MY, Sutcliffe IC, Van Sorge NM. Bacterial glycobiology: rhamnose-containing cell wall polysaccharides in Gram-positive bacteria. FEMS Microbiology Reviews, 2016, 40(4): 464-479. DOI:10.1093/femsre/fuw006 |
| [21] | Dunne M, Hupfeld M, Klumpp J, Loessner MJ. Molecular basis of bacterial host interactions by Gram-positive targeting bacteriophages. Viruses, 2018, 10(8): 397. DOI:10.3390/v10080397 |
| [22] | Farley MM, Tu J, Kearns DB, Molineux IJ, Liu J. Ultrastructural analysis of bacteriophage φ29 during infection of Bacillus subtilis. Journal of Structural Biology, 2017, 197(2): 163-171. DOI:10.1016/j.jsb.2016.07.019 |
| [23] | Li C, Yuan X, Li N, Wang J, Yu S, Zeng H, Zhang J, Wu Q, Ding Y. Isolation and characterization of Bacillus cereus phage vB_BceP-DLc1 reveals the largest member of the φ29-Like phages. Microorganisms, 2020, 8(11): 1750. DOI:10.3390/microorganisms8111750 |
| [24] | Wakinaka T, Matsutani M, Watanabe J, Mogi Y, Tokuoka M, Ohnishi A. Ribitol-containing wall teichoic acid of Tetragenococcus halophilus is targeted by bacteriophage phiWJ7 as a binding receptor. Microbiology Spectrum, 2022, 10(2): e00336-22. |
| [25] | Li X, Gerlach D, Du X, Larsen J, Stegger M, Kühner P, Peschel A, Xia G, Winstel V. An accessory wall teichoic acid glycosyltransferase protects Staphylococcus aureus from the lytic activity of Podoviridae. Scientific Reports, 2015, 5(1): 1-10. DOI:10.9734/JSRR/2015/14076 |
| [26] | Winstel V, Xia G, Peschel A. Pathways and roles of wall teichoic acid glycosylation in Staphylococcus aureus. International Journal of Medical Microbiology, 2014, 304(3/4): 215-221. |
| [27] | Holtzman T, Globus R, Molshanski-Mor S, Ben-Shem A, Yosef I, Qimron U. A continuous evolution system for contracting the host range of bacteriophage T7. Scientific Reports, 2020, 10(1): 307. DOI:10.1038/s41598-019-57221-0 |
| [28] | Tu J, Park T, Morado DR, Hughes KT, Molineux IJ, Liu J. Dual host specificity of phage SP6 is facilitated by tailspike rotation. Virology, 2017, 507: 206-215. DOI:10.1016/j.virol.2017.04.017 |
| [29] | Andres D, Baxa U, Hanke C, Seckler R, Barbirz S. Carbohydrate binding of Salmonella phage P22 tailspike protein and its role during host cell infection. Biochemical Society Transactions, 2010, 38(5): 1386-1389. DOI:10.1042/BST0381386 |
| [30] | Broeker NK, Kiele F, Casjens SR, Gilcrease EB, Thalhammer A, Koetz J, Barbirz S. In vitro studies of lipopolysaccharide-mediated DNA release of podovirus HK620. Viruses, 2018, 10(6): 289. DOI:10.3390/v10060289 |
| [31] | Teh MY, Tran ENH, Morona R. Bacteriophage Sf6 host range mutant that infects Shigella flexneri serotype 2a2 strains. FEMS Microbiology Letters, 2022, 369(1): fnac020. DOI:10.1093/femsle/fnac020 |
| [32] | McPartland J, Rothman-Denes LB. The tail sheath of bacteriophage N4 interacts with the Escherichia coli receptor. Journal of Bacteriology, 2009, 191(2): 525-532. DOI:10.1128/JB.01423-08 |
| [33] | Prokhorov NS, Riccio C, Zdorovenko EL, Shneider MM, Browning C, Knirel YA, Leiman PG, Letarov AV. Function of bacteriophage G7C esterase tailspike in host cell adsorption. Molecular Microbiology, 2017, 105(3): 385-398. DOI:10.1111/mmi.13710 |
| [34] | Pyra A, Brzozowska E, Pawlik K, Gamian A, Dauter M, Dauter Z. Tail tubular protein A: a dual-function tail protein of Klebsiella pneumoniae bacteriophage KP32. Scientific Reports, 2017, 7(1): 2223. DOI:10.1038/s41598-017-02451-3 |
| [35] | Dai W, Hodes A, Hui WH, Gingery M, Miller JF, Zhou ZH. Three-dimensional structure of tropism-switching Bordetella bacteriophage. PNAS, 2010, 107(9): 4347-4452. DOI:10.1073/pnas.0915008107 |
| [36] | Witte S, Zinsli LV, Gonzalez-Serrano R, Matter CI, Loessner MJ, Van Mierlo JT, Dunne M. Structural and functional characterization of the receptor binding proteins of Escherichia coli O157 phages EP75 and EP335. Computational and Structural Biotechnology Journal, 2021, 19: 3416-3426. DOI:10.1016/j.csbj.2021.06.001 |
| [37] | Cuervo A, Fàbrega-Ferrer M, Machón C, Conesa JJ, Fernández FJ, Pérez-Luque R, Pérez-Ruiz M, Pous J, Vega MC, Carrascosa JL, Coll M. Structures of T7 bacteriophage portal and tail suggest a viral DNA retention and ejection mechanism. Nature Communications, 2019, 10(1): 3746. DOI:10.1038/s41467-019-11705-9 |
| [38] | Garcia-Doval C, Van Raaij MJ. Crystallization of the C-terminal domain of the bacteriophage T7 fibre protein gp17. Acta Crystallographica Section F: Structural Biology and Crystallization Communications, 2012, 68(2): 166-171. DOI:10.1107/S1744309111051049 |
| [39] | Cornelissen A, Ceyssens PJ, Krylov VN, Noben JP, Volckaert G, Lavigne R. Identification of EPS-degrading activity within the tail spikes of the novel Pseudomonas putida phage AF. Virology, 2012, 434(2): 251-256. DOI:10.1016/j.virol.2012.09.030 |
| [40] | Guo H, Arambula D, Ghosh P, Miller JF. Diversity-generating retroelements in phage and bacterial genomes. Microbiology Spectrum, 2014, 2(6): 2-6. |
| [41] | Leavitt JC, Gogokhia L, Gilcrease EB, Bhardwaj A, Cingolani G, Casjens SR. The tip of the tail needle affects the rate of DNA delivery by bacteriophage P22. PLoS One, 2013, 8(8): e70936. DOI:10.1371/journal.pone.0070936 |
| [42] | Wang CY, Tu JG, Liu J, Molineux IJ. Structural dynamics of bacteriophage P22 infection initiation revealed by cryo-electron tomography. Nature Microbiology, 2019, 4(6): 1049-1056. DOI:10.1038/s41564-019-0403-z |
| [43] | Olia AS, Prevelige PE, Johnson JE, Cingolani G. Three-dimensional structure of a viral genome-delivery portal vertex. Nature Structural & Molecular Biology, 2011, 18(5): 597-603. |
| [44] | Andres D, Hanke C, Baxa U, Seul A, Barbirz S, Seckler R. Tailspike interactions with lipopolysaccharide effect DNA ejection from phage P22 particles in vitro. Journal of Biological Chemistry, 2010, 285(47): 36768-36775. DOI:10.1074/jbc.M110.169003 |
| [45] | Barbirz S, Müller JJ, Uetrecht C, Clark AJ, Heinemann U, Seckler R. Crystal structure of Escherichia coli phage HK620 tailspike: podoviral tailspike endoglycosidase modules are evolutionarily related. Molecular Microbiology, 2008, 69(2): 303-316. DOI:10.1111/j.1365-2958.2008.06311.x |
| [46] | Zaccheus MV, Broeker NK, Lundborg M, Uetrecht C, Barbirz S, Widmalm G. Structural studies of the O-antigen polysaccharide from Escherichia coli TD2158 having O18 serogroup specificity and aspects of its interaction with the tailspike endoglycosidase of the infecting bacteriophage HK620. Carbohydrate Research, 2012, 357: 118-125. DOI:10.1016/j.carres.2012.05.022 |
| [47] | Broeker NK, Gohlke U, Müller JJ, Uetrecht C, Heinemann U, Seckler R, Barbirz S. Single amino acid exchange in bacteriophage HK620 tailspike protein results in thousand-fold increase of its oligosaccharide affinity. Glycobiology, 2012, 23(1): 59-68. |
| [48] | Simpson DJ, Sacher JC, Szymanski CM. Exploring the interactions between bacteriophage-encoded glycan binding proteins and carbohydrates. Current Opinion in Structural Biology, 2015, 34: 69-77. DOI:10.1016/j.sbi.2015.07.006 |
| [49] | Xu JW, Wang DH, Gui M, Xiang Y. Structural assembly of the tailed bacteriophage ϕ29. Nature Communications, 2019, 10(1): 2366. DOI:10.1038/s41467-019-10272-3 |
| [50] | Aksyuk AA, Bowman VD, Kaufmann B, Fields C, Klose T, Holdaway HA, Fischetti VA, Rossmann MG. Structural investigations of a Podoviridae Streptococcus phage C1, implications for the mechanism of viral entry. PNAS, 2012, 109(35): 14001-14006. DOI:10.1073/pnas.1207730109 |
| [51] | Gebhart D, Williams SR, Scholl D. Bacteriophage SP6 encodes a second tailspike protein that recognizes Salmonella enterica serogroups C2 and C3. Virology, 2017, 507: 263-266. DOI:10.1016/j.virol.2017.02.025 |
| [52] | Zhao HY, Sequeira RD, Galeva NA, Tang L. The host outer membrane proteins OmpA and OmpC are associated with the Shigella phage Sf6 virion. Virology, 2011, 409(2): 319-327. DOI:10.1016/j.virol.2010.10.030 |
| [53] | Kęsik-Szeloch A, Drulis-Kawa Z, Weber-Dąbrowska B, Kassner J, Majkowska-Skrobek G, Augustyniak D, Lusiak-Szelachowska M, Zaczek M, Górski A, Kropinski AM. Characterising the biology of novel lytic bacteriophages infecting multidrug resistant Klebsiella pneumoniae. Virology Journal, 2013, 10: 100. DOI:10.1186/1743-422X-10-100 |
| [54] | Latka A, Lemire S, Grimon D, Dams D, Maciejewska B, Lu T, Drulis-Kawa Z, Briers Y. Engineering the modular receptor-binding proteins of Klebsiella phages switches their capsule serotype specificity. Mbio, 2021, 12(3): e00455-21. |
| [55] | Majkowska-Skrobek G, Latka A, Berisio R, Squeglia F, Maciejewska B, Briers Y, Drulis-Kawa Z. Phage-borne depolymerases decrease Klebsiella pneumoniae resistance to innate defense mechanisms. Frontiers in Microbiology, 2018, 9: 2517. DOI:10.3389/fmicb.2018.02517 |
| [56] | Brzozowska E, Pyra A, Pawlik K, Janik M, Górska S, Urbańska N, Drulis-Kawa Z, Gamian A. Hydrolytic activity determination of tail tubular protein A of Klebsiella pneumoniae bacteriophages towards saccharide substrates. Scientific Reports, 2017, 7(1): 18048. DOI:10.1038/s41598-017-18096-1 |
| [57] | Fortuna MA, Barbour MA, Zaman L, Hall AR, Buckling A, Bascompte J. Coevolutionary dynamics shape the structure of bacteria-phage infection networks. Evolution; International Journal of Organic Evolution, 2019, 73(5): 1001-1011. DOI:10.1111/evo.13731 |
| [58] | Laanto E, Mäkelä K, Hoikkala V, Ravantti JJ, Sundberg LR. Adapting a phage to combat phage resistance. Antibiotics, 2020, 9(6): 291. DOI:10.3390/antibiotics9060291 |
| [59] | Azam AH, Tanji Y. Bacteriophage-host arm race: an update on the mechanism of phage resistance in bacteria and revenge of the phage with the perspective for phage therapy. Applied Microbiology and Biotechnology, 2019, 103(5): 2121-2131. DOI:10.1007/s00253-019-09629-x |
| [60] | Latino L, Midoux C, Vergnaud G, Pourcel C. Investigation of Pseudomonas aeruginosa strain PcyII-10 variants resisting infection by N4-like phage Ab09 in search for genes involved in phage adsorption. PLoS One, 2019, 14(4): e0215456. DOI:10.1371/journal.pone.0215456 |
| [61] | Dekel-Bird NP, Sabehi G, Mosevitzky B, Lindell D. Host-dependent differences in abundance, composition and host range of cyanophages from the Red Sea. Environmental Microbiology, 2015, 17(4): 1286-1299. DOI:10.1111/1462-2920.12569 |
| [62] | Avrani S, Wurtzel O, Sharon I, Sorek R, Lindell D. Genomic island variability facilitates Prochlorococcus– virus coexistence. Nature, 2011, 474(7353): 604-608. DOI:10.1038/nature10172 |
| [63] | Schwartz DA, Lindell D. Genetic hurdles limit the arms race between Prochlorococcus and the T7-like podoviruses infecting them. The ISME Journal, 2017, 11(8): 1836-1851. DOI:10.1038/ismej.2017.47 |
| [64] | Yehl K, Lemire S, Yang AC, Ando H, Mimee M, Torres MDT, De La Fuente-Nunez C, Lu TK. Engineering phage host-range and suppressing bacterial resistance through phage tail fiber mutagenesis. Cell, 2019, 179(2): 459-469.e9. DOI:10.1016/j.cell.2019.09.015 |
| [65] | Peng H, Chen IA. Phage engineering and the evolutionary arms race. Current Opinion in Biotechnology, 2021, 68: 23-29. DOI:10.1016/j.copbio.2020.09.009 |
 2022, Vol. 62
2022, Vol. 62