中国科学院微生物研究所,中国微生物学会
文章信息
- 王飞, 潘灵婷, 钱敏桦, 蔡汝倩, 林威, 洪斌鑫, 杨佳豪, 李靓, 李登峰, 童贻刚. 2022
- WANG Fei, PAN Lingting, QIAN Minhua, CAI Ruqian, LIN Wei, HONG Binxin, YANG Jiahao, LI Liang, LI Dengfeng, TONG Yigang.
- 新型淡水微囊藻噬藻体vB_MweS-yong2的分离与基因组分析
- Isolation and genome analysis of a new freshwater Microcystis cyanophage vB_MweS-yong2
- 微生物学报, 62(10): 3784-3800
- Acta Microbiologica Sinica, 62(10): 3784-3800
-
文章历史
- 收稿日期:2022-02-16
- 修回日期:2022-04-13
- 网络出版日期:2022-06-07
2. 北京化工大学生命科学与技术学院,北京 100029
2. College of Life Science and Technology, Beijing University of Chemical Technology, Beijing 100029, China
水华是淡水水体中浮游生物尤其是蓝藻(又名蓝细菌,cyanobacteria)异常增殖并高度聚集于水面,引起水体变色的一种灾害性生态现象[1]。随着社会经济的迅速发展,水体富营养化日益普遍和严重,导致蓝藻水华频繁、广泛地发生,使水生生物大量死亡,且一些蓝藻的代谢产物藻毒素具有肝毒性、神经毒性、生殖毒性、遗传毒性和肿瘤促进作用,造成饮用水安全风险;藻毒素可在水生生物中富集,造成水产品食用安全风险。蓝藻水华已成为世界性的环境难题。我国的蓝藻水华已经达到了非常严重的地步,十余年来,太湖[2]、巢湖[3]、滇池[4]等频繁地暴发大面积水华,甚至长江、黄河也在近年来出现了蓝藻藻华问题。解决蓝藻水华造成的环境问题刻不容缓。
当前,蓝藻水华治理常用手段主要有物理法和化学法。物理手段主要有曝气、人工换水、过滤打捞、超声波除藻等[5]。物理法需要消耗大量的人力与物力,成本高、效率低。化学法主要是利用金属元素抑制蓝藻的繁殖[6],或用化学杀藻剂、絮凝剂杀除蓝藻[7]。化学法“好坏通杀”,过量、繁多的化学物质进入水体容易对水环境造成破坏,危害有益微生物和水生动植物。相较于物理和化学法,生物法更为环保,符合环境友好的原则,已成为研究热点。生物法主要包括以下几种方法:一是利用水生高等植物对氮、磷等的吸收进行治理;二是利用滤食生物对藻类的捕食和消化来进行治理;三是利用微藻间的化感拮抗作用调节水体中的微藻种类;四是利用微生物(细菌或病毒)直接或间接控制水华藻类[8]。水生高等植物生长相对缓慢,不能有效杀抑蓝藻,难以高效快速净化水体;蓝藻细胞常聚集成大的群落,超过滤食尺寸,难滤食,且细胞群落外被胶被,难消化,因而较少被纤毛虫、轮虫、枝角类、桡足类和虑食性鱼类食用,采用滤食法很难得到快速有效的控制效果;高温季节,蓝藻较其他微藻在富营养化水体中具有显著的生态优势,其分泌的藻毒素对其他水生生物具有化感拮抗或毒性作用,难以利用微藻间拮抗作用来有效控制蓝藻水华。噬藻体(cyanophage)是特异性侵染蓝藻的病毒,在调控生物生产量、藻类多样性及结构演替、生物地球化学循环和调节水生生态系统中微生物个体之间的基因转移等方面起到非常重要的作用[7]。烈性噬藻体可以选择性地快速裂解蓝藻而不感染其他生物,可快速调控藻相,被认为是极具潜力的蓝藻水华生物防控因子[9‒10]。噬藻体已成为水生微生物、生态领域的研究热点。不同的噬藻体具有不同的基因组特性和生物学特性[11‒12]。噬藻体的生物学特性很大程度决定于基因组特性。噬藻体的分离与基因组测定是噬藻体研究及其应用生态学研究的基础。
以往,有关噬藻体的分离与基因组分析的研究报道主要集中于海洋噬藻体,其宿主聚焦于聚球藻(Synechococcus)和原绿球藻(Prochlorococcus)[7]。淡水噬藻体的研究远远滞后于海洋噬藻体,已报道的淡水噬藻体少之又少。在GenBank等数据库已报道的近250株噬藻体的基因组中,仅18株来自淡水水体,其中微囊藻噬藻体仅6株,分别为MaMV-DC、Ma-LMM01、Mic1、vB_MelS-Me-ZS1、Mae-Yong924-1和PhiMa05[13–17]。
惠氏微囊藻(Microcystins wesenbergii)是一种分布广泛的、常见的产毒蓝藻[18]。我国的淡水水体中常常含有大量的惠氏微囊藻,它与水华微囊藻(M. fles-quae)、铜绿微囊藻(M. aeruginosa)被并称为三大水华蓝藻,是水华水体中最常见的优势藻种之一,经常引起大面积的蓝藻水华暴发[19]。然而,上述6种淡水微囊藻噬藻体均为铜绿微囊藻、华美微囊藻或未定种微囊藻的噬藻体,迄今尚无惠氏微囊藻噬藻体的研究报道。本研究以惠氏微囊藻为指示菌,运用双层琼脂平板法,从淡水中分离获得了一株噬藻体,将其命名为vB_MweS-yong2。
迄今为止,在已报道的绝大部分噬藻体基因组中,ORF可以被NCBI等数据库注释的不超过50%,其他ORF只能被注释为假设蛋白(hypothetical protein)基因[20]。例如,从新加坡实龙岗水库中分离出来的蓝藻噬藻体PA-SR01中有166个ORF,其中仅有47个功能可注释,占28%[21]。分离自波罗的海的噬藻体vB_AphaS-CL131基因组所含149个ORF中仅34个的功能可被注释,占23%[22]。分离自巢湖的侵染铜绿微囊藻的长尾噬藻体Mic1基因组的98个ORF中仅36个蛋白的功能被注释,占37%[23]。
噬藻体基因组全序列的测定对于噬藻体的生物学特性分析、分类鉴定、应用开发及其与宿主间相互作用关系研究具有重要的意义。随着分子生物学技术的不断发展,各种分子生物学手段已逐渐运用到噬藻体的研究中。噬藻体遗传多样性研究中常用的靶基因是g20基因[24]、光合基因和DNA聚合酶基因。在部分噬藻体中存在一些特异性基因,如MazG基因和g91基因等。但是由于噬藻体种类很多,并没有统一的保守序列,尤其是淡水噬藻体,往往缺乏海洋噬藻体中常见的核心基因。如科学家们发现88%的海洋噬藻体中存在光合基因[25],而淡水噬藻体却罕见光合相关基因;再如g20基因主要存在于肌尾噬藻体。迄今,尚未找到适用于所有噬藻体的靶基因。此外,由于噬菌体与宿主间长期互作,可能会获得一些辅助代谢基因(AMGs),如抗生素抗性基因、毒力因子基因等,这些基因的存在与表达有损于噬菌体的应用安全性。再者,近年来,科学家们惊讶地发现地球水生浮游病毒资源极其丰富,具有巨大的研发价值[12]。综上所述,对淡水噬藻体进行基因组测定与分析意义重大。
本研究针对新分离的惠氏微囊藻噬藻体vB_MweS-yong2,提取核酸、构建测序文库、测定了其全基因组序列,通过生物信息学分析对其进行注释,未发现抗生素抗性基因、毒力因子基因;通过系统发生分析讨论了其分类学地位。本研究填补了惠氏微囊藻噬藻体研究空白,为今后研究该噬藻体及其功能基因以及治理以惠氏微囊藻为优势种引起的水华问题奠定了基础。
1 材料与方法 1.1 实验藻种惠氏微囊藻(M. wesenbergii) FACHB-1112,购自中国科学院淡水藻种库。
1.2 培养基BG11液体培养基(配方见表 1);BG11半固体培养基:含0.75% (W/V)琼脂的BG11培养基;BG11固体培养基:含1.5% (W/V)琼脂的BG11培养基。
| Ingredients | Concentration/(g/L) |
| NaNO3 | 1.500 |
| K2HPO4·3H2O | 0.040 |
| MgSO4·7H2O | 0.075 |
| CaCl2·2(H2O) | 0.036 |
| C6H8O7 | 0.006 |
| C7H13FeNO7 | 0.006 |
| EDTA | 0.001 |
| Na2CO3 | 0.020 |
| H3BO3 | 0.002 860 |
| MnCl2·H2O | 0.001 810 |
| ZnSO4·7H2O | 0.000 222 |
| CuSO4·5H2O | 0.000 079 |
| Na2MoO4·2H2O | 0.000 390 |
| CoH12N2O12 | 0.000 049 |
1.3 藻种培养
将M. wesenbergii FACHB-1112藻种接种在BG11液体培养基中,放置于25 ℃的控温光照培养箱中培养,光照强度为2 000 lx,光暗周期为12 h/12 h。待藻生长到OD680值为0.6–0.8 (对数生长期)时,用于后续实验。
1.4 水样采集及处理2019年3月10日从浙江宁波月湖公园内开阔水域水样(北纬29.868 094°,东经121.543 259°),经低温冷藏转运到实验室后,立即对水样进行处理。首先4 ℃、10 000×g离心20 min去除沉淀,再取上清依次经0.80 μm和0.45 μm孔径的微孔滤膜过滤。取滤液进行后续实验。
1.5 噬藻体富集往锥形瓶中加入80 mL上述处理好的水样滤液、20 mL的5× BG11培养基和20 mL对数期M. wesenbergii FACHB-1112藻液,混匀,置于光照培养箱中培养,每天手动摇藻2–3次。同时用纯水代替水样上清液与藻液混合作为对照组。感染组藻液裂解黄化后,将其经10 000×g离心10 min,取上清依次经0.45 μm和0.22 μm孔径的硝酸纤维素滤膜过滤。取滤液加到2–10倍体积的对数生长期的FACHB-1112藻液中混匀,培养至藻液出现黄化。
1.6 噬藻体纯化制备1:20–1:40倍的浓缩藻细胞悬液:取对数期FACHB-1112藻液,10 000×g离心10–20 min,保留相当于原体积1:20–1:40的上清,悬浮沉淀。
将高温灭菌的BG11半固体培养基分装成8 mL/管,置于42 ℃的恒温水浴锅中30 min,至半固体培养基内温度恒定。取由1.5方法获得的噬藻体富集液,用BG11液体培养基做系列梯度稀释(10–1–10–6);取各稀释液按体积比1:9的比例添加到0.9 mL浓缩靶标藻液FACHB-1112中,混匀,置于转速70 r/min的摇床中,25 ℃孵育20–30 min,使病毒充分吸附到藻细胞;取出半固体培养基,分别将上述噬藻体-藻混合液倒入各管,用旋涡振荡器振荡3–5 s使其充分混匀后,倒至含1.5%琼脂的BG11培养基底层平板上,凝固后封口,封口膜上戳孔,倒置于培养箱中培养。培养条件为2 000 lx,25 ℃,光照周期为12 h/12 h。每日观察。实验组于10–15 d长出清晰、透明的噬藻斑。
挖取独立的噬藻斑,悬浮于500 μL的BG11培养基中,放置于4 ℃冰箱过夜或25 ℃避光孵育1 h,使其彻底释放出噬藻体,12 000×g离心10 min,取上清即得到第一代噬藻体。将其以体积比1:9与对数期FACHB-1112藻液混匀,置于光照培养箱中培养,黄化后,取藻裂解液离心、过滤(条件同1.5所述),10倍梯度稀释。取10–6稀释液按体积比1:9的比例添加到0.9 mL浓缩靶标藻液FACHB-1112中,按照上述流程制备双层琼脂平板,获得第二代清晰、透亮的噬藻斑后,挖取独立的噬藻斑重复上述操作,制作第3代噬藻斑。如此重复操作,直至得到大小、形态均一的噬藻斑,挖取最后一代噬藻斑至新鲜的对数期FACHB-1112藻液中培养,待藻液黄化后,取藻裂解液于10 000×g离心10 min,取上清,依次经0.45 μm和0.22 μm孔径的硝酸纤维素滤膜过滤。取滤液加到9倍体积的对数生长期FACHB-1112藻液中混匀,培养至藻液出现黄化,如此逐级扩大培养。将藻裂解液于4 ℃避光保存备用。
1.7 噬藻体基因组提取 1.7.1 噬藻体DNA提取取上述噬藻体保存液30 mL于10 000×g离心10 min,取上清,依次经0.45 μm和0.22 μm硝酸纤维素滤膜过滤。往噬藻体滤液中加入Dnase Ⅰ和RNase A至终浓度1 μg/mL,37 ℃过夜消化,80 ℃灭活15 min。向体系中加入裂解液(0.5% SDS,50 μg/mL蛋白酶K,20 nmol/L EDTA,均为终浓度),56 ℃孵育1 h。加入等体积平衡酚,温和振荡后4 ℃、10 000×g离心5 min。收集上层液体,加入等体积酚-氯仿-异戊醇(25:24:1),温和振荡后4 ℃、10 000×g离心5 min。收集上层液体,加入等体积氯仿,充分混匀后10 000×g离心5 min,收集上层液体,重复2次。加入等体积异丙醇,‒20 ℃放置至少30 min,4 ℃、10 000×g离心20 min,沉淀用75%乙醇洗2次。将核酸沉淀用去离子水重悬。
1.7.2 噬藻体的基因组文库构建使用试剂盒NEBNext Ultra Ⅱ DNA Library Prep Kit for Illumina (#E7645)按照说明书构建基因组文库。步骤包括:基因组片段化;末端修复;添加测序接头;磁珠片段筛选;添加Index、PCR扩增;PCR产物纯化。
1.8 噬藻体的全基因组测序与拼接上机测序:按照Agilent High sensitivity DNA Kit 2100说明书鉴定纯化后PCR产物的片段分布。通过实时荧光定量PCR进行定量,根据上机需求量将DNA样品进行混合,最后加入NaOH使DNA变性为单链。使用Illumina PE300试剂盒,在Illumina MiSeq上进行测序。
序列拼接:运用FastQC (http://www.bioinformatics.babraham.ac.uk/projects/fastqc/)对测序数据进行质量评估,然后用Trimmomatic v0.36软件去除低质量值的测序reads (Q-value < 20),使用SPAdes v3.13.0软件对过滤后的数据进行拼接。
基因组末端分析:利用基因组的高通量测序结果分析噬藻体末端序列。首先,将各reads截成20个碱基的序列片段,并按出现频率由高到低进行排列。然后,将高通量测序原始数据与拼接好的全基因组进行比对。查看是否存在测序深度明显高于其上下游的区域。若高频序列位于高测序深度区域,该高频序列所在区域即为噬藻体基因组的末端[26]。
1.9 生物信息学分析首先用在线工具RAST (Rapid Annotation using Subsystem Technology,http://rast.nmpdr.org)对vB_MweS-yong2基因组进行初步注释。然后,所有的ORF均使用NCBI (National Center for Biotechnology Information)的蛋白比对工具BLASTp (Protein BLAST,https://blast.ncbi.nlm.nih.gov/Blast.cgi)进行手动验证(E-value < 0.5);并进一步使用Hhpred (https://toolkit.tuebingen.mpg.de/#/tools/hhpred) (参数设定为posbility > 96%)、使用HMMER (https://www.ebi.ac.uk/Tools/hmmer/search/hmmscan) (以domain完整、且E-value < 0.5为基准),进一步校正ORF注释结果。分别在CARD数据库(http://arpcard.mcmaster.ca)和VFDB数据库(http://www.mgc.ac.Cn/VFs/main.htm)中搜索噬菌体是否含有毒力因子和抗生素抗性基因。运用在线工具PASC (http://www.ncbi.nlm.nih.gov/sutils/pasc/)分析全基因组核苷酸序列相似度。从国际病毒分类委员会(the International Committee on Taxonomy of Viruses,ICTV)中有尾噬菌体目(Caudovirales)下的14个科中选取52个代表种,从GenBank数据库下载其基因组序列,和vB_MweS-yong2及引言所述的6个微囊藻噬藻体、其他5株淡水噬藻体的基因组一起用于进化树的构建。所述的有尾目14科是Myoviridae、Ackermannviridae、Herelleviridae、Chaseviridae、Podoviridae、Autographiviridae、Siphoviridae、Demerecviridae、Drexlerviridae、Zobellviridae、Schitoviridae、Salasmaviridae、Guelinviridae和Rountreeviridae。运用在线软件ViPTree1.9 (https://www.genome.jp/viptree),基于这些病毒的全基因组构建蛋白谱树(proteomic tree)。运用MEGA 11软件采用neighbor-joining法构建噬藻体DNA聚合酶的系统发育树。
2 结果与分析 2.1 噬藻体的分离和鉴定从2019年3月10号取自浙江宁波月湖公园内月湖开阔水域的水样中,分离到一株惠氏微囊藻噬藻体,该噬藻体能在一周内使惠氏微囊藻藻液裂解黄化。将该噬藻体命名为微囊藻噬藻体Microcystis phage vB_MweS-yong2,简称为vB_MweS-yong2。
2.2 噬藻体的全基因组分析噬藻体vB_MweS-yong2的基因组全长为44 530 bp,G+C含量为71.6%。测序深度为333.9×。在高通量测序数据中,未发现高频率的reads,噬藻体vB_MweS-yong2的基因组可能没有固定的末端。已将vB_MweS-yong2的完整基因组序列提交并保存在GenBank中,接收号为OM681334。将惠氏微囊藻噬藻体vB_MweS-yong2全基因组进行BLASTn比对,结果GenBank数据库中与其相似度最高的序列是噬菌体vB_RieM_PPF1的基因组(GenBank:KJ746502.1),相似度为80.1%,但是同源覆盖度显示为0%,表明二者只有极少的序列有相似性。结果显示vB_MweS-yong2的基因组序列很新颖,在现有数据库中没有与其高度同源的病毒。
噬藻体vB_MweS-yong2的基因组含有61个预测的开放阅读框(ORF) (表 2)。利用tRNAscan-SE (http://lowelab.ucsc.edu/tRNAscan-SE)在线软件预测,发现其含有1个tRNA。CARD和VFDB分析,没有发现已知的毒力因子和抗生素耐药基因,显示了该噬藻体在基因水平上的安全性。
| ORF | Size/aa | Prediction function | Top BLASTp hita | Identityb/aa | E valuec |
| 1 | 210 | Terminase small subunit | ref|TCR69703.1|hypothetical protein EV560_101100 [Bosea sp. BK604] | 48% (95/197) | 8.00E−46 |
| 2 | 683 | Terminase large subunit | ref|WP_126112365.1|MULTISPECIES: phage terminase large subunit family protein [unclassified Bosea] | 65% (437/674) | 0.0 |
| 3 | 67 | Head-to-tail joining protein W (GpW) | ref|WP_192641905.1|unnamed protein product [Bosea sp. OAE506] | 64% (45/70) | 4.00E−22 |
| 4 | 497 | Portal protein | ref|WP_126112360.1|MULTISPECIES: phage portal protein [unclassified Bosea] | 64% (315/490) | 0.0 |
| 5 | 666 | Major head protein | ref|WP_131852925.1|HK97 family phage prohead protease [Bosea sp. BK604] | 65% (429/663) | 0.0 |
| 6 | 109 | Phage recombinase | ref|WP_192641902.1|DUF2190 family protein [Bosea sp. OAE506] |
63% (69/109) | 3.00E−36 |
| 7 | 53 | Hypothetical protein | No nits | ||
| 8 | 197 | Minor tail protein | ref|WP_043240993.1|hypothetical protein [Bosea sp. LC85] |
63% (123/196) | 8.00E−72 |
| 9 | 135 | Phage head-tail attachment | ref|WP_126112348.1|MULTISPECIES: hypothetical protein [unclassified Bosea] | 44% (60/135) | 1.00E−32 |
| 10 | 256 | Tail tube terminator protein | ref|WP_131852917.1|hypothetical protein [Bosea sp. BK604] |
50% (129/259) | 6.00E−58 |
| 11 | 64 | Sheath terminator protein | ref|WP_131311513.1|hypothetical protein [Siculibacillus lacustris] | 51% (29/57) | 2.00E−05 |
| 12 | 501 | Tail sheath protein | ref|WP_052514267.1|hypothetical protein [Bosea sp. LC85] |
49% (246/497) | 7.00E−140 |
| 13 | 127 | Tail tube protein | ref|WP_192641896.1|unnamed protein product [Bosea sp. OAE506] |
59% (75/127) | 5.00E−47 |
| 14 | 128 | Tail assembly protein | ref|WP_043240981.1|hypothetical protein [Bosea sp. LC85] |
53% (62/116) | 7.00E−33 |
| 15 | 672 | Tape measure protein | ref|WP_043240979.1|phage tail tape measure protein [Bosea sp. LC85] | 51% (351/683) | 0.0 |
| 16 | 395 | DNA circularization protein | ref|WP_126112325.1|MULTISPECIES: DNA circularization N-terminal domain-containing protein [unclassified Bosea] | 48% (193/399) | 2.00E−103 |
| 17 | 367 | Baseplate hub protein gp44 | ref|WP_131852901.1|hypothetical protein [Bosea sp. BK604] |
58% (217/373) | 3.00E−136 |
| 18 | 155 | Mu-like prophage protein gp45 | ref|WP_192641887.1|unnamed protein product [Bosea sp. OAE506] |
58% (95/163) | 2.00E−59 |
| 19 | 175 | Baseplate protein W | ref|WP_149133418.1|phage GP46 family protein [Bosea sp. F3-2] | 68% (119/175) | 5.00E−79 |
| 20 | 371 | Baseplate protein gp47 | ref|PZR94178.1|hypothetical protein DI537_08800 [Pseudomonas stutzeri] | 62% (227/369) | 4.00E−152 |
| 21 | 209 | Baseplate protein gp48 | ref|WP_047576487.1|DUF2313 domain-containing protein [Methylobacterium sp. ZNC0032] | 59% (120/205) | 3.00E−78 |
| 22 | 510 | Hypothetical protein | ref|WP_131852891.1|hypothetical protein [Bosea sp. BK604] |
37% (33/90) | 4.00E−29 |
| 23 | 68 | Hypothetical protein | No nits | ||
| 24 | 113 | L-shaped tail fiber protein | ref|WP_149821310.1|hypothetical protein [Salinarimonas sp. BN140002] | 39% (45/116) | 1.00E−16 |
| 25 | 1 038 | Carbohydrate esterase | ref|WP_141985777.1|hypothetical protein [Sphingomonas trueperi] | 36% (312/864) | 5.00E−108 |
| 26 | 132 | Hypothetical protein | ref|WP_088582296.1|hypothetical protein [Brevundimonas vesicularis] | 47% (64/136) | 3.00E−25 |
| 27 | 197 | Hypothetical protein | ref|WP_068295584.1|hypothetical protein [Labrys sp. WJW] |
36% (66/182) | 4.00E−07 |
| 28 | 286 | Lysozyme | ref|WP_161676058.1| N-acetylmuramoyL-L-alanine amidase [Pannonibacter sp. XCT-34] | 65% (90/138) | 4.00E−57 |
| 29 | 89 | Hypothetical protein | No nits | ||
| 30 | 95 | Hypothetical protein | ref|MBC7148083.1|hypothetical protein [Rhizobium sp.] |
33% (36/109) | 3.00E−08 |
| 31 | 377 | Integrase | ref|WP_139333738.1|hypothetical protein [Bosea sp. TND4EK4] |
48% (184/384) | 6.00E−103 |
| 32 | 81 | Hypothetical protein | ref|WP_074257963.1|hypothetical protein [Vannielia litorea] |
38% (25/65) | 0.01 |
| 33 | 76 | Hypothetical protein | No nits | ||
| 34 | 72 | Hypothetical protein | ref|WP_063703598.1|hypothetical protein [Bradyrhizobium centrolobii] | 59% (34/58) | 4.00E−14 |
| 35 | 80 | Hypothetical protein | No nits | ||
| 36 | 173 | Endonuclease YncB | ref|WP_152015703.1|MULTISPECIES: thermonuclease family protein [unclassified Bosea] | 50% (74/147) | 2.00E−33 |
| 37 | 133 | Pyrophosphohydrolase | ref|WP_047580566.1|DUF550 domain-containing protein [Methylobacterium sp. ZNC0032] | 87% (89/102) | 1.00E−57 |
| 38 | 365 | Hypothetical protein | ref|WP_138423692.1|phage Gp37/Gp68 family protein [Maritimibacter sp. LZ-17] | 60% (231/384) | 4.00E−147 |
| 39 | 219 | HNH endonuclease | ref|WP_133572294.1|HNH endonuclease [Maritalea mobilis] | 59% (124/210) | 3.00E−87 |
| 40 | 92 | Hypothetical protein | ref|WP_172227729.1|hypothetical protein [Mesorhizobium sp. NZP2077] | 66% (51/77) | 7.00E−27 |
| 41 | 186 | Hypothetical protein | ref|PZR82554.1|hypothetical protein DI537_36405 [Pseudomonas stutzeri] | 33% (48/147) | 7.00E−09 |
| 42 | 612 | Chromosome segregation protein | ref|OJV06223.1|hypothetical protein BGO20_08175 [Bosea sp. 67-29] | 36% (222/615) | 7.00E−66 |
| 43 | 134 | DNA gyrase inhibitor YacG | ref|SEE60114.1|hypothetical protein SAMN05519104_6687 [Rhizobiales bacterium GAS188] | 56% (51/91) | 3.00E−23 |
| 44 | 46 | Hypothetical protein | No nits | ||
| 45 | 76 | Hypothetical protein | No nits | ||
| 46 | 157 | Hypothetical protein | No nits | ||
| 47 | 95 | Hypothetical protein | No nits | ||
| 48 | 88 | Hypothetical protein | ref|WP_146237129.1|helix-turN-helix domain-containing protein [Ensifer sp. AP48] | 47% (26/55) | 5.00E−04 |
| 49 | 136 | Hypothetical protein | ref|WP_072715508.1|SHOCT domain-containing protein [Rhodothermus profundi] | 27% (27/100) | 8.00E−08 |
| 50 | 198 | Repressor protein C | ref|WP_141098457.1|helix-turN-helix transcriptional regulator [Rhodoblastus acidophilus] | 51% (99/195) | 2.00E−50 |
| 51 | 91 | Transcriptional regulator | ref|WP_113892636.1|helix-turN-helix transcriptional regulator [Roseiarcus fermentans] | 52% (29/56) | 1.00E−07 |
| 52 | 168 | Regulatory protein CII | ref|WP_068285753.1|hypothetical protein [Labrys sp. WJW] | 50% (75/150) | 8.00E−39 |
| 53 | 392 | DNA polymerase Ⅲ holoenzyme | ref|WP_112310243.1|DNA polymerase Ⅲ subunit beta [Pseudogemmobacter bohemicus] | 34% (130/381) | 3.00E−36 |
| 54 | 138 | Hypothetical protein | ref|WP_186419180.1|DUF2312 domain-containing protein [Bosea sp. CS1GBMeth4] | 86% (66/77) | 2.00E−37 |
| 55 | 216 | Hydrolases of HD superfamily | ref|RYE48308.1|hypothetical protein EOP24_07825 [Rhizobiales bacterium] | 42% (44/106) | 4.00E−12 |
| 56 | 234 | Modification methylase CviRI | ref|WP_047571326.1|hypothetical protein [Mesorhizobium sp. F7] | 58% (126/218) | 1.00E−75 |
| 57 | 203 | Hypothetical protein | No nits | ||
| 58 | 100 | Hypothetical protein | No nits | ||
| 59 | 151 | Type Ⅱ restriction endonuclease | ref|MBA3715646.1|HNH endonuclease [Pyrinomonadaceae bacterium] | 58% (32/55) | 4.00E−10 |
| 60 | 382 | Pyocin large subunit | ref|WP_103873760.1|hypothetical protein [Bosea lathyri] | 38% (150/394) | 3.00E−59 |
| 61 | 221 | Transcription termination/antitermination protein NusG | ref|WP_066878108.1|hypothetical protein [Sinorhizobium saheli] | 35% (55/156) | 8.00E−13 |
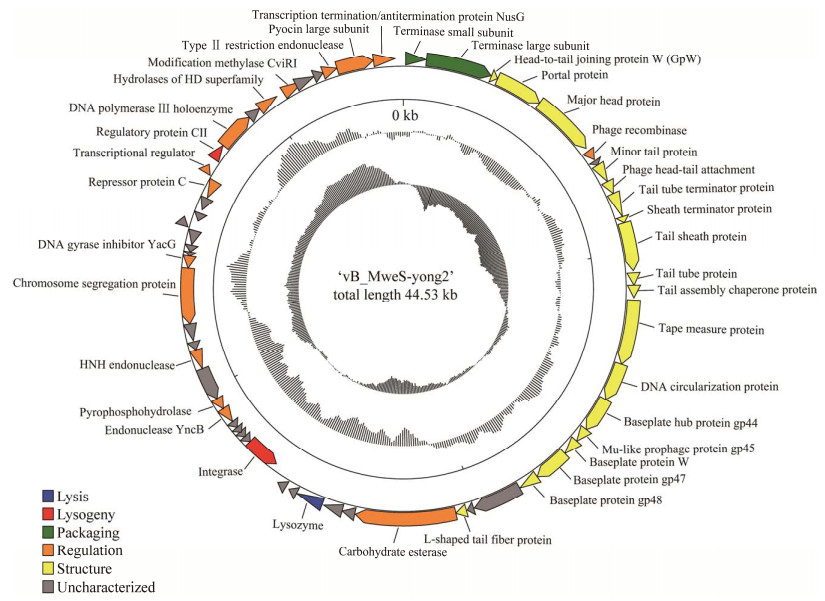
利用RAST、BLASTp、Hhpred、HMMER对噬藻体vB_MweS-yong2编码的ORF注释,发现噬藻体vB_MweS-yong2中有38个ORF可以注释为已知功能基因,约占总ORF的62.3%;余下的23个ORF,约占总ORF的37.7%,只能注释为未知功能蛋白或假设蛋白(表 2)。而对噬藻体vB_MweS-yong2注释的所有功能蛋白进行分类,则主要涉及到5个功能模块:DNA复制与调控(ORFs:6,36,39,42,43,50,51,53,55,56,59,60,61)、结构(ORFs:3,4,5,8,9,10,11,12,13,14,15,16,17,18,19,20,21,24)、包装(ORFs:1,2)、溶原(ORFs:31,52),以及与宿主溶解相关的蛋白(ORF28)。噬藻体vB_MweS-yong2各ORF的大小、预测的功能、BLASTp比对结果、方向等详情汇总于表 2和图 1。
|
| 图 1 微囊藻噬藻体vB_MweS-yong2的基因组图谱 Figure 1 Genome map of Microcystis cyanophage vB_MweS-yong2. The outermost circle represents 61 ORFs encoded in the genome, with different colors represent different functions (clockwise arrow indicates the forward reading frame, counterclockwise arrow indicates the reverse reading frame); the dark circles in the middle represent the GC content ("Black" indicates greater than the average GC content compared with the whole genome, and "Gray" indicates the opposite); the innermost circle represents the GC skew (G–C/G+C: outwards indicates > 0 and inwards indicates < 0). |
在DNA复制和调控模块中,ORF1和ORF2分别编码噬菌体末端酶小亚基(terminase small subunit)和大亚基(terminase large subunit),主要功能是在DNA易位、切割和包装中起作用[27]。ORF6编码重组酶(phage recombinase),在DNA双链断裂修复中起着重要作用[28]。ORF36编码核酸内切酶YncB同源蛋白(endonuclease YncB)。ORF39和ORF59均编码HNH核酸内切酶(HNH endonuclease)。自然界中已经有超过1 000种含有HNH结构域的蛋白被发现,位点特异性的归巢核酸内切酶是HNH蛋白中数量最多的。HNH核酸内切酶广泛分布于细菌、古菌、真核生物、病毒(包括噬菌体)中,具有切割核酸的活性。噬菌体的HNH核酸内切酶在噬菌体的生命周期中发挥着很重要的作用[29]。ORF42被预测为编码染色体分离蛋白质(chromosome segregation protein)。ORF43被预测编码DNA促旋酶抑制剂YacG (DNA gyrase inhibitor YacG)。YacG有抑制促旋酶活性的作用[30]。ORF50和ORF51编码转录抑制因子(repressor protein C、transcriptional regulator),转录抑制因子可以使病毒能够较长时间潜伏而不被宿主发现[31]。ORF53编码DNA聚合酶Ⅲ (DNA polymerase Ⅲ holoenzyme)。ORF55编码HD超家族水解酶(hydrolases of HD superfamily),可以用来水解磷酸酯键,可能与DNA修复有关[32]。ORF56编码甲基化酶(modification methylase CviRI),能够保护噬藻体DNA不被宿主核酸内切酶切割[33]。ORF60编码Pyocin大亚基(Pyocin large subunit),功能是参与次生代谢物的生物合成、运输和分解代谢[34]。ORF61编码转录终止/抗终止蛋白NusG (transcription termination/antitermination protein NusG)。在细菌中转录和翻译是耦合过程,耦合受转录因子NusG和NusA的调节,NusG被认为是桥接RNA聚合酶(RNAP)和核糖体的桥梁[35]。
噬菌体结构蛋白基因模块中,ORF3被注释为编码头尾连接蛋白GpW (head-to-tail joining protein W)。ORF4编码噬藻体门户蛋白(portal protein),portal通常形成一个孔或者门户,使得DNA能够在包装和弹出的过程中顺利通过,它还在噬藻体衣壳和尾巴蛋白之间形成连接。ORF5被注释为编码噬藻体头部主结构蛋白(major head protein)。ORF8编码噬藻体小尾蛋白(minor tail protein)。ORF9编码噬藻体头尾附件(phage head-tail attachment)。ORF10编码噬藻体尾管蛋白(tail tube terminator protein),该蛋白在尾部组装中起着重要作用。ORF11编码噬藻体鞘终止蛋白(sheath terminator protein)。ORF12编码噬藻体尾鞘蛋白(tail sheath protein)。ORF13编码噬藻体尾管蛋白(tail tube protein)。ORF14编码噬藻体尾部组装伴侣蛋白(tail assembly protein)。ORF15编码噬藻体卷尺蛋白(tape measure protein)。ORF16编码噬藻体DNA环化蛋白(DNA circularization protein)。ORF17、ORF18、ORF19、ORF20、ORF21编码噬藻体基板蛋白。ORF8、ORF9、ORF10、ORF11、ORF12、ORF13、ORF14、ORF15、ORF16、ORF17、ORF18、ORF19、ORF20、ORF21、ORF24均编码噬菌体尾部蛋白。
噬菌体溶解与溶原功能模块中,ORF28预测为编码溶菌酶(lysozyme),溶菌酶可以切割细菌细胞壁中的酰胺键,并可与T7 RNA聚合酶结合并抑制其转录[36]。ORF31预测为编码DNA整合酶(integrase),整合酶参与噬菌体基因组整合到宿主染色体中或从宿主染色体中切除、游离出来的过程[37]。ORF52编码噬菌体调节蛋白CII,调节蛋白CII是DNA结合蛋白,参与溶原菌的建立[38]。在本研究中,vB_MweS-yong2对惠氏微囊藻FACHB-1112呈现裂解特性,而非溶原性,这可能与培养条件有关,具体机制有待深入研究。
此外,不少研究发现噬藻体基因组存在一些与宿主新陈代谢相关的同源基因,被称为“辅助代谢基因” (AMGs)[39],可协助微生物驱动生物地球化学循环[40–42]。AMGs主要影响宿主蓝藻的光合作用和磷酸戊糖途径等[43]。如噬藻体P-SSP7中含有负责编码光合系统Ⅱ核心蛋白D1和D2的psbA和psbD基因[44],噬藻体P-SSM2中含有负责编码磷酸盐调节蛋白的phoH基因[45]以及S-CBS2含有可以保护宿主免受强光损伤的hli基因[46]。在噬藻体vB_MweS-yong2中同样也含有AMGs,其中ORF25编码碳水化合物酯酶(carbohydrate esterase),表明病毒可能通过复杂多糖的生物分解直接调控碳循环,揭示了病毒在有机碳分解中的重要作用。ORF37编码焦磷水解酶(pyrophosphohydrolase),该酶能催化三磷酸腺嘌呤脱氧核苷酸(dATP)/三磷酸鸟嘌呤脱氧核苷酸(dGTP)水解为焦磷酸和腺嘌呤脱氧核苷酸(dAMP)/鸟嘌呤脱氧核苷酸(dGMP),使得宿主细胞中的dATP浓度降低,从而更容易地来进行噬菌体DNA合成[47]。
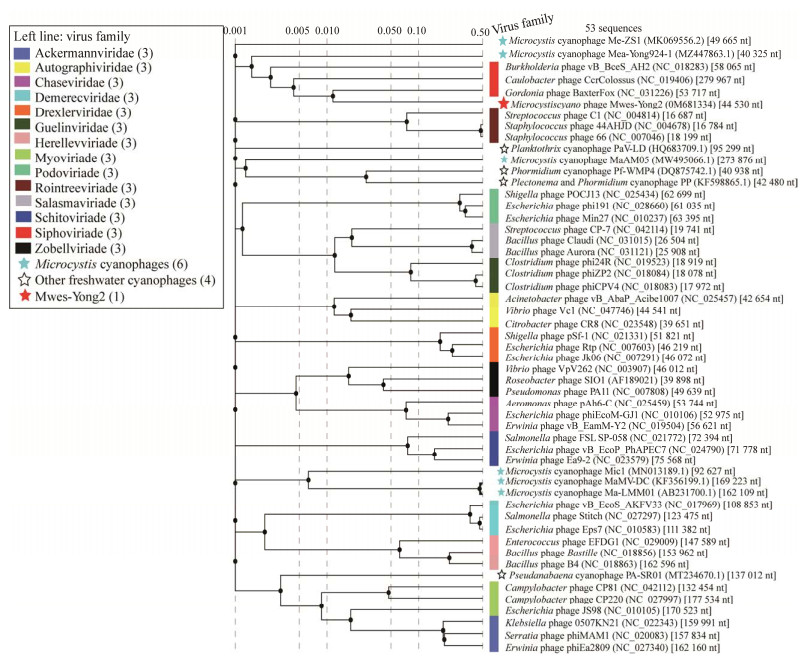
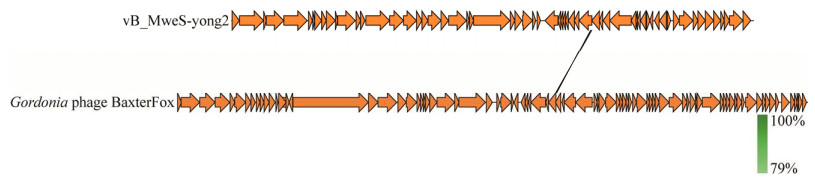
2.3 噬藻体vB_MweS-yong2的系统进化分析国际病毒分类委员会(ICTV)的细菌、古菌病毒分会(Bacterial and Archaeal Viruses Subcommittee,BAVS)关于噬菌体属的界定标准是与其他所有已分类噬菌体的核苷酸序列相似度大于或等于50%,如果低于此值则说明所比对的噬菌体代表一个新的属[48]。PASC (http://www.ncbi.nlm.nih.gov/sutils/pasc/)是ICTV推荐的用于分析核苷酸序列相似度的工具,PASC比对表明vB_MweS-yong2与所有已知噬菌体间的全基因组核苷酸序列相似度最高值为20.21%,这个值远低于50%的属边界值。vB_MweS-yong2代表一个新的属。从ICTV中有尾噬菌体目(Caudovirales)下的14个科中选取42个代表种,从GenBank数据库下载这42个代表种及引言所述的5个微囊藻噬藻体、其他5株淡水噬藻体的基因组序列,运用在线软件ViPTree1.9,基于这些病毒和vB_MweS-yong2的全基因组构建了系统发育进化树proteomic tree (图 2)。结果显示vB_MweS-yong2与长尾科Siphoviridae的成员聚在一起,与Gordonia的噬菌体BaxterFox同源关系最为接近,表明vB_MweS-yong2属于长尾科。vB_MweS-yong2与Gordonia phage BaxterFox间的同源性非常低(图 3),其平均核酸一致性(average nucleotide identity,ANI)值为−1,DNA分子原位杂交值(in silico DNA-DNA hybridization,isDDH value)为0。综上,vB_MweS-yong2在长尾科中代表一个独立、新颖的新属。
|
| 图 2 基于全基因组的proteome tree Figure 2 Phylogenetic proteomic tree of vB_MweS-yong2, 6 reported Microcystis cyanophages, 4 other fresh cyanophages and 42 classified phages of the 14 families. |
|
| 图 3 vB_MweS-yong2和Gordonia phage BaxterFox的基因组比较 Figure 3 Genome comparison of the vB_MweS-yong2 and Gordonia phage BaxterFox. |
DNA聚合酶通常较为保守,也常用作病毒进化分析工具。基于16种噬藻体的DNA聚合酶序列比对,运用MEGA 11软件的neighbor-joining算法构建了DNA聚合酶的系统发育树(图 4)。系统发育树显示噬藻体DNA聚合酶的系统发育与噬藻体的系统分类地位并无耦合关系,噬藻体DNA聚合酶的系统发育与宿主的分类地位并无耦合关系。此外,海水与淡水噬藻体的DNA聚合酶并没有分开成簇,表明噬藻体DNA聚合酶的进化并不一定存在地域隔离。
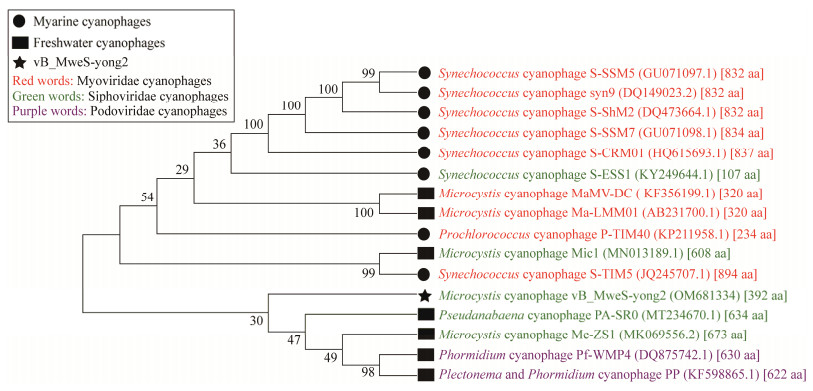
|
| 图 4 噬藻体DNA聚合酶的系统发育树 Figure 4 Phylogenetic tree of cyanophage DNA polymerases. |
3 讨论与结论
惠氏微囊藻噬藻体vB_MweS-yong2的基因组中的基因依其功能而处于不同区域,具有典型的模块化特征。vB_MweS-yong2的基因组序列非常新颖,基于全基因组的BLASTn比对中同源覆盖度为0%、PASC比对显示其与现有病毒序列相似度最高值仅为20.21%,远低于50%的属边界值,进化树中,vB_MweS-yong2虽与长尾科病毒聚在一起,却与其他长尾科病毒有着长远的进化距离。总之,vB_MweS-yong2序列新颖,进化上代表着一个新的噬菌体属,丰富了噬菌体知识体系。
常规的病毒系统进化分析往往借助1–2个保守基因如噬菌体的gp20、DNA聚合酶、末端酶大亚基等的序列比对而构建系统进化树。国际病毒分类委员会(ICTV)曾长期使用末端酶大亚基序列比对作为噬菌体分类的重要依据,由于病毒的高度多样性和不保守性,近年来国际病毒分类委员会推荐[49]使用基于全基因组比对的进化分析如PASC分析和proteomic tree分析,并在此基础上将有尾目3个科改为14个科加1个未分科的属。PASC和proteomic tree是新型原核病毒系统进化分析的有力工具,基于二者所作的分析结果往往较基于单一基因所做的分析更加贴合国际病毒分类委员会的分类界定。本研究所建的噬藻体DNA聚合酶系统发育树中,海水与淡水噬藻体的DNA聚合酶并没有分开成簇。噬藻体DNA聚合酶的进化关系与噬藻体本身的分类地位相矛盾。在Šulčius等基于末端酶大亚基构建的噬藻体系统进化树中,海水与淡水噬藻体的末端酶大亚基也没有按海水和淡水来源分开成簇[22],同时肌尾、长尾和短尾科噬藻体亦没有按照系统分类地位分别成簇,而是各科相互穿插。本课题组前期基于末端酶大亚基构建的噬藻体系统进化树中,这3个科的噬藻体也没有规律性地依据分类地位聚集,而是各科犬牙交错[14]。而在Šulčius等基于全基因组构建的系统进化树中,各科非常规律地按照ICTV的系统分类地位成簇、按照淡水与海水来源分别成簇、按照单细胞宿主和丝状宿主规律成簇。在此,我们呼吁在进行噬藻体系统进化分析中,尽量使用全基因组同源性比较分析法,而非单一基因的比对。
如引言所述,已发现的绝大部分噬藻体基因组中的ORF可以被NCBI等数据库注释的不超50%。本研究中,虽然vB_MweS-yong2序列新颖,却有62.3%的ORF被注释出功能。这可能与本研究中综合运用了多种注释工具有关。
如引言所述,惠氏微囊藻是水体中常见的蓝藻,是常见的水华优势藻,对其进行有效的控制,便可能控制以惠氏微囊藻为优势藻的蓝藻水华暴发。此前,有关惠氏微囊藻噬藻体的分离及基因组的研究报道处于空白状态。引言所述的已被报道基因组序列的6个微囊藻噬藻体中,MaMV-DC、Ma-LMM01、Mic1和Mae-Yong924-1的指示菌是铜绿微囊(M. aeruginosa);vB_MelS-Me-ZS1的指示菌是华美微囊藻(M. elabens);PhiMa05的指示菌是一种未定种的微囊藻(Microcystis sp.)。虽然Mic1最先被报道是使用惠氏微囊藻FACHB-1339株为指示藻分离获得的,后续文献却做了订正,指出该指示藻实为铜绿微囊藻[23]。本研究分离鉴定的淡水微囊藻噬藻体vB_MweS-yong2弥补了有关惠氏微囊藻噬藻体研究的空白,同时因为惠氏微囊藻是淡水水体藻华中常见的优势藻,本研究也为蓝藻水华治理提供了潜在的研发应用材料。在vB_MweS-yong2基因组中没有发现已知的毒力因子和抗生素耐药基因,这为该噬藻体的应用安全性奠定了基础。
噬藻体是以蓝藻为宿主的病毒类群,它在水体中的广泛分布以及对蓝藻种群的丰度和密度的调控作用使它可以成为藻类水华治理的潜在力量,然而有关噬藻体的研究仍处于初级阶段。如果今后的研究中能够分离出更多的噬藻体,并对其进行研究,以及拓宽噬藻体的宿主范围,噬藻体必将会在环境治理领域大放异彩。
| [1] |
Wang YP, Guo X, Xie R, Ji CH, Yu J. Evaluation of the effect of H2O2 on controlling cyanobacteria bloom in a small water body. Pearl River, 2019, 40(11): 95-98.
(in Chinese) 王永平, 郭萧, 谢瑞, 姬昌辉, 于剑. 过氧化氢消除小型水体蓝藻水华的效果评价. 人民珠江, 2019, 40(11): 95-98. DOI:10.3969/j.issn.1001-9235.2019.11.017 |
| [2] |
Fang AH, Huang YZ, Qian J. Causes and harmful effects of blue-green algae disaster in Taihu lake and control measures introduction. Water Purification Technology, 2008, 27(3): 70-72.
(in Chinese) 方爱红, 黄银芝, 钱瑾. 浅谈太湖蓝藻暴发的原因、危害及其预防治理. 净水技术, 2008, 27(3): 70-72. DOI:10.3969/j.issn.1009-0177.2008.03.020 |
| [3] |
Liu W. The water quality analysis of lake Chaohu when the cyanobacteria was occurred. Environmental Monitoring in China, 2007, 23(4): 101-102.
(in Chinese) 刘伟. 巢湖蓝藻重度发生时的水质特征分析. 中国环境监测, 2007, 23(4): 101-102. DOI:10.3969/j.issn.1002-6002.2007.04.031 |
| [4] |
Liu LP. Characteristics of blue algal bloom in Dianchi lake and analysis on its cause. Research of Environmental Sciences, 1999, 12(5): 36-37.
(in Chinese) 刘丽萍. 滇池水华特征及成因分析. 环境科学研究, 1999, 12(5): 36-37. DOI:10.3321/j.issn:1001-6929.1999.05.010 |
| [5] |
Chen HL, Li Y, Chu ZS, Ye BB, Li GH. Present situation and research progress of ultrasonic algae control technology. Journal of Environmental Engineering Technology, 2020, 10(1): 72-78.
(in Chinese) 陈贺林, 李芸, 储昭升, 叶碧碧, 李国宏. 超声波控藻技术现状及研究进展. 环境工程技术学报, 2020, 10(1): 72-78. |
| [6] | Jayme-Torres G, Hansen AM. Nutrient loads in the river mouth of the Río Verde Basin in Jalisco, Mexico: how to prevent eutrophication in the future reservoir?. Environmental Science and Pollution Research International, 2018, 25(21): 20497-20509. DOI:10.1007/s11356-017-0334-2 |
| [7] | Lee TA, Rollwagen-Bollens G, Bollens SM, Faber-Hammond JJ. Environmental influence on cyanobacteria abundance and microcystin toxin production in a shallow temperate lake. Ecotoxicology and Environmental Safety, 2015, 114: 318-325. DOI:10.1016/j.ecoenv.2014.05.004 |
| [8] |
Chen SW, Mao T, Yuan KP, Yu ZH. Advances in the treatment of blooms. Journal of Yangtze University: Natural Science Edition, 2014, 11(35): 69-73.
(in Chinese) 陈识文, 毛涛, 袁科平, 余知和. 水华治理方法研究进展. 长江大学学报: 自科版, 2014, 11(35): 69-73. |
| [9] | Jassim SAA, Limoges RG. Impact of external forces on cyanophage-host interactions in aquatic ecosystems. World Journal of Microbiology & Biotechnology, 2013, 29(10): 1751-1762. |
| [10] | Gerphagnon M, MacArthur DJ, Latour D, Gachon CMM, Van Ogtrop F, Gleason FH, Sime-Ngando T. Microbial players involved in the decline of filamentous and colonial cyanobacterial blooms with a focus on fungal parasitism. Environmental Microbiology, 2015, 17(8): 2573-2587. DOI:10.1111/1462-2920.12860 |
| [11] | Proctor LM, Fuhrman JA. Viral mortality of marine bacteria and cyanobacteria. Nature, 1990, 343(6253): 60-62. DOI:10.1038/343060a0 |
| [12] | Zhang QY, Gui JF. Diversity, evolutionary contribution and ecological roles of aquatic viruses. Science China Life Sciences, 2018, 61(12): 1486-1502. DOI:10.1007/s11427-018-9414-7 |
| [13] |
Zhou K, Du K, Zhu J, Zhou CZ, Li Q. Isolation and characterization of cyanophages from the pond in the west campus of University of Science and Technology of China. Microbiology China, 2020, 47(10): 3161-3170.
(in Chinese) 周科, 杜康, 朱洁, 周丛照, 李琼. 也西湖噬藻体的分离与鉴定. 微生物学通报, 2020, 47(10): 3161-3170. DOI:10.13344/j.microbiol.china.200304 |
| [14] | Lin W, Li DF, Sun ZT, Tong YG, Yan XJ, Wang CL, Zhang X, Pei GQ. A novel freshwater cyanophage vB_MelS-Me-ZS1 infecting bloom-forming cyanobacterium Microcystis elabens. Molecular Biology Reports, 2020, 47(10): 7979-7989. DOI:10.1007/s11033-020-05876-8 |
| [15] | Jaskulska A, Mankiewicz-Boczek J. Cyanophages specific to cyanobacteria from the genus Microcystis. Ecohydrology & Hydrobiology, 2020, 20(1): 83-90. |
| [16] | Naknaen A, Suttinun O, Surachat K, Khan E, Pomwised R. A novel jumbo phage PhiMa05 inhibits harmful Microcystis sp. Frontiers in Microbiology, 2021, 12: 660351. DOI:10.3389/fmicb.2021.660351 |
| [17] | Qian MH, Li DF, Lin W, Pan LT, Liu WC, Zhou Q, Cai RQ, Wang F, Zhu JQ, Tong YG. A novel freshwater cyanophage, mae-Yong924-1, reveals a new family. Viruses, 2022, 14(2): 283. DOI:10.3390/v14020283 |
| [18] |
He JW, Li LP, Yu JL, Zhao YJ, Liu YD. Preliminary studies on a Chinese new record of blue-green algae-Microcystis wesenbergii and its toxicity. Acta Hydrobiologica Sinica, 1996, 20(2): 192-194.
(in Chinese) 何家菀, 李络平, 俞家禄, 赵以军, 刘永定. 我国产毒微囊藻的新发现——惠氏微囊藻及其毒性的初步研究. 水生生物学报, 1996, 20(2): 192-194. |
| [19] |
Wang J, Shi Y, Liu Q, Li Z, Zhang M, Xie SL. Taxonomic and molecular phylogenetics of bloom-forming algae from the Taiyuan section of Fenhe river, China. Journal of Lake Sciences, 2018, 30(5): 1332-1342.
(in Chinese) 王捷, 石瑛, 刘琪, 李砧, 张猛, 谢树莲. 汾河太原河段水华藻类分类及分子系统研究. 湖泊科学, 2018, 30(5): 1332-1342. |
| [20] | Clokie MRJ, Millard AD, Letarov AV, Heaphy S. Phages in nature. Bacteriophage, 2011, 1(1): 31-45. DOI:10.4161/bact.1.1.14942 |
| [21] | Zhang D, You F, He YL, Te SH, Gin KYH. Isolation and characterization of the first freshwater cyanophage infecting Pseudanabaena. Journal of Virology, 2020, 94(17): e00682-20. |
| [22] | Šulčius S, Šimoliūnas E, Alzbutas G, Gasiūnas G, Jauniškis V, Kuznecova J, Miettinen S, Nilsson E, Meškys R, Roine E, Paškauskas R, Holmfeldt K. Genomic characterization of cyanophage vB_AphaS-CL131 infecting filamentous diazotrophic cyanobacterium Aphanizomenon flos-aquae reveals novel insights into virus-bacterium interactions. Applied and Environmental Microbiology, 2019, 85(1): e01311-18. |
| [23] | Yang F, Jin H, Wang XQ, Li Q, Zhang JT, Cui N, Jiang YL, Chen YX, Wu QF, Zhou CZ, Li WF. Genomic analysis of Mic1 reveals a novel freshwater long-tailed cyanophage. Frontiers in Microbiology, 2020, 11: 484. DOI:10.3389/fmicb.2020.00484 |
| [24] | Baena Lozada LP, Hoppert M, Hertel R. Phage vB_BmeM-Goe8 infecting Bacillus megaterium DSM319. Archives of Virology, 2020, 165(2): 515-517. DOI:10.1007/s00705-019-04513-5 |
| [25] | 韩颖. 一株噬藻体的全基因组测序及CO2浓度和温度同步升高对集胞藻6803紫外损伤修复基因转录的影响. 湖北工业大学硕士学位论文, 2017. |
| [26] | Li SS, Fan H, An XP, Fan HH, Jiang HH, Chen YB, Tong YG. Scrutinizing virus genome termini by high-throughput sequencing. PLoS One, 2014, 9(1): e85806. DOI:10.1371/journal.pone.0085806 |
| [27] | Duffy C, Feiss M. The large subunit of bacteriophage λ's terminase plays a role in DNA translocation and packaging termination. Journal of Molecular Biology, 2002, 316(3): 547-561. DOI:10.1006/jmbi.2001.5368 |
| [28] | Leipe DD, Aravind L, Grishin NV, Koonin EV. The bacterial replicative helicase DnaB evolved from a RecA duplication. Genome Research, 2000, 10(1): 5-16. |
| [29] | 徐丹丹. 深海嗜热噬菌体GVE2 HNH核酸内切酶结构与功能的研究. 安徽大学硕士学位论文, 2017. |
| [30] | Sengupta S, Nagaraja V. YacG from Escherichia coli is a specific endogenous inhibitor of DNA gyrase. Nucleic Acids Research, 2008, 36(13): 4310-4316. DOI:10.1093/nar/gkn355 |
| [31] | Villanueva VM, Oldfield LM, Hatfull GF. An unusual phage repressor encoded by mycobacteriophage BPs. PLoS One, 2015, 10(9): e0137187. |
| [32] | Makarova KS, Aravind L, Grishin NV, Rogozin IB, Koonin EV. A DNA repair system specific for thermophilic archaea and bacteria predicted by genomic context analysis. Nucleic Acids Research, 2002, 30(2): 482-496. |
| [33] | Stefan C, Xia YN, Van Etten JL. Molecular cloning and characterization of the gene encoding the adenine methyltransferase M.CviRI from Chlorella virus XZ-6E. Nucleic Acids Research, 1991, 19(2): 307-311. |
| [34] | Lu SN, Wang JY, Chitsaz F, Derbyshire MK, Geer RC, Gonzales NR, Gwadz M, Hurwitz DI, Marchler GH, Song JS, Thanki N, Yamashita RA, Yang MZ, Zhang DC, Zheng CJ, Lanczycki CJ, Marchler-Bauer A. CDD/SPARCLE: the conserved domain database in 2020. Nucleic Acids Research, 2020, 48(D1): D265-D268. |
| [35] | Wang CY, Molodtsov V, Firlar E, Kaelber JT, Blaha G, Su M, Ebright RH. Structural basis of transcription-translation coupling. Science, 2020, 369(6509): 1359-1365. |
| [36] | Cheng X, Zhang X, Pflugrath JW, Studier FW. The structure of bacteriophage T7 lysozyme, a zinc amidase and an inhibitor of T7 RNA polymerase. PNAS, 1994, 91(9): 4034-4038. |
| [37] | Biswas T, Aihara H, Radman-Livaja M, Filman D, Landy A, Ellenberger T. A structural basis for allosteric control of DNA recombination by λ integrase. Nature, 2005, 435(7045): 1059-1066. |
| [38] | Kalionis B, Dodd IB, Egan JB. Control of gene expression in the P2-related template coliphages: Ⅲ. DNA sequence of the major control region of phage 186. Journal of Molecular Biology, 1986, 191(2): 199-209. |
| [39] | Thompson LR, Zeng QL, Kelly L, Huang KH, Singer AU, Stubbe J, Chisholm SW. Phage auxiliary metabolic genes and the redirection of cyanobacterial host carbon metabolism. PNAS, 2011, 108(39): E757-E764. |
| [40] | Anantharaman K, Duhaime MB, Breier JA, Wendt KA, Toner BM, Dick GJ. Sulfur oxidation genes in diverse deep-sea viruses. Science, 2014, 344(6185): 757-760. |
| [41] | York A. Algal virus boosts nitrogen uptake in the ocean. Nature Reviews Microbiology, 2017, 15(10): 573. |
| [42] | Roux S, Brum JR, Dutilh BE, Sunagawa S, Duhaime MB, Loy A, Poulos BT, Solonenko N, Lara E, Poulain J, Pesant S, Kandels-Lewis S, Dimier C, Picheral M, Searson S, Cruaud C, Alberti A, Duarte CM, Gasol JM, Vaqué D, Bork P, Acinas SG, Wincker P, Sullivan MB. Ecogenomics and potential biogeochemical impacts of globally abundant ocean viruses. Nature, 2016, 537(7622): 689-693. |
| [43] |
Gao EB, Ning DG. Advances in researches on cyanophage auxiliary metabolic genes. Microbiology China, 2014, 41(8): 1667-1674.
(in Chinese) 高恶斌, 宁德刚. 噬藻体辅助代谢基因(AMGs)研究进展. 微生物学通报, 2014, 41(8): 1667-1674. |
| [44] | Millard A, Clokie MRJ, Shub DA, Mann NH. Genetic organization of the psbAD region in phages infecting marine Synechococcus strains. PNAS, 2004, 101(30): 11007-11012. |
| [45] | Sullivan MB, Coleman ML, Weigele P, Rohwer F, Chisholm SW. Three Prochlorococcus cyanophage genomes: signature features and ecological interpretations. PLoS Biology, 2005, 3(5): e144. |
| [46] | Huang SJ, Wang K, Jiao NZ, Chen F. Genome sequences of siphoviruses infecting marine Synechococcus unveil a diverse cyanophage group and extensive phage-host genetic exchanges. Environmental Microbiology, 2012, 14(2): 540-558. |
| [47] | Zhou Y, Xu XX, Wei YF, Cheng Y, Guo Y, Khudyakov I, Liu FL, He P, Song ZY, Li Z, Gao Y, Ang EL, Zhao HM, Zhang Y, Zhao SW. A widespread pathway for substitution of adenine by diaminopurine in phage genomes. Science, 2021, 372(6541): 512-516. |
| [48] | Adriaenssens E, Brister JR. How to name and classify your phage: an informal guide. Viruses, 2017, 9(4): 70. |
| [49] | Adriaenssens EM, Sullivan MB, Knezevic P, Van Zyl LJ, Sarkar BL, Dutilh BE, Alfenas-Zerbini P, Łobocka M, Tong YG, Brister JR, Moreno Switt AI, Klumpp J, Aziz RK, Barylski J, Uchiyama J, Edwards RA, Kropinski AM, Petty NK, Clokie MRJ, Kushkina AI, Morozova VV, Duffy S, Gillis A, Rumnieks J, Kurtböke İ, Chanishvili N, Goodridge L, Wittmann J, Lavigne R, Jang HB, Prangishvili D, Enault F, Turner D, Poranen MM, Oksanen HM, Krupovic M. Taxonomy of prokaryotic viruses: 2018-2019 update from the ICTV bacterial and archaeal viruses subcommittee. Archives of Virology, 2020, 165(5): 1253-1260. |
 2022, Vol. 62
2022, Vol. 62