中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 任娜, 朱四东, 陈秀暖, 陈婷, 王景, 徐莹, 杨季芳, 陈吉刚. 2022
- Na REN, Sidong ZHU, Xiunuan CHEN, Ting CHEN, Jing WANG, Ying XU, Jifang YANG, Jigang CHEN. 2022
- 海水来源的548株真菌的近海及远海分布模式及其抑菌活性分析
- Distribution pattern and antibacterial activity of 548 fungi from offshore and pelagic seawater
- 微生物学报, 62(1): 374-387
- Acta Microbiologica Sinica, 62(1): 374-387
-
文章历史
- 收稿日期:2021-05-06
- 修回日期:2021-08-04
- 网络出版日期:2021-11-05
海洋独特的环境条件造就了微生物独特的代谢途径,赋予了微生物产结构新颖、功能独特的生物活性物质的能力,使海洋微生物来源的天然产物成为海洋新药开发的研究热点[1]。海洋真菌的次生代谢产物具有结构多样、生物活性丰富、“创新指数”高、产量大等特点,已经成为化学、生物学和药学等领域关注的焦点[2]。
“海洋来源”的真菌是新颖天然产物的理想来源。在过去的几十年中,从海洋来源真菌中发现了上千种新化合物或已知分子的新衍生物[3]。丝状海洋真菌,尤其是子囊菌(Ascomycetes)已被证明是新天然产物的重要来源[3]。例如,研究人员从海水浸木中分离的Aigialus parvus中鉴定出11种具有多种结构类型的新天然产物[4]。研究者从红树林潮间带木材来源的Hypoxylon oceanicum中发现了3个新的脂肽,5个新的大环聚醚和5种新的线性聚酯[5]。海洋真菌产生的新天然产物已呈现出系列生物活性。如,来源于海底泥样的Zopfiella marina能产生高活性的抗真菌代谢产物zofimarin (一种粪壳菌素衍生物)[6]。红树林来源的Aigialus parvus产生的aigialomycin D呈现出抗疟原虫活性和细胞毒性[7]。海洋浮木来源的Corollospora pulchella能产生具有CD45抑制活性的pulchellalactam[8]。一种海藻(Ceramium sp.)来源的Phaeosphaeria spartinae能产生8种新的天然产物,其中包括对人白细胞弹性蛋白酶有抑制活性的spartinoxide和spartinol C[9]。上述实例说明专性海洋真菌可以来源于各种海洋基质,并可以产生独特的未被发现的天然产物[10]。
虽然近年来新发现的海洋来源真菌活性次生代谢产物的数目持续增加,但这些产活性产物的真菌大都属于具有耐渗/耐盐能力的且与腐殖质相关的种属,如曲霉属(Aspergillus)和青霉属(Penicillium)[3, 11-13]。导致上述结果的主要原因是用于海洋来源真菌分离的方法大都以平板涂布法为主导[14]。因此,建立适合海洋真菌的高效分离策略对新颖海洋天然产物的发现十分必要。2019年,Overy等[10]撰写了海洋来源真菌分离的“最佳实践指南(best- practices guide)”,其中涉及海洋真菌的新颖分离培养方法包括膜过滤、极限稀释涂布、潮湿箱式培养、站位诱取和原位培养,并证明根据不同的样品类型选择相应的海洋真菌分离培养方法,可以显著提高海洋真菌,尤其是专性海洋真菌的种群多样性。
本研究依托大洋科考平台,采用膜过滤法对近海和远海海水样品中的真菌进行了大规模分离培养与鉴定,并对所获菌株的抑菌活性进行分析,以期挖掘尚未被发现的海洋真菌,为海洋天然产物开发提供菌种资源。
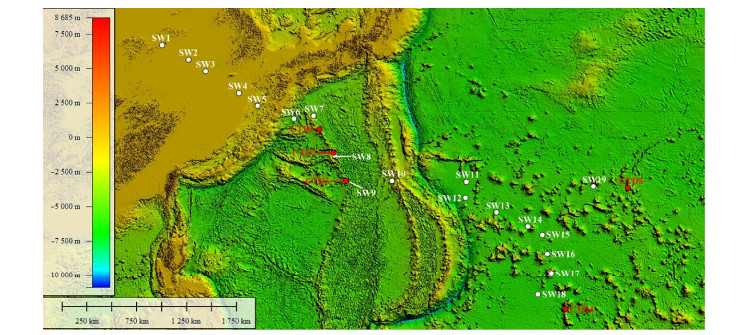
1 材料与方法 1.1 材料 1.1.1 海水样品来源:海水样品为大洋一号科考船在锚地停靠和航渡期间(大洋61航次)用无菌蜀牛瓶采集的19个表层水样(SW1-SW19),以及在科考作业区用Niskin瓶采集的5个CTD站位水样(CTD1- CTD5) (图 1)。采样点横跨浅海带(水深 < 200 m)、半深海带(水深200-2 000 m)和深海带(水深 > 2 000 m)。每个CTD站位采集的水样均包含海洋上层/真光层(从海面到水下200 m)中的5个层次(2、50、100、150、200 m),海洋中层(水深200-1 000 m)中的2个层次(500 m和1 000 m)和海洋深层(水深1 000-4 000 m)中的1个层次(1 500 m)。
|
| 图 1 海水样品站位基本信息 Figure 1 Basic site information of seawater sample. |
1.1.2 培养基:
(1) PDA培养基:200 g去皮马铃薯,切成1 cm3小块,加入800 mL蒸馏水煮沸10 min至马铃薯块熟而不烂,过滤收集滤液,加入20 g葡萄糖和15.0 g琼脂后,用陈海水定容至1 000 mL,121 ℃灭菌20 min。补加终浓度为50 mg/L氯霉素和10 mg/L链霉素。
(2) LB培养基(g/L):蛋白胨10,酵母粉5,氯化钠10,琼脂15.0,蒸馏水定容至1 000 mL,121 ℃灭菌20 min。
(3) 2216培养基(g/L):蛋白胨5,酵母粉1,磷酸铁0.01,琼脂15.0,陈海水: 人工海水: 蒸馏水=2:1:1定容至1 000 mL,121 ℃灭菌20 min。
1.1.3 靶细菌:购自海洋微生物菌种保藏中心(Marine Culture Collection of China, MCCC)的金黄色葡萄球菌(Staphylococcus aureus) MCCC1A0646;本实验室保藏的杀香鱼假单胞菌(Pseudomonas plecoglossicida) NB2011和哈维氏弧菌(Vibrio harveyi) Zj2008;浙江万里学院徐洁皓博士惠赠的嗜水气单胞菌(Aeromonas hydrophila) T4;购自中国普通微生物菌种保藏管理中心(China General Microbiological Culture Collection Center,CGMCC)的副溶血弧菌(Vibrio parahemolyticus) CGMCC1.1997。
1.1.4 主要试剂和仪器:E×Taq酶、dNTPs Mixture、10×Buffer I购自TaKaRa公司;蛋白胨、酵母粉购自OXOID公司;葡萄糖、真菌DNA提取试剂盒购自上海生工生物工程有限公司;核酸提取仪为Tissuelyser LT公司产品。
1.2 真菌的分离纯化样品采集后,于无菌环境将500 mL水样抽滤至聚碳酸脂膜(0.22 μm,Φ47 mm),待滤膜上仍保留少量海水时立刻关闭抽滤泵,并用无菌镊子将湿润的滤膜(含菌面朝上)转移至含氯霉素和链霉素的PDA培养基表面,置于20 ℃恒温培养箱培养至膜表面有菌落形成。挑取膜上不同形态的菌落,用PDA平板进行菌株分离纯化。
1.3 菌株的物种鉴定根据菌株的菌落特征,以及菌株的菌丝、孢子及产孢结构特征对分离纯化的菌株进行排重后,采用试剂盒结合机械破壁法提取菌株的总DNA,并使用引物ITS1 (5ʹ-TCCGTAGGTGAACC TGCGG-3ʹ)和ITS4 (5ʹ-TCCTCCGCTTATTGATA TGC-3ʹ) PCR扩增ITS基因序列[15]。PCR反应体系(25.0 μL)含上、下游引物各0.5 μL (10 μmol/L),10×buffer I 2.5 μL,dNTPs Mixture 2.0 μL (2.5 mmol/L),E×Taq酶0.25 μL (5 U/μL),DNA模板1.0 μL,ddH2O 18.25 μL。PCR扩增反应条件为:95 ℃ 5 min;95 ℃ 45 s,47 ℃ 45 s,72 ℃ 2 min,35个循环;72 ℃ 10 min。PCR产物经琼脂糖凝胶电泳检测后,送至北京擎科新业生物技术有限公司进行单向测序。采用Chromas软件对测序获得的ITS基因序列进行分析,去除质量不佳的测序碱基后,将获得的有效序列提交至GenBank数据库中进行在线BLAST比对。下载与对比菌株ITS基因序列同源性最高的模式菌株的ITS基因序列,将所有测序菌株及其最相近的模式菌株的ITS基因序列整合到一个Fasta文件,将Fasta文件导入到MEGA 7.0软件中,由Clustal W程序对序列进行人工校正及比对分析,然后基于MEGA 7.0软件中的邻接法(NJ)构建系统发育树。
1.4 抑菌菌株筛选本实验通过固体平板法测定形态排重后所获真菌菌株的抑菌活性。将保存于甘油管中的靶细菌接种至新鲜的LB/2216培养基上活化,37 ℃培养18 h。将平板上的单菌落接种到试管中培养一段时间后,调整菌悬液的浓度为107 CFU/mL。取2 mL菌悬液覆盖PDA培养基30 s后弃去未被吸收的菌液。将分离的真菌在PDA平板上活化至成熟单菌落,用打孔器制备成6 mm直径的菌饼接种于靶细菌平板上,20 ℃共培养4-5 d,观察结果并测量抑菌圈直径。每个样本设置2个平行重复,结果取其平均值。
2 结果与分析 2.1 可培养真菌的多样性分析本实验从54份海水样品中分离纯化获得548株真菌,根据形态学排重结果,选取其中菌落形态各异的85株菌株进行了ITS基因序列扩增与测序(GenBank登录号为MW856691- MW856775)。ITS基因序列一致性比对结果显示,只有3株真菌ITS基因序列与数据库中真菌模式菌株ITS序列的一致性<97% (93.4%-94.9%),应代表新的分类单元。其余82株真菌的ITS基因序列与数据库中已有真菌的ITS序列的一致性 > 97% (位于97.7%-100% 之间)。基于ITS基因序列同源比对分析结果,548株真菌分属于子囊菌门(Ascomycota)和担子菌门(Basidiomycota)中的24个属(表 1),其中隶属于外瓶霉属(Exophiala)的菌株数量最多(74株),其次为枝孢属(Cladosporium;68株)和拟青霉属(Simplicillium;68株),而隶属于Dichotomopilus属(1株)、Nigrograna属(1株)和Neocucurbitaria属(1株)的菌株数量最少。
| Family | Genus | Species* | Isolate |
| Sporidiobolaceae | Rhodotorula | diobovata (99.57%-100%), mucilaginosa (100%), taiwanensis (100%) | 39 |
| Aspergillaceae | Aspergillus | versicolor (100%), pachycaulis (100%), sydowii (100%) | 35 |
| Penicillium | griseofulvum (100%), oxalicum (100%), rubens (100%), chrysogenum (100%) | 50 | |
| Trichocomaceae | Talaromyces | variabilis (98.29%), juglandicola (98.08%) | 16 |
| Herpotrichiellaceae | Exophiala | xenobiotica (98.38%-100%), equina (99.77%) | 74 |
| Cystobasidiaceae | Cystobasidium | pinicola (97.82%), calyptogenae (99.8%), benthicum (99.8%-100%), minutum (100%) | 45 |
| Ustilaginaceae | Pseudozyma | rugulosus (98.45%), aphidis (99.83%) | 17 |
| Nigrogranaceae | Nigrograna | mackinnonii (98.74%) | 1 |
| Periconiaceae | Periconia | submersa (94.89%) | 4 |
| Pleosporaceae | Alternaria | alstroemeriae (100%) | 12 |
| Didymellaceae | Didymella | gardeniae (100%) | 14 |
| Cucurbitariaceae | Neocucurbitaria | irregularis (98.18%) | 1 |
| Cladosporiaceae | Cladosporium | phaenocomae (100%), sphaerospermum (100%), puyae (100%), chasmanthicola (99.8%-100%), halotolerans (99.74%-100%), oxysporum (100%) | 68 |
| Teratosphaeriaceae | Hortaea | werneckii (97.68%) | 20 |
| Mycosphaerellaceae | Phaeophleospora | hymenocallidicola (100%) | 4 |
| Sporocadaceae | Pestalotiopsis | rhododendri (99.8%) | 14 |
| Cordycipitaceae | Simplicillium | cylindrosporum (98.61%) | 68 |
| Lecanicillium | kalimantanense (93.63%) | 8 | |
| Chaetomiaceae | Botryotrichum | verrucosum (99.31%) | 5 |
| Chaetomium | globosum (99.5%-100%), nepalense (99.04%), subaffine (100%) | 12 | |
| Dichotomopilus | funicola (100%) | 1 | |
| Nectriaceae | Fusarium | concentricum (99.71%-99.76%) | 23 |
| Thyronectria | caudata (93.66%) | 3 | |
| Bionectriaceae | Clonostachys | eriocamporesii (99.75%) | 14 |
| *: The number in brackets is the highest homology between the ITS gene sequence of the isolated strain and the ITS gene of the type fungus. | |||
2.2 可培养真菌的系统发育分析
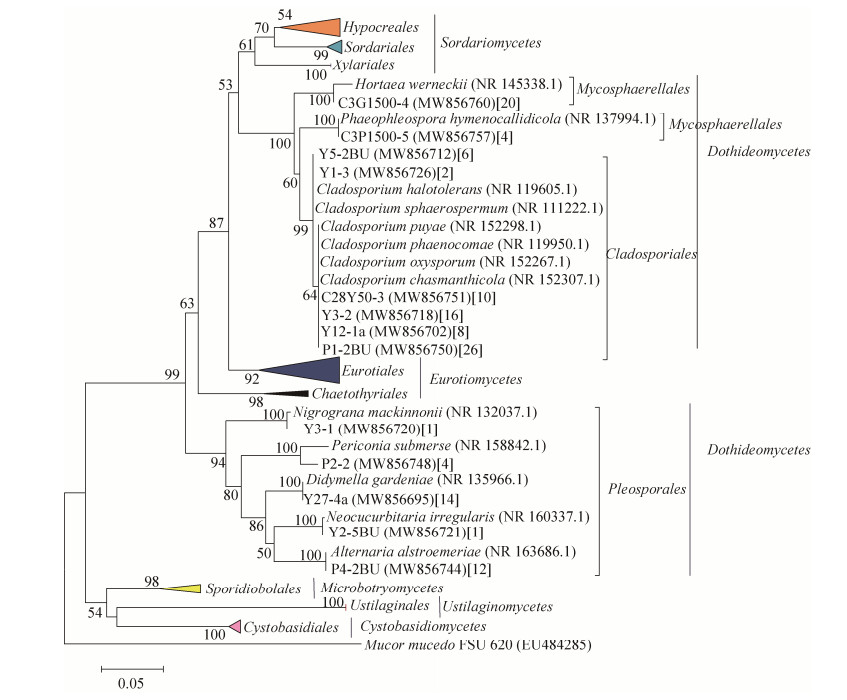
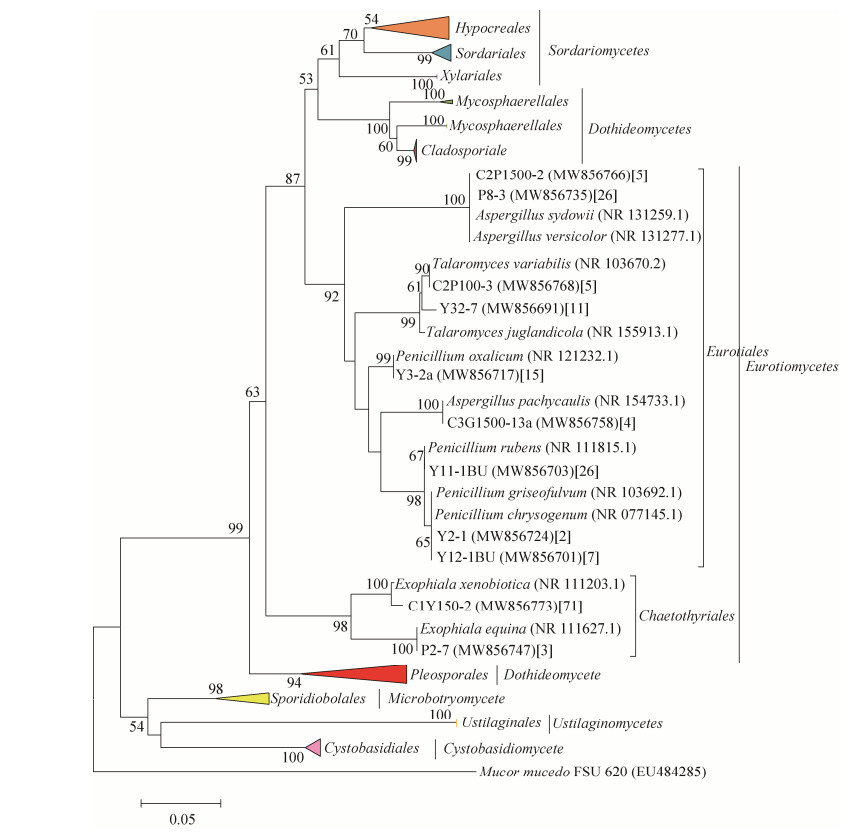
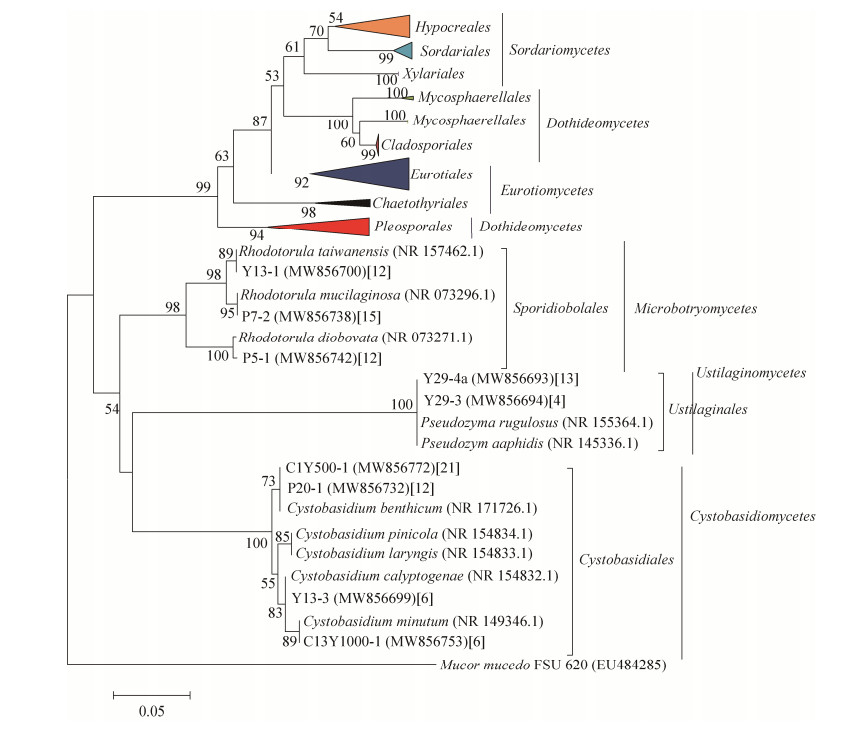
基于ITS基因序列构建的系统发育树显示,548株真菌隶属于2门6纲11目19科24属44种(图 2-5)。在纲水平上,座囊菌纲(Dothideomycetes)包含的种的数量最多(13个,占29.5%),其次为粪壳菌纲(Sordariomycetes;11个,占25%)和散囊菌纲(Eurotiomycetes;11个,占25%),而黑粉菌纲(Ustilaginomycetes)包含的种数量最少(2个,占4.5%)。
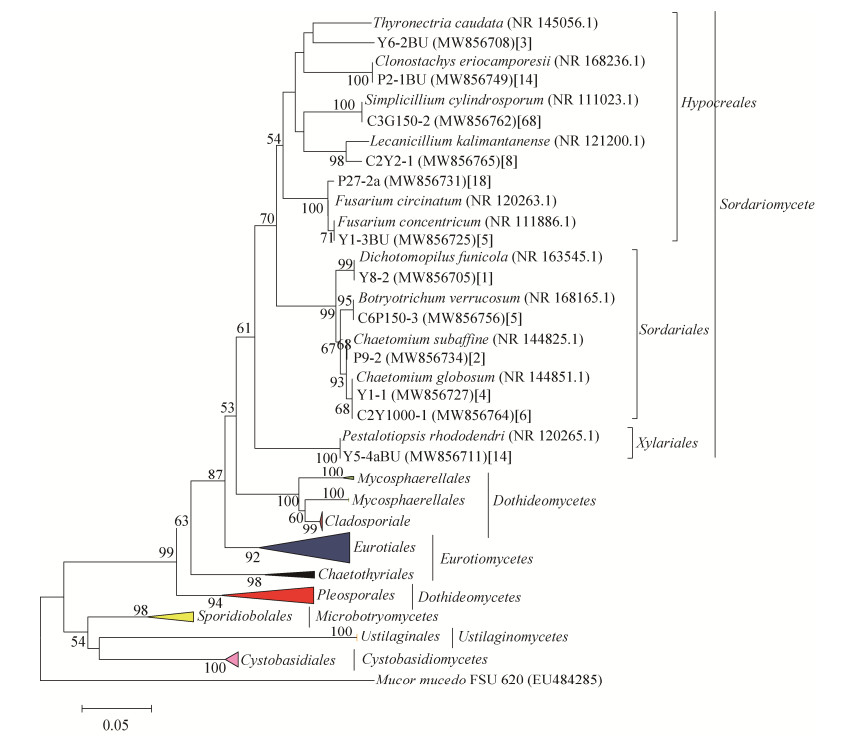
|
| 图 2 隶属于粪壳菌纲11个种真菌的ITS rDNA基因序列系统发育树 Figure 2 Neighbour-joining (NJ) tree for 11 species in Sordariomycetes based on ITS rDNA sequences. Numbers in the bracket represent the number of isolated fungal strains. |
|
| 图 3 隶属于座囊菌纲13个种真菌的ITS rDNA基因序列系统发育树 Figure 3 Neighbour-joining (NJ) tree for 13 species in Dothideomycetes based on ITS rDNA sequences. Numbers in the bracket represent the number of isolated fungal strains. |
|
| 图 4 隶属于散囊菌纲11个种真菌的ITS rDNA基因序列系统发育树 Figure 4 Neighbour-joining (NJ) tree for 11 species in Eurotiomycetes based on ITS rDNA sequences. Numbers in the bracket represent the number of isolated fungal strains. |
|
| 图 5 隶属于微球黑粉菌纲、黑粉菌纲和囊担子菌纲9个种真菌的ITS rDNA基因序列系统发育树 Figure 5 Neighbour-joining (NJ) tree for 9 species in Microbotryomycetes, Ustilaginomycetes and Cystobasidiomycetes based on ITS rDNA sequences. Numbers in the bracket represent the number of isolated fungal strains. |
2.3 可培养真菌的地理分布模式
44个种(含3个潜在新分类单元)呈现5种地理分布模式(图 6),包括:(1) 在浅海带、半深海带和深海带均有分布的15个种,包括聚多曲霉(Aspergillus sydowii)、Cystobasidium benthicum、Cladosporium chamanthicola、Clonostachys eriocamporesi、耐盐枝孢(Cladosporium halotolerans)、尖枝孢(Cladosporium oxysporum)、Cladosporium phaenocomae、异物外瓶霉(Exophiala xenobiotica)、轮纹镰刀菌(Fusarium concentricum)、产黄青霉(Penicillium chrysogenum)、Rhodotorula diobovata、胶红酵母(Rhodotorula mucilaginosa)、Rhodotorula taiwanensis、柱孢单梗霉(Simplicillium cylindrosporum)和Talaromyces juglandicola;(2)未在浅海带发现的12个种,包括Aspergillus pachycaulis、Botryotrichum verrucosum、Cystobasidium calyptogenae、Chaetomium globosum、微小囊担菌(Cystobasidium minutum)、Chaetomium subaffine、Dichotomopilus funicola、Didymella gardeniae、Hortaea werneckii、产红青霉(Penicillium rubens)、Pseudozyma rugulosus和Talaromyces variabilis,其中A. pachycaulis、C. subaffine、D. funicola仅在半深海带水样中发现,而B. verrucosum、C. minutum、P. rugulosus仅在深海带水样中发现;(3) 仅在浅海带分布的4个种和1个潜在新分类单元,包括Neocucurbitaria irregulari、Nigrograna mackinnonii、灰黄青霉(Penicillium griseofulvum)、Pestalotiopsis rhododendri和Periconia sp.;(4) 存在于浅海带和半深海带水样中,但未在深海带分布的5个种和2个潜在新分类单元,包括杂色曲霉(Aspergillus versicolor)、Chaetomium nepalense、球孢枝孢(Cladosporium sphaerospermum)、Exophiala equina、Phaeophleospora hymenocallidicola、Lecanicillium sp.和Thyronectria sp.;(5) 存在于浅海带和深海带水样中,但未在半深海带分布的5个种,包括Alternaria alstroemeriae、松树生泡囊担孢酵母(Cystobasidium pinicola)、Cladosporium puyae、Pseudozyma aphidis和草酸青霉(Penicillium oxalicum)。
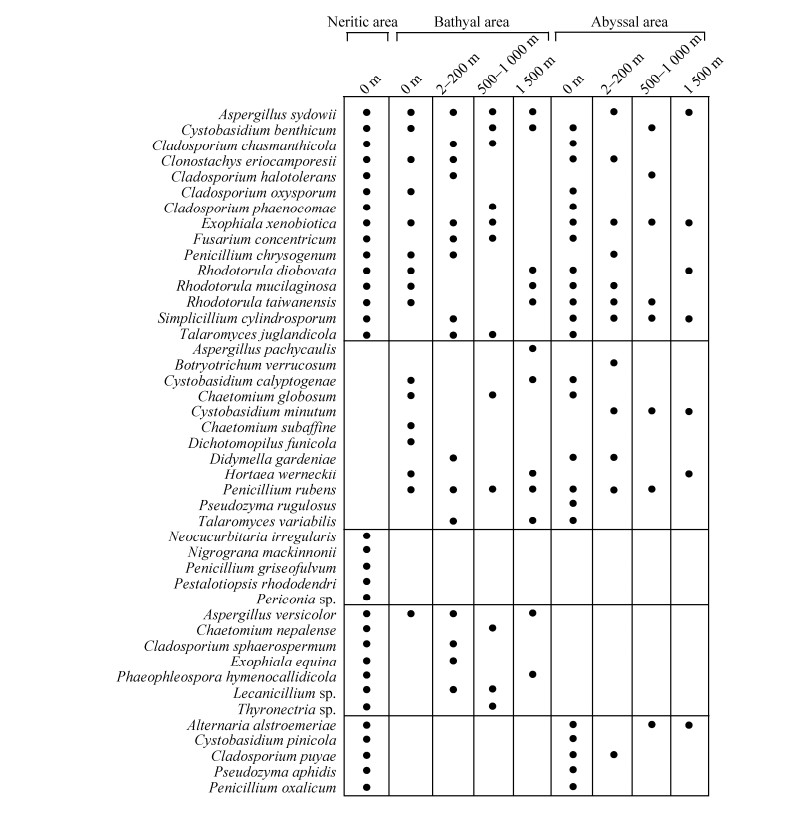
|
| 图 6 44个真菌种的地理分布模式 Figure 6 Geographical distribution patterns of 44 fungal species. Filled circles indicate that the species was isolated from the corresponding seawater samples. |
2.4 抑菌菌株筛选
利用固体平板法对隶属于24属44种的85株代表性真菌进行了抑菌活性测试,发现隶属于10属12种的真菌对5种靶细菌具有不同程度的抑菌作用(表 2)。菌株Simplicillium cylindrosporum C3G150-2不但对S. aureus MCCC1A0646具有强抑菌活性,且对P. plecoglossicida NB2011和A. hydrophila T4有不同程度抑菌作用;菌株Hortaea werneckii C3G1500-4对S. aureus MCCC1A0646、P. plecoglossicida NB2011和A. hydrophila T4均有强抑菌活性,以及对V. parahemolyticus CGMCC 1.1997和V. harveyi Zj2008有较弱抑菌活性;菌株Cladosporium halotolerans Y6-2BU和Didymella gardenia Y27-4a对5株靶细菌各有不同程度的抑菌活性;其他真菌至少对5种靶细菌中的1种有抑菌活性。
| Isolates | Indicator | ||||
| 1 | 2 | 3 | 4 | 5 | |
| Pestalotiopsis rhododendri Y5-4aBU | + | - | - | - | - |
| Penicillium oxalicum P4-2 | ++ | - | - | - | - |
| Alternaria alstroemeriae Y3-4BU | + | - | - | - | - |
| Aspergillus pachycaulis C3G1500-13a | - | ++ | - | - | - |
| Simplicillium cylindrosporum C3G150-2 | +++ | ++ | + | - | - |
| Hortaea werneckii C3G1500-4 | +++ | +++ | +++ | + | + |
| Cladosporium halotolerans C2M100-1 | ++ | ++ | - | + | + |
| Cladosporium phaenocomae C2P1000-2a | ++ | - | - | - | - |
| Cladosporium puyae C28Y50-2 | - | - | ++ | + | + |
| Didymella gardeniae Y27-4a | ++ | + | - | - | + |
| Thyronectria sp.Y6-2BU | - | ++ | + | - | - |
| Talaromyces variabilis C2P100-3 | + | - | - | - | - |
| -: no transparent circle; +: 6≤Φ<12 mm; ++: 12 mm≤Φ<18 mm; +++: Φ≥18 mm; Φ: diameter of antimicrobe circle. 1: Staphylococcus aureus MCCC1A0646; 2: Pseudomonas plecoglossicida NB2011; 3: Aeromonas hydrophila T4; 4: Vibrio parahemolyticus CGMCC1.1997; 5: Vibrio harveyi Zj2008. The results were the average of two replicates. | |||||
3 讨论与结论
本文采用膜过滤法,对采集自浅海带、半深海带和深海带的54份海水样品中的真菌进行了分离培养,共获得真菌菌株548株,它们隶属于24个属44个种(含3个潜在新分类单元),呈现出较高的多样性。基于ITS基因序列构建的系统进化树显示,大部分分离菌株与陆源真菌具有较近的亲缘关系。对本实验所获得的44个种的生态分布模式分析发现,不同种之间的地理分布模式存在较大差异,主要包括:(1) 15个种普遍存在于浅海带、半深海带和深海带的海水样品中,这暗示这些种已具备了强大的海洋环境适应能力,它们有可能在不同海域均广泛存在;(2) 12个种不存于浅海带海水样品中,其原因可能是它们是从其他海域传播至此;(3) 4个种和1个潜在分类单元仅出现于浅海带水样中,且分离频率较低(表 1),暗示这些种均为陆地来源,它们尚没有适应海洋环境,因此不能在海洋环境中有效扩散;(4) 5个种和2个潜在新分类单元存在于浅海带和半深海带水样,但不存在于深海带水样中,这暗示这些种群已经具备了向深海扩散的能力,但尚不能在远海环境中有效生存;(5) 5个种出现于浅海带和深海带表层海水样品中,甚至某些种还出现在深海带深海层水样中,但在半深海带样品中却未能发现它们的踪迹,其中原因可能与半深海带的采集点和采集的样本数量有关。
真菌的生态可塑性给海洋真菌的定义提出了重大挑战[16]。Pang等[17]提出,海洋真菌是指那些能够从海洋栖息地中被反复分离的任何真菌,并且它们能够在海洋环境中生长和/或形成孢子,或与其他海洋生物存在共生关系,或在遗传水平上显示出海洋环境适应和进化能力,或在海洋环境中具有代谢活性。2015年,Jones等[18]对海洋真菌进行了整理,统计出隶属于65个目19个科472个属中的1 112个种的海洋真菌,包括:子囊菌门(Ascomycota)中的387个属943个种、担子菌门(Basidiomycota)中的43个属96个种、壶菌门(Chytridiomycota)和相关门中的13属26种、接合菌门(Zygomycota)中的2属3种和芽枝霉门(Blastocladiomycota)中的1个属1个种。在科水平上,海壳菌科(Halosphaeriaceae)是海洋真菌的最大家族,该科包含59个属141个种,而假丝酵母属(Candida)、曲霉属和青霉属则是较大的属,分别含有64、47和39个种。以Jones等的海洋真菌统计数据为参考,本文分离获得的41个真菌种(不包含3个潜在新分类单元)中有13个种隶属于海洋种,它们是C. oxysporum、C. sphaerospermum、A. sydowii、A. versicolor、P. chrysogenum、P. griseofulvum、P. oxalicum、R. mucilaginosa、R. cladiensis、C. globosum、C. benthicum、C. minutum和P. aphidis。除上述13个海洋真菌种外,本实验室分离获得的C. halotolerans和H. werneckii也极有可能是海洋真菌种,因为近期多个团队在海洋中发现了这些种群,且这2个分离自海洋环境的种均能产生生物活性物质[19-20]。
对本实验分离的41个种和3个潜在新分类单元的抑菌实验表明,11个种和1个潜在新分类单元(Thyronectria sp. Y6-2BU)呈现出抑菌活性(表 2),其中分离自远海海水样品中的S. cylindrosporum C3G150-2和H. werneckii C3G1500-4不但具有较广的抑菌谱,且均对金黄色葡萄球菌具有较高的抑菌活性,值得深入研究。此外,虽然在13个海洋种中仅草酸青霉呈现出抑菌活性,但已有研究表明这些海洋真菌种中的大部分能产生新颖天然产物。如:分离自红树林的尖枝孢能产生抑菌活性的大环内酯类化合物[21];分离自海洋环境的杂色曲霉能产生生物碱类、蒽醌类、氧杂蒽酮类、二苯醚类、核苷衍生物类、内酯类、多肽类、聚酮化合物类、真菌生物碱类、杂色曲霉素类、萜类、水硫酚类、二酮哌嗪类、asperversiamides和indoloditerpene等20余种结构新颖的化合物[22-25];产黄青霉、灰黄青霉和草酸青霉能产生戊烯酮衍生物、羟基苯乙酸衍生物、萜烯类、混源萜类、赤藓糖醇衍生物、聚酮类、二苯甲酮类、sorbicillinoids、二萜类、多氯取代类固醇类、黄酮类、chaetoglobosins、桔霉素类似物、氧杂蒽酮衍生物、二苯醚类、脑苷脂类、苯腙和喹唑啉衍生物、色酮类、酚醛烯胺类、叠氮基酮类等二十余种结构新颖的化合物,其中某些化合物具有抗炎活性、抗病毒活性、DNA Topo I酶抑制活性、抗菌活性、抗真菌活性、抗细菌群体感应活性和抗肿瘤活性[26-27];海洋来源的球毛壳菌能产生白杨素、cytochalasans、嗜氮酮类化合物、chaetomugilins、溶菌酶和链球菌球蛋白等生物活性物质;深海沉积物来源的胶红酵母能高产类胡萝卜素[28]。
总之,本研究首次大尺度开展了近海及远海海域海水样品中真菌的分离培养,筛选获得44个真菌种,并分析了所获菌种的地理分布模式,以及筛选出了多株具有抑菌活性的真菌。这些研究结果不但加深了我们对海水可培养真菌多样性的认识,同时也丰富了海洋真菌菌种资源。
| [1] | Molinski TF, Dalisay DS, Lievens SL, Saludes JP. Drug development from marine natural products. Nature Reviews Drug Discovery, 2009, 8(1): 69-85. DOI:10.1038/nrd2487 |
| [2] |
Ma LL, Tian XP, Li GJ, Zhao YQ, Yin JP. Research status and development trends of natural products from marine microorganisms. Journal of Tropical Oceanography, 2021, 40(5): 134-146.
(in Chinese) 马丽丽, 田新朋, 李桂菊, 赵晏强, 殷建平. 海洋微生物来源天然产物研究现状与态势. 热带海洋学报, 2021, 40(5): 134-146. |
| [3] | Overy DP, Bayman P, Kerr RG, Bills GF. An assessment of natural product discovery from marine (sensu strictu) and marine-derived fungi. Mycology, 2014, 5(3): 145-167. DOI:10.1080/21501203.2014.931308 |
| [4] | Isaka M, Yangchum A, Intamas S, Kocharin K, Jones EBG, Kongsaeree P, Prabpai S. Aigialomycins and related polyketide metabolites from the mangrove fungus Aigialus parvus BCC 5311. Tetrahedron, 2009, 65(22): 4396-4403. DOI:10.1016/j.tet.2009.03.050 |
| [5] | Abbanat D, Leighton M, Maiese W, Jones EBG, Pearce C, Greenstein M. Cell wall active antifungal compounds produced by the marine fungus Hypoxylon oceanicum LL-15G256. I. taxonomy and fermentation. The Journal of Antibiotics, 1998, 51(3): 296-302. DOI:10.7164/antibiotics.51.296 |
| [6] | Chokpaiboon S, Unagul P, Nithithanasilp S, Komwijit S, Somyong W, Ratiarpakul T, Isaka M, Bunyapaiboonsri T. Salicylaldehyde and dihydroisobenzofuran derivatives from the marine fungus Zopfiella marina. Natural Product Research, 2018, 32(2): 149-153. DOI:10.1080/14786419.2017.1342083 |
| [7] | Barluenga S, Dakas PY, Ferandin Y, Meijer L, Winssinger N. Modular asymmetric synthesis of aigialomycin D, a kinase-inhibitory scaffold. Angewandte Chemie International Edition, 2006, 45(24): 3951-3954. DOI:10.1002/anie.200600593 |
| [8] | Alvi KA, Casey A, Nair BG. Pulchellalactam: a CD45 protein tyrosine phosphatase inhibitor from the marine fungus Corollospora pulchella. The Journal of Antibiotics, 1998, 51(5): 515-517. DOI:10.7164/antibiotics.51.515 |
| [9] | Elsebai MF, Kehraus S, Gütschow M, König GM. Spartinoxide, a new enantiomer of A82775C with inhibitory activity toward HLE from the marine-derived fungus Phaeosphaeria spartinae. Natural Product Communications, 2010, 5(7): 1071-1076. |
| [10] | Overy DP, Rämä T, Oosterhuis R, Walker AK, Pang KL. The neglected marine fungi, Sensu stricto, and their isolation for natural products' discovery. Marine Drugs, 2019, 17(1): E42. DOI:10.3390/md17010042 |
| [11] | Wang C, Lu HY, Lan JZ, Zaman KH, Cao SG. A review: halogenated compounds from marine fungi. Molecules, 2021, 26(2): 458. DOI:10.3390/molecules26020458 |
| [12] | Youssef FS, Ashour ML, Singab ANB, Wink M. A comprehensive review of bioactive peptides from marine fungi and their biological significance. Marine Drugs, 2019, 17(10): 559. DOI:10.3390/md17100559 |
| [13] | Xu JZ, Yi MQ, Ding LJ, He S. A review of anti-inflammatory compounds from marine fungi, 2000-2018. Marine Drugs, 2019, 17(11): 636. DOI:10.3390/md17110636 |
| [14] | Karwehl S, Stadler M. Exploitation of fungal biodiversity for discovery of novel antibiotics. Current Topics in Microbiology and Immunology, 2016, 398: 303-338. |
| [15] | Stielow JB, Lévesque CA, Seifert KA, Meyer W, Irinyi L, Smits D, Renfurm R, Verkley GJM, Groenewald M, Chaduli D, Lomascolo A, Welti S, Lesage-Meessen L, Favel A, Al-Hatmi AMS, Damm U, Yilmaz N, Houbraken J, Lombard L, Quaedvlieg W, Binder M, Vaas LAI, Vu D, Yurkov A, Begerow D, Roehl O, Guerreiro M, Fonseca A, Samerpitak K, van Diepeningen AD, Dolatabadi S, Moreno LF, Casaregola S, Mallet S, Jacques N, Roscini L, Egidi E, Bizet C, Garcia-Hermoso D, Martín MP, Deng S, Groenewald JZ, Boekhout T, De Beer ZW, Barnes I, Duong TA, Wingfield MJ, de Hoog GS, Crous PW, Lewis CT, Hambleton S, Moussa TAA, Al-Zahrani HS, Almaghrabi OA, Louis-Seize G, Assabgui R, McCormick W, Omer G, Dukik K, Cardinali G, Eberhardt U, De Vries M, Robert V. One fungus, which genes? Development and assessment of universal primers for potential secondary fungal DNA barcodes. Persoonia - Molecular Phylogeny and Evolution of Fungi, 2015, 35(1): 242-263. DOI:10.3767/003158515X689135 |
| [16] | Schoch CL, Crous PW, Groenewald JZ, Boehm EWA, Burgess TI, De Gruyter J, De Hoog GS, Dixon LJ, Grube M, Gueidan C, Harada Y, Hatakeyama S, Hirayama K, Hosoya T, Huhndorf SM, Hyde KD, Jones EBG, Kohlmeyer J, Kruys A, Li YM, Lücking R, Lumbsch HT, Marvanová L, Mbatchou JS, McVay AH, Miller AN, Mugambi GK, Muggia L, Nelsen MP, Nelson P, Owensby CA, Phillips AJL, Phongpaichit S, Pointing SB, Pujade-Renaud V, Raja HA, Plata ER, Robbertse B, Ruibal C, Sakayaroj J, Sano T, Selbmann L, Shearer CA, Shirouzu T, Slippers B, Suetrong S, Tanaka K, Volkmann-Kohlmeyer B, Wingfield MJ, Wood AR, Woudenberg JHC, Yonezawa H, Zhang Y, Spatafora JW. A class-wide phylogenetic assessment of Dothideomycetes. Studies in Mycology, 2009, 64: 1-15S. DOI:10.3114/sim.2009.64.01 |
| [17] | Pang KL, Overy DP, Jones EBG, Calado MDL, Burgaud G, Walker AK, Johnson JA, Kerr RG, Cha HJ, Bills GF. 'Marine fungi' and 'marine-derived fungi' in natural product chemistry research: toward a new consensual definition. Fungal Biology Reviews, 2016, 30(4): 163-175. DOI:10.1016/j.fbr.2016.08.001 |
| [18] | Jones EBG, Suetrong S, Sakayaroj J, Bahkali AH, Abdel-Wahab MA, Boekhout T, Pang KL. Classification of marine Ascomycota, Basidiomycota, Blastocladiomycota and Chytridiomycota. Fungal Diversity, 2015, 73(1): 1-72. DOI:10.1007/s13225-015-0339-4 |
| [19] | Wang CN, Lu HM, Gao CH, Guo L, Zhan ZY, Wang JJ, Liu YH, Xiang ST, Wang J, Luo XW. Cytotoxic benzopyranone and xanthone derivatives from a coral symbiotic fungus Cladosporium halotolerans GXIMD 02502. Natural Product Research, 2020: 1-8. |
| [20] | Romeo O, Marchetta A, Giosa D, Giuffrè L, Urzì C, De Leo F. Whole genome sequencing and comparative genome analysis of the halotolerant deep sea black yeast Hortaea werneckii. Life, 2020, 10(10): 229. DOI:10.3390/life10100229 |
| [21] | Wang WX, Feng HM, Sun CX, Che Q, Zhang GJ, Zhu TJ, Li DH. Thiocladospolides F-J, antibacterial sulfur containing 12-membered macrolides from the mangrove endophytic fungus Cladosporium oxysporum HDN13-314. Phytochemistry, 2020, 178: 112462. DOI:10.1016/j.phytochem.2020.112462 |
| [22] | Hu JS, Li Z, Gao JY, He HT, Dai HQ, Xia XK, Liu CH, Zhang LX, Song FH. New diketopiperazines from a marine-derived fungus strain Aspergillus versicolor MF180151. Marine Drugs, 2019, 17(5): 262. DOI:10.3390/md17050262 |
| [23] | Ding Y, Zhu XJ, Hao LL, Zhao MY, Hua Q, An FL. Bioactive indolyl diketopiperazines from the marine derived endophytic Aspergillus versicolor DY180635. Marine Drugs, 2020, 18(7): 338. DOI:10.3390/md18070338 |
| [24] | Zhang JY, Liu C, Lv Y, Wei JH, Li B, Liao GF, Lu RM, Yang XZ. A pair of new isocoumarin enantiomers of Ludwigia hyssopifolia. Natural Product Research, 2020: 1-8. |
| [25] | Amin M, Liang X, Ma X, Dong JD, Qi SH. New pyrone and cyclopentenone derivatives from marine-derived fungus Aspergillus sydowii SCSIO 00305. Natural Product Research, 2021, 35(2): 318-326. DOI:10.1080/14786419.2019.1629919 |
| [26] | Liu YP, Fang ST, Shi ZZ, Wang BG, Li XN, Ji NY. Phenylhydrazone and quinazoline derivatives from the cold-seep-derived fungus Penicillium oxalicum. Marine Drugs, 2020, 19(1): 9. DOI:10.3390/md19010009 |
| [27] | Jiang LL, Tang JX, Bo YH, Li YZ, Feng T, Zhu HW, Yu X, Zhang XX, Zhang JL, Wang WY. Cytotoxic secondary metabolites isolated from the marine alga-associated fungus Penicillium chrysogenum LD-201810. Marine Drugs, 2020, 18(5): 276. DOI:10.3390/md18050276 |
| [28] | Buedenbender L, Kumar A, Blümel M, Kempken F, Tasdemir D. Genomics- and metabolomics-based investigation of the deep-sea sediment-derived yeast, Rhodotorula mucilaginosa 50-3-19/20B. Marine Drugs, 2020, 19(1): 14. DOI:10.3390/md19010014 |
 2022, Vol. 62
2022, Vol. 62