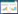中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 孙红波, 姜军, 陈丽莹, 宋泽润, 刘达, 杨杰, 霍岩, 方荣祥, 张莉莉. 2022
- Hongbo SUN, Jun JIANG, Liying CHEN, Zerun SONG, Da LIU, Jie YANG, Yan HUO, Rongxiang FANG, Lili ZHANG. 2022
- 灰飞虱核心共生菌的鉴定
- Identification of the core symbiotic bacteria of Laodelphax striatellus
- 微生物学报, 62(1): 160-175
- Acta Microbiologica Sinica, 62(1): 160-175
-
文章历史
- 收稿日期:2021-03-16
- 修回日期:2021-05-24
- 网络出版日期:2021-06-03
2. 中国科学院大学, 北京 100049;
3. 开封市祥符区农业科学研究所, 河南 开封 475100;
4. 浙江大学农业与生物技术学院, 水稻生物学国家重点实验室, 浙江 杭州 310058
2. University of the Chinese Academy of Sciences, Beijing 100049, China;
3. Institute of Agricultural Science in Xiangfu District, Kaifeng, Kaifeng 475100, Henan, China;
4. State Key Laboratory of Rice Biology, Institute of Biotechnology, Zhejiang University, Hangzhou 310058, Zhejiang, China
媒介昆虫传播多种病原微生物,引起人和动物的疾病以及农作物病害。通过控制病原在虫媒体内的传播过程,能够阻止病原由虫媒向动植物宿主的传播,从而达到抗病的目的。以虫媒体内存在的核心共生菌为载体,表达抗病功能基因,对病原侵染进行干预,是一种有效的新型抗病策略。如按蚊的核心共生菌沙雷氏菌属新菌株Serratia bacterium strain (AS1),兼具垂直和水平传播能力、易于从环境中获得、在按蚊群体中传播、并能在蚊群中代代相传。对AS1菌株进行改造,使之高效分泌表达抗疟效应蛋白,达到了在按蚊肠道内特异性杀灭疟原虫的目标,从而减少了携带疟原虫的蚊子,阻止了疟原虫向人类宿主的传播[1]。沃尔巴克氏体属(Wolbachia)细菌是一类在昆虫中普遍存在的细胞内共生菌,可通过细胞质不相容性使细菌在昆虫种群内传播[2]。将抗登革热病毒的Wolbachia感染埃及伊蚊,可以阻断Ⅱ型登革热病毒在埃及伊蚊中的传播,为控制登革热提供了虫媒共生菌方案[3]。灰飞虱和褐飞虱是水稻害虫,也是水稻病毒的传播媒介。将灰飞虱Wolbachia菌株w Stri导入褐飞虱,有效抑制了褐飞虱对水稻齿叶矮缩病毒的传播[4]。发掘具有特定定殖特性的虫媒核心共生菌,是通过虫媒进行病害防控的重要内容。
鉴定宿主核心微生物组的主要目的是定义一组微生物,为所有或者绝大多数宿主的微生物组共有,且对宿主的生物学功能非常重要。但是,越来越多的研究表明,很少能有一组微生物被所有宿主共享,且共享的微生物也并不总是和宿主的生物学功能相关联。因此,核心微生物组越来越多地通过多种限制条件来进行区分,可分为:常见核心菌(common core)、时空核心菌(temporal core)、生态核心菌(ecological core)、功能核心菌(functional core)和宿主适应性核心菌(host adapted core)。常见核心菌以较高频率出现在宿主种群内;时空核心菌在特定的时间或/和地域或宿主的特定发育阶段出现,具有可预测性;生态核心菌对于塑造宿主菌群结构多样性和稳定性发挥作用;功能核心菌与宿主适应性核心菌均参与宿主的生物学功能,区别在于功能核心菌的功能可被其他微生物取代,而宿主适应性核心菌多为专性或近似于专性共生,其对宿主的功能具有不可替代性[5]。
昆虫细胞内共生菌定殖于昆虫细胞内,可分为初级和次级内共生菌两个类别。其中初级内共生菌为专性共生,常生活在宿主特化的结构组织内,这种组织称为共生菌胞,宿主的正常生命活动离不开初级内共生菌;次级内共生菌为兼性共生,可形成多组织定殖,包括感染肠道、脂肪体和唾液腺等[6]。次级内共生菌虽然与宿主的共生关系非常紧密且能为宿主提供多种生物学功能,但其从昆虫体内缺失却并不影响昆虫的存活。次级内共生菌在昆虫宿主种群中的数量通过多种方式维持,包括垂直传播、不能垂直传播时高的水平传播率、调控性别比例增加携菌后代、提高宿主生殖力以保持对次级内共生菌的选择优势等[7−8]。由于次级内共生菌的选择优势和介导种群替换的特点,其成为基因工程菌较为理想的目标。但是,细胞内生菌不能在无细胞培养基中生长,其遗传操作的条件较为苛刻[9]。
大多数胞外菌,如肠道菌群,则更容易实现在营养培养基上的生长和较为简便的遗传转化[10],肠道是虫媒病毒起始侵染的位点,通过调节肠道微生态系统,可以达到改变昆虫与其传播的病原微生物的互作关系,进而影响病原的侵染[11],因此昆虫宿主体内普遍存在或对宿主有特定功能的核心共生菌,是基因工程菌的潜在候选菌株。
灰飞虱是半翅目飞虱科昆虫,其传播的水稻条纹病毒(rice stripe virus,RSV)引起水稻条纹叶枯病,在中国和亚洲各稻区广泛流行,造成重大的经济损失[12]。目前对灰飞虱共生菌的研究一方面揭示了灰飞虱菌群的组成结构:灰飞虱细胞内生菌Wolbachia为丰度最高的共生菌[13],肠道菌群的组成随地域的不同而有较大变化[14];另一方面阐明了Wolbachia侵染在塑造灰飞虱菌群中发挥的作用,以及灰飞虱在受抗生素处理[15]或宿主植物变化[16]等因素影响下菌群组成的变化。目前的研究尚没有对灰飞虱的核心菌进行分类鉴定。本研究对温室和3个稻区12个条件下的37个灰飞虱样本进行微生物组测序,分析微生物组的组成方式,鉴定常见核心菌及各稻区的优势共生菌,为灰飞虱相关虫媒病毒的防控提供共生工程菌靶标。
1 材料与方法 1.1 实验设计为从多因素多水平测定灰飞虱微生物组结构并鉴定核心共生菌,我们设计了以下样本采集方式:
(1) 在2019及2020年从北京温室(以下简写为GH)以及中国3个稻区,包括云南昆明(以下简写为YN)、河南开封(以下简写为KF)和河南范县(以下简写为FX),分别采集灰飞虱样本,以此代表地域因素对微生物组结构的影响。测序样本数共计16个。
(2) 在河南开封稻区,从2018-2020年间分别采集灰飞虱样本;以此代表同一地域的时间因素对微生物组结构的影响。测序样本数共计11个,其中2020年开封3样本与上述地域因素下开封地区样本为同一批次样本。
(3) 将开封灰飞虱带回北京温室,以蔗糖溶液进行人工饲喂,在饲喂后的0、6和24 h分别采集灰飞虱样本,以此代表改变的食物来源对微生物组结构的影响。测序样本数共计9个。将范县灰飞虱带回北京温室,在温室培养的水稻上饲喂并获得子代昆虫,收集子代幼虫样本,以此代表改变的培养环境及继代对微生物组结构的影响。测序样本数共计4个。
1.2 灰飞虱样本采集温室灰飞虱从浙江稻区采集并在北京的温室长期饲养。稻田灰飞虱采集时间为每年的5月至9月。用捕虫网采集后饥饿1 h,然后放置在含有100%无水乙醇的离心管中[13],干冰低温运输后置于-80 ℃冰箱保存用于DNA提取。温室灰飞虱样本采集后置于1.5 mL离心管,冰上饥饿处理1 h后进行后续实验。10到15头灰飞虱作为一个测序样本。
1.3 灰飞虱全虫基因组DNA提取用70%酒精清洗灰飞虱3次,然后用无菌ddH2O清洗2次,于液氮冷冻研磨成粉末状,采用基因组DNA提取试剂盒(Blood & Cell Culture DNA Mini Kit,Qiagen,USA)提取基因组DNA并溶于无菌ddH2O。
1.4 16S rDNA扩增子样品制备及Illumina二代测序用含有Barcode序列的细菌16S rDNA特异性引物V3F (5′-ACGGHCCARACTCCTACGGA A-3′)和V4R (5′-CTACCMGGGTATCTAATCCK G-3′)[17] PCR扩增16S rDNA的V3-V4区片段。反应体系:10×PCR Buffer 3 μL;10 mmol/L dNTPs 0.6 μL;50 mmol/L MgCl2 0.9 μL;Platinum TM Taq DNA polymerase (Thermo Fisher Scientific) 0.12 μL;优化引物V3F和V4R各0.6 μL;DNA模板50 ng;ddH2O补足至30 μL。扩增程序:94 ℃ 2 min;94 ℃ 30 s,56 ℃ 30 s,72 ℃ 30 s,30个循环;72 ℃ 5 min。每个DNA样品设置3个PCR重复,每个样品的扩增总产物为3份重复PCR产物的总和。扩增产物用1%琼脂糖凝胶电泳分离,切胶回收后通过Nano Drop One荧光定量系统定量(Thermo Fisher Scientific公司)。质检合格后(条带正常,浓度合适),进行相应比例的混合后由上海美吉生物医药科技有限公司通过Illumina MiSeq PE300平台进行上机测序。
1.5 数据分析将不同批次的原始Fastq测序文件整合,采用RDP classifier贝叶斯算法对97%相似水平的OTU代表序列进行分类学分析,并分别在域(domain)、界(kingdom)、门(phylum)、纲(class)、目(order)、科(family)、属(genus)和种(species)分类学水平统计各样本的群落物种组成,并采用silvia138_16s细菌数据库进行分类比对。滤除单个样本中OTU序列数小于5的OTU。利用美吉生物云平台(https://cloud.majorbio.com/)、R语言和Excel等生信分析平台及软件对测序数据进行包括α多样性、β多样性、物种组成、物种组间差异及KEGG功能预测等生物信息学分析。使用Adobe Illustrator对原始图片进行文字处理及格式调整。
2 结果与分析 2.1 测序数据统计本研究对2019年和2020年温室、2018-2020年昆明、开封和范县的37个灰飞虱样本进行微生物组的16S rDNA二代测序及生物信息学分析,共设置12个测序条件。原始数据优化后(97%相似水平的OTU代表序列进行分类学分析,并去除在单个样本中序列条数小于5的OTU),共得到1 849 623条有效序列,组成540条优化OTU,199个属,13个门。
2.2 温室灰飞虱菌群以Wolbachia为主温室代表较为简单可控和相对稳定的培养环境。对2019年和2020年温室灰飞虱的菌群组成进行分析发现:温室灰飞虱菌群以变形菌门为主,在所有测序样本中的相对丰度为98.90%-99.76%,平均相对丰度为99.24%;其次是厚壁菌门,相对丰度为0.02%-0.7% (图 1A)。在属水平,Wolbachia相对丰度高达87.62%-97.62%,平均相对丰度为92.46%。除Wolbachia之外,其他相对丰度较高的细菌均为细胞外共生。其中不动杆菌属(Acinetobacter)、泛菌属(Pantoea)、代尔夫特菌属(Delftia)和根瘤菌属(Rhizobium)存在于所有的测序样本中,平均相对丰度分别为0.9%、0.45%、1.47%和0.31%。肠杆菌(Enterobacter)存在于67%的样本中,平均相对丰度为2.68% (图 1B)。β多样性分析表明:温室灰飞虱菌群组成在不同的测序样本之间差异较大,但与取样年份之间的关联较小(图 1C)。
|
| 图 1 温室灰飞虱菌群结构解析 Figure 1 Analysis of the bacterial community structure of the greenhouse L. striatellus. A: relative abundance at phylum level. B: relative abundance at genus level. Taxa with relative abundance > 0.1% are shown. GH19_1-GH19_3, L. striatellus sample No. 1-3 in 2019; GH20_1−GH20_3, L. striatellus sample No. 1−3 in 2020. C: β-diversity using principal component analysis (PCA) analysis at the genus level. |
2.3 不同稻区灰飞虱的菌群组成差异显著
不同地域水稻种植区的灰飞虱菌群组成差别较大,这些差别表现在优势菌组成、α多样性和β多样性(图 2A-D)。在门水平,昆明灰飞虱主要是变形菌门,占比99.95%,另有少量的放线菌门、拟杆菌门和厚壁菌门;范县以变形菌门和拟杆菌门为主,占比分别为93.22%和6.76%,厚壁菌门和放线菌门含量极低;开封灰飞虱菌群的变形菌门、拟杆菌门、放线菌门和厚壁菌门分别占比为72.01%、26.78%、0.53%和0.52% (图 2A)。在属水平,Wolbachia存在于所有样本,占比为29.02%-98.69% (图 2B)。除Wolbachia外,不同稻区的灰飞虱各有优势菌,这一现象与之前的报道相似[14]。其中昆明稻区的优势菌为立克次体属(Rickettsia),平均占比56.76%;范县优势菌为杀雄菌属(Arsonophonus)和Cardinium,平均占比分别为12.36%和6.76%;开封稻区的优势菌为醋杆菌属(Acetobacterace,12.09%)、Sulcia内生菌(26.72%)和Pantoea (5.1%)。3个稻区总体平均相对丰度前10位的共生菌分别为细胞内生菌Wolbachia、Rickettsia、Sulcia、Cardinium、Nasuia、Arsonophonus和细胞外的Acetobacterace、Pseudomonas、Acinetobacter、Pantoea (图 2B)。开封地区灰飞虱菌群物种多样性和丰富度(α多样性)明显高于云南和范县地区(图 2C),β多样性分析显示云南、开封、范县三地灰飞虱菌群结构差异显著(图 2D)。

|
| 图 2 不同地域灰飞虱菌群结构解析 Figure 2 Analysis of the bacterial community structure of L. striatellus in different rice areas. A: relative abundance at phylum level. B: relative abundance at genus level. C: Shannon index on OTU level. Abscissa represents the group name, ordinate represents mean value of Shannon index of each group. For the two groups with significant differences is marked as *, *: P < 0.05. D: PCA analysis on genus level. YN, KF and FX: insects collected from Kunming, Yunnan; Kaifeng, Henan; and Fanxian, Henan, respectively. Taxa with relative abundance > 0.1% are shown in Figure A and B. |
2.4 同一稻区不同时间灰飞虱的优势菌组成差别较大
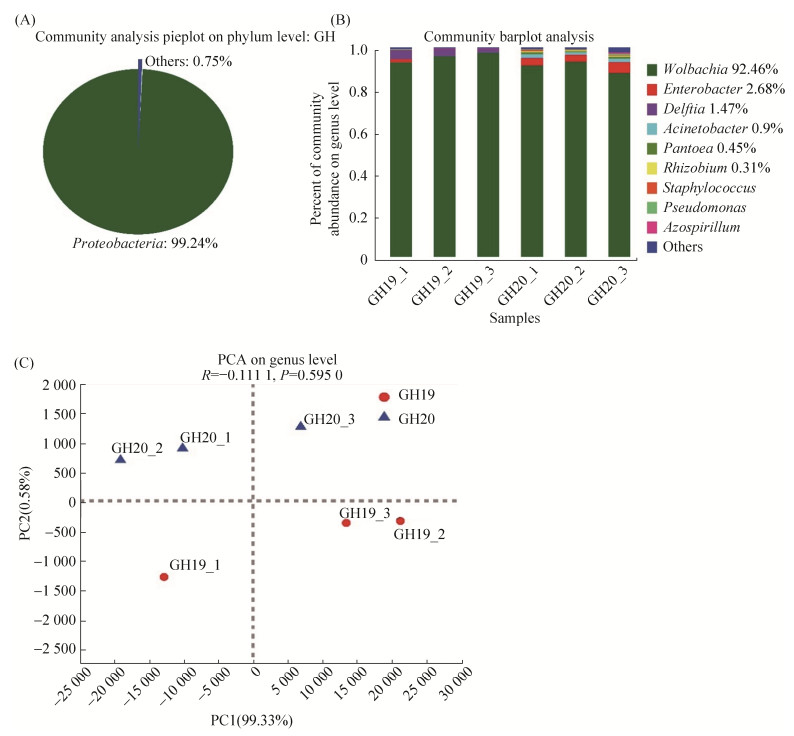
对连续三年从开封稻区采集的灰飞虱进行菌群测序,发现不同年份或不同月份的灰飞虱样本均以变形菌门为主(> 95%),仅2020年的样本有较高丰度的拟杆菌门(26.78%),为细胞内生菌Sulcia属在其中一个样本中的高丰度所致。三年所有样本中,变形菌门、拟杆菌门、厚壁菌门和放线菌门平均占比分别为88.61%、9.79%、1.02%和0.49% (图 3A)。
|
| 图 3 时间变量下灰飞虱菌群结构解析 Figure 3 Analysis of the bacterial community structure of L. striatellus under time variables. A: the average relative abundance on phylum level (left) and genus level (right). KF18_07, KF18_09, KF19, KF20, the Kaifeng L. striatellus samples collected in July 2018, September 2018, 2019 and 2020, respectively. B: PCA analysis on OTU level. C: analysis of the bacterial community structure of each L. striatellus sequencing sample on genus level. KF18-1-KF18-2, the two L. striatellus samples collected in July 2018; KF18-3-KF18-5, the three L. striatellus samples collected in September 2018; KF19-1-KF19-3 and KF20-1-KF20-3, the three L. striatellus samples collected in 2019 and 2020, respectively. Taxa with relative abundance > 0.1% are displayed in Figure A. |
在属水平,Wolbachia在2018和2019年的样本中相对丰度很低,分别为0.31%和5.21%。除Wolbachia外,2018年9月的灰飞虱样本优势菌为Nasuia、Acetobacter、Pantoea、Rickettsia、Sulcia;2018年7月优势菌为Diplorickettsia、Acetobacter、Nasuia、Pantoea;2019年优势菌为Acetobacter、Unclassified Gammaproteobacteria、Cosenzaea、Enterobacter、Rhodobacter和Rickettsia (图 3A)。这些优势菌的相对丰度在不同样本之间差异较大(图 3C),例如:Acetobacter属共生菌在6个样本中的相对丰度为29.76%-86.61%;Nasuia在3个样本丰度较高,分别为5.30%、5.51%和91.36%;2个样本中Diplorickettsia丰度较高,分别为5.60%和90.20%;4个样本Pantoea丰度较高,分别为2.30%、9.62%、12.36%和68.34%;一个样本Unclassified Gammaproteobacteria较高,为61.33% (图 3C)。β多样性分析显示2019和2020年的灰飞虱样本菌群结构相近,与2018年样本差别较大(图 3B)。Anosim相似性分析显示R2为0.279,而残差超过0.7,说明时间变量不是影响灰飞虱共生菌组成的主要因素,P值0.02,说明不同时间点菌群组成差异显著。4个时间点共有的细菌为29个属,其中平均相对丰度高于1%的共有属有5个,分别为:Wolbachia、Pseudomonas、Rickettsia、Enterobacter和Acinetobacter。
2.5 稻田灰飞虱在温室饲养后菌群结构的变化将温室与稻田灰飞虱菌群进行比较发现:所有的温室和稻田灰飞虱样本,其菌群均由细胞内和细胞外共生菌共同组成。温室灰飞虱显著的特点为,Wolbachia是主要的细胞内生菌(在少数样本中检测到极低丰度的Rickettsia),其相对丰度显著高于多种来源的稻田灰飞虱Wolbachia,而大多数稻田灰飞虱则携带超过一种细胞内生菌。
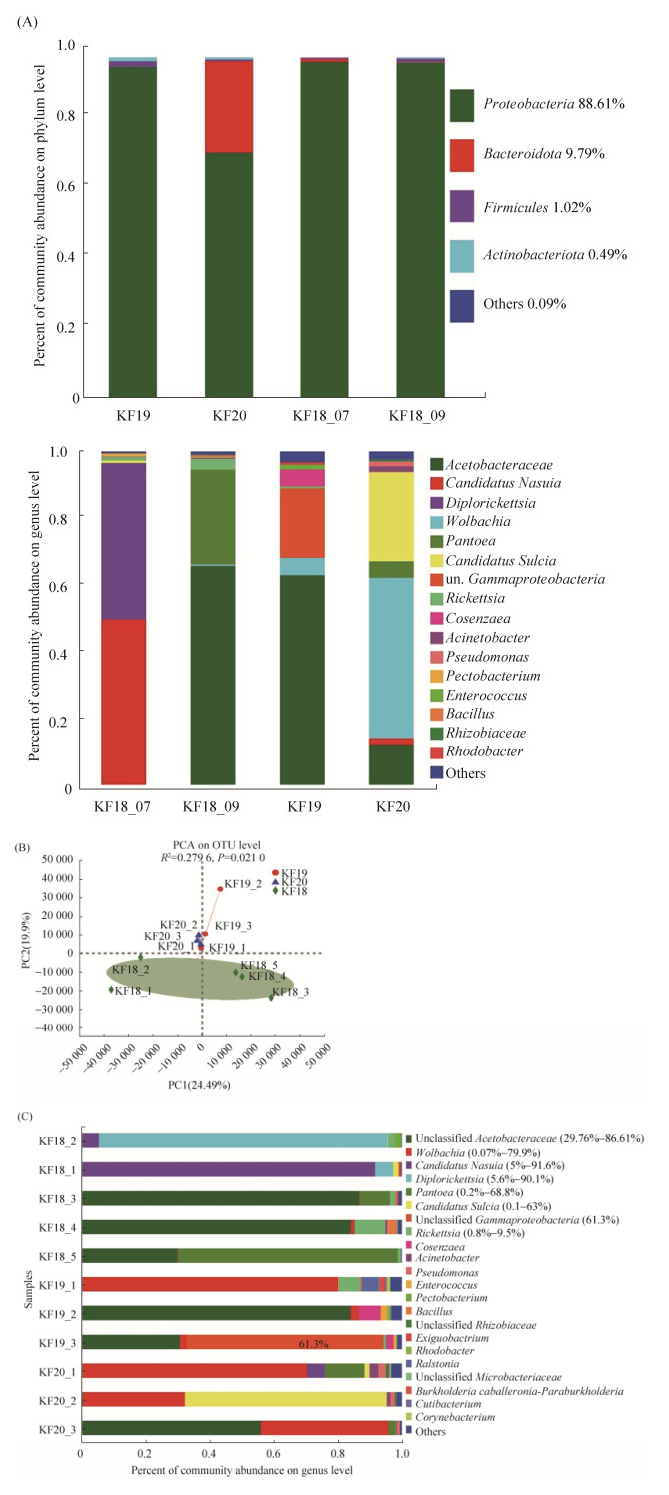
为检测环境变化对菌群结构的影响,我们采集开封稻田灰飞虱并置于温室中,以无菌蔗糖溶液饲喂24 h。属水平菌群结构分析显示:蔗糖饲喂后Wolbachia的相对丰度上升明显,多种其他细胞内生菌丰度降低(图 4A),而β多样性分析显示蔗糖饲喂后的灰飞虱与大田灰飞虱菌群差异不显著,(R2=0.358, P=0.234,图 4B)。我们进而将范县稻田的灰飞虱以温室水稻继代培养,发现:子代灰飞虱Wolbachia的相对丰度进一步升高,而其他细胞内生菌,如Arsonophonus和Cardinium,则检测不到(图 4C),β多样性显示继代培养后菌群结构与大田来源灰飞虱差异显著(图 4D)。这些结果表明:改变的环境条件使得多种共生菌的相对丰度发生了显著变化,相应于温室条件,Wolbachia相对丰度升高而多种稻田优势菌相对丰度下降(图 4E),其中,田间灰飞虱通过取食获得的多种昆虫细胞内生菌在缺乏这些内生菌的环境中被灰飞虱逐渐丢失,可能原因是其无法被灰飞虱垂直传播。
|
| 图 4 稻田灰飞虱在温室蔗糖饲喂与继代培养后菌群结构变化 Figure 4 Microbiome structures of sucrose-fed field insects or their offspring. A-B: after Kaifeng L. striatellus was fed with sucrose in greenhouse, the bacterial community structure was analyzed at the genus level (A) and the β-diversity was analyzed using PCA at the OTU level (B). SH0: without sucrose feeding, S6: 6 hours post sucrose feeding, S24: 24 hours post sucrose feeding. C-D: after FanXian L. striatellus generates offsprings in greenhouse, the bacterial community structure was analyzed at the genus level (C) and the β-diversity was analyzed using PCA at the OTU level (D). FX(F1L1-F1L4): FanXian insects, FXF1(FX 1-FX 4): offsprings of Fanxian insects. E: Kruskal-Wallis rank sum test to show the differentially abundant taxa. The X-axis represents the genus name, the Y-axis represents the average relative abundance. The number on the top is the P value, *P≤0.05. |
2.6 灰飞虱核心共生菌的鉴定
为鉴定不随时间、地域、生活环境变化的常见核心菌,我们通过韦恩图在属水平分析所有12个测序条件共有的细菌属(图 5),鉴定到10个常见核心细菌属,包括Wolbachia、Sphingomona、Rhizobium、Pseudomonas、Enterobacter、Acinetobacter、Pantoea、Delftia、Stenotrophomonas和Microbacterium。其中Wolbachia丰度最高,其次是Pantoea (4.97%)、Enterobacter (0.77%)和Acinetobacter (0.67%) (图 5A和C)。对每个细菌属进行OTU水平的分析,获得8个核心OTU。其中OTU45为Wolbachia代表性序列,占核心OTU序列总数的93.82%,Pantoea代表序列OTU141占4.67% (图 5B和D)。
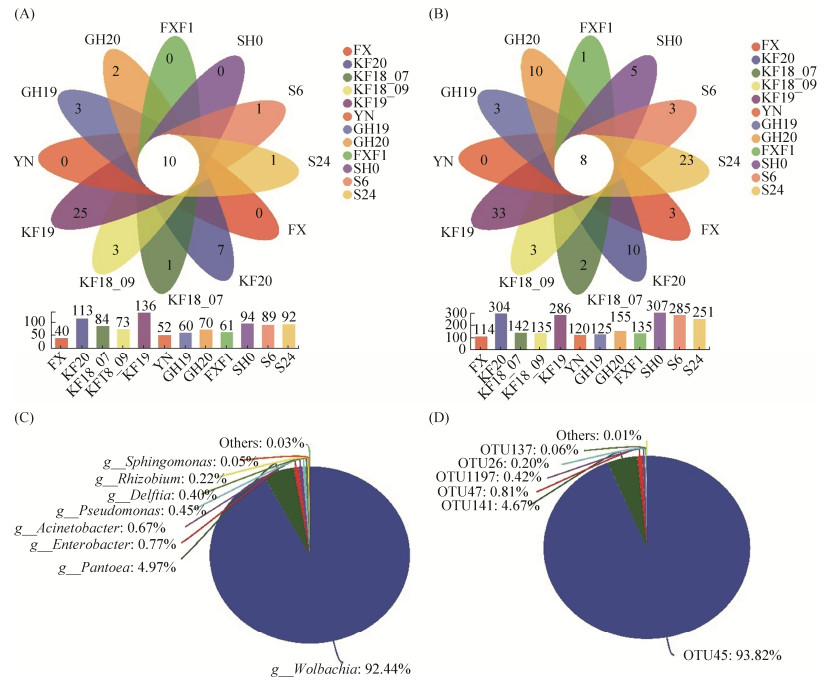
|
| 图 5 韦恩图展示灰飞虱的常见核心菌 Figure 5 Venn diagram shows the common core bacteria of L. striatellus. A-B: Venn diagrams show the common core bacterial genera (A) and the common core OTUs (B). C-D: relative abundance of the 10 common core genera (C) and the 8 core OTUs (D). |
不同的稻田环境下存在一些在特定样本中高丰度的共生菌,包括开封稻区出现的Acetobacter、Diplorickettsia、Nasuia、Sulcia、Pantoea和Unclassified Gammaproteobacteria,昆明稻区的Rickettsia,范县稻区的Arsenophonus和Cardinium。这些高丰度的共生菌多为细胞内生菌,可能是稻田有多种昆虫取食水稻,促进了其他昆虫所携带的细胞内生菌的水平传播。
在OTU水平分析10种常见核心菌和8种在不同地域内出现的高丰度优势菌(表 1)。灰飞虱常见核心菌Wolbachia共检测到85条OTU序列,其中OTU45占据该属OTU序列总数的96.55%,除了在2018年7月开封样本中分布较少,其余样本中该条OTU均以较高的丰度存在。
| Genus | Species | OTU numbers | Representive OTU | Proportion/% |
| Unclassified Acetobacteraceae | Unclassified Acetobacteraceae | 112 | OTU1277 | 9.23 |
| Unclassified Acetobacteraceae | Unclassified Acetobacteraceae | 112 | OTU3757 | 12.91 |
| Unclassified Acetobacteraceae | Unclassified Acetobacteraceae | 112 | OTU3090 | 48.86 |
| Unclassified Acetobacteraceae | Unclassified Acetobacteraceae | 112 | OTU3637 | 22.60 |
| Diplorickettsia | Uncultured Rickettsiella sp. Diplorickettsia | 1 | OTU596 | 100.00 |
| Candidatus Nasuia | Unclassified Candidatus Nasuia | 3 | OTU598 | 96.65 |
| Candidatus Sulcia | Uncultured Candidatus Sulcia sp. | 15 | OTU1208 | 96.00 |
| Candidatus Cardinium | Uncultured Candidatus Cardinium | 5 | OTU143 | 87.00 |
| Unclassified Gammaproteobacteria | Unclassified c Gammaproteobacteria | 1 | OTU1215 | 100.00 |
| Wolbachia | Uncultured bacterium Wolbachia | 88 | OTU45 | 96.55 |
| Acinetobacter | Acinetobacter pittii | 8 | OTU1251 | 4.83 |
| Acinetobacter | Acinetobacter johnsonii | 8 | OTU137 | 5.08 |
| Acinetobacter | Acinetobacter soli | 8 | OTU42 | 89.27 |
| Enterobacter | Enterobacter cloacae complex sp. | 4 | OTU47 | 95.00 |
| Pantoea | Unclassified Pantoea | 12 | OTU141 | 93.60 |
| Sphingomonas | Sphingomonas | 5 | OTU140 | 66.44 |
| Rhizobium | Allorhizobium vitis | 5 | OTU26 | 85.00 |
| Arsenophonus | Unclassified Arsenophonus | 4 | OTU2364 | 81.00 |
| Arsenophonus | Uncultured bacterium Arsenophonus | 4 | OTU2006 | 9.99 |
| Rickettsia | Unclassified Rickettsia | 3 | OTU3938 | 89.73 |
| Rickettsia | Unclassified Rickettsia | 3 | OTU1189 | 10.26 |
| Pseudomonus | Unclassified Pseudomonas | 8 | OTU 3211 | 81.00 |
| Delftia | Delftia | 1 | OTU1197 | 100.00 |
| Microbacterium | Microbacterium laevaniformans | 3 | OTU2772 | 83.00 |
| Stenotrophomonas | Stenotrophomonas | 1 | OTU1329 | 100.00 |
其他常见核心菌OTU分布情况如下:(1) Pantoea属含12条OTU代表序列,其中核心OTU141占该属总OTU序列的89.25%;(2) Enterobacteria OTU47占其总4条OTU序列的95%,在不同类型样本中分布广泛;(3) Acinetobacter共有5条OTU,其中OTU42在样本间分布较广,占5条OTU序列总数的67.25%;(4) Pseudomonas OTU 3211占该属12条OTU序列总数的81%;(5) Rhizobium OTU26占该属5条OTU序列总数的85%;(6) Sphingomonas含有5条OTU,序列占比较低;(7) Delftia仅1条OTU序列,OTU1197,多集中在2019年温室样本中,其他样本有少量分布;(8) Microbacterium含量较低,相对丰度低于0.03%,3条OTU总计220条测序序列;(9) Stenotrophomonas仅65条测序序列,由OTU1329这一条OTU组成。
稻田高丰度共生菌OTU分布情况如下:(1) Acetobacter属共计112条OUT,其中4条OTU组成总OTU序列总数的93.60%,该菌在云南及温室环境下均不存在,但开封稻田灰飞虱在温室饲养后,同代次内该属细菌并不丢失;(2) Diplorickettsia仅含1条OTU代表序列OTU596;(3) Candidatus_Nasuia属共有3条OTU序列,其中OTU598占比96.65%,集中在2018年及2020年开封样本中;(4) Candidatus_Sulcia属含有15条OTU序列,其中OTU1208在2020年开封多个测序样本中存在,占该属OTU序列总数的96%;(5) Unclassified Gammaproteobacteria仅有1条OTU1215,以较高丰度存在于2019年开封第3号样本中;(6) Arsenophonus属含有4条OTU代表序列,其中OTU2364占比最高达81%,所有序列集中在范县灰飞虱FX_01,FX_02两个样本;(7) Candidatus_Cardinium含有5条OTU代表序列,其中OTU143占比87%,序列多集中在范县灰飞虱FX_02和FX_03两个样本,其他个别样本有少量分布;(8) Rickettsia属核心OTU为OTU3938,占3种Rickettsia OTU序列总数的89.24%;Rickettsia OTU1189在昆明3个样本中不存在,但在开封不同年份的样本中有广泛分布,相对丰度较低。
3 讨论本研究通过对不同地域、不同时间及温室的灰飞虱进行菌群测序和分析,初步阐明了灰飞虱共生菌的基本组成和变化规律,并鉴定了常见核心菌及3个中国稻区的优势菌。灰飞虱的菌群结构表现出一些显著的特点:一是本研究中所涉及的所有灰飞虱样本,均携带细胞内和细胞外两种类别的共生菌。在温室条件下,细胞内生菌以Wolbachia为主,其相对丰度高于90%以上。除了Wolbachia之外,在极少数的样本中能检测到很低丰度的Rickettsia细胞内生菌。在稻田环境下,灰飞虱的细胞内生菌种类丰富。其二是灰飞虱菌群受环境影响较大,随着地域和时间的不同,优势菌的组成变化明显。本研究所采集的3个地区的稻田灰飞虱,分别有特定的优势菌,如仅在开封稻区检测到高丰度的Acetobacter、在部分开封样本中检测到高丰度的细胞内生菌Nasuia和Cardinium、仅在范县检测到高丰度的Arsenophonus、以及仅在昆明灰飞虱体内占据较高相对丰度的Rickettsia。将携带这些高丰度优势菌的灰飞虱置于温室条件下培养24 h,在部分样本中优势菌保持高丰度,而在多数样本中,优势菌的丰度会下降。综合不同样本的菌群组成及变化规律,我们初步鉴定了灰飞虱的核心共生菌,包括在不同条件下以一定丰度存在的10种常见核心菌和特定稻田样本中高丰度存在的8种优势菌。其中,Wolbachia为灰飞虱的次级细胞内共生菌,可以在多种组织,包括中肠、脂肪体和唾液腺中定殖,并能侵入卵巢进行垂直传播。其他各共生菌的定殖位置及传播特征,目前均未有报道。
本研究中,Acetobacter属在开封稻田灰飞虱体内连续3年都检测到较高的相对丰度,在OTU水平与已报道的按蚊共生菌Asaia以及白背飞虱共生菌Asaia在16S rDNA序列上高度同源。该株Asaia菌株均能体外培养并以较高的丰度在昆虫肠道定殖,是潜在的基因工程菌[18]。其中Asaia SF2.1菌株已在抗疟研究中得到较好的应用[19]。Wolbachia是重要的昆虫细胞内共生菌[2],在多种昆虫宿主中发挥抗病毒[3, 20-22]、具有增强宿主对杀虫剂的适应性[23]、塑造宿主菌群结构[13]、调节宿主固有免疫系统[24]等功能。灰飞虱Wolbachia在温室和稻田样本中的核心OTU序列完全相同,该菌株在稻田其他昆虫,如褐飞虱、叶蝉等体内占据生态位的能力值得探索。Acinetobacter、Pantoea和Rhizobium在灰飞虱体内稳定存在,这3个属的细菌均在部分样本中表现出较高的相对丰度,其中Acinetobacter在多数样本中相对丰度高于1%,Pantoea在开封灰飞虱体内最高相对丰度达到68.8%。这3个属的细菌作为基因工程菌需要更明确的组织定位和定量分析。稻田灰飞虱携带较多的细胞内生菌,可能的原因是稻田环境有多种昆虫生活,导致了昆虫胞内菌的水平传播,例如Nasuia、Sulcia、Cardinium是蚜虫和叶蝉常见的细胞内生菌[25-27]。温室饲养的媒介昆虫如何更真实地反映田间的复杂情况,将是媒介昆虫与虫媒病毒互作及载体工程菌使用中需要探究的重要问题。
| [1] | Wang SB, Dos-Santos ALA, Huang W, Liu KC, Oshaghi MA, Wei G, Agre P, Jacobs-Lorena M. Driving mosquito refractoriness to Plasmodium falciparum with engineered symbiotic bacteria. Science, 2017, 357(6358): 1399-1402. DOI:10.1126/science.aan5478 |
| [2] | Werren JH, Baldo L, Clark ME. Wolbachia: master manipulators of invertebrate biology. Nature Reviews Microbiology, 2008, 6(10): 741-751. DOI:10.1038/nrmicro1969 |
| [3] | Walker T, Johnson PH, Moreira LA, Iturbe-Ormaetxe I, Frentiu FD, McMeniman CJ, Leong YS, Dong Y, Axford J, Kriesner P, Lloyd AL, Ritchie SA, O'Neill SL, Hoffmann AA. The w Mel Wolbachia strain blocks dengue and invades caged Aedes aegypti populations. Nature, 2011, 476(7361): 450-453. DOI:10.1038/nature10355 |
| [4] | Gong JT, Li YJ, Li TP, Liang YK, Hu LC, Zhang DJ, Zhou CY, Yang C, Zhang X, Zha SS, Duan XZ, Baton LA, Hong XY, Hoffmann AA, Xi ZY. Stable introduction of plant-virus-inhibiting Wolbachia into planthoppers for rice protection. Current Biology, 2020, 30(24): 4837-4845. DOI:10.1016/j.cub.2020.09.033 |
| [5] | Risely A. Applying the core microbiome to understand host-microbe systems. Journal of Animal Ecology, 2020, 89(7): 1549-1558. DOI:10.1111/1365-2656.13229 |
| [6] | Dobson SL, Bourtzis K, Braig HR, Jones BF, Zhou WG, Rousset F, O'Neill SL. Wolbachia infections are distributed throughout insect somatic and germ line tissues. Insect Biochemistry and Molecular Biology, 1999, 29(2): 153-160. DOI:10.1016/S0965-1748(98)00119-2 |
| [7] | Hurst GD, Frost CL. Reproductive parasitism: maternally inherited symbionts in a biparental world. Cold Spring Harbor Perspectives in Biology, 2015, 7(5): a017699. DOI:10.1101/cshperspect.a017699 |
| [8] | Yin CH, Sun P, Yu X, Wang PH, Cheng G. Roles of symbiotic microorganisms in arboviral infection of arthropod vectors. Trends in Parasitology, 2020, 36(7): 607-615. DOI:10.1016/j.pt.2020.04.009 |
| [9] | McCutcheon JP. The bacterial essence of tiny symbiont genomes. Current Opinion in Microbiology, 2010, 13(1): 73-78. DOI:10.1016/j.mib.2009.12.002 |
| [10] | Hosokawa T, Ishii Y, Nikoh N, Fujie M, Satoh N, Fukatsu T. Obligate bacterial mutualists evolving from environmental bacteria in natural insect populations. Nature Microbiology, 2016, 1: 15011. DOI:10.1038/nmicrobiol.2015.11 |
| [11] | Wu P, Yu X, Wang PH, Cheng G. Arbovirus lifecycle in mosquito: acquisition, propagation and transmission. Expert Reviews in Molecular Medicine, 2019, 21: e1. DOI:10.1017/erm.2018.6 |
| [12] | Nault LR. Transmission biology, vector specificity and evolution of planthopper-transmitted plant viruses. Planthoppers. Boston, MA: Springer US, 1994: 429-448. |
| [13] | Duan XZ, Sun JT, Wang LT, Shu XH, Guo Y, Keiichiro M, Zhu YX, Bing XL, Hoffmann AA, Hong XY. Recent infection by Wolbachia alters microbial communities in wild Laodelphax striatellus populations. Microbiome, 2020, 8(1): 104. DOI:10.1186/s40168-020-00878-x |
| [14] | Liu WW, Zhang XW, Wu N, Ren YD, Wang XF. High diversity and functional complementation of alimentary canal microbiota ensure small brown planthopper to adapt different biogeographic environments. Frontiers in Microbiology, 2019, 10: 2953. |
| [15] | Bing XL, Zhao DS, Peng CW, Huang HJ, Hong XY. Similarities and spatial variations of bacterial and fungal communities in field rice planthopper (Hemiptera: Delphacidae) populations. Insect Science, 2020, 27(5): 947-963. DOI:10.1111/1744-7917.12782 |
| [16] | Pan HB, Li MY, Wu W, Wang ZL, Yu XP. Host-plant induced shifts in microbial community structure in small brown planthopper, Laodelphax striatellus (Homoptera: Delphacidae). Journal of Economic Entomology, 2021, 114(2): 937-946. DOI:10.1093/jee/toaa316 |
| [17] | 张莉莉, 陈丽莹. 一种解析水稻叶际内生细菌菌群结构的方法. 中国, 201710187212.9. 2020-06-23. |
| [18] | Li F, Li P, Hua HX, Hou ML, Wang FL. Diversity, tissue localization, and infection pattern of bacterial symbionts of the white-backed planthopper, Sogatella furcifera (Hemiptera: Delphacidae). Microbial Ecology, 2020, 79(3): 720-730. DOI:10.1007/s00248-019-01433-4 |
| [19] | Shane JL, Grogan CL, Cwalina C, Lampe DJ. Blood meal-induced inhibition of vector-borne disease by transgenic microbiota. Nature Communications, 2018, 9: 4127. DOI:10.1038/s41467-018-06580-9 |
| [20] | Panteleev DY, Goryacheva II, Andrianov BV, Reznik NL, Lazebny OE, Kulikov AM. The endosymbiotic bacterium Wolbachia enhances the nonspecific resistance to insect pathogens and alters behavior of Drosophila melanogaster. Russian Journal of Genetics, 2007, 43(9): 1066-1069. DOI:10.1134/S1022795407090153 |
| [21] | Teixeira L, Ferreira A, Ashburner M. The bacterial symbiont Wolbachia induces resistance to RNA viral infections in Drosophila melanogaster. PLoS Biology, 2008, 6(12): e2. |
| [22] | Martinez J, Bruner-Montero G, Arunkumar R, Smith SCL, Day JP, Longdon B, Jiggins FM. Virus evolution in Wolbachia-infected Drosophila. Proceedings of the Royal Society B: Biological Sciences, 2019, 286(1914): 20192117. DOI:10.1098/rspb.2019.2117 |
| [23] | Liu XD, Guo HF. Importance of endosymbionts Wolbachia and Rickettsia in insect resistance development. Current Opinion in Insect Science, 2019, 33: 84-90. DOI:10.1016/j.cois.2019.05.003 |
| [24] | Horak RD, Leonard SP, Moran NA. Symbionts shape host innate immunity in honeybees. Proceedings Biological Sciences, 2020, 287(1933): 20201184. |
| [25] | Kobiaka M, Michalik A, Walczak M, Junkiert U, Szklarzewicz T. Sulcia symbiont of the leafhopper Macrosteles laevis (Ribaut, 1927) (Insecta, Hemiptera, Cicadellidae: Deltocephalinae) harbors Arsenophonus bacteria. Protoplasma, 2016, 253(3): 903-912. DOI:10.1007/s00709-015-0854-x |
| [26] | Konecka E, Olszanowski Z. A new Cardinium group of bacteria found in Achipteria coleoptrata (Acari: Oribatida). Molecular Phylogenetics and Evolution, 2019, 131: 64-71. DOI:10.1016/j.ympev.2018.10.043 |
| [27] | Kobiałka M, Michalik A, Szwedo J, Szklarzewicz T. Diversity of symbiotic microbiota in Deltocephalinae leafhoppers (Insecta, hemiptera, cicadellidae). Arthropod Structure & Development, 2018, 47(3): 268-278. |
 2022, Vol. 62
2022, Vol. 62