中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 张阳, 程鹏, 熊静, 李晓芬, 李会, 王士源, 彭练慈, 韩玉竹, 刘娟, 陈红伟. 2021
- Yang Zhang, Peng Cheng, Jing Xiong, Xiaofen Li, Hui Li, Shiyuan Wang, Lianci Peng, Yuzhu Han, Juan Liu, Hongwei Chen. 2021
- 基于RNA-Seq技术分析鼠源宿主防御肽对铜绿假单胞菌成熟生物被膜的清除作用
- Eradication effect of Pseudomonas aeruginosa biofilms by mouse host defense peptide based on RNA-Seq technology
- 微生物学报, 61(9): 2843-2853
- Acta Microbiologica Sinica, 61(9): 2843-2853
-
文章历史
- 收稿日期:2020-11-16
- 修回日期:2021-03-10
- 网络出版日期:2021-03-19
2. 西南大学动物医学院, 重庆 402460;
3. 西南大学动物科技学院, 重庆 402460
2. College of Veterinary Medicine, Southwest University, Chongqing 402460, China;
3. College of Animal Science and Technology, Southwest University, Chongqing 402460, China
铜绿假单胞菌易形成生物被膜,常导致慢性、反复性感染。生物被膜由生物被膜细菌及其胞外多糖复合物(EPS)组成,是由多个系统调控而产生的一个动态生物学过程[1]。有研究报道,自然界中80%微生物能以生物被膜形式存在,高达75%的人类感染与生物被膜相关,生物被膜细菌能够导致比浮游菌高10–1000倍的抗生素耐药[2-3]。
宿主防御肽是机体免疫的第一道防线,具有调节机体免疫和抵御外来微生物入侵的功能。其广谱的抗微生物活性和独特的抗菌机制使得它被应用于广泛的领域。近年来,有研究者将其应用于对抗细菌生物被膜领域,人源宿主防御肽LL-37及其修饰物已被报道具有抗生物被膜活性[4-6]。而CRAMP是在小鼠中发现的宿主防御肽,其结构和功能与LL-37极为相似[7]。
本研究通过前期试验发现CRAMP修饰肽对铜绿假单胞菌PAO1具有一定的清除作用,通过激光共聚焦扫描显微镜发现CRAMP修饰肽作用后的PAO1生物被膜体积和数量明显变小[8],推测CRAMP修饰肽可能对PAO1生物被膜的形成和分散过程有影响。考虑到生物被膜的形成是一个动态的、复杂的过程。因此利用转录组学方法对CRAMP修饰肽干预后的成熟生物被膜展开进一步研究,探寻CRAMP修饰肽对细菌生物被膜调控的具体机制。
1 材料和方法 1.1 菌株和药品铜绿假单胞菌PAO1株,购自中国普通微生物菌种保藏管理中心。
CRAMP修饰肽,由苏州强耀生物科技有限公司合成和修饰,氨基酸序列和修饰见国家发明专利(201810701474.7)。
1.2 主要试剂MHB、TSA培养基等购自青岛海博生物技术有限公司。1%结晶紫、Triton X-100购自北京索莱宝科技有限公司。96孔细胞培养板(3599)、6孔细胞培养板(3516)、细胞培养瓶(430168)购自美国康宁公司。Lab-TekTMⅡ腔室盖玻片、FilmTracerTM LIVE/DEAD Biofilm Viability Kit购自赛默飞世尔科技(中国)有限公司。海藻酸钠、1, 3-萘二酚购自上海麦克林生化科技有限公司。
1.3 CRAMP修饰肽对铜绿假单胞菌成熟生物被膜的影响将PAO1菌株隔夜培养,10000 r/min离心10 min,弃上清重悬于等体积MHB中。调整OD600=0.1,并稀释100倍,作为工作菌液。将工作菌液加入96孔板中,37 ℃培养24 h,形成成熟生物被膜。弃去培养基,用PBS缓冲液洗净浮游菌,分别加入0.97–62.50 μg/mL的CRAMP修饰肽,1 h后进行后续试验。选择合适浓度后转移至6孔板验证结果。所有试验均独立3次以上重复,下同。
1.4 生物被膜量和生物被膜活菌计数吸净药液,用PBS缓冲液洗涤3次。加入甲醇固定10 min。吸净甲醇,待甲醇挥发后加入结晶紫溶液染色20 min。洗涤生物被膜后加入醋酸溶液溶解生物被膜,在630 nm处测定OD值,检测生物被膜量(crystal violet,CV)。同法洗涤生物被膜。加入Triton,吹打混匀,充分破坏生物被膜,取25 μL进行10倍梯度稀释。最后取100 μL涂布于TSA板上进行生物被膜活菌计数(colony count,CC)。
1.5 激光共聚焦扫描显微镜(CLSM) 表征生物被膜形态用MHB培养铜绿假单胞菌培养过夜,调整其OD600=0.1。将工作菌液加入Lab-TekTM Ⅱ腔室盖玻片中。在37 ℃条件下静置培养5 d,每隔24 h更换培养基。培养5 d后,吸净培养基并用0.9 %的NaCl溶液洗去未附着的细菌。在暗室中用FilmtracerTM LIVE/DEADTM Biofilm Viability Kit染色。用灭菌水洗去多余的染料,在Zeiss-800共聚焦激光显微镜10倍镜下观察生物被膜。激发波长分别为488 nm (SYTO)和561 nm (PI),发射波长分别为500/32 nm (SYTO)和617/40 nm (PI)。试验随机选取5个独立重复视野,采集图像后采用荧光强度反映生物被膜的量。
1.6 转录组测序将PAO1菌株隔夜培养,10000 r/min离心10 min,弃上清重悬于等体积MHB中。调整OD600=0.1,并稀释100倍,作为工作菌液。在细胞培养瓶中形成成熟生物被膜,CRAMP修饰肽干预1 h后,用PBS缓冲液洗涤3次,用细胞刮刀刮取生物被膜,干冰保存送样。委托北京奥维森基因科技有限公司对上述样品进行测序。测序平台为Illumina HiSeqTM2500/4000。研究测得的转录组数据已上传国家微生物科学数据中心(NMDC),编号为NMDC40001263。
1.7 real-time PCR采用PrimeScriptTM RT reagent Kit with gDNA Eraser进行cDNA反转录,所用引物见表 1。其中16S作为管家基因。采用SYBR® Premix Ex TaqTM Ⅱ (Tli RNaseH Plus),ROX plus进行real-time PCR,real-time PCR体系(20 μL):2×Master Mix 10 μL、正向引物0.5 μL、反向引物0.5 μL、cDNA 2 μL、ddH2O补足至20 μL。按以下程序进行real-time PCR反应:95 ℃ 30 s;95 ℃ 5 s,60 ℃ 40 s (收集荧光),40个循环。熔解曲线:95 ℃ 10 s;60 ℃ 60 s;95 ℃ 15 s每个样品设置3次平行,利用2–ΔΔCt法分析基因的相对表达量。
| Primer | Primer sequences (5′→3′) |
| algU-F | CGATGTGACCGCAGAGGATG |
| algU-R | ACTGCTGGATGGTCTGGTGC |
| mucA-F | ATGCCGAGCTGCGTTCCACC |
| mucA-R | TCCGCCACGGTCCCTTCTCC |
| mvfR-F | GACATGCTGCGTCTGGTGGA |
| mvfR-R | CGCTGAGGACTGCCAGGGTA |
| lasR-F | AAGTGGAAAATTGGAGTGGAGC |
| lasR-R | GGGTAGTTGCCGACGATGAA |
| bifA-F | GATTTCGGCACCGGCTATTC |
| bifA-R | GCGATGATGTACGCCTCCTG |
| amrZ-F | GCCCACTGAAACAGGCAACT |
| amrZ-R | CCAGGCGAACACCGAGATTG |
| mexC-F | CAAGGCTGGCGACCTGCTGT |
| mexC-R | GGCGGTGGCGGTATCGAAGT |
| oprJ-F | CAAACCTGCTTTCGGCGTAT |
| oprJ-R | GCAGCGAGCGGTTGTTATCC |
| rhlA-F | ACGAGACCGTCGGCAAATAC |
| rhlA-R | GCTCCAGGCAAGCCAAGTAG |
| tpbA-F | TCAGCGTCCTCCGCCAGTTG |
| tpbA-R | TCGTCCTCGTCGCCGAAACC |
| 16S-F | CAGCMGCCGCGGTAATWC |
| 16S-R | CCGTCAATTCMTTTRAGTTT |
1.8 CRAMP修饰肽对PAO1成熟生物被膜藻酸盐的影响
参考文献[9],取藻酸钠对照品建立含量检测标准曲线。采用硫酸铜-盐酸反应体系和1, 3-萘二酚试剂作为反应试剂,乙酸乙酯提取后,在565 nm处测量OD值。按1.3方法制备成熟生物被膜,按上述方法用CRAMP修饰肽处理1 h。利用标准曲线同法测定藻酸盐含量。
1.9 生物信息学分析 1.9.1 质量控制:将测序得到的原始序列进行原始读段过滤,过滤掉带有测序接头的读段、不确定碱基含量大于1%的读段和低质量碱基含量大于50%的读段。同时统计计算干净读段的GC含量和Q20、Q30碱基的比例。
1.9.2 基因表达水平分析:采用HTSeq软件对各样品进行基因表达水平分析,模型为union。结果文件分别统计了不同表达水平下基因的数量以及单个基因的表达水平。一般情况下,使用FPKM值为0.1或者1作为判断基因是否表达的阈值,在我们的流程中,后续分析只分析FPKM > 1的基因。
1.9.3 差异基因分析:采用DEGseq软件进行两组基因差异表达分析,基于泊松分布计算模型确定差异表达基因,利用TMM标准化方法,使用BH (fdr correction with Benjamini/Hochberg)进行多重假设检验校正。对检测结果以log2|Fold Change|>1、P<0.005作为标准进行筛选,对差异基因表达情况进行具体分析。
1.9.4 差异基因功能富集分析:采用GOseq R包软件对差异基因进行GO (Gene Ontology)富集分析[10],校正P < 0.05的OG term为富集显著的差异基因。通过KOBAS软件对KEGG (Kyoto Encyclopedia of Genes and Genomes)中的pathway进行pathway显著性富集分析,P < 0.05的pathway为候选靶基因中显著富集的pathway。
2 结果和分析 2.1 CRAMP修饰肽对铜绿假单胞菌成熟生物被膜的影响在96孔板中CRAMP修饰肽在62.5 μg/mL存在条件下PAO1生物被膜减少率为56.56% (P < 0.01),31.25 μg/mL存在条件下PAO1生物被膜减少率为50.25% (P < 0.01),15.63 μg/mL存在条件下PAO1生物被膜减少率为34.78% (P < 0.05),7.81 μg/mL存在条件下PAO1生物被膜减少率为33.53% (P < 0.05),3.91 μg/mL存在条件下PAO1生物被膜减少率为38.79% (P < 0.01),1.95 μg/mL存在条件下PAO1生物被膜减少率为30.48% (P < 0.05),详见图 1-A。

|
| 图 1 CRAMP修饰肽对PAO1成熟生物被膜的影响 Figure 1 Effect of modified peptide CRAMP on PAO1 mature biofilms. A: the effect of different concentrations of CRAMP modified peptides on the amount of PAO1 mature biofilm in a 96-well plate; B: the effect of CRAMP modified peptide (62.5 μg/mL) on the amount of PAO1 biomass and biofilm cells in the 6-well plate. *: P < 0.05, **: P < 0.01 compare with the control well. |
通过6孔板验证,在62.5 μg/mL条件下,生物被膜量比空白对照组极显著减少了77.17% (P < 0.01)。并且能够极显著地减少生物被膜的活菌数量(P < 0.01),减少了1.16个log10CFU/mL,即90%以上生物被膜细菌被抑杀,详见图 1-B。
2.2 CLSM观察生物被膜形态学变化采用CLSM观察62.5 μg/mL CRAMP修饰肽干预1 h后的成熟生物被膜形态变化,主要包括生物被膜厚度、密度、活菌(SYTO9染色,绿色)和死菌(PI染色,红色)的荧光强度等。结果显示,CRAMP修饰肽能够明显降低生物被膜厚度和密度(图 2-A),显著减少细菌总荧光强度,即SYTO9和PI染色的总荧光强度(P < 0.05,减少了38.94%),详见图 2-B。

|
| 图 2 CLSM观察生物被膜形态学 Figure 2 Biofilms was observed by CLSM. A: representative CLSM orthogonal diagram; B: fluorescence intensity of SYTO and PI. *: P < 0.05, **: P < 0.01, compared with the control well. |
2.3 差异基因表达分析
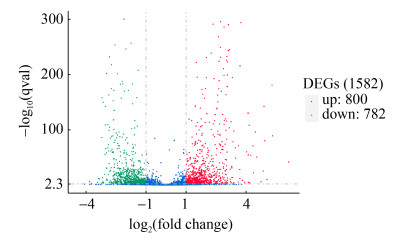
差异表达基因筛选:由图 3可知,上调差异表达显著基因有800个,下调差异表达基因有782个(log2|Fold Change|>1、P<0.005)。
|
| 图 3 差异基因上下调情况 Figure 3 Statistics of up-regulated and down- regulated genes. Each dot represents a gene: blue dots show genes with no significant differences; red dots are significantly up-regulated genes; green dots are significantly down-regulated genes. |
2.4 Real-Time PCR验证分析
对差异基因进行Real-Time PCR验证分析,详见表 2。Real-Time PCR结果显示,选取的10个基因有7个基因同测序结果上下调关系具有一致性,说明高通量测序结果具有可信性。
| Gene ID | Gene | Function | log2|Fold Change| | P-value | Real-time PCR |
| gene-PA1431 | rsaL | Regulatory protein | –1.1429 | 8.15×10–5 | |
| gene-PA1430 | lasR | Transcriptional regulator | –1.1260 | 1.86×10–30 | 6.57 |
| gene-PA1003 | mvfR/pqsR | Transcriptional regulator | –1.6086 | 4.18×10–26 | 2.61 |
| gene-PA3622 | rpoS | RNA polymerase sigma factor | –1.9800 | 3.00×10–247 | |
| gene-PA2227 | vqsM | HTH-type transcriptional regulator | –1.3897 | 2.74×10–4 | |
| gene-PA0425 | mexA | Multidrug resistance protein | 2.1864 | 1.29×10–114 | |
| gene-PA0426 | mexB | Multidrug resistance protein | 2.2789 | 1.51×10–239 | |
| gene-PA0427 | oprM | Outer membrane protein | 2.2742 | 8.21×10–102 | |
| gene-PA4599 | mexC | Resistance-nodulation-cell division (RND) multidrug efflux membrane | 5.3377 | 4.27×10–90 | 7.37 |
| gene-PA4598 | mexD | Resistance-nodulation-cell division (RND) multidrug efflux transporter | 5.3142 | 7.27×10–182 | |
| gene-PA4597 | oprJ | Multidrug efflux outer membrane protein | 6.1423 | 8.2510–43 | 10.98 |
| gene-PA4296 | pprB | Two-component response regulator | –1.5827 | 1.55×10–23 | |
| gene-PA3479 | rhlA | Rhamnosyltransferase subunit A | 1.2056 | 1.01×10–7 | 4.45 |
| gene-PA3478 | rhlB | Rhamnosyltransferase subunit B | 1.1490 | 2.73×10–3 | |
| gene-PA5255 | algQ | Anti-RNA polymerase sigma 70 factor | –1.2800 | 1.34×10–44 | |
| gene-PA5253 | algP | Alginate regulatory protein | –1.4422 | 5.21×10–159 | |
| gene-PA0762 | algU | RNA polymerase sigma factor | 1.5305 | 1.42×10–224 | 6.51 |
| gene-PA0763 | mucA | Sigma factor AlgU negative regulator | 1.1757 | 6.54×10–60 | 11.69 |
| gene-PA0764 | mucB | Sigma factor AlgU regulator | 1.3942 | 1.29×10–49 | |
| gene-PA0766 | mucD | Serine protease MucD | 1.3400 | 8.65×10–76 | |
| gene-PA2241 | pslK | Biofilm formation protein | 1.7675 | 3.19×10–3 | |
| gene-PA2242 | pslL | 1.0941 | 1.59×10–4 | ||
| gene-PA2244 | pslN | –2.1678 | 1.62×10–14 | ||
| gene-PA4108 | PA4108 | Cyclic di-GMP phosphodiesterase | –2.1320 | 9.15×10–16 | |
| gene-PA4367 | bifA | Protein BifA | –1.2398 | 1.52×10–10 | 44.74 |
| gene-PA3385 | amrZ | Alginate and motility regulator Z | 1.2143 | 6.05×10–50 | 26.53 |
| gene-PA3885 | tpbA | Protein tyrosine phosphatase | 4.2479 | 5.44×10–55 | 1.56 |
2.5 差异基因功能富集分析
GO分析是一个国际标准化的基因功能分类体系,包括分子功能(molecular function)、生物过程(biological process)和细胞组成(cellular component) 3个功能描述。以term为基本单位,每一个term代表一个功能分类。由图 4 (只展示了前30个富集最显著的GO term)可知,CRAMP修饰肽处理后的生物被膜细菌生物过程中有15个组为显著富集GO term,细胞组分中有13个组为显著富集GO term,分子功能中有2个组为显著富集GO term。KEGG (Kyoto Encyclopedia of Genes and Genomes)是系统分析基因功能和基因组信息数据的数据库,研究了KEGG中关于生物被膜形成相关的差异显著基因。由表 2可知,CRAMP修饰肽处理后的PAO1成熟生物被膜细菌QS系统pathway中相关基因显著下调。同时对与生物被膜形成调控相关的多糖pathway和c-di-GMP pathway进行了分析。

|
| 图 4 CRAMP修饰肽干预后差异基因GO富集分析 Figure 4 GO enrichment analysis of differentially expressed genes after CRAMP-derived peptide treatment. |
2.6 CRAMP修饰肽对PAO1成熟生物被膜藻酸盐的影响
空白对照组藻酸盐含量为43.96±26.31 (x±s) μg/mL,CRAMP修饰肽干预后藻酸盐含量为9.49±7.61 (x±s) μg/mL,PAO1成熟生物被膜中藻酸盐含量显著下降了78.41% (P < 0.05)。
3 讨论经过96孔板筛选发现CRAMP修饰肽在一定程度上对于PAO1成熟生物被膜呈浓度依赖性抑制,在试验浓度条件下,62.5 μg/mL时达到最高(抑制率56.56%)。而通过6孔板验证其生物被膜量和细菌计数结果显示PAO1成熟生物被膜量抑制率有所上升,这可能是因为两个细胞培养板之间的生物被膜构建体系和接触面不一致导致生物被膜形成的三维结构差异导致的。但总体来讲,CRAMP修饰肽能够极显著地减少PAO1成熟生物被膜。而且通过细菌计数结果显示,生物被膜细菌减少了90%以上。随后,通过激光共聚焦扫描显微镜(CLSM)观察CRAMP修饰肽在62.5 μg/mL存在条件下对PAO1成熟生物被膜的作用。由于细胞培养板材质为聚苯乙烯,会影响CLSM成像时的清晰度,而选用的CLSM专用的腔室盖玻片的底板为玻璃材质,不利于细菌粘附和生物被膜的形成,因此延长了生物被膜形成的时间。结果显示,SYTO9荧光强度与6孔板试验结果具有一致性,生物被膜细菌SYTO9荧光强度显著减少(49.92%),表明PAO1成熟生物被膜细菌已被大量清除,与课题组前期研究相一致。我们课题组成员曾通过对13种宿主防御肽进行筛选,发现仅有CRAMP能明显清除P. aeruginosa (ATCC27853)成熟生物被膜(清除率达50%)[8]。
群体密度感应(quorum sensing,QS)系统能通过信号因子和受体来调节细菌之间的通讯,并调控多种毒力因子和生物被膜的产生。QS系统包括las、rhl、pqs、iqs系统,是一个复杂的调控网络,系统与系统之间存在着调节和负调节关系。本试验中发现,rhlR控制的rhlA和rhlB基因显著上调,其功能主要是控制鼠李糖脂的合成。有研究报道,高浓度的鼠李糖脂能抑制生物被膜的形成和有助于生物被膜的分散[11-12],这与我们观察到的生物被膜量、生物被膜活菌数和CLSM形态学观察等表型结果一致。除此之外,许多转录调节因子也能参与QS系统的调控过程。如QS调节因子VqsM,可通过VqsR调控多种QS关键基因及其高丝氨酸内酯(AHL)的表达[13];QS抑制因子RsaL,其表达下调能够促进3-Oxo-C12-HSL水平增高来激活lasR基因的表达从而维持菌体QS系统的平衡[14-15];压力感受器RpoS,能够通过RpoS增强铜绿假单胞菌耐受性[16]。但CRAMP修饰肽如何导致PAO1生物被膜明显减少,具体是通过减少生物被膜形成还是促进生物被膜分散仍需进一步探索。
环二鸟苷酸(c-di-GMP)是广泛存在于细菌中的第二信使,能调控细菌的运动、生物被膜形成、毒力等多种生物学过程[17]。c-di-GMP主要由二鸟苷酸环化酶(DGC)合成并由磷酸二酯酶(PDE)降解。高水平的c-di-GMP能促使生物被膜的形成,使细菌聚积,减少细菌的运动,低水平的c-di-GMP则与之相反。结果显示,未观察到DGC相关基因显著变化,而PDE相关基因PA4108显著减少。PDE基因的下调会导致c-di-GMP一定程度的上升。但同时还观察到了tbpA基因表达上调,log2|Fold Change|值达到了4.24。TbpA蛋白具有酪氨酸磷酸酶活性,能够使TbpB脱磷酸化使得c-di-GMP含量下降[18]。因此推测由于TbpA过表达使得c-di-GMP含量出现了剧烈下降,导致生物被膜细菌菌体应激性下调了PDE的表达来对抗c-di-GMP含量的下降。另外CRAMP修饰肽还影响了amrZ基因的表达,amrZ基因表达的情况与细菌的生活状态改变相关。最初发现amrZ的功能是参与藻酸盐的生物合成过程,后来发现其对于调节细菌的运动功能至关重要[19]。有研究表明,在浮游细菌中amrZ表达水平更高,而在生物被膜细菌中观察到amrZ基因下调,amrZ缺失株表现为c-di-GMP水平增高[20]。由此推测CRAMP修饰肽有可能通过减少细菌中c-di-GMP的含量来增强细菌的运动能力,促进生物被膜的分散。
生物被膜主要由生物被膜细菌及其胞外多糖复合物(EPS)组成,EPS又由多糖、蛋白质和eDNA组成。铜绿假单胞菌胞外多糖通常由Psl、Pel和藻酸盐(alginate)组成。藻多糖是生物被膜成熟阶段EPS的主要成分,具有维持生物被膜结构及保护生物被膜细菌的双重功能[21]。Psl多糖合成相关的基因pslK、pslL基因表达显著升高、pslN基因表达显著下降,说明生物被膜中Psl多糖含量可能上调[22]。而关于藻酸盐和Pel合成基因并没有观察到显著变化。但c-di-GMP含量变化也能够影响藻酸盐的形成过程。c-di-GMP能够结合Alg44的PliZ区域激活藻酸盐聚合过程,通过抑制Alg44与c-di-GMP结合,可以抑制藻酸盐的分泌[23]。另外,RpoS可介导pprB基因转录,PprB过表达可导致细菌-细菌黏附和细菌-表面黏附增加,这与细菌多糖产生有关。有研究发现,与pfrA基因相关的pprA和pprB基因与algR和algP相似,但不参与铁离子调控[24-25]。提示RpoS可能影响了藻酸盐的生物合成。上述分析推测,CRAMP修饰肽能够减少成熟生物被膜中藻酸盐的含量。为了进一步验证推测结果,对CRAMP修饰肽干预后的PAO1成熟生物被膜藻酸盐含量进行了测定。结果表明CRAMP修饰肽显著减少了PAO1成熟生物被膜中藻酸盐的含量,初步证实了前文的假设,但是否是通过影响c-di-GMP含量的变化来导致藻多糖含量下降还有待进一步研究。
综上所述,CRAMP修饰肽能够减少PAO1成熟生物被膜的量。通过转录组学分析发现,参与PAO1生物被膜调控的几条途径均有变化。通过藻酸盐含量测定,推测CRAMP修饰肽可能通过下调铜绿假单胞菌PAO1的c-di-GMP水平,导致藻酸盐含量减少引起PAO1生物被膜量下降。但c-di-GMP的合成和分解涉及多种不同的DGC酶、PDE酶及转录调控因子的调控,c-di-GMP菌体水平的变化究竟如何还需进一步验证。QS系统、多糖合成系统和c-di-GMP之间应该有着更加复杂且紧密的关系,c-di-GMP系统与QS系统之间的调控通路与网络关系还有待进一步探索。除此之外压力感受系统、双组分调控系统和ppGpp系统也可能参与生物被膜调控过程,还需要进一步挖掘。
| [1] | Rasamiravaka T, Labtani Q, Duez P, El Jaziri M. The formation of biofilms by Pseudomonas aeruginosa: a review of the natural and synthetic compounds interfering with control mechanisms. BioMed Research International, 2015, 2015: 759348. |
| [2] | Haney EF, Trimble MJ, Cheng JT, Valle Q, Hancock REW. Critical Assessment of Methods to Quantify Biofilm Growth and Evaluate Antibiofilm Activity of Host Defence Peptides. Biomolecules, 2018, 8(2): 29. DOI:10.3390/biom8020029 |
| [3] | Jacques M, Aragon V, Tremblay YDN. Biofilm Formation in Bacterial Pathogens of Veterinary Importance. Animal Health Research Reviews, 2010, 11(2): 97-121. DOI:10.1017/S1466252310000149 |
| [4] | Mwangi J, Yin YZ, Wang G, Yang M, Li Y, Zhang ZY, Lai R. The Antimicrobial Peptide ZY4 Combats Multidrug-Resistant Pseudomonas aeruginosa and Acinetobacter baumannii Infection. Proceedings of the National Academy of Sciences, 2019, 116(52): 26516-26522. DOI:10.1073/pnas.1909585117 |
| [5] | Mnif S, Jardak M, Graiet I, Abid S, Driss D, Kharrat N. The novel cationic cell-penetrating peptide PEP-NJSM is highly active against Staphylococcus epidermidis biofilm. International Journal of Biological Macromolecules, 2019, 125: 262-269. DOI:10.1016/j.ijbiomac.2018.12.008 |
| [6] | Wang GS, Mishra B, Epand RF, Epand RM. High-Quality 3D Structures Shine Light On Antibacterial, Anti-Biofilm and Antiviral Activities of Human Cathelicidin LL-37 and its Fragments. Biochimica et Biophysica Acta (BBA)-Biomembranes, 2014, 1838(9): 2160-2172. |
| [7] | 李杨. Cathelicidin相关抗菌肽(CRAMP)在炎性肠病及哮喘小鼠模型中的免疫调控作用与机制. 北京协和医学院博士学位论文, 2016. |
| [8] | Chen HW, Wubbolts RW, Haagsman HP, Veldhuizen EJA. Inhibition and Eradication of Pseudomonas aeruginosa Biofilms by Host Defence Peptides. Scientific Reports, 2018, 8(1). |
| [9] | 陈一强. 绿原酸对铜绿假单胞菌生物膜抑制作用及其机制的体内外研究. 广西医科大学博士学位论文, 2010. |
| [10] |
Qi XZ, Liu L, Wang J. RNA-Seq Reveals Changes of Gene Expression and Cellular Metabolism Caused by Exogenous Oxidative Stress (H2O2) in Foc4. Acta Microbiologica Sinica, 2019, 59(5): 891-906.
(in Chinese) 齐兴柱, 刘磊, 汪军. RNA-Seq揭示Foc4在外源氧化胁迫(H2O2)下的基因表达及细胞代谢变化. 微生物学报, 2019, 59(5): 891-906. |
| [11] | Zheng HW, Singh N, Shetye GS, Jin YC, Li D, Luk YY. Synthetic analogs of rhamnolipids modulate structured biofilms formed by rhamnolipid-nonproducing mutant of Pseudomonas aeruginosa. Bioorganic & Medicinal Chemistry, 2017, 25(6): 1830-1838. |
| [12] |
Li B, Li B, Tian YW, Wang YR, Dou J, Wang H, Zhou CL. The anti-biofilm activity against drug-resistance bacterial of antimicrobial peptide BF-30in vitro. Pharmaceutical Biotechnology, 2015, 22(1): 14-19.
(in Chinese) 李博, 李冰, 田玉伟, 王嫣蓉, 窦洁, 王慧, 周长林. 抗菌肽BF-30体外抗耐药细菌生物被膜的研究. 药物生物技术, 2015, 22(1): 14-19. |
| [13] | Dong YH, Zhang XF, Xu JL, Tan AT, Zhang LH. VqsM, a novel AraC-type global regulator of quorum-sensing signalling and virulence in Pseudomonas aeruginosa. Molecular Microbiology, 2005, 58(2): 552-564. DOI:10.1111/j.1365-2958.2005.04851.x |
| [14] | Rampioni G, Schuster M, Greenberg EP, Zennaro E, Leoni L. Contribution of the RsaL global regulator to Pseudomonas aeruginosa virulence and biofilm formation. FEMS Microbiology Letters, 2009, 301(2): 210-217. DOI:10.1111/j.1574-6968.2009.01817.x |
| [15] | Kang HP, Gan JH, Zhao JR, Kong WN, Zhang J, Zhu M, Li F, Song YQ, Qin J, Liang HH. Crystal structure of Pseudomonas aeruginosa RsaL bound to promoter DNA reaffirms its role as a global regulator involved in quorum-sensing. Nucleic Acids Research, 2017, 45(2): 699-710. DOI:10.1093/nar/gkw954 |
| [16] | Cochran W L, Suh S J, Mcfeters G A, Stewart P S. Role of RpoS and AlgT in Pseudomonas aeruginosa Biofilm Resistance to Hydrogen Peroxide and Monochloramine. Journal of Applied Microbiology, 2000, 88(3): 546-553. DOI:10.1046/j.1365-2672.2000.00995.x |
| [17] |
Zhao LM, Sun HF, Liu ZJ, Lin C, Mao ZZ. Regulation in EPS biosynthesis and transportation by cyclic diguanylate. Microbiology China, 2017, 44(5): 1196-1205.
(in Chinese) 赵腊梅, 孙惠芳, 刘正杰, 林春, 毛自朝. C-di-GMP对细菌胞外多糖合成与运输的调控. 微生物学通报, 2017, 44(5): 1196-1205. |
| [18] | Ueda A, Wood TK. Connecting quorum sensing, c-di-GMP, pel polysaccharide, and biofilm formation in Pseudomonas aeruginosa through tyrosine phosphatase TpbA (PA3885). PLoS Pathogens, 2009, 5(6): e1000483. DOI:10.1371/journal.ppat.1000483 |
| [19] | Baynham PJ, Ramsey DM, Gvozdyev BV, Cordonnier EM, Wozniak DJ. The Pseudomonas aeruginosa Ribbon-Helix-Helix DNA-binding Protein AlgZ (AmrZ) Controls Twitching Motility and Biogenesis of Type Ⅳ Pili. JournaL of Bacteriology, 2006, 188(1): 132-140. DOI:10.1128/JB.188.1.132-140.2006 |
| [20] | Hou LL, Debru A, Chen QQ, Bao QY, Li KW. AmrZ Regulates Swarming Motility through Cyclic di-GMP-Dependent Motility Inhibition and Controlling Pel Polysaccharide Production in Pseudomonas aeruginosa PA14. Frontiers in Microbiology, 2019: 10. |
| [21] | Schurr MJ. Which bacterial biofilm exopolysaccharide is preferred, Psl or alginate?. Journal of Bacteriology, 2013, 195(8): 1623-1626. DOI:10.1128/JB.00173-13 |
| [22] | Byrd MS, Sadovskaya I, Vinogradov E, Lu HP, Sprinkle AB, Richardson SH, Ma LY, Ralston B, Parsek MR, Anderson EM, Lam JS, Wozniak DJ. Genetic and biochemical analyses of the Pseudomonas aeruginosa Psl exopolysaccharide reveal overlapping roles for polysaccharide synthesis enzymes in Psl and LPS production. Molecular Microbiology, 2009, 73(4): 622-638. DOI:10.1111/j.1365-2958.2009.06795.x |
| [23] | Zhou E, Seminara AB, Kim SK, Hall CL, Wang Y, Lee VT. Thiol-benzo-triazolo-quinazolinone inhibits Alg44 binding to c-di-GMP and reduces alginate production by Pseudomonas aeruginosa. ACS Chemical Biology, 2017, 12(12): 3076-3085. |
| [24] | Wang CC, Chen WH, Xia AG, Zhang RR, Huang YJ, Yang S, Ni L, Jin F. Carbon Starvation Induces the Expression of PprB-Regulated Genes in Pseudomonas aeruginosa. Applied and Environmental Microbiology, 2019, 85(22). DOI:10.1101/639112 |
| [25] | Venturi V, Otten M, Korse V, Brouwer B, Leong J, Weisbeek P. Alginate Regulatory and Biosynthetic Gene Homologs in Pseudomonas Putida WCS358:Correlation with the Siderophore Regulatory Gene pfrA. Gene, 1995, 155(1): 83-88. |
 2021, Vol. 61
2021, Vol. 61




