中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 袁文杰, 郭亚琼, 韩毅, 朱宝利. 2021
- Wenjie Yuan, Yaqiong Guo, Yi Han, Baoli Zhu. 2021
- 非小细胞肺癌患者肠道微生物特征分析
- Analysis on characteristics of microbiome structure in non-small cell lung cancer patients
- 微生物学报, 61(9): 2776-2790
- Acta Microbiologica Sinica, 61(9): 2776-2790
-
文章历史
- 收稿日期:2020-11-06
- 修回日期:2021-01-23
- 网络出版日期:2021-08-27
2. 中国科学院微生物研究所, 北京 100101;
3. 中国科学院大学, 北京 100049
2. Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China;
3. University of Chinese Academy of Sciences, Beijing 100049, China
肺癌是目前最常见的恶性肿瘤之一,在男性恶性肿瘤发病中居首位,每年新发病例约52.0万,在女性中每年发病约为30.4万,仅次于乳腺癌,高居第二[1-3]。肺癌发病隐匿,患者早期可以没有任何症状或症状缺乏特异性,当患者出现症状就诊时,往往已处于疾病进展期,甚至已经发生局部或远处转移。目前随着低剂量CT的普及,加上纤维支气管镜和胸腔镜等诊疗手段的应用,已经大大提高了早期肺癌的检出率,但肺癌依旧面临难发现、难治疗和预后差等困境,5年生存率仅为15%–18%[4]。人体内寄居着数以万计的微生物群落,这些微生物群落可以帮助人体消化吸收营养物质、参与机体的代谢调节过程、合成维生素或调控机体免疫应答。自“肠-肺轴”的存在被发现以来,不断有研究证明肠道菌群在肺部疾病的发生发展过程中有着重要的作用[5-10]。Zhuang及其团队发现非小细胞肺癌患者与健康人群有着不同的肠道菌群构成并且某些菌群有可能成为潜在的非小细胞肺癌预测标志物[11]。另一方面,Ji及其团队也利用特定的肠道菌群标志物建立菌群预测模型来预测早期非小细胞肺癌[12]。Liu等则将目光聚焦在肿瘤标志物与肠道菌群之间的关系,不同的肿瘤标志物可以对非小细胞肺癌病理类型的诊断有辅助作用,他们认为如果能找到肠道菌群与肿瘤标志物之间的关系以后可以将肠道菌群作为一个新型诊断方法。随着靶向治疗的逐渐完善,免疫治疗的兴起,非小细胞肺癌治疗已经取得巨大进展。然而这些治疗方法都只针对一部分患者,并且在一段时间之后都会出现不同程度的耐药[13-14]。Lu等分析了免疫治疗的非小细胞肺癌患者的肠道微生物之后发现,拥有更高肠道菌群多样性患者的免疫治疗效果更好,并且无进展生存期(PFS)显著延长[15]。因此人体微生物群落的临床价值受到了前所未有的重视,展现出了很好的临床应用前景。有研究认为微生物促进肿瘤发生的潜在机制包括:菌群失调、基因毒性及毒性作用、代谢、炎症和免疫反应等。以上研究均关注非小细胞肺癌患者与健康人群的肠道菌群,而忽略了肺部良性病变人群。在某些肿瘤中有明确的癌前病变阶段,临床上常见的癌前病变有萎缩性胃炎、宫颈糜烂、乳腺囊性增生和胃肠道息肉等。在这个阶段组织细胞形态与起源组织的细胞形态存在着差异并逐渐加重,但是尚未成为癌细胞。肺癌的癌前病变尚未见报道,但是有研究表明慢性阻塞性肺疾病(COPD)是肺癌的危险因素之一[16-18]。因此我们希望从肠道微生物的角度出发探究非小细胞肺癌与肺部良性病变之间的关系,希望能对后续的研究有一定的启发。
本研究收集在2019年1月至2019年12月就诊于首都医科大学附属北京胸科医院的39例非小细胞肺癌患者(non-small-cell lung cancer,NSCLC)以及24例肺部良性病变患者(benign lesions,BL)总共63例粪便样本。采用16S rDNA测序手段对粪便标本进行高通量二代测序,并对不同性别、年龄、吸烟史、临床分期及病理类型进行分析比较,拟进行菌群微生态在非小细胞肺癌患者及肺部良性病变患者肠道中的变化及关联研究,为判断菌群在疾病发生发展过程中的作用提供理论依据。
1 材料和方法 1.1 纳入和排除标准 1.1.1 纳入标准:(1) 年龄大于18岁;(2) 知情同意参加本课题研究;(3) 临床怀疑非小细胞肺癌患者、肺部良性病变患者;(4) 无消化道相关手术史,无其他器官肿瘤病史。
1.1.2 排除标准:(1) 一个月内使用过抗生素者;(2) 目前患有急、慢性胃肠疾病;(3) 有精神疾患史;(4) 怀孕期、哺乳期妇女。
1.2 一般资料收集2019年1月至2019年12月在首都医科大学附属北京胸科医院确诊的39例非小细胞肺癌患者,其中肺腺癌(lung adenocarcinoma,ADC) 32例,肺鳞癌(lung squamous cell carcinomas,SCC) 7例以及24例肺部良性病变患者的粪便标本以及临床资料。男性36例,女性27例,年龄平均57 (±13),从未吸烟的患者39例,吸烟患者24例(曾经吸烟患者归到吸烟组)。分别对性别、年龄和吸烟史进行统计学分析,其中非小细胞肺癌患者与肺部良性病变患者之间的性别、年龄和吸烟史并无统计学差异(表 1)。而在临床分期中,Ⅰ、Ⅱ、Ⅲ、Ⅳ期患者分别占比59%、3%、19%、19%,Ⅰ期患者占了绝大多数。
| Characteristics | NSCLC | NSCLC vs BL P value | |||
| Total (n=63) | ADC (n=32) | SCC (n=7) | BL (n=24) | ||
| Sex | 1.0000 | ||||
| Female | 27 | 17 (53.1) | 0 (0) | 10 (41.7) | |
| Male | 36 | 15 (46.9) | 7 (100) | 14 (58.3) | |
| Age (years) | 0.2484 | ||||
| Mean (±SD) | 57 (±13) | 58 (±10) | 63 (±12) | 55 (±17) | |
| Smoking status | 1.0000 | ||||
| Never smoker | 39 | 24 (75.0) | 0 (0) | 15 (62.5) | |
| Smoker | 24 | 8 (25.0) | 7 (100) | 9 (37.5) | |
| Disease stage | |||||
| Ⅰ | 20 | 19 (59.4) | 1 (14.3) | NA | |
| Ⅱ | 2 | 1 (3.1) | 1 (14.3) | NA | |
| Ⅲ | 9 | 6 (18.8) | 3 (42.9) | NA | |
| Ⅳ | 8 | 6 (18.8) | 2 (28.6) | NA | |
| Distant metastasis | |||||
| Yes | 8 | 6 (18.8) | 2 (28.6) | NA | |
| No | 30 | 26 (81.2) | 4 (57.1) | NA | |
| Uncertain | 1 | 0 (0) | 1 (14.3) | NA | |
| Mean±SD was used for continuous variable data, and percentage was used for categorical variables; continuous variables were compared using the Student’s t test, categorical variables were analyzed using Fisher’s test; ADC: lung adenocarcinoma; SCC: lung squamous cell carcinoma; NSCLS: non-small-cell lung cancer; BL: benign lesions. P < 0.05 indicated statistical significance. | |||||
1.3 实验仪器和材料
非小细胞肺癌患者和肺部良性病变患者粪便样本(加有保存液);DNA提取试剂盒:QIAamp® PowerFecal® DNA Kit (50);各种规格移液器及吸头、涡旋振荡器、高速离心机、多样品组织研磨机、微波炉、Nanodrop检测仪、电泳仪和凝胶成像仪。
1.4 粪便采集处理向患者分发粪便采集套装并指导患者根据套装说明书上的步骤正确采集粪便样本。之后尽快放置于–20 ℃冰箱保存,待后续实验室处理检测。
1.5 粪便DNA提取将适量粪便(150–250 mg)加入PowerBead Tube并利用高通量组织研磨机将粪便标本在60 Hz破碎1 min。之后根据DNA提取试剂盒说明书严格按实验步骤提取粪便中的DNA,取2 μL DNA使用Nanodrop测量DNA浓度。之后琼脂糖凝胶电泳检测DNA质量。
1.5.1 构建PCR反应体系:从–20 ℃冰箱中取出KAPA HiFi酶、5×GC buffer、dNTPs、通用引物F (5′-CCTACGGGNBGCASCAG-3′)和引物R (5′-GTGCCAGCMGCCGCGGTAA-3′)、ddH2O及模板DNA,放在冰上融化,根据事先计算好的剂量在PCR管中加入反应体系。
第一次PCR反应体系:GC buffer 5 µL,dNTPs 0.5 µL,DNA 50–100 ng,扩增引物F (1st3F,1×10–11 mol/L) 0.5 µL,扩增引物R (1st4R,1×10–11 mol/L) 0.5 µL,KAPA HiFi 0.5 µL,ddH2O补足,Total 25 µL。
第二次PCR反应体系:ddH2O 10.75 µL、GC buffer 5 µL、dNTPs 0.75 µL、DNA 5 µL、扩增引物F (2stXF,5×10–12 mol/L) 1.5 µL、扩增引物R (2stXR,5×10–12 mol/L) 1.5 µL、KAPA HiFi 0.5 µL、Total 25 µL。
1.5.2 PCR条件:预变性95 ℃ 3 min,变性95 ℃ 30 s,退火55 ℃ 30 s,延伸72 ℃ 30 s,终末延伸72 ℃ 5 min,25个循环。第一次PCR需要25个循环,第二次PCR需要8个循环。琼脂糖凝胶电泳检测PCR扩增结果,电泳图谱保存为.tiff格式。
1.5.3 上机测序:按照步骤纯化扩增成功的样本。管盖上注明样本名或样本编号,管壁上注明文库构建日期及barcode号码。利用Illumina NovaSeq 6000高通量测序平台对样本微生物16S rDNA的V3–V4区进行二代测序。
1.6 生物学分析通过操作分类单元(operational taxonomic units,OTU)聚类及物种注释分析、alpha多样性分析、beta多样性分析来比较样本中的微生物组成及丰度。
1.7 统计学分析计数指标进行正态性检验,对服从正态分布的数据使用均数±标准差。病人一般资料数据中的连续型变量使用Student’s t test检验方法,分类变量使用Fisher’s test检验;alpha多样性指数以及菌群在患者中的差异是基于Wilcoxon rank-sum test得出的统计结果。Beta多样性则是使用基于Bray-Cutis距离的PCoA分析。P < 0.05时认为有统计学意义,P < 0.01时认为在统计学上有显著性差异。所有统计分析均使用SPSS 20版本进行统计学处理分析。
2 结果和分析 2.1 OTU聚类分析63例样本总共得到8681个OTU,有431个OTU对应到门水平,825个OTU对应到纲水平,864个OTU对应到目水平,1490个OTU对应到科水平,5071个OTU对应到属水平(图 1)。测序覆盖率(coverage)均值为99.72%,测序深度使用alpha多样性中的chao1及Shannon稀释曲线来衡量。曲线逐渐平缓,当测序量继续增加,对OTU数的增长贡献不大,说明测序量已经足够,增加测序量不能提供更多新的OTU。

|
| 图 1 OTU物种注释结果统计图 Figure 1 OTU taxonomy bar plot. |
2.2 物种丰度分析
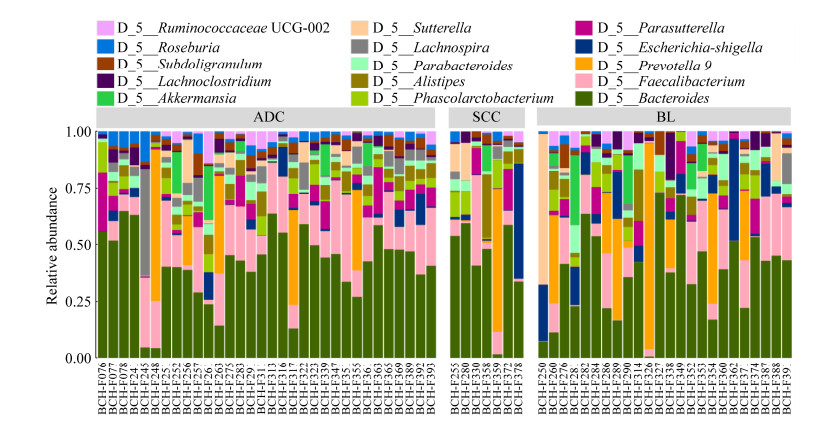
根据OTU物种注释分析结果,非小细胞肺癌患者与肺部良性病变患者两组人群中最主要的菌门为:拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)、变形菌门(Proteobacteria),最主要的菌纲为:拟杆菌纲(Bacteroidia)、梭菌纲(Clostridia)、厚壁菌纲(Negativicutes)、beta-变形菌纲(Betaproteobacteria)、γ-变形菌纲(Gammaproteobacteria),最主要的菌目为:拟杆菌目(Bacteroidales)、梭菌目(Clostridiales)、伯克氏菌目(Burkholderiales)、肠杆菌目(Enterobacteriales)、梭杆菌目(Fusobacteriales),最主要的菌科为:拟杆菌科(Bacteroidaceae)、瘤胃菌科(Ruminococcaceae)、毛螺菌科(Lachnospiraceae)、普雷沃氏菌科(Prevotellaceae)、产碱杆菌科(Alcaligenaceae)、酸乳杆菌科(Acidaminococcaceae),最主要的菌属为(图 2):拟杆菌属(Bacteroides)、粪杆菌属(Faecalibacterium)、普雷沃菌属(Prevotella)、副萨特氏菌属(Parasutterella)、考拉杆菌属(Phascolarctobacterium)、罕见小球菌属(Subdoligranulum) (表 2)。我们使用Wilcoxon秩和检验初步对2组样本进行统计分析后发现,3种菌门与3种菌属在2组间的占比存在统计学差异,分别为厚壁菌门(Firmicutes)、放线菌门(Actinobacteria)、变形菌门(Proteobacteria)、粪杆菌属(Faecalibacterium)、毛螺菌属(Lachnospira)以及罗氏菌属(Roseburia)。其中厚壁菌门(Firmicutes)在肺腺癌患者与肺部良性病变患者中丰度增加。变形菌门(Proteobacteria)在非小细胞肺癌患者中丰度增加,放线菌门(Actinobacteria)则是在肺部良性病变患者中显著增加(表 3)。
|
| 图 2 属层次菌落相对丰度柱状图 Figure 2 Relative abundance of gut bacteria at the genus level in each sample. |
| Taxa | ADC mean/% | SCC mean/% | BL mean/% |
| Phylum | |||
| Bacteroidetes | 49.90 | 51.59 | 43.16 |
| Firmicutes | 28.61 | 31.74 | 53.52 |
| Proteobacteria | 23.25 | 15.92 | 2.18 |
| Class | |||
| Bacteroidia | 47.90 | 36.53 | 9.38 |
| Clostridia | 17.07 | 50.50 | 32.99 |
| Negativicutes | 11.17 | 0.31 | 5.82 |
| Betaproteobacteria | 21.57 | 9.48 | 34.47 |
| Gammaproteobacteria | 0.53 | 0.15 | 16.10 |
| Order | |||
| Bacteroidales | 47.90 | 47.03 | 45.46 |
| Clostridiales | 17.07 | 7.52 | 40.03 |
| Selenomonadales | 11.17 | 6.76 | 0.03 |
| Burkholderiales | 21.57 | 10.91 | 0.96 |
| Enterobacteriales | 0.53 | 3.22 | 11.41 |
| Fusobacteriales | – | 24.00 | 1.42 |
| Family | |||
| Bacteroidaceae | 46.74 | 34.87 | 7.86 |
| Ruminococcaceae | 2.58 | 39.95 | 20.95 |
| Lachnospiraceae | 14.36 | 7.90 | 16.10 |
| Prevotellaceae | – | 1.14 | 27.63 |
| Alcaligenaceae | 21.64 | 10.02 | 1.26 |
| Porphyromonadaceae | 1.32 | 2.10 | 5.99 |
| Acidaminococcaceae | 11.16 | 0.31 | 6.52 |
| Genus | |||
| Bacteroides | 55.80 | 40.81 | 41.40 |
| Faecalibacterium | 0.04 | 39.79 | 20.28 |
| Parasutterella | 25.84 | 11.70 | 7.36 |
| Phascolarctobacterium | 13.32 | 0.36 | 0.94 |
| Alistipes | – | 0.54 | 7.98 |
| Subdoligranulum | – | – | 10.14 |
| Bacterial | NSCLC mean/% | BL mean/% | P value |
| Firmicutes | 28.6 | 53.52 | 0.001 |
| Actinobacteria | 0.14 | 1.12 | 0.04 |
| Proteobacteria | 23.35 | 2.18 | 0.0029 |
| Faecalibacterium | 37.79 | 20.28 | 0.04 |
| Lachnospira | 0.03 | 0.21 | 0.0001 |
| Roseburia | 1.31 | 3.96 | 0.001 |
| Wilcoxon rank-sum test was used to test the difference between groups. P < 0.05 indicated statistical significance. | |||
2.3 组内特有及组间共有OTU分析
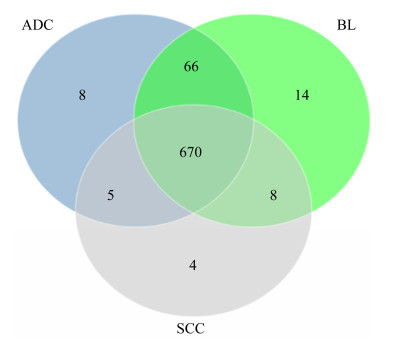
OTU聚类分析后,我们发现63例样本中有6个OTU是共有的(覆盖100%样本的微生物菌群),它们对应的注释菌群主要是:粪杆菌属(Faecalibacterium)、副萨特氏菌属(Parasutterella)、拟杆菌属(Bacteroides)、埃希氏菌-志贺氏菌属(Escherichia-Shigella),这提示上述菌群有可能是人体肠道菌群的基本组成部分。分析样本中OTU的数量,3组共有775个OTU,肺腺癌患者有8个特有OTU,肺鳞癌患者有4个特有OTU,肺部良性病变患者有14个特有OTU。而3种人群共有的OTU为670,说明大部分菌群为三者共有,小部分特殊菌群为部分人群特有(图 3)。
|
| 图 3 三组间韦恩图 Figure 3 Venn diagram between three groups. |
2.4 Alpha多样性分析
分别对alpha多样性的各个指数进行秩和检验(表 4)。Chao1指数体现物种丰富度,Shannon和Simpson指数则反映物种多样性和均匀度。我们可以看出非小细胞肺癌组与肺部良性病变组的物种丰富度无明显差异,而物种多样性则有显著差异。肺腺癌同肺部良性病变患者相比,Shannon指数升高,提示肺腺癌患者的肠道菌群多样性升高。
| Alpha-diversity | ADC mean | SCC mean | BL mean | ADC vs BL (P value) | SCC vs BL (P value) | ADC vs SCC (P value) |
| Observed species | 352 | 318.57 | 293.46 | 0.051 | 0.8 | 0.089 |
| Chao1 | 420.34 | 419.61 | 377.72 | 0.15 | 0.295 | 0.957 |
| Shannon | 5.25 | 4.71 | 4.76 | 0.038* | 0.695 | 0.058 |
| Simpson | 0.93 | 0.90 | 0.91 | 0.108 | 0.532 | 0.082 |
| Goods coverage | 0.998 | 0.998 | 0.997 | 0.446 | 0.341 | 0.359 |
| ADC: lung adenocarcinoma; SCC: lung squamous cell carcinoma; NSCLS: non-small-cell lung cancer; BL: benign lesions; Kruskal-Wallis test was used to test the difference between groups. P < 0.05 indicated statistical significance. | ||||||
2.5 Beta多样性分析
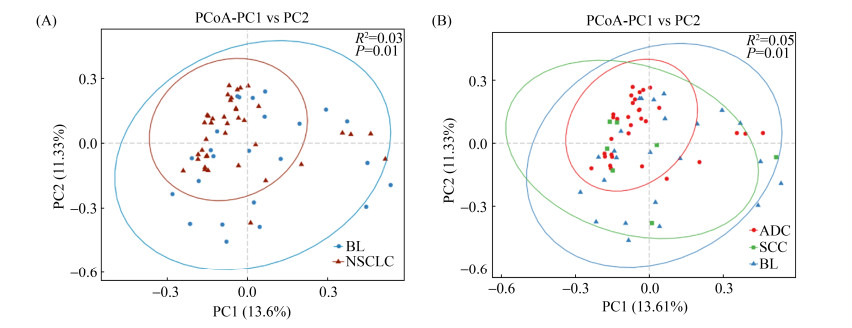
我们使用主坐标分析(principal coordinate analysis,PCoA)将多维数据降维,对样本之间的物种多样性关系进行描述。如果样本的物种丰度和构成越相似,则它们在PCoA图中的距离越近。我们从结果中可以看出,非小细胞肺癌患者与肺部良性病变患者之间的肠道菌群物种多样性存在差异且组间差异大于组内差异(图 4)。
|
| 图 4 PCoA图 Figure 4 PCoA between groups based on the Bray-Cutis. A: non-small cell lung cancer and benign lesion; B: ADC, SCC and benign lesion. Significant differences were observed with Adonis test. |
2.6 差异分析 2.6.1 LDA effect size (LEfSe)分析:
由beta多样性分析结果可知,非小细胞肺癌患者与肺部良性病变患者的肠道菌群存在一定的差异。我们通过LEfSe分析得到具有统计学差异的biomarker,即不同组间丰度有显著差异的菌群。结果显示(图 5),属水平中的毛螺菌属(Lachnospira)、瘤胃球菌属(Ruminococcus)、粪杆菌属(Faecalibacterium)、罗氏菌属(Roseburia)和纺锤链杆属(Fusicatenibacter)在肺腺癌患者的肠道菌群中显著富集,变化存在统计学差异。嗜血杆菌属(Haemophilus)、普雷沃菌属(Prevotella)、链球菌属(Streptococcus)和柠檬酸杆菌属(Citrobacter)在肺部良性病变患者肠道中显著富集,变化存在统计学差异。经分析未发现在肺鳞癌中显著富集的细菌。

|
| 图 5 LDA值分布柱状图 Figure 5 LDA bar between ADC and BL. |
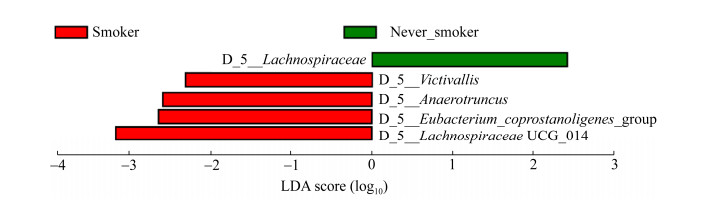
在表 1中,吸烟因素在非小细胞肺癌患者与肺部良性病变患者之间没有显著差异,但吸烟已经被证实是非小细胞肺癌发病非常重要的危险因素,于是我们进一步分析了与吸烟相关的差异菌群(图 6)。我们发现属水平食物谷菌属(Victivallis)、瘤胃球菌属(Ruminococcus)、厌氧棍状菌属(Anaerotruncus)和真杆菌属(Eubacterium)在非小细胞肺癌吸烟患者肠道菌群中显著富集。
|
| 图 6 LDA值分布柱状图 Figure 6 LDA bar between smoker and non-smoker. |
2.6.2 非小细胞肺癌分期及转移情况相关的肠道微生物组成比较:
我们接着根据非小细胞肺癌患者的临床分期以及是否有远处转移来比较肠道微生物的组成(图 7、8)。发现只有韦荣球菌(Erysipelotrichaceae UCG-003)与非小细胞肺癌患者临床分期有关。接着又比对分析发生转移(metastasis,M)与未发生转移(nonmetastasis,NM)非小细胞肺癌患者肠道菌群的丰度差异,发现纺锤链杆属(Fusicatenibacter)在未发生转移的非小细胞肺癌患者中丰度升高,瘤胃球菌属1 (Ruminococcus 1)则是在发生转移的患者中丰度升高。
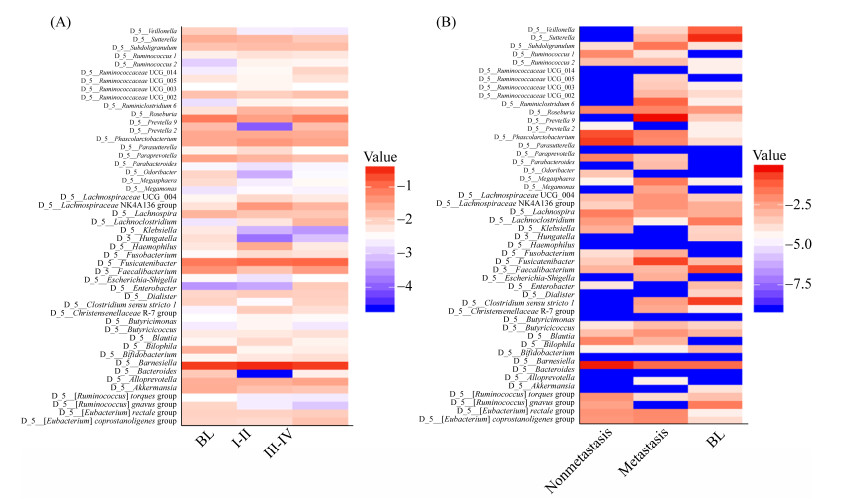
|
| 图 7 基于分期、远处转移的肠道菌群热图 Figure 7 Heat map based on (A) clinical stages and (B) metastatic status. Heat map showing the relative abundances of the 50 most abundant species in samples grouped by clinical stages and metastatic status, species were in rows, and the relative abundance was indicated by color gradient. |

|
| 图 8 分期、远处转移相关肠道菌群 Figure 8 Gut microbiota associated with clinical variables. The most significantly changed species were further analyzed in each group (A, B). P values were adjusted by FDR method. *: P < 0.05, **: P < 0.01, ***: P < 0.001. |
3 讨论
通过二代高通量测序手段,分析非小细胞肺癌患者与肺部良性病变患者肠道微生物群落的特异性及多样性,这对研究微生物与人类疾病的关系有着重要的理论和现实意义。许多研究对肺部疾病患者和健康人群的肺部微生物群落结构进行了描述,发现肺部疾病患者有其独特的微生物结构并且与健康人群存在显著差异[5, 19]。我们的结果表明,两组患者在门水平的菌群主要是:拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)。在属水平主要是:拟杆菌属(Bacteroides)、粪杆菌属(Faecalibacterium)、普雷沃菌属(Prevotella)、副萨特氏菌属(Parasutterella)、考拉杆菌属(Phascolarctobacterium)、罕见小球菌属(Subdoligranulum)。我们发现非小细胞肺癌患者与肺部良性病变患者的alpha多样性并无明显差别,而beta多样性则有显著差异。这与Zhuang等[11]的研究结论相类似,不过他们研究的并不是肺部良性病变患者,而是健康人群。根据我们的beta多样性分析与LefSe差异分析结果,非小细胞肺癌患者与肺部良性病变患者的肠道菌群存在差异且拥有其特异菌群。之后我们根据患者临床分期与肿瘤转移状态再次对两组患者进行分析,发现不同分期、不同转移状态下的非小细胞肺癌患者同样存在差异菌群。我们发现放线菌门(Actinobacteria)、嗜血杆菌属(Haemophilus)、普雷沃菌属(Prevotella)、链球菌属(Streptococcus)、柠檬酸杆菌属(Citrobacter)在肺部良性病变患者中富集。Kundu等的研究结果认为,放线菌门(Actinobacteria)包含大量的共生物种,是健康人群体内微生物群落的重要组成部分。Rangan团队提出一项假设,他们认为肠道放线菌的二次代谢产物对恶性肿瘤细胞有抑制作用[20]。之后另一项研究结果证实了这个假设,研究者们发现从健康孩子的肠道中分离提取出来的放线菌二次代谢产物有潜在的肿瘤抑制作用[21]。同时一项关于乳腺癌的研究结果发现在良性乳腺病变患者中也拥有更高丰度的链球菌属(Streptococcus)[22]。同我们的结果相似,链球菌属(Streptococcus)也同样在肺部良性病变患者中显著富集。但是与此同时也有研究认为链球菌可以侵入人体,靶向体内的纤维连接蛋白,在特定的环境下诱导细胞因子反应,从而引起炎症和感染的发生,而持续的肺部感染和炎症已被证实有致癌作用[23-25]。我们猜测出现这种结果有两种可能性,一是这些肺部良性病变患者可能处于癌前病变阶段。在病灶还未转变成肿瘤之前就被手术切除。二是,链球菌是引起肺炎的主要微生物,肺部良性病变患者组中也包括肺炎患者,有可能是这些肺炎患者对研究结果产生了影响。后续的差异分析结果令我们感到意外,我们发现毛螺菌属(Lachnospira)、瘤胃球菌属(Ruminococcus)在肺腺癌患者中富集,因为许多文献表明这两者都是可以帮助人体对抗肿瘤生成的。毛螺菌属(Lachnospira)可以通过产生丁酸来保护人体免受肿瘤侵害,而瘤胃球菌属(Ruminococcus)则与免疫检查点活性与毒性有关[26]。我们猜测这两种菌群是肿瘤引起的富集菌群,是机体在对抗肿瘤形成的过程中出现的改变,后续需要更多的研究来对这两种菌群进行一些功能通路或代谢组学的分析从而来进一步明确这两种菌群在肿瘤发生发展中所扮演的角色。而粪杆菌属(Faecalibacterium)在肺腺癌患者中的富集是令我们欣喜的,因为粪杆菌属(Faecalibacterium)同抗PD-1治疗有协同作用,可以改善非小细胞肺癌免疫治疗的疗效。越来越多的证据表明,我们可以利用肠道微生物来改善抗肿瘤治疗疗效以及减轻化疗相关副作用。这是因为肠道微生物可以在维生素代谢、食物代谢中起到重要作用,还可以阻止肠道病原体的扩张及传播[27-28]。但是遗憾的是我们未能发现肺鳞癌患者肠道中的特异菌群,或许这同我们的肺鳞癌患者样本偏少有关,希望后续的研究能增加肺鳞癌患者的数量。
根据“肠-肺轴”理论,肠道微生物可以维持促肿瘤炎症反应和抗肿瘤免疫反应之间的平衡,细菌和它的代谢产物都有可能对人体有着系统性的影响[29-31]。一项大型流行病学研究表明,在反复使用抗生素的情况下,非小细胞肺癌的发病率会增加。这为菌群与非小细胞肺癌之间可能存在的联系提供了间接的证据。有研究证实短链脂肪酸有可能是肠道远距离作用的潜在介质,可以通过激活肠道/循环免疫系统间接或直接影响靶器官[14, 32-34]。例如:肠道粘膜以及肠系膜淋巴结中都存在着大量巨噬细胞和其他免疫细胞,细菌会被粘膜下层或肠系膜淋巴结里面的免疫细胞或巨噬细胞吞噬并消灭,如果不能通过这第一道防线将细菌消灭,这些幸存的细菌、细胞壁碎片或死亡细菌的蛋白碎片会与肠道产生的细胞因子和化学因子一起逃逸,经肠系膜淋巴系统进入乳糜池,从而进入血液循环系统。因此它们可以进入肺循环引起局部树突状细胞和巨噬细胞的激活以及T细胞的激活和分化[14, 35-36]。肠道菌群影响肺部的另一种机制可能是在肠粘膜受到抗原攻击之后,产生和激活的免疫细胞发生了自身转移。例如在结肠腺瘤小鼠模型中,粘膜上皮屏障的破坏会诱发促炎症因子的分泌,进而引起肿瘤发生[37-38]。微生物群落对免疫系统的作用不单单只有促进肿瘤发生,它们也可以激活抗肿瘤监视来预防癌症或至少可以限制肿瘤的生长。微生物产物可以塑造T细胞储藏库,通过产生对细菌及肿瘤抗原的特异性交叉反应T细胞来调节肿瘤反应[39-41]。从肿瘤部位中分离出的某些细菌可以引起DNA双链的断裂,从而诱导癌症的发生,但是肠道菌群失调与非小细胞肺癌之间是否也存在这种机制仍然不明确。
本研究的几个局限性:(1) 我们只收集了肠道样本,没有进行多部位的菌群分析。对说明微生物与肿瘤之间关系的作用有限。(2) 没有收集健康人群的样本作为对照组进行分析,结果缺乏一定的代表性。(3) 由于肿瘤诊断的特殊性,我们无法在收样前获取准确的病理诊断,导致样本病理分型之间存在不平衡。肺腺癌样本明显多于肺鳞癌样本,可能会对结果产生一定影响。(4) 没有事先对患者饮食习惯做详细调查,而饮食对肠道菌群有着重要的影响。
总的来说,本次研究只是对非小细胞肺癌患者及肺部良性病变患者进行了一次描述性研究,接下来仍然需要对微生物在肿瘤发生发展中的作用机制进行研究。希望我们的结果能对后续的微生物研究提供一个新的思路,同时也能够为肿瘤的诊断和治疗提供新的策略和方法。
| [1] |
Zheng RS, Sun KX, Zhang SW, Zeng HM, Zou XN, Chen R, Gu XY, Wei WQ, He J. Report of cancer epidemiology in China, 2015. Chinese Journal of Oncology, 2019, 41(1): 19-28.
(in Chinese) 郑荣寿, 孙可欣, 张思维, 曾红梅, 邹小农, 陈茹, 顾秀瑛, 魏文强, 赫捷. 2015年中国恶性肿瘤流行情况分析. 中华肿瘤杂志, 2019, 41(1): 19-28. DOI:10.3760/cma.j.issn.0253-3766.2019.01.005 |
| [2] | Mao Y, Yang D, He J, Krasna M J. Epidemiology of lung cancer. Surgical Oncology Clinics of North America, 2016, 25(3): 439-445. DOI:10.1016/j.soc.2016.02.001 |
| [3] | Chen WQ, Zheng RS, Zeng HM, Zhang SW. Epidemiology of lung cancer in China. Thoracic Cancer, 2015, 6(2): 209-215. DOI:10.1111/1759-7714.12169 |
| [4] | de Groot P, Munden RF. Lung cancer epidemiology, risk factors, and prevention. Radiologic Clinics of North America, 2012, 50(5): 863-876. DOI:10.1016/j.rcl.2012.06.006 |
| [5] | Lee JT, Kim CM, Ramakrishnan V. Microbiome and disease in the upper airway. Current Opinion in Allergy and Clinical Iimmunology, 2019, 19(1): 1-6. DOI:10.1097/ACI.0000000000000495 |
| [6] | Lynch SV. The lung microbiome and airway disease. Annals of the American Thoracic Society, 2016, 13 Suppl 2(Suppl 5): S462-5. |
| [7] | Budden KF, Gellatly SL, Wood DLA, Cooper MA, Morrison M, Hugenholtz P, Hansbro PM. Emerging pathogenic links between microbiota and the gut-lung axis. Nature Reviews Microbiology, 2017, 15(1): 55-63. DOI:10.1038/nrmicro.2016.142 |
| [8] | Enaud R, Prevel R, Ciarlo E, Beaufils F, Wieërs G, Guery B, Delhaes L. The gut-lung axis in health and respiratory diseases: a place for inter-organ and inter-kingdom crosstalks. Frontiers in Cellular and Infection Microbiology, 2020, 10: 9. DOI:10.3389/fcimb.2020.00009 |
| [9] | He Y, Wen Q, Yao FF, Xu D, Huang YC, Wang JS. Gut-lung axis: the microbial contributions and clinical implications. Critical Reviews in Microbiology, 2017, 43(1): 81-95. DOI:10.1080/1040841X.2016.1176988 |
| [10] | Dumas A, Bernard L, Poquet Y, Lugo-Villarino G, Neyrolles O. The role of the lung microbiota and the gut-lung axis in respiratory infectious diseases. Cellular Microbiology, 2018, 20(12): e12966. DOI:10.1111/cmi.12966 |
| [11] | Zhuang H, Cheng L, Wang Y, Zhang YK, Zhao MF, Liang GD, Zhang MC, Li YG, Zhao JB, Gao YN, Zhou YJ, Liu SL. Dysbiosis of the gut microbiome in lung cancer. Frontiers in Cellular and Infection Microbiology, 2019, 9: 112. DOI:10.3389/fcimb.2019.00112 |
| [12] | Zheng YJ, Fang ZY, Xue Y, Zhang J, Zhu JJ, Gao RY, Yao S, Ye Y, Wang SH, Lin CD, Chen SY, Huang H, Hu L, Jiang GN, Qin HL, Zhang P, Chen JF, Ji HB. Specific gut microbiome signature predicts the early-stage lung cancer. Gut Microbes, 2020, 11(4): 1030-1042. DOI:10.1080/19490976.2020.1737487 |
| [13] | Schadendorf D, Hodi FS, Robert C, Weber JS, Margolin K, Hamid O, Patt D, Chen TT, Berman DM, Wolchok JD. Pooled analysis of long-term survival data from phase Ⅱ and phase Ⅲ trials of ipilimumab in unresectable or metastatic melanoma. Journal of Clinical Oncology, 2015, 33(17): 1889-1894. DOI:10.1200/JCO.2014.56.2736 |
| [14] | Hansen JJ. Immune responses to intestinal microbes in inflammatory bowel diseases. Current Allergy and Asthma Reports, 2015, 15(10): 1-8. |
| [15] | Jin YP, Dong H, Xia LL, Yang Y, Zhu YQ, Shen Y, Zheng HJ, Yao CC, Wang Y, Lu S. The diversity of gut microbiome is associated with favorable responses to anti-programmed death 1 immunotherapy in Chinese patients with NSCLC. Journal of Thoracic Oncology, 2019, 14(8): 1378-1389. DOI:10.1016/j.jtho.2019.04.007 |
| [16] | Mouronte-Roibás C, Leiro-Fernández V, Fernández-Villar A, Botana-Rial M, Ramos-Hernández C, Ruano-Ravina A. COPD, emphysema and the onset of lung cancer. A systematic review.. Cancer Letters, 2016, 382(2): 240-244. DOI:10.1016/j.canlet.2016.09.002 |
| [17] | Tyl M, Domagała-Kulawik J. Lung cancer and copd-growing clinical problem. Polski Merkuriusz Lekarski: Organ Polskiego Towarzystwa Lekarskiego, 2017, 43(253): 5-9. |
| [18] | Houghton AM. Mechanistic links between COPD and lung cancer. Nature Reviews Cancer, 2013, 13(4): 233-245. DOI:10.1038/nrc3477 |
| [19] | Pang T, Leach ST, Katz T, Day AS, Ooi CY. Fecal biomarkers of intestinal health and disease in children. Frontiers in Pediatrics, 2014, 2: 6. |
| [20] | Rangan KJ, Hang HC. Biochemical mechanisms of pathogen restriction by intestinal bacteria. Trends in Biochemical Sciences, 2017, 42(11): 887-898. DOI:10.1016/j.tibs.2017.08.005 |
| [21] | Zhou YJ, Zhao DD, Liu HD, Chen HT, Li JJ, Mu XQ, Liu Z, Li X, Tang L, Zhao ZY, Wu JH, Cai YX, Huang YZ, Wang PG, Jia YY, Liang PQ, Peng X, Chen SY, Yue ZL, Yuan XY, Lu T, Yao BQ, Li YG, Liu GR, Liu SL. Cancer killers in the human gut microbiota: diverse phylogeny and broad spectra. Oncotarget, 2017, 8(30): 49574-49591. DOI:10.18632/oncotarget.17319 |
| [22] | Urbaniak C, Gloor GB, Brackstone M, Scott L, Tangney M, Reid G. The microbiota of breast tissue and its association with breast cancer. Applied and Environmental Microbiology, 2016, 82(16): 5039-5048. DOI:10.1128/AEM.01235-16 |
| [23] | Zhan P, Suo LJ, Qian Q, Shen XK, Qiu LX, Yu LK, Song Y. Chlamydia pneumoniae infection and lung cancer risk: a meta-analysis. European Journal of Cancer: Oxford, England, 2011, 47(5): 742-747. |
| [24] | Zhai K, Ding J, Shi HZ. HPV and lung cancer risk: a meta-analysis. Journal of Clinical Virology: the Official Publication of the Pan American Society for Clinical Virology, 2015(63): 84-89. |
| [25] | Khan S, Imran A, Khan AA, Abul Kalam M, Alshamsan A. Systems biology approaches for the prediction of possible role of chlamydia pneumoniae proteins in the etiology of lung cancer. PLoS ONE, 2016, 11(2): e0148530. DOI:10.1371/journal.pone.0148530 |
| [26] | Larsen JM. The immune response to Prevotella bacteria in chronic inflammatory disease. Immunology, 2017, 151(4): 363-374. DOI:10.1111/imm.12760 |
| [27] | Daniel S G, Ball C L, Besselsen D G, Doetschman T, Hurwitz B L. Functional changes in the gut microbiome contribute to transforming growth factor β-deficient colon cancer. mSystems, 2017, 2(5): e00065-17. |
| [28] | Vivarelli S, Salemi R, Candido S, Falzone L, Santagati M, Stefani S, Torino F, Banna G L, Tonini G, Libra M. Gut microbiota and cancer: from pathogenesis to therapy. Cancers, 2019, 11(1): 38. DOI:10.3390/cancers11010038 |
| [29] | Biagi E, Nylund L, Candela M, Ostan R, Bucci L, Pini E, Nikkïla J, Monti D, Satokari R, Franceschi C, Brigidi P, De Vos W. Through ageing, and beyond: gut microbiota and inflammatory status in seniors and centenarians. PLoS ONE, 2010, 5(5): e10667. DOI:10.1371/journal.pone.0010667 |
| [30] | Bingula R, Filaire M, Radosevic-Robin N, Bey M, Berthon J Y, Bernalier-Donadille A, Vasson M P, Filaire E. Desired turbulence? Gut-lung axis, immunity, and lung cancer. Journal of Oncology, 2017, 2017(5035371). |
| [31] | Cuervo A, Salazar N, Ruas-Madiedo P, Gueimonde M, González S. Fiber from a regular diet is directly associated with fecal short-chain fatty acid concentrations in the elderly. Nutrition Research (New York, NY), 2013, 33(10): 811-816. DOI:10.1016/j.nutres.2013.05.016 |
| [32] | Atarashi K, Tanoue T, Oshima K, Suda W, Nagano Y, Nishikawa H, Fukuda S, Saito T, Narushima S, Hase K, Kim S, Fritz JV, Wilmes P, Ueha S, Matsushima K, Ohno H, Olle B, Sakaguchi S, Taniguchi T, Morita H, Hattori M, Honda K. Treg induction by a rationally selected mixture of Clostridia strains from the human microbiota. Nature, 2013, 500(7461): 232-236. DOI:10.1038/nature12331 |
| [33] | David LA, Maurice CF, Carmody RN, Gootenberg DB, Button JE, Wolfe BE, Ling AV, Devlin AS, Varma Y, Fischbach MA, Biddinger SB, Dutton RJ, Turnbaugh PJ. Diet rapidly and reproducibly alters the human gut microbiome. Nature, 2014, 505(7484): 559-563. DOI:10.1038/nature12820 |
| [34] | Lippitz BE. Cytokine patterns in patients with cancer: a systematic review. The Lancet Oncology, 2013, 14(6): 218-228. DOI:10.1016/S1470-2045(12)70582-X |
| [35] | Nishida A, Inoue R, Inatomi O, Bamba S, Naito Y, Andoh A. Gut microbiota in the pathogenesis of inflammatory bowel disease. Clinical Journal of Gastroenterology, 2018, 11(1): 1-10. DOI:10.1007/s12328-017-0813-5 |
| [36] | Jangi S, Gandhi R, Cox LM, Li N, von Glehn F, Yan R, Patel B, Mazzola MA, Liu SR, Glanz BL, Cook S, Tankou S, Stuart F, Melo K, Nejad P, Smith K, Topçuolu BD, Holden J, Kivisäkk P, Chitnis T, de Jager PL, Quintana FJ, Gerber GK, Bry L, Weiner HL. Alterations of the human gut microbiome in multiple sclerosis. Nature Communications, 2016, 7: 12015. DOI:10.1038/ncomms12015 |
| [37] | Kostic A D, Chun E, Robertson L, Glickman J N, Gallini C A, Michaud M, Clancy T E, Chung D C, Lochhead P, Hold G L, El-Omar E M, Brenner D, Fuchs C S, Meyerson M, Garrett W S. Fusobacterium nucleatum potentiates intestinal tumorigenesis and modulates the tumor-immune microenvironment. Cell Host & Microbe, 2013, 14(2): 207-215. |
| [38] | Blander J M, Longman R S, Iliev I D, Sonnenberg G F, Artis D. Regulation of inflammation by microbiota interactions with the host. Nature Immunology, 2017, 18(8): 851-860. DOI:10.1038/ni.3780 |
| [39] | Zitvogel L, Ayyoub M, Routy B, Kroemer G. Microbiome and anticancer immunosurveillance. Cell, 2016, 165(2): 276-287. DOI:10.1016/j.cell.2016.03.001 |
| [40] | Gopalakrishnan V, Spencer CN, Nezi L, Reuben A, Andrews MC, Karpinets TV, Prieto PA, Vicente D, Hoffman K, Wei SC, Cogdill AP, Zhao L, Hudgens CW, Hutchinson DS, Manzo T, Petaccia de Macedo M, Cotechini T, Kumar T, Chen WS, Reddy SM, Szczepaniak Sloane R, Galloway-Pena J, Jiang H, Chen PL, Shpall EJ, Rezvani K, Alousi AM, Chemaly RF, Shelburne S, Vence LM, Okhuysen PC, Jensen VB, Swennes AG, McAllister F, Marcelo Riquelme Sanchez E, Zhang Y, Le Chatelier E, Zitvogel L, Pons N, Austin-Breneman JL, Haydu LE, Burton EM, Gardner JM, Sirmans E, Hu J, Lazar AJ, Tsujikawa T, Diab A, Tawbi H, Glitza IC, Hwu WJ, Patel SP, Woodman SE, Amaria RN, Davies MA, Gershenwald JE, Hwu P, Lee JE, Zhang J, Coussens LM, Cooper ZA, Futreal PA, Daniel CR, Ajami NJ, Petrosino JF, Tetzlaff MT, Sharma P, Allison JP, Jenq RR, Wargo JA. Gut microbiome modulates response to anti-PD-1 immunotherapy in melanoma patients. Science, 2018, 359(6371): 97-103. DOI:10.1126/science.aan4236 |
| [41] | Gong J, Chehrazi-Raffle A, Placencio-Hickok V, Guan M, Hendifar A, Salgia R. The gut microbiome and response to immune checkpoint inhibitors: preclinical and clinical strategies. Clinical and Translational Medicine, 2019, 8(1): 9. |
 2021, Vol. 61
2021, Vol. 61




