中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 贾云, 钮成拓, 郑飞云, 刘春凤, 王金晶, 李崎. 2021
- Yun Jia, Chengtuo Niu, Feiyun Zheng, Chunfeng Liu, Jinjing Wang, Qi Li. 2021
- 传统豆瓣酱微生物群落发酵演替规律及其功能分析
- Succession and function analysis of microbial community during traditional broad bean paste fermentation
- 微生物学报, 61(9): 2749-2764
- Acta Microbiologica Sinica, 61(9): 2749-2764
-
文章历史
- 收稿日期:2020-11-05
- 修回日期:2021-01-15
- 网络出版日期:2021-05-21
2. 江南大学生物工程学院, 酿酒科学与工程研究室, 江苏 无锡 214122
2. Laboratory of Brewing Science and Technology, School of Biotechnology, Jiangnan University, Wuxi 214122, Jiangsu Province, China
豆类发酵食品是一种具有上千年历史的传统发酵调味品,常见的豆类发酵食品有豆瓣酱、黄豆酱、酱油、纳豆、腐乳等。其中豆瓣酱是以蚕豆和面粉为主要原料,经米曲霉(Aspergillus oryzae)和各种微生物发酵而成。传统的豆瓣酱生产工艺一般采用多菌种混合固态发酵开放式工艺,主要包括制曲和酱醅发酵两个步骤。首先是成曲的制作,蚕豆经蒸煮预处理,然后接种A. oryzae发酵2–3 d。第二步中,将成曲与盐水在陶瓷罐中混合后再次发酵,得到的酱醅在阳光下曝晒3–6个月。
由于传统豆瓣酱的生产属于开放式的固态发酵过程,因此传统豆瓣酱的生产涉及多种微生物和代谢产物的相互作用。其发酵本质是蚕豆和面粉中的蛋白质、淀粉、脂肪等成分在真菌和细菌的作用下降解为低分子化合物,并被进一步合成为各种风味化合物,最终形成了豆瓣酱独特的风味和功能。通常,微生物之间复杂的相互作用以及微生物与代谢产物之间的相关性共同决定了发酵食品的最终味道、风味、质地、保质期和功能特性[1]。但是,由于较长的生产周期和粗放的生产模式,在传统食品工厂中可能存在发酵批次不稳定的问题,甚至有存在某些潜在有害微生物的安全隐患[2]。应用确定的起始发酵剂是标准化发酵和确保质量稳定的最有效方法之一。因此,研究风味形成的关键功能微生物、开发新的发酵菌剂具有重要意义,这将有利于指导传统豆瓣酱发酵产业的转型。
近年来,高通量测序以及多组学技术已经广泛应用于多个微生物相关的研究领域中。已有大量的研究工作基于纯培养、变性梯度凝胶电泳(PCR-DGGE)和高通量测序等手段,初步解析了豆类发酵产品中存在的微生物群落结构[3-8]。如Yan等采用扩增子和纯培养的方法研究了成曲制作过程中的微生物组成,并确定了主要的细菌和真菌微生物为Weissella、Staphylococcus、Aspergillus和Candida[3]。马岩石等采用高通量测序技术对东北市售豆酱中的微生物群落结构进行解析,发现细菌主要为Staphylococcus、Enterobacter、Leuconostoc、Bacillus、Chromohalobacter和Lactobacillus,真菌主要为Zygosaccharomyces、Aspergillus、Gibberella、Mucor和Penicillium[9]。然而,目前对豆酱发酵过程中复杂微生物群落的功能还缺乏系统的研究。近年来,相关性的统计学方法为研究微生物群落的功能提供了机会,为弥合发酵生态系统中表型和基因型之间的差距提供了一个方法[10-11]。如徐岩等采用微生物群落与挥发性化合物轮廓关联分析确定了白酒发酵过程中的风味代谢功能微生物[12]。本研究的目的在于通过解析中国传统豆瓣酱发酵过程中的微生物群落演替和理化代谢物质变化规律,明确豆瓣酱发酵过程中影响豆瓣酱风味的核心功能微生物,并对核心物种进行体外分离和功能验证。
1 材料和方法 1.1 样本采集本研究中使用的豆瓣酱均由胡玉美酿造食品有限责任公司生产。为了获得具有代表性的样品,根据翻醅时间来确定取样时间,新下的酱醅要连续7 d每天同时间倒缸1次,之后每隔7 d进行倒缸1次。所以在每次翻醅后,收集发酵第0、1、2、3、4、5、6、13、20、27、34、41、48和55天同一批次发酵缸的上层、中层和底层各一份酱醅混匀,并设置3个生物学重复。样品收集于无菌瓶中,于–80 ℃保存,然后进行进一步分析。
1.2 理化指标和风味物质分析5 g样本与45 mL蒸馏水混合,100 r/min、30 ℃均质1 h,然后4 ℃、5000× g离心10 min。随后,收集上清液并过滤,测定可滴定酸度、氨基酸态氮含量、还原糖含量、蛋白酶活和葡萄糖淀粉酶活性。用酸碱滴定法和甲醛滴定法分别测定可滴定酸度(以乳酸计)和氨基酸态氮含量[13]。采用福林酚法测定蛋白酶活性(以酪氨酸计)[14],采用3, 5-二硝基水杨酸(DNS)法测定还原糖含量和葡萄糖淀粉酶活性(以葡萄糖计)[15]。葡萄糖淀粉酶活性的一个单位(U)被定义为在40 ℃的条件下,1 min内从淀粉中释放1 mg葡萄糖的酶的数量。通过高效液相色谱法测定了7种有机酸(草酸、酒石酸、丙酮酸、乳酸、乙酸、柠檬酸、琥珀酸)和17种游离氨基酸(Glu、Arg、Lys、Leu、Val、Phe、Asp、Tyr、Ile、Ala、Thr、His、Pro、Gly、Met、Ser、Cys-s)[16]。
采用气相色谱-质谱联用顶空固相微萃取技术(HS-SPME/GC-MS)测定酱醅中的挥发性风味化合物。准确称取2 g样品,添加2 g NaCl,并加入62.5 μg/kg的2-辛醇作为内标,密封在专用瓶中。分析前,样品在55 ℃水浴中预热30 min,SPME纤维头萃取挥发性成分40 min,萃取结束后在气相色谱仪的进样口解吸5 min (Trace GC-1310-ISQ LT,美国),挥发性化合物的分离是在DB-WAX毛细管柱上进行的(30 m×0.25 mm×0.25 mm)。程序升温条件:初始温度40 ℃保持2 min,然后以5 ℃/min升到230 ℃,最后在230 ℃保持8 min。以氦气为载气,流速为1.2 mL/min。离子源温度为260 ℃,采用电子轰击(EI)模式,电离电压为70 eV。离子扫描范围25–500 amu,扫描速率0.2 scan/s。根据Wiley和NIST数据库进行化合物鉴定,并根据特定化合物的峰面积与内标物2-辛醇峰面积的比值计算化合物的含量。
1.3 DNA提取及扩增子测序样品在液氮中研磨成粉末,然后使用Powersoil DNA提取试剂盒提取DNA。用Nanodrop 2000和琼脂糖凝胶电泳检测提取DNA的纯度和完整性。在进一步分析之前,DNA被保存在–80 ℃。分别用带有特异序列的引物338-F/806-R和ITS1-f/ITS2-R扩增细菌16S rRNA基因的V3–V4区和真菌的ITS1区,然后通过Illumina HiSeq 2500平台对文库进行测序。使用QIIME将有效序列按97%的相似度聚成操作分类单元(OTUs)。利用Greengenes细菌数据库和UNITE真菌数据库对具有代表性的OTU序列进行注释。Alpha多样性指数和beta多样性在QIIME中计算[17]。
1.4 微生物的分离和功能验证为了分离微生物,将10 g酱醅与90 mL无菌生理盐水(0.85%)混合,于200 r/min 30 ℃下孵育1 h,然后在无菌生理盐水中梯度稀释,并将100 μL稀释液涂布于LB、MRS、YAP和PDA平板上,分别在好氧和厌氧条件下于30 ℃或37 ℃培养3 d。最后,根据先前研究中描述的方法对分离到的单菌落进行菌种鉴定[18]。
采用体外模拟发酵法验证微生物(A. oryzae、Z. rouxii、S. gallinarum、B. subtilis和W. confusa)的代谢特性。将10 g蚕豆粉、2.5 g小麦粉、50 g豆酱加入1 L去离子水中煮30 min,过滤后制成模拟液体培养基。然后将上述5株菌接种到液体培养基中,模拟豆瓣酱的发酵过程,初始细胞浓度调整为1×106 CFU/g。所有发酵均在30 ℃下进行3 d。以未接种的发酵培养基为对照,对菌株发酵液的理化风味物质进行检测。所有实验均一式三份。
1.5 数据处理与分析用R语言软件包ade4和ggplot2进行主成分分析(principal co-ordinates analysis,PCoA)。用SIMCA-14.1软件进行偏最小二乘判别分析(partial least squares discriminant analysis,PLS-DA)建模,生成变量重要性投影(variable importance in the projection,VIP),基于VIP > 0.7选择判别性差异代谢物。为了确定最有可能解释不同发酵时期之间差异的细菌和真菌分类群,使用LEfSe (LDA Effect Size)差异分析来鉴定多个组之间的差异物种,并将LDA的筛选值设置为4[19]。使用R语言软件包psych和corr.test函数计算斯皮尔曼(Spearman)两两相关性并分析相关性的显著性,通过Cytoscape软件将显著性P值< 0.05且相关系数|r| > 0.6的高度相关性可视化。使用Excel 2017软件和GraphPad Prism 8.0软件进行进一步的统计分析和图形处理。
2 结果和分析 2.1 风味理化物质的测定对发酵过程酱醅样品的可滴定酸、氨基酸态氮、还原糖和酶活等理化指标进行了跟踪检测。发现可滴定酸和氨基酸态氮含量在发酵过程中呈先增加后趋于平缓的趋势(图 1-A)。可滴定酸度从3.1%增加到5.7% (W/W),氨基酸氮的含量在第41天达到最高水平2.0% (W/W)。当成曲与高浓度盐水混合时[约20%–22% (W/V) NaCl],蛋白酶活性急剧下降(图 1-C)。氨基酸态氮含量是判定发酵程度和豆类发酵产品质量好坏的重要指标,而蛋白酶活性的高低直接影响氨基酸态氮的形成[20]。此外还原糖含量在发酵前2 d从14.0%迅速增至24.7% (W/W),这应当是由于淀粉被Aspergillus分泌的葡萄糖淀粉酶分解,且还原糖的生成速率远远大于消耗速率。之后由于葡糖淀粉酶活性的降低、微生物对还原糖的利用以及美拉德反应的发生,使得还原糖含量在发酵中基本维持稳定。我们还检测了豆瓣酱发酵过程中的风味代谢产物,包括氨基酸、有机酸和挥发性化合物。如图 1-D所示,氨基酸含量在发酵中期达到最大,然后在发酵后期有所减少。其中谷氨酸是含量最高的氨基酸,占总含量的16.8%,对于豆类产品的鲜味至关重要[21]。图 1-E显示乙酸和乳酸是主要的有机酸,占总含量的90%以上,其不仅可以通过调节酸度来平衡豆瓣酱的味道,还可以作为前体物质参与芳香酯类的合成[22]。通过HS-SPME/GC-MS检测分析发现,豆瓣酱发酵过程中共检测出107种挥发性化合物,主要包括19种醇类、41种酯类、12种醛类、12种酮类、6种酚类、2种酸类和15种低含量其他挥发性化合物。如图 1-F所示,总挥发性化合物含量在发酵过程中逐渐积累。醇类和酯类是豆瓣酱的主体风味,其中醇类化合物占挥发性化合物总含量的50%以上。
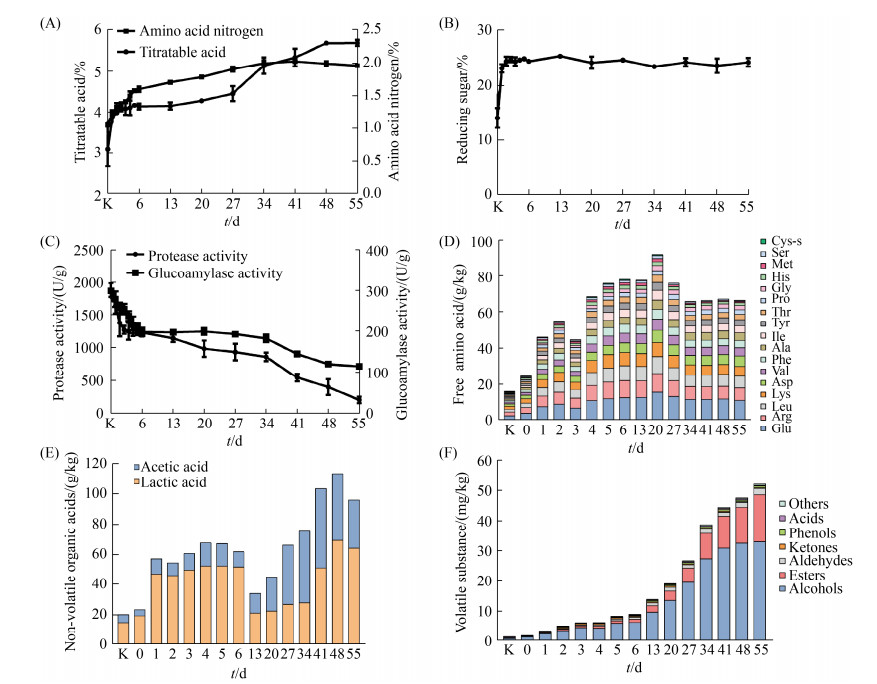
|
| 图 1 传统豆瓣酱发酵过程中风味理化的动态变化 Figure 1 Temporal changes of physicochemical parameters and flavor metabolites during the broad bean paste fermentation. A: titratable acid and amino acid nitrogen content; B: reducing sugar content; C: protease activity and glucoamylase activity; D: free amino acids; E: non-volatile organic acids; F: volatile flavor compounds; K: koji. |
挥发性化合物的热图表明,大多数挥发性化合物的含量在发酵过程中呈上升趋势,发酵中后期是风味物质形成和积累的重要时期(图 2-A)。通过PLS-DA模型(VIP > 0.7)筛选出35种差异代谢物,主要包括10种醇类、11种酯类、7种醛类、1种酮类、2种酸类、2种酚类和2种其他化合物(图 2-B)。如表1所示,醇类和酯类化合物在发酵后期显著积累。众所周知,酯类广泛存在于水果和葡萄酒中,可以增加果香和花香以及香气复杂性。酱醅中酯类物质的存在对豆瓣酱的香气有积极作用[23],如异戊酸乙酯、乙酸乙酯、棕榈酸乙酯、亚油酸乙酯等。醇类会显著影响酱醅的香气和口感,同时它们也可以作为前体物质参与酯类的合成[22],如乙醇、3-甲基-1-丁醇、苯乙醇等。特别是苯乙醇,由于其和醇厚的玫瑰香气在酱油和豆酱中起到增强香气的作用,而且由于其抗菌、防腐的特性被视为天然防腐剂[24]。此外,检测到的醛类主要有3-甲基丁醛、苯甲醛、2-甲基丁醛、苯乙醛等,这些化合物具有类似坚果的气味,并且已知具有传统大酱的独特风味[25]。酚类物质如2-甲氧基-4-乙烯基苯酚和丁香酚,它们具有烟熏、木头和烘焙的气味。豆瓣酱中挥发酸的种类很少,仅检测到异戊酸和乙酸,它们可以赋予豆瓣酱一定的酸味。在35种差异代谢物中,发现3-辛酮、1-辛烯-3-醇和2-辛烯-1-醇的含量在发酵过程中呈先升高后降低的趋势,其分别具有霉味、泥土和花香味。

|
| 图 2 传统发酵过程中挥发性风味物质的变化 Figure 2 Temporal changes of volatile flavor compounds during the broad bean paste fermentation. A: heatmap showing the succession of volatile flavor compounds. The peak area of each volatile flavor compound was normalized using Z-score. The color intensity is proportional to the relative abundance of volatile compounds. B: PLS-DA VIP plot for volatile compounds. K represents koji. |
2.2 微生物群落结构及演替
通过扩增子测序对发酵过程中的细菌和真菌群落进行了分析。首先对样本的测序结果进行了统计分析,具体的测序信息见表 2。每个样品的覆盖率(goods coverage)均在99%以上,表明足够的测序深度和可靠的数据质量。从多样性指数(Shannon、Chao1、Observed OTUs)可以发现由于环境微生物的引入,细菌群落的微生物多样性和丰富度在第1天达到最大,之后由于各种环境压力胁迫导致多样性指数开始降低。而在真菌群落中,微生物多样性和丰富度在发酵中期有一定的波动,但相较于细菌群落整体相对比较稳定。
| Sample ID | 16S | ITS | |||||||
| Shannon | Chao1 | Observed OTUs | Goods coverage/% | Shannon | Chao1 | Observed OTUs | Goods coverage/% | ||
| koji | 2.96 | 180.63 | 157 | 100 | 0.11 | 20.40 | 18 | 100 | |
| 0 | 3.34 | 233.45 | 199 | 100 | 0.21 | 55.90 | 40 | 100 | |
| 1 | 3.34 | 264.71 | 218 | 100 | 0.16 | 58.23 | 40 | 100 | |
| 2 | 3.27 | 262.50 | 213 | 100 | 0.15 | 38.42 | 32 | 100 | |
| 3 | 2.51 | 244.11 | 200 | 100 | 0.10 | 28.33 | 24 | 100 | |
| 4 | 2.45 | 221.91 | 186 | 100 | 0.12 | 48.33 | 34 | 100 | |
| 5 | 2.43 | 248.67 | 204 | 100 | 0.22 | 70.48 | 48 | 100 | |
| 6 | 2.22 | 253.33 | 194 | 100 | 0.16 | 68.83 | 47 | 100 | |
| 13 | 1.92 | 218.54 | 175 | 100 | 0.36 | 67.16 | 59 | 100 | |
| 20 | 2.04 | 224.58 | 184 | 100 | 0.22 | 51.33 | 44 | 100 | |
| 27 | 1.69 | 210.27 | 167 | 100 | 0.13 | 35.07 | 28 | 100 | |
| 34 | 1.88 | 245.50 | 181 | 100 | 0.38 | 66.15 | 54 | 100 | |
| 41 | 1.99 | 246.20 | 191 | 100 | 0.21 | 58.95 | 43 | 100 | |
| 48 | 1.82 | 234.98 | 184 | 100 | 0.17 | 47.87 | 37 | 100 | |
| 55 | 1.77 | 231.02 | 177 | 100 | 0.12 | 34.10 | 28 | 100 | |
从整体上对样本的微生物组成进行了PCoA分析,第一个主坐标轴(PCoA1)对细菌和真菌群落总变化的解释度分别为65.56%和48.77%。随着发酵的进行,样本在PCoA1上随时间逐渐移动,且相邻时间点的间隔距离越来越小,到发酵后期样本聚集在一起,表明微生物群落结构逐渐趋于稳定(图 3-A和图 3-B)。通过聚类,可以将发酵过程分为3个阶段,分别是:成曲阶段(koji)、发酵前期(0–5 d) 和发酵中后期(6–55 d) (图 3-C)。
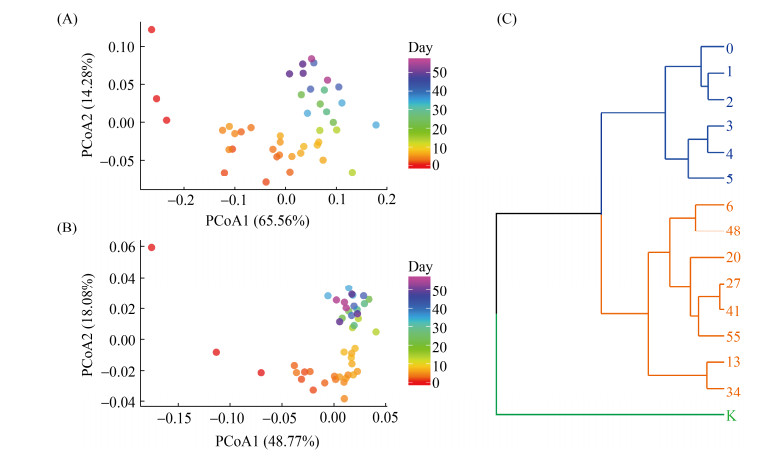
|
| 图 3 豆瓣酱发酵过程中微生物组成的主成分和聚类分析 Figure 3 Principal component and clustering analysis of microbial diversity during Chinese broad bean paste fermentation. Principal component analysis of bacterial (A) and fungal (B) community. C: clustering analysis of microbial community. K represents koji. |
高丰度优势微生物通常被认为是食品发酵的重要组成部分。通过对样本的统计分析,发现只有5个细菌属和2个真菌属的平均丰度大于1%且存在于50%的样本中,它们被定义为优势微生物。如图 4-A柱状图所示,细菌群落中的优势属主要是Staphylococcus (79.8%)、Bacillus (7.5%)、Weisiella (5.9%)、unidentified Bacillales (3.0%)和unidentified Bacilli (1.7%),其中Staphylococcus在整个发酵过程中呈上升趋势并占主导地位,而Bacillus、Weissella在发酵前期呈逐渐消亡状态,在发酵中后期细菌群落结构达到稳定状态。此外,真菌的群落结构较为简单且稳定,其中Aspergillus的平均丰度占真菌总群落的97%以上,Zygosaccharomyces呈先上升后下降的趋势(图 4-B)。
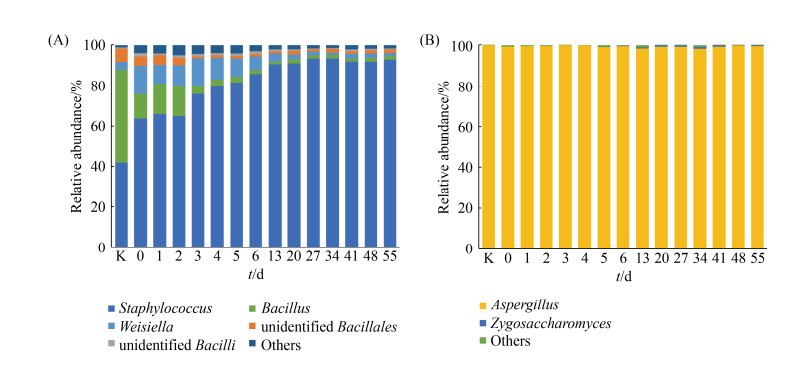
|
| 图 4 传统豆瓣酱发酵过程中微生物群落结构演替 Figure 4 Successions and distributions of bacterial (A) and fungal (B) community during the traditional fermentation at the genus level. Only those genera above 1% at total relative abundance are shown. K: Koji. |
随后,基于物种丰度通过LEfSe分析来确定不同发酵阶段(成曲阶段、发酵前期和发酵中后期)的差异物种。如表 3所示,成曲阶段的差异物种主要是Aspergillus和Bacillus,发酵前期的差异物种主要是Weissella和Pichia,而发酵中后期主要是Staphylococcus和Zygosaccharomyces。
| Fermentation stage | Kingdom | Class | Family | Genus |
| Koji stage (koji) | Bacteria | Bacilli | Bacillaceae | Bacillus |
| Fungi | Eurotiomycetes | Trichocomaceae | Aspergillus | |
| Itial stage (0–5 d) | Bacteria | Bacilli | Leuconostocaceae | Weissella |
| Fungi | Saccharomycetes | Pichiaceae | Pichia | |
| Mid-late stage (6–55 d) | Bacteria | Bacilli | Staphylococcaceae | Staphylococcus |
| Fungi | Saccharomycetes | Saccharomycetaceae | Zygosaccharomyces |
2.3 微生物的功能预测
接下来我们通过对优势属相对丰度与理化代谢物之间的相关性分析研究了微生物群落的功能潜力,发现Aspergillus、Bacillus、Weissella和unidentified Bacillales、unidentified Bacilli主要与酶活性呈正相关,而Staphylococcus和Zygosaccharomyces与可滴定酸、氨基酸态氮、有机酸、游离氨基酸和挥发性风味物质呈正相关性(图 5)。
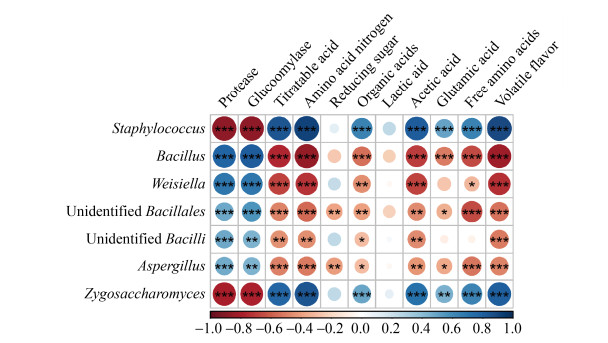
|
| 图 5 发酵过程中微生物属和理化代谢之间的相关性分析 Figure 5 Spearman correlation between dominant genera and physicochemical metabolites during the fermentation process. The correlation coefficient is represented by the color and size of the circles. Dark blue indicates positive correlation and dark red indicates negative correlation. P values were calculated using Spearman's rank correlation test, *: P < 0.05; **: P < 0.01; ***: P < 0.001. |
在网络相关性分析中,我们计算了68个微生物属和107种风味化合物之间的Spearman相关系数,并选择了系数(|r|) > 0.6和显著性(P) < 0.05作为网络的强相关节点。结果表明优势属Staphylococcus和Zygosaccharomyces分别与64种和56种风味物质显著正相关(图 6)。综上我们可以初步得出结论,在成曲阶段和发酵前期主要是Aspergillus、Bacillus和Weissella分泌各种酶来降解大分子物质,而在发酵中后期主要是耐盐的Staphylococcus和Zygosaccharomyces对风味物质的形成起主要作用。然而,这些功能微生物在群落环境中的代谢特性还需要进一步的实验验证。
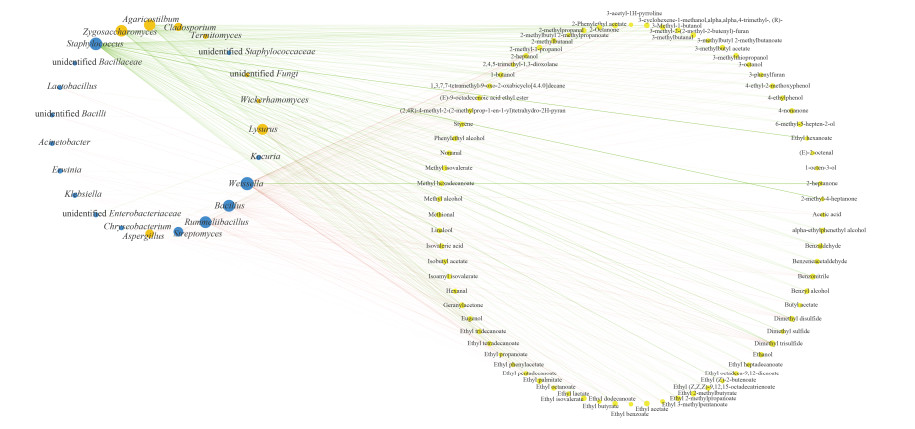
|
| 图 6 发酵过程中微生物属和挥发性风味物质之间的相关性分析 Figure 6 Correlation network between microbial genera and volatile flavor compounds analyzed by Spearman's correlation rank test. The size of the nodes indicates the degree of connections. Blue, orange, and yellow nodes indicate bacteria, fungi, and volatile flavor compounds, respectively. Edge thickness represents the proportional to the value of Spearman's correlation. |
2.4 微生物的分离和功能验证
统计方法为缩小豆瓣酱发酵生态系统中表型和基因型之间的差距提供了一个视角,并指出了以前未知的相互作用。但是,相关性分析并不意味着直接的因果关系,应进一步通过体外模拟发酵来验证核心微生物群的功能特性。因此,我们从酱醅中分离出核心微生物,并获得了各微生物属中相对丰度最高的代表物种(A. oryzae、B. subtilis、S. gallinarum、W. confusa和Z. rouxii),结果见表 4。由于微生物属的物种多样性和微生物培养技术的局限性,这5个物种可以被用来代表核心微生物属[26-27]。
| Dominant genera | Dominant OTUs | Relative abundance of dominant OTU in related genus/% | Isolates | Similarity/% |
| Staphylococcus | OTU432 | 84.2 | S. gallinarum | 100 |
| Bacillus | OTU401 | 67.0 | B. subtilis | 100 |
| Weissella | OTU1 | 81.9 | W. confusa | 100 |
| Aspergillus | OTU140 | 99.1 | A. oryzae | 100 |
| Zygosaccharomyces | OTU1 | 71.7 | Z. rouxii | 100 |
| a: sequence similarity between the isolate and dominant OTU in each corresponding genus was compared by NCBI Blast. | ||||
随后,通过体外模拟发酵验证了核心物种的代谢特性(图 7),发现A. oryzae是分泌蛋白酶和葡萄糖淀粉酶的主要微生物。此外,虽然B. subtilis会抑制A. oryzae的生长,但其本身也可以分泌一定的蛋白酶和葡萄糖淀粉酶,有可能在发酵前期对大分子物质起到一定的降解作用。我们还发现,A. oryzae和S. gallinarum都能产生乙酸,而W. confusa不仅能产生乙酸,而且能产生高含量的乳酸。这表明,在豆瓣酱发酵过程中,W. confusa、A. oryzae和S. gallinarum具有较高的产酸能力,这些有机酸将导致发酵过程中pH值的降低。W. confusa、A. oryzae和S. gallinarum也表现出较高的氨基酸合成能力或蛋白质降解能力。此外,在接种Z. rouxii的培养基中,酯类和醇类化合物的含量显著增加。总的来说,培养组学提供了一个将原位功能预测与体外功能验证联系起来的机会,并且从体外试验获得的部分微生物功能结果与基于原位模式相关性的预测结果是一致的。例如,A. oryzae和B. subtilis能分泌高蛋白酶活和葡萄糖淀粉酶活,这有助于酱醅中大分子的降解。Z. rouxii是大部分挥发性风味物质形成的直接原因,可能直接导致豆瓣酱的独特风味。然而,我们还观察到一些明显的差异,这些差异可能是由于微生物代谢活性的滞后效应或多菌株的协同作用,导致微生物演替和理化代谢动力学之间的不一致。

|
| 图 7 核心物种的功能特性 Figure 7 Functions of core species in liquid simulation fermentation. A: enzyme activity; B: non-volatile organic acid; C: free amino acid; D: volatile substance. The capital letters represent abbreviations for dominant species. A: A. oryzae; B: B. subtilis; S: S. gallinarum; W: W. confusa; Z: Z. rouxii; CK: control. |
3 讨论
优势微生物可以被定义为比其他物种具有更高相对丰度的广泛分布的物种,它们可能在生物过程中起着重要的作用[26]。扩增子测序分析揭示了在传统豆瓣酱发酵过程中有7个广泛分布的属(Staphylococcus、Bacillus、Weissella、unidentified Bacillales、unidentified Bacilli、Aspergillus和Zygosaccharomyces)为优势菌群成员。此外,由于所使用的样品和方法不同,与其他研究人员报道的结果也有所差异。例如Tanaka等通过PCR-DGGE分析了酱油生产过程中涉及的微生物群落,并在成曲中检测到了Weissella、Staphylococcus、Aspergillus和几种酵母,而Tetragenococcus和Zygosaccharomyces出现在早期发酵阶段,Candida在发酵中期和成熟阶段被检测到[4]。Kim等比较了中日两国发酵豆酱中的微生物组成,结果表明在日本样品中以Tetragenococcus和Staphylococcus占主导,而Bacillus是中国样品中的主要细菌,Aspergillus和Zygosaccharomyces共同存在于中国和日本发酵豆酱中[5]。尽管不同地区的样品或基于不同豆类的发酵食品之间的微生物群落结构存在差异,但优势微生物属通常是相同的,且具有普遍性,主要包括Staphylococcus、Bacillus、Weissella、Aspergillus和Zygosaccharomyces[4-6]。此外,Staphylococcus也被确定为其他以蛋白质为主要成分的发酵食品(如奶酪)的主要微生物属[26]。
在研究中我们还探索了中国传统豆瓣酱发酵过程中不同发酵阶段的差异物种和微生物的功能,结果表明这7个优势属可能在不同发酵阶段发挥着不同的关键作用。Aspergillus、Weissella和Bacillus主要在成曲阶段和发酵前期分泌各种酶来降解大分子物质,芽孢杆菌纲的部分未知属(unidentified Bacillales和unidentified Bacilli)也可能与蛋白酶和葡萄糖淀粉酶活的分泌相关,但受测序技术的局限未能得到有效鉴定,仍需依赖传统培养分离技术对其进行进一步研究。此外,耐盐的Staphylococcus和Zygosaccharomyces主要在发酵中后期对风味物质的形成起主要作用,这与先前庞惟俏的研究结果是一致的[28]。然而,微生物代谢的滞后效应或多菌种的协同效应会导致微生物演替与理化代谢动力学不一致,预测结果不能代表微生物的实际功能。因此,我们分离了核心物种并对其代谢特性进行了评估,发现A. oryzae和B. subtilis的主要功能是在成曲和发酵前期释放各种酶来水解蚕豆和小麦中的大分子物质(蛋白质和淀粉),为酱醅发酵阶段细菌和酵母的生长提供前体物质,它们已被广泛用作豆类发酵食品的微生物接种剂[29]。此外,A. oryzae、S. gallinarum和W. confusa具有较高的有机酸和氨基酸产生能力,这导致了酱醅的酸化,并为豆瓣酱提供了独特的风味。然后成曲与高浓度盐水混合,微生物群落开始从成曲阶段转变为酱醅发酵阶段。在这个阶段,由于盐水的加入导致酱醅发酵过程中不耐受微生物菌群(Aspergillus、Bacillus、Weissella)的丰度和该类群微生物所产酶活性显著降低。在中后期,细菌群落相对稳定,Z. rouxii开始发挥作用,生成了许多挥发性风味成分,从而赋予豆瓣酱特有的风味。但是由于发酵酱醅中盐含量很高,对Z. rouxii生长活性有一定的抑制作用,从而导致Z. rouxii的丰度和代谢活性也较低。因此,我们推断通过在发酵阶段强化Z. rouxii可以提高豆瓣酱风味品质并缩短发酵时间[20]。Staphylococcus是丰度最高的优势菌,并且在整个发酵过程中逐渐增加,加入盐水不会抑制酱醅中Staphylococcus的生长,可以推断出Staphylococcus具有很强的耐受渗透压的能力。正如先前的研究,Staphylococcus中的几个物种也被报道是参与其他高渗发酵食品的优势菌之一[3]。由于其对碳水化合物的发酵、蛋白质的水解、脂肪的降解和氨基酸的转化,凝乳酶阴性的Staphylococcus被认为对于发酵食品的风味形成和感官品质至关重要[30]。在本研究中,Staphylococcus的代表种S. gallinarum虽然具有一定的有机酸和氨基酸产生能力,但其属于条件致病菌,不能用于食品生产。因此,后续应进一步研究S. gallinarum在酱醅发酵中广泛分布的机制,从而采取合适的措施(如添加抑菌微生物、天然防腐剂等)抑制其生长。
发酵过程中的微生物控制是影响最终豆类发酵产品质量和稳定性的关键。由于开放的发酵环境和较长的发酵周期,豆瓣酱发酵所涉及的微生物群落表现出明显的多样性和动态性。这项研究对豆瓣酱发酵过程中微生物群落的演替和核心功能微生物提供了更广泛的了解,有利于指导豆瓣酱工业生产的优化和提高,确保豆瓣酱的质量和安全性。然而,本研究只关注了豆瓣酱发酵过程中不同优势微生物属对发酵功能的积极影响,酱醅发酵过程中还存在大量的低丰度物种,这些物种在环境发生变化时还可能具有一定的生态系统恢复力和生态系统功能[27-28]。因此,未来应进一步对豆瓣酱中低丰度的微生物进行分离和功能的验证。
| [1] | Singh D, Lee S, Lee CH. Metabolomics for empirical delineation of the traditional Korean fermented foods and beverages. Trends in Food Science & Technology, 2017, 61: 103-115. |
| [2] | Park HK, Shukla S, Lee JS, Kim JK, Kim M. Reduction of foodborne pathogens and aflatoxins in doenjang samples using defined meju. Journal of Food Safety, 2014, 34(2): 161-167. DOI:10.1111/jfs.12109 |
| [3] | Yan YZ, Qian YL, Ji FD, Chen JY, Han BZ. Microbial composition during Chinese soy sauce koji-making based on culture dependent and independent methods. Food Microbiology, 2013, 34(1): 189-195. DOI:10.1016/j.fm.2012.12.009 |
| [4] | Tanaka Y, Watanabe J, Mogi Y. Monitoring of the microbial communities involved in the soy sauce manufacturing process by PCR-denaturing gradient gel electrophoresis. Food Microbiology, 2012, 31(1): 100-106. DOI:10.1016/j.fm.2012.02.005 |
| [5] | Kim TW, Lee JH, Park MH, Kim HY. Analysis of bacterial and fungal communities in Japanese- and Chinese-fermented soybean pastes using nested PCR-DGGE. Current Microbiology, 2010, 60(5): 315-320. DOI:10.1007/s00284-009-9542-4 |
| [6] | Nam YD, Lee SY, Lim SI. Microbial community analysis of Korean soybean pastes by next-generation sequencing. International Journal of Food Microbiology, 2012, 155(1/2): 36-42. |
| [7] |
Liu CF, Liu JX, Jiang LS, Cheng HS, Li Q. Analysis on microbial community in mature soybean sauce mash during traditional fermentation. Science and Technology of Food Industry, 2012, 33(13): 122-126.
(in Chinese) 刘春凤, 刘金霞, 蒋立胜, 程华曙, 李崎. 传统发酵成熟期豆瓣酱醅中的微生物群落分析. 食品工业科技, 2012, 33(13): 122-126. |
| [8] |
Wu YT, Du MY, He HH, Kan JQ, Cheng FF, Yin N, Liu WB, Ding CY, Yin XQ, Wu Y. Microbial diversity analysis of natural fermented Chili Sauce from different regions in Xinjiang by high-throughput sequencing. Food and Fermentation Industries, 2019, 45(21): 221-228.
(in Chinese) 武亚婷, 杜木英, 何欢欢, 阚建全, 程方方, 殷娜, 刘维兵, 丁承焱, 尹小庆, 武运. 基于高通量测序技术分析新疆不同地区自然发酵辣椒酱微生物群落多样性. 食品与发酵工业, 2019, 45(21): 221-228. |
| [9] |
Ma YS, Jiang M, Li H, Pei FY. Analysis of microbial diversity of northeast soy sauce based on high-throughput sequencing technology. Science and Technology of Food Industry, 2020, 41(12): 100-105.
(in Chinese) 马岩石, 姜明, 李慧, 裴芳艺. 基于高通量测序技术分析东北豆酱的微生物多样性. 食品工业科技, 2020, 41(12): 100-105. |
| [10] | Vilanova C, Porcar M. Are multi-omics enough?. Nature Microbiology, 2016, 1: 16101. DOI:10.1038/nmicrobiol.2016.101 |
| [11] | Hall EK, Bernhardt ES, Bier RL, Bradford MA, Boot CM, Cotner JB, del Giorgio PA, Evans SE, Graham EB, Jones SE, Lennon JT, Locey KJ, Nemergut D, Osborne BB, Rocca JD, Schimel JP, Waldrop MP, Wallenstein MD. Understanding how microbiomes influence the systems they inhabit. Nature Microbiology, 2018, 3(9): 977-982. DOI:10.1038/s41564-018-0201-z |
| [12] |
Wang P, Wu Q, Xu Y. Core microbiota in Chinese liquor fermentation and associations with environmental factors. Acta Microbiologica Sinica, 2018, 58(1): 142-153.
(in Chinese) 王鹏, 吴群, 徐岩. 中国白酒发酵过程中的核心微生物群及其与环境因子的关系. 微生物学报, 2018, 58(1): 142-153. |
| [13] | Jang YK, Shin GR, Jung ES, Lee S, Lee S, Singh D, Jang ES, Shin DJ, Kim HJ, Shin HW, Moon BS, Lee CH. Process specific differential metabolomes for industrial gochujang types (pepper paste) manufactured using white rice, brown rice, and wheat. Food Chemistry, 2017, 234: 416-424. DOI:10.1016/j.foodchem.2017.04.154 |
| [14] | Gao XL, Zhao HF, Feng YZ, Zhao MM. A comparative study on physicochemical properties of Chinese-type soy sauces prepared using pure koji and mixed kojis. African Journal of Biotechnology, 2010, 9(40): 6740-6747. |
| [15] | Liu ZB, Wang ZY, Lv X, Zhu XP, Chen LL, Ni L. Comparison study of the volatile profiles and microbial communities of Wuyi Qu and Gutian Qu, two major types of traditional fermentation starters of Hong Qu glutinous rice wine. Food Microbiology, 2018, 69: 105-115. DOI:10.1016/j.fm.2017.07.019 |
| [16] | Wu LH, Lu ZM, Zhang XJ, Wang ZM, Yu YJ, Shi JS, Xu ZH. Metagenomics reveals flavour metabolic network of cereal vinegar microbiota. Food Microbiology, 2017, 62: 23-31. DOI:10.1016/j.fm.2016.09.010 |
| [17] | Li P, Lin WF, Liu X, Wang XW, Gan X, Luo LX, Lin WT. Effect of bioaugmented inoculation on microbiota dynamics during solid-state fermentation of Daqu starter using autochthonous of Bacillus, Pediococcus, Wickerhamomyces and Saccharomycopsis. Food Microbiology, 2017, 61: 83-92. DOI:10.1016/j.fm.2016.09.004 |
| [18] | Wang XS, Du H, Zhang Y, Xu Y. Environmental microbiota drives microbial succession and metabolic profiles during Chinese liquor fermentation. Applied and Environmental Microbiology, 2017, 84(4): e02369-17. DOI:10.1128/aem.02369-17 |
| [19] | Segata N, Izard J, Waldron L, Gevers D, Miropolsky L, Garrett WS, Huttenhower C. Metagenomic biomarker discovery and explanation. Genome Biology, 2011, 12(6): R60. DOI:10.1186/gb-2011-12-6-r60 |
| [20] | Zhong YY, Lu X, Xing L, Ho SWA, Kwan HS. Genomic and transcriptomic comparison of Aspergillus oryzae strains: a case study in soy sauce koji fermentation. Journal of Industrial Microbiology & Biotechnology, 2018, 45(9): 839-853. |
| [21] | Zhu Y, Tramper J. Koji-where East meets West in fermentation. Biotechnology Advances, 2013, 31(8): 1448-1457. DOI:10.1016/j.biotechadv.2013.07.001 |
| [22] | Qi W, Guo HL, Wang CL, Hou LH, Cao XH, Liu JF, Lu FP. Comparative study on fermentation performance in the genome shuffledCandida versatilisand wild-type salt tolerant yeast strain. Journal of the Science of Food and Agriculture, 2017, 97(1): 284-290. DOI:10.1002/jsfa.7728 |
| [23] | Huang ZR, Hong JL, Xu JX, Li L, Guo WL, Pan YY, Chen SJ, Bai WD, Rao PF, Ni L, Zhao LN, Liu B, Lv XC. Exploring core functional microbiota responsible for the production of volatile flavour during the traditional brewing of Wuyi Hong Qu glutinous rice wine. Food Microbiology, 2018, 76: 487-496. DOI:10.1016/j.fm.2018.07.014 |
| [24] | Yang Y, Deng Y, Jin YL, Liu YX, Xia BX, Sun Q. Dynamics of microbial community during the extremely long-term fermentation process of a traditional soy sauce. Journal of the Science of Food and Agriculture, 2017, 97(10): 3220-3227. DOI:10.1002/jsfa.8169 |
| [25] | Chang M, Hae CC. Characteristics of bacterial-koji and doenjang(soybean paste) made by using Bacillus subtilis DJI. Korean Journal of Microbiology and Biotechnology, 2007, 35(4): 325-333. |
| [26] | Wolfe BE, Button JE, Santarelli M, Dutton RJ. Cheese rind communities provide tractable systems for in situ and in vitro studies of microbial diversity. Cell, 2014, 158(2): 422-433. DOI:10.1016/j.cell.2014.05.041 |
| [27] | Wang SL, Wu Q, Nie Y, Wu JF, Xu Y. Construction of synthetic microbiota for reproducible flavor compound metabolism in Chinese light-aroma-type liquor produced by solid-state fermentation. Applied and Environmental Microbiology, 2019, 85(10): e03090-e03018. |
| [28] | 庞惟俏. 黑龙江大豆酱中微生物群落与挥发性成分关系的研究. 黑龙江八一农垦大学硕士学位论文, 2018. |
| [29] | Seo HS, Lee S, Singh D, Shin HW, Cho SA, Lee CH. Untargeted metabolite profiling for koji-fermentative bioprocess unravels the effects of varying substrate types and microbial inocula. Food Chemistry, 2018, 266: 161-169. DOI:10.1016/j.foodchem.2018.05.048 |
| [30] | Sánchez Mainar M, Xhaferi R, Samapundo S, Devlieghere F, Leroy F. Opportunities and limitations for the production of safe fermented meats without nitrate and nitrite using an antibacterial Staphylococcus sciuri starter culture. Food Control, 2016, 69: 267-274. DOI:10.1016/j.foodcont.2016.04.056 |
 2021, Vol. 61
2021, Vol. 61




