中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 王帅涛, 牛艳婷, 张恒, 李盼欣, 张宁梅, 成娟丽, 林金水. 2021
- Shuaitao Wang, Yanting Niu, Heng Zhang, Panxin Li, Ningmei Zhang, Juanli Cheng, Jinshui Lin. 2021
- 靶向破坏铜绿假单胞菌生物被膜的定向蛋白投放技术
- An engineered bacterium for the targeted delivery of proteins to destroy Pseudomonas aeruginosa biofilms
- 微生物学报, 61(9): 2726-2748
- Acta Microbiologica Sinica, 61(9): 2726-2748
-
文章历史
- 收稿日期:2020-11-04
- 修回日期:2020-12-30
- 网络出版日期:2021-01-06
2. 陕西省红枣重点实验室(延安大学), 陕西 延安 716000
2. Shaanxi Key Laboratory of Chinese Jujube(Yan'an University), Yan'an 716000, Shaanxi Province, China
合成生物学目的在于利用基因技术和工程学概念改造已有的生物系统或者设计合成新的生物系统,并且这些系统可以执行自然界中不存在的新功能[1]。随着基因重组技术、分子系统生物学的发展,这种新型生物学系统的构建成为可预测和可靠的系统过程。合成生物学可以应用于广泛的领域,如通过适度的遗传途径控制细胞分裂、动物发育和衰老、经济有效地生产药物等[2-4]。只需将具有不同功能的生物部件组合起来,就可构建出许多具有不同特定生物功能的系统,利用这些系统可以生产材料、加工化学物质、生产能源、提供食物,帮助维持或改善人类健康以及生态环境[1]。比如,利用活细菌来治疗癌症的肿瘤细菌疗法虽已超过150年的历史,但其仍然存在着治疗效果和细菌自身毒性的矛盾以及抗肿瘤作用时空控制性弱等问题。而随着合成生物学的兴起,设计合成复杂、多功能的基因线路能够弥补传统细菌疗法的缺陷,使肿瘤细菌疗法重新焕发生机[5]。目前已报道的对肿瘤治疗菌株的基因工程改造主要集中于降低细菌天然毒力、提高细菌安全性、增强细菌靶向性、侵袭性和定殖能力及降低细菌在肿瘤微环境的逃逸率等[5]。例如,我国微生物学家牟希亚教授通过生物工程技术改造选育的具有完全自主知识产权的铜绿假单胞菌甘露糖敏感血凝菌毛株(Pseudomonas aeruginosa mannose-sensitive hemagglutinin,PA-MSHA),已被中国国家食品药品监督管理总局(CFDA)批准用于恶性肿瘤的辅助治疗[6];以改造过的铜绿假单胞菌作为微生物工具,利用其Ⅲ型分泌系统(T3SS)将重组抗原蛋白注射到宿主细胞中,可以在抗肿瘤免疫治疗中发挥作用[7]。然而相比革兰氏阳性菌,对革兰氏阴性菌的改造使之能够分泌抗肿瘤重组蛋白的难度更大,因为革兰氏阴性菌包含有两层组分不同的膜结构,即内膜和外膜,其中外膜是重组蛋白分泌的重要屏障,而合成生物学技术是解决这一难题的重要手段[8]。
目前利用合成生物学技术主要通过(1)与已知的分泌蛋白融合表达,(2) 使用细胞外膜缺陷突变株,(3) 表达裂解蛋白以诱导细胞裂解等3种策略来改造革兰氏阴性菌使之能有效地分泌胞外蛋白[9]。其中,第3种策略已被用于构建能够感应并消杀铜绿假单胞菌的工程菌,该工程菌以大肠杆菌为底盘菌,能通过感应铜绿假单胞菌产生的群体感应信号分子(N-酰基高丝氨酸内酯)来诱导E7裂解蛋白的合成而使工程菌裂解,进而释放出工程菌胞内积累的抗铜绿假单胞菌蛋白pyocin S5和抗生物被膜酶DspB,从而实现定向杀死铜绿假单胞菌的目标。但是当工程菌与铜绿假单胞菌共培养时,由于工程菌被快速诱导裂解而不利于功能蛋白在工程菌胞内的积累,因此在动物感染实验中工程菌处理后需要花很长时间才能显示出对铜绿假单胞菌的消杀效果且达到这种消杀效果所需的时间在不同的动物宿主中存在巨大差异[10]。很显然,这种通过在胞内引入外源的基因线路来调控细胞裂解的模式,其作用效果很容易受外界环境的干扰,同时额外的基因线路也增加了细菌的代谢负担。为了克服这些问题,我们尝试了2种新的策略:(1) 利用环境中的噬菌体来裂解细菌细胞;(2) 对细菌内源的裂解元件和调控元件进行重新编排和组装以达到可控裂解细胞的目的。
噬菌体是地球上最丰富多样的生物之一,必须在其专性细菌内寄生,具有严格的宿主特异性[11]。来自临床和自然环境的铜绿假单胞菌分离株中普遍存在丝状噬菌体[12]。Pf4丝状噬菌体以整合到基因组上的方式自然寄生在铜绿假单胞菌PAO1中,对PAO1毒力发挥、生物被膜发育和结构稳定起至关重要的作用[13]。缺失Pf4丝状噬菌体编码基因簇的铜绿假单胞菌对Pf4丝状噬菌体高度敏感[13]。铜绿假单胞菌除了合成能杀死自身的Pf4丝状噬菌体外,还产生各种类型的蛋白类杀菌素–脓菌素(pyocin),这些脓菌素一般分为R型、F型和S型,它们均能专一性地杀死铜绿假单胞菌自身[14]。比如,通过大肠杆菌异源表达铜绿假单胞菌的脓菌素pyocin S5基因后,该菌株显示出高效地杀死铜绿假单胞菌的活性[15]。几乎所有类型脓菌素的生物合成都受prtN调控基因的正调控。prtN基因突变导致铜绿假单胞菌中R2、F2和S2等类型脓菌素的生物合成存在缺陷[14]。PrtN蛋白通过与脓菌素合成基因5′非编码区的保守DNA序列相互作用来激活各种脓菌素合成基因的表达。正常情况下prtN基因的表达却受PrtR抑制因子的抑制,然而,当细胞处在紫外照射等可诱发细胞发生SOS反应的环境胁迫中,由SOS激活的RecA蛋白才将PrtR抑制因子降解而激活prtN基因的表达,进而诱导细胞合成各种脓菌素[14-15]。
本研究正是利用Pf4丝状噬菌体和脓菌素对铜绿假单胞菌自身的杀菌作用构建一种可定向投放蛋白的铜绿假单胞菌工程菌,以该工程菌作为微生物工具,在其胞内过表达不同的功能基因,通过工程菌的自裂解系统将目的蛋白定向投放出来以期实现特定的生物治疗目的,比如抗肿瘤等。本研究还通过在所构建的工程菌中过表达两种胞外多糖水解酶基因pelA和pslG来验证该工程菌的有效性。铜绿假单胞菌至少可以产生3种类型的胞外多糖:海藻酸钠、Pel (由部分乙酰化的N-乙酰-D-氨基葡萄糖和N-乙酰-D-氨基半乳糖组成的阳离子多糖)和Psl (一种中性多糖,由D-甘露糖、L-鼠李糖和D-葡萄糖组成的五糖重复单位)。它们作为生物被膜细胞外基质的关键组分都参与了不同情况下的生物被膜的发育[16]。Pel和Psl型胞外多糖在生物被膜基质的形成和维持中起着关键结构支架和保护屏障的作用[16-17]。PelA和PslG这两种胞外多糖水解酶已经被证实在体外可分别特异性地水解铜绿假单胞菌的Pel和Psl型胞外多糖,从而抑制或破坏铜绿假单胞菌生物被膜[16, 18-20]。此外,铜绿假单胞菌生物被膜内的死细菌也会释放出PslG而破坏生物被膜[20]。形成生物被膜可以为细菌的生存提供保护,特别是可以显著增强细菌对抗生素的抵抗能力[17, 21-22]。因此,通过检测过表达pelA和pslG基因的工程菌对铜绿假单胞菌生物被膜的抑制或破坏效果以及由此导致的铜绿假单胞菌抗生素耐药性的变化情况,可评估所构建的工程菌能否作为一种有效的定向投放蛋白的微生物工具。因此,本研究通过对铜绿假单胞菌上述相关内源基因的重新编排和组装,以期获得能够表达不同功能基因并实现功能蛋白的定向投放以执行不同生物学功能的工程菌。
1 材料和方法 1.1 菌株、质粒见表 1。
| Strains and plasmids | Description | Sources |
| Strains | ||
| Pseudomonas aeruginosa | ||
| PAO1 (ATCC15692) | Wild-type | [23] |
| PAO1∆fabV | Mutant of knockout fabV in PAO1 | This study |
| PAO1∆pf4r | Mutant of knockout pf4r in PAO1 | This study |
| PAO1∆Pf4 | Mutant of knockout pf4 cluster in PAO1 | This study |
| PAO1∆4 | Mutant of knockout pelF, pslA-B, T3SS, fabV in PAO1 | This study |
| PAO1∆5 | Mutant of knockout pelF, pslA-B, T3SS, fabV, pf4 in PAO1 | This study |
| PAO1101 | PAO1∆5 attTn7:: PPA2069-prtN with pBBR1MCS-6 | This study |
| PAO1102 | PAO1∆5 attTn7:: PPA2069-prtN with pBBR1MCS-6-pelA-pslG | This study |
| Escherichia coli | ||
| TG1 | F′[traD36 proAB+ lac Iq lacZΔM15], supE, thi-1, Δ(lac-proAB), Δ(mcrB-hsdSM)5, (rK– mK–) | [23] |
| S17-1 | RP4-2 (Km::Tn7, Tc::Mu-1), pro-82, LAMpir, recA1, endA1, thiE1, hsdR17, creC510 | [23] |
| Transetta(DE3) | Host for expression vector pET28a | [23] |
| Transetta(DE3) 1 | Transetta(DE3) with pBBR1MCS-6-pelA-pslG | This study |
| Transetta(DE3) 2 | Transetta(DE3) with pBBR1MCS-6 | This study |
| Plasmids | ||
| pK18mobSacB | Kmr; sacB-based gene replacement vector | [24] |
| p34s-Gm | Ampr; Gm resistant cassette carrying vector | [25] |
| pK18-∆fabV-Gm | Kmr; Gmr; ∆fabV::Gm in pK18mobSacB | This study |
| pK18-∆pf4r-Gm | Kmr; Gmr; ∆pf4r::Gm in pK18mobSacB | This study |
| pK18-∆Pf4-Gm | Kmr; Gmr; ∆Pf4:: Gm in pK18mobSacB | This study |
| pK18-∆pelF-Gm | Kmr; Gmr; ∆pelF::Gm in pK18mobSacB | This study |
| pK18-∆pslA-B-Gm | Kmr; Gmr; ∆pslA-B::Gm in pK18mobSacB | This study |
| pK18-∆T3SS-Gm | Kmr; Gmr; ∆T3SS::Gm in pK18mobSacB | This study |
| pBBR1MCS-5 | Broad-host-range vector, Gmr | [26] |
| pBBR1MCS-6 | Tac promoter and fabV were cloned into pBBR1MCS-5 | This study |
| pBBR1MCS-6-pelA-pslG | pelA and pslG were cloned into pBBR1MCS-6 | This study |
| pME6032 | Nru I-EcoR I lac Iq-Ptac fragment of pJF118EH subcloned in [BamH I]- EcoR I-digested pME6031; lac Iq- Ptac expression vector | [27] |
| pMini-CTX∷lacZ | Ω-FRT-attP-MCS, ori, int, oriT, Tcr | [28] |
| pMini-CTX-PPA2069∷lacZ | PPA2069 promoter was cloned into pMini-CTX::lacZ | This study |
| pUC18T-mini-Tn7T-Gm | Gmr on mini-Tn7; for gene insertion in Gms bacteria | [29] |
| pUC18T-mini-Tn7T-PPA2069-prtN-Gm | PPA2069-prtN was cloned into pUC18T-mini-Tn7T-Gm | This study |
| pTNS3 pFLP2 | Helper plasmid encoding transposase gene necessary for the chromosomal integration of mini-Tn7 ApR; source of Flp recombinase |
[30] [31] |
| pBBR-gfp mut3 | pBBR1MCS-5 containing PA1/04/03-gfp mut3-To-T1 | [32] |
1.2 引物
见表 2。
| Primers | Sequences (5′→3′) |
| pelF up F | CAGTGGATCCTGTTCCCGGCGCTGTACC |
| pelF up R | CTGCGGATTGATGAAGAACACCGAGAAG |
| pelF low F | GTTCTTCATCAATCCGCAGCGCTGGCAG |
| pelF low R | ACTGAAGCTTAAGTCGAAGGCCACCATG |
| pslA-B up F | CAGTGAATTCCTACGACGCCAAGCCATG |
| pslA-B up R | TCACCAGGTCGTAGTACAGCAGGAACAG |
| pslA-B low F | GCTGTACTACGACCTGGTGATGATCGAG |
| pslA-B low R | CGAGAAGCTTCCAGCAAGCGGTAATC |
| pf4r up F | CTAGGGATCCAGTAATGTGGTTGATCTG |
| pf4r up R | AATCGACGTCGGATAGCGATCACTAGAAAAG |
| pf4r low F | GATCGCTATCCGACGTCGATTTCTTCCGTG |
| pf4r low R | CTAGAAGCTTTTCAGTCGGGAAGACAGC |
| Pf4 up F | CTAGAGATCTACAGCGTCGAGCAAGTCC |
| Pf4 up R | CGCCAGCTAATTACATCATCAGCGAGTAG |
| Pf4 low F | GATGATGTAATTAGCTGGCGAGGGCGGCGAG |
| Pf4 low R | CTAGAAGCTTCGACCACAGCGTCCTTC |
| T3SS up F | TCGAGGATCCTGGAGACGGCTTCGGCAC |
| T3SS up R | CCACTTCCAGCACATAGTCGTAAGGCAG |
| T3SS low F | CGACTATGTGCTGGAAGTGGTCGGCGATG |
| T3SS low R | CTAGAAGCTTGGAAACCTGGTTCAGTG |
| tac F | CTCGGGTACCCCGCTTCCACTTTTTCCC |
| tac R | CAGTCTCGAGGTGTGAAATTGTTATCCG |
| pelA F | CAGTAAGCTTATCCATGCTTCCGACCTG |
| pelA R | CAGTGAATTCCCTGGGGATGCTTGTCAG |
| pslG F | CAGTGAATTCCACTGTTCAACGACATGG |
| pslG R | CAGTGAGCTCAGGATCCAGAGAATACGC |
| PA2069 F | CTGAGGTACCAATACCGACCTGCGACC |
| PA2069 R | ACTGAAGCTTGTGGCATTCGTGATGGTC |
| prtN F | AGTCGGATCCAACGACGTGTAGCCGCAG |
| prtN R | AGTCGAGCTCATGGTGTCGCTCCCTAC |
| fabV F | TACGTTCTGCAATGCGCGACGTCTCTAG |
| fabV R | TGTTAGGTGTCAGGCCTGGATCAGGTTG |
| pBBR1MCS-5 F | CCAGGCCTGACACCTAACAATTCGTTCA |
| pBBR1MCS-5 R | GTCGCGCATTGCAGAACGTAAGCCGCGAG |
| fabV up F | CAGTGAATTCTGGGCAAGAACTACAGC |
| fabV up R | TGTCCCACAGGTAGTCGATCTGTTGCTTG |
| fabV low F | GATCGACTACCTGTGGGACAAGGTAACC |
| fabV low R | AGTCAAGCTTACCTGGAAGGTGGCTCG |
1.3 培养条件
本研究中所使用的大肠杆菌、铜绿假单胞菌PAO1以及构建的各菌株均在37 ℃下培养,在摇床中振荡培养时,使用的转速为220 r/min,实验中使用的培养基为LB培养基(NaCl 10 g/L,YEAST EXTRACT 5 g/L,TRYPTONE 10 g/L)。
1.4 试剂琼脂糖、TRYPTONE、YEAST EXTRACT、TRYPTIC SOY BROTH培养基购自OXOID;琼脂粉购自北京索莱宝科技有限公司;DNA纯化回收试剂盒、质粒小提试剂盒购自天根生化科技有限公司;DNA聚合酶购自全式金生物技术有限公司;限制性内切酶购自TaKaRa生物公司;T4 DNA连接酶购自NEB;O-NITROPHENYL B-D- GALACTOPYRANOSIDE (ONPG)购自Sigma- Aldrich西格玛奥德里奇(上海)贸易有限公司;庆大霉素(gentamicin,Gm)、卡那霉素(kanamycin,Km)、氯霉素(chloramphenicol,Cm)、四环素(tetracyclin,Tc)、三氯生(triclosan,Tri)、托普霉素(tobramycin,Tob)等购自北京索莱宝科技有限公司。其他常规试剂均使用国产分析纯。
1.5 突变株的构建本研究中基因敲除的方法按照参考文献[33]执行,以铜绿假单胞菌基因组DNA为模板分别PCR扩增目的基因的上下游序列,通过重叠延伸PCR或酶切连接的方法构建部分缺失的目的片段,经酶切,连接至自杀载体pK18mobSacB上,从质粒p34s-Gm上酶切庆大霉素抗性基因GmR,连接至上一步获得的重组载体上,并转化大肠杆菌S17-1获得重组菌;通过接合将该重组菌的重组自杀性载体导入受体菌,结合抗性筛选、蔗糖筛选和PCR鉴定,获得无抗性筛选标记的目的基因删除突变株。
1.6 噬菌斑形成实验噬菌斑形成实验结合参考文献[13]的方法执行,将PAO1∆pf4r突变菌株(Pf4r是Pf4丝状噬菌体的切除酶阻遏蛋白,敲除pf4r可以使Pf4丝状噬菌体大量产生)在5 mL补加了0.1%葡萄糖和5 mmol/L CaCl2的液体LB培养基中37 ℃、220 r/min振荡培养8 h;吸取2 mL培养液,12000 r/min离心2 min,吸取上清液并通过0.22 μm无菌滤膜过滤,滤液即为Pf4丝状噬菌体溶液;然后,用LB将获得的Pf4丝状噬菌体溶液通过连续10倍稀释;每个梯度吸取10 μL点板在涂布有PAO1或PAO1∆Pf4菌液的固体平板上,在37 ℃培养箱中培养过夜,观察噬菌斑。
1.7 β-半乳糖苷酶酶活检测实验使用参考文献[34]方法测定转录融合报告菌株的β-半乳糖苷酶。方法如下:在离心管中依次加入Z缓冲液420 µL、待测菌液100 µL、氯仿20 µL、0.1% SDS 10 µL混匀,30 ℃条件下放置至少1 h;将加入了0.004 g/mL ONPG的底物缓冲液,充分溶解后30 ℃放置30 min;开始酶活检测时,每管样品中加入100 µL底物缓冲液,30 ℃条件下开始酶活反应,反应时间为30–90 min;反应完成后,加入250 μL的1 mol/L Na2CO3终止反应,并记录反应时间;最后分别检测反应液在420 nm和550 nm处的吸光值,依据公式(1)计算相应的β-半乳糖苷酶酶活。

|
公式(1) |
Z缓冲液:100 mmol/L Na2HPO4、40 mmol/L NaH2PO4、10 mmol/L KCl、1 mmol/L MgSO4和5.4 µL/mL β-巯基乙醇。
底物缓冲液:60 mmol/L Na2HPO4、40 mmol/L NaH2PO4、2.7 µL/mL β-巯基乙醇和4 mg/mL ONPG。
1.8 静态生物被膜培养实验结合参考文献[16]方法执行,将培养OD600至2.5的细菌,用新鲜培养基按1︰100稀释后,混匀;取100 μL加入96孔PVC板中,37 ℃静置培养24 h;吸去培养液,用PBS洗涤PVC板2次,以除去非粘附细胞和培养基,空气中干燥;室温下加入0.1%结晶紫对形成的生物被膜染色10 min,再用PBS洗涤2次;最后用等体积95%乙醇洗提96孔PVC板上每孔中生物被膜上结合的结晶紫,并用酶标仪测定洗提液的OD570进行生物被膜定量。
1.9 pelA和pslG的异源表达和活性检测实验结合参考文献[14]方法执行,将表达载体pBBR1MCS-6-pelA-pslG热激转化到大肠杆菌Transetta(DE3)中,得到相应的大肠杆菌重组菌在添加有5 μg/mL三氯生的液体LB培养基中37 ℃培养过夜,按照1︰100接种量转接至添加有5 μg/mL三氯生的50 mL液体LB培养基中37 ℃振荡培养OD600至0.5后添加0.5 mmol/L IPTG诱导,接着在18 ℃振荡培养过夜,4000 r/min离心8 min收集菌体,10 mL PBS缓冲液洗涤菌体2次,用5 mL PBS缓冲液重悬,超声波破碎,按20%的比例将30 mg/mL蛋白浓度的细胞裂解液混入PBS,吸取200 μL混合液加入96孔PVC板中孵育8 h (96孔PVC板中含有静态培养完成的铜绿假单胞菌生物被膜),然后用PBS洗涤PVC板2次将反应淬灭,空气中干燥;加入0.1%结晶紫对形成的生物被膜染色10 min,再用PBS洗涤2次;最后用等体积95%乙醇洗提96孔板上每孔中生物被膜上结合的结晶紫,并用酶标仪测定洗提液的OD570进行生物被膜定量。以带有pBBR1MCS-6空载体的大肠杆菌Transetta(DE3)菌株作为对照。
1.10 有剪切力存在下形成生物被膜实验结合参考文献[10]方法执行,将无菌玻璃盖玻片浸没于装有5 mL LB液体培养基的平皿中,按照1︰100接种量,在平皿中转接培养过夜的带有GFP绿色荧光蛋白标记的铜绿假单胞菌(携带pBBR-gfp mut3质粒);37 ℃、100 r/min振荡培养72 h后,取出盖玻片使用PBS缓冲液冲洗2次,以除去非粘附细胞和培养基,空气中干燥,即可得到带有绿色荧光蛋白的铜绿假单胞菌生物被膜;将制好的玻片在激光共聚焦扫描显微镜下观察生物被膜的形成情况。
1.11 细菌共培养菌落计数实验结合参考文献[15]方法执行,为了对共培养的工程菌PAO1102和铜绿假单胞菌PAO1进行菌落计数,将pME6032质粒和pBBR1MCS-5质粒分别转入工程菌PAO1102和PAO1中,构建PAO1102 (pME6032)和PAO1 (pBBR1MCS-5)菌株;将两种细菌分别培养至OD600=2.5,以1︰1混匀,按照1%的接种量将混合菌液接种于5 mL新鲜的LB培养基中,37 ℃、220 r/min振荡培养;将培养的菌液每隔12 h取100 μL,通过连续10倍稀释,每个稀释梯度吸取3 μL菌液,分别在200 μg/mL四环素抗性平板和200 μg/mL庆大霉素抗性平板上点板,对菌液中PAO1102 (pME6032)和PAO1 (pBBR1MCS-5)菌株进行CFU计数,最后用Log10 (CFU/mL)绘制生长曲线;同时为了防止培养过程中因质粒丢失造成菌落计数出现误差,实验以PAO1 (pME6032)和PAO1 (pBBR1MCS-5)共培养的菌落计数作为对照。
1.12 生物被膜破坏实验用1.8和1.10的方法培养获得生物被膜;加入200 μL培养至OD600=4.0的工程菌孵育8 h,然后用PBS洗涤2次将反应淬灭,空气中干燥。对于静态培养形成的生物被膜,加入0.1%结晶紫对形成的生物被膜染色10 min,再用PBS洗涤2次,最后用等体积95%乙醇洗提96孔板上每孔中生物被膜上结合的结晶紫,并用酶标仪测定洗提液的OD570进行生物被膜定量;对于剪切力下形成的生物被膜,直接用激光共聚焦扫描显微镜观察。
1.13 生物被膜预防实验实验结合参考文献[15]方法执行,将铜绿假单胞菌PAO1和工程菌PAO1102分别培养至OD600=2.5,以1︰1混匀成混合菌液,用新鲜培养基按1︰100稀释后,取100 μL混合菌液加入96孔PVC板中,37 ℃静置培养24 h;吸去培养液,用PBS洗涤PVC板2次,空气中干燥;加入0.1%结晶紫对形成的生物膜染色10 min,再用PBS洗涤2次;用等体积95%乙醇洗提96孔板上每孔中生物被膜上结合的结晶紫,并用酶标仪测定洗提液的OD570进行生物被膜定量。以铜绿假单胞菌PAO1和PAO1101菌株共培养形成的生物被膜作为对照。
1.14 生物被膜内细菌的抗生素耐药性实验实验结合参考文献[16]方法执行,将带有四环素抗性筛选标记的铜绿假单胞菌PAO1 (pME6032)振荡培养OD600至2.5,分别用新鲜培养基按1︰100稀释后,取100 μL加入96孔PVC板中,37 ℃静置培养24 h;吸去培养液,用PBS洗涤PVC板2次,得到铜绿假单胞菌的生物被膜;加入200 μL培养至OD600=4.0的工程菌在37 ℃下孵育8 h后,吸去菌液;加入200 μg/mL妥布霉素处理2 h后,使用PBS洗涤PVC板将反应淬灭;用600 μL PBS将4个孔中的生物被膜通过刮擦并用力吸打,收集在一起,确保洗下PVC板上粘附的所有细胞以防止实验偏差,每次实验设置3组重复;通过连续10倍稀释,每个稀释梯度吸取3 μL菌液,在相应四环素抗性平板上点板,通过CFU计数生物被膜中铜绿假单胞菌PAO1的细菌存活数。以不处理、单独加对照菌株PAO1101、单独加工程菌PAO1102、单独加200 μg/mL妥布霉素以及先加入PAO1101菌株孵育再加入200 μg/mL妥布霉素的处理作为对照。
1.15 统计分析实验结果以平均值±标准偏差的形式体现,使用Student’s t-test (双尾不配对)对显著性进行分析。用GraphPad Prism version 5.00软件(GraphPad software Inc.; San Diego,CA,USA)对结果进行统计学分析并作图,P<0.05代表显著。*:P<0.05;**:P<0.01;***:P<0.001。所有的实验都至少设3个生物学平行并且独立重复2次。
2 结果和分析 2.1 Pf4丝状噬菌体介导的细胞被动裂解为了构建一种可定向投放蛋白的铜绿假单胞菌工程菌,本研究首先利用了Pf4丝状噬菌体对铜绿假单胞菌自身的杀菌作用这一特性。在长期进化过程中,铜绿假单胞菌PAO1基因组中整合进一种温和性丝状噬菌体Pf4,它不仅有助于铜绿假单胞菌的毒力发挥,而且在生物被膜的发育和结构完整性中起着重要的作用,同时也是铜绿假单胞菌生物被膜的组成成分之一[13]。相比在浮游生长的细菌中,该丝状噬菌体Pf4的编码基因簇在生物被膜中的表达受到显著诱导[13]。如图 1-A所示,在敲除了铜绿假单胞菌基因组中编码Pf4丝状噬菌体的完整基因簇后,构建PAO1∆Pf4突变株,并以野生铜绿假单胞菌PAO1为对照,进行噬菌斑形成实验,结果如平板上噬菌斑形成表型图(图 1-B)和对应的定量图(图 1-C)所示,Pf4丝状噬菌体按梯度稀释后点在PAO1平板上,最后出现噬菌斑的梯度为10–2;而在PAO1ΔPf4突变株平板上噬菌斑最后出现的梯度为10–6。结果表明:相比于野生PAO1菌株,PAO1ΔPf4突变株对Pf4丝状噬菌体的敏感性提高了104倍。因此可利用野生铜绿假单胞菌产生的Pf4丝状噬菌体具有侵染并裂解PAO1∆Pf4突变株的特性来建立一种具备细胞被动裂解机制的工程菌。利用这种被动裂解机制,当遇到生物被膜中或环境中的Pf4丝状噬菌体时工程菌可以很容易被Pf4丝状噬菌体裂解。
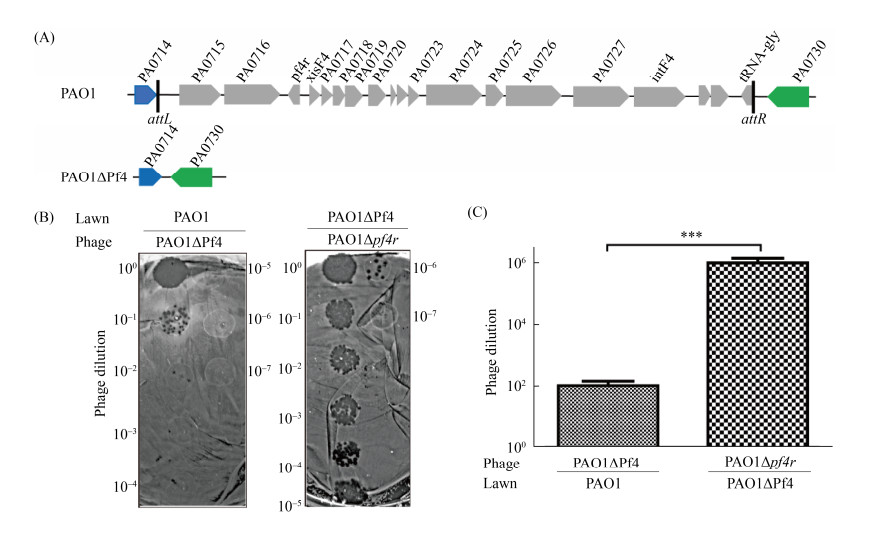
|
| 图 1 PAO1ΔPf4突变株对Pf4丝状噬菌体更敏感 Figure 1 The PAO1ΔPf4 mutant is more sensitive to Pf4 filamentous phage. A: a schematic diagram illustrates the excision of the prophage Pf4 from the PAO1 host chromosome. B: plaque formation by the phage lysates. Phage lysates were serially diluted and 10 μL samples were dropped on PAO1 and ΔPf4 lawns, respectively. C: Pf4 phage titers (phage dilution ratio) were quantified on PAO1 and ΔPf4 lawns using supernatant from PAO1Δpf4r as shown in B. ***: P < 0.001. |
2.2 脓菌素激活蛋白PrtN介导的细胞主动裂解
除了细胞被动裂解机制外,在工程菌中设计一种细胞主动裂解机制以扩大其适用范围,为此本研究选择了铜绿假单胞菌脓菌素激活蛋白PrtN,它可通过激活prf24、prf9等细胞裂解蛋白基因的表达[14]使工程菌裂解。同时,为了使工程菌可以生长到一定细胞密度,让目的基因有充足的时间表达,我们选择铜绿假单胞菌群体感应系统调控的启动子延迟表达prtN基因,形成依赖细胞密度的启动裂解机制。在铜绿假单胞菌中至少有4种不同的群体感应系统:las、rhl、pqs和iqs[35],其中rhl系统中信号分子BHL (N-丁酰-L-高丝氨酸内酯)由RhlI负责合成并与其受体蛋白RhlR结合形成RhlR-BHL复合物而激活相关靶标基因的表达[35]。转录组分析显示,铜绿假单胞菌中功能未知的操纵子PA2066–PA2069,其表达受BHL信号分子的激活,暗示该操纵子的启动子(PPA2069)是RhlR-BHL复合物的激活靶标之一[36]。如图 2-A,将PA2069启动子连接在pMini- CTX::lacZ质粒上构建pMini-PPA2069-CTX::lacZ重组载体,分别转入PAO1、PAO1∆rhlI、PAO1∆pqsE、PAO1∆rhlR中,按照细菌的不同生长时期即不同的OD600检测β-半乳糖苷酶酶活,在PAO1野生菌和PAO1∆pqsE突变株生长前期没有检测到显著水平的β-半乳糖苷酶酶活,而在细菌生长进入稳定期(OD600=2.0)时β-半乳糖苷酶酶活显著上升,并最终达到了17372.82 miller units的水平,而在PAO1∆rhlI和PAO1∆rhlR突变株中最高只能检测到约1430.69 miller units水平的β-半乳糖苷酶酶活。这些结果说明:PA2069启动子受到铜绿假单胞菌RhlRI群体感应系统的正调控,其表达依赖于细菌的群体密度。

|
| 图 2 细胞主动裂解系统的检测 Figure 2 Characterization of active cell lysis system. A: transcription from the promoter of the PA2069 in P. aeruginosa PAO1 and various mutants; B: the effect of expression of prtN on the growth of P. aeruginosa. *: P < 0.05; **: P < 0.01; ***: P < 0.001. |
因此,将脓菌素激活基因prtN和PA2069启动子,经酶切连接在pUC18T-mini-Tn7T-Gm质粒上,构建主动裂解系统的重组载体pUC18T-mini- Tn7T-PPA2069-prtN-Gm。为了验证该系统对铜绿假单胞菌的裂解效果,如图 2-B,在辅助质粒pTNS3的帮助下将重组质粒pUC18T-mini-Tn7T-PPA2069- prtN-Gm电转导入PAO1中获得的基因组整合菌株作为实验菌株,以基因组中整合空质粒的菌株作为对照,每隔6 h在培养基中检测2株菌株的OD600。结果显示在细菌生长前期2株菌株生长没有显著差异,在细菌进入稳定生长期后的36 h,实验菌株较对照菌株开始出现明显的生长衰退现象,说明细菌生长到稳定期后PA2069的启动子才启动prtN基因表达从而诱导细胞裂解。
2.3 pBBR1MCS-6-pelA-pslG表达元件的构建及异源表达本研究在工程菌中过表达2种胞外多糖水解酶基因pelA和pslG,以期通过检测该菌株对铜绿假单胞菌生物被膜的抑制或破坏效果来验证所构建工程菌的有效性。首先,为了使表达系统能最大程度表达目的基因,本研究使用表 2中的引物PCR扩增pME6032载体上的tac启动子,克隆至载体pBBR1MCS-5 (该载体自身带有lac启动子),构建pBBR1MCS-5-Ptac双启动子载体,因该载体不存在lacI基因,它在无lacI基因菌株中的表达是组成型的。同时为了防止构建的工程菌在使用时发生质粒水平转移带来的安全问题,本研究使用铜绿假单胞菌自身的三氯生抗性基因-烯脂酰ACP还原酶编码基因fabV[37]替换pBBR1MCS-5-Ptac重组载体上的庆大霉素(Gm)抗性基因。使用PCR扩增铜绿假单胞菌的fabV基因序列和pBBR1MCS-5-Ptac重组载体上的非Gm抗性基因序列,经酶切和连接构建重组载体pBBR1MCS-5-Ptac-GmR::fabV,命名为pBBR1MCS-6。为了验证是否将抗性基因替换成功,将载体pBBR1MCS-6和pBBR1MCS-5-Ptac经质粒接合转移,分别转入PAO1和PAO1ΔfabV,再经庆大霉素和三氯生抗性检验,如图 3-A,与PAO1∆fabV (pBBR1MCS-5-Ptac)菌株相反,PAO1∆fabV (pBBR1MCS-6)菌株在添加5 μg/mL三氯生的LB培养基中可以生长,而在添加200 μg/mL庆大霉素的LB平板上不能生长,表明三氯生抗性基因fabV已经成功替换pBBR1MCS-5质粒上的Gm抗性基因。
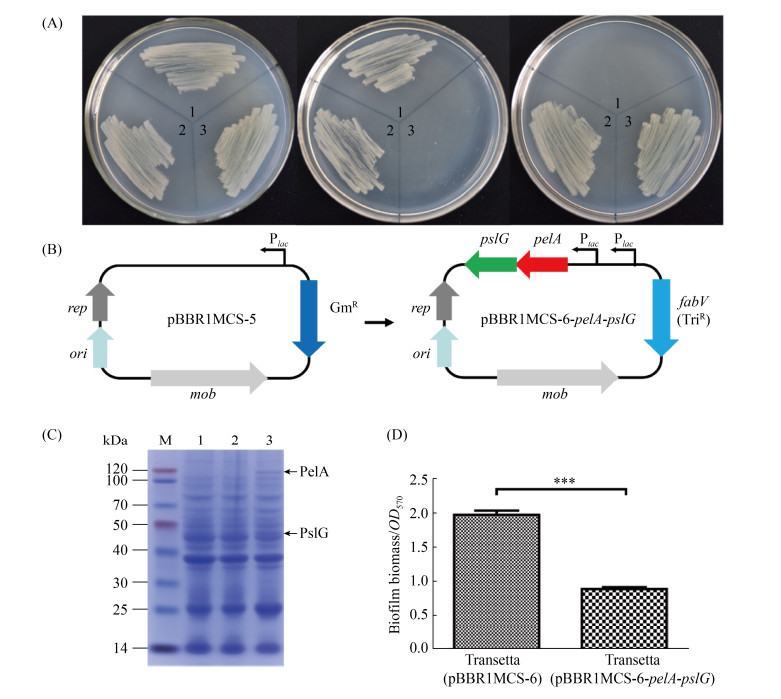
|
| 图 3 pBBR1MCS-6-pelA-pslG表达元件的构建及其在大肠杆菌中的表达 Figure 3 Construction and characterization of pBBR1MCS-6-pelA-pslG. A: fabV, the target of triclosan resistance, replaced gentamicin resistance gene on pBBR1MCS-5 vector. Lane 1: PAO1∆fabV (pBBR1MCS-6); lane 2: PAO1 (pBBR1MCS-5-Ptac); lane 3: PAO1∆fabV (pBBR1MCS-5-Ptac). LB (left): LB+5 μg/mL triclosan (middle), LB+200 μg/mL gentamicin (right). B: construction of pBBR1MCS-6-pelA-pslG. C: SDS-PAGE of total proteins from E. coli. M: protein marker; lane 1: the total proteins from Transetta (pBBR1MCS-6) after induction with IPTG; lane 2 and 3: the total proteins from Transetta (pBBR1MCS-6-pelA-pslG) after induction without IPTG (lane 2) or with IPTG (lane 3). D: biofilm disperse by the lysates from E. coli carrying pBBR1MCS-6 or pBBR1MCS-6-pelA-pslG after induction with IPTG. ***: P < 0.001. |
其次,PCR扩增铜绿假单胞菌PAO1的胞外多糖水解酶基因(包括基因自身的核糖体结合位点) pelA和pslG,经酶切后串联连接到pBBR1MCS-6载体上,获得重组载体pBBR1MCS- 6-pelA-pslG (图 3-B)。为了检测所构建的重组载体是否可以表达出胞外多糖水解酶PelA和PslG,本研究进一步将pBBR1MCS-6-pelA-pslG热激转化到大肠杆菌Transetta(DE3)中,以pBBR1MCS-6空载体作为对照。IPTG诱导后经SDS-PAGE凝胶电泳检测,显示Transetta(DE3) (pBBR1MCS-6- pelA-pslG)菌株能够诱导产生PelA蛋白和PslG蛋白(图 3-C)。最后,将诱导表达菌体的细胞裂解液加入在静态培养形成的生物被膜中处理8 h,结果如图 3-D,与对照菌株处理的铜绿假单胞菌生物被膜相比,经实验组处理的生物被膜的含量下降了约55% (P < 0.001)。说明pBBR1MCS-6-pelA- pslG可以正常表达并产生有活性的胞外多糖水解酶PelA和PslG。
2.4 工程菌的构建及其定向释放功能蛋白的原理由于铜绿假单胞菌生物被膜的主要成分为Pel型和Psl型胞外多糖,因此敲除pelF和pslA-B可丧失铜绿假单胞菌形成生物被膜的能力[16, 38];作为铜绿假单胞菌重要毒力因子的Ⅲ型分泌系统(T3SS),敲除其针筒状结构蛋白编码基因pscF可失活T3SS,从而降低铜绿假单胞菌的致病性[39];敲除作为三氯生抗性靶点的烯脂酰ACP还原酶编码基因fabV,可使铜绿假单胞菌对三氯生敏感[37]。所以在铜绿假单胞菌PAO1基因组上连续敲除pelF、pslA-B、T3SS和fabV,获得的四重基因突变株命名为PAO1Δ4。
在PAO1Δ4的基础上再敲除Pf4丝状噬菌体编码基因簇,得到PAO1Δ5突变株;将pUT18C- mini-Tn7T-PPA2069-prtN-Gm与辅助质粒pTNS3共转化至PAO1Δ5菌株中,经抗性筛选获得基因组融合菌株PAO1Δ5:: pUT18C-mini-Tn7T-PPA2069- prtN-Gm;将S17-1 pFLP2菌株中的pFLP2质粒导入PAO1Δ5:: pUT18C-mini-Tn7T-PPA2069-prtN-Gm菌株中,结合抗性筛选、蔗糖筛选,获得无Gm抗性筛选标记的菌株PAO1Δ5 attTn7::PPA2069-prtN;最后再将重组质粒pBBR1MCS-6-pelA-pslG导入PAO1Δ5 attTn7::PPA2069-prtN菌株中,通过抗性筛选,获得最终的铜绿假单胞菌工程菌PAO1102。将pBBR1MCS-6载体转入PAO1Δ5 attTn7::PPA2069- prtN菌株中,通过抗性筛选,获得最终的菌株命名为PAO1101,作为工程菌PAO1102的实验对照菌株。
铜绿假单胞菌工程菌PAO1102定向投放功能蛋白原理如图 4所示:在培养过程中,工程菌大量表达并在胞内积累胞外多糖水解酶PelA和PslG;当细胞密度达到阈值时,rhl群体感应系统诱导PA2069启动子表达脓菌素激活蛋白基因prtN,PrtN激活细胞产生Prf9和Prf24等裂解蛋白而诱导工程菌裂解,使积累在胞内的胞外多糖水解酶PelA和PslG大量投放到胞外,经酶解反应破坏生物被膜的骨架成分Pel和Psl型胞外多糖,从而破坏铜绿假单胞菌的生物被膜。此外,当工程菌接触到生物被膜中或者环境中的Pf4丝状噬菌体时,Pf4丝状噬菌体会侵染并裂解工程菌,以投放胞内积累的胞外多糖水解酶PelA和PslG,从而破坏铜绿假单胞菌的生物被膜。
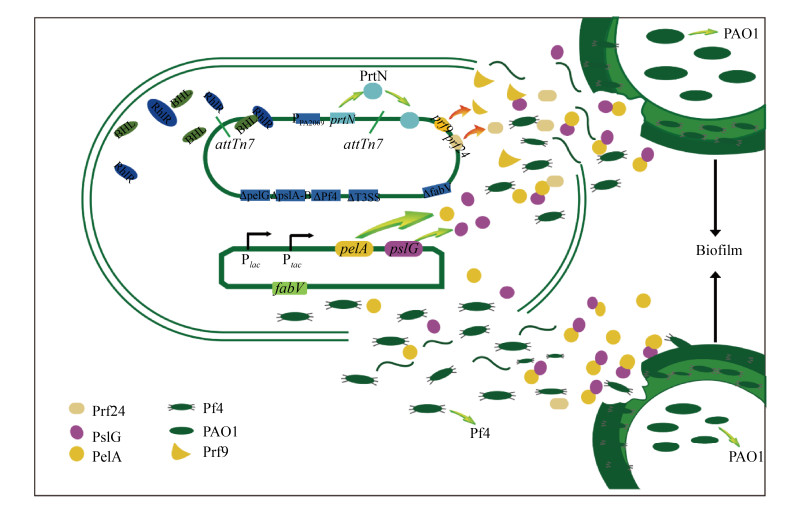
|
| 图 4 工程菌PAO1102定向投放功能蛋白的模型图 Figure 4 The schematic representation of engineered strain PAO1102 for targeted delivery of functional protein. The engineered bacteria expressed and accumulated the extracellular polysaccharide hydrolase PelA and PslG in large quantities during culture. When the cell density reaches the threshold, the RHL quorum-sensing system induced PA2069 promoter to express the pyocin activating protein gene prtN. And then PrtN activates the cells to produce lytic proteins such as Prf9 and Prf24, and induces lysis of engineering bacteria, so that the PelA and PslG accumulated in the cell are released into the extracellular. The cytoskeletal components of the biofilm Pel and Psl exopolysaccharide were destroyed by enzymatic hydrolysis reaction of the PelA and PslG, thus destroying the biofilm of P. aeruginosa. In addition, when the engineered bacteria contact the Pf4 filamentous phage in the biofilm or in the environment, the Pf4 filamentous phage will infect and lyse the engineered bacteria, to release the PelA and PslG accumulated in the cell, thereby destroying the P. aeruginosa biofilm. |
2.5 工程菌PAO1102的表型分析
在开始验证工程菌PAO1102对铜绿假单胞菌生物被膜的破坏效果之前,首先检测了在工程菌PAO1102构建的生物模块是否使细菌自身受到损害,工程菌PAO1102自身形成生物被膜的情况以及工程菌PAO1102的裂解元件是否起作用。如图 5-A所示,工程菌PAO1102的生长与铜绿假单胞菌生长状况在稳定生长期之前无显著差异,说明在铜绿假单胞菌内构建的这些生物模块并不会对细菌的生长产生毒害作用。工程菌PAO1102的生物被膜形成情况如图 5-B所示,与野生铜绿假单胞菌PAO1相比工程菌PAO1102产生的生物被膜下降了90.2% (P < 0.001),说明工程菌PAO1102基本丧失了形成生物被膜的能力。因此,在后续实验中不会因工程菌产生生物被膜而影响实验结果。为了检测所构建的裂解元件介导工程菌PAO1102的自我裂解效果,本研究将工程菌PAO1102 (pME6032)分别与野生菌PAO1 (pBBR1MCS-5)和Pf4丝状噬菌体基因簇缺失突变株PAO1ΔPf4 (pBBR1MCS-5)共培养并对混合菌液中两种细菌进行菌落计数。结果如图 5-C所示,工程菌PAO1102 (pME6032)与野生菌PAO1 (pBBR1MCS-5)共培养48 h后开始出现显著的生长差异,此时相比PAO1 (pBBR1MCS-5)菌株,工程菌PAO1102 (pME6032)的活菌数显著下降,并且在72 h时下降了约107倍(P<0.01);而同样的条件下,工程菌PAO1102 (pME6032)与Pf4丝状噬菌体基因簇缺失突变株PAO1ΔPf4 (pBBR1MCS-5)共培养时出现生长差异的时间明显延后,需要在72 h后才出现明显的生长差异现象,此时相比PAO1ΔPf4 (pBBR1MCS-5)菌株,工程菌PAO1102 (pME6032)的活菌数显著下降,下降了约103倍(P<0.05) (图 5-D);同时图 5-E对照组PAO1 (pME6032)和PAO1 (pBBR1MCS-5)共培养的细菌菌落计数无显著差异,表明图 5-C和图 5-D中2种菌株的生长差异不是由细菌在培养过程中pME6032质粒的丢失造成的。这些结果说明工程菌PAO1102可以通过所构建的裂解元件介导自身有效裂解,并且这种裂解效果主要由环境中Pf4丝状噬菌体介导的被动裂解过程造成的。
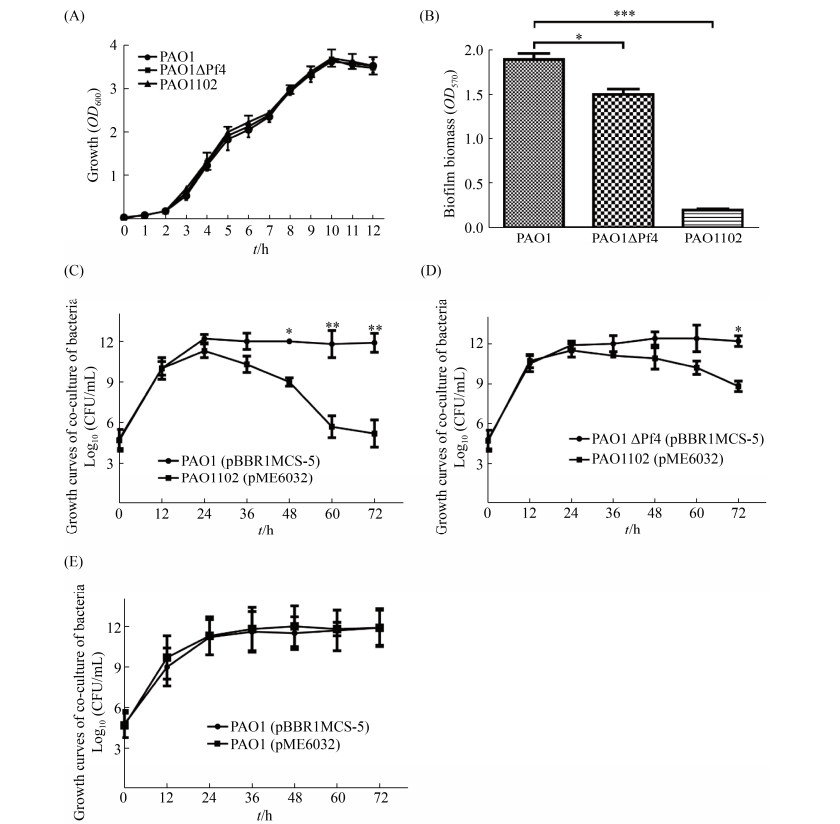
|
| 图 5 工程菌PAO1102的表型分析 Figure 5 Characterization of the engineered strain PAO1102. A: the growth curve of P. aeruginosa PAO1 strain and its derivatives; B: biofilm biomass of PAO1, ΔPf4 and PAO1102 after 24 h of growth on 96-well PVC plates. C, D and E: growth curves of co-culture between PAO1 strain and its derivative. *: P < 0.05; **: P < 0.01; ***: P < 0.001. |
2.6 工程菌PAO1102可破坏已形成的铜绿假单胞菌生物被膜
在得到工程菌PAO1102后,接着本研究分别检验了PAO1102对已经形成的野生型PAO1生物被膜和PAO1ΔPf4生物被膜的破坏效果。在将工程菌PAO1102加入在静态培养形成的野生型PAO1生物被膜中处理8 h后,结果如图 6-A所示,与对照菌株PAO1101处理的铜绿假单胞菌生物被膜相比,经工程菌PAO1102处理的生物被膜的含量下降了47.3% (P < 0.001)。同样的,在激光共聚焦扫描显微镜下可以观察到,与对照菌株PAO1101相比,经工程菌PAO1102处理的有剪切力存在的条件下形成的生物被膜显著减少(图 6-B)。而在使用PAO1102处理的PAO1ΔPf4菌株形成的生物被膜后,对PAO1ΔPf4静态培养(图 6-C)和有剪切力培养(图 6-D)形成的生物被膜均无显著破坏作用。虽然PAO1和PAO1ΔPf4所形成的生物被膜在结构上有所区别,但是它们经工程菌处理后表型出现显著差异。这些结果表明在接触到野生型铜绿假单胞菌生物被膜后,PAO1102工程菌可以按照预期方式,定向投放胞外多糖水解酶PelA和PslG,以破坏已经形成的铜绿假单胞菌生物被膜,而这个过程主要由生物被膜中Pf4丝状噬菌体诱导的PAO1102细胞裂解所介导的。

|
| 图 6 工程菌PAO1102破坏已形成生物被膜的能力 Figure 6 Anti-biofilm activity of engineered strain PAO1102 against mature P. aeruginosa biofilm. A: the anti-biofilm activity of engineered strain PAO1102 against mature PAO1 biofilm produced by static culture; B: the anti-biofilm activity of engineered strain PAO1102 against mature PAO1 biofilm formed in the presence of shear force; C: the anti-biofilm activity of engineered strain PAO1102 against mature PAO1ΔPf4 biofilm produced by static culture; D: the anti-biofilm activity of engineered strain PAO1102 against mature PAO1ΔPf4 biofilm formed in the presence of shear force. A & C: anti-biofilm activity was measured by quantifying biofilm mass (by crystal violet staining) after treating mature biofilm with engineered strain PAO1102; B & D: anti-biofilm activity was measured by visualizing biofilm with confocal laser scanning microscopy (CLSM) after treating mature biofilm with engineered strain PAO1102. Excited by argon laser (488 nm) and photographed under objective lens×10, and the scale in the figure is 50 μm. ***: P < 0.001. |
2.7 工程菌PAO1102可有效抑制铜绿假单胞菌生物被膜的形成能力
由于胞外多糖水解酶能有效地破坏已建立的生物被膜,本研究进一步确定工程菌PAO1102是否可以抑制铜绿假单胞菌在生长过程中形成的生物被膜。由于工程菌基本不形成生物被膜(图 5-B),以PAO1101为对照,在将工程菌PAO1102分别与野生菌PAO1和PAO1ΔPf4菌株共培养,并检测静态条件下和有剪切力存在的条件下生物被膜形成情况。结果显示与对照菌株PAO1101相比,与工程菌PAO1102共培养时,PAO1菌株生物被膜的形成量下降了52.8% (P < 0.001),如图 7-A;在激光共聚焦扫描显微镜下可以观察到,经工程菌PAO1102处理的有剪切力存在的条件下形成的生物被膜也显著减少(图 7-B)。而将工程菌PAO1102与PAO1ΔPf4共培养时,PAO1ΔPf4菌株静态培养形成的生物被膜(图 7-C)和激光共聚焦扫描的有剪切力形成的生物被膜(图 7-D)均无显著差异。虽然PAO1和PAO1ΔPf4所形成的生物被膜在结构上有所区别,但是它们经工程菌处理后表型出现显著差异。这些结果说明,工程菌PAO1102与野生铜绿假单胞菌共培养形成生物被膜的过程中,由于丝状噬菌体Pf4的编码基因簇在生物被膜中的表达受到显著诱导,因此产生的Pf4丝状噬菌体可以将工程菌PAO1102裂解,从而释放胞外多糖水解酶PelA和PslG,抑制铜绿假单胞菌PAO1生物被膜的形成。
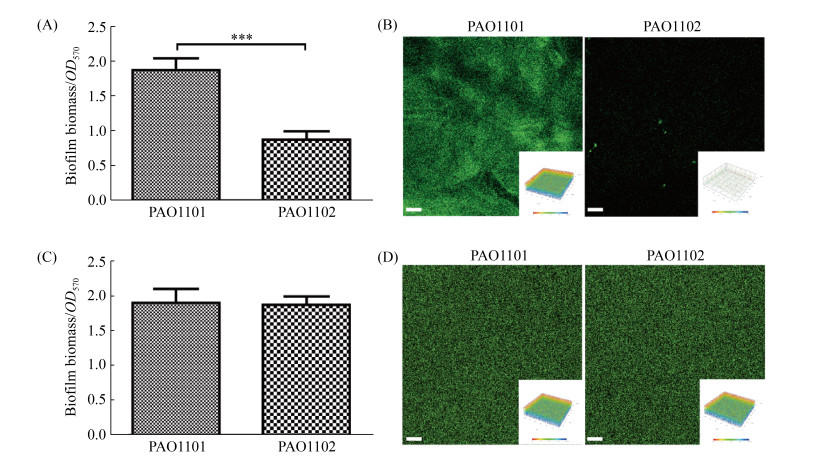
|
| 图 7 工程菌PAO1102预防生物被膜的形成 Figure 7 Anti-biofilm activity of engineered strain PAO1102 against P. aeruginosa biofilm formation. A: P. aeruginosa PAO1 was co-cultured with engineered strain PAO1102 or its control PA1101 under static conditions to evaluate its effect on biofilm formation by crystal violet staining; B: P. aeruginosa PAO1 was co-cultured with engineered strain PAO1102 or its control PA1101 in the presence of shear force to evaluate its effect on biofilm formation by visualizing biofilm with confocal laser scanning microscopy (CLSM); C: P. aeruginosa PAO1ΔPf4 was co-cultured with engineered strain PAO1102 or its control PA1101 under static conditions to evaluate its effect on biofilm formation by crystal violet staining; D: P. aeruginosa PAO1ΔPf4 was co-cultured with engineered strain PAO1102 or its control PA1101 in the presence of shear force to evaluate its effect on biofilm formation by visualizing biofilm with confocal laser scanning microscopy (CLSM). Excited by argon laser (488 nm) and photographed under objective lens×10, and the scale in the figure is 50 μm. ***: P < 0.001. |
2.8 工程菌PAO1102可降低生物被膜内细菌对抗生素的耐药性
之前研究表明,细菌产生的生物被膜可以将细菌包裹在生物被膜基质中,从而提高细菌对于抗生素的耐药性[22]。那么细菌生物被膜破坏之后,可能导致生物被膜中的细菌对抗生素更加敏感。为此本研究将带有四环素抗性基因的pME6032空载体转入铜绿假单胞菌PAO1中,通过静置培养的方式形成带有四环素抗性的铜绿假单胞菌生物被膜,接着通过四环素抗性平板的CFU计数执行该生物被膜对妥布霉素的耐药性实验。结果如图 8所示,图中前5组分别显示了不处理、单独加对照菌株PAO1101、单独加工程菌PAO1102、单独加200 μg/mL妥布霉素以及先加入对照菌株孵育再加入200 μg/mL妥布霉素处理时生物被膜中的存活菌数,结果显示在这些条件下生物被膜中的存活菌数没明显差异;而相比前五组对照实验,第6组中先加入工程菌PAO1102孵育再加入200 μg/mL妥布霉素处理后,生物被膜中存活菌数降低了大约1000倍。这些结果表明,通过先添加工程菌PAO1102破坏细菌生物被膜再使用抗生素,可以将被破坏的生物被膜中裸露出来的细菌杀死,达到根除生物被膜中的病原细菌,实现通过破坏生物被膜来降低病原细菌抗生素耐药性的目标。
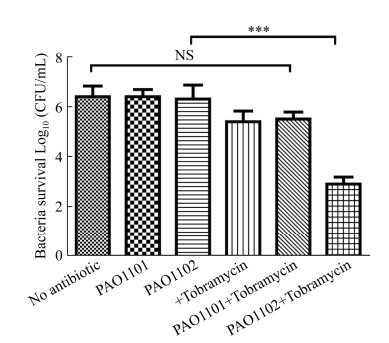
|
| 图 8 工程菌PAO1102可降低生物被膜内细菌对抗生素的耐药性 Figure 8 Combined engineered strain PAO1102 treatment of PAO1 biofilms improves antibiotic efficacy. ***: P < 0.001; NS: not significant difference. |
3 讨论
合成生物学在近年来得到越来越多的关注,并且通过合成生物学构建的一些生物系统,成功应用于能源、环境和医疗保健等领域[40]。本研究在铜绿假单胞菌PAO1∆4中构建了表达元件和自裂解系统,使构建的工程菌可以作为一种微生物工具,用于表达各种不同功能的目的基因,并通过工程菌的基于Pf4丝状噬菌体的细胞被动裂解机制和基于脓菌素的细胞主动裂解机制定向投放胞内积累的目的蛋白以执行特定的生物学功能。
通过在工程菌中过表达胞外多糖水解酶基因pelA和pslG,用于检测对铜绿假单胞菌生物被膜抑制或破坏效果,以此来评估工程菌的有效性。通过生物被膜的破坏实验,结果显示了工程菌PAO1102分别可以在8 h内显著破坏铜绿假单胞菌静态培养产生的生物被膜(图 6-A)和有剪切力条件下形成的生物被膜(图 6-B),同时共培养时工程菌PAO1102也可以显著抑制铜绿假单胞菌在静态(图 7-A)和有剪切力条件下(图 7-B)生物被膜的形成,而且PAO1102的这些功能主要依赖于Pf4噬菌体介导的细胞被动裂解过程,相反,由脓菌素激活蛋白PrtN介导的细胞主动裂解过程则显得可有可无。为了进一步比较被动裂解装置和主动裂解装置之间工作效率的差异情况,我们通过数学建模的方法,使用Michaedis-Menten动力学方程对这2种裂解方式进行模拟分析,结果显示以Pf4丝状噬菌体介导的工程菌被动裂解释放2种水解酶PelA和PslG完全破坏生物被膜需要1 h,而以脓菌素激活蛋白PrtN介导的工程菌主动裂解释放2种水解酶PelA和PslG完全破坏生物被膜则需要100 h (数据未展示)。这些结果说明,相比于依赖脓菌素激活蛋白PrtN介导的细胞主动裂解过程,依赖于Pf4丝状噬菌体介导的细胞被动裂解过程在工程菌PAO1102破坏生物被膜过程中的作用更加高效。
一直以来,细菌生物被膜是临床上彻底治愈病原菌感染的最大障碍之一[17, 20]。本研究通过生物被膜内细菌的抗生素耐药性实验(图 8),结果显示相比对照,工程菌PAO1102处理后使铜绿假单胞菌PAO1生物被膜中的细菌对妥布霉素的敏感性增加了近1000倍,这说明工程菌PAO1102通过破坏生物被膜可有效降低生物被膜中细菌的耐药性。这为细菌生物被膜相关感染(特别是慢性伤口细菌生物被膜)的治疗提供了一种新的思路。
这些结果说明本研究所构建的铜绿假单胞菌工程菌可以作为一种微生物工具,用于靶向运输投放功能蛋白。同时,由于自裂解系统的存在,可解决因工程菌逃逸带来的潜在威胁;pBBR1MCS-6载体中携带的是铜绿假单胞菌自身的三氯生抗性基因,因此不会造成因质粒丢失而带来的抗性基因的水平转移问题;工程菌本身丧失了形成生物被膜的能力、缺失了Ⅲ型分泌系统(T3SS)的功能,所以工程菌PAO1102在应用过程中安全性有了很大的保障,但是铜绿假单胞菌毕竟是一种病原菌,对其改造使用仍需经过后续严格的宿主安全性评价。另外,Pf4丝状噬菌体是铜绿假单胞菌PAO1的专性噬菌体,不侵染其他铜绿假单胞菌菌株,如PA14,故通过Pf4丝状噬菌体介导工程菌裂解投放功能蛋白的方式,在应用上还具有一定的局限性。因此,选择其它宿主范围比较广的噬菌体作为改造对象则更有应用价值。
人们一直设法用药物消除细菌来治疗疾病,现在越来越多的研究发现,细菌并不是只会导致疾病,也是身体健康的重要因素,这方面研究比较热门的就是肠道菌群。在对细菌的研究中,还有一个重要方向比较违反直觉,就是对细菌进行基因改造,让细菌变成可以治疗疾病的工具[41]。使用改造的细菌作为治疗传递系统,选择性地在体内释放有效治疗成分正是肿瘤细菌疗法的主要原理,而通过与合成生物学的结合则大大提高了该疗法的适用度和可操作性[42-43]。有研究报道,一种工程化的大肠杆菌可以在肿瘤微环境中特异性裂解、释放一种纳米拮抗剂CD47nb,它是专门阻断在多种肿瘤细胞中过度产生的抗吞噬受体CD47,以刺激抗肿瘤免疫和促进肿瘤消退[42]。由于肿瘤微环境菌群中存在大量的铜绿假单胞菌[44],因此将来可在工程菌PAO1102中表达合成一些对肿瘤细胞有抑制或者杀伤作用的蛋白(如铜绿假单胞菌的天青蛋白(azurin),它可以直接进入黑素瘤UISO-Mel-2细胞并诱导其凋亡[43]),并利用工程菌自身的主动裂解或肿瘤微环境中野生铜绿假单胞菌产生的Pf4丝状噬菌体介导的被动裂解在肿瘤组织中定向投放抗肿瘤蛋白,以期实现用活菌治疗肿瘤的目的。另外,由于蛋白类杀菌素和丝状噬菌体广泛存在于各种革兰氏阴性菌和部分革兰氏阳性菌中[12, 45],这使本研究靶向投放蛋白的工程菌构建思路和方法可推广应用到其他细菌。
致谢
本研究工作参加了2019年国际基因工程机器大赛(iGEM)并获大赛银奖,感谢在此期间参赛队伍每一位成员的辛勤付出以及延安大学各部门对本研究的大力支持。
| [1] | Endy D. Foundations for engineering biology. Nature, 2005, 438(7067): 449-453. DOI:10.1038/nature04342 |
| [2] | Levskaya A, Chevalier AA, Tabor JJ, Simpson ZB, Lavery LA, Levy M, Davidson EA, Scouras A, Ellington AD, Marcotte EM, Voigt CA. Engineering Escherichia coli to see light. Nature, 2005, 438(7067): 441-442. DOI:10.1038/nature04405 |
| [3] | Elowitz MB, Leibler S. A synthetic oscillatory network of transcriptional regulators. Nature, 2000, 403(6767): 335-338. DOI:10.1038/35002125 |
| [4] | Martin VJJ, Pitera DJ, Withers ST, Newman JD, Keasling JD. Engineering a mevalonate pathway in Escherichia coli for production of terpenoids. Nature Biotechnology, 2003, 21(7): 796-802. DOI:10.1038/nbt833 |
| [5] |
Dong YX, Zeng ZY, Xia L, Liu CL. Bacterial anti-cancer therapy in the era of synthetic biology. Chinese Bulletin of Life Sciences, 2019, 31(4): 332-342.
(in Chinese) 董宇轩, 曾正阳, 夏霖, 刘陈立. 肿瘤细菌疗法迎来合成生物学时代. 生命科学, 2019, 31(4): 332-342. |
| [6] |
Hong YD, Hu N, Bian BX, Song ZY, Wu FL. Research advances in the mechanisms for anti-tumor effect of Pseudomonas aeruginosa injection. Chinese Journal of Modern Applied Pharmacy, 2019, 36(2): 256-259.
(in Chinese) 洪义东, 胡楠, 卞宝祥, 宋子琰, 吴风雷. 铜绿假单胞菌注射液抗肿瘤作用及其相关机制的研究进展. 中国现代应用药学, 2019, 36(2): 256-259. |
| [7] | Epaulard O, Toussaint B, Quenee L, Derouazi M, Bosco N, Villiers C, Berre RL, Guery B, Filopon D, Crombez L, Marche PN, Polack B. Anti-tumor immunotherapy via antigen delivery from a live attenuated genetically engineered Pseudomonas aeruginosa type Ⅲ secretion system-based vector. Molecular Therapy, 2006, 14(5): 656-661. DOI:10.1016/j.ymthe.2006.06.011 |
| [8] | Reed B, Chen R. Biotechnological applications of bacterial protein secretion: from therapeutics to biofuel production. Research in Microbiology, 2013, 164(6): 675-682. DOI:10.1016/j.resmic.2013.03.006 |
| [9] | Ni Y, Chen R. Extracellular recombinant protein production from Escherichia coli. Biotechnology Letters, 2009, 31(11): 1661. DOI:10.1007/s10529-009-0077-3 |
| [10] | Hwang IY, Koh E, Wong A, March JC, Bentley WE, Lee YS, Chang MW. Engineered probiotic Escherichia coli can eliminate and prevent Pseudomonas aeruginosa gut infection in animal models. Nature Communications, 2017, 8(1): 15028. DOI:10.1038/ncomms15028 |
| [11] | Hanlon GW. Bacteriophages: an appraisal of their role in the treatment of bacterial infections. International Journal of Antimicrobial Agents, 2007, 30(2): 118-128. DOI:10.1016/j.ijantimicag.2007.04.006 |
| [12] | Mai-Prochnow A, Hui JGK, Kjelleberg S, Rakonjac J, McDougald D, Rice SA. Big things in small packages: the genetics of filamentous phage and effects on fitness of their host. FEMS Microbiology Reviews, 2015, 39(4): 465-487. DOI:10.1093/femsre/fuu007 |
| [13] | Li YM, Liu XX, Tang KH, Wang PX, Zeng ZS, Guo YX, Wang XX. Excisionase in Pf filamentous prophage controls lysis-lysogeny decision-making in Pseudomonas aeruginosa. Molecular Microbiology, 2018, 111(2): 495-513. |
| [14] | Matsui H, Sano Y, Ishihara H, Shinomiya T. Regulation of pyocin genes in Pseudomonas aeruginosa by positive (prtN) and negative (prtR) regulatory genes. Journal of Bacteriology, 1993, 175(5): 1257-1263. DOI:10.1128/jb.175.5.1257-1263.1993 |
| [15] | Saeidi N, Wong CK, Lo TM, Nguyen HX, Ling H, Leong SSJ, Poh CL, Chang MW. Engineering microbes to sense and eradicate Pseudomonas aeruginosa, a human pathogen. Molecular Systems Biology, 2011, 7(1): 521-521. DOI:10.1038/msb.2011.55 |
| [16] | Baker P, Hill PJ, Snarr BD, Alnabelseya N, Pestrak MJ, Lee MJ, Jennings LK, Tam J, Melnyk RA, Parsek MR, Sheppard DC, Wozniak DJ, Howell LP. Exopolysaccharide biosynthetic glycoside hydrolases can be utilized to disrupt and prevent Pseudomonas aeruginosa biofilms. Science Advances, 2016, 2(5): e1501632. DOI:10.1126/sciadv.1501632 |
| [17] | Ma LY, Conover M, Lu HP, Parsek MR, Bayles K, Wozniak DJ. Assembly and development of the Pseudomonas aeruginosa biofilm matrix. PLoS Pathogens, 2009, 5(3): e1000354. DOI:10.1371/journal.ppat.1000354 |
| [18] | Yu S, Su TT, Wu HJ, Liu SH, Wang D, Zhao TH, Jin ZJ, Du WB, Zhu MJ, Chua SL, Yang L, Zhu DY, Gu LC, Ma LZ. PslG, a self-produced glycosyl hydrolase, triggers biofilm disassembly by disrupting exopolysaccharide matrix. Cell Research, 2015, 25(12): 1352-1367. DOI:10.1038/cr.2015.129 |
| [19] | Zhang JC, He J, Zhai CH, Ma LZ, Gu LC, Zhao K. Effects of PslG on the surface movement of Pseudomonas aeruginosa. Applied and Environmental Microbiology, 2018, 84(13): e00219-18. |
| [20] | Zhao TH, Zhang YH, Wu HJ, Wang D, Chen YH, Zhu MJ, Ma LZ. Extracellular aminopeptidase modulates biofilm development of Pseudomonas aeruginosa by affecting matrix exopolysaccharide and bacterial cell death. Environmental Microbiology Reports, 2018, 10(5): 583-593. DOI:10.1111/1758-2229.12682 |
| [21] | Wang SW, Liu X, Liu HS, Zhang L, Guo Y, Yu S, Wozniak DJ, Ma LZ. The exopolysaccharide Psl-eDNA interaction enables the formation of a biofilm skeleton in Pseudomonas aeruginosa. Environmental Microbiology Reports, 2015, 7(2): 330-340. DOI:10.1111/1758-2229.12252 |
| [22] | Costerton JW, Stewart PS, Greenberg EP. Bacterial biofilms: a common cause of persistent infections. Science, 1999, 284(5418): 1318-1322. DOI:10.1126/science.284.5418.1318 |
| [23] |
Lin JS, Niu YT, Wang ST, Wang GF, Tian Y, Zhang H, Zhu XF, Si QP, Cheng JL, Ai YN, Zhao WJ, Zhang XQ. Characterization of zinc ion uptake mediated by cntRLMN operon in Pseudomonas aeruginosa. Acta Microbiologica Sinica, 2020, 60(4): 789-804.
(in Chinese) 林金水, 牛艳婷, 王帅涛, 王贵锋, 田野, 张恒, 朱旭飞, 司青坡, 成娟丽, 艾亚楠, 赵文静, 张向前. 铜绿假单胞菌cntRLMN操纵子介导锌离子摄取的功能鉴定. 微生物学报, 2020, 60(4): 789-804. |
| [24] | Schäfer A, Tauch A, Jäger W, Kalinowski J, Thierbach G, Pühler A. Small mobilizable multi-purpose cloning vectors derived from the Escherichia coli plasmids pK18 and pK19:selection of defined deletions in the chromosome of Corynebacterium glutamicum. Gene, 1994, 145(1): 69-73. DOI:10.1016/0378-1119(94)90324-7 |
| [25] | Dennis JJ, Zylstra GJ. Plasposons: modular self-cloning minitransposon derivatives for rapid genetic analysis of gram-negative bacterial genomes. Applied and Environmental Microbiology, 1998, 64(7): 2710-2715. DOI:10.1128/AEM.64.7.2710-2715.1998 |
| [26] | Kovach ME, Elzer PH, Hill DS, Robertson GT, Farris MA, Martin Roop II R, Peterson KM. Four new derivatives of the broad-host-range cloning vector pBBR1MCS, carrying different antibiotic-resistance cassettes. Gene, 1995, 166(1): 175-176. DOI:10.1016/0378-1119(95)00584-1 |
| [27] | Heeb S, Blumer C, Haas D. Regulatory RNA as mediator in GacA/RsmA-dependent global control of exoproduct formation in Pseudomonas fluorescens CHA0. Journal of Bacteriology, 2002, 184(4): 1046-1056. DOI:10.1128/jb.184.4.1046-1056.2002 |
| [28] | Becher A, Schweizer HP. Integration-proficient Pseudomonas aeruginosa vectors for isolation of single-copy chromosomal lacZ and lux gene fusions. Biotechniques, 2000, 29(5): 948-950. DOI:10.2144/00295bm04 |
| [29] | Choi KH, Schweizer HP. mini-Tn7 insertion in bacteria with single attTn7 sites: example Pseudomonas aeruginosa. Nature Protocols, 2006, 1(1): 153-161. DOI:10.1038/nprot.2006.24 |
| [30] | Choi KH, Mima T, Casart Y, Rholl D, Kumar A, Beacham IR, Schweizer HP. Genetic tools for select-agent-compliant manipulation of Burkholderia pseudomallei. Applied and Environmental Microbiology, 2008, 74(4): 1064-1075. DOI:10.1128/AEM.02430-07 |
| [31] | Hoang TT, Karkhoff-Schweizer RR, Kutchma AJ, Schweizer HP. A broad-host-range Flp-FRT recombination system for site-specific excision of chromosomally-located DNA sequences: application for isolation of unmarked Pseudomonas aeruginosa mutants. Gene, 1998, 212(1): 77-86. DOI:10.1016/S0378-1119(98)00130-9 |
| [32] | Huber B, Riedel K, Köthe M, Givskov M, Molin S, Eberl L. Genetic analysis of functions involved in the late stages of biofilm development in Burkholderia cepacia H111. Molecular Microbiology, 2002, 46(2): 411-426. DOI:10.1046/j.1365-2958.2002.03182.x |
| [33] | Lin JS, Cheng JL, Chen KQ, Guo CH, Zhang WP, Yang X, Ding W, Ma L, Wang Y, Shen XH. The icmF3 locus is involved in multiple adaptation- and virulence-related characteristics in Pseudomonas aeruginosa PAO1. Frontiers in Cellular and Infection Microbiology, 2015, 5: 70. |
| [34] | Miller JH. A short course in bacterial genetics: a laboratory manual and handbook for Escherichia coli and related bacteria. Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press, 1992. |
| [35] | Lin JS, Cheng JL, Wang Y, Shen XH. The Pseudomonas quinolone signal (PQS): not just for quorum sensing anymore. Frontiers in Cellular and Infection Microbiology, 2018, 8: 230. DOI:10.3389/fcimb.2018.00230 |
| [36] | Schuster M, Phoebe Lostroh C, Ogi T, Greenberg EP. Identification, timing, and signal specificity of Pseudomonas aeruginosa quorum-controlled genes: a transcriptome analysis. Journal of Bacteriology, 2003, 185(7): 2066-2079. DOI:10.1128/JB.185.7.2066-2079.2003 |
| [37] | Zhu L, Lin JS, Ma JC, Cronan JE, Wang HH. Triclosan resistance of Pseudomonas aeruginosa PAO1 is due to FabV, a triclosan-resistant enoyl-acyl carrier protein reductase. Antimicrobial Agents and Chemotherapy, 2010, 54(2): 689-698. DOI:10.1128/AAC.01152-09 |
| [38] | Jackson KD, Starkey M, Kremer S, Parsek MR, Wozniak DJ. Identification of psl, a locus encoding a potential exopolysaccharide that is essential for Pseudomonas aeruginosa PAO1 biofilm formation. Journal of Bacteriology, 2004, 186(14): 4466-4475. DOI:10.1128/JB.186.14.4466-4475.2004 |
| [39] | Pastor A, Chabert J, Louwagie M, Garin J, Attree I. PscF is a major component of the Pseudomonas aeruginosa type Ⅲ secretion needle. FEMS Microbiology Letters, 2005, 253(1): 95-101. DOI:10.1016/j.femsle.2005.09.028 |
| [40] | Ro DK, Paradise EM, Ouellet M, Fisher KJ, Newman KL, Ndungu JM, Ho KA, Eachus RA, Ham TS, Kirby J, Chang MCY, Withers ST, Shiba Y, Sarpong R, Keasling JD. Production of the antimalarial drug precursor artemisinic acid in engineered yeast. Nature, 2006, 440(7086): 940-943. DOI:10.1038/nature04640 |
| [41] | Reardon S. Genetically modified bacteria enlisted in fight against disease. Nature, 2018, 558(7711): 497-498. DOI:10.1038/d41586-018-05476-4 |
| [42] | Chowdhury S, Castro S, Coker C, Hinchliffe TE, Arpaia N, Danino T. Programmable bacteria induce durable tumor regression and systemic antitumor immunity. Nature Medicine, 2019, 25(7): 1057-1063. DOI:10.1038/s41591-019-0498-z |
| [43] | Yamada T, Goto M, Punj V, Zaborina O, Mei LC, Kimbara K, Majumdar D, Cunningham E, Gupta TKD, Chakrabarty AM. Bacterial redox protein azurin, tumor suppressor protein p53, and regression of cancer. Proceeding of National Academy of Sciences of the United States of America, 2002, 99(22): 14098-14103. DOI:10.1073/pnas.222539699 |
| [44] | Chiba A, Bawaneh A, Velazquez C, Clear KYJ, Wilson AS, Howard-McNatt M, Levine EA, Levi-Polyachenko N, Yates-Alston SA, Diggle SP, Soto-Pantoja DR, Cook KL. Neoadjuvant chemotherapy shifts breast tumor microbiota populations to regulate drug responsiveness and the development of metastasis. Molecular Cancer Research, 2020, 18(1): 130-139. DOI:10.1158/1541-7786.MCR-19-0451 |
| [45] | Behrens HM, Six A, Walker D, Kleanthous C. The therapeutic potential of bacteriocins as protein antibiotics. Emerging Topics in Life Sciences, 2017, 1(1): 65-74. DOI:10.1042/ETLS20160016 |
 2021, Vol. 61
2021, Vol. 61




