中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 孙延瑜, 宋增磊, 刘鹏远, 张海坤, 胡晓珂. 2021
- Yanyu Sun, Zenglei Song, Pengyuan Liu, Haikun Zhang, Xiaoke Hu. 2021
- 威海天鹅湖大叶藻(Zostera marina)与日本鳗草(Zostera japonica)根际微生物群落结构及其驱动机制
- Rhizosphere microbial community structure and driving mechanism of Zostera marina and Zostera japonica in Swan Lake, Weihai
- 微生物学报, 61(9): 2675-2692
- Acta Microbiologica Sinica, 61(9): 2675-2692
-
文章历史
- 收稿日期:2020-09-27
- 修回日期:2021-01-07
- 网络出版日期:2021-02-24
2. 青岛海洋科学与技术试点国家实验室, 海洋生物学与生物技术功能实验室, 山东 青岛 266237;
3. 中国科学院大学, 北京 100049
2. Laboratory for Marine Biology and Biotechnology, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266237, Shandong Province, China;
3. University of Chinese Academy of Sciences, Beijing 100049, China
海草属于大型沉水被子植物,具有高等植物的一般特征,能完全适应水中生活[1]。海草通过长期进化,在结构与生理特性上都成功适应了海洋环境,是唯一可以在海水中完成开花、结实以及萌发这一生长发育过程的被子植物。海草的进化包括完全丢失控制气孔、紫外保护和红外感知色素基因;此外,海草为了适应海洋环境中的高盐度,也进化出与大型海藻相似的具有低甲基化果胶、硫酸化半乳聚糖和阴电性的细胞壁;这些都有利于海草细胞的离子稳态、养分摄取以及O2/CO2的交换[2]。
海草多生长于潮下带浅海区域至潮间带,形成一个独特的生态系统——海草床生态系统,与红树林以及珊瑚礁生态系统并称为3大典型的海洋生态系统。虽然海草床在全球分布面积较少,却具有极高的初级生产力,并为海洋环境提供了许多重要的生态系统服务。首先,海草生态系统是生物圈中碳储存的关键场所,能够捕获和储存多达19.9 Pg的有机碳,从而起到蓝色碳汇的作用[3]。其次,海草是许多经济物种的庇护所、育幼场和食物来源,通过碎屑循环和直接食草成为海草床食物网的基础。仅一英亩海草就可以养活多达5000万无脊椎动物和40000条鱼类[4]。第三,海草可以抵御海浪侵蚀海岸,保持沉积物稳定。海草叶子起到过滤作用,能够捕获悬浮的颗粒和细小沉积物,从而净化水体[5]。
微生物是海草床生态系统中重要的组成部分,在生态系统的物质、元素循环和能量流动中发挥重要作用。微生物附着在海草叶片上,或者生活于海草根际,参与海草床生物地球化学循环[6]。海草为多种附生微生物提供了基质,其中包括自养生物(硅藻、蓝藻)[7]和异养生物(微生物、无脊椎动物)[8]。海草的地上和地下组织都存在丰富的微生物,叶片的微生物量为1×106– 8.5×106细胞/cm−2,根际和根茎组织微生物量为105–106细胞/cm2[9]。此外,海草附生微生物存在核心微生物群,与周围环境中的微生物群落存在明显差异[10–12]。一方面,海草的叶子和根分泌出可被附生微生物代谢的营养物质,包括氨基酸、维生素和铁等[13];另一方面,特定微生物的代谢活动可以对海草的生长、健康和生产力产生重大影响。例如,长期缺氧沉积物中的硫化物氧化或营养贫乏生态系统中的氮固定,无疑可以提高海草的适应能力[14–16];海草附生微生物可产生IAA和细胞分裂素等植物激素,促进海草生长发育[17]。微生物群落在维持海草健康中起着多方面的作用,阐明宿主-微生物相互作用可以为海草床的恢复提供指导。
海草共附生微生物是高度动态的群落。在微观尺度上,微生物群落的变化可由宿主的营养盐供给以及氧气可利用性所驱动[18–19]。共附生微生物群落在时间和空间尺度上的变化及其驱动机制的研究,往往存在争议。有研究表明,共附生微生物的稳定性和多样性是随着时间和空间而变化的[18–20],但也有文献证明,微生物群落高度依赖宿主的生理和环境条件[21–22]。微生物群落分布模式的争议使得我们无法对宿主相关微生物群落的稳定性或可变性作出概括。在本研究中,大叶藻与日本鳗草同时生长在威海天鹅湖中,是共附生微生物分布模式的良好研究对象。相同时间微生物群落差异可以分析不同宿主对微生物群落多样性的影响;而不同海草生长时期下的微生物差异可以探究时间尺度对微生物群落的驱动机制。
海草可以通过无性繁殖和有性繁殖两种方式增殖[23]。无性繁殖是海草床种群补充的主要因素。然而,有性繁殖是保持种群遗传多样性的唯一途径[24]。海草有性繁殖种群补充机制,则受到海草生殖周期(如开花、结果、种子萌发和幼苗存活率)的制约。通过传粉和开花,有性生殖循环产生了与亲代不同的海草种子,种子被埋在沉积物中,从而形成了一个种子库。经过一段时间的休眠和种子萌发,幼苗存活并生长,海草将陆续进入下一个花期,有性繁殖周期继续[25]。有性繁殖将提高海草种群的遗传多样性,提高其对干扰的抗逆性[26]。
威海天鹅湖中海草的有性繁殖在种群补充中发挥了重要作用,当前大多数海草附生微生物研究集中于单一时期,而海草不同生活时期的根际微生物群落结构尚未见报道。基于此,本实验选择于4月(海草幼苗期)、9月(海草成熟期)以及1月(海草衰亡期)对天鹅湖大叶藻和日本鳗草根际沉积物进行样品采集。利用高通量测序手段并结合基因功能预测,探究天鹅湖海草床2种海草不同生活时期的根际与非草区的微生物多样性、菌群结构及分布。拟探究的科学问题为:(1) 在海草床根际沉积物环境中驱动微生物群落变化的机制是什么;(2) 是否存在海草核心微生物组,其功能是什么;(3) 海草生命周期早期阶段的微生物是否影响成熟海草的性能和健康。本研究通过解析根际效应与生活时期对海草附生微生物群落结构的影响机制,结合核心微生物组以及关键功能微生物,提供管理或操控微生物的群落和生态功能,从而保护和修复海草床的思路和基础。
1 材料和方法 1.1 样品采集于2019年4月17日,9月7日以及2020年1月8日在山东威海荣成天鹅湖(37°20′00′′– 37°22′00′′N,122°32′45′′–122°35′30′′E)采集日本鳗草(Zostera japonica)、大叶藻(Zostera marina)根际沉积物以及无海草生长的表层沉积物。天鹅湖是典型的泻湖(4.8 km2),水深度相对较浅,平均潮差约为0.9 m[27]。天鹅湖是中国北方典型的海草床,在20世纪70年代初发现了大叶藻和日本鳗草。以大叶藻为优势海草种,覆盖面积约为2.0 km2,主要生长在天鹅湖中部,而日本鳗草生活在浅水或潮间带区域[28]。在低潮时随机采集大叶藻和日本鳗草,对所选海草的根进行振荡以去除松散沉积物,并收集附着在根上的沉积物。海草退化区表层沉积物样本采集于距海草床约5 m的无植被地区。将采集到的沉积物样品置于冰上带回实验室,用于提取环境DNA的样品于–80 ℃中储存;而用于测定理化因子的沉积物在冷冻干燥机中处理72 h。
1.2 沉积物理化性质检测海水温度和盐度通过手持YSI水质分析仪原位测定。沉积物中的有机碳氮(TOC、TON):冷冻干燥后的沉积物样品磨至100目,取1–2 g,放到玻璃离心管,加盐酸(1 mol/L),先滴加,然后加过量,反应8 h以上,期间要振荡或超声,离心,加新的盐酸,待样品中的无机碳反应充分后,用超纯水清洗,离心,倒掉上清液,继续用超纯水清洗、离心,重复2–3遍,冷冻干燥,包样后在元素分析仪(Elementar,德国)测定含量。
1.3 环境DNA提取及高通量测序沉积物样品DNA采用PowerSoilTM DNA Isolation Kit试剂盒提取,实验步骤参照试剂盒说明书。使用正向引物515F (5′-GTGCCAGCMGCC GCGG-3′)以及反向引物806R (5′-GGACTACH VGGGTWTCTAAT-3′)扩增细菌16S rRNA基因的V4–V5区,在Illumina Miseq平台进行测序(上海美吉生物医药科技有限公司)。
1.4 数据分析及统计检验使用QIIME (version 1.9.1)对序列进行质控。以97%相似度进行OTU聚类,并根据SILVA数据库进行分类(Release132 http://www.arb-silva.de)。alpha多样性分析,包括群落丰富度参数(Chao、Sobs、ACE),群落多样性参数(Shannon、Simpson)和测序深度指数(Good’s coverage),使用Mothur软件(version v.1.30.1)计算。利用R软件包软件显示样品的微生物分类学结构。采用非度量多维标度(nMDS)排序,以揭示OTUs中微生物群落组成变化的聚类情况。采用Wilcoxon秩和检验分析组间差异,采用fdr法对P值进行多重检验校正。P < 0.05被认为有统计学意义。
2 结果和分析 2.1 天鹅湖海草床环境因子及微生物多样性特征由表 1可知,海草不同生长时期,环境因子有明显变化。在威海天鹅湖海草床开放海域,各个时期样品之间的海水温度与盐度并无差异,但海草根际沉积物总有机碳氮含量大于非草区,且大叶藻根际沉积物的总有机碳氮含量要大于日本鳗草。这主要是由于大叶藻植株大,根系更为发达所致。
| Samples | T/℃ | Salinity/‰ | TOC/% | TON/% |
| Seeding-Un | 6.6±0 | 32.2900±0.0071 | 0.1020±0.0088 | 0.0206±0.0021 |
| Seeding-Zj | 6.6700±0.0471 | 32.3500±0.0492 | 0.1491±0.0753 | 0.0260±0.0101 |
| Seeding-Zm | 6.5700±0.0943 | 32.3200±0.0943 | 0.1788±0.0988 | 0.0272±0.0057 |
| Maturation-Un | 13.4±0 | 33.03±0.01 | 0.4393±0.0311 | 0.0485±0.0092 |
| Maturation-Zj | 13.3700±0.1247 | 32.9700±0.0294 | 0.4799±0.1139 | 0.0527±0.0173 |
| Maturation-Zm | 13.3700±0.1699 | 32.9900±0.0408 | 0.6535±0.0234 | 0.0683±0.0061 |
| Decline-Un | 3.05±0.05 | 30.3650±0.0304 | 0.1560±0.0179 | 0.0444±0.0092 |
| Decline-Zj | 3.1300±0.0471 | 30.3500±0.0216 | 0.1455±0.0048 | 0.0392±0.0042 |
| Decline-Zm | 3.1000±0.0816 | 30.3000±0.0616 | 0.1988±0.0035 | 0.0448±0.0029 |
| Zj: Zostera japonica; Zm: Zostera marina; Un: unvegetated. | ||||
总体而言,沉积物中有机碳氮含量呈现成熟期 > 衰退期 > 幼苗期的趋势。有机物质经过冬季消耗,含量相对匮乏;随着气温回升,海草及藻类旺长,有机质含量增加;秋季海草碎片不断在沉积物中积累,并在冬季腐烂,为微生物提供颗粒性有机碳(POC)如纤维素和半纤维素等。
27个沉积物样品经过测序共得到1765359条有效序列,平均序列长度为253 bp,且原始数据已上传至NCBI数据库,生成的序列号为PRJNA646607。研究环境中微生物的多样性,可以通过α多样性分析反映微生物群落的丰富度和多样性。Sobs指数反映丰富度的实际观测值,Ace指数和Chao指数都能够反映群落丰富度,Shannon指数反映的则是群落多样性,而Coverage指数则反映的是群落覆盖度。
由表 2可知,幼苗期(4月)非草区Sobs指数最高(5187.00±87.05),其次为幼苗期日本鳗草根际沉积物(4786.00±562.66)。同样,Shannon指数最高的样品为幼苗期非草区样品(6.66±0.15),其次为幼苗期日本鳗草根际样品(6.55±0.18)。总体而言,微生物群落丰富度与多样性呈现幼苗期 > 衰退期 > 成熟期的特性,说明在海草成熟期具有特定的微生物类群占据较高的相对丰度。
| Sample/Estimators | Sobs | Shannon | Ace | Chao | Coverage |
| Decline unvegetated sediment | 3857.50±914.30 | 6.44±0.38 | 7412.37±1539.46 | 6150.05±1358.49 | 96.17±0.51% |
| Decline Z. japonica rhizosphere | 4603.67±811.03 | 6.49±0.27 | 5888.08±1121.14 | 7188.09±1116.05 | 96.47±0.39% |
| Decline Z. marina rhizosphere | 4343.67±538.35 | 6.43±0.55 | 8487.03±1602.96 | 6897.94±1057.61 | 95.72±1.83% |
| Seeding unvegetated sediment | 5187.00±87.05 | 6.66±0.15 | 8160.60±671.87 | 7516.57±45.75 | 96.86±0.05% |
| Seeding Z. japonica rhizosphere | 4786.00±562.66 | 6.55±0.18 | 7932.95±1523.00 | 7116.37±875.14 | 97.14±0.48% |
| Seeding Z. marina rhizosphere | 3821.33±452.79 | 6.10±0.23 | 7068.53±485.79 | 5905.02±494.38 | 97.39±0.20% |
| Maturation unvegetated sediment | 4053.50±609.50 | 5.88±0.63 | 7581.34±1113.37 | 6288.61±780.03 | 96.96±0.48% |
| Maturation Z. japonica rhizosphere | 4142.67±108.43 | 5.80±0.47 | 7363.62±819.29 | 6330.69±112.31 | 97.48±0.04% |
| Maturation Z. marina rhizosphere | 3033.67±941.10 | 5.06±1.21 | 5377.58±1992.26 | 4630.62±1546.06 | 98.04±0.61% |
2.2 天鹅湖海草床沉积物微生物群落结构及差异性分析
天鹅湖海草床沉积物微生物群落结构如图 1所示。在门水平上(图 1-A),微生物相对丰度由高到低依次为变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、绿湾菌门(Chloroflexi)、浮霉菌门(Planctomycetes)、放线菌门(Actinobacteria)、厚壁菌门(Firmicutes)以及梭杆菌门(Fusobacteria)。其中较为明显的变化是在成熟期时,大叶藻与日本鳗草根际沉积物中梭杆菌门(Fusobacteria)相对丰度较非草区有大幅增加,而非草区中厚壁菌门(Firmicutes)相对丰度较高。

|
| 图 1 海草床沉积物微生物类群在门(A)科(B)属(C)分类阶元上的相对丰度 Figure 1 Relative abundance of microbial taxa in seagrass bed at phylum (A), family (B), genus (C) levels. M: maturation; S: seedling; D: decline; Zj: Zostera japonica; Zm: Zostera marina; Un: unvegetated. |
在科水平上(图 1-B),微生物相对丰度由高到低依次为脱硫球茎菌科(Desulfobulbaceae)、浮霉菌科(Planctomycetaceae)、黄杆菌科(Flavobacteriaceae)、螺杆菌科(Helicobacteraceae)、厌氧绳菌科(Anaerolineaceae)、脱硫杆菌科(Desulfobacteraceae)以及梭杆菌科(Fusobacteriaceae)。
在属水平上(图 1-C),在所有样品中都占有较高相对丰度的是硫卵菌属(Sulfurovum)以及未分类的脱硫球茎菌科(norank_f_Desulfobulbaceae)和未分类的厌氧绳菌科(norank_f_Anaerolineaceae)。值得注意的是,在海草成熟期时,丙酸菌属(Propionigenium)在海草根际沉积物中占有较大丰度,这一结果与门和科水平上的相对丰度变化是相符的。
利用Wilcoxon秩和检验得到了海草不同生长时期根际沉积物与非草区相对丰度前15的OTU,并标注其分类信息(图 2)。在海草生长的各个时期,微生物群落结构有极大差异。在海草成熟期,丙酸菌属(Propionigenium)、硫卵菌属(Sulfurovum)、芽孢杆菌目(Bacillales)、脱硫球茎菌科(Desulfobulbaceae)、海边杆菌属(Actibacter)以及黄杆菌科(Flavobacteriaceae)占有较大丰度;而在海草幼苗期,脱硫球茎菌科(Desulfobulbaceae)、假单胞菌属(Pseudomonas)、SVa1033、硫卵菌属(Sulfurovum)、需盐海仙菌属(Haliea)占有较大丰度;在海草衰退期,硫卵菌属(Sulfurovum)、脱硫球茎菌科(Desulfobulbaceae)、SVa1033以及脱硫杆菌科(Desulfobacteraceae)具有较高的相对丰度,暗示在海草衰退期微生物驱动的硫循环在海草床具有重要意义。
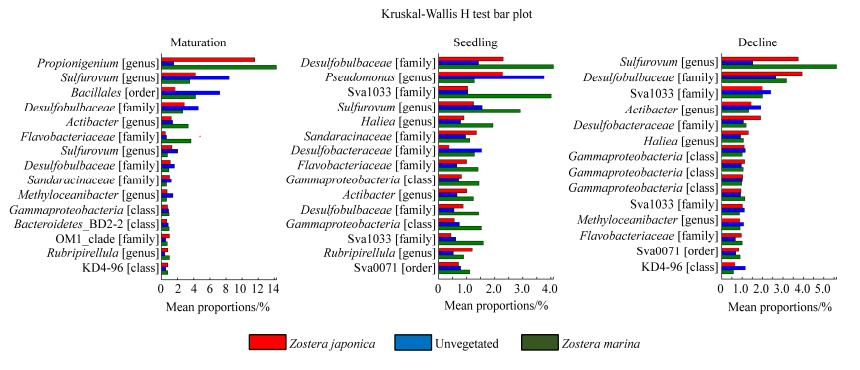
|
| 图 2 海草不同生长时期下微生物相对丰度的组间差异比较 Figure 2 Statistical comparison of the relative abundance of microbiota among unvegetated sediment, Z. marina rhizosphere and Z. japonica rhizosphere groups. |
此外,由图 2可知,微生物群落结构在根际沉积物与非草区有显著差异。在海草成熟期,丙酸菌属(Propionigenium)在大叶藻与日本鳗草根际有明显富集,其相对丰度分别为11.58%和14.26%,而在非草区,其相对丰度仅为1.513%。与之不同的是,硫卵菌属(Sulfurovum)芽孢杆菌目(Bacillales)以及脱硫球茎菌科(Desulfobulbaceae)在非草区相对丰度较根际高,分别为8.41%、7.242%、4.586%。在海草幼苗期,脱硫球茎菌科(Desulfobulbaceae)在海草根际富集,在大叶藻与日本鳗草根际沉积物中,其相对丰度分别为2.299%和4.092%。而假单胞菌属(Pseudomonas)则在非草区相对丰度更高(3.763%)。在海草衰退期时,硫卵菌属(Sulfurovum)的相对丰度在根际较高(大叶藻:5.624%;日本鳗草:3.749%),而在非草区,其相对丰度为1.519%。由此可知,海草床微生物群落结构具有明显的根际效应。
将海草不同生长时期日本鳗草和大叶藻根际沉积物微生物群落结构做两组检验,得到丰度前15的类群及其差异分析结果(图 3)。由检验结果可知,在海草成熟期以及衰退期时,日本鳗草和大叶藻根际沉积物微生物群落极为相近;而在海草幼苗期,两种海草根际沉积物微生物群落结构略有差异,这种差异是由微生物丰度差异造成的。其中,脱硫球茎菌科、Sva1033、硫卵菌属、需盐洒仙菌属和黄杆菌科等都在大叶藻根际沉积物中丰度高,可能与大叶藻的生物量较大和根茎较粗壮有关。总体而言,日本鳗草和大叶藻根际沉积物微生物群落结构无显著性差异(P > 0.05)。

|
| 图 3 日本鳗草和大叶藻根际微生物相对丰度的组间差异比较 Figure 3 Statistical comparison of the relative abundance of microbiota between Z. japonica and Z. marina rhizosphere. A: maturation; B: decline; C: seedling. |
OTU 20239 (硫卵菌属)、OTU 20696 (脱硫球茎菌科)、OTU 3473 (海边杆菌属)、OTU 840 (黄杆菌科)、OTU 462 (γ-变形菌纲)、OTU 19736 (Sandaracinaceae)、OTU 20174 (Sva1033)和OTU 19775 (Sva0071)在海草生长的不同时期都有出现且丰度较高。说明上述类群是海草根际核心微生物类群,在海草根际长时间存在且具有较高丰度,不会随海草生长时期变化而消失。核心微生物群是与宿主相关微生物群系中持久的、功能上必不可少的成员,可能是决定宿主健康以及整个生态系统功能和健康的关键因素。
通过计算环境因子与科水平上丰度为前20的物种之间的Spearman相关系数并对物种层级求平均值的方式进行聚类,将获得的数值矩阵通过Heatmap图展示(图 4),颜色变化表示相关程度大小。结果表明,脱硫杆菌科(Desulfobacteraceae)、厌氧绳菌科(Anaerolineaceae)、Halieaceae以及Sva1033生长条件类似,与温度、盐度和总有机碳氮呈现明显的负相关关系。其中脱硫杆菌科与总有机碳呈现显著的负相关关系(R=–0.53358,P=0.00415)。

|
| 图 4 科水平上的相关性热图 Figure 4 Heatmap correlationship analysis on the family level. *: P≤0.05; **: P≤0.01. |
而梭杆菌科(Fusobacteriaceae)、norank_c_ Bacteroidetes BD2-2、unclassified_o_Bacillales、红杆菌科(Rhodobacteraceae)、螺旋杆菌科(Helicobacteraceae)、酸微菌科(Acidimicrobiaceae)、黄杆菌科(Flavobacteriaceae)以及OM1_clade与温度、盐度和总有机碳呈现明显的正相关关系,与总有机氮相关关系不显著。另外,JTB225_marine_benthic_group与总有机氮呈显著负相关关系(R=–0.442,P=0.2098)。
2.3 海草生长时期与定殖对微生物群落结构解释度分析我们按照不同生长时期(幼苗期-成熟期-衰退期)和海草定殖情况(非草区-日本鳗草根际-大叶藻根际)对样品进行非度量多维尺度排序(non-metric multidimensional scaling,nMDS)分析。由图 5可知,以海草不同生长期对样品进行划分,样品聚类更为明显,P=0.001;而以海草定殖情况作为分组条件,虽然对样本有一定的解释意义,但非草区样本更为分散,P=0.025。

|
| 图 5 基于Bray-Curtis距离的微生物不同分组条件下的nMDS分析 Figure 5 The nMDS analysis of microorganisms under different grouping conditions based on Bray-Curtis distance. |
为了进一步量化分组结果对样品差异的解释度,我们对样品进行了Permanova分析(表 3)。PERMANOVA (permutational multivariate analysis of variance,置换多元方差分析),又称Adonis分析,利用Bray-Curtis距离矩阵对总方差进行分解,分析不同分组因素或环境因子对样品差异的解释度,并使用置换检验对划分的统计学意义进行显著性分析。由结果可知,海草生长时期对样品的解释度最大,R2=0.20335,P=0.002;其次,沉积物中总有机碳(TOC)、海水温度、盐度、以及沉积物总有机氮含量(TON)都对样品有一定的解释度,而以海草定殖情况分组样品解释度最低,R2=0.07416,P=0.548。结合图 5和表 3的结果显示,海草生长时期相较于定殖情况对微生物群落结构解释度更大。
| Characteristics | Sums of Sqs | Mean Sqs | F_Model | R2 | P_value | P_adjust |
| Life history | 0.26360 | 0.13180 | 2.93553 | 0.20335 | 0.002 | 0.0120 |
| TOC | 0.18876 | 0.18876 | 4.09049 | 0.14562 | 0.006 | 0.0132 |
| Temperature | 0.17038 | 0.17038 | 3.63201 | 0.13144 | 0.008 | 0.0132 |
| Salinity | 0.15550 | 0.15550 | 3.27154 | 0.11996 | 0.010 | 0.0132 |
| TON | 0.15895 | 0.15895 | 3.35425 | 0.12262 | 0.011 | 0.0132 |
| Type | 0.09613 | 0.04807 | 0.92119 | 0.07416 | 0.548 | 0.5480 |
2.4 海草不同生长时期标志类群以及功能预测分析
除了确定微生物种类和多样性之外,比较微生物群落的另一个目标是确定样本中的特殊群落。LEfSe分析可用来估算每个菌种丰度对差异贡献度的大小,从而找出每组中的优势菌种及每组间具有显著性差异的菌群。LDA判别柱形图统计多组中有显著作用的微生物类群,通过LDA分析(线性回归分析)获得的LDA分值,LDA分值越大,代表物种丰度对差异效果影响越大。本文选择LDA分值大于3.5的标志类群进行讨论。
由LEfSe分析可知(图 6),在海草幼苗期时,γ-变形菌纲(Gammaproteobacteria)、脱硫单胞菌目(Desulfuromonadales)、假单胞菌目(Pseudomonadales)、脱硫球茎菌属(Desulfobulbus)等是具有重要意义的标志类群。厚壁菌门(Firmicutes)、芽孢杆菌目(Bacillales)、梭菌纲(Clostridia)、弧菌科(Vibrioaceae)、发光杆菌属(Photobacterium)、微小杆菌属(Exiguobacterium)等是海草成熟期时的标志类群。此外,在海草衰退期,δ-变形菌纲(Deltaproteobacteria)、脱硫杆菌科(Desulfobacteraceae)、Sva0081_sediment_group、酸杆菌门(Acidobacteria)以及Phycisphaerae等是具有显著性差异的菌群。
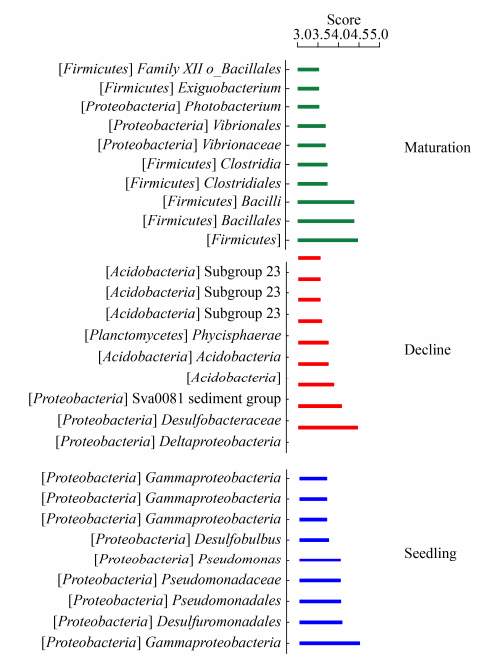
|
| 图 6 海草不同生长时期的沉积物LEfSe分析 Figure 6 LEfSe analysis of sediment in different growth periods of seagrass. |
对不同时期海草床沉积物样品进行PICRUSt2功能预测(图 7),结果表明各功能基因在海草不同生长时期所呈现的趋势一致,但丰度上呈现出幼苗期 > 成熟期 > 衰退期的结果。其中包括氨基酸合成以及碳代谢等的代谢通路以及ABC转运蛋白等的膜转运通路,此外,信号转导以及能量代谢方面也呈现相同的趋势。由图 7结果可知,在海草幼苗期,微生物代谢更为活跃,而在成熟期和衰退期,微生物代谢放缓,与海草生长史相吻合。
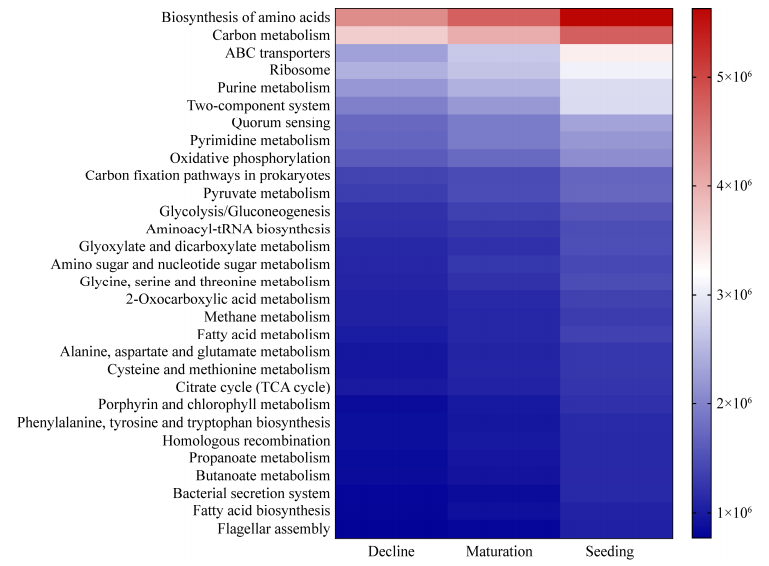
|
| 图 7 海草不同生长时期的微生物PICRUSt2功能预测分析 Figure 7 PICRUSt2 function prediction analysis of microbe in different growth stages of seagrass, the ordinate is the function names of KEGG pathway level 3, and the color gradient is used to show the function abundance. |
3 讨论
威海天鹅湖海草的生长史以及种群特征前人已有研究,其中大叶藻研究较多。研究表明,有性生殖在威海天鹅湖海草生长中起着相对重要的作用。天鹅湖大叶藻种子在3月即开始萌发,但萌发量较少,种苗密度较低;4月种子大量萌发,种苗密度不断升高,并达到峰值,为(481.77±303.42)株/m2[29]。种子库最大密度为552.21±204.94种子/m2 (10月)。天鹅湖的日本鳗草生长也表现出强烈的季节性变化,海草枝密度在8月份达到峰值,为9880±2786枝/m2[28]。
植物根际是受植物根系分泌的化学物质如溶解有机碳直接影响的狭窄土壤区域(1–2 mm),分布有数目庞大的微生物,并与植物间有密切的联系[30]。根际微生物在海草的生长发育过程中起着重要的作用。首先,海草根系存在好氧与厌氧固氮微生物,显著促进海草床沉积物和海草根系的氮固定[31]。其次,海草叶片和根系内生菌群可产生IAA和细胞分裂素等植物激素,促进海草生长[32]。此外,海草根际的硫氧化细菌能够加速硫化物氧化的过程,从而减轻硫化物对海草的毒害作用[33–34]。海草根际菌群也可产生氧化酶保护自身和海草[35],某些内生菌也能够合成抗菌、抗病毒化合物以提高海草的抗逆性[36]。
一般来说,海草的叶片和根际具有核心附生微生物,与周围环境中的微生物存在显著差异。Cúcio发现不同区域海草(Z. marina、Z. noltii和Cymodocea nodosa)的根际微生物群落与周围非根际沉积物的群落结构显著不同,且同一区域内不同海草种类的根际微生物没有显著差异,但不同地理位置的海草根际微生物差异较大[22]。Fahimipour等[18]研究发现,大叶藻根系微生物群落与沉积物群落有很大差异的原因是与沉积物相比根际沉积物中有高丰度的硫氧化菌(硫单胞菌属,Sulfurimonas)。海草根际的核心微生物群主要为参与硫循环的细菌,硫循环相关过程在海草生态系统中具有重要作用[5, 14, 22]。
在本研究中,不同生长时期下的大叶藻与日本鳗草存在不同的根际富集效应。在海草成熟期,丙酸菌属(Propionigenium)在海草根际有明显富集,其相对丰度分别为11.58%和14.26%。在海草幼苗期,脱硫球茎菌科(Desulfobulbaceae)在海草根际富集,在大叶藻与日本鳗草根际沉积物中,其相对丰度分别为2.299%和4.092%。在海草衰退期时,硫卵菌属(Sulfurovum)的相对丰度在根际较高(大叶藻:5.624%;日本鳗草:3.749%),而在非草区,其相对丰度为1.519%。由此可知,海草床微生物群落结构具有明显的根际效应,且大叶藻与日本鳗草根际富集的微生物种类一致,说明在同一区域海草根际微生物群落结构与宿主种类没有显著相关,与前人研究结果一致[22]。
迄今为止,丙酸菌属(Propionigenium)由2个物种组成,即P. modestum和P. maris。P. modestum最初是从意大利威尼斯格兰德运河中缺氧沉积物样本中分离得到的,其丰度达到100个细胞/mL沉积物[37]。此外,P. maris也从海洋沉积物中分离[38],缺氧的海洋沉积物是该菌株的典型栖息地。近年来,也有文献报道在中国北方渤海的沉积物样品中也发现了丙酸菌属[39-41],该属是严格的厌氧菌,可以通过琥珀酸脱羧为丙酸提供生长所需能源[42]。Hasler-Sheetal等[43]研究了Z. marina对水体缺氧的响应、适应和耐受机制,结果表明琥珀酸在缺氧和黑暗条件下积累。海草根际具有解剖结构适应海洋环境,但其仍定期暴露于夜间以及缺氧环境中。虽然目前还没有海草根际沉积物丙酸菌属富集的详细报道,但我们推测这是由于缺氧时琥珀酸积累造成的。丙酸菌属有可能作为一种有益细菌来缓解根际有机酸的压力。此外,也有报道称丙酸菌属与泥滩沉积物中大型底栖生物的生物扰动有关[39]。
脱硫球茎菌科(Desulfobulbaceae)与硫卵菌属(Sulfurovum)都是参与硫循环的功能微生物[44]。在缺氧的沉积物环境中,硫酸盐的还原是有机质矿化的最主要终端过程。硫酸盐还原菌(sulfate reducing bacteria,SRB)以有机物为电子供体、以SO42–为末端电子受体进行有机物的矿化作用;其产物H2S被硫氧化菌(sulfur oxidizing bacteria,SOB)利用进行碳元素的固定作用[45]。同时,硫酸盐还原过程耦合甲烷厌氧氧化,促进碳循环的代谢。微生物中很多硫酸盐还原菌的还原过程与生物固氮反应有着密切的联系。海水中具有高浓度的硫酸盐,硫酸盐还原反应产生的能量可以部分满足生物固氮途径中的能量需求。大叶藻生态系统中硫酸盐还原反应抑制实验发现该系统的固氮效率在抑制实验后下降了80%左右;同时对从海草沉积物中分离出来的所有硫酸盐还原菌进行固氮活性测定,结果表明群落中超过60%的菌群都具有固氮能力[46]。海草幼苗期时脱硫球茎菌科在根际沉积物微生物中的高丰度可能正是由于其固氮功能,能够满足海草生长所需要的大量氮源。
δ-变形菌纲(Deltaproteobacteria)含有大部分已知硫酸盐还原菌属和其他生理功能的厌氧菌,而γ-变形菌纲(Gammaproteobacteria)是许多海草床沉积物中占主要丰度的微生物群体[47]。绿弯菌门(Chloroflexi)在有机质丰富的土壤和沉积物生境中通常具有较高丰度,其中的厌氧绳菌纲(Anaerolineae)是严格厌氧菌,具有降解纤维素的功能[48]。拟杆菌门(Bacteroidetes)中的黄杆菌科(Flavobacteriaceae)是重要的海洋异养菌类群,在海水食物链和碳循环中扮演重要角色,能够降解大分子有机质如纤维素、木聚糖和几丁质等[49–50]。
从图 5的nMDS分析以及置换多元方差分析(表 3)中可知,海草微生物群落结构受到海草生长时期的影响更为明显。本课题组在研究黄渤海地区(东营、威海、大连)日本鳗草根际微生物群落结构的时空分布时发现,从季节上看,各细菌类群随季节变化程度显著不同,造成群落丰度上的季节性差异。通过对比环境因子有机碳、氮含量,作者猜测这可能是因为在不同季节外界环境发生变化,造成温度以及沉积物有机质的变化,进而使得微生物群落结构在不同时期呈现出不同的多样性特征[51]。
综上所述,天鹅湖海草床沉积物微生物群落结构受到海草生长时期以及海草定殖的影响,且海草生长时期影响更为显著。在海草幼苗期、成熟期以及衰退期时,脱硫球茎菌科、丙酸菌属以及硫卵菌属分别在海草根际沉积物微生物中占据高丰度,是各时期的代表菌属。核心微生物组是海草共附生微生物群系中持久的、功能上必不可少的成员,硫卵菌属、脱硫球茎菌科、海边杆菌属、黄杆菌科、Sandaracinaceae、Sva1033、Sva0071等在海草生长的不同时期都有出现且丰度较高。在大叶藻以及日本鳗草根际,参与硫循环的微生物占据相对较高丰度,说明参与硫循环微生物对海草生长以及维持海草根际微环境具有重要意义。
| [1] |
Wang SM, Cui YN, Liu JX, Xia ZR. Research progress on seagrass and seagrass ecosystems. Acta Prataculturae Sinica, 2016, 25(11): 149-159.
(in Chinese) 王锁民, 崔彦农, 刘金祥, 夏曾润. 海草及海草场生态系统研究进展. 草业学报, 2016, 25(11): 149-159. DOI:10.11686/cyxb2016025 |
| [2] | Olsen JL, Rouzé P, Verhelst B, Lin YC, Bayer T, Collen J, Dattolo E, De Paoli E, Dittami S, Maumus F, Michel G, Kersting A, Lauritano C, Lohaus R, Töpel M, Tonon T, Vanneste K, Amirebrahimi M, Brakel J, Boström C, Chovatia M, Grimwood J, Jenkins WJ, Jueterbock A, Mraz A, Stam TW, Tice H, Bornberg-Bauer E, Green PJ, Pearson GA, Procaccini G, Duarte CM, Schmutz J, Reusch TBH, Van De Peer Y. The genome of the seagrass Zostera marina reveals angiosperm adaptation to the sea. Nature, 2016, 530(7590): 331-335. DOI:10.1038/nature16548 |
| [3] | Fourqurean JW, Duarte CM, Kennedy H, Marbà N, Holmer M, Mateo MA, Apostolaki ET, Kendrick GA, Krause-Jensen D, McGlathery KJ, Serrano O. Seagrass ecosystems as a globally significant carbon stock. Nature Geoscience, 2012, 5(7): 505-509. DOI:10.1038/ngeo1477 |
| [4] | Ugarelli K, Chakrabarti S, Laas P, Stingl U. The seagrass holobiont and its microbiome. Microorganisms, 2017, 5(4): 81. DOI:10.3390/microorganisms5040081 |
| [5] | van der Heide T, Govers LL, de Fouw J, Olff H, van der Geest M, van Katwijk MM, Piersma T, van de Koppel J, Silliman BR, Smolders AJP, van Gils JA. A three-stage symbiosis forms the foundation of seagrass ecosystems. Science, 2012, 336(6087): 1432-1434. DOI:10.1126/science.1219973 |
| [6] |
Zhou WG, Ding DW, Ling J, Lin XC, Yang QS, Zhang Y, Ahmad M, Zhang YY, Dong JD. Seagrass-microbial interactions in the rhizosphere. Acta Microbiologica Sinica, 2019, 59(11): 2117-2129.
(in Chinese) 周卫国, 丁德文, 凌娟, 林显程, 杨清松, 张颖, Manzoor Ahmad, 张燕英, 董俊德. 海草根际微生物与海草植株的互作效应. 微生物学报, 2019, 59(11): 2117-2129. |
| [7] | Hamisi M, Díez B, Lyimo T, Ininbergs K, Bergman B. Epiphytic cyanobacteria of the seagrass Cymodocea rotundata: diversity, diel nifH expression and nitrogenase activity. Environmental Microbiology Reports, 2013, 5(3): 367-376. DOI:10.1111/1758-2229.12031 |
| [8] | Trevathan-Tackett S.M., et al. A horizon scan of priorities for coastal marine microbiome research.. Nature Ecology Evolution, 2019, 3: 1509-1520. DOI:10.1038/s41559-019-0999-7 |
| [9] | Tarquinio F, Hyndes GA, Laverock B, Koenders A, Säwström C. The seagrass holobiont: understanding seagrass-bacteria interactions and their role in seagrass ecosystem functioning. FEMS Microbiology Letters, 2019, 366(6): 1-15. |
| [10] | Sun YY, Song ZL, Zhang HK, Liu PY, Hu XK. Seagrass vegetation affect the vertical organization of microbial communities in sediment. Marine Environmental Research, 2020, 162: 105174. DOI:10.1016/j.marenvres.2020.105174 |
| [11] | Apostolaki ET, Holmer M, Santinelli V, Karakassis I. Species-specific response to sulfide intrusion in native and exotic Mediterranean seagrasses under stress. Marine Environmental Research, 2018, 134: 85-95. DOI:10.1016/j.marenvres.2017.12.006 |
| [12] | Ugarelli K, Laas P, Stingl U. The microbial communities of leaves and roots associated with turtle grass (Thalassia testudinum) and manatee grass (Syringodium filliforme) are distinct from seawater and sediment communities, but are similar between species and sampling sites. Microorganisms, 2019, 7(1). |
| [13] | Tarquinio F, Bourgoure J, Koenders A, Laverock B, Säwström C, Hyndes GA. Microorganisms facilitate uptake of dissolved organic nitrogen by seagrass leaves. ISME Journal, 2018, 12(11): 2796-2800. DOI:10.1038/s41396-018-0218-6 |
| [14] | Martin BC, Bougoure J, Ryan MH, Bennett WW, Colmer TD, Joyce NK, Olsen YS, Kendrick GA. Oxygen loss from seagrass roots coincides with colonisation of sulphide-oxidising cable bacteria and reduces sulphide stress. The ISME Journal, 2019, 13(3): 707-719. DOI:10.1038/s41396-018-0308-5 |
| [15] | Brodersen KE, Siboni N, Nielsen DA, Pernice M, Ralph PJ, Seymour J, Kühl M. Seagrass rhizosphere microenvironment alters plant-associated microbial community composition. Environmental Microbiology, 2018, 20(8): 2854-2864. DOI:10.1111/1462-2920.14245 |
| [16] | NSR A, Ferriol P, Cryer C, Alcon E, Busquets A, Sintes E, Vidal C, Moyà G. Significant nitrogen fixation activity associated with the phyllosphere of Mediterranean seagrass Posidonia oceanica: first report. Marine Ecology Progress Series, 2016, 551: 53-62. DOI:10.3354/meps11755 |
| [17] | Crump BC, Wojahn JM, Tomas F, Mueller RS. Metatranscriptomics and amplicon sequencing reveal mutualisms in seagrass microbiomes. Frontiers in Microbiology, 2018, 9(3): 1-14. |
| [18] | Fahimipour AK, Kardish MR, Lang JM, Green JL, Eisen JA, Stachowicz JJ. Globalscale structure of the eelgrass microbiome. Applied and Environmental Microbiology, 2017, 83: 1-12. |
| [19] | Garcias-Bonet N, Arrieta JM, Duarte CM, Marbà N. Nitrogen-fixing bacteria in Mediterranean seagrass (Posidonia oceanica) roots. Aquatic Botany, 2016, 131: 57-60. DOI:10.1016/j.aquabot.2016.03.002 |
| [20] | Lachnit T, Meske D, Wahl M, Harder T, Schmitz R. Epibacterial community patterns on marine macroalgae are host-specific but temporally variable. Environmental Microbiology, 2011, 13: 655-665. DOI:10.1111/j.1462-2920.2010.02371.x |
| [21] | Marzinelli EM, Campbell AH, Valdes EZ, Vergés A, Nielsen S, Wernberg T, Bettibnies TD, Bennett S, Caporaso JG, Thomas T, Steinberg PD. Continental-scale variation in seaweed host-associated bacterial communities is a function of host condition, not geography. Environmental Microbiology, 2015, 17: 4078-4088. DOI:10.1111/1462-2920.12972 |
| [22] | Cúcio C, Engelen AH, Costa R, Muyzer G. Rhizosphere microbiomes of European seagrasses are selected by the plant, but are not species specific. Frontiers in Microbiology, 2016, 7: 440. |
| [23] | Johnson AJ, Orth RJ, Moore KA. The role of sexual reproduction in the maintenance of established Zostera marina meadows. Journal of Ecology, 2020, 108(3): 945-957. DOI:10.1111/1365-2745.13362 |
| [24] | Furman BT, Jackson LJ, Bricker E, Peterson BJ. Sexual recruitment in Zostera marina: a patch to landscape-scale investigation. Limnology and Oceanography, 2015, 60(2): 584-599. DOI:10.1002/lno.10043 |
| [25] | Zhang X, Zhou Y, Xu S, Wang P, Zhao P, Yue S, Gu R, Song X, Xu S, Liu JX, Wang X. Differences in reproductive effort and sexual recruitment of the seagrass Zostera japonica between two geographic populations in northern China. Marine Ecology Progress Series, 2020, 638: 65-81. DOI:10.3354/meps13248 |
| [26] | Jiang K, Tsang PKE, Xu NN, Chen XY. High genetic diversity and strong differentiation in dramatically fluctuating populations of Zostera japonica (Zosteraceae): implication for conservation. Journal of Plant Ecology, 2018, 11(5): 789-797. DOI:10.1093/jpe/rtx053 |
| [27] | Hu CY, Dong JY, Gao LJ, Yang XL, Wang Z, Zhang XM. Macrobenthos functional trait responses to heavy metal pollution gradients in a temperate lagoon. Environmental Pollution: Barking, Essex, 2019, 253: 1107-1116. DOI:10.1016/j.envpol.2019.06.117 |
| [28] | Zhang XM, Zhou Y, Liu P, Wang F, Liu BJ, Liu XJ, Yang HS. Temporal pattern in biometrics and nutrient stoichiometry of the intertidal seagrass Zostera japonica and its adaptation to air exposure in a temperate marine lagoon (China): implications for restoration and management. Marine Pollution Bulletin, 2015, 94(1/2): 103-113. |
| [29] |
Wang PM, Zhou Y, Zhang XM, Wang F, Gu RT, Xu SC, Song XY. Supplementary investigation of eelgrass seedlings (Zostera marina L.)/in Swan lake.. Marine Sciences, 2016, 40(6): 49-55.
(in Chinese) 王朋梅, 周毅, 张晓梅, 王峰, 顾瑞婷, 徐少春, 宋肖跃. 天鹅湖大叶藻种苗补充情况调查. 海洋科学, 2016, 40(6): 49-55. |
| [30] | Turner TR, James EK, Poole PS. The plant microbiome. Genome Biology, 2013, 14(6): 1-10. |
| [31] | Matse DT, Huang CH, Huang YM, Yen MY. Effects of coinoculation of Rhizobium with plant growth promoting rhizobacteria on the nitrogen fixation and nutrient uptake of Trifolium repens in low phosphorus soil. Journal of Plant Nutrition, 2020, 43(5): 739-752. DOI:10.1080/01904167.2019.1702205 |
| [32] | Yamamoto K, Shiwa Y, Ishige T, Sakamoto H, Tanaka K, Uchino M, Tanaka N, Oguri S, Saitoh H, Tsushima S. Bacterial diversity associated with the rhizosphere and endosphere of two halophytes: Glaux maritima and Salicornia europaea.. Frontiers in Microbiology, 2018, 9: 2878. DOI:10.3389/fmicb.2018.02878 |
| [33] | Hasler-Sheetal H, Holmer M. Sulfide intrusion and detoxification in the seagrass Zostera marina.. PLoS ONE, 2015, 10(6): 1-19. |
| [34] | Scholz VV, Müller H, Koren K, Nielsen LP, Meckenstock RU. The rhizosphere of aquatic plants is a habitat for cable bacteria. FEMS Microbiology Ecology, 2019, 95(6): fiz062. DOI:10.1093/femsec/fiz062 |
| [35] | Sanchez-Amat A, Solano F, Lucas-Elío P. Finding new enzymes from bacterial physiology: a successful approach illustrated by the detection of novel oxidases in Marinomonas mediterranea. Marine Drugs, 2010, 8(3): 519-541. DOI:10.3390/md8030519 |
| [36] | Houbo W. Culture-dependent diversity of Actinobacteria associated with seagrass (Thalassia hemprichii). African Journal of Microbiology Research, 2012, 6(1): 87-94. |
| [37] | Schink B, Pfennig N. Propionigenium modestum gen. nov. sp. nov. a new strictly anaerobic, nonsporing bacterium growing on succinate.. Archives of Microbiology, 1982, 133: 209-216. DOI:10.1007/BF00415003 |
| [38] | Janssen PH, Liesack W. Succinate decarboxylation by Propionigenium maris sp. nov., a new anaerobic bacterium from an estuarine sediment.. Archives of Microbiology, 1995, 164(1): 29-35. DOI:10.1007/BF02568731 |
| [39] | Ma Y, Hu AY, Yu CP, Yan QP, Yan XZ, Wang YZ, Deng F, Xiong HJ. Response of microbial communities to bioturbation by artificially introducing macrobenthos to mudflat sediments for in situ bioremediation in a typical semi-enclosed bay, southeast China. Marine Pollution Bulletin, 2015, 94(1/2): 114-122. |
| [40] | Zheng B, Wang L, Liu L. Bacterial community structure and its regulating factors in the intertidal sediment along the Liaodong Bay of Bohai Sea, China. Microbiological Research, 2014, 169(7/8): 585-592. |
| [41] | He H, Li MY, Zhen Y, Mi TZ, Yu ZG. Bacterial and archaeal communities in sediments from the adjacent waters of Rushan bay (China) revealed by illumina sequencing. Geomicrobiology Journal, 2020, 37(1): 86-100. DOI:10.1080/01490451.2019.1666193 |
| [42] | Dworkin M, Falkow S, Rosenberg E, Schleifer KH, Stackebrandt E. The Prokaryotes-delta and epsilon subclasses. 2006. |
| [43] | Hasler-Sheetal H, Fragner L, Holmer M, Weckwerth W. Diurnal effects of Anoxia on the metabolome of the seagrass Zostera marina. Metabolomics, 2015, 11(5): 1208-1218. DOI:10.1007/s11306-015-0776-9 |
| [44] |
Wan YY, Zhao GP. Research on sulfur microorganisms in prokaryotes. Microbiology, 2017, 44(6): 1471-1480.
(in Chinese) 万云洋, 赵国屏. 原核微生物的硫功能菌. 微生物学通报, 2017, 44(6): 1471-1480. |
| [45] | Cúcio C, Overmars L, Engelen AH, Muyzer G. Metagenomic analysis shows the presence of bacteria related to free-living forms of sulfur-oxidizing chemolithoautotrophic symbionts in the rhizosphere of the seagrass Zostera marina. Frontiers in Marine Science, 2018, 5(5): 1-15. |
| [46] |
Ling J, Dong JD, Zhang YY, Wang YS, Long LJ, Chen L, Huang XF, Pan H, Zhang S. Research on the status and prospects of N2-fixation microorganisms in seagrass beds ecosystem. Journal of Biology, 2012, 29(3): 62-65.
(in Chinese) 凌娟, 董俊德, 张燕英, 王友绍, 龙丽娟, 陈蕾, 黄小芳, 潘虎, 张偲. 海草床生态系统固氮微生物研究现状与展望. 生物学杂志, 2012, 29(3): 62-65. DOI:10.3969/j.issn.2095-1736.2012.03.062 |
| [47] |
Zheng PF, Zhang XL, Gong J. Vertical patterns of bacterial and archaeal abundance and community structure in seagrass (Zostera marina) meadow sediments. Microbiology, 2020, 47(6): 1662-1674.
(in Chinese) 郑鹏飞, 张晓黎, 龚骏. 大叶藻(Zostera marina)海草床沉积物细菌和古菌丰度及组成的垂直剖面特征. 微生物学通报, 2020, 47(6): 1662-1674. |
| [48] | Hug LA, Castelle CJ, Wrighton KC, Thomas BC, Sharon I, Frischkorn KR, Williams KH, Tringe SG, Banfield JF. Community genomic analyses constrain the distribution of metabolic traits across the Chloroflexi phylum and indicate roles in sediment carbon cycling. Microbiome, 2013, 1(1): 22. DOI:10.1186/2049-2618-1-22 |
| [49] | Zhang H, Yoshizawa S, Sun Y, Huang YJ, Chu X, González JM, Pinhassi J, Luo HW. Repeated evolutionary transitions of flavobacteria from marine to non-marine habitats. Environmental Microbiology, 2019, 21(2): 648-666. DOI:10.1111/1462-2920.14509 |
| [50] | Xue CX, Zhang H, Lin HY, Sun Y, Luo DL, Huang YJ, Zhang XH, Luo HW. Ancestral niche separation and evolutionary rate differentiation between sister marine flavobacteria lineages. Environmental Microbiology, 2020, 22(8): 3234-3247. DOI:10.1111/1462-2920.15065 |
| [51] |
Pengyuan L, Haikun Z, Lin C, Yanyu S, Caixia W, Hong S, Xiaoke H. Rhizosphere microbial community structure and function of Zostera japonica in the distribution area of seagrass beds in the Yellow Sea and Bohai Sea. Acta Microbiologica Sinica, 2019, 59(8): 1484-1499.
(in Chinese) 刘鹏远, 张海坤, 陈琳, 孙延瑜, 王彩霞, 宋虹, 胡晓珂. 黄渤海海草分布区日本鳗草根际微生物群落结构特征及其功能分析. 微生物学报, 2019, 59(8): 1484-1499. |
 2021, Vol. 61
2021, Vol. 61




