中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 周杰, 谢泰祥, 刘江枫, 兰琳英, 徐艳芳, 刘志毅, 艾叶, 张清华. 2021
- Zhou Jie, Xie Taixiang, Liu Jiangfeng, Lan Linying, Xu Yanfang, Liu Zhiyi, Ai Ye, Zhang Qinghua. 2021
- 野生建兰根系共生真菌群落结构及生物学功能
- Community structure and biological function of the root symbiotic fungi of wild Cymbidium ensifolium
- 微生物学报, 61(7): 2136-2153
- Acta Microbiologica Sinica, 61(7): 2136-2153
-
文章历史
- 收稿日期:2020-11-07
- 修回日期:2020-12-31
- 网络出版日期:2021-01-19
2. 福建农林大学林学院, 福建 福州 350002;
3. 福州市于山风景区管理处, 福建 福州 350001
2. College of Forestry, Fujian Agriculture and Forestry University, Fuzhou 350002, Fujian Province, China;
3. Management Office of Yushan Scenic Area, Fuzhou 350001, Fujian Province, China
真菌广泛分布在植物根、茎、叶、花和果等器官组织内外,特别是菌根真菌、深色有隔内生真菌(dark septate endophytic fungi,DSE)和印度梨形孢Piriformospora indica等为代表的植物共生真菌(plant symbiotic fungi)与陆地植物的根系形成的共生体系伴随着陆地生态系统的变迁[1-3]。兰科植物作为最大开花植物类群之一,其种类超过28000种[4-5]。兰科植物与菌根真菌构建共生关系,菌根真菌伴随着兰科植物几乎整个生活史[6],兰科植物种子缺乏胚乳,在种子萌发和原球茎时期无法自行供养,高度依赖菌根真菌提供碳源和其他营养物质[7-8]。按碳源的来源,兰科植物可划分3种营养型:光合自养型、部分真菌异养型和完全真菌异养型[9],光合自养型是指兰科植物成年后进行光合作用供给碳源;部分真菌异养型,是指兰科植物成年后通过光合作用和真菌共生获取碳源和其他营养物质;完全真菌异养型兰科植物因无法光合作用在其整个生活史完全依赖真菌提供营养。自1840年Link首次发现兰科植物的根内真菌以来,兰科植物的菌根真菌不断被发现,现已知的兰科菌根真菌主要集中在担子菌门Basidiomycota、子囊菌门Ascomycota和毛霉门Mucoromycota[10-11],其中最常见的菌根真菌为胶膜菌科Tulasnellaceae、角担菌科Ceratobasidiaceae和蜡壳耳科Sebacinaceae,它们均为担子菌门[12-13]。
目前研究发现,除菌根真菌外,植物根系其他共生真菌同样对植物的生长发育有着促进作用[14-15]。植物共生真菌是指能与植株形成共生关系的真菌类群,包括菌根真菌(Mycorrhizal fungi)、内生真菌(Endophytic fungi)、根际土壤真菌(Rhizosphere fungi)和附生真菌(Epiphytic fungi),是自然界普遍存在的一种共生现象[3]。其中绿僵菌属(Metarhizium)作为重要的昆虫病原真菌,现阶段主要集中于防治害虫研究和应用方面,然而一些绿僵菌的主要栖息处不为昆虫,而是大量存在于植物根际土壤环境并兼具腐生性,对植物根系的发育和植株生长有促进作用[16-17]。植物内生真菌白色产气霉Muscodor crispans能产生挥发性有机物,对终极腐霉Pythium ultimum、Phytophthora cinnamomi和Sclerotinia sclerotiorum等多种植物病原菌有抑制和致死作用,此外对一些人类病原菌也有较好的抗性[18]。
建兰Cymbidium ensifolium为兰科兰属地生兰,广泛分布在东南亚和南亚各国,在中国有着悠久的栽培历史,其叶姿优雅、形态美观、香气扑鼻,具有极高的观赏、文化和经济价值,常作为盆栽和切花植物栽培[19]。近些年,由于人为活动频繁和气候变化异常,野生建兰资源遭受严重破坏,被列入《濒危野生动植物种国际贸易公约》(CITES)[7],对建兰的野生资源恢复和快速繁殖的相关研究迫切。大量研究表明,从兰科植物根内分离出的菌根真菌能对兰科植物的种子萌发、幼苗形成及其后续生长发育产生促生作用[7-9]。然而建兰共生真菌的研究多集中在菌根真菌分离鉴定[20-21]、促生[22]等方面,受限于现有的真菌分离培养技术,人们仅对极小部分的建兰根系共生真菌进行了分离,缺乏对建兰共生真菌群落多样性和其与寄主间营养关系的全面了解,阻碍了建兰生长有益真菌的开发与利用,本研究采用高通量测序技术和生物信息学对不同地域分布的野生建兰根围土壤、根表与根内真菌的种类多样性、群落结构及优势真菌的生态功能进行分析,从不同地点、不同生态位对建兰根系共生真菌进行全面的探究,深入探讨了建兰与真菌间的共生关系,对针对性地分离有益真菌资源、建兰种质资源的保护、野生种群的生态恢复以及生产实践有着重要的指导意义。
1 材料和方法 1.1 试验材料在2018年11月至12月从湖南、贵州、云南和福建四地陆续采集处于花果期的野生建兰样品,具体为2018年11月25日采集湖南样品;2018年11月27日采集贵州样品;2018年11月30日采集云南样品;2018年12月2日采集福建样品。采集点的地理位置分别为:湖南省株洲市天元区长源村(27°39′N,113°4′E),缩写为HN;福建省福州市晋安区寿山乡溪下村(26°13′N,119°17′E),缩写为FJ;贵州省黔南布依族苗族自治州都匀市高源村(26°14′N,107°33′E),缩写为GZ;云南省文山壮族苗族自治州文山市科麻粟村(23°20′N,104°24′E),缩写为YN。除样品FJ的建兰植株生于疏林陡坡(坡度65°),其他样品HN、GZ和YN的建兰植株均生长在密林缓地中,土壤湿润,有丰富的植物凋落物覆盖层。
取样方法,将土壤表面的植物残体移除,把建兰根及根周围3 mm土壤装进50 mL的离心管,立刻放入干冰中,参考Edwards等的样品处理方式[23],将根共生真菌分为根围土壤(Rs)、根表(Rp)、根内(Ep) 3个生态位进行真菌种群结构鉴定。根围土壤是采集根表3 mm范围的土壤,通过振荡洗涤沉淀获得的;根表附着微生物群落来自根表面,不能被振荡洗涤获取,通过超声波清洗使其脱离根表面;而根内微生物群落仅指寄生在根内部的微生物,通过超声波清洗3次以后,干净的根用于内生菌的检测。
用离心管中加入100 mL无菌PBS缓冲液振荡洗涤,收集土壤悬浊液,即为Rs生态位;清洗后的根加入100 ml无菌PBS缓冲液,并用超声波清洗30 s,2次,收集液体,作为Rp生态位;清洗后的根再次加入100 mL无菌PBS缓冲液,再次超声波清洗30 s后,放置无菌吸水纸上晾干,作为Ep生态位。
1.2 DNA提取及真菌ITS区域扩增测序使用土壤样品DNA试剂盒(12888,MoBio公司)分别提取根围土壤(Rs)和根表土壤(Rp)的总DNA;使用植物组织DNA试剂盒(DP305,天根)提取根部的总DNA。检测各生态位提取DNA的纯度和浓度,合格的DNA样品用无菌水稀释至1 ng/μL作为真菌核糖体内部DNA转录间隔区(internal transcribed spacer,ITS)扩增的模板,使用ITS2-2043R (5'-GCTGCGTTCTTCATCGAT GC-3')和ITS5-1737F (5'-GGAAGTAAAAGTCGT AACAAGG-3')引物扩增真菌ITS1。PCR产物委托北京诺和致源科技股份有限公司,基于Ion S5TMXL (Thermofisher)进行高通量测序。
1.3 数据分析 1.3.1 聚类分析:利用Uparse软件质控和过滤质量序列,在97%相似度下进行聚类,得到分类操作单位OUT (operational taxonomic unit)[24]。按界、门、纲、目、科、属、种的真菌水平进行分类,基于Unit数据库(https://unite.ut.ee/),将聚类的OUT中丰度最高的序列采用Qiime1.91软件物种注释[25-26]。
1.3.2 Alpha多样性分析:单样本的OUT多样性分析(alpha diversity)可以反映样本内微生物群落的丰富度和多样性,利用每个OTU在样品中的有效序列的绝对丰度和相对信息[26],使用Qiime1.9.1计算Observed-OTUs、ACE、Shannon、Simpson和Good’s coverage等指数,评估各样本中微生物群落的物种丰富度和多样性等差异。Shannon指数值越大,该群落多样性越高,范围在0–1;Simpson指数值越大该群落的绝对多样性程度越高;ACE指数值越高,群落物种绝对丰富度越高。使用R软件(Version 2.15.3)绘制稀释曲线(rarefaction curve)和物种箱型图(species accumulation boxplot)评价测序量和样品数量合理性。
1.3.3 Beta多样性分析:利用Qiime软件对所测所有样本的真菌群落构成进行分析,采用共有OUT以Venn图展示,主成分分析法(PCoA)和分子方差分析法(AMOVA)对样本间的真菌种群结构差异显著性进行分析。
1.3.4 生物功能注释:基于已经获得的建兰的根围土壤、根表和根内真菌物种分类和丰度,利用FunGuild数据库查询真菌生态学功能,根据已知的真菌功能,推断该真菌在建兰生长发育过程中所起的作用,数据分析方法参考Nguyen等的论文[27]。
2 结果和分析 2.1 测序结果分析分别对HN、FJ、GZ、YN共4个样品的野生建兰根系的3个生态位点(根围土壤Rs、根表Rp、根内Ep)共12个生态位的真菌核糖体DNA (rDNA)基因ITS1区域分别进行高通量测序,各生态位的测序clean reads数据量有效率均超93%。稀释曲线显示,在测序clean reads未达到80000条时,各生态位的稀释曲线已趋于稳定(图 1-A);所有测序生态位深度指数Good’s coverage指数均超过0.997 (表 1),表明测序数据完整性较好,各生态位真菌菌群未被测出的序列的概率极低。物种积累箱型图表明,持续抽样下新observed species出现的速率,随着样本数的增加箱型图趋于平缓(图 1-B),表示该环境中的真菌物种数并不会随样本数的增加而显著增多,该测序的样本数充足,能够覆盖测试样品的真菌种群。

|
| 图 1 建兰根共生真菌高通量测序稀释性曲线和物种积累箱型图 Figure 1 Rarefaction curve of high throughput sequencing results and species accumulation boxplot of Cymbidium ensifolium root symbiotic fungi. Rs: rhizosphere soil; Rp: rhizoplane; Ep: endoplane. |
| Samples | OTUs | ACE | Simpson | Shannon | Goods coverage | |
| GZ | Rs | 848 | 840.306 | 0.883 | 4.966 | 0.998 |
| Rp | 770 | 746.761 | 0.904 | 5.166 | 0.999 | |
| Ep | 913 | 901.686 | 0.974 | 6.561 | 0.999 | |
| FJ | Rs | 551 | 565.288 | 0.680 | 2.867 | 0.998 |
| Rp | 739 | 944.712 | 0.735 | 3.939 | 0.997 | |
| Ep | 376 | 366.858 | 0.893 | 4.083 | 0.999 | |
| YN | Rs | 1283 | 1266.38 | 0.961 | 7.056 | 0.999 |
| Rp | 1098 | 1087.78 | 0.961 | 6.883 | 0.999 | |
| Ep | 454 | 475.107 | 0.925 | 4.849 | 0.999 | |
| HN | Rs | 784 | 772.104 | 0.913 | 5.815 | 0.999 |
| Rp | 715 | 703.207 | 0.883 | 5.719 | 0.999 | |
| Ep | 493 | 479.505 | 0.914 | 4.764 | 0.999 | |
| Rs: rhizosphere soil; Rp: rhizoplane; Ep: endoplane. | ||||||
2.2 建兰根共生真菌群落结构 2.2.1 建兰各样品间真菌群落结构差异:
对建兰的根系共生真菌进行高通量测序,检测出建兰所有样品共获得2840个OTU。对不同样品进行比较分析,云南(YN)样品鉴定的OTU数量最多,共鉴定了1607个,香农指数6.263;贵州(GZ)样品检测到1267个OTUs,香农指数5.564;湖南(HN)样品鉴定了1126个OTU,香农指数5.433;而福建样品(FJ)检测到的OTU数量最少,仅994个,香农指数3.63。显示YN样品的根共生真菌多样性最高,其次为GZ、HN,FJ样品的根共生真菌多样性最低;云南、贵州、湖南三地样品的辛普森指数均超过0.9 (表 1),揭示了这3个建兰样品的根共生真菌有着高的多样性。
对各样品的生态位间进行比较分析,发现YN和HN样品生态位间的ACE指数和香农指数均按照:根围土壤、根表、根内的顺序渐减,说明YN和HN样品的各生态位真菌丰度和多样性均按根围土壤、根表、根内由外向内递减;GZ样品根内生态位的ACE指数和香农指数均高于根围土壤和根表的生态位,说明GZ样品根内真菌的丰度和种类多样性均高于根围土壤和根表;FJ样品的根表真菌ACE指数显著高于根围土壤和根内真菌,而根内生态位的香农指数最高(表 1),说明该样品建兰根表附生丰富的真菌,而根内真菌多样性程度高。
建兰根共生真菌包含有12门44纲103目241科432属,这12个门分别为子囊菌门Ascomycota、担子菌门Basidiomycota、壶菌门Chytridiomycota、虫霉菌门Entomophthoromycota、球囊菌门Glomeromycota、单毛壶菌门Monoblepharomycota、被孢霉门Mortierellomycota、毛霉菌门Mucoromycota、梳霉菌门Kickxellomycota、油壶菌门Olpidiomycota、隐菌门Rozellomycota、捕虫霉门Zoopagomycota,其中担子菌门、子囊菌门和被孢霉门为优势菌门,分别占建兰根系共生真菌总丰度的49.51%、27.39%和20.22%。在门水平下,GZ、YN和HN样品的优势菌门都为子囊菌门和担子菌门,2门相对丰度之和均超总丰度的85%,而被孢霉门均不超总丰度的10% (图 2-A);而在FJ样品中被孢霉门丰度占绝对优势为54.22%,其次为担子菌门和子囊菌门分别为26.10%、18.14% (图 2-A),说明在门水平下,样品FJ与样品GZ、YN、HN真菌群落结构差异大,且被孢霉属(Mortierella)在FJ样品丰度显著高于在GZ、YH和HN样品的含量。根系共生真菌丰度排名前20种的真菌分布在子囊菌门、担子菌门和被孢霉门中(图 3),合计占建兰共生真菌丰度的36.34%,其中被孢霉属、Saitozyma和Papiliotrema在4个样品均有分布,且丰度显著高于其他属(图 4),分别占建兰共生真菌总丰度的11.75%、11.45%和7.93%。

|
| 图 2 各样品与各生态位点在门水平下相对丰度 Figure 2 Relative abundance map at phylum level between each sample and each niche point. A: relative abundance map at phylum level between each sample; B: relative abundance map at phylum level between each niche. Rs: rhizosphere soil; Rp: rhizoplane; Ep: endoplane. |
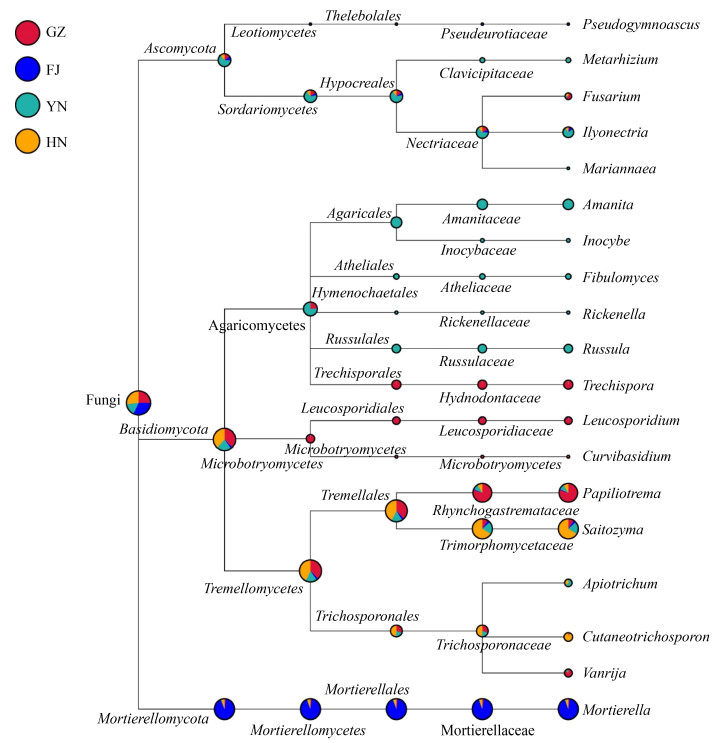
|
| 图 3 建兰根共生真菌优势种类 Figure 3 Dominant taxa of Cymbidium ensifolium root symbiotic fungi. |
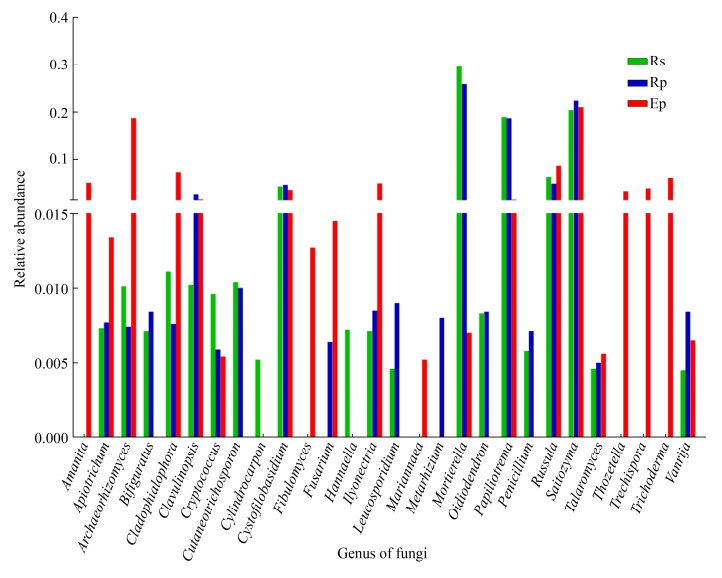
|
| 图 4 建兰根系各生态位点丰度前20的真菌属 Figure 4 The top 20 fungal genera in each niche point of Cymbidium ensifolium root. Rs: rhizosphere soil; Rp: rhizoplane; Ep: endoplane. |
2.2.2 建兰各生态位间真菌群落结构差异:
对不同生态位进行分析,根围土壤、根表和根内分别检测到2141、2043和1303个OTU,而根围土壤、根表和根内真菌的辛普森指数分别为0.860、0.871和0.927,说明建兰根系存在着种类丰富的共生真菌;根围土壤、根表和根内生态位点的香农指数分别为5.176、5.426、5.064 (表 1),各生态位间物种多样性水平无显著差异(P > 0.05)。根内真菌平均ACE值为555.789,低于根围土壤(ACE=861.019)和根表真菌(ACE=870.615) (表 1),说明建兰的根内真菌丰度低于根围土壤和根表真菌,根表真菌丰度略高于根围土壤真菌,但3个生态位间丰度无显著差异(P > 0.05)。
建兰根系各生态位的优势门类为担子菌门、被孢霉门和子囊菌门,三者之和占种群结构总丰度95%以上(图 2-B),说明各生态位点种群结构有一定的相似性。其中担子菌门在根围土壤、根表和根内真菌间所占丰度无显著差异(47.85%,53.10%,47.74%);子囊菌门真菌在根围土壤和根表的丰度差异极小分别为19.86%、19.09%,而根内所占丰度显著增幅(45.18%);被孢霉门真菌在根内仅占3.14%,而在根围土壤和根表中占比高达30.20%和25.64% (图 2-B)。综上可知,在门水平下,根内真菌与根围土壤和根表的真菌群落结构存在一定的差异,而根围土壤和根表真菌的群落结构无显著差异;但在根围土壤中没有检测出虫霉菌门,根表真菌无梳霉菌门和捕虫霉门,根内真菌没有发现虫霉菌门和油壶菌门,说明不同生态位都有相应特殊的真菌存在。
各生态位丰度前20的真菌分布在28个属中(图 4),担子菌门有14个属,子囊菌门有12个属,被孢霉门和毛霉菌门各有1个属,且每个生态位各自丰度前20的真菌种类丰度之和均超过89%。其中根围土壤的优势菌属(相对丰度大于5%)为被孢霉属、Saitozyma、Papiliotrema和红菇属(Russula)分别占其丰度的29.59%、20.32%、18.88%、6.32%,并且被孢霉属、Saitozyma、Papiliotrema和红菇属在根表菌群也为优势菌属,占相对丰度的25.83%、22.33%、18.63%、5.01%,根内菌群的优势菌属较多有Saitozyma、Archaeorhizomyces、红菇属、Cladophialophora、木霉属(Trichoderma)和鹅膏属(Amanita)分别占根内真菌丰度的20.95%、18.69%、8.62%、7.30%、6.10%和5.10% (图 4)。各生态位点不仅优势菌属不同,并且存在特有属,在丰度前20的真菌属中,汉纳酵母属(Hannaella)、柱孢属(Cylindrocarpon)真菌仅分布在根围土壤;绿僵菌属仅在建兰根表分布;木霉属、粗糙孔菌属(Trechispora)、Thozetella、马利娅霉属(Mariannaea)、Fibulomyces和鹅膏属仅在建兰根内检测到存在(图 4),说明建兰根共生真菌存在生态位点偏好性。
2.3 建兰根共生真菌差异分析 2.3.1 建兰根共生真菌种类丰度分析:对不同样品建兰根系共生真菌进行差异分析,发现4个样品的共有OTU数量(275个)低于YN、HN和GZ样品的独有OTU数量(662个,355个,350个) (图 5-A),高于FJ样品的独有OTU (183个),说明建兰不同样品间的真菌种类差异较大,其中各样品间的真菌种类独特性为YN > HN > GZ > FJ;然而根围土壤、根表和根内真菌中特有OTU分别为457、364、204个,明显低于各生态位共有830个OTU (图 5-B),说明建兰不同生态位间的真菌种类差异较小,各生态位的真菌种类独特性为根围土壤 > 根表 > 根内。

|
| 图 5 样品间和生态位点间真菌OTU分布韦恩图 Figure 5 Venn figure of fungal OTU between each sample and niche point. A: Venn figure of fungal OTU between each sample point; B: Venn figure of fungal OTU between each niche. Rs: rhizosphere soil; Rp: rhizoplane; Ep: endoplane. |
建兰12个生态位丰度排名前10的真菌共有44个属,其中GZ样品独有属为隐球菌属(Cryptococcus)、汉纳酵母属、Krasilnikovozyma、白冬孢酵母属(Leucosporidium)、粗糙孔菌属、Vanrija 6个属;枝顶孢属(Acremonium)、节丛孢属(Arthrobotrys)、Chaunopycnis、斜盖伞属(Clitopilus)、长毛孔菌属(Funalia)、Luellia、新丛赤壳属(Neonectria)、假裸囊菌属(Pseudogymnoascus)、孢子丝菌属(Sporothrix)、Thozetella 10个属仅在FJ;单独在YN样品中检测到的属有鹅膏属、Fibulomyces、马利娅霉属、绿僵菌属、青霉属(Penicillium)、革菌属(Thelephora) 6属;仅分布在HN样品的属有Bulleribasidium、拟锁瑚菌属(Clavulinopsis)、柱孢属、Dactylonectria、Solicoccozyma (图 6),说明建兰4个样品的真菌群落结构在属水平上差异显著;FJ样品独有的真菌种类最多。根围土壤的独有属仅有柱孢属,根表独有属有枝顶孢属、绿僵菌属,根内有13个独有属分别为粗糙孔菌属、Thozetella、革菌属、玛利亚霉属(Mariannaea)、Luellia、长毛孔菌属、Fibulomyces、Diutina、Dactylonectria、斜盖伞属、Bulleribasidium、节丛孢属和鹅膏属(图 6),说明在属水平上,建兰根内与根围土壤、根表生态位的真菌群落结构差异显著,但根围土壤和根表真菌的群落结构差异不明显。

|
| 图 6 建兰各生态位丰度前10的真菌属 Figure 6 The top 10 fungal genera of each Cymbidium ensifolium root niche. Rs: rhizosphere soil; Rp: rhizoplane; Ep: endoplane. |
2.3.2 建兰根共生真菌群落差异分析:
基于Weight Unifrac距离进行PCoA分析,样品间的聚集距离越近,它们的物种群落结构越相似,横坐标表示一个主成分,纵坐标表示另一个主成分,百分比则表示对样品差异的贡献率。对建兰的各样品进行PCoA分析表明,第一主成分解释了群落差异的31.55%,第二主成分解释了群落差异的24.74%,共解释了真菌群落结构差异的56.29%,且各样品间呈各自分离状态(图 7),并用分子方差分析法(AMOVA)进行检验GZ-YN-HN-FJ组间差异(P < 0.001),说明GZ、FJ、YN和HN样品间根系共生真菌群落结构差异极显著。分别对4个样品的各生态位进行PCoA分析发现,每个样品的根围土壤、根表和根内生态位间呈显著分离,且第一、第二主成分的贡献率之和均为100%,完全解释了真菌群落结构的差异性(图 8),此外,用AMOVA验证每个样品的根围土壤、根表和根内生态位间的差异,均为P < 0.001,说明4个样品的根围土壤、根表和根内间的共生真菌结构均具极显著差异性。

|
| 图 7 不同样品间真菌群落PCoA分析 Figure 7 Principal component analysis of fungal communities between different samples. |

|
| 图 8 不同样品各生态位的真菌群落PCoA分析 Figure 8 Principal component analysis of fungal communities in different niches of different samples. Rs: rhizosphere soil; Rp: rhizoplane; Ep: endoplane. |
2.4 建兰根共生真菌生物学功能
基于环境功能数据库(FunGuild)对建兰根系共生真菌群落的营养型进行鉴定,发现以腐生营养型(Saprotroph)、病理-腐生-共生混合型(Pathotroph-Saprotroph-Symbiotroph)和共生营养型(Symbiotroph)为主要类型(图 9),分别占建兰根系共生真菌总丰度的45.31%、40.49%、9.76%。建兰4个样品间的真菌营养型构成差异较大,但每个样品在各自根围土壤和根表间的营养型结构高度相似,其中仅FJ样品3个生态位的营养型结构相似(图 9)。

|
| 图 9 建兰根共生真菌的生物学功能 Figure 9 Biological function of Cymbidium ensifolium root symbiotic fungi. A (Rs): rhizosphere soil; B (Rp): rhizoplane; C (Ep): endoplane. |
样品FJ在各生态位均以腐生型为绝对优势,相对丰度范围为85.98%–94.76%,其中被孢霉属为根围土壤和根表真菌的绝对优势菌属,相对丰度为96.73%,94.95%,而根内则以Thozetella为绝对优势菌属,相对丰度为83.67%;GZ样品在根围土壤和根表均以病理-腐生-共生混合型和共生营养型为主,其中病理-腐生-共生混合型均以Papiliotrema、Saitozyma为主要类型,两者相对丰度之和超93%,共生营养型主要种类均为红菇属,相对丰度均达97%;样品YN的根围土壤和根表均以腐生型、病理-腐生-共生混合型和共生营养型为主,腐生型优势属均为被孢霉属和Cladophialophora,病理-腐生-共生混合型的主要类型均为Saitozyma、Papiliotrema,共生营养型的主要类型均为红菇属、腊壳菌属(Sebacina)、丝盖伞属(Inocybe)和鹅膏属;样品HN根围土壤和根表也都以腐生型、病理-腐生-共生混合型为主要类型,腐生型均以被孢霉属、拟锁瑚菌属和Apiotrichum为优势菌,病理-腐生-共生混合型都以Saitozyma为绝对优势菌属(图 9)。
根内生态位点真菌以腐生营养型、共生营养型和病原-腐生-共生营养型为主要类型,其中腐生型为绝对优势类型,各样品相对丰度范围50.11%–85.98% (图 9-C),而病原-腐生-共生混合型和共生型分别占根内真菌群落总丰度的25.12%、15.26%。其中腐生营养型真菌的优势菌属为Archaeorhizomyces、Cladophialophora、土赤壳属(Ilyonectria)、Thozetella、粗糙孔菌属和木霉属;共生营养型真菌以鹅膏属和红菇属为主;Saitozyma在病理-腐生-共生混合类型中丰度最高,这9个属均为根内丰度排名前20的种类(图 3)。
3 讨论自Link发现兰科植物根内真菌以来,对兰科植物与其菌根真菌间的研究不断开展[10-11],本文采用高通量测序技术,对4个样品的野生建兰的根围土壤、根表和根内的共生真菌种类进行鉴定,分析真菌群落结构、种类多样性,预测真菌的生物学功能。结果显示,建兰根系共生真菌的种类多样性高,与兰科植物相关研究结论相同[28];建兰根系共生真菌分布在12个门中,以子囊菌门、担子菌门和被孢霉门为优势门类;建兰各样品的根系共生真菌营养型结构差异极大,但每个样品根围土壤和根表间营养型结构相似度高。有研究表明,兰科植物共生真菌群落结构会受到栖息地环境的影响[29-30],来自GZ、FJ、YN和HN的样品间的根系共生真菌群落结构呈极显著差异(P < 0.001),充分说明生长环境对兰花植物共生真菌群落会产生重大影响。所有样品的根围土壤、根表和根内间真菌结构差异性极显著,表明了野生建兰的根围土壤、根表和根内间的真菌群落结构差异极显著。在自然环境中,林下植被多样性和土壤含水量等因素会影响植物根系生长,对植物菌根真菌群落结构也会产生重大影响[31-32],然而FJ样品的根围土壤、根表和根内的真菌种类多样性均低于样品YN、GZ和HN对应的各生态位,样品FJ生长在疏林山地陡坡(坡度65°),土壤层薄、根系土壤持水量少,而YN、GZ和HN样品的建兰均生长密林缓地中,真菌的孢子萌发、菌丝生长大多数需要高湿度[33],推断林下植被多样性和土壤的含水量会对建兰根系共生真菌群落结构产生影响。
建兰根系共生真菌以子囊菌门、担子菌门和被孢霉门为优势菌门,其中根内真菌以子囊菌门和担子菌门数量占绝对优势,与Anacamptis morio、竹叶兰等兰科植物根内真菌群落结构相似[11, 28, 34]。建兰根系共生真菌中的被孢霉门仅有被孢霉属1属,被孢霉属在FJ样品的根系共生真菌中丰度高,而在其他样品各生态位中种类丰度低,FJ样品的根围土壤和根表真菌群落中被孢霉属为绝对优势菌属,与GZ、HN和YN样品建兰根围土壤和根表真菌以子囊菌门和担子菌门为主的结果不同,该样点建兰生长正常,因此作者认为建兰对根围土壤和根表真菌的选择不会偏好某一特定类群,在不同环境中会选择不同类型种类的真菌,首选具有促进建兰生长发育功能的类群。胶膜菌科、角担菌科和蜡壳菌科为兰科植物的重要菌根真菌[12-13],但本研究未在建兰根系共生真菌检测到胶膜菌科真菌,分析其原因可能是采用的DNA测序区域不合适,已有研究表明,不同引物对扩增不同的真菌类群有一些偏好[35],因此为了更加全面了解建兰根共生菌种群结构,采用不同引物分别对其进行高通量测序是必要的;角担菌科在建兰根系共生真菌丰度极低,然而杨前宇[22]等在建兰根内分离出了胶膜菌科和角担菌科真菌,说明了高通测序需与分离技术相结合才能更加全面地了解兰科植物根系共生真菌类群。红菇科作为多种兰花植物菌根真菌,在珊瑚兰属Corallorhiza、天麻属Gastrodia等兰科植物根内存在[36-37],红菇属真菌也与松属Larix、松属Pinus和豆科Fabaceae等多种植物形成外生菌根,能促进植物根系的生长和营养吸收并提高植物的抗逆性[38],本研究检测到红菇属在建兰的根围土壤、根表和根内均为优势真菌种类,红菇属真菌可能在建兰的种子萌发、生长发育过程中起着重要的作用。银耳目Tremellales的Saitozyma和Papiliotrema多存在于土壤和植物根际中[14, 29, 39],有研究发现Papiliotrema中存在菌株能与丛枝菌根真菌进行协同作用,增加植物磷、氮、叶绿素的吸收并能产生酚类化合物,从而对植物生长发育产生有利影响[14],但未见Saitozyma和Papiliotrema作为兰科植物菌根真菌的报道,且Saitozyma和Papiliotrema在建兰的根围土壤、根表和根内均大量存在,Saitozyma和Papiliotrem是否能对建兰生长发育产生有利影响还有待进一步研究。
按照生态类型的不同,兰科植物被划分为地生兰、附生兰和腐生兰,建兰属地生兰,其种子萌发、原球茎的形成及营养吸收等生理过程需共生真菌进行协作。本文中的建兰根系共生真菌以腐生营养型、共生营养型和病理-腐生-共生混合型为主要类型,样品GZ、FJ、YN和HN间的真菌群落营养型结构差异较大,其中各样品的根围土壤和根表真菌的营养类型结构高度相似,除样品FJ外,其他样品的根内与根围土壤和根表营养型结构差异较大。建兰根内真菌营养类型结构以腐生营养型、共生营养型和病理-腐生-共生混合型为主,腐生营养型为优势类型丰度最高,这与植物内生菌大都与植物是共生关系的普遍认知有所不同,当然,FunGuild是基于已知文献进行真菌生物学功能进行预测,而同一种真菌在不同环境中对不同寄主的生物学功能不同,已知的腐生型真菌在建兰根内是否也是腐生营养还需要进一步的研究。样品FJ各生态位的营养类型均以腐生营养型为绝对优势型,其中根围土壤和根表真菌的腐生营养型中绝对优势菌属被孢霉属,而根内真菌的被孢霉属丰度极低。被孢霉属真菌分布在土壤、植物根际和根内等环境中[15, 40],一些被孢霉真菌(M. elongata,M. antarctica,M. verticillata)能促进植物产生赤霉酸、吲哚乙酸等植物激素,并为共生植物提供氮、磷等营养物质[41]。被孢霉属作为腐生菌,能促进土壤的碳循环,并释放有机酸来溶解土壤中的磷的潜力[42-43]。王涵等[44]在对金线莲Anoectochilus roxburghii幼苗与其根内真菌的促生实验中,发现从根内分离出的Cladophialophora一菌株对其幼苗有一定的促生作用,并且Cladophialophora也寄生在竹叶兰和二叶舌唇兰Platanthera chlorantha等兰科植物根内[28, 45],在建兰根围土壤、根表和根内的腐生类型中大量存在,推测Cladophialophora对建兰生长发育也有一定的促进作用。土壤真菌Archaeorhizomyces普遍存在于植物根系环境中具有生长缓慢的特征,其营养机制尚未可知[46],在竹叶兰的根表也有分布[28],为建兰根内腐生营养型的优势菌属,也是建兰根内优势属,Archaeorhizomyces与建兰的营养机制值得进一步探讨。鹅膏属和红菇属作为常见的外生菌根真菌(Ectomycorrhizal fungi)[3, 47-48],为建兰的根内优势菌属,说明建兰对菌根真菌有着筛选,但鹅膏属和红菇属侵入建兰根内的机制有待探索。
兰科植物种子的萌发和幼苗的生长均需要真菌不同程度的参与[7-8],长期以来,用于兰科植物种子共生萌发相关研究的真菌多是从野生成年植株的根、原球茎中分离和筛选获得,但大部分的真菌不能进行分离培养[5, 10],而且环境中真菌种类繁多,腐生型、生长迅速的真菌往往覆盖生长慢的真菌,导致分离方法遗漏了具有重要生物学功能的真菌种类。该文应用高通测序技术对建兰共生真菌群落结构及其生物功能进行探索,为分离建兰根共生菌中某些可能有促生能力的真菌提供理论依据,并为针对性地分离这些真菌而设计选择性分离培养基提供支持。为建兰的种子萌发、幼苗促生及植株生长发育提供了理论依据。
| [1] | Simon L, Bousquet J, Lévesque RC, Lalonde M. Origin and diversification of endomycorrhizal fungi and coincidence with vascular land plants. Nature, 1993, 363(6424): 67-69. DOI:10.1038/363067a0 |
| [2] |
Liang Y, Guo LD, Ma KP. The role of mycorrhizal fungi in ecosystems. Acta Phytoecologica Sinica, 2002, 26(6): 739-745.
(in Chinese) 梁宇, 郭良栋, 马克平. 菌根真菌在生态系统中的作用. 植物生态学报, 2002, 26(6): 739-745. DOI:10.3321/j.issn:1005-264X.2002.06.016 |
| [3] | 刘润进, 王琳. 生物共生学. 北京: 科学出版社, 2018: 8-87. |
| [4] | Chase MW, Cameron KM, Freudenstein JV, Pridgeon AM, Salazar G, van den Berg C, Schuiteman A. An updated classification of Orchidaceae. Botanical Journal of the Linnean Society, 2015, 177(2): 151-174. DOI:10.1111/boj.12234 |
| [5] |
Gao YY, Hang Y, Liu JD, Li Y, Liu Z, Li L, Tian HL, Zhang MS. Advances on orchid symbiotic fungi promoting seeds germination. Plant Physiology Journal, 2020, 56(2): 141-150.
(in Chinese) 高燕燕, 杭烨, 刘剑东, 李扬, 刘智, 李林, 田海露, 张明生. 兰科药用植物共生真菌促进种子萌发研究进展. 植物生理学报, 2020, 56(2): 141-150. |
| [6] | Dearnaley JDW. Further advances in orchid mycorrhizal research. Mycorrhiza, 2007, 17(6): 475-486. DOI:10.1007/s00572-007-0138-1 |
| [7] | Valadares RB, Pereira MC, Otero JT, Cardoso EJ. Narrow fungal mycorrhizal diversity in a population of the orchid Coppensia doniana. Biotropica, 2012, 44(1): 114-122. DOI:10.1111/j.1744-7429.2011.00769.x |
| [8] | Fochi V, Chitarra W, Kohler A, Voyron S, Singan VR, Lindquist EA, Barry KW, Girlanda M, Grigoriev IV, Martin F, Balestrini R, Perotto S. Fungal and plant gene expression in the Tulasnella calospora-Serapias vomeracea symbiosis provides clues about nitrogen pathways in orchid mycorrhizas. New Phytologist, 2017, 213(1): 365-379. DOI:10.1111/nph.14279 |
| [9] | Stöckel M, Těšitelová T, Jersáková J, Bidartondo MI, Gebauer G. Carbon and nitrogen gain during the growth of orchid seedlings in nature. New Phytologist, 2014, 202(2): 606-615. DOI:10.1111/nph.12688 |
| [10] |
Wang MN, Hu Y, Li HJ, Li J, Chen JB, Lan SR. New insights into orchids mycorrhizal fungi research. Guihaia, 2021, 41(4): 487-502.
(in Chinese) 王美娜, 胡玥, 李鹤娟, 李健, 陈建兵, 兰思仁. 兰科植物菌根真菌研究新见解. 广西植物, 2021, 41(4): 487-502. |
| [11] | Ercole E, Adamo M, Rodda M, Gebauer G, Girlanda M, Perotto S. Temporal variation in mycorrhizal diversity and carbon and nitrogen stable isotope abundance in the wintergreen meadow orchid Anacamptis morio. New Phytologist, 2015, 205(3): 1308-1319. DOI:10.1111/nph.13109 |
| [12] |
Gai XG, Xing XK, Guo SX. Ecological research of orchid mycorrhizae: a review. Mycosystema, 2014, 33(4): 753-767.
(in Chinese) 盖雪鸽, 邢晓科, 郭顺星. 兰科菌根的生态学研究进展. 菌物学报, 2014, 33(4): 753-767. |
| [13] | Cevallos S, Sánchez-Rodríguez A, Decock C, Declerck S, Suárez JP. Are there keystone mycorrhizal fungi associated to tropical epiphytic orchids?. Mycorrhiza, 2017, 27(3): 225-232. DOI:10.1007/s00572-016-0746-8 |
| [14] | del V., Leguina AC, Barrios AC, del M., Rosales Soro M, Lacosegliaz MJ, Pajot HF, Castellanos de Figueroa LI, Nieto-Peñalver CG. Copper alters the physiology of tomato rhizospheric isolates of Papiliotrema laurentii. Scientia Horticulturae, 2019, 243: 376-384. DOI:10.1016/j.scienta.2018.08.057 |
| [15] | 黄芳芳. 农药对三七根际土壤被孢霉的影响及其田间定殖能力. 云南大学硕士学位论文, 2017. |
| [16] | Leger RJS, Wang CS, Fang WG. New perspectives on insect pathogens. Fungal Biology Reviews, 2011, 25(2): 84-88. DOI:10.1016/j.fbr.2011.04.005 |
| [17] |
Li XJ, Feng XJ, Nong XQ, Cai N, Wang GJ, Zhou G, Tu XB, Zhang ZH. Effect of metarhizium anisopliae as a biocontrol agent on soil microbial community in peanut field. Chinese Journal of Biological Control, 2019, 35(6): 867-875.
(in Chinese) 李兴佳, 冯晓洁, 农向群, 蔡霓, 王广君, 周淦, 涂雄兵, 张泽华. 花生田施用绿僵菌对土壤微生物群落的影响. 中国生物防治学报, 2019, 35(6): 867-875. |
| [18] | Mitchell AM, Strobel GA, Moore E, Robison R, Sears J. Volatile antimicrobials from Muscodor crispans, a novel endophytic fungus. Microbiology, 2010, 156(1): 270-277. DOI:10.1099/mic.0.032540-0 |
| [19] | 陈心启. 国兰及其品种全书. 北京: 中国林业出版社, 2011: 1-42. |
| [20] |
Li J, Wang ZN, Kuang P, Zhang YJ. Isolation and phenotype identification of mycorrhizal fungi associated with wild Cymbidium plants. Subtropical Agriculture Research, 2013, 9(4): 254-257.
(in Chinese) 李杰, 王芝娜, 匡萍, 张银洁. 野生兰属植物菌根真菌的分离和表型鉴定. 亚热带农业研究, 2013, 9(4): 254-257. |
| [21] |
Hang F, Zhang CY. Effects of different sterilizations and media on the isolation of orchid mycorrhizal fungi. Journal of Biology, 2013, 30(5): 93-95.
(in Chinese) 黄芳, 张春英. 不同消毒和培养基条件对兰花菌根真菌分离的影响. 生物学杂志, 2013, 30(5): 93-95. DOI:10.3969/j.issn.2095-1736.2013.05.093 |
| [22] |
Yang QY, He CF, Liang LX, Wang T, Li LB, Liu L. Effect of mycorrhizal fungi on the plantlets growth of three species of orchid. Journal of Nuclear Agricultural Sciences, 2019, 33(4): 687-695.
(in Chinese) 杨前宇, 何聪芬, 梁立雄, 王涛, 李潞滨, 刘蕾. 菌根真菌对3种兰花幼苗生长作用研究. 核农学报, 2019, 33(4): 687-695. |
| [23] | Edwards J, Johnson C, Santos-Medellín C, Lurie E, Podishetty NK, Bhatnagar S, Eisen JA, Sundaresan V. Structure, variation, and assembly of the root-associated microbiomes of rice. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(8): E911-E920. DOI:10.1073/pnas.1414592112 |
| [24] | Rognes T, Flouri T, Nichols B, Quince C, Mahé F. VSEARCH: a versatile open source tool for metagenomics. PeerJ, 2016(4): e2584. |
| [25] | Haas BJ, Gevers D, Earl AM, Feldgarden M, Ward DV, Giannoukos G, Ciulla D, Tabbaa D, Highlander SK, Sodergren E, Methé B, DeSantis TZ, Consortium HM, Petrosino JF, Knight R, Birren BW. Chimeric 16S rRNA sequence formation and detection in Sanger and 454-pyrosequenced PCR amplicons. Genome Research, 2011, 21(3): 494-504. DOI:10.1101/gr.112730.110 |
| [26] | Edgar RC. UPARSE: highly accurate OTU sequences from microbial amplicon reads. Nature Methods, 2013, 10(10): 996-998. DOI:10.1038/nmeth.2604 |
| [27] | Nguyen NH, Song ZW, Bates ST, Branco S, Tedersoo L, Menke J, Schilling JS, Kennedy PG. FUNGuild: an open annotation tool for parsing fungal community datasets by ecological guild. Fungal Ecology, 2015(20): 241-248. |
| [28] |
Ai Y, Xie TX, Liu JF, Lan SR, Peng DH, Zhang QH. Community structure and biological function of the root symbiotic fungi of Arundina graminifolia. Mycosystema, 2019, 38(10): 1631-1642.
(in Chinese) 艾叶, 谢泰祥, 刘江枫, 兰思仁, 彭东辉, 张清华. 竹叶兰根系共生真菌群落结构及生物学功能初探. 菌物学报, 2019, 38(10): 1631-1642. |
| [29] | 周珊. 扇脉杓兰(Cypripedium japonicum Thunb. )内生和根际真菌多样性研究. 华东师范大学硕士学位论文, 2020. |
| [30] | Bunch WD, Cowden CC, Wurzburger N, Shefferson RP. Geography and soil chemistry drive the distribution of fungal associations in lady's slipper orchid, Cypripedium acaule. Botany, 2013, 91(12): 850-856. DOI:10.1139/cjb-2013-0079 |
| [31] | Shi LL, Mortimer PE, Ferry Slik JW, Zou XM, Xu JC, Feng WT, Qiao L. Variation in forest soil fungal diversity along a latitudinal gradient. Fungal Diversity, 2014, 64(1): 305-315. DOI:10.1007/s13225-013-0270-5 |
| [32] | Bahram M, Peay KG, Tedersoo L. Local-scale biogeography and spatiotemporal variability in communities of mycorrhizal fungi. New Phytologist, 2015, 205(4): 1454-1463. DOI:10.1111/nph.13206 |
| [33] | 邢来君, 李明春, 魏东盛. 普通真菌学. 第3版. 北京: 高等教育出版社, 2010: 84. |
| [34] | Rasmussen HN, Dixon KW, Jersáková J, Tĕšitelová T. Germination and seedling establishment in orchids: a complex of requirements. Annals of Botany, 2015, 116(3): 391-402. DOI:10.1093/aob/mcv087 |
| [35] |
Jiang SJ, Shi GX, Mao L, Pan JB, An LZ, Liu YJ, Feng HY. Comparison of different PCR primers on detecting arbuscular mycorrhizal communities inside plant roots. Acta Microbiologica Sinica, 2015, 55(7): 916-925.
(in Chinese) 蒋胜竞, 石国玺, 毛琳, 潘建斌, 安黎哲, 刘永俊, 冯虎元. 不同PCR引物在根系丛枝菌根真菌群落研究中的应用比较(英文). 微生物学报, 2015, 55(7): 916-925. |
| [36] | Taylor DL, Bruns TD. Population, habitat and genetic correlates of mycorrhizal specialization in the 'cheating' orchids Corallorhiza maculata and C. mertensiana. Molecular Ecology, 1998, 8(10): 1719-1732. |
| [37] | Lee YI, Yang CK, Gebauer G. The importance of associations with saprotrophic non-Rhizoctonia fungi among fully mycoheterotrophic orchids is currently under-estimated: novel evidence from sub-tropical Asia. Annals of Botany, 2015, 116(3): 423-435. DOI:10.1093/aob/mcv085 |
| [38] |
Li GJ, Li SF, Zhao D, Wen HA. Recent research progress of Russula (Russulales, Agaricomycetes): a review. Mycosystema, 2015, 34(5): 821-848.
(in Chinese) 李国杰, 李赛飞, 赵东, 文华安. 红菇属研究进展. 菌物学报, 2015, 34(5): 821-848. |
| [39] |
Lou JX, Liu H, Sheng SJ, Feng ZH, He DD, Wu SS, Chen W, Jiang GH, Chang PF, Peng GH. Effects of different agronomic measures on the structure and function of soil fungi community in tobacco fields. Chinese Tobacco Science, 2020, 41(1): 38-43, 55.
(in Chinese) 娄俊鑫, 刘泓, 沈少君, 冯芷菡, 何冬冬, 吴树松, 陈炜, 江桂花, 常鹏飞, 彭国华. 不同农艺措施对烟田土壤真菌群落结构和功能的影响. 中国烟草科学, 2020, 41(1): 38-43, 55. |
| [40] | Summerbell RC. From Lamarckian fertilizers to fungal castles: recapturing the pre-1985 literature on endophytic and saprotrophic fungi associated with ectomycorrhizal root systems. Studies in Mycology, 2005(53): 191-256. |
| [41] | Zhang KL, Bonito G, Hsu CM, Hameed K, Vilgalys R, Liao HL. Mortierella elongata increases plant biomass among non-leguminous crop species. Agronomy, 2020, 10(5): 754. DOI:10.3390/agronomy10050754 |
| [42] | Osorio NW, Habte M. Soil phosphate desorption induced by a phosphate-solubilizing fungus. Communications in Soil Science and Plant Analysis, 2014, 45(4): 451-460. DOI:10.1080/00103624.2013.870190 |
| [43] | Brabcová V, Nováková M, Davidová A, Baldrian P. Dead fungal mycelium in forest soil represents a decomposition hotspot and a habitat for a specific microbial community. New Phytologist, 2016, 210(4): 1369-1381. DOI:10.1111/nph.13849 |
| [44] |
Wang H, Lin QQ, Hu XJ, Wu GH, Xu Q. Screening of Anoectochilus roxburghii growth-promoting endophytic fungi and their promoting mechanism. Journal of Fujian Normal University (Natural Science Edition), 2019, 35(3): 72-79, 95.
(in Chinese) 王涵, 林清强, 胡雪娇, 武广珩, 徐庆. 金线莲内生促生真菌的筛选及其促生机制探讨. 福建师范大学学报(自然科学版), 2019, 35(3): 72-79, 95. |
| [45] | 蒋玉玲. 辽宁省内九种兰科植物菌根真菌多样性研究. 沈阳农业大学硕士学位论文, 2018. |
| [46] | Menkis A, Urbina H, James TY, Rosling A. Archaeorhizomyces borealis sp. nov. and a sequence-based classification of related soil fungal species. Fungal Biology, 2014, 118(12): 943-955. DOI:10.1016/j.funbio.2014.08.005 |
| [47] | Redecker D, Szaro TM, Bowman RJ, Bruns TD. Small genets of Lactarius xanthogalactus, Russula cremoricolor and Amanita francheti in late-stage ectomycorrhizal successions. Molecular Ecology, 2001, 10(4): 1025-1034. DOI:10.1046/j.1365-294X.2001.01230.x |
| [48] | Taylor DL, Bruns TD. Community structure of ectomycorrhizal fungi in a Pinus muricata forest: minimal overlap between the mature forest and resistant propagule communities. Molecular Ecology, 1999, 8(11): 1837-1850. DOI:10.1046/j.1365-294x.1999.00773.x |
 2021, Vol. 61
2021, Vol. 61




