中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 高利利, 王聪聪, 杨帆, 曹伟军, 张向乐, 刘湘涛, 朱紫祥, 郑海学. 2021
- Lili Gao, Congcong Wang, Fan Yang, Weijun Cao, Xiangle Zhang, Xiangtao Liu, Zixiang Zhu, Haixue Zheng. 2021
- CRISPR/Cas9介导的RPSA基因敲除细胞系的建立及其应用
- Construction of RPSA gene knockout BHK21 cell line using the CRISPR/Cas9 system
- 微生物学报, 61(7): 1945-1956
- Acta Microbiologica Sinica, 61(7): 1945-1956
-
文章历史
- 收稿日期:2020-06-21
- 修回日期:2020-08-18
- 网络出版日期:2021-06-15
基因组编辑(genome editing)是对基因组进行精确修饰和定向编辑的一种技术,是指通过人为操作在基因组特定碱基位置引入插入、缺失或DNA序列替换等修饰的方法,从而实现对编辑受体基因组序列的突变,最终达到改变基因的结构或功能的目的。最初的基因组编辑技术是利用基因枪、显微注射或者电转的方法将外源供体的遗传物质导入细胞中,借助自身的修复系统,通过同源重组方法将导入的DNA片段插入到细胞基因组中,进而完成基因的转移或修饰。但这些早期技术因插入效率较低、打靶定点编辑能力差等缺点,使其应用以及推广受到了很大程度的限制。伴随着人工核酸内切酶在基因组编辑中逐步得以应用,基于锌指核酸酶编辑技术(zinc finger nucleases,ZFNs)和基于植物病原体-黄单胞菌分泌的一种转录激活子样效应因子而建立的类转录激活因子效应物核酸酶编辑技术(transcription activator-like effector nucleases,TALENs)成为更为简便和高效的基因组编辑技术[1–2]。ZFNs和TALENs技术在基因编辑精准性及编辑效率等方面和早期的物理打靶技术相比有了大幅度提升,但是这两种技术在使用过程中由于操作难度大,载体构建复杂、实验周期冗长、成本高和结果难控等弊端也逐步限制了其广泛应用[3]。
CRISPR/Cas9基因编辑技术是利用小分子RNA介导的一种靶向基因组编辑技术,是继ZFNs和TALENs技术之后出现的第三代新型基因编辑技术[3–4]。该技术是基于广泛存在于古细菌及细菌中的一种免疫系统经由人工改造开发建立[5]。CRISPR/Cas9技术通过导向RNA序列在靶标基因特定位点进行切割形成DNA双链断裂[6–7],进而诱发细胞固有的同源重组和非同源末端连接的自我修复过程,实现对基因组特定靶标位点进行编辑或修饰[8]。该技术使用便捷,重复性好,而且合成简单、周期短、插入效率高、操作灵活,能够实现基因精准编辑,而且近年来通过逐步升级改造使得其实用性不断提升[9–10],已在医学、药物和生命科学等多领域得以广泛应用[11–13]。
宿主核糖体蛋白SA (ribosomal protein SA,RPSA)是核糖体亚基的RPSA的组成部分,也被称为层粘连蛋白受体1[14–15],在细胞中广泛表达,并且在各种细胞反应中起着多种作用。RPSA参与RNA合成[16]、细胞迁移[17]、血管生成[18]和脾脏发育等生物学反应过程[18]。同时,分布于细胞表面的RPSA能够被登革热病毒、猪瘟病毒和委内瑞拉马脑脊髓炎病毒利用作为受体感染靶标细胞[19–20]。在口蹄疫病毒感染宿主细胞后,RPSA可抑制MAPK通路,从而抑制病毒的复制[21]。因此,RPSA与病毒在感染细胞中存在着重要关联,并且在不同病毒感染过程中RPSA可能发挥着不同的作用。我国于2015年鉴定了1种新发的猪水泡病病原塞内卡病毒(Senecavirus A,SVA),其感染引起的临床症状与口蹄疫、猪泡状病、泡状口炎或泡状疹的临床症状难以区分[22]。至今关于SVA感染致病机制仍不清楚,有待深入阐明。为了研究SVA感染过程中,宿主RPSA蛋白是否能够调控病毒的复制,本研究利用CRISPR/Cas9技术建立RPSA基因敲除细胞系,为深入研究SVA致病机制提供试验材料。
1 材料和方法 1.1 细胞、病毒及载体BHK21细胞由本实验室提供。细胞培养使用含有10%胎牛血清和1%双抗的DMEM培养基进行培养。塞内卡病毒CH-FJ-2017毒株由中国农业科学院兰州兽医研究所口蹄疫与新发病流行病学团队分离并保存。PX330载体为中国农业大学赵要风教授惠赠。
1.2 试剂及抗体大肠杆菌Trans5α感受态、LA Taq DNA聚合酶、限制性核酸内切酶Kpn I和Xba I、T4 DNA连接酶、Trizol试剂、TB GreenⅡ购自宝生物工程大连有限公司。Trizol试剂、蛋白预染Marker和Lipofectamine 3000转染试剂购自Invitrogen公司。Opti-MEM、0.25% EDTA胰酶和新生牛血清(FBS)均购自Gibco公司。DMEM细胞培养液和PBS溶液购自Hyclone公司。胶回收试剂盒、微量DNA提取试剂盒购自OMEGA公司。大提质粒试剂盒购自MACHEREY-NAGEL公司。RPSA抗体(Laminin Receptor 1)购自Abcam,羊抗兔IgG (IgG-HRP)购自Invitrogen公司。塞内卡病毒VP2兔抗多抗由本实验室制备保存。
1.3 sgRNA靶点的设计在NCBI中查询RPSA基因序列,并找到仓鼠全基因组,定位RPS在基因组中不同转录本的重叠区第一个外显子,通过http://tolls.genomeengineering.org (http://crispr.mit.edu/)网站设计sgRAN,根据评分设计20 bp的引导RNA (sgRNA),并在sgRNA序列5′端加CACC/AAAC粘性末端,共选取了评分较高的4对sgRNA序列,分别命名为BHK-sgRNA-1、BHK-sgRNA-2、BHK-sgRNA-3、BHK-sgRNA-4。sgRNA序列见表 1。
| SgRNA names | Sequences (5'→3') |
| sgRNA1 | F: CACCGCTCAATAGCAACGATGGCCC |
| R: AAACGGGCCATCGTTGCTATTGAGC | |
| sgRNA2 | F: CACCGCATCGTTGCTATTGAGAACC |
| R: AAACGGTTCTCAATAGCAACGATGC | |
| sgRNA3 | F: CACCGTCTCAATAGCAACGATGGCC |
| R: AAACTCGTTGCTATTGAGAACCCGC | |
| sgRNA4 | F: CACCGTCTCAATAGCAACGATGGCC |
| R: AAACGGCCATCGTTGCTATTGAGAC |
1.4 PX330-sgRNA重组表达质粒构建
PX330-sgRNA重组表达质粒构建步骤:(1) sgRNA的退火。将sgRNA上下游序列合成产物稀释至100 μmol/L后,取上下游引物各2.5 μL,并加入ddH2O 4 μL、Taq buffer I 1 μL,共10 μL体系按照0.3 ℃/s梯度降温进行退火处理:95 ℃ 3 min;95 ℃ 1 min,85 ℃ 1 min,75 ℃ 1 min,65 ℃ 1 min,55 ℃ 1 min,45 ℃ 1 min,35 ℃ 1 min,25 ℃ 1 min,16 ℃ 1 h,使上下游引物形成双链。(2) 利用BBSI内切酶酶切PX330载体。反应体系如下:PX330载体5 μL,BBSI 1 μL、10×buffer 2 μL、ddH2O 12 μL,共20 μL体系;37 ℃、2 h酶切。转化回收酶切片段。(3) 酶切载体与sgRNA的连接。T4连接酶1 μL、10×T4连接酶buffer 1 μL、PX330酶切片段1.5 μL、退火后的sgRNA各6.5 μL,共10 μL体系。16 ℃过夜连接。(4) PX330-sgRNA连接产物转化。将连接产物转化Trans5α感受态细胞,提取质粒,送样测序。
1.5 细胞的转染在转染前复苏BHK21细胞于T25细胞瓶中,使用含有10%的FBS、1%双抗的DMEM培养基培养,当细胞传代2–3次性状稳定且状态较好时,将细胞消化后铺于细胞板中,待细胞融合度至70%–80%时,将质粒与Lipofectamine 3000 (按照比例1 μg: 2 μL)分别加至Opti-MEM中,静止15 min后将两者混合;将脂质体-质粒复合物混合静置5 min后直接加至细胞培养基中;将细胞重新置于37 ℃、5% CO2培养箱中培养48 h。
1.6 细胞DNA的提取及T7EI实验按照微量DNA提取试剂盒的操作说明提取细胞DNA,使用check引物进行扩增,对扩增出的目的片段进行胶回收,并按照上述的退火体系和程序进行变性再退火。取再退火DNA产物8 μL、T7核酸内切酶1 μL和相应buffer 1 μL,共10 μL体系,按照T7核酸内切酶说明书推荐酶切温度和时间进行酶切,使用10×loading buffer终止反应后,使用1%琼脂糖胶进行鉴定其是否具有切割片段,分析切割效率。设计check引物进行检测,引物序列为Check-F:5′-GTCTGCCT TCCTGTAGTGTCCT-3′;Check-R:5′-CAACCTA ACTCAGCCAGCCTAT-3′。
1.7 有限稀释法T7EI实验中具有切割条带的细胞进行有限稀释挑选单克隆细胞。利用0.25%胰酶消化细胞,轻拍脱落后,加入培养基吹打均匀;留取100 μL细胞悬液用于后续测序使用以确定打靶作用。剩余细胞使用无血清培养基将细胞稀释至1×103/mL,再使用5% FBS培养基稀释到10个细胞/mL,即每0.1 mL培养基1个细胞;使用100 μL排枪将稀释后细胞悬液每孔0.1 mL加入96孔板中;培养4–5 d后观察,除去双细胞孔、多细胞孔,对细胞形态正常且单一细胞孔标记;继续培养7–9 d,将96孔中细胞传代转入48孔板继续培养,待细胞长满后依次传至24孔板、6孔板进行放大培养。
1.8 Western blot蛋白检测取野生型细胞与各个候选单克隆细胞株分别进行培养,待细胞长满后,收取细胞放置冰上。加入适量1×SDS loading buffer充分搅拌裂解,待细胞全部脱落后吸取至EP管中,做好标记;金属浴变性10 min,4 ℃离心后,取上清进行SDS-PAGE。电泳后通过湿转法转印至NC膜上,转印后使用5%的脱脂奶粉封闭,用购买的67 kDa laminin为一抗,羊抗兔IgG (IgG-HRP)为二抗,进行抗原抗体复合物检测,对RPSA基因敲除的BHK21单细胞系在蛋白表达水平上进行验证。
1.9 Sanger测序鉴定取野生型细胞与各个候选单克隆细胞株分别进行培养,待细胞长满后,收取细胞放置冰上。按照细胞微量DNA提取试剂盒说明书操作提取DNA,使用check引物扩增含有sgRNA靶向位点的片段,胶回收纯化后送样进行测序鉴定。将返回结果中显示为套峰的片段,或无套峰、但发生了编辑的样品,进行进一步分析,将扩增片段连接T载体,进行连接转化,分别随机挑取5个单克隆菌进行再次测序鉴定。
1.10 细胞接毒感染将正常传代2–3代的野生型BHK21和RPSA基因敲除细胞分别铺于培养板中,待其密度长至90%时,使用无血清培养基润洗细胞,洗去残留血清;用无血清培养基将病毒稀释至适当浓度,充分混匀后加入细胞;37 ℃、5% CO2细胞培养箱中孵育1 h,去掉含有病毒的培养基,更换为含有1%血清的维持液,放置于培养箱中继续培养。
1.11 Trizol法提取RNA及反转录收取细胞样品(如6孔细胞培养板),加入1 mL Trizol,反复吹打使得细胞充分裂解,室温静置5 min,将裂解细胞液转入EP管中;在上述EP管中加入250 μL氯仿,振荡30 s,4 ℃放置15 min,4 ℃、12000 r/min离心15 min;取450 μL上清(勿吸取下层沉淀)到新的无RNase的1.5 mL EP管中,并加入等量的异丙醇以沉淀RNA,–20 ℃放置30 min,4 ℃、12000 r/min离心15 min,弃去上清;加入1 mL的75%乙醇,轻轻上下颠倒3次,4 ℃、12000 r/min离心5 min,弃去上清,将EP管倒置于超净台中晾干;加入适量DEPC水溶解RNA,并测定浓度,放置于–80 ℃保存。取5×HiScript Ⅱ qRT SuperMix Ⅱ 4 μL,RNA 4 μL,ddH2O 12 μL,共20 μL体系,按照50 ℃ 15 min,85 ℃ 2 min,16 ℃ 10 s进行反转录。
1.12 荧光定量PCR (TB GreenⅡ法)收集细胞样品,提取总RNA,反转录获得cDNA。按照TB GreenⅡ Premix Ex Taq 10 μL,上下游引物各1 μL,cDNA 1 μL,ddH2O 7 μL,共10 μL的体系配置预混反应,并按照CFX96TM real-time system操作流程,95 ℃ 3 min;95 ℃ 10 s,60 ℃ 34 s,共40个循环进行荧光定量PCR反应,利用GAPDH作为内参进行不同基因的mRNA的相对定量。利用t-test检验对不同处理组病毒RNA复制水平差异进行统计学分析,“**”表示P < 0.01,统计学分析差异极显著。
2 结果和分析 2.1 sgRNA重组表达质粒的鉴定将PX330酶切产物与sgRNA连接转化,提取质粒后,使用PX330载体引物和相应的sgRNA分别作为上下游引物进行PCR扩增鉴定,且将有条带的质粒送测序。使用BLAST软件对测序结果与目的基因序列进行对比,成功构建具有靶向不同目的基因外显子的sgRNA重组质粒,分别命名为:PX330-RPSA-sgRNA1、PX330-RPSA-sgRNA2、PX330-RPSA-sgRNA3、PX330-RPSA-sgRNA4。重组质粒模式结构见图 1。并使用1%琼脂糖核酸胶对质粒大小进行鉴定,结果见图 2,其大小符合预期结果。表明重组质粒构建成功。
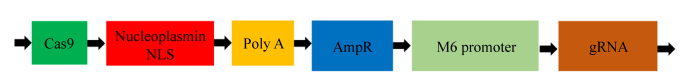
|
| 图 1 PX330-RPSA-sgRNA重组质粒模式图 Figure 1 The model of PX330-RPSA-sgRNA recombinant plasmid. |
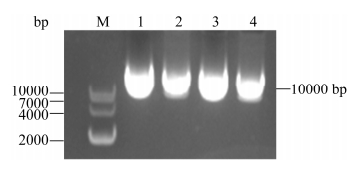
|
| 图 2 PX330-RPSA-sgRNA重组质粒的核酸电泳检测 Figure 2 Identification of PX330-RPSA-sgRNA recombinant plasmid. M: DNA maker; 1: PX330- RPSA-sgRNA1; 2: PX330-RPSA-sgRNA2; 3: PX330- RPSA-sgRNA3; 4: PX330-RPSA-sgRNA4. |
2.2 不同RPSA基因缺失细胞株的筛选和鉴定 2.2.1 不同sgRNA酶切效率鉴定:
将构建成功的4种PX330-sgRNA重组质粒分别转染BHK21细胞,48 h后收取细胞样品并提取细胞基因组DNA。使用sgRNA check引物进行PCR扩增。通过对DNA目的片段胶回收后,进行变性再退火,使用T7核酸内切酶检测切割效应和切割效率,结果如图 3所示,4对sgRNA均有酶切条带。

|
| 图 3 不同sgRNA酶切效率的检测分析 Figure 3 The analysis of the editing efficiency of each sgRNA. A: the RPSA gene fragments amplified using the check primer. M: DNA maker; Co: WT BHK21; lane 1: PX330-RPSA-sgRNA1; lane 2: PX330-RPSA-sgRNA2; lane 3: PX330-RPSA-sgRNA3; lane 4: PX330-RPSA-sgRNA4; B: the electrophoresis analysis of the re-annealed DNA products digested by the T7E1 endonuclease. M: DNA Maker; Co: negative control; lane 1: PX330- RPSA-sgRNA1; lane 2: PX330-RPSA-sgRNA2; lane 3: PX330-RPSA-sgRNA3; lane 4: PX330-RPSA-sgRNA4. |
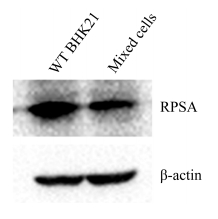
选取酶切效率较高的PX330-RPSA-sgRNA2进行进一步后续实验。将PX330-RPSA-sgRNA2转染细胞中扩增的片段送样进行DNA测序,结果如图 4所示,测序峰图中出现了大量的套峰,表明扩增片段中发生了有效的基因编辑,sgRNA2成功发挥了打靶效应。同时对PX330-RPSA- sgRNA2转染的细胞收样,利用Western blot检测RPSA表达情况,结果如图 5所示,RPSA-sgRNA2转染后,RPSA的表达量显著下降,进一步证实sgRNA2成功发挥编辑效应。
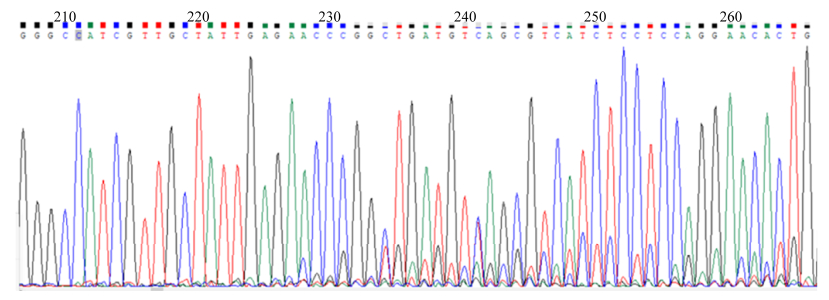
|
| 图 4 PX330-RPSA-sgRNA2转染BHK21细胞后,check引物扩增RPSA基因片段测序峰图 Figure 4 The sequencing data of the RPSA fragment amplified by the check primers from the PX330-RPSA-sgRNA2-transfected BHK21 cells. |
|
| 图 5 转染PX330-RPSA-sgRNA2能够降低BHK21细胞RPSA的表达 Figure 5 Transfection of PX330-RPSA-sgRNA2 decreased RPSA expression in BHK21 cells. |
2.2.2 RPSA基因敲除单克隆细胞候选株的筛选:
利用有限稀释法将PX330-RPSA-sgRNA2转染后的细胞接种到96孔细胞培养板中,正常培养7–9 d,去除双细胞孔、多细胞孔,仅挑选标记单克隆细胞群并进行后续的扩大培养。经过有限稀释法筛选出的10株候选单克隆细胞分别标记为#1、#2、#3、#4、#5、#6、#7、#8、#9、#10。如图 6所示为培养8 d后的部分单克隆细胞团。

|
| 图 6 有限稀释法获取的单克隆细胞团 Figure 6 Screening the single-cell clones by the limited dilution method. A: single-cell clone #1; B: single-cell clone #3; C: single-cell clone #6; D: single-cell clone #7. |
2.2.3 不同单克隆细胞株基因水平的鉴定:
经过扩大培养的细胞,收取单克隆细胞样品进行Western blot验证,其结果如图 7所示,与转染空载体的野生型BHK21相比,#1、#6、#7细胞株中RPSA蛋白的表达发生了缺失。

|
| 图 7 RPSA蛋白在候选基因敲除细胞中表达的Western blot检测 Figure 7 Detection of RPSA expression in the candidate KO cell lines by Western blot. |
2.2.4 不同单克隆细胞株RPSA基因编辑效果检测:
不同单克隆细胞的DNA为模板,使用check引物进行目的条带的扩增,条带单一且明亮,回收#1、#6、#7后进行测序。结果如图 8所示,#1、#7等结果显示为套峰,#6无套峰,但发生了基因编辑。将扩增的#1、#6、#7目的片段,连接T载体,转化后随机挑选5个单克隆菌落,提取质粒再次进行测序鉴定。使用MegAlign对测序结果进行分析,证实其基因组发生移码突变,结合蛋白水平结果,成功筛选出#6细胞株为RPSA基因敲除细胞株。

|
| 图 8 #1、#6、#7细胞系check引物扩增RPSA基因片段测序峰图 Figure 8 The sequencing data of the RPSA fragment amplified by the check primers from the #1, #6 and #7 BHK21 KO cells. |
2.3 RPSA基因敲除细胞系的初步应用 2.3.1 敲除RPSA抑制SVA的复制:
在获得RPSA基因敲除细胞系后,首先测定比较了基因敲除细胞系的生长速率和状态与野生型BHK21细胞的差异,细胞生长曲线、细胞活性测定结果表明在BHK-21细胞中敲除RPSA没有显著影响细胞的正常生长特性(图 9-A–B)。因此,本研究进一步利用建立的细胞系初步开展了RPSA对SVA复制调控影响的研究。使用SVA感染野生型BHK21细胞和#6 RPSA基因敲除细胞,结果显示RPSA基因缺失后,SVA的复制水平显著下降。结果如图 9-C与9-D所示,表明RPSA对SVA的复制具有重要的调控作用,将RPSA敲除后,SVA的复制能力受到严重影响。

|
| 图 9 敲除RPSA能够显著抑制SVA在BHK21细胞中的复制 Figure 9 Knockout of RPSA in BHK21 cells considerably inhibited SVA replication. A: the growth curve of wild-type BHK21 and #6 RPSA KO cells. Same amount of WT or KO cells were seeded equally in the 6-Well culturing plates. The cell number was calculated at 0, 12, 24 and 48 h respectively. B: the cell viability was measured at 0, 12, 24 and 48 h respectively. C: the wild-type BHK21 and #6 RPSA KO cells were infected with SVA for 0, 4, 8, 12, 16 or 24 h, and the expression of viral VP2 protein was detected by Western blotting at the indicated time point.; D: the wild-type BHK21 and #6 RPSA KO cells were infected with SVA for 0, 6 or 12 h, the total RNA was extracted and the SVA transcripts were detected by qPCR. The error bar indicated mean±SD: ** P < 0.01 (highly statistically significant). |
2.3.2 RPSA激活ERK通路调控SVA复制:
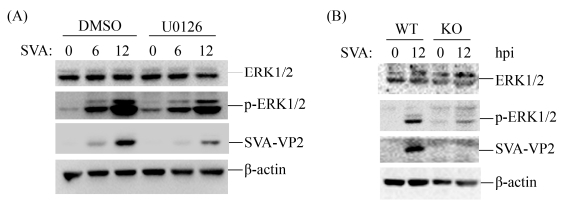
RPSA能够调控MAPK通路参与肿瘤形成,而且RPSA能够靶向MAPK通路调控口蹄疫病毒复制[21]。为了研究RPSA在SVA复制过程中是否能够靶向MAPK发挥调控作用,本研究首先研究了MAPK通路的活化是否影响SVA的复制,利用SVA分别感染BHK-21细胞及MAPK通路抑制剂Μ0126处理的BHK-21细胞,结果表明SVA复制能够激活MAPK通路的活化(ERK1/2发生了磷酸化),而利用Μ0126抑制MAPK通路激活后,SVA的复制明显被抑制(图 10-A),表明MAPK通路的活化能够促进SVA的复制,抑制MAPK通路活化能够降低SVA的复制水平。我们进一步研究了RSPA敲除后MAPK通路的活化情况,结果发现RPSA基因缺失后,MAPK通路活化显著下降,SVA复制明显受抑制(图 10-B)。因此,研究结果表明RSPA能够靶向MAPK通路活化调控SVA复制,敲除RPSA抑制了MAPK通路的活化,进而导致SVA复制能力的下降。
|
| 图 10 敲除RPSA能够抑制MAPK通路的活化,抑制SVA的复制 Figure 10 Knockout of RPSA in BHK21 cells inhibited the activation of MAPK pathway, resulting in the decreased replication of SVA. A: wild-type BHK21 were pretreated with DMSO (solvent control) or 20 μmol/L of U0126 for 1 h and then infected with SVA for 0, 6, 12 h. The cells were then lysed and subjected to Western blotting analysis. The expression levels of ERK1/2, p-ERK1/2, SVA VP2 were detected. B: wild-type BHK21 and #6 RPSA KO cells were infected with SVA for 0, 12 h. The cells were then lysed and subjected to Western blotting analysis. The expression of ERK1/2, p-ERK1/2, SVA VP2 were detected by anti-ERK1/2, anti-p-ERK1/2 and anti-SVA VP2 antibodies respectively. |
3 讨论
本研究采用CRISPR/Cas9技术建立RPSA基因敲除的BHK21细胞株,为进一步建立RPSA其他基因敲除细胞系提供了参考。RPSA基因敲除细胞系的建立过程中构建了PX330-sgRNA重组质粒,转染BHK21细胞后,经有限稀释法得到备选单克隆细胞株,通过蛋白、基因水平检测鉴定,成功获得了RPSA基因敲除细胞系。在筛选基因敲除细胞株过程中仅通过有限稀释法进行盲筛,未使用任何抗生素进行筛选,细胞生长曲线和细胞活性测定结果表明所得的基因敲除细胞系生长速率和状态与野生型BHK21细胞基本相同,最大程度保持了细胞的正常生理性状。为在RPSA基因敲除细胞系中进一步通过抗性筛选进行多基因敲除奠定基础。同时,在利用CRISPR/Cas9技术进行实际操作过程中,对转染方式、试剂和剂量,有限稀释法等一系列的操作和方法进行了摸索,总结了有效的实验操作流程,提高了敲除细胞系的建立效率。
本实验中利用RPSA敲除细胞系,在正常传代培养后开展SVA感染实验,与正常野生型BHK21细胞相比,我们发现病毒复制水平大幅度下降。表明敲除RPSA基因能够限制SVA在细胞中的复制,表明RPSA对SVA的复制具有重要的调控作用,其可以作为抑制SVA感染复制的一个重要靶点。而进一步研究表明RPSA能够靶向MAPK通路活化调控SVA的复制。
RPSA为高度保守的核糖体蛋白质,其不仅在多种细胞中广泛表达,还参与细胞迁移[16]、血管生成[18]和脾脏发育[23]等,具有多形性、多位点和多功能的特点。RPSA可作为消化道结合外源性致病朊蛋白的候选蛋白,参与朊病毒蛋白的结合和内化[14]。在肿瘤细胞扩散中,RPSA参与丝裂原活化蛋白激酶(MAPK)信号通路的相关调节[17]。同时RPSA是登革热病毒进入肝脏细胞的血清型特异性受体,也是猪瘟病毒感染过程中进入宿主细胞的细胞附着受体。RPSA也被称为37 kDa层粘连蛋白受体前体/67 kDa层粘连蛋白受体[20, 24–25]。其是否是SVA感染宿主细胞的一个潜在受体,有待进一步研究。我们将利用成功建立的RPSA基因敲除细胞系对RPSA是否作为SVA感染其宿主细胞的潜在受体进行进一步鉴定,深入开展RPSA调控SVA复制的分子机制,挖掘RPSA蛋白的功能。
综上所述,我们利用CRISPR/Cas9基因编辑技术实现了RPSA基因的高效编辑,为其他基因敲除细胞系的建立提供了参考。RPSA基因敲除细胞系的建立能够为RPSA分子功能的深入研究提供实验材料,RPSA调控SVA复制的研究能够为进一步开展RSPA调控其他病毒复制的机制研究提供思路。
| [1] | Joung JK, Sander JD. TALENs: a widely applicable technology for targeted genome editing. Nature Reviews Molecular Cell Biology, 2013, 14(1): 49-55. DOI:10.1038/nrm3486 |
| [2] | Kianianmomeni A. Genome editing using engineered zinc finger nucleases. Clinical Biochemistry, 2011, 44(S13): S28. |
| [3] | Zhang F, Wen Y, Guo X. CRISPR/Cas9 for genome editing: progress, implications and challenges. Human Molecular Genetics, 2014, 23(R1): R40-R46. DOI:10.1093/hmg/ddu125 |
| [4] | Cong L, Ran FA, Cox D, Lin SL, Barretto R, Habib N, Hsu PD, Wu XB, Jiang WY, Marraffini LA, Zhang F. Multiplex genome engineering using CRISPR/Cas systems. Science, 2013, 339(6121): 819-823. DOI:10.1126/science.1231143 |
| [5] | Ishino Y, Shinagawa H, Makino K, Amemura M, Nakata A. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product. Journal of Bacteriology, 1987, 169(12): 5429-5433. DOI:10.1128/jb.169.12.5429-5433.1987 |
| [6] | Plagens A, Richter H, Charpentier E, Randau L. DNA and RNA interference mechanisms by CRISPR-Cas surveillance complexes. FEMS Microbiology Reviews, 2015, 39(3): 442-463. DOI:10.1093/femsre/fuv019 |
| [7] | Christian M, Cermak T, Doyle EL, Schmidt C, Zhang F, Hummel A, Bogdanove AJ, Voytas DF. Targeting DNA double-strand breaks with TAL effector nucleases. Genetics, 2010, 186(2): 757-761. DOI:10.1534/genetics.110.120717 |
| [8] | Bolotin A, Quinquis B, Sorokin A, Ehrlich DS. Clustered regularly interspaced short palindrome repeats (CRISPRs) have spacers of extrachromosomal origin. Microbiology, 2005, 151(8): 2551-2561. DOI:10.1099/mic.0.28048-0 |
| [9] | Sternberg SH, Haurwitz RE, Doudna JA. Mechanism of substrate selection by a highly specific CRISPR endoribonuclease. RNA, 2012, 18(4): 661-672. DOI:10.1261/rna.030882.111 |
| [10] | Horii T, Hatada I. Genome engineering using the CRISPR/Cas system. World Journal of Medical Genetics, 2014, 4(3): 69-76. DOI:10.5496/wjmg.v4.i3.69 |
| [11] | Yao SH, He ZY, Chen C. CRISPR/Cas9-mediated genome editing of epigenetic factors for cancer therapy. Human Gene Therapy, 2015, 26(7): 463-471. DOI:10.1089/hum.2015.067 |
| [12] | Karimian A, Azizian K, Parsian H, Rafieian S, Shafiei-Irannejad V, Kheyrollah M, Yousefi M, Majidinia M, Yousefi B. CRISPR/Cas9 technology as a potent molecular tool for gene therapy. Journal of Cellular Physiology, 2019, 234(8): 12267-12277. DOI:10.1002/jcp.27972 |
| [13] | Swiech L, Heidenreich M, Banerjee A, Habib N, Li YQ, Trombetta J, Sur M, Zhang F. In vivo interrogation of gene function in the mammalian brain using CRISPR-Cas9. Nature Biotechnology, 2015, 33(1): 102-106. DOI:10.1038/nbt.3055 |
| [14] | Knorr C, Beuermann C, Beck J, Brenig B. Characterization of the porcine multicopy ribosomal protein SA/37-kDa laminin receptor gene family. Gene, 2007, 395(1/2): 135-143. |
| [15] | Ben-shem A, de Loubresse NG, Melnikov S, Jenner L, Yusupova G, Yusupov M. The structure of the eukaryotic ribosome at 3.0Å resolution.. Science, 2011, 334(6062): 1524-1529. DOI:10.1126/science.1212642 |
| [16] | O'Donohue MF, Choesmel V, Faubladier M, Fichant G, Gleizes PE. Functional dichotomy of ribosomal proteins during the synthesis of mammalian 40S ribosomal subunits. Journal of Cell Biology, 2010, 190(5): 853-866. DOI:10.1083/jcb.201005117 |
| [17] | Rea VEA, Rossi FW, de Paulis A, Ragno P, Selleri C, Montuori N. 67 kDa laminin receptor: structure, function and role in cancer and infection. Le Infezioni in Medicina, 2012, 20(Suppl 2): 8-12. |
| [18] | Bernard A, Gao-li J, Franco CA, Bouceba T, Huet A, Li ZL. Laminin receptor involvement in the anti-angiogenic activity of pigment epithelium-derived factor. The Journal of Biological Chemistry, 2009, 284(16): 10480-10490. DOI:10.1074/jbc.M809259200 |
| [19] | Brassart B, da Silva J, Donet M, Seurat E, Hague F, Terryn C, Velard F, Michel J, Ouadid-Ahidouch H, Monboisse JC, Hinek A, Maquart FX, Ramont L, Brassart-Pasco S. Tumour cell blebbing and extracellular vesicle shedding: key role of matrikines and ribosomal protein SA. British Journal of Cancer, 2019, 120(4): 453-465. DOI:10.1038/s41416-019-0382-0 |
| [20] | Ould-Abeih MB, Petit-Topin I, Zidane N, Baron B, Bedouelle H. Multiple folding states and disorder of ribosomal protein SA, a membrane receptor for laminin, anticarcinogens, and pathogens. Biochemistry, 2012, 51(24): 4807-4821. DOI:10.1021/bi300335r |
| [21] | Zhu ZX, Li WW, Zhang XL, Wang CC, Gao LL, Yang F, Cao WJ, Li KL, Tian H, Liu XT, Zhang KS, Zheng HX. Foot-and-Mouth disease virus capsid protein VP1 interacts with host ribosomal protein sa to maintain activation of the MAPK signal pathway and promote virus replication. Journal of Virology, 2020, 94(3): e01350-19. |
| [22] | Zhu Z, Yang F, Chen P, Liu H, Cao W, Zhang K, Liu X, Zheng H. Emergence of novel Seneca valley virus strains in China, 2017. Transboundary and Emerging Diseases, 2017, 64(4): 1024-1029. DOI:10.1111/tbed.12662 |
| [23] | Bolze A, Mahlaoui N, Byun M, Turner B, Trede N, Ellis SR, Abhyankar A, Itan Y, Patin E, Brebner S, Sackstein P, Puel A, Picard C, Abel L, Quintana-Murci L, Faust SN, Williams AP, Baretto R, Duddridge M, Kini U, Pollard AJ, Gaud C, Frange P, Orbach D, Emile JF, Stephan JL, Sorensen R, Plebani A, Hammarstrom L, Conley ME, Selleri L, Casanova JL. Ribosomal protein SA haploinsufficiency in humans with isolated congenital asplenia. Science, 2013, 340(6135): 976-978. DOI:10.1126/science.1234864 |
| [24] | Chen JN, He WR, Shen L, Dong H, Yu JH, Wang X, Yu SX, Li YF, Luo YZ, Sun Y, Qiu HJ. The laminin receptor is a cellular attachment receptor for classical swine fever virus. Journal of Virology, 2015, 89(9): 4894-4906. DOI:10.1128/JVI.00019-15 |
| [25] | Thepparit C, Smith DR. Serotype-specific entry of dengue virus into liver cells: identification of the 37-kilodalton/67-kilodalton high-affinity laminin receptor as a dengue virus serotype 1 receptor. Journal of Virology, 2004, 78(22): 12647-12656. DOI:10.1128/JVI.78.22.12647-12656.2004 |
 2021, Vol. 61
2021, Vol. 61




