中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 赵燕妮, 李悦, 安靖, 刘欢, 王希越. 2021
- Yanni Zhao, Yue Li, Jing An, Huan Liu, Xiyue Wang. 2021
- 代谢组学技术在微藻研究中的应用
- Application of metabolomics approaches in microalgae research
- 微生物学报, 61(7): 1799-1815
- Acta Microbiologica Sinica, 61(7): 1799-1815
-
文章历史
- 收稿日期:2020-07-13
- 修回日期:2020-08-20
- 网络出版日期:2021-01-28
2. 吉林化工学院化学与制药工程学院, 吉林 吉林 132022
2. College of Chemistry and Pharmaceutical Engineering, Jilin Institute of Chemical Technology, Jilin 132022, Jilin Province, China
代谢组学(metabolomics/metabonomics)是通过分析生物体受外部环境刺激、病理生理或基因变异等因素引起的内源性小分子代谢物变化来研究其生理功能与代谢之间关系的一门科学[1-3]。它是继基因组学(genomics)、转录组学(transcriptomics)和蛋白质组学(proteomics)之后系统生物学的另一个重要分支[2],可以对生物体内的小分子代谢物(≤1500 Da)进行全面、整体的定性和(半)定量分析。这些小分子物质是细胞代谢活动的最终执行者,它们的代谢变化可以直接反映生物体的状态,所以相对于转录组学和蛋白质组学,代谢组学的研究可更直接、准确地揭示细胞的生理状态[4]。
代谢组学发展初期,研究工作主要围绕基础分析方法与组学研究策略的建立。近年来,针对生命科学领域的研究产生了各种问题,例如新型分析仪器设备的发展、复杂代谢物的分离分析、海量数据的处理等,代谢组学的研究技术与方法的不断发展,反过来又极大推动了以解决实际问题为目标的代谢组学应用研究的蓬勃发展。目前,代谢组学研究已被广泛地应用到生命科学的各个领域,如医药学、微生物学、环境毒理学、食品科学和植物学等[5-8]。
微藻是一类含有光合色素(叶绿素a)可进行光合作用的微小生物的总称,具有光合利用率高、生长速度快、环境适应性强和生物质含量高等特点[9]。相比于代谢组学在动物和植物等领域的研究,微藻代谢组学起步虽晚,但发展迅速。作为一类重要的物种,微藻具有结构单一、易于进行遗传操作等优势,通过基因工程优化或加速进化进行微藻的选育已成为微藻研究的重要领域。其中,蓝藻和衣藻的基因组信息相对完善[10],是微藻代谢工程研究的主要对象。目前代谢组学技术在微藻研究中的应用主要涉及到医药[11]、食品[12]、化妆品[13]、生物燃料[14]及环境治理[15]等方面。
1 代谢组学技术 1.1 代谢组学概况根据代谢物覆盖尺度的不同,代谢组学研究可以划分为无偏向性全尺度表征的代谢组学分析(metabolomics/metabonomcis)、局部表征的代谢轮廓分析(metabolic profiling analysis)、较大尺度表征的代谢指纹分析(metabolic fingerprinting analysis)与针对少数特定代谢物的代谢靶标分析(metabolites target analysis)等不同层次[16-17]。
1.2 代谢组学研究路线及策略代谢组学的主要目标是系统地研究生物代谢组的变化特征及趋势,从中寻找可靠的关键代谢物和代谢通路,对生物系统的改变进行鉴定和跟踪。代谢组学主要包括以下流程:样本的采集和保存、样本预处理、分析检测、数据处理和分析、代谢物结构鉴定和生物学解释等,如图 1所示。
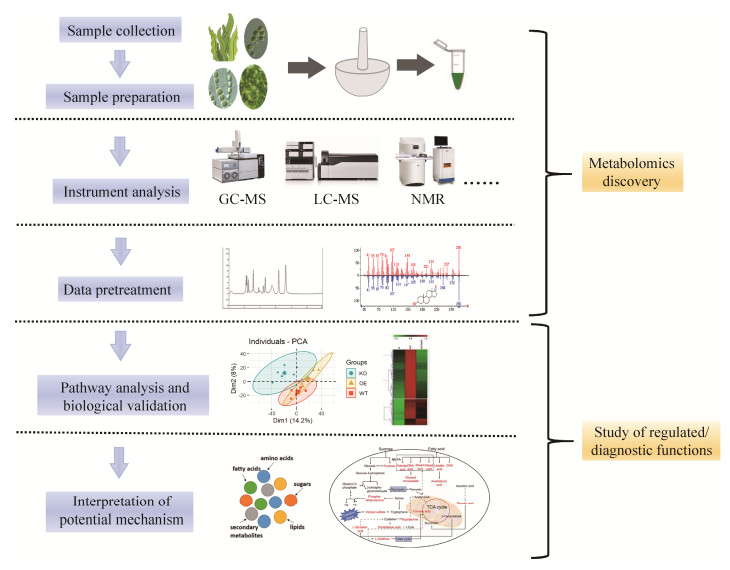
|
| 图 1 代谢组学研究流程 Figure 1 The workflow of metabolomics study. |
1.2.1 样品采集和保存:
样品的采集是代谢组学实验开展的第一步,样品采集方式及样品的质量会影响所得结论的正确与否。藻体代谢物对外部环境的变化刺激非常敏感,因此研究者需要在严格控制的条件下培养实验所需的藻类样本(如组织、细胞等),固定采样方式、时间和部位以减少藻类样品间的个体差异。
当快速取样时即藻体脱离培养环境后,细胞内的代谢会迅速发生改变,这时候需要对细胞进行快速灭活来获取样品的“代谢快照”[18]。常用的细胞灭活方法有液氮淬灭、冷甲醇淬灭和酸碱淬灭等。其中酸碱淬灭容易促使代谢产物发生理化反应而失真,因此在代谢轮廓分析中不常用[19]。液氮淬灭方法容易导致细胞破裂死亡,胞内和胞外代谢物混合,不适合胞内代谢指纹和胞外代谢足迹分析。使用冷甲醇[20]进行淬灭是目前微生物代谢组学中常用的淬灭方法,为了尽量减少细胞内代谢物的外泄,通常在冷甲醇溶液中加入缓冲盐(如4-羟乙基哌嗪乙磺酸(HEPES),磷酸盐缓冲溶液(PBS)、碳酸铵)。
此外,水会作为酶介导反应的介质导致藻体内的代谢物发生分解反应,而藻类样品中水含量的差异会显著影响代谢物定量的准确性[21]。因此在预处理前需要进行样品的干燥以去除多余的水分。目前样品干燥方法有空气干燥、烘干、冷冻干燥等。其中冷冻干燥方法可在低温下将样品中的固态水分直接转化为气态除去,是目前常用的生物样品干燥方法。
1.2.2 样品预处理:样本预处理是将采集到的样本通过一定的预处理过程转变成可供仪器分析的样品,这个过程需要满足:(1)对代谢物提取的无偏向性和高覆盖度;(2)方法简单、速度快、重复性好、通量高。
预处理的流程一般包括提取、浓缩冻干和衍生化等。生物组织常用的提取方式是固-液萃取,包括索式提取、超临界流体萃取、超声辅助萃取和微波辅助萃取等。常用的极性提取溶剂包括甲醇、水和乙醇等;非极性溶剂包括氯仿、甲基叔丁基醚等。根据检测目的的不同,需要对提取溶剂的选择及溶剂比例进行优化。Werner等[43]采用基于MTBE: MeOH: H2O (2:1:1.6,V/V/V)的双相提取系统同时提取了蓝细菌藻(Synechocystis sp. PCC 6803)中的极性和非极性代谢物,其中非极性代谢物在MTBE层,主要包括脂类、色素等,极性代谢物在甲醇水层,包括氨基酸、有机酸和碳水化合物等。
萃取结束后,对样品进行冷冻浓缩可以提高代谢物的浓度。之后再根据不同的检测技术对样品进行相应的预处理方式。如含有羟基、巯基和氨基等极性代谢物常常需要衍生化处理后才能进行液相色谱质谱或气相色谱质谱的检测。其中,气相色谱质谱常用硅烷化试剂(trimethylsilyl reagents,TMS)对含有活泼氢的代谢物进行衍生化,来增加化合物的挥发性[22]。此外,氨基类的化合物在反相液相色谱中不易被保留,离子被抑制,造成对此类化合物定量和定性分析困难[23]。目前常用于氨基酸及胺类化合物的衍生化试剂包括邻苯二甲醛[24]、6-氨基喹啉-N-琥珀酰亚胺甲酯(6-aminoquinolyl-N-hydroxysuccinimidyl carbamate, AQC)[25]和苯甲酰氯[26]等。
1.2.3 样品分析检测:由于代谢产物官能团、极性、带电性和稳定性等理化性质的不同,应选择合适的分析平台。每种分析方法都有其各自的优势及适用范围[27-28]。核磁共振(nuclear magnetic resonance,NMR)技术与质谱(mass spectrometry,MS)检测技术是代谢组学研究的主要分析平台[29]。基于NMR的代谢组学研究方法可实现生物样本的无偏向性、无损和高通量分析,方法简单重复性好,通过核磁数据还可获得原子之间的连接和空间结构,方便未知代谢物的结构鉴定。但该分析方法的灵敏度低、动态范围有限,代谢物检测覆盖范围较小,使其应用受到一定的局限。
相对于NMR,MS技术具有灵敏度高、动态范围广和分辨率高等优点,能实现对多种代谢物的同步快速分析与鉴定。MS主要由进样系统、离子源、质量分析器和检测器等系统构成。其中,质谱的离子源可为样品离子化提供能量,将样品分子电离,形成具有不同质核比的离子束。根据质谱离子源电离能量大小的不同,可将离子源分为“硬”电离源和“软”电离源。其中,电子轰击电离源(electron ionization,EI)是常见的一种“硬”电离源,电离能量通常为70 eV,主要用于气相色谱-质谱联用仪(GC-MS)[30]。对于大部分有机物来说,EI源的电离方式可以得到丰富的碎片离子信息,便于进行结构解析,此外也有许多标准的质谱图库可用来对照定性[31]。然而当样品分子稳定性不高时,EI源产生的分子离子峰的强度低,甚至没有分子离子峰,对未知化合物的元素组成与结构分析都带来不便。化学电离源(chemical ionization,CI)主要是通过试剂离子与样品分子之间的分子离子反应实现样品分子的离子化,是GC-MS联用方法中常用的一种“软”电离技术,所得到的多为分子离子或准分子离子信号,谱图简单,电离效率较高,主要用于测定化合物分子量[32]。但EI、CI源都需要在真空环境下工作,且主要用于易挥发性样品的分析,限制了液体样品检测。大气压离子源(atmospheric pressure ionization,API)可以在常压条件下对液体样品进行电离,并将MS和液体分离技术结合起来,对复杂生物样品进行分析鉴定。常用的API源主要包括电喷雾离子源(electrospray ionization,ESI)、大气压化学电离源(atmospheric pressure chemical ionization,APCI)和大气压光电离源(atmospheric pressure photoionization,APPI)等[33]。其中,ESI离子源结构简单,流速大,维护方便,易获得分子离子峰,能产生多电荷离子,是目前液相色谱质谱联用(LC-MS)最常用的离子源[34]。质量分析器位于离子源和检测器之间,可以将不同m/z的离子在时间或空间上分离,是质谱仪的核心组成部件。代谢组学研究中常见的质量分析器有:飞行时间(time-of-flight analyzer,TOF)、四级杆(quadrupole mass analyzer,Q)、离子阱(ion trap analyzer,IT)和傅里叶变换离子回旋共振(Fourier-transform ion-cyclotron resonance,FT-ICR)等[35]。其中四级杆分析器体积小、重量轻、扫描速度快,常与其他质谱连接,形成串接质谱(如Q/TOF、QQQ等)[36],是目前最成熟、应用最广泛的一种质量分析器,但它的质量范围和分辨率有限。飞行时间质量分析器具有结构简单、无需外加电场和磁场、扫描速度快、对检测离子的质荷比没有上限要求、适合检测大分子等优点,目前已广泛应用于GC-MS和LC-MS质谱仪中[37]。离子阱结构小巧,质量轻,灵敏度高,而且具有多级质谱分析(MSn)的功能,对物质结构鉴定非常有用,但其存在分辨率低和线性范围窄等不足[38]。傅里叶变换离子回旋共振质量分析器的质量分辨率极高,可以完成多级时间上串联质谱操作,且灵敏度较高,是现代质谱学中可用于重要用途的质量分析器[27]。但由于其使用超导磁铁需要液氦,维护费用昂贵,此外不能联用GC,动态范围稍窄,因此目前还不太作为常规仪器使用。
虽然质谱有良好的鉴别物质结构的能力,但对混合物的分离能力有限,因此,为了减小基质干扰、提高检测灵敏度,在质谱分析前,通常会通过气相色谱(gas chromatography,GC)、液相色谱(liquid chromatography,LC)和毛细管电泳(capillary electrophoresis,CE)进行样品的分离。其中基于GC-MS的代谢组学分析技术主要用于挥发性和半挥发性代谢物的检测。该方法灵敏度高、重复性好且有较完备的标准谱库,如NIST、Wiley等,在代谢物定性方面具有明显的优势,已广泛应用于植物和微生物的研究中[39]。但GC-MS方法不适合对高沸点、热不稳定和分子量较大的代谢物进行分析。LC-MS联用分析技术同时具有液相色谱的高效选择性、分离能力和质谱的高分辨率、高灵敏度等优点,是目前应用最为广泛的代谢组分析方法,适用对高沸点、强极性、热稳定性差及高分子量代谢物的检测。此外,LC-MS分析方法的应用范围广,可以通过改变液相色谱的流动相种类、填料类型和离子化方式等,建立适合不同代谢物的分析检测方法[40]。与GC-MS相比,LC-MS技术存在保留时间的重复性差、离子抑制强及代谢物定性困难等问题。CE-MS相对发展较晚,主要应用于强极性离子化合物,如有机酸、氨基酸和核苷酸等极性代谢物及带电物质的研究。CE-MS进样量小,检测灵敏度高,但是其重复性差,迁移时间漂移严重。目前,单一方法无法实现代谢物的全面分析,整合多种分析平台可扩大代谢物的检测覆盖度并提供综合的结构化数据,已成为代谢组学分析发展的一大趋势[41-43]。Bhinderwala等[42]研究比较了基于NMR和MS的代谢组学分析方法对莱茵衣藻(Chlamydomonas reinhardtii)中的小分子代谢物的检测及鉴定的覆盖度,结果表明基于GC-MS及NMR的代谢组学技术可分别对莱茵衣藻中82、20个代谢物进行分析检测,其中有16个代谢物仅由GC-MS平台检测鉴定、14个代谢物仅由NMR平台检测鉴定,且主要涉及到戊糖磷酸途径、卡尔文循环、三羧酸循环和氨基酸生物合成等关键代谢通路。该研究表明整合NMR和MS代谢组学技术不但能扩大代谢物检测覆盖度,而且还能发现更为多样的关键代谢物。Werner等[43]采用甲基叔丁基醚/甲醇/水双相提取系统对蓝藻(Synechocystis sp. PCC 6803)中极性、非极性代谢物进行提取,进一步采用基于LC-MS非靶向代谢组学方法(non-targeted LC-MS)对蓝细菌中非极性物质(如脂类、色素等)进行定性定量分析;采用HILIC TQS-MS靶向及GC-MS非靶向代谢组学技术(non-targeted GC-MS)分别对蓝细菌中极性次级代谢物及初级代谢物进行分析;此外,采用半定量GC-MS及非靶向GC-MS技术对蓝细菌中聚合物进行分析检测。
1.2.4 数据处理与分析:仪器分析后获得海量代谢数据,需采用合适的方法对数据进行处理,获取其中的代谢信息。数据处理过程包括数据方法验证、数据预处理、统计学分析和特征代谢物筛选等。
方法验证主要是为了确保数据的可靠性[44]。常用的数据验证方法包括质量控制样品(QC)验证法[45]和内标验证法[46]。QC是将所有待测样品等量均匀混合后得到的特殊样品,在仪器分析过程中,均匀地插入QC样品,用于评价分析方法的重复性和仪器的稳定性。内标验证法是选用一定质量测试样品中不含有的纯物质作为内标物加入需要分析的样品中,对样品进行色谱分析来评价分析方法的稳定性。对数据进行预处理是为了使其转化成可直接用于数据统计学分析的形式。首先对代谢物积分获得峰面积,将峰面积进行滤噪、基线校正、峰提取、峰匹配和归一化等数据预处理后再进行单变量分析或多变量分析。目前常用的数据预处理软件有MET-IDEA[47]和XCMS[48]等。
数据预处理后即可对数据进行多变量和单变量分析。多变量分析是通过降低维度的算法减小数据的复杂度,随后通过相关软件将结果可视化。多变量分析包括无监督模式识别和有监督模式识别方法,其中无监督模式识别方法包括主成分分析(principal components analysis,PCA)、分层聚类分析(hierarchical cluster analysis,HCA)等;有监督识别模式方法主要有偏最小二乘-判别分析(partial least squares-discrimination analysis,PLS-DA)、正交偏最小二乘-判别分析(orthogonal signal correction partial least squares-discriminant analysis,OPLS-DA)和随机森林分析(random forests,RF)等。常见的单变量分析有t-检验、非参数检验和ANOVA分析等。对于两组之间符合正态分布的数据用t-检验方法,两组之间不符合正态分布的数据用非参数检验,而ANOVA常用于3组以上的数据分析[49]。
1.2.5 代谢物的鉴定与生物学解释:代谢物的结构鉴定是代谢组学研究的重要环节。GC-MS分析平台是利用能量固定的EI电离源(70 eV)获得丰富的化合物质谱碎片信息,方便代谢物的定性。为了防止共流出峰的干扰,GC-MS的原始数据首先需要通过去卷积软件(如AMDIS和ChromaTOF软件等)进行化合物的峰识别,然后与标准谱图库(如NIST、Wiley、Fiehn等)进行匹配,获得可能的代谢物结构信息,然后利用标准样品进行结构确认。LC-MS分析平台由于其存在不同的离子化方式(如ESI、APCI、APPI、NESI等)及碰撞模式(如CID、PQD、HCD、ETD等),且同一碰撞模式下,不同仪器获得的碎片离子的丰度比例也有差异,因此很难建立通用的LC-MS标准谱库。通常基于LC-MS分析平台对其进行结构鉴定时,首先需要根据准确的分子量、同位素丰度比、二级碎片信息与数据库(如METLIN、HMDB、MassBank)中谱图进行比对,得到候选化合物,最后用获得的标样进行保留时间的验证。对于未知的代谢物,可以应用制备色谱、核磁等方法进行定性。
在确定特征代谢物后,为了更好地理解其生理功能,需要进行代谢途径或代谢网络分析。目前已有一些网络数据库收录了基因、蛋白及代谢物等的信息(如HMDB[50]、Metaboanalyst[51]、KEGG[52]等),可以快速追溯到他们所在的代谢途径,并找到与其密切相关的基因和蛋白,为代谢物生理功能的解释提供了丰富的参考资源[53]。
2 代谢组学技术在微藻研究中的应用创始至今的数十年间,代谢组学的触角不断延伸至生命科学的各个领域,在疾病诊断、新药研发、植物生理学、微生物工程、环境毒理学、营养学和食品品质评价等领域蓬勃发展,取得了很大的进展,但对微藻领域的功能应用研究尚不普遍。微藻含有丰富的高价值营养成分和独特的生物活性功能,在许多领域都有巨大的经济利用价值。近年来随着代谢组学技术的快速发展和微藻研究热度的提升,微藻代谢组学的研究受到研究者的广泛关注,研究内容主要涉及到生物燃料、环境治理、逆境胁迫和毒理学等方面。
2.1 在生物燃料研究中的应用生物燃料是传统化石燃料的理想替代物。其中,微藻代谢物可用于生产乙醇、氢和生物柴油等生物燃料,是当前最具潜力的生物燃料来源。因此,对微藻副产品(如醇类、脂质、氢气、乙烯等)的开发为缓解化石燃料引起的能源危机和环境危机提供新的方向。Liu等[14]建立一种基于超高效液相色谱质谱联用(UHPLC-MS)的脂质分析新方法,实现了绿藻莱茵衣藻(Chlamydomonas reinhardtii)和海洋绿藻(Nannochloropsis oceanica)中三酰甘油(TAG)结构的详细表征,此外,脱镁叶绿素可作为藻类TAG定量分析的内标,实现TAG的准确定量。Xiao等[54]利用GC-MS方法对海洋绿藻(Nannochloropsis oceanica)的脂类进行了研究,结果表明营养缺乏的条件下,该绿藻可通过酰基辅酶a依赖途径积累中性脂TAG,而极性脂类可通过酰基辅酶a非依赖途径转变为TAG,该研究为微藻定向积累油脂、脂肪酸等能源物质提供了理论支持。Zhang等[55]采用基于GC-MS和LC-MS的代谢组学分析方法,对氮缺乏条件下湛江等鞭金藻(Isochrysis zhangjiangensis)胞内代谢物随时间的变化规律进行了研究,共鉴定出包括有机酸、碳水化合物和氨基酸在内的44个小分子代谢物,统计学分析发现缺氮胁迫后大多数碳代谢的中间体下调,而丙酮酸发生上调,提示氮限制条件会促进碳流向脂类。为了进一步对上述结果进行验证,作者在缺氮条件下,配合使用β-1, 3-葡聚糖酶抑制剂(β-1, 3-glucanase inhibitor)来阻碍金藻内多糖的形成,结果发现加入抑制剂后多糖含量减少10%,脂质含量增加50%,此外,γ-氨基丁酸(gamma-aminobutyric acid, GABA)发生显著变化,可以作为提高金藻脂类产量的关键靶点,该研究为微藻积累脂质生产生物燃料提供了新方向。羰基氰化物间氯苯腙(carbonyl cyanide m-chlorophenylhydrazone,CCCP)是一种氧化磷酸化抑制剂,会影响线粒体中蛋白质的合成,损害细胞构成,最终导致细胞死亡,但研究发现CCCP添加会促进部分微藻产氢(H2),目前有关CCCP对微藻产H2的代谢调控研究较为关注。Yang等[56]在莱茵衣藻(Chlamydomonas reinhardtii)生长过程中添加CCCP,通过基于GC-TOF/MS代谢组学分析方法研究发现,添加CCCP 10 min后,藻类细胞中游离氨基酸、不饱和游离脂肪酸和核苷酸等代谢物水平显著降低,随着CCCP暴露时间的延长,藻类细胞中脂肪酸含量持续增加,且氢气(H2)稳定产生,该研究为微藻定向积累氢气提供了理论基础。纪超凡等[57]采用基于GC-MS的方法研究了抑制剂CCCP调控海洋绿藻亚心型四爿藻产氢过程的代谢途径,对光、暗两种环境下亚心型四爿藻产氢过程中乙酸、乙醇和丙酸等代谢产物进行分析,发现与暗反应过程相比,光反应下藻细胞的乙醇代谢途径受到抑制,而乙酸代谢途径增加。而乙醇的代谢途径与氢酶竞争还原当量NAD(P)H,不利于氢气的产生,因此四爿藻在光下的产氢量远高于暗处。Timmins等[58]利用基于NMR和GC-MS的代谢组学方法分析了莱茵衣藻(Chlamydomonas reinhardtii)在硫耗竭产H2过程中细胞代谢组的变化,结果表明在硫耗竭24 h之内,莱茵衣藻细胞主要进行有氧代谢,并以淀粉和TG的形式储存能量,该阶段细胞并未产生H2;硫耗竭24–72 h是细胞的主要产H2阶段,细胞通过降解内源性代谢物(如淀粉和氨基酸)进行无氧发酵代谢,产生大量发酵代谢产物(如乙醇和甲酸盐);随着大量发酵产物的产生,在硫耗竭72–120 h之内,细胞中淀粉分解速率降低,溶液pH值稳定,H2的生成速度减缓。该研究可为揭示微藻产H2机理及进行代谢工程改造提供理论指导。
2.2 在医药领域中的应用微藻富含多种营养成分和各种生物活性物质,如类胡萝卜素、多糖、维生素、矿物质和多不饱和脂肪酸等[59],具有抗肿瘤、抗病毒和抗感染等作用[60]。研究发现部分微藻和酵母可产生抗病毒化合物虾青素,其具有抗癌[31]、抗炎症[61]和增强免疫系统等作用。Yu等[11]利用LC-MS和GC-MS测定了8种化学诱导剂对雨生红球藻(Haematococcus pluvialis)中虾青素含量的影响,PLS-DA和权重代谢网络分析(WGCNA)表明,化学诱导剂对雨生红球藻中虾青素代谢具有显著调控作用,进一步筛选出包括“谷胱甘肽代谢途径”和“不饱和脂肪酸生物合成途径”等在内的9条与虾青素代谢密切相关的关键代谢通路,该研究可为抗病毒化合物虾青素生物合成提供基因改造的靶点。Marco等[62]从海洋硅藻(Skeletonema marinoi)中分离纯化得到一种对血液肿瘤细胞系U-937和结肠癌细胞系HCT-116有选择性杀伤作用的单酰甘油三酯(MAGs),LC-MS分析结果表明,藻类中MAGs主要以2-单酰基衍生物形式存在,而在分离纯化过程中2-单酰基转变为1-单酰基,形成1-单酰基类衍生物。此外,MAGs类物质抗癌机制研究表明MAGs物质可通过激活半胱天冬酶3/7诱发癌细胞凋亡。
2.3 在化妆品研究中的应用化妆品是一类含有药物和类药物功效的生物活性成分的产品[13]。据报道,微藻代谢物和生物活性分子(如多糖、维生素、氨基酸等)可治疗皮肤烧伤、防止皮肤老化和维持皮肤水平衡等[63-64]。Xu等[65]分析了6种红藻(Porphyra haitanensis,Gracilaria chouae,Gracilaria blodgettii,Gracilaria lemaneiformis,Eucheuma galetinae,Gelidium amansii)中多糖组成的差异,并对其潜在的功效进行了研究,结果表明对于不同类别的红藻,其多糖中6-脱水半乳糖(AnGal)和半乳糖(Gal)具有差异,酪氨酸酶抑制活性实验表明红藻多糖及其降解产物具有潜在的美白功效,可作为化妆品原材料使用。类菌胞素氨基酸(MAAs)是以环己烯酮为基本骨架,与不同氨基酸缩合所形成的一类功能性氨基酸,能在海洋藻类和真核藻类细胞中合成,具有紫外光防护、渗透和抗氧化等功能,可作为化妆品中的活性成分[66]。Vanessa等[67]建立了一种基于LC-MS/MS的快速、高通量的MAAs类化合物的定量分析新方法,该方法可以在6 min内准确定量分析蓝藻细菌内的3种高含量MAAs (porphyra- 334、shinorine和mycosporine-glycine-alanine),该研究可用于紫外线诱导藻体细胞中MAAs生物合成调控机制的研究,为化妆品产业的发展提供新的方向。
2.4 在环境治理研究中的应用近年来,随着人口增长和工业发展,大量有机物、重金属和病毒等污染物进入水体,污染水质,破坏生态系统平衡,因此亟需开发高效、经济的污染治理方法。微藻具有生长速度快、光合效率高等特点,可以有效吸收污水中的有机碳、氮和磷等营养物质用于微藻生长,同时产生大量生物活性物质,实现污水的资源化,具有低成本、高效率和环境友好型等优点[68-69]。Tengda等[15]采用GC-MS技术研究绿藻四尾栅藻(Scenedesmus quadricauda)和硅藻纳氏藻(Navicula sp.)对废水中葛兰素内酯(HHBC)的去除作用,结果表明两种藻对HHBC的去除率均可达到72.9%–100%,基于UPLC-MS/MS方法筛选鉴定出4种HHCB降解的中间产物(TP 300,TP 319,TP 392和TP 284),HHCB在藻体中的代谢涉及到羟基化、甲氧基化、甲基化和酮基化等。Chen等[70]利用基于GC-MS的代谢组学方法分析了索式小球藻(Chlorella sorokiniana)和细菌联合培养对废水营养物的去除情况,结果表明,联合处理方法和单一小球藻处理方法对营养物的去除率分别为72.8%和94.4%,已鉴定代谢物的PLS-DA分析发现,联合处理方法会阻碍小球藻的代谢过程,该研究揭示了微藻代谢调控与营养物质降解的可能关系,可为藻类净化污水的研究提供理论基础。
由于化学燃料、农药的使用、电子工业开发等原因,我国重金属污染事故频发,其中最严重的是2012年发生在广西龙江镉超标事件,高峰时超标80多倍,影响范围达100 km[71]。重金属对环境造成了负面影响,但其自身含有较高的经济价值,因此对重金属的处理和回收尤为重要。传统的金属处理方法有离子交换、石灰沉淀、化学沉淀和吸附,这些方法成本昂贵,效率低下,尤其面对金属浓度较高的废水[72]。生物吸附作用是将重金属元素被动地结合到一些生物表面,从而将其从废水中去除的过程[73]。藻类生长环境恶劣,经长期进化形成了快速适应环境的调节机制,不仅将自身的危害降到最低,而且可以吸附废水中的重金属,再加上其生长迅速和成本低廉的特点,可作为良好的生物吸附材料[74]。研究发现微藻对重金属的修复作用与藻体内的植物络合素、谷胱甘肽等密切相关。在重金属存在时,植物络合素在藻体内会转化成小分子多肽,用于螯合重金属,使微藻耐受重金属的能力提高[75]。MoreHie等[76]将硅藻三角褐指藻(Phaeodactylum tricornutum)培养于含有重金属的培养液中,发现三角褐指藻中50%的谷胱甘肽转化成为植物络合素。除此之外,其他藻类已表现出对重金属的吸附能力,如小球藻对铜(Cu)和锌(Zn)的吸附[77],螺旋藻对铅(Pb)的吸附[78]。
2.5 在环境适应机制研究中的应用迄今已知的藻类约有3万余种,其中微藻约占70%,大多数微藻为光能自养型生物,在陆地、海洋中均有分布。此外,一些生长在极端环境(如南北极、火山口和盐碱度高的海水)中的微藻相比于其他微藻具有特殊的环境适应机制。系统研究胁迫环境下微藻响应和适应的分子机制,对发展生态生理学、微生物资源学等研究具有重要科学意义。雪衣藻(Chlamydomonas nivalis)是一种能够适应极地和高山雪地等极端环境的单细胞绿藻,它能抵御低温、高光照、强紫外辐射、高盐和贫营养等胁迫条件。Lu等采用多平台代谢组学技术系统地研究了雪衣藻响应氮磷营养缺乏及盐胁迫的代谢调控机制。基于GC-MS的脂肪酸代谢轮廓分析方法对盐胁迫下雪衣藻细胞脂肪酸代谢变化进行研究,多变量统计分析表明胁迫组与对照组具有显著差异,进一步筛选获得与盐胁迫密切相关的6个脂肪酸类标志物(C16:0、C16:3、C18:0、C18:1、C18:2和C18:3),这些脂肪酸的变化可调控细胞膜的流动性和渗透性,从而有利于细胞抵抗外界胁迫环境[79];采用基于UPLC-MS的脂质组学方法对雪衣藻响应盐胁迫、氮缺乏及磷缺乏相关的脂类标志物进行分析,研究发现与胁迫和营养缺乏密切相关的标志物主要涵盖磷脂、糖脂等,这些脂类的变化可能与细胞膜的稳定性、信号传导等密切相关[80-81]。Lu等采用基于GC/TOF-MS的非靶向代谢组学分析方法对氮磷营养缺乏下雪衣藻的初级代谢物进行了全面表征,筛选获得与氮缺乏和磷缺乏相关的响应标志物分别有30个和39个,研究发现营养缺乏后,藻体细胞可以通过调节糖代谢、TCA循环和糖酵解等通路适应胁迫[82]。Huseby等[83]采用超高效液相色谱-高分辨质谱(UHPLC-MS)技术研究了从大西洋水体和地中海水体中分离出的聚生角毛藻(Chaetoceros socialis)的代谢指纹差异,主成分分析(PCA)表明2个不同生长环境下分离出的聚生角毛藻代谢组具有明显的分离趋势;此外,两种聚生角毛藻菌株的生长速率、光合效率及表型等也具有显著的差异。吕和鑫等[84]采用基于GC-MS的代谢组学方法研究了嗜盐微藻-杜氏藻(Dunaliella viridis)对碳、氮、磷元素共同胁迫时的环境适应机制,共检测定性出80种代谢物,其中饱和脂肪酸、蔗糖和麦芽糖含量升高,而卡尔文循环、三羧酸循环和尿素循环的一些中间代谢物含量降低,该研究可为揭示杜氏藻耐盐响应机制及构建杜氏藻代谢工厂提供理论参考。
2.6 在毒理学研究中的应用近年来沿海地区的工业和经济快速发展,一些有毒材料被排放进入环境,使得环境污染状况加剧,不仅引起生态系统中各级生物的不良反应,甚至危害到各类生物的生存。微藻对毒理因子的反应可用于考察水体污染程度,通常将莱茵衣藻(Chlamydomonas reinhardtii)作为淡水环境中的毒理学研究的受试生物。纳米氧化铈(NPs)是一种燃料催化剂,但可通过各种途径进入环境中,对环境造成负面影响。Taylor等[85]研究了不同浓度NPs对莱茵衣藻生长及代谢的影响,结果表明NPs暴露浓度对莱茵衣藻的生长几乎没有影响,但在超浓度NPs下会干扰莱茵衣藻光合作用和固氮代谢过程。Wai等[86]应用GC-MS和LC-MS方法研究了镍胁迫条件下四叶栅藻(Chlorophyceae)的代谢特征,结果表明,镍胁迫后,藻体中硫代谢、氨基酸和氮代谢等受到显著影响,此外,温度也会影响四叶栅藻对镍的吸收量,25 ℃时四叶栅藻吸收的镍量高于35 ℃。丁醇胁迫条件对光合蓝细菌细胞的毒害严重,阻碍了其作为工程宿主菌生产生物丁醇。Niu等[87]采用基于LC-MS及GC-MS的代谢组学方法研究了反应调控因子Sll0039在集胞藻(Synechocystis sp.)丁醇耐受机制中的作用,结果表明sll0039基因缺失会引起集胞藻细胞中二磷酸葡萄糖腺苷(ADP-glucose)、二羟基丙酮磷酸(DHAP)、D-核糖5-磷酸(R5P)、D-葡萄糖-6-磷酸(G6P)和D-果糖-6-磷酸(F6P)等代谢物发生显著变化。此外,对两种缺失不同基因的集胞藻样品(Δsll0039和Δslr1037)进行代谢组学研究,结果表明两个反应调控因子Sll0039和Slr1037在集胞藻耐受丁醇胁迫条件的机制是相互独立的。
2.7 在微藻培养研究中的应用微藻细胞富含蛋白质、多糖、脂类及色素等营养物质,在食品、饲料、医药等领域得到了广泛应用。然而,由于微藻培养技术的不成熟导致的生产成本高、效率低是限制微藻产业化培养的主要因素。优化微藻培养条件,建立高效、低成本的微藻培养技术,是目前微藻研究的热点领域之一。Bogaert等[88]基于GC-MS代谢组学方法对不同醋酸盐浓度(17、31、44、57 mmol/L)胁迫下莱茵衣藻(Chlamydomonas reinhardtii)的代谢轮廓进行分析,结果表明,随着醋酸盐浓度的增加,藻体细胞中淀粉、脂肪酸、叶绿素和蛋白质含量均上调,且57 mmol/L浓度下藻体生物质产量相比于17 mmol/L时增加3倍,这可能与醋酸盐代谢途径的上调密切相关。Aguilera-Saez等[89]采用基于NMR的代谢组学技术研究了海洋微藻-球等鞭金藻(Isochrysis galbana)在不同培养温度(15– 30 ℃)及不同光照强度(250–1600 µmol/m2s)下的代谢变化,结果表明高的光照强度可导致叶绿素a、岩藻黄质和氨基酸的下降,促进极性脂肪酸向中性脂肪酸的转变;高温和高光照强度会降低多不饱和脂肪酸(PUFAs)的浓度;低温及低光照强度会引起有机渗透调节物质(如龙虾肌碱、甘油、二甲基巯基丙酸DMSP、甘氨酸甜菜碱GBT)和糖类(如葡萄糖、海藻糖、半乳糖)等下降。Lv等[90]采用基于GC-MS代谢组学技术对不同光照条件下雨生红球藻(Haematococcus pluvialis)代谢响应进行研究,共鉴定出雨生红球藻中81种代谢产物,其中氨基酸23种,糖19种,有机酸15种,醇类8种,胺类7种,核酸4种,其他5种,对强光处理后产生的3类雨生红球藻样品(黄色、红色、绿色株系)进行多变量和单变量分析,结果表明在强光处理后获得的黄色株系中卡尔文循环和TCA循环加强,促进了一些参与细胞保护的代谢物发生了显著上调(如糖、脯氨酸和谷氨酸等)。
3 展望代谢组学主要针对基因表达的末端代谢产物在生物体受外界刺激扰动、基因突变或生理病理变化后出现的多元动态变化进行分析。借助于代谢组学对复杂生物的整体代谢通路高通量表征方面的巨大优势,代谢组学技术日益成为生命科学研究的热点。近年来,依赖于分析检测技术和生物信息学的快速发展,代谢组学技术在微藻应用领域的研究越来越多,主要涉及到生物燃料和高价值产物开发、环境治理等多个方面。但代谢组学技术在微藻资源的开发和应用方面还面临诸多问题和挑战。
(1) 首先,如何有效分离微藻细胞的胞内和胞外代谢物、扩大代谢物检测覆盖度、建立丰富的藻类代谢物数据库及海量代谢数据快速高效挖掘等是目前微藻代谢组学技术发展面临的主要问题。
(2) 其次,目前常规的代谢组学分析获取的是代谢物的静态结果,表征了生物反应的最终结果,而微藻生物活性物质的调控机制研究需要通过代谢流和代谢通量实现,这就需要发展新的动态代谢组学技术,追踪代谢物的合成、分解和转化等动态过程,用于微藻中特定代谢靶点的精准调控。
(3) 代谢组学技术依赖于分析技术的快速发展,关键核心技术掌握在分析化学家手中,而微藻生物特性及资源开发等为生物学家所熟知,这在一定程度上阻碍了微藻代谢组学的发展,因此亟需有效整合代谢组学和生物信息学,发展代谢组学生物信息学这一交叉学科,为代谢组学技术在不同学科中的应用提供更广阔的空间。
| [1] | Nicholson JK, Lindon JC, Holmes E. 'Metabonomics': understanding the metabolic responses of living systems to pathophysiological stimuli via multivariate statistical analysis of biological NMR spectroscopic data. Xenobiotica, 1999, 29(11): 1181-1189. DOI:10.1080/004982599238047 |
| [2] | Oliver SG, Winson MK, Kell DB, Baganz F. Systematic functional analysis of the yeast genome. Trends in Biotechnology, 1998, 16(10): 373-378. |
| [3] | Fiehn O. Metabolomics-the link between genotypes and phenotypes. Plant Molecular Biology, 2002, 48(1/2): 155-171. DOI:10.1023/A:1013713905833 |
| [4] | German JB, Bauman DE, Burrin DG, Failla ML, Freake HC, King JC, Klein S, Milner JA, Pelto GH, Rasmussen KM, Zeisel SH. Metabolomics in the opening decade of the 21st century: building the roads to individualized health. The Journal of Nutrition, 2004, 134(10): 2729-2732. DOI:10.1093/jn/134.10.2729 |
| [5] | Ren SC, Shao YP, Zhao XJ, Hong CS, Wang FB, Lu X, Li J, Ye GZ, Yan M, Zhuang ZP, Xu CL, Xu GW, Sun YH. Integration of metabolomics and transcriptomics reveals major metabolic pathways and potential biomarker involved in prostate cancer. Molecular & Cellular Proteomics, 2016, 15(1): 154-163. |
| [6] | Liu H, Liu W, He XX, Chen XF, Yang JF, Wang Y, Li Y, Ren JM, Xu WS, Zhao YN. Characterization of a cell density-dependent sRNA, Qrr, and its roles in the regulation of the quorum sensing and metabolism in Vibrio alginolyticus. Applied Microbiology and Biotechnology, 2020, 104(4): 1707-1720. DOI:10.1007/s00253-019-10278-3 |
| [7] | Ye GZ, Ding DX, Gao H, Chi YL, Chen JS, Wu ZM, Lin Y, Dong SJ. Comprehensive metabolic responses of HepG2 cells to fine particulate matter exposure: insights from an untargeted metabolomics. Science of the Total Environment, 2019(691): 874-884. |
| [8] | Zeng J, Liu SSY, Cai WC, Jiang HR, Lu X, Li GL, Li J, Liu JW. Emerging lipidome patterns associated with marine Emiliania huxleyi-virus model system. Science of the Total Environment, 2019(688): 521-528. |
| [9] | Hirashima T, Toyoshima M, Moriyama T, Sato N. Evolution of the phosphatidylcholine biosynthesis pathways in green algae: combinatorial diversity of methyltransferases. Journal of Molecular Evolution, 2018, 86(1): 68-76. DOI:10.1007/s00239-017-9826-4 |
| [10] | Grossman AR, Croft M, Gladyshev VN, Merchant SS, Posewitz MC, Prochnik S, Spalding MH. Novel metabolism in Chlamydomonas through the lens of genomics. Current Opinion in Plant Biology, 2007, 10(2): 190-198. DOI:10.1016/j.pbi.2007.01.012 |
| [11] | Yu XH, Niu XF, Zhang XQ, Pei GS, Liu J, Chen L, Zhang WW. Identification and mechanism analysis of chemical modulators enhancing astaxanthin accumulation in Haematococcus pluvialis. Algal Research, 2015(11): 284-293. |
| [12] | Pinu FR, De Carvalho-Silva S, Uetanabaro APT, Villas-Boas SG. Vinegar metabolomics: an explorative study of commercial balsamic vinegars using gas chromatography-mass spectrometry. Metabolites, 2016, 6(3): 22. DOI:10.3390/metabo6030022 |
| [13] | Reszko AE, Berson D, Lupo MP. Cosmeceuticals: practical applications. Obstetrics and Gynecology Clinics of North America, 2010, 37(4): 547-569. DOI:10.1016/j.ogc.2010.09.006 |
| [14] | Liu BS, Vieler A, Li C, Jones AD, Benning C. Triacylglycerol profiling of microalgae Chlamydomonas reinhardtii and Nannochloropsis oceanica. Bioresource Technology, 2013(146): 310-316. |
| [15] | Ding TD, Li W, Cai M, Jia XF, Yang MT, Yang B, Li JY. Algal toxicity, accumulation and metabolic pathways of galaxolide. Journal of Hazardous Materials, 2020(384): 121360. |
| [16] | Fiehn O, Kopka J, Trethewey RN, Willmitzer L. Identification of uncommon plant metabolites based on calculation of elemental compositions using gas chromatography and quadrupole mass spectrometry. Analytical Chemistry, 2000, 72(15): 3573-3580. DOI:10.1021/ac991142i |
| [17] | Shulaev V. Metabolomics technology and bioinformatics. Briefings in Bioinformatics, 2006, 7(2): 128-139. DOI:10.1093/bib/bbl012 |
| [18] | Tsutsaeva AA, Anan'ina AE, Balyberdina LM, Stepanyuk LV, Pavlenko NV. Long-term storage of industrial microbial strains. Microbiology, 2008, 77(5): 621-624. DOI:10.1134/S0026261708050172 |
| [19] |
Wu QP, Li YD, Zhang JM. Advances in metabolomics of common food-borne pathogen. Microbiology China, 2016, 43(3): 609-613.
(in Chinese) 吴清平, 李玉冬, 张菊梅. 常见食源性致病菌代谢组学研究进展. 微生物学通报, 2016, 43(3): 609-613. |
| [20] | Faijes M, Mars AE, Smid EJ. Comparison of quenching and extraction methodologies for metabolome analysis of Lactobacillus plantarum. Microbial Cell Factories, 2007, 6(1): 27. DOI:10.1186/1475-2859-6-27 |
| [21] | Kim HK, Verpoorte R. Sample preparation for plant metabolomics. Phytochemical Analysis, 2010, 21(1): 4-13. DOI:10.1002/pca.1188 |
| [22] | Zhao YN, Zhang L, Zhao CX, Hu CX, Li YL, Zhao JY, Zhang JJ, Li LL, Chang YW, Wang F, Lu X, Zhu Z, Xu GW. Metabolic responses of rice leaves and seeds under transgenic backcross breeding and pesticide stress by pseudotargeted metabolomics. Metabolomics, 2015, 11(6): 1802-1814. DOI:10.1007/s11306-015-0834-3 |
| [23] | Erim FB. Recent analytical approaches to the analysis of biogenic amines in food samples. TrAC Trends in Analytical Chemistry, 2013(52): 239-247. |
| [24] | Kutlán D, Molnár-Perl I. New aspects of the simultaneous analysis of amino acids and amines as their o-phthaldialdehyde derivatives by high-performance liquid chromatography: analysis of wine, beer and vinegar. Journal of Chromatography A, 2003, 987(1/2): 311-322. |
| [25] | Gaudin Z, Cerveau D, Marnet N, Bouchereau A, Delavault P, Simier P, Pouvreau JB. Robust method for investigating nitrogen metabolism of 15N labeled amino acids using AccQ•Tag ultra performance liquid chromatography- photodiode array-electrospray ionization-mass spectrometry: application to a parasitic plant-plant interaction. Analytical Chemistry, 2014, 86(2): 1138-1145. DOI:10.1021/ac403067w |
| [26] | Aflaki F, Ghoulipour V, Saemian N, Sheibani S. Biogenic amine contents in non-alcoholic beers: screening and optimization of derivatization. Food Analytical Methods, 2014, 7(3): 713-720. DOI:10.1007/s12161-013-9746-x |
| [27] | Dettmer K, Aronov PA, Hammock BD. Mass spectrometry-based metabolomics. Mass Spectrometry Reviews, 2007, 26(1): 51-78. DOI:10.1002/mas.20108 |
| [28] | Ackermann BL, Hale JE, Duffin KL. The role of mass spectrometry in biomarker discovery and measurement. Current Drug Metabolism, 2006, 7(5): 525-539. DOI:10.2174/138920006777697918 |
| [29] | Baidoo EEK, Benke PI, Keasling JD. Mass spectrometry-based microbial metabolomics//Walker John M. Methods in Molecular Biology. Berlin: Springer, 2012(881): 215-278. |
| [30] | Jarukas L, Mykhailenko O, Baranauskaite J, Marksa M, Ivanauskas L. Investigation of organic acids in saffron stigmas (Crocus sativus L.) extract by derivatization method and determination by GC/MS. Molecules, 2020, 25(15): 3427. DOI:10.3390/molecules25153427 |
| [31] | Tanaka T, Kawamori T, Ohnishi M, Makita H, Mori H, Satoh K, Hara A. Suppression of azoxymethane-induced rat colon carcinogenesis by dietary administration of naturally occurring xanthophylls astaxanthin and canthaxanthin during the postinitiation phase. Carcinogenesis, 1995, 16(12): 2957-2963. DOI:10.1093/carcin/16.12.2957 |
| [32] | Yang Y, Lin MQ, Tang J, Ma ST, Yu YX. Derivatization gas chromatography negative chemical ionization mass spectrometry for the analysis of trace organic pollutants and their metabolites in human biological samples. Analytical and Bioanalytical Chemistry, 2020, 412(25): 6679-6690. DOI:10.1007/s00216-020-02762-x |
| [33] |
Ren XN, Liang QL. Advance in metabolomics based on mass spectrometry. Journal of Instrumental Analysis, 2017, 36(2): 161-169.
(in Chinese) 任向楠, 梁琼麟. 基于质谱分析的代谢组学研究进展. 分析测试学报, 2017, 36(2): 161-169. DOI:10.3969/j.issn.1004-4957.2017.02.002 |
| [34] | Chetwynd AJ, David A. A review of nanoscale LC-ESI for metabolomics and its potential to enhance the metabolome coverage. Talanta, 2018(182): 380-390. |
| [35] | Arevalo Jr R, Ni ZQ, Danell RM. Mass spectrometry and planetary exploration: a brief review and future projection. Journal of Mass Spectrometry, 2020, 55(1): e4454. |
| [36] | Herrero M, Vicente MJ, Cifuentes A, Ibáñez E. Characterization by high-performance liquid chromatography/electrospray ionization quadrupole time-of-flight mass spectrometry of the lipid fraction of Spirulina platensis pressurized ethanol extract. Rapid Communications in Mass Spectrometry, 2007, 21(11): 1729-1738. DOI:10.1002/rcm.3017 |
| [37] | Arevalo Jr R, Ni ZQ, Danell RM. Mass spectrometry and planetary exploration: a brief review and future projection. Journal of Mass Spectrometry, 2020, 55(1): e4454. |
| [38] | Nolting D, Malek R, Makarov A. Ion traps in modern mass spectrometry. Mass Spectrometry Review, 2019, 38(2): 150-168. DOI:10.1002/mas.21549 |
| [39] | Fiehn O, Kristal B, van Ommen B, Sumner LW, Sansone SA, Taylor C, Hardy N, Kaddurah-Daouk R. Establishing reporting standards for metabolomic and metabonomic studies: a call for participation. OMICS: A Journal of Integrative Biology, 2016, 10(2): 158-163. |
| [40] | Wang SY, Qiao LZ, Shi XZ, Hu CX, Kong HW, Xu GW. On-line stop-flow two-dimensional liquid chromatography- mass spectrometry method for the separation and identification of triterpenoid saponins from ginseng extract. Analytical and Bioanalytical Chemistry, 2015, 407(1): 331-341. DOI:10.1007/s00216-014-8219-4 |
| [41] | Tang HR, Xiao CN, Wang YL. Important roles of the hyphenated HPLC-DAD-MS-SPE-NMR technique in metabonomics. Magnetic Resonance in Chemistry, 2009, 47(S1): S157-S162. DOI:10.1002/mrc.2513 |
| [42] | Bhinderwala F, Wase N, DiRusso C, Powers R. Combining mass spectrometry and NMR improves metabolite detection and annotation. Journal of Proteome Research, 2018, 17(11): 4017-4022. DOI:10.1021/acs.jproteome.8b00567 |
| [43] | Werner A, Broeckling CD, Prasad A, Peebles CAM. A comprehensive time-course metabolite profiling of the model cyanobacterium Synechocystis sp. PCC 6803 under diurnal light: dark cycles. The Plant Journal, 2019, 99(2): 379-388. DOI:10.1111/tpj.14320 |
| [44] | Wu YM, Li L. Sample normalization methods in quantitative metabolomics. Journal of Chromatography A, 2016(1430): 80-95. |
| [45] | Zhao YN, Zhao CX, Li YL, Chang YW, Zhang JJ, Zeng Z, Lu X, Xu GW. Study of metabolite differences of flue-cured tobacco from different regions using a pseudotargeted gas chromatography with mass spectrometry selected-ion monitoring method. Journal of Separation Science, 2014, 37(16): 2177-2184. DOI:10.1002/jssc.201400097 |
| [46] | Naz S, Garcia A, Rusak M, Barbas C. Method development and validation for rat serum fingerprinting with CE-MS: application to ventilator-induced-lung-injury study. Analytical and Bioanalytical Chemistry, 2013, 405(14): 4849-4858. DOI:10.1007/s00216-013-6882-5 |
| [47] | Lei ZT, Li HQ, Chang J, Zhao PX, Sumner LW. MET-IDEA version 2.06; improved efficiency and additional functions for mass spectrometry-based metabolomics data processing. Metabolomics, 2012, 8(1): 105-110. DOI:10.1007/s11306-012-0397-5 |
| [48] | Eliasson M, Rännar S, Madsen R, Donten MA, Marsden-Edwards E, Moritz T, Shockcor JP, Johansson E, Trygg J. Strategy for optimizing LC-MS data processing in metabolomics: a design of experiments approach. Analytical Chemistry, 2012, 84(15): 6869-6876. DOI:10.1021/ac301482k |
| [49] | Kim HY. Statistical notes for clinical researchers: nonparametric statistical methods: 1. nonparametric methods for comparing two groups. Restorative Dentistry & Endodontics, 2014, 39(3): 235-239. |
| [50] | Wishart DS, Tzur D, Knox C, Eisner R, Guo AC, Young N, Cheng DA, Jewell K, Arndt D, Sawhney S, Fung C, Nikolai L, Lewis M, Coutouly MA, Forsythe I, Tang P, Shrivastava S, Jeroncic K, Stothard P, Amegbey G, Block D, Hau DD, Wagner J, Miniaci J, Clements M, Gebremedhin M, Guo N, Zhang Y, Duggan GE, Macinnis GD, Weljie AM, Dowlatabadi R, Bamforth F, Clive D, Greiner R, Li L, Marrie T, Sykes BD, Vogel HJ, Querengesser L. HMDB: the human metabolome database. Nucleic Acids Research, 2007, 35(S1): D521-D526. |
| [51] | Xia JG, Sinelnikov IV, Han B, Wishart DS. MetaboAnalyst 3.0-making metabolomics more meaningful. Nucleic Acids Research, 2015, 43(W1): W251-W257. DOI:10.1093/nar/gkv380 |
| [52] | Kanehisa M, Goto S, Kawashima S, Okuno Y, Hattori M. The KEGG resource for deciphering the genome. Nucleic Acids Research, 2004, 32(S1): D277-D280. |
| [53] | D'Alessandro A, Slaughter AL, Peltz ED, Moore EE, Silliman CC, Wither M, Nemkov T, Bacon AW, Fragoso M, Banerjee A, Hansen KC. Trauma/hemorrhagic shock instigates aberrant metabolic flux through glycolytic pathways, as revealed by preliminary 13C-glucose labeling metabolomics. Journal of Translational Medicine, 2015, 13(1): 253. DOI:10.1186/s12967-015-0612-z |
| [54] | Xiao Y, Zhang JT, Cui JT, Feng YG, Cui Q. Metabolic profiles of Nannochloropsis oceanica IMET1 under nitrogen-deficiency stress. Bioresource Technology, 2013(130): 731-738. |
| [55] | Zhang YS, Liu YT, Cao XP, Gao P, Liu XY, Wang XY, Zhang JJ, Zhou JN, Xue S, Xu GW, Tian J. Free amino acids and small molecular acids profiling of marine microalga Isochrysis zhangjiangensis under nitrogen deficiency. Algal Research, 2016(13): 207-217. |
| [56] | Yang DW, Zhang YT, Barupal DK, Fan XL, Gustafson R, Guo RB, Fiehn O. Metabolomics of photobiological hydrogen production induced by CCCP in Chlamydomonas reinhardtii. International Journal of Hydrogen Energy, 2014, 39(1): 150-158. DOI:10.1016/j.ijhydene.2013.09.116 |
| [57] |
Ji CF, Yu XJ, Xue S, Chen ZA, Zhang W. Electron transport pathways of hydrogen photobiological production metabolism in CCCP-treated Tetraselmis subcordiformis. Microbiology China, 2011, 38(11): 1666-1672.
(in Chinese) 纪超凡, 虞星炬, 薛松, 陈兆安, 张卫. 亚心型四爿藻在CCCP作用下的光生物产氢的代谢途径. 微生物学通报, 2011, 38(11): 1666-1672. |
| [58] | Matthew T, Zhou WX, Rupprecht J, Lim L, Thomas-Hall SR, Doebbe A, Kruse O, Hankamer B, Marx UC, Smith SM, Schenk PM. The metabolome of Chlamydomonas reinhardtii following induction of anaerobic H2 production by sulfur depletion. Journal of Biological Chemistry, 2009, 284(35): 23415-23425. DOI:10.1074/jbc.M109.003541 |
| [59] | Brennan L, Owende P. Biofuels from microalgae-a review of technologies for production, processing, and extractions of biofuels and co-products. Renewable and Sustainable Energy Reviews, 2010, 14(2): 557-577. DOI:10.1016/j.rser.2009.10.009 |
| [60] | Lauritano C, Ferrante MI, Rogato A. Marine natural products from microalgae: an -omics overview. Marine Drugs, 2019, 17(5): 269. DOI:10.3390/md17050269 |
| [61] | Guedes AC, Amaro HM, Malcata FX. Microalgae as sources of high added-value compounds-a brief review of recent work. Biotechnology Progress, 2011, 27(3): 597-613. DOI:10.1002/btpr.575 |
| [62] | Miceli M, Cutignano A, Conte M, Ummarino R, Romanelli A, Ruvo M, Leone M, Mercurio FA, Doti N, Manzo E, Romano G, Altucci L, Ianora A. Monoacylglycerides from the diatom Skeletonema marinoi induce selective cell death in cancer cells. Marine Drugs, 2019, 17(11): 625. DOI:10.3390/md17110625 |
| [63] | Wijesinghe WAJP, Jeon YJ. Biological activities and potential cosmeceutical applications of bioactive components from brown seaweeds: a review. Phytochemistry Reviews, 2011, 10(3): 431-443. DOI:10.1007/s11101-011-9214-4 |
| [64] | Mayer AMS, Rodríguez AD, Berlinck RGS, Fusetani N. Marine pharmacology in 2007-8:marine compounds with antibacterial, anticoagulant, antifungal, anti-inflammatory, antimalarial, antiprotozoal, antituberculosis, and antiviral activities; affecting the immune and nervous system, and other miscellaneous mechanisms of action. Comparative Biochemistry and Physiology C: Toxicology & Pharmacology, 2011, 153(2): 191-222. |
| [65] | Xie XT, Zhang X, Liu Y, Chen XQ, Cheong KL. Quantification of 3, 6-anhydro-galactose in red seaweed polysaccharides and their potential skin-whitening activity. 3 Biotech, 2020, 10(4): 189. DOI:10.1007/s13205-020-02175-8 |
| [66] | Fleming ED, Castenholz RW. Effects of periodic desiccation on the synthesis of the UV-screening compound, scytonemin, in cyanobacteria. Environmental Microbiology, 2007, 9(6): 1448-1455. DOI:10.1111/j.1462-2920.2007.01261.x |
| [67] | Geraldes V, de Medeiros LS, Jacinavicius FR, Long PF, Pinto E. Development and validation of a rapid LC-MS/MS method for the quantification of mycosporines and mycosporine-like amino acids (MAAs) from cyanobacteria. Algal Research, 2020(46): 101796. |
| [68] | Pacheco MM, Hoeltz M, Moraes MSA, Schneider RCS. Microalgae: cultivation techniques and wastewater phycoremediation. Journal of Environmental Science and Health, Part A, 2015, 50(6): 585-601. |
| [69] | Medhi K, Thakur IS. Bioremoval of nutrients from wastewater by a denitrifier Paracoccus denitrificans ISTOD1. Bioresource Technology Reports, 2018(1): 56-60. |
| [70] | Chen TJ, Zhao QY, Wang L, Xu YF, Wei W. Comparative metabolomic analysis of the green microalga Chlorella sorokiniana cultivated in the single culture and a consortium with bacteria for wastewater remediation. Applied Biochemistry and Biotechnology, 2017, 183(3): 1062-1075. DOI:10.1007/s12010-017-2484-6 |
| [71] |
Wang YB, Ke K, Zhang RC, Gao CH, He BJ. Present study and perspective of heavy metal biosorption on marine algae. Advances in Marine Science, 2013, 31(4): 574-582.
(in Chinese) 王一兵, 柯珂, 张荣灿, 高程海, 何碧娟. 海藻生物吸附重金属研究现状及展望. 海洋科学进展, 2013, 31(4): 574-582. DOI:10.3969/j.issn.1671-6647.2013.04.017 |
| [72] | Salama ES, Govindwar SP, Khandare RV, Roh HS, Jeon BH, Li XK. Can omics approaches improve microalgal biofuels under abiotic stress?. Trends in Plant Science, 2019, 24(7): 611-624. DOI:10.1016/j.tplants.2019.04.001 |
| [73] | Volesky B. Biosorption and me. Water Research, 2007, 41(18): 4017-4029. DOI:10.1016/j.watres.2007.05.062 |
| [74] | Martensson AM, Aulin C, Wahlberg O, Agren S. Effect of humic substances on the mobility of toxic metals in a mature landfill. Waste Management and Research, 1999, 17(4): 296-304. DOI:10.1177/0734242X9901700406 |
| [75] |
Tan WN, Li ZA, Zou B. Molecular mechanisms of plant tolerance to heavy metals. Journal of Plant Ecology, 2006, 30(4): 703-712.
(in Chinese) 谭万能, 李志安, 邹碧. 植物对重金属耐性的分子生态机理. 植物生态学报, 2006, 30(4): 703-712. DOI:10.3321/j.issn:1005-264X.2006.04.022 |
| [76] | Morelli E, Scarano G. Synthesis and stability of phytochelatins induced by cadmium and lead in the marine diatom Phaeodactylum tricornutum. Marine Environmental Research, 2001, 52(4): 383-395. DOI:10.1016/S0141-1136(01)00093-9 |
| [77] | Mishra A, Medhi K, Maheshwari N, Srivastava S, Thakur IS. Biofuel production and phycoremediation by Chlorella sp. ISTLA1 isolated from landfill site. Bioresource Technology, 2018(253): 121-129. |
| [78] | Al-Homaidan AA, Al-Abbad AF, Al-Hazzani AA, Al-Ghanayem AA, Alabdullatif JA. Lead removal by Spirulina platensis biomass. International Journal of Phytoremediation, 2016, 18(2): 184-189. DOI:10.1080/15226514.2015.1073673 |
| [79] | Lu N, Wei D, Jiang XL, Chen F, Yang ST. Fatty acids profiling and biomarker identification in snow alga Chlamydomonas nivalis by NaCl stress using GC/MS and multivariate statistical analysis. Analytical Letters, 2012, 45(10): 1172-1183. DOI:10.1080/00032719.2012.673094 |
| [80] | Lu N, Wei D, Chen F, Yang ST. Lipidomic profiling reveals lipid regulation in the snow alga Chlamydomonas nivalis in response to nitrate or phosphate deprivation. Process Biochemistry, 2013, 48(4): 605-613. DOI:10.1016/j.procbio.2013.02.028 |
| [81] | Lu N, Wei D, Chen F, Yang ST. Lipidomic profiling and discovery of lipid biomarkers in snow alga Chlamydomonas nivalis under salt stress. European Journal of Lipid Science and Technology, 2012, 114(3): 253-265. DOI:10.1002/ejlt.201100248 |
| [82] | Lu N, Chen JH, Wei D, Chen F, Chen G. Global metabolic regulation of the snow alga Chlamydomonas nivalis in response to nitrate or phosphate deprivation by a metabolome profile analysis. International Journal of Molecular Sciences, 2016, 17(5): 694. DOI:10.3390/ijms17050694 |
| [83] | Huseby S, Degerlund M, Zingone A, Hansen E. Metabolic fingerprinting reveals differences between northern and southern strains of the cryptic diatom Chaetoceros socialis. European Journal of Phycology, 2012, 47(4): 480-489. DOI:10.1080/09670262.2012.741714 |
| [84] |
Lv HX, Qi BB, Cui XG, Jia SR. Metabolic analysis of response of Dunaliella salina to nitrogen, phosphorus and sulfur deprivation. Journal of Tianjin University of Science & Technology, 2018, 33(6): 9-15.
(in Chinese) 吕和鑫, 齐兵兵, 崔相敢, 贾士儒. 氮源、磷源、硫源共同饥饿胁迫下盐生杜氏藻代谢响应分析. 天津科技大学学报, 2018, 33(6): 9-15. |
| [85] | Taylor NS, Merrifield R, Williams TD, Chipman JK, Lead JR, Viant MR. Molecular toxicity of cerium oxide nanoparticles to the freshwater alga Chlamydomonas reinhardtii is associated with supra-environmental exposure concentrations. Nanotoxicology, 2016, 10(1): 32-41. |
| [86] | Yong WK, Sim KS, Poong SW, Wei D, Phang SM, Lim PE. Physiological and metabolic responses of Scenedesmus quadricauda (Chlorophyceae) to nickel toxicity and warming. 3 Biotech, 2019, 9(8): 315. DOI:10.1007/s13205-019-1848-8 |
| [87] | Niu XF, Zhu Y, Pei GS, Wu LN, Chen L, Zhang WW. Elucidating butanol tolerance mediated by a response regulator Sll0039 in Synechocystis sp. PCC 6803 using a metabolomic approach. Applied Microbiology and Biotechnology, 2015, 99(4): 1845-1857. DOI:10.1007/s00253-015-6374-y |
| [88] | Bogaert KA, Perez E, Rumin J, Giltay A, Carone M, Coosemans N, Radoux M, Eppe G, Levine RD, Remacle F, Remacle C. Metabolic, physiological, and transcriptomics analysis of batch cultures of the green microalga Chlamydomonas grown on different acetate concentrations. Cells, 2019, 8(11): 1367. DOI:10.3390/cells8111367 |
| [89] | Aguilera-Sáez LM, Abreu AC, Camacho-Rodríguez J, González-López CV, Cerón-García M, Fernández I. NMR Metabolomics as an effective tool to unravel the effect of light intensity and temperature on the composition of the marine microalgae Isochrysis galbana. Journal of Agricultural and Food Chemistry, 2019, 67(14): 3879-3889. DOI:10.1021/acs.jafc.8b06840 |
| [90] | Lv HX, Xia F, Liu M, Cui XG, Wahid F, Jia SR. Metabolomic profiling of the astaxanthin accumulation process induced by high light in Haematococcus pluvialis. Algal Research, 2016(20): 35-43. |
 2021, Vol. 61
2021, Vol. 61




