中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 林丹丹, 刘一凡, 刘忠林, 周蕾, 牟伯中. 2021
- Dandan Lin, Yifan Liu, Zhonglin Liu, Lei Zhou, Bo-Zhong Mu. 2021
- 古丸菌Archaeoglobi的代谢特征
- Metabolic characteristics of Archaeoglobi
- 微生物学报, 61(6): 1399-1415
- Acta Microbiologica Sinica, 61(6): 1399-1415
-
文章历史
- 收稿日期:2021-03-26
- 修回日期:2021-04-20
- 网络出版日期:2021-05-11
古丸菌目(Archaeoglobaceae),作为古丸菌纲(Archaeoglobi)下的唯一的科,包含3个属:古丸菌属(Archaeoglobus)、地丸菌属(Geoglobus)和铁丸菌属(Ferroglobus),三者均为嗜热型专性厌氧菌,包含化能自养型(氢营养型)和异养型两种营养模式;同时,基于可利用的外源末端电子受体分类,Archaeoglobi还可分为硫酸盐还原、硝酸盐还原和铁还原菌等,分别可以使用硫氧阴离子、结晶性较弱的三价铁(Fe3+)和硝酸盐等作为代谢末端电子受体进行代谢[1]。
从1987年发现第一株古丸菌Archaeoglobus fulgidus VC-16[2]到现在为止,已分离得到11株Archaeoglobi菌株。目前分离到的Archaeoglobus都是严格的厌氧菌,其中大多数是超嗜热硫酸盐还原菌。Archaeoglobus作为一种化能自养型(氢营养型)或化能异养型微生物,可利用硫酸盐、亚硫酸盐或硫代硫酸盐作为电子受体代谢,释放硫化氢(H2S)。Geoglobus和Ferroglobus可利用Fe3+作为电子受体生长,与Geoglobus不同,Ferroglobus还能利用其他的电子受体进行生长,如硫代硫酸盐和硝酸盐等。
虽然在系统发育上接近产甲烷古菌,但Archaeoglobus一直被认为是异养硫酸盐还原古菌。不同于氢营养型产甲烷菌的是,虽然在Archaeoglobi基因组中发现了涉及古菌型Wood-Ljungdahl (WL,即乙酰辅酶A途径)途径中的多个基因,但在目前分离的菌株基因组中都没发现产甲烷途径中的关键酶甲基辅酶M还原酶(MCR)和甲基转移酶复合物(MTR)的基因,同时相关生理生化实验也证明这些菌株不具备产甲烷功能。最近研究发现环境样品宏基因组中存在多个未培养的Archaeoglobi基因组,在这些基因组中存在编码MCR和MTR的基因,形成一条完整的产甲烷通路[3-4]。进一步基于基因组进化发育分析的研究推测Archaeoglobi的MCR和MTR可能在进化过程中丢失[3],同时向其他更有效的能量代谢进化,如异化硫酸盐还原方向。来自Archaeoglobi的异化硫酸盐还原序列(dsrAB)的系统发育分析也表明Archaeoglobi中的dsrAB基因可能是通过水平基因转移机制从其他细菌处获得的[3]。“Archaeoglobi保留古菌WL途径的一些或全部酶”这一发现使Archaeoglobi成为微生物学研究热点。因此本文总结了关于Archaeoglobi菌的研究进展,并对今后的研究进行了一些展望。
1 古丸菌的分类及系统发育 1.1 分类及菌株发现1987年,Stetter[2]从来自意大利Vulcano和Stufe di Nerone附近海洋热液系统的高温沉积物中分离到第一株古丸菌,即A. fulgidus VC-16。随后从北海的油藏环境中也分离得到其他A. fulgidus菌株。Archaeoglobus是一类严格厌氧的极端嗜热古菌(最适生长温度60–95 ℃),能够通过还原硫酸盐来氧化多种有机碳源[5]。除了海洋环境,陆上油藏环境具有高温和缺氧两个特点,也成为此类菌的适宜生存环境之一,其中一株A. fulgidus strain 7324就是从北海油藏水环境中分离得到[6]。迄今为止,Archaeoglobus属包含Archaeoglobus fulgidus[2]、Archaeoglobus profundus[7]、Archaeoglobus veneficus[8]、Archaeoglobus infectus[9]、“Archaeoglobus lithotrophicus”[10]、Archaeoglobus sulfaticallidus[11] 6个种,其中“Archaeoglobus lithotrophicus”尚未得到充分描述;Geoglobus仅发现两种菌株,分别为Geoglobus ahangari和Geoglobus acetivorans。目前已报道的Archaeoglobi各菌株生理生化特征可参见表 1,可见在许多海洋(如热液喷泉)、陆地(热液系统和油藏)和深层环境样品中均检测到了Archaeoglobi的16S rRNA基因序列,表明该纲的成员广泛分布于高温环境中。目前发现的Archaeoglobi主要存在于海洋热液,其次在南海热油环境中。
| Characteristic | Morphology/μm | NaCl (W/V)(opt) | pH (opt) | T (opt)/℃ | G+C/% | Location | References |
| 1 | 0.4-1.2 regular to irregular coccoid | 0.5-4.5 | 5.5-8.0 | 60-95 (83) | 46.0 | Marine hydrothermal system, Italy | [1] |
| 2 | 1.0-1.5 irregular coccoid | (7.0) | 60-90 (75-80) | 45.0 | Volcanic environment in Vulcano, Italy | [12] | |
| 3 | 0.3-1.0 irregular coccoid | 0.03-5.0 (3.0) | 6.4-7.4 (6.9) | 60-85 (76) | 47.0 | Hot North Sea Oil Field Water, Italy | [10] |
| 4 | 1.3 irregular coccoid | 0.9-3.6 (1.8) | 4.5-7.5 (6.0) | 65-90 (82) | 41.0 | Marine hydrothermal system, Guaymas, Mexico | [7] |
| 5 | 0.5-1.2 irregular coccoid | 0.5-4.0 (2.0) | 6.5-8.0 (7.0) | 65-85 (80) | 45.0 | Mid-Atlantic ridge | [8] |
| 6 | 0.5-1.0 irregular coccoid | 1.0-4.0 (3.0) | 6.5-7.0 (6.5) | 60-75 (70) | 45.9 | Deep-sea hydrothermal field Suiyo Seamount, Izu-Bonin Arc, western Pacific Ocean | [9] |
| 7 | 0.4-1.2 regular to irregular coccoid | 0.6-4.8 | 6.0-8.5 | 63-89 (80) | 40.0 | Fluids in a North Sea oilfield | [10] |
| 8 | 0.4-2.2 irregular lobes or triangles | 0.5-3.5 (2.0) | 6.3-7.6 (7.0) | 60-80 (75) | 42.0 | Eastern flank of Juan de Fuca Ridge, eastern Pacific Ocean | [11] |
| 9 | 0.3-0.5 irregular coccoid | 0.9-3.8 (1.9) | 5.0-7.6(7.0) | 65-90 (88) | 58.7 | Guaymas Basin hydrothermal system, Mexico | [13] |
| 10 | 0.3-0.5 coccoid | 1.0-6.0 (2.5) | 5.0-7.5 (6.8) | 50-85 (81) | 46.8 | Deep-sea hydrothermal vent, Ashadzefield, Mid-Atlantic Ridge | [14-15] |
| 11 | 0.7-1.3 coccoid | 0.5-4.5 (1.8-2.0) | 6.0-8.5 (7.0) | 65-95 (85) | 43.0 | Hydrothermal vent system at Vulcano Island, Italy | [16] |
| 1: A. fulgidus VC-16; 2: A. fulgidus Z; 3: A. fulgidus Strain 7324; 4: A. profundus AV18T; 5: A. veneficus SNP6; 6: A. infectus Arc51T; 7: A. lithotrophicus TF2; 8: A. sulfaticallidus PM70-1T; 9: G. ahangari 234T; 10: G. acetivorans SBH6T; 11: F. placidus AEDII12DOT. | |||||||
1.2 系统发育
最初针对A. fulgidus VC-16的16S rRNA基因的进化发育学分析结果显示,Archaeoglobus在系统发育树上位于甲烷球菌(Methanococcales)和热球菌目(Thermococcales)的分支之间[17]。随着Archaeoglobi成员的不断扩充,在这个基础上重新分析了它们的16S rRNA基因的进化发育分类,发现Archaeoglobi分支被划分在了甲烷杆菌目(Methanobacteriales)和甲烷微菌目(Methanomicrobiales)两大簇之间。此外,系统发育分析还显示Archaeoglobus分支与Methanococcales更加紧密(图 1)[18]。A. profundus与F. placidus高度紧密,具有96.5%的序列相似性,虽然序列同一性足够高,指示这两个生物体可包含在同一属中,但它们的生理差异是巨大的[1]。Geoglobus与Archaeoglobus和Ferroglobus差距较大,G. acetivorans与其最接近的亲缘种G. ahangari有97%的相似性,与其他Archaeoglobus相似性为94.9%–95.3%[14]。另外G. ahangari与A. fulgidus最相似(96.2%),与其他Archaeoglobi具有94.1%–95.1%相似性[13]。
2 化能自养
自养微生物可以通过多种生化途径进行无机碳的固定,目前已发现有6种自养固碳途径,分别为Wood-Ljungdal途径、卡尔文循环、还原性柠檬酸循环、3-羟基丙酸途径、3-羟基丙酸/4-羟基丁酸循环和二羧酸/4-羟基丁酸循环(图 2)[20]。其中Wood-Ljungdal途径可能是最古老的自养固碳途径,同时也是唯一的非循环型途径,该途径上的酶都是严格的厌氧酶。Wood-Ljungdal途径利用两分子CO2合成乙酰-CoA,其中的关键酶为一氧化碳脱氢酶,通过催化CoASH,CO和甲基转化为乙酰-CoA,这是此途径的最后一步[21-23]。该途径通常存在于产甲烷菌和产乙酸菌中,此外在Archaeoglobi中(除A. profundus)也利用该途径进行CO2的固定[20, 24]。
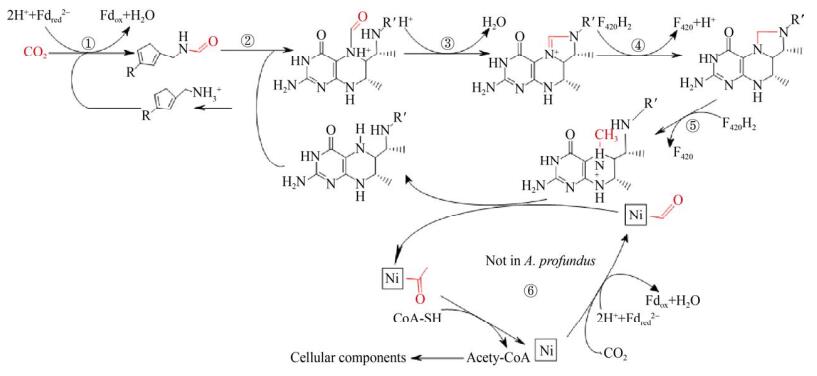
|
| 图 2 在Archaeoglobi中的CO2固定途径[20] Figure 2 CO2 fixation pathway in Archaeoglobi[20]. The enzymes involved in each reaction are: ① formylmethylfuran dehydrogenase; ② formylmethylfuran: H4MPT formyl transferase; ③ N5, N10-methenyl- H4MPT cyclohydrolase; ④ N5, N10-methylene- H4MPT dehydrogenase; ⑤ N5, N10-methylene- H4MPT reductase; ⑥ CO dehydrogenase- acetyl-CoA-synthase. |
在厌氧条件下,Archaeoglobi通过氧化有机底物或分子态氢(在某些情况下利用Fe2+或S2–) 进行代谢。除A. profundus和A. infectus外,Archaeoglobi中所有的菌株均能以化能自养的方式生长,其中“A. lithotrophicus”仅以CO2作为碳源,以H2作为能源和电子供体,硫酸盐为电子受体进行化能自养生长,不能进行异养生长[9]。
Archaeoglobi中含有用于自养型CO2固定的所有酶(图 2),其中催化CH3-H4MPT+还原为CH2=H4MPT (图 2中的第4步)和CH2=H4MPT还原为CH3-H4MPT的酶(图 2中的第5步)需要还原型的辅酶F420作为电子供体,而还原型的辅酶F420的再生需要通过H2的氧化来完成。CO2的固定需要还原2个CO2,一个在CO脱氢酶的作用下被还原为镍原子结合的CO,另一个还原为与四氢蝶呤载体结合的甲基。CO脱氢酶还充当乙酰-CoA合酶,通过甲基化的类卟啉蛋白从甲基化的四氢蝶呤接受甲基,与CO结合形成酶结合的Ni-乙酰基,然后与CoA释放该基团以形成乙酰-CoA[20, 25],参与细胞合成。自养型Archaeoglobi由于严格受限于厌氧条件,需要大量的一碳单位来进行能量代谢,WL途径产生的乙酰-CoA正好解决了这个问题。
在A. fulgidus的基因组中除了含有编码Wood- Ljungdal途径相关酶的基因以外,还存在编码其他CO2固定方式的基因:羟基丙酸酯-羟基丁酸酯循环、二羧酸-羟基丁酸酯循环和核糖-1, 5-双磷酸羧化酶/加氧酶(RubisCO)[25-27],但研究结果显示这些酶没有参与到Archaeoglobus的CO2固定中[28],在F. placidus基因组中同样存在编码RubisCO途径相关酶的基因,然而缺少编码Calvin-Benson循环相关酶的基因,所以F. placidus中的RubisCO可能参与到AMP循环途径而非CO2固定[29],在G. acetivorans和G. ahangari基因组中同样存在编码Wood-Ljungdahl途径相关的基因[15, 30]。
3 化能异养 3.1 糖类代谢A. fulgidus strain 7324能利用腺苷二磷酸(ADP)依赖性葡萄糖激酶进行淀粉代谢,该酶在A. fulgidusVC-16中不存在[31-32]。除淀粉以外,β-环糊精也能作为A. fulgidus的底物被降解代谢,而其他的糖类如葡萄糖、果糖和麦芽糖等则不能被利用。
A. fulgidus strain 7324能将淀粉降解为6-磷酸葡萄糖和葡萄糖,然而A. fulgidus的基因组中普遍缺少编码经典的淀粉降解途径Embden-Meyerhof途径的α淀粉酶和支链淀粉酶的基因[33]。根据这个现象,有学者提出A. fulgidus strain 7324中可能是通过修饰的Embden-Meyerhof途径将淀粉降解,转化为6-磷酸葡萄糖,最终生成丙酮酸[31]。
2007年,Labes[34]证实了A. fulgidus strain 724能通过以环糊精作为中间产物降解淀粉(图 3)。该途径涉及环糊精葡聚糖转移酶(CGTase)的细胞外作用,以及细胞内环糊精通过环糊精酶(CDase)、麦芽糖糊精磷酸化酶(Mal-P)和磷酸葡糖变性酶(PGM)降解为6-磷酸葡萄糖的过程。
在Geoglobus基因组中存在着部分修饰Embden-Meyerhof途径酶的编码基因,但是由于缺乏编码丙酮酸激酶的基因,也缺乏葡萄糖氧化相关的Entner-Doudoroff途径关键基因和戊糖氧化途径的相关基因,同样也没有相关生理生化实验验证Geoglobus具有代谢碳氢化合物的功能[15]。
3.2 乳酸氧化在A. fulgidus VC-16中乳酸通过丙酮酸和乙酰-CoA作为中间代谢物完全氧化生成CO2[35]。乳酸在乳酸脱氢酶作用下生成丙酮酸,丙酮酸:铁氧还蛋白氧化还原酶(PFOR)催化丙酮酸的氧化脱羧生成乙酰-CoA和CO2,从而将异养代谢与Wood-Ljungdahl途径相关联[24]。其中膜结合的D-乳酸脱氢酶(Dld) 需要Zn2+和核黄素辅酶FAD作为辅因子[36]。乙酰-CoA进一步氧化成CO2的过程中涉及C-C键的裂解反应,这步反应的关键酶为一氧化碳脱氢酶,它能在体外催化紫精染料将游离CO加H2O氧化为CO2,并催化CO2和乙酰-CoA的羰基之间的交换。交换反应既涉及乙酰-CoA的可逆碳-碳裂解,也涉及羰基向CO2的氧化[35]。乳酸的厌氧氧化途径类似于CO2固定的逆反应(图 4),以四氢甲蝶呤(H4MPT)作为甲基载体,复合体铁氧还蛋白作为电子载体,乙酰-CoA裂解的CH3-基团进一步氧化为CO2。该途径的独特特征是辅因子F420和甲呋喃的参与[37],F420充当N5, N10-亚甲基-H4MPT还原酶和N5, N10-亚甲基-H4MPT脱氢酶利用电子受体H2的媒介,然而到目前为止,甲酰甲烷呋喃脱氢酶的天然电子受体还是未知的。

|
| 图 4 乳酸完全代谢途径 Figure 4 Lactate is completey oxidiation pathways. ① Lactate dehydrogenase; ② Pyruvate: ferredoxin oxidoreductase; ③ Carbon monoxide dehydrogenase; ④ N5, N10-methylene-H4MPT reductase; ⑤ N5, N10- methylene-H4MPT dehydrogenase; ⑥ N5, N10-methylenyl- H4MPT cyclohydrolase; ⑦ Formylmethylfuran: H4MPT formyl transferase; ⑧ Formylmethylfuran dehydrogenase. |
A. fulgidus VC-16在硫酸盐还原条件下能将1分子的乳酸完全氧化为3分子CO2,而在A. fulgidus Z中,乳酸被不完全代谢成乙酸盐和CO2[38],从图 5中可看出,在不同的硫酸盐浓度条件下,乳酸代谢途径存在差异。
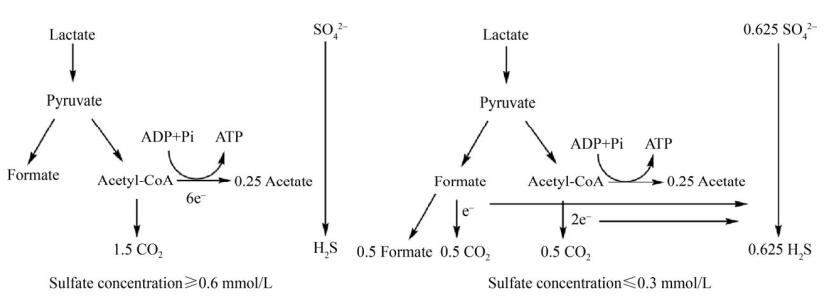
|
| 图 5 A. fulgidus strain Z分别在高浓度硫酸盐和低浓度硫酸盐条件下的乳酸代谢途径 Figure 5 Lactate metabolism pathway of A. fulgidus strain Z under the conditions of high concentration of sulfate and low concentration of sulfate, respectively. |
3.3 长链烷烃厌氧降解
Archaeoglobi能以原油为底物生长[39],我们对嗜热产甲烷厌氧氧化长链烷烃的富集体系进行测序分析,发现在没有硫酸盐作为末端电子受体的情况下,Archaeoglobi占比较大,如A. fulgidus和F. placidus等,表明Archaeoglobi参与长链烷烃的厌氧降解[39]。
A. fulgidus VC-16在硫代硫酸盐或硫酸盐的存在下能够氧化长链饱和烷烃(C10–C21),然而具体的降解机理目前还是未知。通过转录组分析对比发现VC-16基因组中pflD编码的丙酮酸甲酸酯裂解酶在烷烃降解体系中表达量比在脂肪酸降解体系中显著提高[5]。同时在进化发育树上,该类pflD基因与催化烷烃富马酸加成反应的烷基琥珀酸盐合成酶(assA)基因亲缘性较高[40]。因此推测在厌氧条件下,VC-16可能通过在烷烃次末端碳上加入一个富马酸分子方式来活化烷烃,生成的脂肪酸类产物通过β-氧化途径进一步降解为CO2[41]。然而,PflD酶的具体功能还需要进一步通过体外实验来验证。
3.4 脂类代谢早在1997年对A. fulgidus VC-16的全基因组分析时发现存在与β-氧化相关的基因,但直到2010年Archaeoglobus能利用长链脂肪酸(C4–C18)生长才得到验证[42]。在以硫代硫酸盐作为电子受体的情况下,A. fulgidus通过β-氧化降解代谢脂肪酸,首先生成脂酰CoA,然后进入β-氧化代谢,最终代谢生成CO2和硫化物。A. fulgidus利用脂肪酸代谢的能力与和它在系统发育树上亲缘相近的G. ahangari类似。
3.5 芳香族化合物降解F. placidus作为迄今为止唯一具有厌氧代谢芳香族化合物能力的嗜热古菌,可利用Fe3+为电子受体厌氧降解芳香族化合物,如苯、苯甲酸和苯酚等[29, 43]。在羧化酶的作用下,F. placidus以苯为单一的电子供体,代谢生成苯甲酸[44]。苯甲酸作为中间代谢物,在一系列酶的作用下生成3-羟基亚氨酰-CoA[44] (图 6)。终产物3-羟基亚氨酰-CoA通过β-氧化转化为乙酰-CoA,最后经三羧酸循环完全氧化为CO2[15]。

|
| 图 6 F. placidus中苯的代谢途径 Figure 6 Metabolic pathway of benzene in F. placidus. ① Carboxylase; ② Benzoate-CoA ligase; ③ Benzoyl-CoA reductase; ④ Cyclohex-1-ene-1-carboxy-CoA hydratase; ⑤ 2-hydroxycyclohexane carboxyl-CoA dehydrogenase; ⑥ 2-ketocyclohexane carboxy-CoA hydrolase; ⑦ Pimelyl-CoA dehydrogenase; ⑧ Didehydro-pyroyl-CoA hydratase. |
虽然G. acetivorans存在苯甲酸降解的相关基因,但是并未观察到代谢苯甲酸的功能[45],猜测可能是其他芳香族底物经由这一途径进行代谢;也可能是苯甲酸代谢需要特定的环境,而这个环境是目前未知的[15]。
3.6 其他有机物代谢丙酮酸是乳酸降解过程中的一种产物,通过相关基因组序列以及丙酮酸降解实验,表明在不完全代谢乳酸情况下,A. fulgidus存在类似丙酮酸甲酸酯裂解酶(PFL),催化丙酮酸生成甲酸盐与乙酰-CoA,甲酸盐被代谢生成CO2[26, 38]。而在A. fulgidus VC-16完全氧化乳酸的过程中,PFOR将丙酮酸直接氧化成CO2和乙酰-CoA[31]。丙酮酸发酵过程中产生的乙酰-CoA约有75%可能会通过C1-CO脱氢酶途径被完全氧化成CO2[35],而其余部分可能转化为乙酸盐,这步可能是由形成ADP的乙酰-CoA合成酶催化的[31]。
在硫代硫酸盐存在下,A. fulgidus Z和VC-16均可利用甲酸盐生长[12]。菌株Z可能仅在低水平的硫酸盐下氧化甲酸盐;当培养基中不存在硫酸盐时,A. fulgidus停止生长[12]。除A. veneficus和A. fulgidus能代谢甲酸盐以外,G. ahangari和G. acetivorans在以Fe3+为电子供体的条件下,也能代谢甲酸盐。
4 硫化物呼吸在厌氧环境中,硫酸盐还原是Archaeglobus主要的呼吸方式,并且是厌氧生态系统中硫循环的重要组成部分。Archaeoglobus可以利用硫酸盐或其他硫氧阴离子如硫代硫酸盐、亚硫酸盐作为电子受体氧化乙酸盐、乳酸和氢气等电子供体,同时产生二价硫化合物[46]。
硫酸盐还原被划分为异化硫酸盐还原和同化硫酸盐还原两类,异化硫酸盐还原途径是现代海洋沉积物菌厌氧降解有机物的主要方式,Archaeglobus作为典型的厌氧微生物,异化硫酸盐还原为能量代谢的重要方式[47-48]。
4.1 硫酸盐还原在Archaeoglobi中,硫酸盐(SO42–)通过高度保守的硫酸盐还原菌(sulfate-reducing prokaryotes,SRP)经异化硫酸盐还原途径还原为硫化物(S2–),这可能是Archaeoglobi通过SRP的早期祖先多个侧向基因转移事件获得的[49]。
如在硫酸盐还原菌(sulfate-reducing bacterial,SRB)中所表征的,A. fulgidus包含完整的保守的异化硫酸盐还原途径[26]。在硫酸盐还原途径中,硫酸盐进入细胞后,首先在腺苷三磷酸(ATP)的作用下接受电子激活生成腺苷硫酸盐(APS),在此反应过程中会释放出无机焦磷酸盐(PPi),随后通过编码腺苷酸硫酸还原酶的AprAB基因产物将APS还原为亚硫酸盐(SO32–)和腺苷一磷酸(AMP),最后经6个电子转移,dsrABD基因的作用下被还原成硫化物,硫化物以硫化氢H2S气体形式释放[18, 26]。和醌相互作用的膜结合的氧化还原酶(QmoABC)复合物可能将电子转移连接5′-磷酸腺苷(APS),还原酶(AprAB)催化的硫酸盐还原的第一步还原步骤[49]。在A. fulgidus中,硫代硫酸盐(S2O32–)和亚硫酸盐(SO32–)都可代替硫酸盐作为电子受体,但不能被元素硫取代[26]。SO32–转变为S2–为不可逆反应,是决定硫酸盐还原最重要的一步,起着限速的作用。
硫酸盐还原过程中获得的能量用于细胞中的4个主要生命活动:(i) 维持细胞成分,(ii) 碳代谢和生长,(iii) 细胞的输出和输入,以及(iv)硫酸盐还原,其中ATP激活硫酸盐以形成5′-磷酸腺苷(APS),然后将其还原为硫化物[38](式1)。

|
公式(1) |
硫代硫酸盐的利用是所有Archaeoglobus和F. placidus的共同特性,除A. veneficus外,其不能还原硫酸盐,而只能还原硫酸盐[8]。虽然都能利用硫代硫酸盐,但不同的Archaeoglobus似乎有不同的酶系统可以还原硫代硫酸盐。
硫代硫酸盐得到两个电子,生成亚硫酸盐和硫化物,随后亚硫酸盐进入胞内,在亚硫酸盐还原酶(dsrABD)的作用下进行还原生成硫化物。此过程中,需要编码钼蝶呤氧化还原酶和铁硫聚簇。
5 产乙酸Archaeoglobus是目前所知的唯一一种能利用一氧化碳的硫酸盐还原古菌,并且具有CO高耐受性,在CO为电子受体、没有外部电子受体的生长条件下,A. fulgidus以产乙酸菌的形式利用CO生长,需要CO脱氢酶复合体(CODH) 转化CO为CO2[50-51]。Archaeoglobus通过乙酰-CoA途径将CO2还原为乙酸盐,其中关键酶为乙酰CoA/CO脱氢酶复合体(ACS/CODH)。A. fulgidus的基因组测序表明存在3个[Ni-Fe]-CODH,1个“细菌”型的单体CooS和2个古菌型(CdhAB)型的CODH[50]。由于编码酶复合物的基因的组成型表达,双功能ACS/CODH参与了A. fulgidus中CO的初级氧化。相反,单功能CODH (CooS)似乎在CO代谢中起较小的作用。在转录表达和差异调节的基础上,CdhAB-2可能催化CO氧化和CO同化。
CO2通常被甲氧甲烷呋喃脱氢酶还原成甲氧甲烷呋喃,随后将甲酰基从甲烷呋喃辅因子转移至四氢甲蝶呤,甲酰四氢甲蝶呤在A. fulgidus中经水解作用形成甲酸盐。生成的甲酸盐与CO在CdhAB-2/ACS的催化下生成乙酰CoA,随后乙酰-COA经ACS催化的底物水平磷酸化生成乙酸。
6 产甲烷 6.1 Wood-Ljungdahl代谢途径作为重要的代谢方式之一,Wood-Ljungdahl途径用于甲烷生成和CO2的固定[52-53]。
研究表明,在硫酸盐还原过程中,A. fulgidus在DsrC蛋白上生成2个二硫键(形成DsrC三硫),并通过膜结合的异二硫键还原酶样酶进行还原。Archaeoglobi还具有非常类似于产甲烷菌中Hdr/ Mvh复合物的胞质复合物[54]。因此可以作出这样的假设:Archaeoglobus碳固定过程中所需的Fdred可能是通过类似于产甲烷过程中发生的电子歧化机制产生的,即用DsrC三硫化物代替了产甲烷菌中的CoM-S-S-CoB[55] (图 7)。另外,异养生长的Archaeoglobales也可以利用古菌WL途径的反向反应,来氧化有机化合物[26]。
Euryarchaeota内的大多数非甲烷源谱系失去从甲烷生成代谢中获取能量的能力[56],导致产甲烷和非产甲烷古菌形成明显不同的谱系。然而目前为止还没有证据证明产甲烷的损失造成的原因,可能是由于产甲烷菌不能从另一种代谢中获取能量,这可能是迈向产甲烷损失的必要中间步骤。摆脱甲烷代谢可能会采取截然不同的方向,例如,Halobacteriales (嗜盐古菌目)借助细菌在进化过程中的侧向基因转移失去了MTR和MCR复合物以及WL途径,并适应不同的条件[57];相反,Archaeoglobi保留古菌WL途径的一些或全部酶,这是其祖先甲烷循环生活方式的残余[55, 58]。
尽管在系统发育上接近产甲烷的古菌,但Archaeoglobi的成员长期以来被认为是非产甲烷型古菌,在Archaeoglobi基因组中存在氢营养型甲烷生成和古菌典型的Wood-Ljungdahl (WL)途径中的基因[3]。
虽然尚不清楚Archaeoglobi的LCA编码的是常规还是不同的MCR,但以下4种情况解释了dsrAB和MCR在该谱系中的当前分布:(i) dsrAB经基因水平转移(HGT)到Archaeoglobi的LCA后,含不同的MCR复合体在Ca. P. marinifundus (Archaeoglobi MAG,Ca. Polytropus marinifundus gen. nov. sp. nov.)丧失这种代谢功能;(ii)将发散的MCR复合物经HGT进入Archaeoglobi的LCA中,然后在Archaeoglobaceae获得dsrAB后失去复合物;(iii) Ca. P. marinifundus和Archaeoglobaceae分别获得发散的MCR复合物和dsrAB;或(iv) dsrAB基因和MCR从祖先到Archaeoglobi的不同损失(编码2个操纵子)[59]。
通过对MCR的进化历史进行探索,发现传统的产甲烷菌在很大程度上与分支顺序一致,这表明已知产甲烷菌中的MCR的进化历史主要遵循垂直遗传。鉴于Archaeoglobi和甲烷之间的代谢相似之处,推测Archaeoglobi的最后共同祖先(LCA)编码了MCR,该复合体随后在细菌的dsrAB基因发生HGT后丢失。Archaeoglobi庞大的基因组信息和比较基因组学有利于阐述进化史,尤其是Archaeoglobi中发现编码MCR的谱系。MCR的进化历史非常复杂,有垂直遗传和HGT的证据。Archaeoglobi谱系编码不同的MCR将持续发现,并且随着它们在公共数据库中相关基因组表示形式的不断增加,它们在水热地下生物圈中的进化历史和代谢作用将变得越来越清晰[59]。
6.2 未分离菌株中的潜在代谢功能最近,从甲烷或烷烃代谢不相关的古菌谱系中鉴定出MCR基因[59-64],在Archaeoglobi中也得到了这样的菌株。到目前为止,有11株Archaeoglobi菌株得到分离,然而还存在大量未得到分离的菌,这里我们主要收集了7种Archaeoglobi:MAG-Bin 16、Bin 11、Bin 17、WYZ-LMO 1–3、JdFR-42,前6种MAG在系统发育学上显示靠近A. fulgidus,而JdFR-42更接近于A. sulfaticallidus[3-4]。
对收集的样品进行的宏基因组学分析表明,微生物群落结果表明许多新的Archaeoglobi菌株:Archaeoglobi WYZ-LMO 1–3、Bin 16、Bin 11、Bin 17,这几株均含有MCR和MTR复合体,存在完整的甲烷生成途径基因组。Liu等[3]将Bin16、Bin11、Bin74和LMO2进行命名为“Ca. Methanomixophus hydrogenotrophicum”。在“Ca. Methanomixophus hydrogenotrophicum” MAG中均发现操纵子mtrABCDEFGH,该操纵子由编码完整的甲基H4M(S)PT︰辅酶M甲基转移酶复合物相关基因组成。将MTR复合体的基因系统发育与基因组系统发育进行了比较,结果表明“Ca. Methanomixophus hydrogenotrophicum”的进化方式为垂直遗传。另外在“Ca. M. hydrogenotrophicum”中缺乏编码具有硫酸盐异化还原相关联的酶基因,即硫酸腺苷酸转移酶(Sat)、腺苷酸硫酸还原酶(aprAB)和异化亚硫酸盐还原酶(dsrAB)。“Ca. Methanomixophus hydrogenotrophicum”除了编码N5, N10-亚甲基-H4MPT还原酶(Mer)的基因外,基因组编码几乎完整的古菌型Wood-Ljungdahl途径。将LMO1和LMO3命名为“Ca. Methanomixophus dualitatem”,与“Ca. Methanomixophus hydrogenotrophicum”的基因组特征具有高度的相似性,含有MTR,MCR和F420非还原性氢酶(Mvh/Hdr)复合物。此外在“Ca. Methanomixophus dualitatem”中发现了编码Mer的基因。“Ca. Methanomixophus dualitatem”使用WL途径进行CO2固定,基因组具有额外的硫酸盐还原基因,包括Sat、aprAB和dsrAB,此外还有含有编码DsrC蛋白和DsrMKJOP复合物的基因,这表明“Ca. Methanomixophus dualitatem”成员可能会在单个细胞中进行硫酸盐依赖性厌氧甲烷氧化[3]。
此外,Archaeoglobi JdFR-42编码Wood- Ljungdahl途径和脂肪酸氧化途径的相关基因,这些途径与Ca. Syntrophoarchaeum spp.存在的代谢途径类似。因此,Archaeoglobi JdFR-42可能是另一种多碳烷烃降解菌。同样,Archaeoglobi JdFR-42不携带编码硫酸盐还原酶的基因,例如dsrAB和aprAB[4]。
7 展望随着单菌分离技术的成熟,越来越多的Archaeoglobi菌株得到分离,通过对Archaeoglobi生理生化和基因组序列的深入了解,有利于发现古菌与细菌和真核生物的差异。Archaeoglobi广泛的地理分布和多样的代谢潜能使其成为微生物生态学研究的一个热点。考虑到其在深海热液和油藏中参与复杂的碳代谢(如复杂化合物降解、产乙酸过程、产甲烷过程等),Archaeoglobi在全球碳元素的循环过程中可能发挥着重要作用。随着基因组测序手段的不断发现,Archaeoglobi在公共数据库中宏基因组数量的逐渐增加,结合生物信息技术,可以更好地了解Archaeoglobi中祖先的来源,其进化历史和代谢作用将变得越发清晰。代谢重组显示新型Archaeoglobi能氧化碳氢化合物,进行氨基酸发酵代谢并能利用多种电子受体进行代谢。系统发育学分析为新型MCR复合体在古菌之间的分布提出HGT假说,为MCR复合体和Archaeoglobi的进化提供新的见解。虽然对Archaeoglobi中存在的多种代谢途径有了初步的了解,但是到目前为止对这些途径还缺乏系统的认知,可能的原因是:(1) 实验室无法完全还原菌株生长环境,菌株在实验室条件下生长与环境中存在差异;(2) 极端的采样环境导致菌株的基因组发生变化,因此寻找最有利菌株生长的环境和采样手段是至关重要的,对代谢途径相关的功能基因和代谢潜能进行进一步探索将是未来的主要研究内容之一。
| [1] | Rosenberg E, Delong EF, Lory S, Stackebrandt EThompson F. The Prokaryotes. Springer Heidelberg New York Dordrecht London, Springer, 2014. |
| [2] | Stetter KO, Lauerer G, Thomm M, Neuner A. Isolation of extremely thermophilic sulfate reducers: evidence for a novel branch of archaebacteria. Science, 1987, 236(4803): 822-824. DOI:10.1126/science.236.4803.822 |
| [3] | Liu YF, Chen J, Zaramela LS, Wang LY, Mbadinga SM, Hou ZW, Wu XL, Gu JD, Zengler K, Mu BZ. Genomic and transcriptomic evidence supports methane metabolism in Archaeoglobi. mSystems, 2020, 5(2). DOI:10.1128/msystems.00651-19 |
| [4] | Wang YZ, Wegener G, Hou JL, Wang FP, Xiao X. Expanding anaerobic alkane metabolism in the domain of Archaea. Nature Microbiology, 2019, 4(4): 595-602. DOI:10.1038/s41564-019-0364-2 |
| [5] | Klenk HP, Clayton RA, Tomb JF, White O, Nelson KE, Ketchum KA, Dodson RJ, Gwinn M, Hickey EK, Peterson JD, Richardson DL, Kerlavage AR, Graham DE, Kyrpides NC, Fleischmann RD, Quackenbush J, Lee NH, Sutton GG, Gill S, Kirkness EF, Dougherty BA, McKenney K, Adams MD, Loftus B, Peterson S, Reich CI, McNeil LK, Badger JH, Glodek A, Zhou L, Overbeek R, Gocayne JD, Weidman JF, McDonald L, Utterback T, Cotton MD, Spriggs T, Artiach P, Kaine BP, Sykes SM, Sadow PW, D'Andrea KP, Bowman C, Fujii C, Garland SA, Mason TM, Olsen GJ, Fraser CM, Smith HO, Woese CR, Venter JC. The complete genome sequence of the hyperthermophilic, sulphate-reducing archaeon Archaeoglobus fulgidus. Nature, 1997, 390(6658): 364-370. DOI:10.1038/37052 |
| [6] | Beeder J, Nilsen RK, Rosnes JT, Torsvik T, Lien T. Archaeoglobus fulgidus isolated from hot north sea oil field waters. Applied and Environmental Microbiology, 1994, 60(4): 1227-1231. DOI:10.1128/AEM.60.4.1227-1231.1994 |
| [7] | Burggraf S, Jannasch HW, Nicolaus B, Stetter KO. Archaeoglobus profundus sp. nov., Represents a New Species within the Sulfate-reducing Archaebacteria. Systematic and Applied Microbiology, 1990, 13(1): 24-28. DOI:10.1016/S0723-2020(11)80176-1 |
| [8] | Huber H, Jannasch H, Rachel R, Fuchs T, Stetter KO. Archaeoglobus veneficus sp. nov., a novel facultative chemolithoautotrophic hyperthermophilic sulfite reducer, isolated from abyssal black smokers. Systematic and Applied Microbiology, 1997, 20(3): 374-380. DOI:10.1016/S0723-2020(97)80005-7 |
| [9] | Mori K, Maruyama A, Urabe T, Suzuki KI, Hanada S. Archaeoglobus infectus sp. nov., a novel thermophilic, chemolithoheterotrophic archaeon isolated from a deep-sea rock collected at Suiyo Seamount, Izu-Bonin Arc, western Pacific Ocean. International Journal of Systematic and Evolutionary Microbiology, 2008, 58(4): 810-816. DOI:10.1099/ijs.0.65422-0 |
| [10] | Stetter KO, Huber R, Blöchl E, Kurr M, Eden RD, Fielder M, Cash H, Vance I. Hyperthermophilic Archaea are thriving in deep North Sea and Alaskan oil reservoirs. Nature, 1993, 365(6448): 743-745. DOI:10.1038/365743a0 |
| [11] | Steinsbu BO, Thorseth IH, Nakagawa S, Inagaki F, Lever MA, Engelen B, Ovreas L, Pedersen RB. Archaeoglobus sulfaticallidus sp. nov., a thermophilic and facultatively lithoautotrophic sulfate-reducer isolated from black rust exposed to hot ridge flank crustal fluids. International Journal of Systematic and Evolutionary Microbiology, 2010, 60(12): 2745-2752. DOI:10.1099/ijs.0.016105-0 |
| [12] | Zellner G, Stackebrandt E, Kneifel H, Messner P, Sleytr UB, de Macario EC, Zabel HP, Stetter KO, Winter J. Isolation and characterization of a thermophilic, sulfate reducing archaebacterium, Archaeoglobus fulgidus strain Z. Systematic and Applied Microbiology, 1989, 11(2): 151-160. DOI:10.1016/S0723-2020(89)80055-4 |
| [13] | Kashefi K. Geoglobus ahangari gen. nov., sp. nov., a novel hyperthermophilic archaeon capable of oxidizing organic acids and growing autotrophically on hydrogen with Fe(III) serving as the sole electron acceptor. International Journal of Systematic and Evolutionary Microbiology, 2002, 52(3): 719-728. |
| [14] | Slobodkina GB, Kolganova TV, Querellou J, Bonch-Osmolovskaya EA, Slobodkin AI. Geoglobus acetivorans sp. nov., an iron(III)-reducing archaeon from a deep-sea hydrothermal vent. International Journal of Systematic and Evolutionary Microbiology, 2009, 59(11): 2880-2883. DOI:10.1099/ijs.0.011080-0 |
| [15] | Mardanov AV, Slododkina GB, Slobodkin AI, Beletsky AV, Gavrilov SN, Kublanov IV, Bonch-Osmolovskaya EA, Skryabin KG, Ravin NV. The Geoglobus acetivorans genome: Fe(III) reduction, acetate utilization, autotrophic growth, and degradation of aromatic compounds in a hyperthermophilic archaeon. Applied and Environmental Microbiology, 2015, 81(3): 1003-1012. DOI:10.1128/AEM.02705-14 |
| [16] | Hafenbradl D, Keller M, Dirmeier R, Rachel R, Roßnagel P, Burggraf S, Huber H, Stetter KO. Ferroglobus placidus gen. nov., sp. nov., a novel hyperthermophilic archaeum that oxidizes Fe2+ at neutral pH under anoxic conditions. Archives of Microbiology, 1996, 166(5): 308-314. DOI:10.1007/s002030050388 |
| [17] | Achenbach-Richter L, Stetter KO, Woese CR. A possible biochemical missing link among archaebacteria. Nature, 1987, 327(6120): 348-349. DOI:10.1038/327348a0 |
| [18] | Hartzell P, Reed DW. The genus Archaeoglobus. The Prokaryotes. New York, NY: Springer New York, 2006: 82-100. |
| [19] | Labes A, Ein ungewöhnlicher Stärkeabbauweg im hyperthermophilen archaeellen Sulfatreduzierer Archaeoglobus fulgidus Stamm 7324 und Charakterisierung Stärke-abbauender Enzyme aus geothermalen Habitaten. Christian-Albrechts-Universität, 2005. |
| [20] | Berg IA, Kockelkorn D, Ramos-Vera WH, Say RF, Zarzycki J, Hügler M, Alber BE, Fuchs G. Autotrophic carbon fixation in Archaea. Nature Reviews Microbiology, 2010, 8(6): 447-460. DOI:10.1038/nrmicro2365 |
| [21] | Ljungdahl LG. The autotrophic pathway of acetate synthesis in acetogenic bacteria. Annual Review of Microbiology, 1986, 40: 415-450. DOI:10.1146/annurev.mi.40.100186.002215 |
| [22] | Wood HG. Life with CO or CO2 and H2 as a source of carbon and energy. The FASEB Journal, 1991, 5(2): 156-163. DOI:10.1096/fasebj.5.2.1900793 |
| [23] | Thauer RK. MICROBIOLOGY: a fifth pathway of carbon fixation. Science, 2007, 318(5857): 1732-1733. DOI:10.1126/science.1152209 |
| [24] | Ragsdale SW. Enzymology of the wood-ljungdahl pathway of acetogenesis. Annals of the New York Academy of Sciences, 2008, 1125(1): 129-136. DOI:10.1196/annals.1419.015 |
| [25] | Vornolt J, Kunow J, Stetter KO, Thauer RK. Enzymes and coenzymes of the carbon monoxide dehydrogenase pathway for autotrophic CO2 fixation in Archaeoglobus lithotrophicus and the lack of carbon monoxide dehydrogenase in the heterotrophic A. profundus. Archives of Microbiology, 1995, 163(2): 112-118. DOI:10.1007/BF00381784 |
| [26] | Klenk HP, Clayton RA, Tomb JF, White O, Nelson KE, Ketchum KA, Dodson RJ, Gwinn M, Hickey EK, Peterson JD, Richardson DL, Kerlavage AR, Graham DE, Kyrpides NC, Fleischmann RD, Quackenbush J, Lee NH, Sutton GG, Gill S, Kirkness EF, Dougherty BA, McKenney K, Adams MD, Loftus B, Peterson S, Reich CI, McNeil LK, Badger JH, Glodek A, Zhou LX, Overbeek R, Gocayne JD, Weidman JF, McDonald L, Utterback T, Cotton MD, Spriggs T, Artiach P, Kaine BP, Sykes SM, Sadow PW, D'Andrea KP, Bowman C, Fujii C, Garland SA, Mason TM, Olsen GJ, Fraser CM, Smith HO, Woese CR, Venter JC. The complete genome sequence of the hyperthermophilic, sulphate-reducing archaeon Archaeoglobus fulgidus. Nature, 1997, 390(6658): 364-370. DOI:10.1038/37052 |
| [27] | Finn MW, Tabita FR. Synthesis of catalytically active form III ribulose 1, 5-bisphosphate carboxylase/oxygenase in Archaea. Journal of Bacteriology, 2003, 185(10): 3049-3059. DOI:10.1128/JB.185.10.3049-3059.2003 |
| [28] | Estelmann S, Ramos-Vera WH, Gad'On N, Huber H, Berg IA, Fuchs G. Carbon dioxide fixation in 'Archaeoglobus lithotrophicus': are there multiple autotrophic pathways?. FEMS Microbiology Letters, 2011, 319(1): 65-72. DOI:10.1111/j.1574-6968.2011.02268.x |
| [29] | Anderson I, Risso C, Holmes D, Lucas S, Copeland A, Lapidus A, Cheng JF, Bruce D, Goodwin L, Pitluck S, Saunders E, Brettin T, Detter JC, Han C, Tapia R, Larimer F, Land M, Hauser L, Woyke T, Lovley D, Kyrpides N, Ivanova N. Complete genome sequence of Ferroglobus placidus AEDII12DO. Standards in Genomic Sciences, 2011, 5(1): 50-60. DOI:10.4056/sigs.2225018 |
| [30] | Manzella MP, Holmes DE, Rocheleau JM, Chung A, Reguera G, Kashefi K. The complete genome sequence and emendation of the hyperthermophilic, obligate iron-reducing archaeon "Geoglobus ahangari" strain 234T. Standards in Genomic Sciences, 2015, 10(1): 1-19. DOI:10.1186/1944-3277-10-1 |
| [31] | Labes A, Schönheit P. Sugar utilization in the hyperthermophilic, sulfate-reducing archaeon Archaeoglobus fulgidus strain 7324:starch degradation to acetate and CO2 via a modified Embden-Meyerhof pathway and acetyl-CoA synthetase (ADP-forming). Archives of Microbiology, 2001, 176(5): 329-338. DOI:10.1007/s002030100330 |
| [32] | Labes A, Schönheit P. ADP-dependent glucokinase from the hyperthermophilic sulfate-reducing archaeon Archaeoglobus fulgidus strain 7324. Archives of Microbiology, 2003, 180(1): 69-75. DOI:10.1007/s00203-003-0563-2 |
| [33] | Bender H. Cyclodextrin-Glucanotransferase von Klebsiella pneumoniae.. Archives of Microbiology, 1977, 111(3): 271-282. DOI:10.1007/BF00549366 |
| [34] | Labes A, Schönheit P. Unusual starch degradation pathway via cyclodextrins in the hyperthermophilic sulfate-reducing archaeon Archaeoglobus fulgidus strain 7324. Journal of Bacteriology, 2007, 189(24): 8901-8913. DOI:10.1128/JB.01136-07 |
| [35] | Möller-Zinkhan D, Thauer RK. Anaerobic lactate oxidation to 3 CO2 by Archaeoglobus fulgidus via the carbon monoxide dehydrogenase pathway: demonstration of the acetyl-CoA carbon-carbon cleavage reaction in cell extracts. Archives of Microbiology, 1990, 153(3): 215-218. DOI:10.1007/BF00249070 |
| [36] | Reed DW, Hartzell PL. The Archaeoglobus fulgidus d-lactate dehydrogenase is a Zn2+Flavoprotein. Journal of Bacteriology, 1999, 181(24): 7580-7587. DOI:10.1128/JB.181.24.7580-7587.1999 |
| [37] | Rabus R, Hansen TA, Widdel F. Dissimilatory Sulfate-and Sulfur-Reducing Prokaryotes. The Prokaryotes, Springer, New York: 659-768. |
| [38] | Habicht KS, Salling L, Thamdrup B, Canfield DE. Effect of low sulfate concentrations on lactate oxidation and isotope fractionation during sulfate reduction by Archaeoglobus fulgidus strain Z. Applied and Environmental Microbiology, 2005, 71(7): 3770-3777. DOI:10.1128/AEM.71.7.3770-3777.2005 |
| [39] | Mbadinga SM, Li KP, Zhou L, Wang LY, Yang SZ, Liu JF, Gu JD, Mu BZ. Analysis of alkane-dependent methanogenic community derived from production water of a high-temperature petroleum reservoir. Applied Microbiology and Biotechnology, 2012, 96(2): 531-542. DOI:10.1007/s00253-011-3828-8 |
| [40] | Khelifi N, Amin Ali O, Roche P, Grossi V, Brochier-Armanet C, Valette O, Ollivier B, Dolla A, Hirschler-Réa A. Anaerobic oxidation of long-chain n-alkanes by the hyperthermophilic sulfate-reducing archaeon, Archaeoglobus fulgidus. The ISME Journal, 2014, 8(11): 2153-2166. DOI:10.1038/ismej.2014.58 |
| [41] | Liu YF, Galzerani DD, Mbadinga SM, Zaramela LS, Gu JD, Mu BZ, Zengler K. Metabolic capability and in situ activity of microorganisms in an oil reservoir. Microbiome, 2018, 6(1): 5. DOI:10.1186/s40168-017-0392-1 |
| [42] | Khelifi N, Grossi V, Hamdi M, Dolla A, Tholozan JL, Ollivier B, Hirschler-Réa A. Anaerobic oxidation of fatty acids and alkenes by the hyperthermophilic sulfate-reducing archaeon Archaeoglobus fulgidus. Applied and Environmental Microbiology, 2010, 76(9): 3057-3060. DOI:10.1128/AEM.02810-09 |
| [43] | Tor JM, Lovley DR. Anaerobic degradation of aromatic compounds coupled to Fe(III) reduction by Ferroglobus placidus. Environmental Microbiology, 2001, 3(4): 281-287. DOI:10.1046/j.1462-2920.2001.00192.x |
| [44] | Holmes DE, Risso C, Smith JA, Lovley DR. Anaerobic oxidation of benzene by the hyperthermophilic archaeon Ferroglobus placidus. Applied and Environmental Microbiology, 2011, 77(17): 5926-5933. DOI:10.1128/AEM.05452-11 |
| [45] | Schleinitz KM, Schmeling S, Jehmlich N, von Bergen M, Harms H, Kleinsteuber S, Vogt C, Fuchs G. Phenol degradation in the strictly anaerobic iron-reducing bacterium Geobacter metallireducens GS-15. Applied and Environmental Microbiology, 2009, 75(12): 3912-3919. DOI:10.1128/AEM.01525-08 |
| [46] | Cassarini C, Rene ER, Bhattarai S, Esposito G, Lens PNL. Anaerobic oxidation of methane coupled to thiosulfate reduction in a biotrickling filter. Bioresource Technology, 2017, 240: 214-222. DOI:10.1016/j.biortech.2017.03.003 |
| [47] | Parey K, Warkentin E, Kroneck PMH, Ermler U. Reaction cycle of the dissimilatory sulfite reductase from Archaeoglobus fulgidus. Biochemistry, 2010, 49(41): 8912-8921. DOI:10.1021/bi100781f |
| [48] | Sim MS. Effect of sulfate limitation on sulfur isotope fractionation in batch cultures of sulfate reducing bacteria. Geosciences Journal, 2019, 23(5): 687-694. DOI:10.1007/s12303-019-0015-x |
| [49] | Hocking WP, Stokke R, Roalkvam I, Steen IH. Identification of key components in the energy metabolism of the hyperthermophilic sulfate-reducing archaeon Archaeoglobus fulgidus by transcriptome analyses. Frontiers in Microbiology, 2014, 5: 95. |
| [50] | Hocking WP, Roalkvam I, Magnussen C, Stokke R, Steen IH. Assessment of the carbon monoxide metabolism of the hyperthermophilic sulfate-reducing archaeon Archaeoglobus fulgidus VC-16 by comparative transcriptome analyses. Archaea: Vancouver, B C, 2015: 235384. |
| [51] | Henstra AM, Dijkema C, Stams AJM. Archaeoglobus fulgidus couples CO oxidation to sulfate reduction and acetogenesis with transient formate accumulation. Environmental Microbiology, 2007, 9(7): 1836-1841. DOI:10.1111/j.1462-2920.2007.01306.x |
| [52] | Berg IA. Ecological aspects of the distribution of different autotrophic CO2 fixation pathways. Applied and Environmental Microbiology, 2011, 77(6): 1925-1936. DOI:10.1128/AEM.02473-10 |
| [53] | Adam PS, Borrel G, Gribaldo S. An archaeal origin of the Wood-Ljungdahl H4MPT branch and the emergence of bacterial methylotrophy. Nature Microbiology, 2019, 4(12): 2155-2163. DOI:10.1038/s41564-019-0534-2 |
| [54] | Mander GJ, Pierik AJ, Huber H, Hedderich R. Two distinct heterodisulfide reductase-like enzymes in the sulfate-reducing archaeon Archaeoglobus profundus. European Journal of Biochemistry, 2004, 271(6): 1106-1116. DOI:10.1111/j.1432-1033.2004.04013.x |
| [55] | Borrel G, Adam PS, Gribaldo S. Methanogenesis and the wood-ljungdahl pathway: an ancient, versatile, and fragile association. Genome Biology and Evolution, 2016, 8(6): 1706-1711. DOI:10.1093/gbe/evw114 |
| [56] | Brochier C, Forterre P, Gribaldo S. Archaeal phylogeny based on proteins of the transcription and translation machineries: tackling the Methanopyrus kandleri paradox. Genome Biology, 2004, 5(3): 1-12. |
| [57] | Becker EA, Seitzer PM, Tritt A, Larsen D, Krusor M, Yao AI, Wu DY, Madern D, Eisen JA, Darling AE, Facciotti MT. Phylogenetically driven sequencing of extremely halophilic Archaea reveals strategies for static and dynamic osmo-response. PLoS Genetics, 2014, 10(11). |
| [58] | Bapteste É, Brochier C, Boucher Y. Higher-level classification of the Archaea: evolution of methanogenesis and methanogens. Archaea, 2005, 1(5): 353-363. DOI:10.1155/2005/859728 |
| [59] | Boyd JA, Jungbluth SP, Leu AO, Evans PN, Woodcroft BJ, Chadwick GL, Orphan VJ, Amend JP, Rappé MS, Tyson GW. Divergent methyl-coenzyme M reductase genes in a deep-subseafloor Archaeoglobi. The ISME Journal, 2019, 13(5): 1269-1279. DOI:10.1038/s41396-018-0343-2 |
| [60] | Berghuis BA, Yu FB, Schulz F, Blainey PC, Woyke T, Quake SR. Hydrogenotrophic methanogenesis in archaeal Phylum Verstraetearchaeota reveals the shared ancestry of all methanogens. Proceedings of the National Academy of Sciences of the United States of America, 2019, 116(11): 5037-5044. DOI:10.1073/pnas.1815631116 |
| [61] | Borrel G, Adam PS, McKay LJ, Chen LX, Sierra-García IN, Sieber CMK, Letourneur Q, Ghozlane A, Andersen GL, Li WJ, Hallam SJ, Muyzer G, de Oliveira VM, Inskeep WP, Banfield JF, Gribaldo S. Wide diversity of methane and short-chain alkane metabolisms in uncultured Archaea. Nature Microbiology, 2019, 4(4): 603-613. DOI:10.1038/s41564-019-0363-3 |
| [62] | McKay LJ, Dlakić M, Fields MW, Delmont TO, Eren AM, Jay ZJ, Klingelsmith KB, Rusch DB, Inskeep WP. Co-occurring genomic capacity for anaerobic methane and dissimilatory sulfur metabolisms discovered in the Korarchaeota. Nature Microbiology, 2019, 4(4): 614-622. DOI:10.1038/s41564-019-0362-4 |
| [63] | Seitz KW, Dombrowski N, Eme L, Spang A, Lombard J, Sieber JR, Teske AP, Ettema TJG, Baker BJ. Asgard Archaea capable of anaerobic hydrocarbon cycling. Nature Communications, 2019, 10(1): 1822. DOI:10.1038/s41467-019-09364-x |
| [64] | Chen SC, Musat N, Lechtenfeld OJ, Paschke H, Schmidt M, Said N, Popp D, Calabrese F, Stryhanyuk H, Jaekel U, Zhu YG, Joye SB, Richnow HH, Widdel F, Musat F. Anaerobic oxidation of ethane by Archaea from a marine hydrocarbon seep. Nature, 2019, 568(7750): 108-111. DOI:10.1038/s41586-019-1063-0 |
 2021, Vol. 61
2021, Vol. 61







