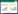中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 华康敏, 刘向阳, 邓子新, 贺新义, 蒋明. 2021
- Kangmin Hua, Xiangyang Liu, Zixin Deng, Xinyi He, Ming Jiang. 2021
- 酮基合酶AlpRQ影响醌那霉素的产量
- Ketosynthetases AlpRQ affects the yield of kinamycin
- 微生物学报, 61(5): 1328-1337
- Acta Microbiologica Sinica, 61(5): 1328-1337
-
文章历史
- 收稿日期:2020-07-06
- 修回日期:2020-08-31
- 网络出版日期:2021-01-28
聚酮化合物(polyketide,PK)是聚酮合酶(polyketide synthase,PKS)以短链羧酸为延伸单元,通过一系列重复的脱羧缩合反应生成的天然产物,其合成过程类似于脂肪酸的合成[1-2]。许多PK类的天然产物可用于临床治疗,比如抗细菌类、抗真菌类、抗癌类;降胆固醇;免疫抑制剂类;兽用抗生素等等[3-9]。除了已经投入生产和使用的聚酮类药物,还有很多具有生物活性的聚酮化合物尚未成药,值得科学家们进行更深入的研究。
根据合成过程中聚酮合酶的不同,聚酮化合物通常可分为三类,即Ⅰ型聚酮化合物(type Ⅰ PK),Ⅱ型聚酮化合物(type Ⅱ PK)和Ⅲ型聚酮化合物(type Ⅲ PK)。Ⅱ型聚酮化合物又被称为芳香型或迭代型聚酮化合物,它们多数是从革兰氏阳性菌放线菌中分离得到的,如柔红霉素(daunorubicin),土霉素oxytetracycline和醌那霉素(kinamycin)[10-13]。Ⅱ型聚酮类化合物的合成酶中包含了一个最小聚酮合酶(minimal PKS),它由聚酮合酶(KSα)、链长决定因子(KSβ)和酰基载体蛋白(ACP)组成。Ⅱ型聚酮化合物的起始单元首先是在这3个酶的作用下通过重复的脱羧缩合反应生成聚酮链的。
醌那霉素(kinamycin)是由Omura等于1970年在Streptomyces murayamaensis KA-295的发酵液中首次分离得到。之后,Gould等完成了几乎所有醌那霉素早期的生物合成研究。1985年,他们通过同位素喂养实验,即将[1, 2-13C2]标记的乙酸盐添加到S. murayamaensis菌株的发酵液中,继续发酵到结束后再用13C NMR进行检测与分析。检测结果表明醌那霉素是由乙酸盐单元组成的,说明醌那霉素是聚酮类化合物[14]。他们随后在S. murayamaensis发酵液中分离到了一个不属于醌那霉素家族的新化合物——dehydrorabelomycin (DHR),同样通过同位素喂养证明了DHR是醌那霉素合成过程中的一个中间体。之后Gould的团队又陆续从S. murayamaensis的发酵液中分离并鉴定了一些属于醌那霉素家族的化合物及其生物合成过程中生成的一系列中间体,其中包括了pre-kinamycin,keto-anhydrokinamycin,kinobscurinone,stealthin C,kinamycin E和kinamycin F[15-17]。
2011年,Bunet等证明了Streptomyces ambofaciens也能合成醌那霉素[18]。但在该菌株中,醌那霉素的生物合成基因簇是双拷贝的,这会给遗传操作带来不便。2018年,本课题组从构建好的BAC文库中钓取了包含醌那霉素完整生物合成基因簇的BAC,并在S. albus J1074中对醌那霉素进行了异源表达。与此同时还确定了醌那霉素合成基因簇的边界,其生物合成基因簇是从alpZ到alp2H,总长约72 kb,共有62个ORF[19]。在该菌株中,醌那霉素异源生物合成体系的建立为我们后续研究其每个基因所编码蛋白的具体功能提供了便利。醌那霉素合成基因簇如图 1所示。

|
| 图 1 醌那霉素生物合成基因簇 Figure 1 Biosynthetic gene cluster of kinamycin (alp cluster). |
Ⅱ型聚酮类天然产物的生物合成基因簇中包含了“minimal PKS”,通常这样的“minimal PKS”在芳香型聚酮化合物的生物合成基因簇中有且只有一个,但醌那霉素的生物合成基因簇比较特殊,它包含了两套酮基合酶(KSα和KSβ)的编码基因,即图 1中的alpA,alpB和alpR,alpQ。此前,已经有文献报导了alpA和alpB是醌那霉素合成过程中所必需的酮基合酶基因,alpR和alpQ则被认为是另一个Ⅱ型聚酮化合物生物合成的KSα和KSβ的必需基因,但没有对这两个蛋白进行研究的报道[20]。
近几年,在研究醌那霉素的生物合成机制方面有了很大的进展,科学家们探究了它的苯并芴骨架部分的生物合成过程,并对其生物合成途径中许多后修饰酶的功能进行了鉴定[19, 21-23]。图 2是在现有研究基础上对醌那霉素生物合成途径的一个推测。醌那霉素首先在最小聚酮合酶的作用下生产聚酮链骨架,然后在聚酮还原酶、环化酶、芳香化酶的作用下形成中间体UWM6,最后在多种后修饰酶的作用下形成高度氧化的A环和重氮官能团。本研究通过体内遗传学实验对醌那霉素生物合成基因簇中两套酮基合酶之间的关系进行了初步研究。

|
| 图 2 推测的醌那霉素合成途径 Figure 2 The proposed biosynthetic pathway of kinamycin. |
1 材料和方法 1.1 实验材料 1.1.1 菌种及质粒:
大肠杆菌DH10B(克隆宿主),ET12567/pUB307[24-25] (用于三亲本接合转移)和BW25113[26] (K12衍生菌株,用于基因敲除);链霉菌S. albus J1074 (异源表达宿主),S. albus/ΔalpW-2E9 (转入ΔalpW-2E9 BAC的出发菌株,产kinamycin D)以及本研究构建的S. albus/ ΔalpABΔalpW-2E9和S. albus/ΔalpRQΔalpW-2E9 (2个突变菌株),所有菌株均由上海交通大学分子微生物学实验室保存。
ΔalpW-2E9:包含醌那霉素完整合成基因簇的人工细菌染色体(BAC)2E9上,敲除负调基因alpW。在此基础上构建了另外2个BAC质粒(ΔalpABΔalpW-2E9和ΔalpRQΔalpW-2E9)。
1.1.2 培养基:大肠杆菌的液体培养基为LB (g/L):酵母提取物5,NaCl 5,胰蛋白胨10,加入去离子水使之完全溶解,121 ℃、1×105 Pa灭菌30 min;大肠杆菌的固体培养基为LA (g/L):即在LB的配方中添加1.5% (15 g)的琼脂粉,121 ℃、1×105 Pa灭菌30 min。
链霉菌的孢子活化培养基(g/L):黄豆饼粉20–25,加入1 L去离子水,121 ℃、1×105 Pa灭菌30 min。灭菌后过滤(六层纱布)到新的烧杯中,并加入甘露醇20 g,完全溶解后调pH至7.2–7.4,接着加入去离子水定容至1 L,最后加入2% (20 g)的琼脂粉,121 ℃、1×105 Pa灭菌30 min。
链霉菌的种子培养基为TSBY (g/L):蔗糖103,胰蛋白胨30,酵母提取物5,加入去离子水使之完全溶解,最后定容至1 L,115 ℃、1×105 Pa灭菌20 min;链霉菌的发酵培养基为R2 (g/L):蔗糖103,葡萄糖10,TES 5.6,酪蛋白氨基酸(Difco Casaminoacids) 0.1,MgCl2·6H2O 10.12,K2SO4 0.25,加入去离子水(950 mL左右)使之完全溶解,然后调pH至7.2,最后定容至970 mL,115 ℃、1×105 Pa灭菌20 min。在使用前加入15 mL 20%的L-脯氨酸,10 mL 0.5%的KH2PO4,5 mL 5 mol/L的CaCl2·2H2O以及2 mL微量元素溶液。微量元素溶液(1 L):FeCl3·6H2O 200 mg,ZnCl2 40 mg,CuCl2·2H2O 10 mg,MnCl2·4H2O 10 mg,Na2B4O7·10H2O 10 mg,(NH4)6Mo7O24·4H2O 10 mg,加入去离子水使之完全溶解,最后定容至1 L,121 ℃、1×105 Pa灭菌30 min。
1.1.3 PCR-targeting引物:设计了2对敲除引物,即S. gt-ΔalpAB-F:5ʹ-CGCGATGGCCACGCGCTT CTACAGCTGGGAGGCCTCCCGATTCCGGGGATCCGTCGACC-3ʹ;S. gt-ΔalpAB-R:5ʹ-GCGGTTG CGGTGCGTCGGTCGGGCGGTGTGCGGGGGTCCTGTAGGCTGGAGCTGCTTC-3ʹ;S. gt-ΔalpRQ- F:5ʹ-GTGAACCGCGTCGTGATCACCGGGATCGG CGTCGTCGCCCATTCCGGGGATCCGTCGACC-3ʹ;S. gt-ΔalpRQ-R: 5ʹ-TCACTCCGTCTGAGGGG CCCGGAGCACTGCGGCGGAGTTTGTAGGCTGGAGCTGCTTC-3ʹ。此外,还设计了2对验证引物,即S. gt-alpAB-flank-F:5ʹ-CCACCTCCAGGAC TTCGACGAGG-3ʹ;S. gt-alpAB-flank-R:5ʹ-CCCG CAGGGTGCGCTTGAGG-3ʹ;S. gt-alpRQ-flank-F:5ʹ-GCCCGTCCTGGTCTTCGC-3ʹ;S. gt-alpRQ-flank- R:5ʹ-AAGGCCAGGAGCCGGAAG-3ʹ。
1.1.4 主要试剂和仪器:PCR所用试剂、限制性内切酶购自TaKaRa公司;质粒提取试剂盒、胶回收试剂盒购自南京诺唯赞生物科技有限公司;HPLC购自安捷伦科技有限公司。
1.2 基因敲除本研究中基因敲除采用的是PCR-targeting方法。抗性基因模板是带有neo抗性(编码卡纳霉素或新霉素抗性)的长度为1.4 kb的片段,是用Xba I/EcoR I双酶切从pJTU4659质粒上切下来的。敲除引物与目的基因的同源臂长度均为39 bp。
1.3 三亲本接合转移本研究中基因敲除实验的接合转移步骤都是采用了三亲本接合转移的方式。敲除了目的基因并完成验证的BAC质粒首先是转入了大肠杆菌ET12567。在接合转移实验的前一天将该菌株与含有辅助质粒的ET12567/pUB307分别接种到添加了相应抗生素的LB培养基中,在摇床中培养过夜(37 ℃,220 r/min)。第2天分别取500 μL菌液,转接到添加了相应抗生素的5 mL LB培养基中,摇床(37 ℃,220 r/min)培养至OD600=0.6 (4 h左右)。培养结束后分别取1 mL菌液,12000 r/min离心1 min,倒掉上清,菌体用1 mL无抗LB洗涤2次(重悬,12000 r/min离心1 min,弃上清)。然后分别用300 μL无抗LB对菌体进行重悬。
接着取适量(1/3MS板)活化的链霉菌孢子到装有1 mL TES缓冲液的EP管中,12000 r/min离心30 s,弃上清。孢子用1 mL TES缓冲液洗涤2次(重悬,12000 r/min离心30 s,弃上清)。然后用500 μL TES缓冲液将孢子重悬起来,热激10 min (50 ℃),再加入500 μL的2×YT培养基,用枪头吸打混匀后摇床(37 ℃,220 r/min)培养1 h。1 h后离心(12000 r/min离心30 s),弃上清,孢子用300 μL的无抗LB重悬起来。最后将三管重悬的菌液混匀,均匀的涂在MS板上,待其吹干后恒温(30 ℃)培养13 h左右。第2天将相应抗生素添加到1 mL ddH2O中混匀,然后均匀覆盖到MS板上,恒温(30 ℃)培养3–4 d。
1.4 链霉菌的发酵和萃取接合转移3–4 d后,挑选单个的接合子进行抗性复证。接着取适量复证后的链霉菌孢子接种到含有相应抗生素的30 mL TSBY培养基中,摇床(30 ℃,220 r/min)培养1–2 d,即为种子液。然后取1 mL种子液加入装有30 mL无抗R2培养基和1.5 g HP-20填料的三棱瓶中(250 mL),摇床(30 ℃,220 r/min)培养2–3 d。
发酵完成后,每瓶发酵液中先加入300 μL冰醋酸,接着加入30 mL乙酸乙酯进行萃取。萃取过程中需要每隔10 min左右晃动混合液,使发酵液能够萃取充分,总的萃取时间为1–2 h。然后将混合液进行分离,3000 r/min离心5 min,离心之后将上层有机相转移到旋蒸瓶中进行旋转蒸发。有机相完全蒸发后,用600 μL甲醇充分溶解悬蒸瓶中的化合物,将溶解液转入1.5 mL EP管中,–30 ℃保存。
2 结果和分析 2.1 醌那霉素生物合成中两套聚酮合酶的序列分析醌那霉素完整生物合成基因簇中包含有2套聚酮合酶基因alpA/alpB和alpR/alpQ,这在Ⅱ型聚酮化合物的生物合成途径中是非常罕见的。然而,在alpR/alpQ的附近并没有找到编码酰基载体蛋白的基因,因此无法确定alpR/alpQ编码的蛋白是否参与生物合成。据报道,在Streptomyces ambofaciens中,alpR/alpQ编码的蛋白不是醌那霉素生物合成的必需基因。为了探究这两组聚酮合酶之间可能存在的关联,我们首先通过生物信息学的方法对它们进行了研究。前人通过同源对比,发现AlpR/AlpQ与已报道的光辉霉素(mithramycin)的聚酮合酶非常相似,同源性分别为80%和72%。从进化树结果来看,链长决定因子AlpB和AlpQ均属于聚酮骨架链长为20的分支(图 3)。前人的研究显示,AlpR/AlpQ对于醌那霉素的生物合成并不是必需的,不过由于AlpR/AlpQ可能参与合成链长20的Ⅱ型聚酮类化合物,我们也不能排除它们对于醌那霉素的生物合成具有补充作用。
|
| 图 3 MEGA-7软件以极大似然法绘制的AlpB,AlpQ与其他Ⅱ型聚酮化合物合成基因簇中KSβ的系统进化树分析 Figure 3 Phylogenetic tree geneterated by MEGA-7 with maximum likelihood method to cluster AlpB and AlpQ with other ketosynthases involved in other type Ⅱ polyketide biosynthesis. |
2.2 S. albus/ΔalpRQΔalpW-2E9和S. albus/ΔalpABΔalpW-2E9菌株的构建结果
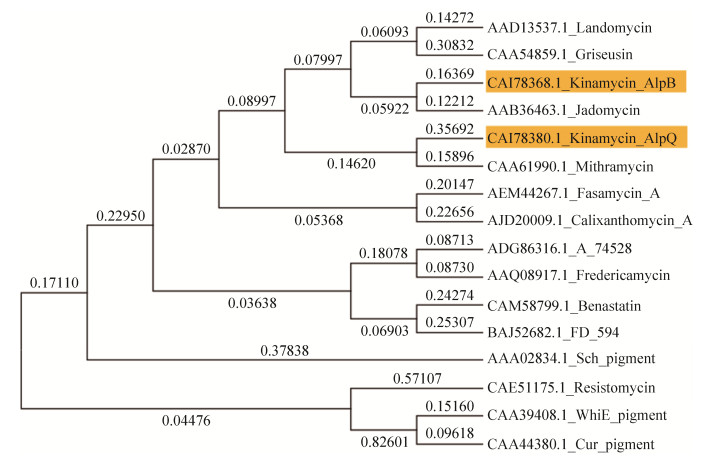
为了研究alpR/alpQ与醌那霉素的生物合成的潜在关系,我们在包含BAC ΔalpW-2E9的大肠杆菌BW25113中,利用PCR-targeting的方法分别同框缺失了alpA/alpB和alpR/alpQ;基因缺失过程及验证结果如图 4和图 5所示。然后通过三亲本接合转移的方式将BAC质粒ΔalpABΔalpW- 2E9和ΔalpRQΔalpW-2 E9分别转入异源表达菌株S. albus J1074中,得到了分别敲除两套酮基合酶的突变株,S. albus/ΔalpABΔalpW-2E9和S. albus/ ΔalpRQΔalpW-2E9。
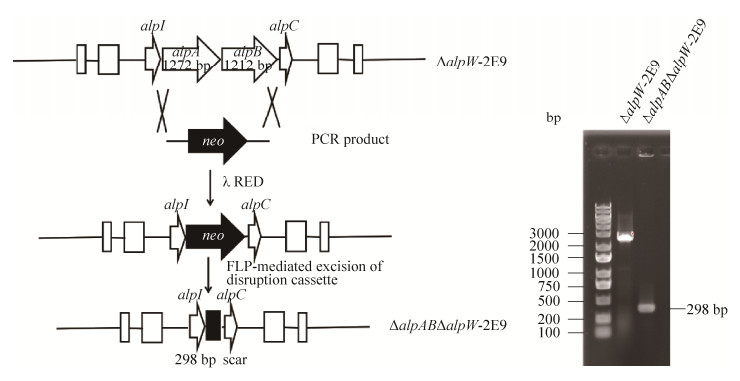
|
| 图 4 ΔalpABΔalpW-2E9 BAC质粒构建示意图及PCR验证 Figure 4 Schematic representation of mutant BAC of ΔalpABΔalpW-2E9 and PCR validation (S. gt-alpAB-flank-F and S. gt-alpAB-flank-R were used). |

|
| 图 5 ΔalpRQΔalpW-2E9 BAC质粒构建示意图及PCR验证 Figure 5 Schematic representation of mutant BAC of ΔalpRQΔalpW-2E9 and PCR validation (S. gt-alpRQ-flank-F and S. gt-alpRQ-flank-R were used). |
2.3 alpAB和alpRQ缺失突变对醌那霉素合成的影响
将构建好的突变BAC ΔalpABΔalpW-2E9和ΔalpABΔalpW-2E9三亲本接合转移至S. albus J1074中得到醌那霉素对应的突变菌,将它们与S. albus/ΔalpW-2E9 (可产生醌那霉素的菌株)在同样条件下进行发酵并检测发酵产物,检测结果如图 6所示。

|
| 图 6 HPLC检测alpAB或alpRQ敲除菌株的发酵产物 Figure 6 HPLC analysis of the products for kinamycin gene cluster with ΔalpAB or ΔalpRQ in S. albus J1074. i: S. albus/ΔalpW-2E9 (control); ii: S. albus/ΔalpABΔalpW-2E9; iii: S. albus/ΔalpRQΔalpW-2E9, HPLC traces were recorded at 276 nm. |
从发酵结果HPLC的分析可以看出,alpA/alpB敲除突变株失去了生物合成醌那霉素的能力(图 6,ii),说明它们编码的酮基合酶确实如文献报道的那样,是该基因簇合成醌那霉素聚酮链所必需的聚酮合酶。另一方面,该突变株完全丧失醌那霉素产生能力,也确证了AlpR/AlpQ并不能互补AlpA/AlpB的功能。突变菌株S. albus/ΔalpRQΔalpW-2E9不仅能够生产醌那霉素,其产量是出发菌株(S. albus/ΔalpW-2E9)的3.2倍。
alpR/alpQ的缺失使得醌那霉素的产量大幅提高,可能的原因是AlpR/AlpQ参与了另一种Ⅱ型聚酮的生物合成。它们的敲除使得AlpR/AlpQ参与的生物合成途径中断,导致与醌那霉素生物合成的共有前体流向了醌那霉素的生物合成中,从而有效地提高了醌那霉素的产量。为了验证AlpR/AlpQ是否真的参与合成了一种新的聚酮化合物,我们对S. albus/ΔalpABΔalpW-2E9和S. albus/ΔalpW-2E9的发酵液进行了仔细分析。除了醌那霉素及其相关产物外,并没有找到其他可能的Ⅱ型聚酮类产物。与突变菌株S. albus/ΔalpRQ ΔalpW-2E9相比,S. albus/ΔalpW-2E9的发酵液中也没有发现不同的化合物峰。
3 讨论和结论很多天然产物的生物合成基因簇都很大,有些甚至超过100 kb,包含数十个基因。醌那霉素的生物合成基因簇跨越长度约72 kb,包含了62个开放阅读框。其中,很多基因编码蛋白的功能得到了确认,比如那些参与到醌那霉素聚酮骨架合成的基因。但还是有一些基因的功能目前并不清楚。醌那霉素生物合成基因簇中包含有两组聚酮合酶,本研究通过体内遗传学实验证实了这两组聚酮合酶功能上并不相同。体内敲除实验证明:合成基因簇中的alpR和alpQ基因不直接影响醌那霉素的合成,即它们编码的蛋白不是醌那霉素生物合成中所必需的酮基合酶,可能与另一未知Ⅱ型聚酮化合物的合成相关。但本研究发现alpR/alpQ缺失突变株的醌那霉素产量显著提高,约为出发菌株的3倍。推测可能是因为AlpR、AlpQ和AlpA,AlpB这两套酮基合酶之间存在合成前体的竞争关系,所以敲除alpRQ之后,用于醌那霉素合成途径的底物增加了,从而使得终产量提高。
随着遗传操作技术的进步,提高天然产物产量的方法越来越多。一个常见的策略是通过调控蛋白提高参与天然产物合成的蛋白表达量。另外一个重要手段就是:阻断与目标产物竞争前体物质的代谢途径,从而增加目标产物生物合成途径的代谢流。许多天然产物的生物合成都是通过这种方式使得产量显著提高的。不过,通常都是敲除位于染色体上其他位置的生物合成基因簇。而在本研究中,通过敲除醌那霉素生物合成基因簇内部的聚酮合酶基因(alpR/alpQ),使得醌那霉素的产量得到了大幅提高,这是之前没有报道过的。
Ⅱ型聚酮化合物的合成酶系是由多个独立的酶组成的,其中最核心的成员又被称为最小聚酮合酶,包括酮基合酶、链长决定因子和酰基载体蛋白。alpR/alpQ附近并没有酰基载体蛋白,因此该Ⅱ型聚酮体系缺少了关键的酰基载体蛋白。这种情况在少数Ⅱ型聚酮孤儿基因簇中也发现过[27]。人们推测,这类型基因簇可能可以利用基因簇外的酰基载体蛋白,或者采用一个新的生物合成机制,也有可能基因簇是没有功能的。alpR/alpQ的缺失使得醌那霉素的产量大幅增加这一现象暗示着alpR/alpQ应该有功能。可是本研究中并不能发现alpR/alpQ的相关联产物,我们推测其关联产物在发酵液中没有检测到可能是相关产物不稳定,进一步的研究探索工作正在进行。在alpR/alpQ附近我们还发现了一个环化酶基因(alpM),乙酰辅酶脱氢酶基因(alpN),肽合成酶基因(alpP),它们形成一个相对独立的簇,前期研究显示它们可能均不与醌那霉素的生物合成相关。对这些基因编码蛋白功能的研究,能帮助我们揭示AlpR/AlpQ的功能以及它们和醌那霉素生物合成的关联。
| [1] | Hopwood DA. Genetic contributions to understanding polyketide synthases. Chemical Reviews, 1997, 97(7): 2465-2498. DOI:10.1021/cr960034i |
| [2] | Staunton J, Weissman KJ. Polyketide biosynthesis: a millennium review. Natural Product Reports, 2001, 18(4): 380-416. DOI:10.1039/a909079g |
| [3] | Du LC, Lou LL. PKS and NRPS release mechanisms. Natural Product Reports, 2010, 27(2): 255-278. DOI:10.1039/B912037H |
| [4] | Minotti G, Menna P, Salvatorelli E, Cairo G, Gianni L. Anthracyclines: molecular advances and pharmacologic developments in antitumor activity and cardiotoxicity. Pharmacological Reviews, 2004, 56(2): 185-229. DOI:10.1124/pr.56.2.6 |
| [5] | Crow RT, Rosenbaum B, Smith Ⅲ R, Guo Y, Ramos KS, Sulikowski GA. Landomycin A inhibits DNA synthesis and G1S cell cycle progression. Bioorganic & Medicinal Chemistry Letters, 1999, 9(12): 1663-1666. |
| [6] | Hou MH, Wang AHJ. Mithramycin forms a stable dimeric complex by chelating with Fe(Ⅱ): DNA-interacting characteristics, cellular permeation and cytotoxicity. Nucleic Acids Research, 2005, 33(4): 1352-1361. DOI:10.1093/nar/gki276 |
| [7] | Menéndez N, Nur-e-Alam M, Braña AF, Rohr J, Salas JA, Méndez C. Biosynthesis of the antitumor chromomycin A3 in Streptomyces griseus: analysis of the gene cluster and rational design of novel chromomycin analogs. Chemistry & Biology, 2004, 11(1): 21-32. |
| [8] | Keniry MA, Owen EA, Shafer RH. The three-dimensional structure of the 4:1 mithramycin: d(ACCCGGGT)2 complex: evidence for an interaction between the E saccharides. Biopolymers, 2000, 54(2): 104-114. DOI:10.1002/1097-0282(200008)54:2<104::AID-BIP3>3.0.CO;2-2 |
| [9] | Pioletti M, Schlünzen F, Harms J, Zarivach R, Glühmann M, Avila H, Bashan A, Bartels H, Auerbach T, Jacobi C, Hartsch T, Yonath A, Franceschi F. Crystal structures of complexes of the small ribosomal subunit with tetracycline, edeine and IF3. The EMBO Journal, 2001, 20(8): 1829-1839. DOI:10.1093/emboj/20.8.1829 |
| [10] | Chopra I, Roberts M. Tetracycline antibiotics: mode of action, applications, molecular biology, and epidemiology of bacterial resistance. Microbiology and Molecular Biology Reviews, 2001, 65(2): 232-260. DOI:10.1128/MMBR.65.2.232-260.2001 |
| [11] | Bartlett JJ, Trivedi PC, Yeung P, Kienesberger PC, Pulinilkunnil T. Doxorubicin impairs cardiomyocyte viability by suppressing transcription factor EB expression and disrupting autophagy. Biochemical Journal, 2016, 473(21): 3769-3789. DOI:10.1042/BCJ20160385 |
| [12] | Hasinoff BB, Wu X, Yalowich JC, Goodfellow V, Laufer RS, Adedayo O, Dmitrienko GI. Kinamycins A and C, bacterial metabolites that contain an unusual diazo group, as potential new anticancer agents: antiproliferative and cell cycle effects. Anti-Cancer Drugs, 2006, 17(7): 825-837. DOI:10.1097/01.cad.0000224442.78211.27 |
| [13] | Herzon SB, Woo CM. The diazofluorene antitumor antibiotics: structural elucidation, biosynthetic, synthetic, and chemical biological studies. Natural Product Reports, 2012, 29(1): 87-118. DOI:10.1039/C1NP00052G |
| [14] | Sato Y, Gould SJ. Biosynthesis of the kinamycin antibiotics by Streptomyces murayamaensis. determination of the origin of carbon, hydrogen, and oxygen atoms by carbon-13 NMR spectroscopy. Journal of the American Chemical Society, 1986, 108(15): 4625-4631. DOI:10.1021/ja00275a058 |
| [15] | Seaton PJ, Gould SJ. New products related to kinamycin from Streptomyces murayamaensis. Ⅱ. structures of pre-kinamycin, keto-anhydrokinamycin, and kinamycins E and F. The Journal of Antibiotics, 1989, 42(2): 189-197. DOI:10.7164/antibiotics.42.189 |
| [16] | Gould SJ, Melville CR. Kinamycin biosytheesis. Synthesis, detection, and incorporation of kinobscurinone, a benzo[b]fluorenone. Bioorganic & Medicinal Chemistry Letters, 1995, 5(1): 51-54. |
| [17] | Gould SJ, Melville CR, Cone MC, Chen J, Carney JR. Kinamycin biosynthesis. synthesis, isolation, and incorporation of stealthin C, an aminobenzo[b]fluorene. The Journal of Organic Chemistry, 1997, 62(2): 320-324. DOI:10.1021/jo961486y |
| [18] | Bunet R, Song LJ, Mendes MV, Corre C, Hotel L, Rouhier N, Framboisier X, Leblond P, Challis GL, Aigle B. Characterization and manipulation of the pathway-specific late regulator AlpW reveals Streptomyces ambofaciens as a new producer of Kinamycins. Journal of Bacteriology, 2011, 193(5): 1142-1153. DOI:10.1128/JB.01269-10 |
| [19] | Liu XY, Liu DX, Xu M, Tao MF, Bai LQ, Deng ZX, Pfeifer BA, Jiang M. Reconstitution of kinamycin biosynthesis within the heterologous host Streptomyces albus J1074. Journal of Natural Products, 2018, 81(1): 72-77. DOI:10.1021/acs.jnatprod.7b00652 |
| [20] | Pang XH, Aigle B, Girardet JM, Mangenot S, Pernodet JL, Decaris B, Leblond P. Functional angucycline-like antibiotic gene cluster in the terminal inverted repeats of the Streptomyces ambofaciens linear chromosome. Antimicrobial Agents and Chemotherapy, 2004, 48(2): 575-588. DOI:10.1128/AAC.48.2.575-588.2004 |
| [21] | Wang B, Guo F, Ren JW, Ai GM, Aigle B, Fan KQ, Yang KQ. Identification of Alp1U and Lom6 as epoxy hydrolases and implications for kinamycin and lomaiviticin biosynthesis. Nature Communications, 2015, 6: 7674. DOI:10.1038/ncomms8674 |
| [22] | Wang KKA, Ng TL, Wang P, Huang ZD, Balskus EP, van der Donk WA. Glutamic acid is a carrier for hydrazine during the biosyntheses of fosfazinomycin and kinamycin. Nature Communications, 2018, 9(1): 3687. DOI:10.1038/s41467-018-06083-7 |
| [23] | Wang B, Ren JW, Li LY, Guo F, Pan GH, Ai GM, Aigle B, Fan KQ, Yang KQ. Kinamycin biosynthesis employs a conserved pair of oxidases for B-ring contraction. Chemical Communications, 2015, 51(42): 8845-8848. DOI:10.1039/C5CC01986A |
| [24] | MacNeil DJ, Gewain KM, Ruby CL, Dezeny G, Gibbons PH, MacNeil T. Analysis of Streptomyces avermitilis genes required for avermectin biosynthesis utilizing a novel integration vector. Gene, 1992, 111(1): 61-68. DOI:10.1016/0378-1119(92)90603-M |
| [25] | Flett F, Mersinias V, Smith CP. High efficiency intergeneric conjugal transfer of plasmid DNA from Escherichia coli to methyl DNA-restricting streptomycetes. FEMS Microbiology Letters, 1997, 155(2): 223-229. DOI:10.1111/j.1574-6968.1997.tb13882.x |
| [26] | Datsenko KA, Wanner BL. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proceedings of the National Academy of Sciences of the United States of America, 2000, 97(12): 6640-6645. DOI:10.1073/pnas.120163297 |
| [27] | Hillenmeyer ME, Vandova GA, Berlew EE, Charkoudian LK. Evolution of chemical diversity by coordinated gene swaps in type Ⅱ polyketide gene clusters. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(45): 13952-13957. DOI:10.1073/pnas.1511688112 |
 2021, Vol. 61
2021, Vol. 61