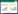中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 陈华枝, 蒋海宾, 祝智威, 范元婵, 许雅静, 孙明会, 刘佳美, 熊翠玲, 郑燕珍, 付中民, 徐国钧, 陈大福, 郭睿. 2021
- Huazhi Chen, Haibin Jiang, Zhiwei Zhu, Yuanchan Fan, Yajing Xu, Minghui Sun, Jiamei Liu, Cuiling Xiong, Yanzhen Zheng, Zhongmin Fu, Guojun Xu, Dafu Chen, Rui Guo. 2021
- 蜜蜂球囊菌菌丝和孢子中环状RNA的鉴定及比较分析
- Identification and comparison of circular RNAs in Ascosphaera apis mycelium and spore
- 微生物学报, 61(5): 1299-1314
- Acta Microbiologica Sinica, 61(5): 1299-1314
-
文章历史
- 收稿日期:2020-06-19
- 修回日期:2020-09-02
- 网络出版日期:2021-02-03
2. 福建农林大学蜂疗研究所, 福建 福州 350002;
3. 福建农林大学蜂产品加工与应用教育部工程研究中心, 福建 福州 350002
2. Apitherapy Research Institution, Fujian Agriculture and Forestry University, Fuzhou 350002, Fujian Province, China;
3. Engineering Research Center of Processing and Application of Bee Products of Ministry of Education, Fuzhou 350002, Fujian Province, China
蜜蜂球囊菌(Ascosphera apis,简称球囊菌)侵染蜜蜂幼虫而导致白垩病,严重影响蜂群群势和养蜂生产[1]。该病能引起成年蜜蜂数量、蜂群群势和蜂产品产量的大幅下降[2]。球囊菌孢子随食物被蜜蜂幼虫经口摄入,在中肠肠腔中CO2的刺激下低水平萌发,并生长出少量菌丝,至预蛹期中肠与后肠隔膜消失,孢子随食物残渣进入后肠,在O2的刺激下剧烈萌发,大量生长的菌丝先后穿透幼虫肠壁和体壁,进而包裹整个幼虫,形成白垩状虫尸[2]。2006年公布的球囊菌基因组版本仅有序列信息,缺少功能注释信息[3],导致球囊菌的组学和分子生物学研究举步维艰。2016年公布的球囊菌基因组版本同时包含了完整的序列和功能注释信息[4],为球囊菌的相关研究奠定了基础。
近年来,笔者团队对球囊菌开展了一系列组学研究,例如de novo组装和注释了球囊菌的参考转录组,并大规模地开发了球囊菌的SSR[5];基于高质量转录组数据对球囊菌的已注释基因进行了结构优化,并对未注释基因进行了鉴定和功能注释[6];通过比较转录组分析筛选出球囊菌菌丝和孢子的共有、特有和差异表达的mRNA (differentially expressed mRNA,DEmRNA)、微小RNA (microRNA,miRNA)[7-8]及长链非编码RNA (long noncoding RNA,lncRNA)(未发表数据)并初步解析了它们的潜在功能。环状RNA (circular RNA,circRNA)是一类新近发现的非编码RNA (non-coding RNA,ncRNA),广泛存在于真核生物细胞,已被较多研究证实具有多种重要的生物学功能[9]。共价闭合的环形结构赋予circRNA良好的稳定性,使其耐受RNase R的消化,因而可作为理想的生物标志物[10]。随着高通量测序技术和生物信息学算法的持续革新与迅速发展,人类[11]、蜜蜂[12]、秀丽隐杆线虫(Caenorhabditis elegans)[13]、古细菌[14]等物种的circRNA被陆续鉴定出来。circRNA具有物种保守性、稳定性、表达丰富性、组织和时序表达特异性等特点[15]。circRNA已被证实可通过调控来源基因的转录,充当miRNA的“分子海绵”,以及与RNA结合蛋白质相互作用等方式参与诸多生命活动[16]。近期的研究表明,含有核糖体进入位点(intemal ribosome entry site,IRES)和N6-甲基腺嘌呤(N6-methyladenosine,m6A)甲基化位点的circRNA能够翻译多肽和蛋白质[17-18]。含miRNA反应元件(miRNA response element,MRE)的circRNA可作为竞争性内源RNA (competing endogenous RNA,ceRNA)与其他RNA (mRNA和lncRNA等)竞争性结合miRNA,从而间接调控下游靶mRNA的丰度[12, 19-21]。CHEN等[12]对意大利蜜蜂(Apis mellifera ligustica,简称意蜂) 4种不同状态的蜂王卵巢进行转录组测序和circRNA预测,鉴定到12211个circRNA;作者进一步对DEcircRNA的来源基因进行功能注释,并构建和分析了DEcircRNA的ceRNA调控网络,发现多个DEcircRNA可能与卵巢产卵存在潜在关联性。前期研究中,笔者全基因组鉴定和分析了意蜂中肠的circRNA[22],并对意蜂和中华蜜蜂(Apis cerana cerana,简称中蜂)工蜂中肠发育过程的circRNA差异表达谱、调控网络及潜在作用进行系统解析[23-24]。目前,对于球囊菌菌丝和孢子中circRNA的数量和种类差异,以及circRNA在菌丝生长、孢子萌发和有性生殖中的作用,相关研究仍然缺失。
近期,笔者团队利用基于链特异性cDNA建库的RNA-seq和small RNA-seq (sRNA-seq)技术对球囊菌的纯化菌丝(AaM)和孢子(AaS)样品分别进行测序和分析,系统解析了二者的共有mRNA、特有mRNA和DEmRNA的潜在功能,共有miRNA、特有miRNA和差异表达miRNA (differentially expressed miRNA,DEmiRNA)的调控网络及潜在功能[7-8],共有lncRNA、特有lncRNA和差异表达lncRNA (differentially expressed lncRNA,DElncRNA)的调控方式和潜在作用(未发表数据)。本研究基于前期获得的高质量的全转录组数据[7-8],对球囊菌菌丝和孢子中的circRNA进行鉴定,通过比较分析筛选出菌丝和孢子的共有circRNA、特有circRNA和DEcircRNA并进行来源基因的功能和通路注释,进而构建和分析ceRNA的调控网络,以期解析球囊菌菌丝和孢子中circRNA的数量和种类差异,挖掘菌丝生长、孢子萌发、有性生殖和致病性相关的关键circRNA并探讨其潜在功能。
1 材料和方法 1.1 基于链特异性cDNA建库的RNA-seq数据来源前期研究中,笔者团队已利用基于链特异性cDNA建库的RNA-seq技术对AaM和AaS分别进行深度测序,获得了高质量的测序数据,包含mRNA组学数据[7]、lncRNA组学数据(未发表数据)和circRNA组学数据。其中,高质量的circRNA组学数据可为本研究中AaM和AaS中的circRNA鉴定和分析提供可靠的数据基础。
1.2 sRNA-seq数据来源笔者团队前期已利用sRNA-seq技术分别对AaM和AaS进行测序,获得了高质量的miRNA组学数据[8],可为本研究中的共有circRNA、特有circRNA及DEcircRNA的靶miRNA预测及调控网络构建和分析提供可靠的数据支撑。
1.3 circRNA的鉴定及种类分析参照GUO等[25]的方法对AaM和AaS中的circRNA进行生物信息学预测,方法简述如下:将前期获得的去除了比对上核糖体数据库的有效读段(clean reads),利用TopHat2比对球囊菌的参考基因组(assembly AAP 1.0),去除比对上的clean reads;将剩余的未比对上的clean reads的两端分别截取20 nt,得到短序列读段(anchors reads);将anchors reads比对球囊菌参考基因组(assembly AAP 1.0),利用find_circ软件[26]在比对上的短序列读段中预测circRNA。按照circRNA序列在参考基因组的位置,将预测到的circRNA分为以下类型:已注释的外显子circRNA (annotated and exonic circRNA)、单一外显子circRNA (single exonic circRNA)、外显子和内含子circRNA (exonic and intronic circRNA)、内含子circRNA (intronic circRNA)、反义链circRNA (antisense circRNA)以及基因间区circRNA (intergenic circRNA)。
1.4 共有circRNA、特有circRNA及DEcircRNA的来源基因分析根据RPM (每百万比对上的读段中可比对上反向剪切位点的读段数)算法计算AaM和AaS中circRNA的表达量。利用omicshare平台(http://www.omichsare.com)的Venn工具对AaM和AaS中的circRNA进行Venn分析,筛选出共有circRNA和特有circRNA。利用edgeR软件[27]筛选AaM vs. AaS比较组DEcircRNA,筛选标准为P≤0.05且|log2 fold change|≥1。
circRNA可与RNA聚合酶Ⅱ、U1小核核糖核蛋白及基因启动子相互作用调控来源基因的表达[28-29]。circRNA的来源基因通过使用Bowtie2[30]将上述circRNA两端的anchors reads比对球囊菌参考基因组进行确定。再将来源基因映射到GO数据库和KEGG数据库,分别统计映射上的功能条目和KEGG通路。
1.5 共有circRNA、特有circRNA及DEcircRNA的靶向预测及调控网络分析基于前期获得的AaM和AaS的miRNA组学数据[8],利用TargetFinder软件[31]预测共有circRNA、特有circRNA和DEcircRNA靶向结合的miRNA。按照P≤0.05,自由能≤35的标准进一步从上述预测结果中提取共有circRNA、特有circRNA和DEcircRNA的靶miRNA,获得共有circRNA-miRNA、特有circRNA-miRNA以及DEcircRNA-miRNA的调控关系。
结合前期获得的AaM和AaS的mRNA组学数据[7]预测共有circRNA、特有circRNA和DEcircRNA靶向miRNA的靶mRNA,根据上述靶向结合关系获得共有circRNA-miRNA-mRNA、特有circRNA-miRNA-mRNA以及DEcircRNA- miRNA-mRNA的调控关系。通过Cytoscape软件[32]对各调控网络进行可视化,参数采用默认参数。进一步对共有circRNA、特有circRNA和DEcircRNA的ceRNA调控网络中的靶mRNA进行GO和KEGG数据库注释。
1.6 DEcircRNA的RT-qPCR验证随机选取10个DEcircRNA (novel_circ_000116、novel_circ_000206、novel_circ_002810、novel_circ_ 000018、novel_circ_000079、novel_circ_002976、novel_circ_002726、novel_circ_001780、novel_circ_ 000474和novel_circ_002752)进行RT-qPCR验证。参照Guo等[25]的方法,设计和合成上述DEcircRNA的特异性反向引物(表 1)。利用RNA抽提试剂盒(天漠,中国)分别提取AaM和AaS的总RNA,分为2份,其中1份总RNA经RNase R酶(吉赛,中国)处理后以随机引物进行反转录,获得circRNA的cDNA模板;另一份总RNA直接作为模板,以Oligo (dT)18作为引物进行反转录,获得内参基因actin的cDNA模板。按照SYBR Green Dye (诺唯赞,中国)说明书配制qPCR的反应体系(20 μL)。反应在ABI Q3荧光定量PCR仪(ABI公司,美国)上进行,每个反应进行3次重复。反应条件:94 ℃ 5 min;94℃ 50 s,60℃ 30 s,45个循环。利用2–△△Ct法对上述DEcircRNA的相对表达量进行计算。通过GraphPad Prism 7软件(GraphPad公司,美国)进行相关数据分析及绘图。
| Primer names | Primer sequences (5′→3′) |
| Novel_circ_000116-F | CACTTCGCTGATTTGACG |
| Novel_circ_000116-R | TGATGTGCTGGTGGTTTG |
| Novel_circ_000206-F | GAGACAGCATTTCGCAGAC |
| Novel_circ_000206-R | GGATTGGTGATAAGTTTGGC |
| Novel_circ_002810-F | TCCCTCTTCTATCATCTCTGC |
| Novel_circ_002810-R | CATCATCGTCATCTTTCTGG |
| Novel_circ_000018-F | CGAATACATTGGGAAAGGAG |
| Novel_circ_000018-R | AATCAAACTACTGCCGCC |
| Novel_circ_000079-F | TAAGGGTGTTCCTGTGGGA |
| Novel_circ_000079-R | TGATGTGCTGGTGGTTTGA |
| Novel_circ_002976-F | CCTTGTCCTCAAAACCAGC |
| Novel_circ_002976-R | ACAGCAACGAATAGCGATG |
| Novel_circ_002726-F | AGTTTCCTCTATCTGCGTG |
| Novel_circ_002726-R | GTCAGTGGTCCGACTTCCG |
| Novel_circ_001780-F | CTTCATCGCCCAACTTCTG |
| Novel_circ_001780-R | AAACCTCCACGCCTGCCTA |
| Novel_circ_000474-F | TGCCAAGGGCGTCCACACC |
| Novel_circ_000474-R | CTCGCGCGGGGATAGATCC |
| Novel_circ_002752-F | AAGAAGGTGCGTCGACCGA |
| Novel_circ_002752-R | CCCCACAACAGCGAAATGG |
| actin-F | CAGGAAAGGCTATGTTCGC |
| actin-R | ATTACCGAGGAGCAAGACG |
2 结果和分析 2.1 球囊菌菌丝和孢子中circRNA的预测及分析
AaM和AaS的anchors reads数分别为13210156和19011000条,比对上参考基因组的anchors reads数分别为6124922 (46.37%)条,11392886 (59.93%)。在AaM和AaS中分别鉴定到1868个和2225个circRNA。其中AaM中表达量最高的是novel_circ_000216,其次是novel_circ_ 000217和novel_circ_002913 (表 2);AaS中表达量最高的是novel_circ_000216,其次是novel_circ_ 000217和novel_circ_001553 (表 3)。
| CircRNA ID | RPM values |
| Novel_circ_000216 | 956073.87 |
| Novel_circ_000217 | 10185.83 |
| Novel_circ_002913 | 433.85 |
| Novel_circ_001553 | 276.43 |
| Novel_circ_001554 | 195.81 |
| Novel_circ_000650 | 180.45 |
| Novel_circ_001468 | 180.45 |
| Novel_circ_001884 | 176.61 |
| Novel_circ_000652 | 168.93 |
| Novel_circ_001382 | 145.89 |
| CircRNA ID | RPM values |
| Novel_circ_000216 | 926438.18 |
| Novel_circ_000217 | 9431.94 |
| Novel_circ_001553 | 661.65 |
| Novel_circ_001554 | 428.93 |
| Novel_circ_000682 | 287.48 |
| Novel_circ_000652 | 282.91 |
| Novel_circ_002913 | 278.35 |
| Novel_circ_000986 | 264.66 |
| Novel_circ_000552 | 260.10 |
| Novel_circ_001363 | 246.41 |
Venn分析结果表示,AaM和AaS共有的circNRA为1098个,特有的circRNA分别为770和1127个。在AaM和AaS中,分布在1000–2000 nt范围的circRNA数量最多,分别达到598和690个;其次为分布在2000–3000 nt和0–500 nt的circRNA (图 1)。AaM和AaS中最长的circRNA分别为61627 nt和90611 nt,最短的circRNA分别为90 nt和87 nt。进一步分析发现,对于AaM和AaS中的circRNA,最丰富的环化类型均为基因间区circRNA,其次是外显子和内含子circRNA、单一外显子circRNA (图 2)。

|
| 图 1 球囊菌菌丝和孢子中circRNA的长度分布 Figure 1 Length distribution of circRNAs in A. apis mycelium and spore. |
|
| 图 2 球囊菌菌丝和孢子circRNA环化类型分布 Figure 2 Cyclization type of circRNAs in A. apis mycelium and spore. |
2.2 球囊菌菌丝和孢子中共有circRNA、特有circRNA和DEcircRNA的来源基因的数据库注释
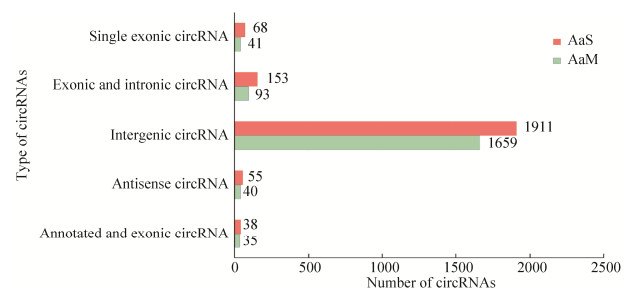
共有circRNA的来源基因可注释到GO数据库的29个功能条目,其中在生物学进程大类中,注释数量最多的条目分别是代谢进程(32个)、细胞进程(30个)及单一组织进程(19个);在分子功能大类中,注释数量最多的为催化活性(17个)、结合(16个)及转运器活性(4个);在细胞组分大类中,注释数量最多的为细胞组分(21个)、细胞(21个)及细胞器(16个)(图 3)。
|
| 图 3 球囊菌菌丝和孢子共有和特有circRNA的来源基因的GO数据库注释 Figure 3 GO database annotation of source genes of common and specific circRNAs in A. apis mycelium and spore. |
AaM的特有circRNA的来源基因可注释到31个功能条目,包括细胞进程、代谢进程和单一组织进程等15个生物学进程相关条目;结合、催化活性和转运活性等7个分子功能相关条目;细胞、细胞组分和细胞器等9个细胞组分相关条目(图 2)。AaS的特有circRNA的来源基因可注释到34个功能条目,包括代谢进程、细胞进程和单一组织进程等16个生物学进程相关条目;结合、催化活性和结构分子活性等6个分子功能相关条目;细胞、细胞组分和细胞器等12个细胞组分相关条目(图 3)。
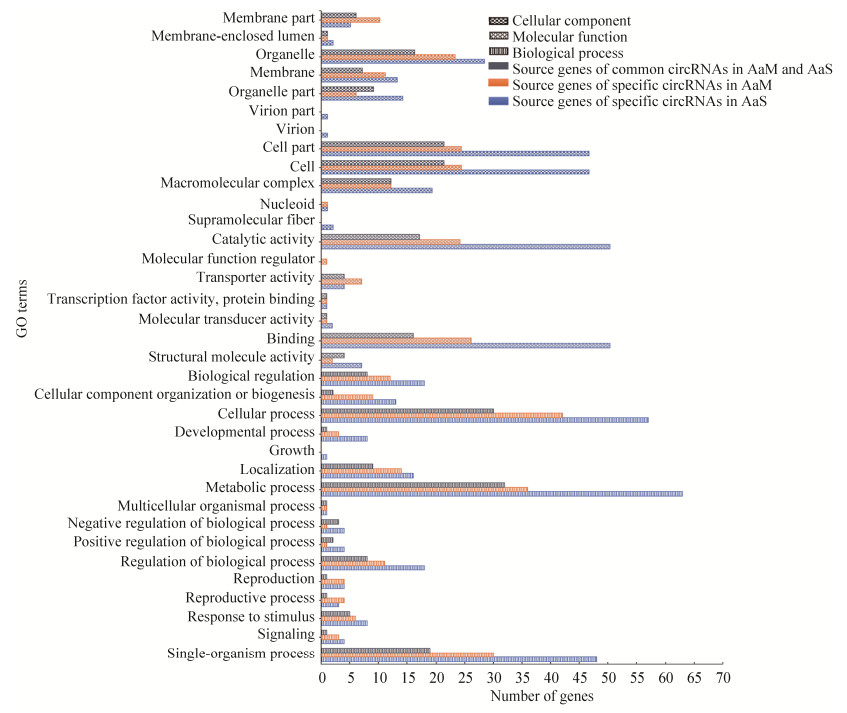
此外,上述共有circRNA的来源基因可注释到KEGG数据库中与代谢、遗传信息处理、环境信息处理和细胞进程相关的14条通路,包括为代谢通路(9条)、次级代谢产物的生物合成(8条)、抗生素的生物合成(6条)、碳代谢(5条)和不同环境中的微生物代谢(5条)等(图 4)。AaM的特有circRNA的来源基因可注释到17类通路,包括代谢通路(17条)、次级代谢产物的生物合成(7条)、内吞作用(4条)、氧化磷酸化(4条)和抗生素的生物合成(4条)(图 4)。AaS的特有circRNA的来源基因可注释到16类通路,包括代谢通路(27条)、次级代谢产物的生物合成(15条)、核糖体(11条)、抗生素的生物合成(11条)和不同环境中的微生物代谢(8条)(图 4)。
|
| 图 4 球囊菌菌丝和孢子共有和特有circRNA的来源基因的KEGG数据库注释 Figure 4 KEGG database annotation of source genes of common and specific circRNAs in A. apis mycelium and spore. |
2.3 DEcircRNA的筛选及来源基因分析
AaM vs. AaS比较组共筛选出553个DEcircRNA,包括456个上调circRNA和97个下调circRNA。这些DEcircRNA的来源基因可注释到29个功能条目,包括细胞进程、代谢进程和单一组织进程等12个生物学进程相关条目;结合、催化活性和结构分子活性等6个分子功能相关条目;细胞、细胞组分和细胞器等11个细胞组分相关条目。此外,还可以注释到代谢通路(11个)、次级代谢产物的生物合成(6个)、核糖体(5个)、内吞作用(4个)和不同环境中的微生物代谢(4个)等40条通路。
2.4 球囊菌菌丝和孢子的共有circRNA、特有circRNA和DEcircRNA的ceRNA调控网络构建及分析223个共有circRNA可靶向22个miRNA进而结合310个靶mRNA;194个AaM的特有circRNA可靶向9个miRNA进而结合264个靶mRNA;282个AaS的特有circRNA可靶向17个miRNA进而结合28个靶mRNA;68个DEcircRNA可靶向3个miRNA进而结合1082个靶mRNA。
对ceRNA调控网络中的靶mRNA进行功能注释,结果显示共有circRNA的靶mRNA可注释细胞进程、代谢进程和催化活性等31个功能条目,以及代谢通路、抗生素的生物合成和次级代谢产物的生物合成等72条通路;AaM的特有circRNA的靶mRNA可注释细胞进程、代谢进程和催化活性等30个功能条目,以及代谢通路、抗生素的生物合成和次级代谢产物的生物合成等61条通路;AaS的特有circRNA的靶mRNA可注释细胞进程、代谢进程和结合等21个功能条目,以及RNA转运、碳代谢和不同环境中的微生物代谢等24条通路;DEcircRNA的靶mRNA可注释细胞进程、代谢进程和催化活性等41个功能条目,以及代谢通路、次级代谢产物生物合成和抗生素的生物合成等106条通路。
进一步分析发现,36个共有circRNA可靶向4个miRNA进而调控6个内吞作用相关的靶mRNA (图 5-A);AaM的4个特有circRNA可靶向2个miRNA进而调控8个次级代谢产物生物合成通路相关的靶mRNA (图 5-B);AaS的255个特有circRNA可靶向2个miRNA进而调控2个次级代谢产物生物合成通路相关的靶mRNA (图 5-C);9个DEcircRNA可靶向2个DEmiRNA进而调控3个MAPK信号通路相关的DEmRNA (图 6)。
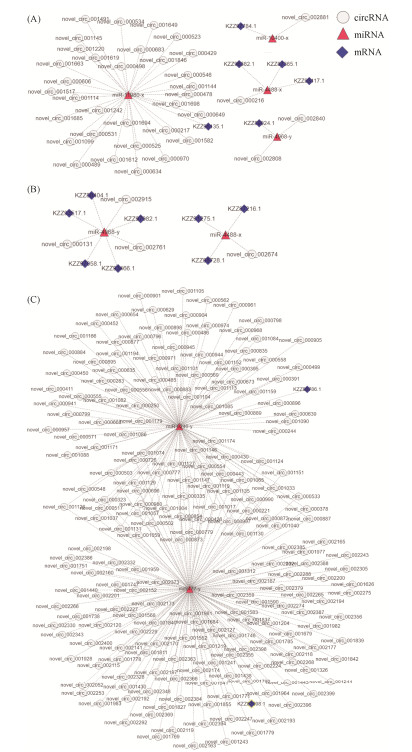
|
| 图 5 球囊菌菌丝和孢子的共有和特有circRNA的ceRNA调控网络 Figure 5 CeRNA regulation networks of common and specific circRNAs in A. apis mycelium and spore. A: ceRNA network of common circRNAs in A. apis mycelium and spore and their target mRNAs relative to endocytosis pathway; B: ceRNA network of specific circRNAs in A. apis mycelium and their target mRNAs relative to biosynthesis of secondary metabolites pathway; C: ceRNA network of specific circRNAs A. apis spore and their target mRNAs relative to biosynthesis of secondary metabolites pathway. |
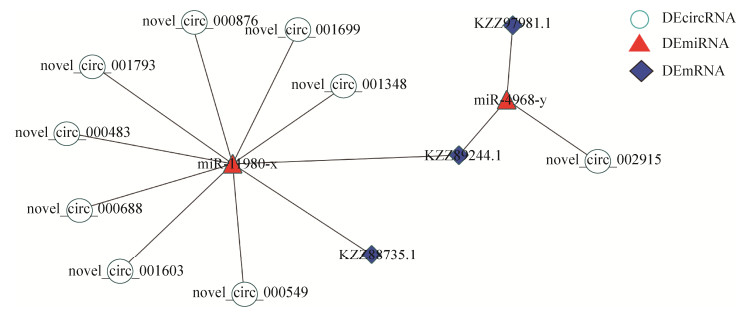
|
| 图 6 球囊菌菌丝和孢子DEcircRNA的ceRNA调控网络 Figure 6 CeRNA regulation network of DEcircRNA between mycelium and spore of A. apis. |
2.5 DEcircRNA的RT-qPCR验证
随机选取10个DEcircRNA进行RT-qPCR验证,结果显示DEcircRNA的表达水平与测序数据中相应的表达水平变化趋势一致(图 7),证明了测序数据的可靠性和DEcircRNA表达趋势的准确性。

|
| 图 7 DEcircRNA的RT-qPCR验证 Figure 7 RT-qPCR validation of DEcircRNAs. *: P < 0.05; **: P < 0.01. |
3 讨论
前期研究中,笔者团队采用链特异性cDNA建库的RNA-seq技术和sRNA-seq技术分别测序了球囊菌菌丝和孢子,同时获得了mRNA[7]、miRNA[8]、lncRNA (未发表数据)和circRNA组学数据。之所以采用链特异性cDNA建库的RNA-seq技术而没有采用去线性RNA的circRNA-seq技术,是因为后者虽然可能检测到更多的低表达circRNA,但因为预先使用RNase R消化去除了绝大多数的线性RNA,导致无法同时计算circRNA与其他RNA的表达量关系[33]。因此,本研究基于已获得的球囊菌菌丝和孢子的全转录组数据筛滤出circRNA组学数据,分别从AaM和AaS中鉴定到1868个和2225个circRNA,它们的长度主要分布在1000–2000 nt,主要环化类型均为基因间区circRNA (88.81%和85.89%)。东方蜜蜂微孢子虫(Nosema ceranae)是养蜂生产中另一种常见的蜜蜂真菌病原,其circRNA的主要环化类型为外显子circRNA (85.29%)和基因间区circRNA (14.71%)[34],表明不同蜜蜂真菌病原的circRNA环化类型存在差异。笔者团队前期研究发现意蜂主要环化类型为已注释外显子circRNA (48.56%)和反义链circRNA (15.83%)[22],而中蜂的主要环化类型均为已注释外显子circRNA (47.45%)和反义链circRNA (18.66%)[24],表明来源于不同物种的circRNA的环化类型具有物种特异性。circRNA的表达具有组织特异性[15, 31]。本研究中,球囊菌菌丝和孢子的共有circRNA为1098个,特有circRNA的数量分别为770和1127个。推测上述共有circRNA可能在球囊菌生活史的不同阶段发挥必要的生物学功能,而特有circRNA在菌丝和孢子中分别发挥不同的调控功能。鉴于球囊菌的circRNA研究目前还十分滞后,本研究的鉴定结果较好地丰富了球囊菌的circRNA信息,也能为其他真菌物种的circRNA研究提供有益的参考信息。
circRNA可与RNA聚合酶Ⅱ、U1小核核糖核蛋白及基因启动子相互作用调控来源基因的表达,从而发挥特定的生物学功能[28-29]。本研究中,AaM和AaS的共有circRNA的来源基因可注释到生物学进程、细胞组分和分子功能相关的29个功能条目,包括代谢进程、细胞进程、催化活性、生殖和生殖进程等;此外还可注释到38条通路,包括赖氨酸降解和半胱氨酸和蛋氨酸的代谢等10条氨基酸代谢相关通路,三羧酸循环和磷酸戊糖途径等5条碳水化合物代谢相关通路,脂肪酸伸长和不饱和脂肪酸的生物合成等2条脂质代谢相关通路,以及1条能量代谢相关通路(氧化磷酸化)。以上结果说明上述共有circRNA可能通过调控相应的来源基因表达广泛调节球囊菌菌丝和孢子的物质和能量代谢。内吞作用是通过质膜的变形运动将胞外物质转运入胞内的过程。真菌菌丝在生长过程中其顶端具有较为活跃的物质合成和分泌过程,而膜泡通过内吞作用将酶类与质膜等关键分子往返运输于菌丝顶端和其他部位[35]。在稻瘟病病原(Magnaporthe oryzae)中,内吞作用相关基因MoARK1和MoPAN1可显著影响细胞壁的完整性、产孢量以及致病性[36]。本研究发现,3个共有circRNA (novel_circ_002895、novel_circ_002896和novel_circ_000146)调控的3个来源基因可注释到内吞作用通路,说明这些circRNA可能通过调控相关来源基因的表达调节菌丝和孢子的内吞作用,进行影响物质代谢、杂交产孢和致病性。
次级代谢产物一般分为抗生素、激素、生物碱和毒素等类型。在侵染宿主时,真菌能够分泌次级代谢物促进自身增殖并杀死宿主[37-38]。卵孢白僵菌(Beauveria tenella)和球孢白僵菌(Beauveria bassiana)合成与分泌的草酸和类草酸晶体等次级代谢物可协同致死宿主[37]。本研究中,球囊菌菌丝的4个特有circRNA调控的来源基因可注释到次级代谢产物生物合成通路。前期研究发现菌丝的4个特有lncRNA可能通过顺式作用调控相关基因表达,从而调节次级代谢产物生物合成通路(未发表数据)。以上结果表明菌丝的特有circRNA和特有lncRNA具有调控次级代谢产物生物合成的潜在功能,可能在侵染和致死宿主的过程中发挥重要作用。此外,真菌的孢子萌发也受次级代谢物的影响[38-39],例如禾谷镰刀菌(Fusarium graminearum)分生孢子和菌落的产生受自身次级代谢物的诱导[39]。本研究发现,孢子的255个特有circRNA调控的来源基因可注释到次级代谢产物通路。前期研究发现孢子的197个特有miRNA调控的288个靶mRNA也注释到了次级代谢产物生物合成通路[8]。上述结果表明孢子的部分特有circRNA和特有miRNA可能通过共同调控次级代谢产物的合成影响孢子萌发,两种不同类型的ncRNA之间或存在一定的关联。
丝裂原活化蛋白激酶(mitogen-activated protein kinase,MAPK)级联反应在真菌的致病性、有性生殖及孢子形成等方面发挥关键调控作用[40-41],其依赖的MAPK激酶可协助病原真菌侵染宿主[42-43]。在前期研究中,我们发现AaS vs AaM比较组中11个DElncRNA通过顺式作用调节9个上下游基因,对MAPK信号通路具有潜在调控性[7]。本研究中,2个DEcircRNA (novel_circ_002736和novel_circ_ 002895)调控的2个来源基因可注释到MAPK信号通路。上述结果表明DEcircRNA和DElncRNA可能通过不同的方式调控基因表达,进而通过调节MAPK信号通路影响球囊菌菌丝和孢子的有性生殖和致病过程。ceRNA机制认为凡含有相同或类似MRE的转录本如circRNA和lncRNA等均可以通过竞争结合miRNA间接调控基因表达[44-47]。ZHANG等[47]证实胶质母细胞中的circRNAMOT1能够作为“分子海绵”吸附结合miR-92,继而上调WWOX信号通路相关基因的表达,进而抑制胶质细胞瘤细胞的增殖。本研究中,ceRNA调控网络分析结果显示223个共有cirRNA、194个菌丝特有circRNA、282个孢子特有circRNA和68个DEcircRNA分别靶向结合22、9、17和3个miRNA,进而调控310、264、28和1082个mRNA。进一步分析发现,菌丝和孢子共有的36个circRNA靶向4个miRNA,进而调控内吞作用相关的6个靶mRNA;菌丝的4个特有circRNA靶向2个miRNA,进而调控次级代谢产物生物合成相关的8个靶mRNA;孢子的255个特有circRNA靶向2个miRNA,进而调控次级代谢产物生物合成通路相关的2个靶mRNA;此外,9个DEcircRNA靶向结合了2个DEmiRNA,进而调控MAPK信号通路相关的3个DEmRNA。以上结果表明球囊菌的circRNA可能通过ceRNA机制调控上述靶mRNA相关的基因表达,进而通过调节内吞作用、次级代谢产物生物合成和MAPK信号通路参与球囊菌的有性生殖和致病过程。但需要强调的是,本研究涉及的球囊菌菌丝和孢子均来源于实验室条件下获得的纯培养,不同于处于侵染过程的球囊菌,因而上述分析结果更多体现的是部分共有circRNA、特有circRNA和DEcircRNA与球囊菌致病性的潜在关联。目前,笔者团队已制备球囊菌侵染的意蜂幼虫肠道和中蜂幼虫肠道样品,下一步将利用链特异性cDNA建库的RNA-seq技术对上述肠道样品进行测序,通过连续比对核糖体数据库、蜜蜂宿主基因组和球囊菌基因组筛选得到病原数据,再结合球囊菌孢子的测序数据进行比较分析,处于侵染过程的球囊菌的DEcircRNA与病原致病性的关系将更为直接和真实。
根癌农杆菌介导(AtMT)的真菌遗传转化体系为真菌基因的分离克隆和功能研究提供了技术平台,目前已经广泛应用于昆虫病原真菌[48-50]的基因功能研究。下一步的工作重点是构建根癌农杆菌介导的球囊菌遗传转化技术体系,并对内吞作用、次级代谢产物生物合成和MAPK信号通路等相关的关键circRNA进行初步的功能研究。
4 结论本研究通过全面分析球囊菌菌丝和孢子的共有circRNA、特有circRNA和DEcircRNA,解析了菌丝和孢子中circRNA的数量和种类差异,揭示了部分circRNA可能通过调控来源基因和充当ceRNA的方式调节内吞作用、次级代谢产物生物合成和MAPK信号通路及相关基因表达,进而影响球囊菌的物质和能量代谢、杂交产孢和致病性。
| [1] | Calderón RA, Rivera G, Sánchez A, Zamora LG. Chalkbrood (Ascosphaera apis) and some other fungi associated with Africanized honey bees (Apis mellifera) in Costa Rica. Journal of Apicultural Research, 2004, 43(4): 187-188. DOI:10.1080/00218839.2004.11101135 |
| [2] | Aronstein KA, Murray KD. Chalkbrood disease in honey bees. Journal of Invertebrate Pathology, 2010, 103(Suppl 1): S20-S29. |
| [3] | Qin X, Evans JD, Aronstein KA, Murray KD, Weinstock GM. Genome sequences of the honey bee pathogens Paenibacillus larvae and Ascosphaera apis. Insect Molecular Biology, 2006, 15(5): 715-718. DOI:10.1111/j.1365-2583.2006.00694.x |
| [4] | Shang YF, Xiao GH, Zheng P, Cen K, Zhan S, Wang CS. Divergent and convergent evolution of fungal pathogenicity. Genome Biology and Evolution, 2016, 8(5): 1374-1387. DOI:10.1093/gbe/evw082 |
| [5] |
Zhang ZN, Xiong CL, Xu XJ, Huang ZJ, Zheng YZ, Luo Q, Liu M, Li WD, Tong XY, Zhang Q, Liang Q, Guo R, Chen DF. De novo assembly of a reference transcriptome and development of SSR markers for Ascosphaera apis. Acta Entomologica Sinica, 2017, 60(1): 34-44.
(in Chinese) 张曌楠, 熊翠玲, 徐细建, 黄枳腱, 郑燕珍, 骆群, 刘敏, 李汶东, 童新宇, 张琦, 梁勤, 郭睿, 陈大福. 蜜蜂球囊菌的参考转录组de novo组装及SSR分子标记开发. 昆虫学报, 2017, 60(1): 34-44. |
| [6] |
Guo R, Chen HZ, Tong XY, Xiong CL, Zheng YZ, Fu ZM, Xie YL, Wang HP, Zhao HX, Chen DF. Structural optimization of annotated genes and identification of novel genes in Ascosphaera apis. Journal of China Agricultural University, 2019, 24(1): 61-68.
(in Chinese) 郭睿, 陈华枝, 童新宇, 熊翠玲, 郑燕珍, 付中民, 解彦玲, 王海朋, 赵红霞, 陈大福. 蜜蜂球囊菌基因结构优化及新基因鉴定. 中国农业大学学报, 2019, 24(1): 61-68. |
| [7] |
Jiang HB, Du Y, Fan XX, Wang J, Zhu ZW, Fan YC, Xiong CL, Fu ZM, Xu GJ, Chen DF, Guo R. Comparative transcriptome analysis of Ascosphaera apis mycelium and spore. Journal of Sichuan University (Natural Science Edition), 2020, 57(6): 1178-1185.
(in Chinese) 蒋海宾, 杜宇, 范小雪, 王杰, 祝智威, 范元婵, 熊翠玲, 付中民, 徐国钧, 陈大福, 郭睿. 蜜蜂球囊菌菌丝和孢子的比较转录组分析. 四川大学学报(自然科学版), 2020, 57(6): 1178-1185. |
| [8] |
Chen HZ, Zhu ZW, Jiang HB, Wang J, Fan YC, Fan XX, Wan JQ, Lu JX, Xiong CL, Zheng YZ, Fu ZM, Chen DF, Guo R. Comparative analysis of microRNAs and corresponding target mRNAs in Ascosphaera apis mycelium and spore. Scientia Agricultura Sinica, 2020, 53(17): 3606-3619.
(in Chinese) 陈华枝, 祝智威, 蒋海宾, 王杰, 范元婵, 范小雪, 万洁琦, 卢家轩, 熊翠玲, 郑燕珍, 付中民, 陈大福, 郭睿. 蜜蜂球囊菌菌丝和孢子中微小RNA及其靶mRNA的比较分析. 中国农业科学, 2020, 53(17): 3606-3619. DOI:10.3864/j.issn.0578-1752.2020.17.017 |
| [9] | Qu SB, Zhong Y, Shang RZ, Zhang X, Song WJ, Kjems J, Li HM. The emerging landscape of circular RNA in life processes. RNA Biology, 2017, 14(8): 992-999. DOI:10.1080/15476286.2016.1220473 |
| [10] | Perkel JM. Assume nothing: the tale of circular RNA. Biotechniques, 2013, 55(2): 55-57. |
| [11] | Jeck WR, Sorrentino JA, Wang K, Slevin MK, Burd CE, Liu JZ, Marzluff WF, Sharpless NE. Circular RNAs are abundant, conserved, and associated with ALU repeats. RNA, 2013, 19(2): 141-157. DOI:10.1261/rna.035667.112 |
| [12] | Chen X, Shi W, Chen C. Differential circular RNAs expression in ovary during oviposition in honey bees. Genomics, 2019, 111(4): 598-606. DOI:10.1016/j.ygeno.2018.03.015 |
| [13] | Liu JW, Chen LL. Analysis of circular RNA in Caenorhabditis elegans. Hans Journal of Computational Biology, 2015, 5(2): 17-28. DOI:10.12677/HJCB.2015.52003 |
| [14] | Danan M, Schwartz S, Edelheit S, Sorek R. Transcriptome-wide discovery of circular RNAs in Archaea. Nucleic Acids Research, 2012, 40(7): 3131-3142. DOI:10.1093/nar/gkr1009 |
| [15] | He J, Xie QC, Xu HL, Li JT, Li YS. Circular RNAs and cancer. Cancer Letters, 2017, 396: 138-144. DOI:10.1016/j.canlet.2017.03.027 |
| [16] | Chen LL. The expanding regulatory mechanisms and cellular functions of circular RNAs. Nature Reviews Molecular Cell Biology, 2020, 21(8): 475-490. DOI:10.1038/s41580-020-0243-y |
| [17] | Yang Y, Fan XJ, Mao MW, Song XW, Wu P, Zhang Y, Jin YF, Yang Y, Chen LL, Wang Y, Wong CC, Xiao XS, Wang ZF. Extensive translation of circular RNAs driven by N6-methyladenosine. Cell Research, 2017, 27(5): 626-641. DOI:10.1038/cr.2017.31 |
| [18] | Pamudurti NR, Bartok O, Jens M, Ashwal-Fluss R, Stottmeister C, Ruhe L, Hanan M, Wyler E, Perez-Hernandez D, Ramberger E, Shenzis S, Samson M, Dittmar G, Landthaler M, Chekulaeva M, Rajewsky N, Kadener S. Translation of circRNAs. Molecular Cell, 2017, 66(1): 9-21. DOI:10.1016/j.molcel.2017.02.021 |
| [19] | Qu SB, Yang XS, Li XL, Wang JL, Gao Y, Shang RZ, Sun W, Dou KF, Li HM. Circular RNA: a new star of noncoding RNAs. Cancer Letters, 2015, 365(2): 141-148. DOI:10.1016/j.canlet.2015.06.003 |
| [20] | Yang J, Li Y, Yu ZC, Zhou YF, Tu JF, Lou J, Wang YH. Circular RNA circ100084 functions as sponge of miR-23a-5p to regulate IGF2 expression in hepatocellular carcinoma. Molecular Medicine Reports, 2020, 21(6): 2395-2404. |
| [21] | Chen DF, Guo R, Xu XJ, Xiong CL, Liang Q, Zheng YZ, Luo Q, Zhang ZN, Huang ZJ, Kumar D, Xi WJ, Zou X, Liu M. Uncovering the immune responses of Apis mellifera ligustica larval gut to Ascosphaera apis infection utilizing transcriptome sequencing. Gene, 2017, 621: 40-50. DOI:10.1016/j.gene.2017.04.022 |
| [22] |
Xiong CL, Chen HZ, Chen DF, Zheng YZ, Fu ZM, Xu GJ, Du Y, Wang HP, Geng SH, Zhou DD, Liu SY, Guo R. Analysis of circular RNAs and their regulatory networks in the midgut of Apis mellifera ligustica workers. Acta Entomologica Sinica, 2018, 61(12): 1363-1375.
(in Chinese) 熊翠玲, 陈华枝, 陈大福, 郑燕珍, 付中民, 徐国钧, 杜宇, 王海朋, 耿四海, 周丁丁, 刘思亚, 郭睿. 意大利蜜蜂工蜂中肠的环状RNA及其调控网络分析. 昆虫学报, 2018, 61(12): 1363-1375. |
| [23] |
Guo R, Chen HZ, Xiong CL, Zheng YZ, Fu ZM, Xu GJ, Du Y, Wang HP, Geng SH, Zhou DD, Liu SY, Chen DF. Analysis of differentially expressed circular RNAs and their regulation networks during the developmental process of Apis mellifera ligustica worker's midgut. Scientia Agricultura Sinica, 2018, 51(23): 4575-4590.
(in Chinese) 郭睿, 陈华枝, 熊翠玲, 郑燕珍, 付中民, 徐国钧, 杜宇, 王海朋, 耿四海, 周丁丁, 刘思亚, 陈大福. 意大利蜜蜂工蜂中肠发育过程中的差异表达环状RNA及其调控网络分析. 中国农业科学, 2018, 51(23): 4575-4590. DOI:10.3864/j.issn.0578-1752.2018.23.015 |
| [24] | Chen DF, Chen HZ, Du Y, Zhu ZW, Wang J, Geng SH, Xiong CL, Zheng YZ, Hou CS, Diao QY, Guo R. Systematic identification of circular RNAs and corresponding regulatory networks unveil their potential roles in the midguts of eastern honeybee workers. Applied Microbiology and Biotechnology, 2020, 104(1): 257-276. DOI:10.1007/s00253-019-10159-9 |
| [25] | Guo R, Chen DF, Chen HZ, Fu ZM, Xiong CL, Hou CS, Zheng YZ, Guo YL, Wang HP, Du Y, Diao QY. Systematic investigation of circular RNAs in Ascosphaera apis, a fungal pathogen of honeybee larvae. Gene, 2018, 678: 17-22. DOI:10.1016/j.gene.2018.07.076 |
| [26] | Memczak S, Jens M, Elefsinioti A, Torti F, Krueger J, Rybak A, Maier L, Mackowiak SD, Gregersen LH, Munschauer M, Loewer A, Ziebold U, Landthaler M, Kocks C, Le Noble F, Rajewsky N. Circular RNAs are a large class of animal RNAs with regulatory potency. Nature, 2013, 495(7441): 333-338. DOI:10.1038/nature11928 |
| [27] | Robinson MD, McCarthy DJ, Smyth GK. edgeR: a bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics, 2010, 26(1): 139-140. DOI:10.1093/bioinformatics/btp616 |
| [28] | Zhang Y, Zhang XO, Chen T, Xiang JF, Yin QF, Xing YH, Zhu SS, Yang L, Chen LL. Circular intronic long noncoding RNAs. Molecular Cell, 2013, 51(6): 792-806. DOI:10.1016/j.molcel.2013.08.017 |
| [29] | Li ZY, Huang C, Bao C, Chen L, Lin M, Wang XL, Zhong GL, Yu B, Hu WC, Dai LM, Zhu PF, Chang ZX, Wu QF, Zhao Y, Jia Y, Xu P, Liu HJ, Shan G. Exon-intron circular RNAs regulate transcription in the nucleus. Nature Structural & Molecular Biology, 2015, 22(3): 256-264. |
| [30] | Langdon WB. Performance of genetic programming optimised Bowtie2 on genome comparison and analytic testing (GCAT) benchmarks. BioData Mining, 2015, 8(1): 1. DOI:10.1186/s13040-014-0034-0 |
| [31] | Allen E, Xie ZX, Gustafson AM, Carrington JC. microRNA-directed phasing during Trans-acting siRNA biogenesis in plants. Cell, 2005, 121(2): 207-221. DOI:10.1016/j.cell.2005.04.004 |
| [32] | Smoot ME, Ono K, Ruscheinski J, Wang PL, Ideker T. Cytoscape 2.8: new features for data integration and network visualization. Bioinformatics, 2011, 27(3): 431-432. DOI:10.1093/bioinformatics/btq675 |
| [33] | Hu XL, Zhu M, Zhang X, Liu B, Liang Z, Huang LX, Xu J, Yu L, Li K, Zar MS, Xue RY, Cao GL, Gong CL. Identification and characterization of circular RNAs in the silkworm midgut following Bombyx mori cytoplasmic polyhedrosis virus infection. RNA Biology, 2018, 15(2): 292-301. DOI:10.1080/15476286.2017.1411461 |
| [34] | Guo R, Chen DF, Chen HZ, Xiong CL, Zheng YZ, Hou CS, Du Y, Geng SH, Wang HP, Zhou DD, Guo YL. Genome-wide identification of circular RNAs in fungal parasite Nosema ceranae. Current Microbiology, 2018, 75(12): 1655-1660. DOI:10.1007/s00284-018-1576-z |
| [35] | Doherty GJ, McMahon HT. Mechanisms of endocytosis. Annual Review of Biochemistry, 2009, 78: 857-902. DOI:10.1146/annurev.biochem.78.081307.110540 |
| [36] |
Wang JM. Characterization of two endocytosis MoARK1 and MoPAN1 in development and pathogenicity of Magnaporthe oryza. Master Dissertation of Nanjing Agricultural University, 2012. (in Chinese) 王佳妹. 细胞内吞作用基因MoARKl和MoPANl在稻瘟病菌生长发育及致病过程中的功能研究. 南京农业大学硕士学位论文, 2012. |
| [37] |
Huang JM, Zhang PZ, Wang F, Li Y, Feng SJ, Yang M. Advanced studies on metabolites and pharmacological of Beauveria bassiana. Chinese Journal of Biochemical Pharmaceutics, 2014, 34(9): 167-173.
(in Chinese) 黄居敏, 张普照, 王芳, 李旸, 冯少俊, 杨明. 白僵菌的代谢产物及药理活性研究进展. 中国生化药物杂志, 2014, 34(9): 167-173. |
| [38] | Calvo AM, Wilson RA, Bok JW, Keller NP. Relationship between secondary metabolism and fungal development. Microbiology and Molecular Biology Reviews, 2002, 66(3): 447-459. DOI:10.1128/MMBR.66.3.447-459.2002 |
| [39] | Wolf JC, Mirocha CJ. Regulation of sexual reproduction in Gibberella zeae (Fusarium roseum 'Graminearum') by F-2 (Zearalenone). Canadian Journal of Microbiology, 1973, 19(6): 725-734. DOI:10.1139/m73-117 |
| [40] | Zhao XH, Mehrabi R, Xu JR. Mitogen-activated protein kinase pathways and fungal pathogenesis. Eukaryotic Cell, 2007, 6(10): 1701-1714. DOI:10.1128/EC.00216-07 |
| [41] | Igbaria A, Lev S, Rose MS, Lee BN, Hadar R, Degani O, Horwitz BA. Distinct and combined roles of the MAP kinases of Cochliobolus heterostrophus in virulence and stress responses. Molecular Plant-Microbe Interactions, 2008, 21(6): 769-780. DOI:10.1094/MPMI-21-6-0769 |
| [42] | Chen XX, Xu C, Qian Y, Liu R, Zhang QQ, Zeng GH, Zhang X, Zhao H, Fang WG. MAPK cascade-mediated regulation of pathogenicity, conidiation and tolerance to abiotic stresses in the entomopathogenic fungus Metarhizium robertsii. Environmental Microbiology, 2016, 18(3): 1048-1062. DOI:10.1111/1462-2920.13198 |
| [43] | Jiang C, Zhang X, Liu HQ, Xu JR. Mitogen-activated protein kinase signaling in plant pathogenic fungi. PLoS Pathogens, 2018, 14(3): e1006875. DOI:10.1371/journal.ppat.1006875 |
| [44] | Salmena L, Poliseno L, Tay Y, Kats L, Pandolfi PP. A ceRNA hypothesis: the Rosetta stone of a hidden RNA language?. Cell, 2011, 146(3): 353-358. DOI:10.1016/j.cell.2011.07.014 |
| [45] | Deng YY, Zhang WP, She JQ, Zhang LS, Chen T, Zhou J, Yuan ZY. GW27-e1167 circular RNA related to PPARγ function as ceRNA of microRNA in human acute myocardial infarction. Journal of the American College of Cardiology, 2016, 68(16S): C51-C52. |
| [46] | Pan RY, Liu P, Zhou HT, Sun WX, Song J, Shu J, Cui GJ, Yang ZJ, Jia EZ. Circular RNAs promote TRPM3 expression by inhibiting hsa-miR-130a-3p in coronary artery disease patients. Oncotarget, 2017, 8(36): 60280-60290. DOI:10.18632/oncotarget.19941 |
| [47] | Zhang XW, Zhong B, Zhang WJ, Wu J, Wang Y. Circular RNA CircMTO1 inhibits proliferation of glioblastoma cells via miR-92/WWOX signaling pathway. Medical Science Monitor: International Medical Journal of Experimental and Clinical Research, 2019, 25: 6454-6461. |
| [48] | Fang WG, Pei Y, Bidochka MJ. Transformation of Metarhizium anisopliae mediated by Agrobacterium tumefaciens. Canadian Journal of Microbiology, 2006, 52(7): 623-626. DOI:10.1139/w06-014 |
| [49] | Leclerque A, Wan H, Abschütz A, Chen SW, Mitina GV, Zimmermann G, Schairer HU. Agrobacterium-mediated insertional mutagenesis (AIM) of the entomopathogenic fungus Beauveria bassiana. Current Genetics, 2004, 45(2): 111-119. DOI:10.1007/s00294-003-0468-2 |
| [50] | Zhang YJ, Zhao JJ, Xie M, Peng DL. Agrobacterium tumefaciens-mediated transformation in the entomopathogenic fungus Lecanicillium lecanii and development of benzimidazole fungicide resistant strains. Journal of Microbiological Methods, 2014, 105: 168-173. DOI:10.1016/j.mimet.2014.07.033 |
 2021, Vol. 61
2021, Vol. 61