中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 何荣荣, 彭婧, 孙悦. 2021
- Rongrong He, Jing Peng, Yue Sun. 2021
- 接种发酵和自然发酵中酿酒酵母菌株多样性比较
- Yeasts diversity of inoculated and spontaneous fermentations
- 微生物学报, 61(5): 1211-1221
- Acta Microbiologica Sinica, 61(5): 1211-1221
-
文章历史
- 收稿日期:2020-05-10
- 修回日期:2020-07-01
- 网络出版日期:2020-12-14
2. 中国农业大学食品科学与营养工程学院, 北京 100089
2. College of Food Science and Nutrition Engineering, China Agricultural University, Beijing 100089, China
葡萄酒的酿造过程主要涉及多酵母属、种及菌株等微生物的参与,以酿酒酵母(Saccharomyces cerevisiae)为主的酒精发酵是葡萄酒生产的重要阶段。根据葡萄酒酒精发酵过程中是否接种酵母菌,可将其分为接种发酵和非接种发酵(即自然发酵)。自然发酵完全依靠葡萄表面的天然菌群和酿酒设备上的微生物来完成整个发酵过程;而接种发酵是由所接种的商业酵母和本土酵母共同完成的。纯培养技术的建立促使葡萄酒的发酵方式由自然发酵向接种发酵转变。
自然发酵生产的葡萄酒通常具有典型的地域风格,但由于不确定因素的影响,会导致葡萄酒质量的不稳定。接种发酵使酒精发酵过程得到有效控制,并实现产品质量的均一化。但这一过程会造成葡萄酒的同质化,从而削弱葡萄酒的整体风味特性。传统观点认为,接种发酵过程中,所接种的商业活性干酵母是相应发酵中的优势菌株。但前人研究发现,接种的酿酒酵母在发酵过程中并不一定占据主导地位,野生酿酒酵母表现出较强的竞争力,处于优势地位、主导完成酒精发酵[1-2]。在接种发酵过程中,酿酒酵母的定殖能力受菌株特性、接种方式(复水活化或干粉直投)、接种温度、发酵温度和接种量等因素的影响[2-8]。不同酿酒酵母菌株的代谢产物存在差异,发酵过程中酿酒酵母菌株多样性组成影响葡萄酒的外观及品质。因此,借助DNA分子标记法对酿酒酵母菌株进行区分至关重要,从而实现对发酵过程的有效监控。Interdelta指纹图谱分析技术具有容易操作、多态性丰富等优点,因此在酿酒酵母菌株的区分和遗传多样性研究,开发特定地区酿酒酵母菌株资源中发挥重要作用[9-10]。
因此,本研究利用Interdelta指纹图谱技术对宁夏银川产区霞多丽葡萄自然发酵和接种不同酿酒酵母菌株NXU17-26、UCD522和UCD2610发酵过程中的酿酒酵母分离株进行分类,比较自然发酵和3个接种发酵中酿酒酵母菌株的遗传多样性。利用UPGMA聚类分析构建相关酿酒酵母菌株的系统发育树,通过计算不同接种发酵过程中酿酒酵母的多样性指数对分离的酿酒酵母多样性进行比较。
1 材料和方法 1.1 材料、试剂及仪器 1.1.1 葡萄材料:霞多丽葡萄(宁夏银川产区):糖210.00 g/L,总酸6.65 g/L,pH 3.99。
1.1.2 酿酒酵母:NXU17-26 (中性菌株,宁夏大学食品与葡萄酒学院葡萄酒微生物资源与遗传育种实验室保藏),UCD522 (敏感菌株,美国加州大学戴维斯分校葡萄栽培与葡萄酒酿造系菌种保藏中心),UCD2610 (嗜杀菌株,美国加州大学戴维斯分校葡萄栽培与葡萄酒酿造系菌种保藏中心),UCD522和UCD2610两株酵母菌株由美国加州大学戴维斯分校葡萄栽培与葡萄酒酿造系菌种保藏中心赠予。
1.1.3 引物:26S rDNA D1/D2区序列引物:使用优化的引物NL1 (5′-GCATATCAATAAGCGG AGGAAAAG-3′)和NL4 (5′-GGTCCGTGTTTCA AGACGG-3′)。
Interdelta扩增引物:使用优化的delta引物delta12 (5′-TCAACAATGGAATCCCAAC-3′)和delta21 (5′-CATCTTAACACCGTATATGA-3′)。
1.1.4 试剂:葡萄糖、蛋白胨、酵母浸粉、琼脂等生物试剂,引物(由生工生物公司合成)、dNTPs (北京全式金生物公司)、Taq酶(北京全式金生物公司)。
1.1.5 仪器设备:超净工作台SW-CJ-2FD (苏州安泰空气技术有限公司);生化培养箱SHP-150B型(常州诺基仪器有限公司);干式恒温器GDH-100 (上海巴玖实业有限公司);JNX-26高速冷冻离心机(贝克曼库尔特股份有限公司);PCR仪844-0069 (德国耶拿分析仪器股份公司);全自动凝胶成像仪ChampGel 15000 (北京赛智创业科技有限公司);电泳仪DYY-6C型(北京六一生物科技有限公司)。
1.1.6 培养基:YEPD培养基(yeast extract peptone dextrose medium):酵母浸粉10 g/L (1%),蛋白胨20 g/L (2%),葡萄糖20 g/L (2%),蒸馏水。121 ℃灭菌20 min,添加100 mg/L的氯霉素。配制固体培养基时加入20 g/L (2%)的琼脂。
WL营养琼脂培养基(wallerstein laboratory nutrient agar medium):用于酵母菌株的初步形态分类[11],调pH至6.5,121 ℃灭菌20 min,添加100 mg/L的氯霉素。
1.2 葡萄汁发酵本试验设置了4种发酵处理,分别是自然发酵、接种中性菌株NXU17-26、敏感菌株UCD522和嗜杀菌株UCD2610的发酵。用无菌自封袋将成熟的葡萄原料在无菌状态下进行除梗破碎,发酵体积为700 mL,每个处理设置2个发酵重复[12-13]。对于接种发酵而言,在葡萄破碎后的第2天进行接种,接种后的细胞浓度为106 CFU/mL。所有的发酵均于25–28 ℃控温发酵,采用CO2失重法对发酵进程进行监控,连续两天失重变化小于0.1 g时视为发酵结束。
1.3 酵母菌分离在发酵的第2,4,6天[2, 14]取样并进行梯度稀释,选取合适的稀释浓度涂布于WL培养基上(加入氯霉素100 mg/L,抑制细菌和霉菌的生长),28 ℃培养3 d,根据菌落的颜色和形态不同,每个时期挑选20个酵母单菌落进行划线纯化[15-17],经纯化后的酵母菌于20%的无菌甘油中于–20 ℃下保藏备用。
1.4 DNA提取DNA的提取采用石英砂破壁法[18]。
1.5 酵母菌26S rDNA D1/D2区的扩增[12]引物NL1和NL4各0.5 μL,10×Easy Tap Buffer 2.5 μL,2.5 mmol/L dNTPs 2 μL,10×Easy Tap Buffer Polymerase 0.25 μL,酵母菌DNA模板1 μL,加ddH2O至25 μL。PCR扩增条件:95 ℃ 5 min;94 ℃ 1 min,52 ℃ 1 min,72 ℃ 80 s,36个循环;72 ℃ 8 min。PCR产物经2%琼脂糖凝胶电泳进行检验,电压110 V,电流90 mA,时间约30 min,然后将PCR产物送生工生物工程(上海)股份有限公司检测。
1.6 酿酒酵母的Interdelta指纹图谱分析[19]引物delta12和delta21各0.5 μL,10×HiFi bufferⅠ 2.5 μL,2.5 mmol/L dNTPs 2 μL,10×HiFi DNA Polymerase 0.25 μL,酵母菌DNA模板1 μL,加ddH2O至25 μL。PCR扩增条件:94 ℃ 2 min;94 ℃ 30 s,56 ℃ 30 s,72 ℃ 90 s,35个循环;72 ℃ 10 min。PCR产物经2%琼脂糖凝胶电泳进行检验,电压100 V,电流90 mA,时间约40 min,用凝胶成像仪观察结果。
对Interdelta PCR电泳图谱进行统计,根据特定位置上有、无DNA条带分别记为“1”和“0”进行统计区分,建立数据矩阵,采用NTsys 2.10e版软件进行非加权算术平均数法(unweighted pair group method with arithmetic mean,UPGMA)构建相关酿酒酵母菌株的系统发育树[20]。
1.7 数据处理使用Office 2007和SPSS Statistics V19.0 (IBM Inc.,Chicago,IL,USA)对所得的数据进行均值和方差分析,采用Ducan检验方法(P < 0.05),绘图软件使用Origin 8.1。
2 结果和分析 2.1 发酵曲线利用CO2失重法监控发酵进程,图 1为不同发酵处理的发酵曲线(CO2每天释放量表示)。从发酵曲线可以看出,接种UCD522和接种UCD2610的发酵曲线相类似,在发酵的第3天和4天时发酵速度最快,接种NXU17-26的发酵速度次之;自然发酵的发酵曲线较为平缓,在发酵的第5天和6天发酵速度较快。
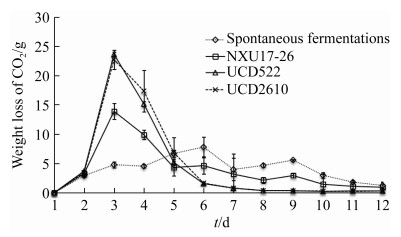
|
| 图 1 接种不同酿酒酵母菌株发酵的CO2失重曲线 Figure 1 Comparison of CO2 weight loss curves of different fermentation. The standard deviation reflects the dispersion of CO2 weight loss per day (until the end of fermentation) in different fermentation treatments. |
2.2 酵母菌种属动态变化
经WL培养基初步分离,自然发酵中共分离到112个酵母单菌落,接种NX11424发酵中分离到114个酵母单菌落,接种UCD522发酵中分离到117个酵母单菌落,接种UCD2610发酵中分离到113个酵母单菌落。利用26S rDNA D1/D2区序列分析,按序列相似性在99%–100%原则进行酵母菌种鉴定。本研究对4个发酵中分离到的456个酵母菌落进行了鉴定,经鉴定共有6属11种,即矮小假丝酵母(Candida humili)、萄葡汁有孢汉逊酵母(Hanseniaspora uvarum)、H. guilliermondii、H. meyeri、Kazachstania hellenica、美极梅奇酵母(Metschnikowia pulcherrima)、Metschnikowia aff、Metschnikowia sp.、库徳毕赤酵母(Pichia kudriavzevii)、克鲁维毕赤酵母(P. kluyveri)和酿酒酵母(S. cerevisiae)。其中,自然发酵中分离的酵母有5属6种,即C. humili、K. hellenica、H. uvarum、Metschnikowia aff、P. kudriavzevii和P. kluyveri,未分离到酿酒酵母;接种NXU17-26的发酵中分离到3种酵母P. kluyveri、Metschnikowia sp.和酿酒酵母;接种UCD522的发酵分离到3种酵母H. uvarum、M. pulcherrima和酿酒酵母;接种UCD2610的发酵分离到3种酵母H. guilliermondii、M. pulcherrima和酿酒酵母。
不同发酵处理的酵母菌种、属组成存在明显差异,自然发酵中非酿酒酵母的比例远高于其他发酵(表 1)。如表所示,随着发酵的进行各接种发酵中非酿酒酵母的比例逐渐下降,酿酒酵母逐渐占据主导地位并完成酒精发酵。在发酵中后期,接种UCD2610的发酵中,酿酒酵母的数量高于其他发酵。
| Yeast species | 2 d | 4 d | 6 d | |||||||||||
| A | B | C | D | A | B | C | D | A | B | C | D | |||
| Pichia | 7.42% | 10.91% | 0 | 9.26% | 50.00% | 0 | 0 | 0 | 12.20% | 0 | 0 | 0 | ||
| Metschnikowia | 7.42% | 3.62% | 3.12% | 3.07% | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | ||
| Hanseniaspora | 85.16% | 84.38% | 90.86% | 80.26% | 50.00% | 13.64% | 8.71% | 0 | 14.83% | 0 | 0 | 0 | ||
| Candida | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 27.84% | 0 | 0 | 0 | ||
| Kazachstania | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 45.13% | 0 | 0 | 0 | ||
| S. cerevisiae | 0 | 1.09% | 6.02% | 7.41% | 0 | 86.36% | 91.29% | 100.00% | 0 | 100.00% | 100.00% | 100.00% | ||
| A: spontaneous fermentation; B: inoculated with NXU17-26; C: inoculated with UCD522; D: inoculated with UCD2610. | ||||||||||||||
2.3 酿酒酵母菌株基因型多样性分析
Interdelta指纹图谱能实现不同菌株的区分。本研究利用Interdelta指纹图谱技术对酿酒酵母的基因型进行分析,从而计算出不同基因型所占的比例,进而对不同发酵方式下酿酒酵母菌株多样性进行比较。自然发酵和接种发酵中共分离到90个酿酒酵母单菌落,经Interdelta指纹图谱即不同基因型的分析后,共分为13种基因型即13株不同的酿酒酵母菌株,除接种的NXU17-26、UCD522和UCD2610 3株酵母以外,分离到10株本土酿酒酵母(图 2)。结合Inerdelta和聚类分析结果可以看出,当欧氏距离为0.61时,分离自接种敏感菌株UCD522的发酵中的5种基因型(NXU19-12,NXU29-22,NXU19-13,NXU29-21和NXU29-23)被聚为一类。而分离自不同发酵中的酿酒酵母菌株间遗传差异性较大。

|
| 图 2 酿酒酵母的Interdelta指纹图谱UPGMA聚类图 Figure 2 The UPGMA cluster of Interdelta fingerprinting patterns of S. cerevisiae strains. |
13个基因型在各发酵时期的分布情况如图 3所示。本研究在不同的接种发酵中,发酵结束时酿酒酵母占主导地位甚至完全是酿酒酵母,但这些酵母的基因型是有差异的。接种中性菌株NXU17-26的发酵中共分离出24株酿酒酵母,表现为3种基因型(NXU18-01、NXU28-15和NXU28-16),NXU18-01是发酵中的优势酵母,其基因型占比为45.83% (11/24);接种敏感菌株UCD522的发酵中共分离出25株酿酒酵母,表现出5种基因型(NXU19-12、NXU29-22、NXU19-13、NXU29-21和NXU29-23),NXU29-21是发酵中的优势酵母,基因型占比为36.00% (9/25);接种嗜杀菌株UCD2610的发酵中分离出41株酿酒酵母,表现为3种基因型(NXU20-14、NXU30-19和NXU30-18),其中类型NXU20-14与所接种的UCD2610具有相同基因型,基因型占比为48.78% (20/41)。接种NXU17-26和UCD522的发酵中,未发现与NXU17-26和UCD522相同的基因型,可能是因为发酵过程中野生本土酵母表现出较强的竞争力,成为发酵中的优势菌株,接种的酿酒酵母生长受到了抑制;而接种的UCD2610发酵中,UCD2610占主导地位,这可能与UCD2610是嗜杀菌株有关。
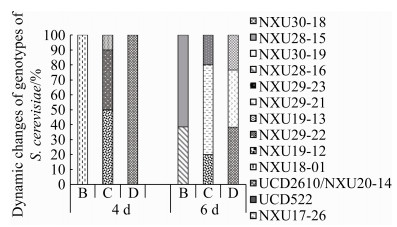
|
| 图 3 接种发酵过程中酿酒酵母的基因型动态变化 Figure 3 Dynamic changes of S. cerevisiae genotypes during inoculated fermentations. B: inoculated with NXU17-26; C: inoculated with UCD522; D: inoculated with UCD2610. |
2.4 酿酒酵母多样性指数
为更好地分析自然发酵和接种发酵对酿酒酵母菌株多样性的影响,将3个接种发酵中分离的90个酿酒酵母的多样性指数进行统计比较。如表 2所示,90个酿酒酵母单菌落共产生了11(3+5+3)种基因型。以基因型数量与分离到的菌株数量之间的比率表示生物多样性(%)[21],接种UCD522发酵过程中分离的酿酒酵母的多态性水平最高,生物多样性为20% (5/25);接种UCD2610发酵过程中分离的酿酒酵母的多态性水平最低,生物多样性为7.3% (3/41)。3个发酵中分离的酿酒酵母的优势度指数:接种UCD2610 > 接种NXU17-26 > 接种UCD522,优势度指数可以反映各物种种群数量的变化情况,优势度指数越大,则说明接种UCD2610的发酵过程中不同基因型酿酒酵母的分布越不均匀,因此,接种UCD2610发酵中的优势菌株(UCD2610)在发酵过程中占据更加突出的地位。接种UCD522发酵中分离的酿酒酵母的Shannon指数大于接种NXU17-26和接种UCD2610中分离的酿酒酵母,Shannon指数越大,说明接种UCD522发酵中酿酒酵母种群中的未知因素越多(表 2)。Simpson指数反映了不同基因型酿酒酵母的集中度,接种NXU17-26和接种UCD2610发酵中的Simpson指数值相似,接种UCD522发酵中的Simpson指数值大于其他两个发酵(表 2),则说明接种NXU17-26和接种UCD2610发酵过程中不同基因型酿酒酵母的集中程度大小基本相同,而接种UCD522发酵过程中不同基因型酿酒酵母的集中度较高。
| Different ferments | Number of S. cerevisiae isolates | Number of interdelta patterns | Biodiversitya/% | Berger-Parker dominance index | Shannon index (H′) | Simpson index (D) |
| NXU17-26 | 24 | 3 | 12.50 | 0.4583 | 1.5157 | 0.6354 |
| UCD522 | 25 | 5 | 20.00 | 0.3600 | 2.0621 | 0.7360 |
| UCD2610 | 41 | 3 | 7.30 | 0.4878 | 1.4906 | 0.6234 |
3 讨论
本文研究了不同发酵类型(接种发酵与自然发酵)对霞多丽葡萄酒发酵中酿酒酵母菌株多样性的影响。自然发酵中分离的酵母菌和商业酵母菌株具有相同的基因型,在我国宁夏、香格里拉产区已有报道[18, 22]。本研究中,自然发酵过程中未分离出与接种酵母相同的基因型,说明自然发酵中本土酵母表现出较强的竞争力,没有受到所接种的酿酒酵母(NXU17-26、UCD522和UCD2610)的影响。因此,亟需对我国本土酵母菌资源进行收集与开发,对发酵中分离的本土酿酵母菌资源进行分离、鉴定,可为后续开发和利用优良本土酵母菌株奠定基础,保护我国本土酵母菌资源的多样性。
葡萄酒的发酵是一个复杂的过程,是由不同的酵母属、种之间相互协作完成的。根据前人研究发现,自然发酵在发酵前期非酿酒酵母占据主导地位,在发酵中后期酿酒酵母成为优势菌。杨美景等[23]对赤霞珠葡萄自然发酵过程中的酵母菌进行了分离和鉴定并探寻赤霞珠葡萄酒发酵过程相关酵母菌群的变化规律,研究发现,酵母属(Saccharomyces)酵母是自然发酵中后期的主导菌,发酵前期主要是非酿酒酵母。Elena等对意大利白葡萄自发发酵过程中的酵母种群的多样性进行了研究,结果显示,发酵早期以Hanseniaspa、Issatchenkia和Candida为优势菌株,发酵中期和末期以酿酒酵母为主[24]。然而,本研究在自然发酵初期和中期Hanseniaspora uvarum为优势菌,发酵末期以Candida humili和Kazachstania hellenica为主,整个发酵过程中并未分离到酿酒酵母。自然发酵过程中的酿酒酵母可能来自酿酒厂、设备仪器和葡萄表皮等,本研究的自然发酵中没有分离到野生酿酒酵母,可能是由于该发酵是在实验室条件下进行的,所使用仪器都经过高压灭菌处理,加之发酵过程中野生非酿酒酵母与酿酒酵母间的竞争,而使野生酿酒酵母受到了抑制。此外,在分类学上,Candida humili与Kazachstania hellenica同位于哈萨克斯坦种属Kazachstania的进化分支上,两者都归类于Kazachstania属[25]。C. humilis是一种公认安全的酵母[26],具有较好的葡萄酒酿造潜力。对非酿酒酵母C. humilis和Kazachstania hellenica的遗传关系以及发酵特性等进行研究,从而进一步探索和开发宁夏产区本土的优良酵母资源。
研究表明,即使在接种商业酵母的情况下,野生酿酒酵母在发酵中也能表现出较强的竞争力,甚至在发酵中占据主导地位。宋育阳等研究接种商业酵母RC212的黑比诺葡萄发酵过程中酿酒酵母菌株动态变化,结果发现,在发酵早期和中期,接种的商业酵母RC212并没有表现出很强的竞争性,直到发酵末期才开始占据主导地位[3]。孙悦等对接种不同嗜杀特性的酿酒酵母的赤霞珠葡萄发酵中的酵母菌多样性进行了研究,结果显示所接种的酿酒酵母是相应发酵中的优势菌[27]。Vigentini等[1]在A厂和B厂接种相同的商业酵母进行发酵,对基酒发酵过程进行微生物监测。在A厂商业酵母在发酵中占据主导地位,而在B厂,发酵过程不是由接种的商业酵母主导完成的。本研究中,在接种NXU17-26和UCD522的发酵后期,未发现与NXU17-26和UCD522相同的基因型,这与接种的酵母与本土酵母间的竞争关系有关,后续研究可通过研究不同酿酒酵母菌株在发酵中的定殖能力,细胞间的相互作用来研究其在发酵中消失的原因。此外,本土酿酒酵母NXU18-01和NXU29-21在相应的发酵中表现出较强的发酵能力和竞争力,通过对本土酿酒酵母NXU18-01和NXU29-21酿酒特性的研究,有望获得适合该地区和该品种葡萄发酵的优良酵母菌系。
不同酵母菌株的酿造特性存在差异,尤其是嗜杀特性,使各发酵中酵母菌株之间的相互作用有所不同。嗜杀酵母在其生长繁殖过程中向体外分泌蛋白或者糖蛋白毒素,从而抑制或杀死某些特定的微生物[26, 28-30]。Villalba等对18个不同的嗜杀酵母进行了分析,结果显示,菌株T. delbrueckii NPCC 1033 (TdKT产生菌)对所有腐败酵母的生长均有明显的抑制作用[31]。Satora等通过评估嗜杀酵母菌株在苹果酒生产中对葡萄酒腐败微生物的抗菌活性,表明在最佳发酵条件下,所分析的嗜杀菌株抑制了11种葡萄酒腐败真菌(包括酵母菌和其他真菌菌株)中7种的生长[32]。在葡萄酒发酵中,嗜杀酵母能够抑制或杀死其近缘酵母,加速近缘酵母细胞自溶,增加葡萄酒风味物质的含量[33-34]。因此,分离筛选出适合宁夏地区的本土嗜杀酵母作为工业生产用菌株,可以有效防止生产过程中的杂菌污染,保证发酵的正常进行,丰富葡萄酒风味并酿造出具有当地特色的葡萄酒。
4 结论本研究以霞多丽葡萄为原料,进行了自然发酵和接种不同酿酒酵母菌株的发酵,通过对酵母菌种类和酿酒酵母菌株遗传多样性进行分析,证实了不同发酵方式对酵母菌的种类多样性具有显著影响,同时接种不同菌株的发酵过程中酿酒酵母的遗传多样性也存在明显差异。26S rDNA D1/D2区序列分析将分离到的酵母菌鉴定为6属11种,自然发酵中分离的酵母有5属6种,均为非酿酒酵母;而接种发酵中的酵母多样性远低于自然发酵,均由酿酒酵母和两种非酿酒酵母组成。此外,本研究发现在接种发酵中,接种所使用的酿酒酵母不一定是相应发酵中的优势菌株。如在接种NXU17-26和UCD522的发酵中,未分离到与NXU17-26和UCD522相同的基因型,本土酿酒酵母NXU18-01和NXU29-21在相应的发酵中表现出较强的竞争力,基因型占比分别为和45.83%和36.00%。通过对其酿酒特性的研究,有望获得适于宁夏地区葡萄酒发酵的优良酵母菌系。
| [1] | Vigentini I, Fabrizio V, Faccincani M, Picozzi C, Comasio A, Foschino R. Dynamics of Saccharomyces cerevisiae populations in controlled and spontaneous fermentations for Franciacorta D.O.C.G. base wine production. Annals of Microbiology, 2014, 64(2): 639-651. DOI:10.1007/s13213-013-0697-7 |
| [2] | Maturano YP, Lerena MC, Mestre MV, Casassa LF, Toro ME, Vazquez F, Mercado L, Combina M. Inoculation strategies to improve persistence and implantation of commercial S. cerevisiae strains in red wines produced with prefermentative cold soak. LWT, 2018, 97: 648-655. DOI:10.1016/j.lwt.2018.07.063 |
| [3] |
Song YY, Pei YF, Wang GP, Liu YL. Monitoring of yeast changes during inoculated fermentation process of pinot noir grape. Journal of Chinese Institute of Food Science and Technology, 2010, 10(2): 125-130.
(in Chinese) 宋育阳, 裴颖芳, 王国平, 刘延琳. 黑比诺葡萄接种发酵过程酵母菌的变化监控. 中国食品学报, 2010, 10(2): 125-130. DOI:10.3969/j.issn.1009-7848.2010.02.018 |
| [4] |
Li MH. Interactions of yeast strains in the fermentation process of cabernet sauvignon. Liquor-Making Science & Technology, 2016(11): 60-64.
(in Chinese) 李梅花. 复合酵母在赤霞珠发酵过程中酵母菌相互作用研究. 酿酒科技, 2016(11): 60-64. |
| [5] |
Sun Y, Ye DQ, Chu Y, Zhang YF, Liu YL. Effects of different inoculation methods & temperature on the fermentation of active dry yeast. Liquor-Making Science & Technology, 2019(3): 24-28.
(in Chinese) 孙悦, 叶冬青, 褚越, 张怡飞, 刘延琳. 不同接种方式及温度对活性干酵母发酵的影响. 酿酒科技, 2019(3): 24-28. |
| [6] |
Liu YL, Li H, Zhang H, Yang H, Liu LY, Deng JJ, Lu F, Liu ZJ, Xiong LJ, Xu YH. Effects of direct inoculation and re-activated inoculation of active dry yeast on wine fermentation. China Brewing, 2019, 38(9): 102-106.
(in Chinese) 刘苑琳, 李辉, 张衡, 杨华, 刘玲彦, 邓娟娟, 卢发, 柳志杰, 熊丽娇, 许引虎. 葡萄酒活性干酵母直投和活化工艺对酒精发酵的影响. 中国酿造, 2019, 38(9): 102-106. |
| [7] | Torija MJ, Beltran G, Novo M, Poblet M, Guillamón JM, Mas A, Rozès N. Effects of fermentation temperature and Saccharomyces species on the cell fatty acid composition and presence of volatile compounds in wine. International Journal of Food Microbiology, 2003, 85(1/2): 127-136. |
| [8] | Rodríguez-Porrata B, Novo M, Guillamón J, Rozès N, Mas A, Otero RC. Vitality enhancement of the rehydrated active dry wine yeast. International Journal of Food Microbiology, 2008, 126(1/2): 116-122. |
| [9] |
Pei YF, Liu YL. Application of DNA molecular markers in genetic diversities of Saccharomyces cerevisiae strains. Scientia Agricultura Sinica, 2010, 43(5): 1023-1030.
(in Chinese) 裴颖芳, 刘延琳. DNA分子标记技术在酿酒酵母菌株遗传多样性分析中的应用. 中国农业科学, 2010, 43(5): 1023-1030. DOI:10.3864/j.issn.0578-1752.2010.05.018 |
| [10] | Ayoub MJ, Legras JL, Saliba R, Gaillardin C. Application of multi locus sequence typing to the analysis of the biodiversity of indigenous Saccharomyces cerevisiae wine yeasts from Lebanon. Journal of Applied Microbiology, 2006, 100(4): 699-711. DOI:10.1111/j.1365-2672.2006.02817.x |
| [11] | Pallmann CL, Brown JA, Olineka TL, Cocolin L, Mills DA, Bisson LF. Use of WL medium to profile native flora fermentations. American Journal of Enology and Viticulture, 2001, 52(3): 198-203. |
| [12] | Wang CX, Liu YL. Dynamic study of yeast species and Saccharomyces cerevisiae strains during the spontaneous fermentations of Muscat blanc in Jingyang, China. Food Microbiology, 2013, 33(2): 172-177. DOI:10.1016/j.fm.2012.09.014 |
| [13] | 黄英子. 冰酒发酵过程中酵母菌的多样性及动态变化研究. 西北农林科技大学硕士学位论文, 2013. |
| [14] | Santamaría P, Garijo P, López R, Tenorio C, Gutiérrez AR. Analysis of yeast population during spontaneous alcoholic fermentation: effect of the age of the cellar and the practice of inoculation. International Journal of Food Microbiology, 2005, 103(1): 49-56. |
| [15] | Valero E, Cambon B, Schuller D, Casal M, Dequin S. Biodiversity of Saccharomyces yeast strains from grape berries of wine-producing areas using starter commercial yeasts. FEMS Yeast Research, 2007, 7(2): 317-329. DOI:10.1111/j.1567-1364.2006.00161.x |
| [16] |
Wang ZJ, Liu YL, Liu AG, Han N. Survey on yeast population dynamics during wine spontaneous fermentation in Xinjiang. Journal of Huazhong Agricultural University, 2008, 27(5): 664-667.
(in Chinese) 王泽举, 刘延琳, 刘爱国, 韩娜. 新疆葡萄酒自然发酵过程酵母菌的种类和动态变化. 华中农业大学学报, 2008, 27(5): 664-667. |
| [17] | Tofalo R, Perpetuini G, Fasoli G, Schirone M, Corsetti A, Suzzi G. Biodiversity study of wine yeasts belonging to the "terroir" of montepulciano d'Abruzzo "Colline Teramane" revealed Saccharomyces cerevisiae strains exhibiting atypical and unique 5.8S-ITS restriction patterns. Food Microbiology, 2014, 39: 7-12. DOI:10.1016/j.fm.2013.10.001 |
| [18] | Sun Y, Qin Y, Pei YF, Wang GP, Joseph CML, Bisson LF, Liu YL. Evaluation of Chinese Saccharomyces cerevisiae wine strains from different geographical origins. American Journal of Enology and Viticulture, 2017, 68(1): 73-80. DOI:10.5344/ajev.2016.16059 |
| [19] | 孙悦. 不同氮素水平对酿酒酵母混合发酵特征的影响及其代谢物研究. 西北农林科技大学博士学位论文, 2016. |
| [20] | Tristezza M, Vetrano C, Bleve G, Spano G, Capozzi V, Logrieco A, Mita G, Grieco F. Biodiversity and safety aspects of yeast strains characterized from vineyards and spontaneous fermentations in the Apulia Region, Italy. Food Microbiology, 2013, 36(2): 335-342. |
| [21] | Tóthmérész B. Comparison of different methods for diversity ordering. Journal of Vegetation Science, 1995, 6(2): 283-290. |
| [22] |
Yang K, Mao RZ, Zhao Y, He C, Wang HL, Cao JH, Su W, He XH. Biodiversity of wine-related yeasts isolated from Shangri-La wine-producing region of Yunnan. Microbiology China, 2018, 45(12): 2708-2721.
(in Chinese) 杨宽, 毛如志, 赵悦, 何迟, 王慧玲, 曹建宏, 速伟, 何霞红. 云南香格里拉葡萄酒产区酿酒相关酵母菌的生物多样性. 微生物学通报, 2018, 45(12): 2708-2721. |
| [23] |
Yang MJ, Chen XB, Zhao JJ, Niu SK, Wang ZC, Li Y. Identification of yeasts fermentation of isolated from spontaneous cabernet sauvignon. Food and Fermentation Industries, 2011, 37(7): 22-27.
(in Chinese) 杨美景, 陈小波, 赵静静, 牛帅科, 王照淳, 李艳. 赤霞珠葡萄自然发酵过程中酵母菌的分离与鉴定. 食品与发酵工业, 2011, 37(7): 22-27. |
| [24] | Di Maro E, Ercolini D, Coppola S. Yeast dynamics during spontaneous wine fermentation of the Catalanesca grape. International Journal of Food Microbiology, 2007, 117(2): 201-210. |
| [25] | Vaughan-Martini A, Lachance MA, Kurtzman CP. Kazachstania zubkova (1971)//Kurtzman CP, Fell JW, Boekhout T. The yeasts. 5th ed. Amsterdam: Elsevier, 2010: 439–470. |
| [26] | de Vuyst L, Harth H, van Kerrebroeck S, Leroy F. Yeast diversity of sourdoughs and associated metabolic properties and functionalities. International Journal of Food Microbiology, 2016, 239: 26-34. |
| [27] |
Sun Y, Zhang FF, Chu SX, Li JX, Shao S, Zhang JX. Effects of saccharomyces cerevisiae strains with different killer activities on yeast diversity during inoculated fermentation of cabernet sauvignon. Food Science, 2020, 41(2): 166-172.
(in Chinese) 孙悦, 张方方, 褚遂兴, 李佳幸, 邵帅, 张军翔. 接种不同嗜杀特性的酿酒酵母对赤霞珠发酵中酵母多样性的影响. 食品科学, 2020, 41(2): 166-172. |
| [28] | Zagorc T, Maráz A, Cadez N, Jemec KP, Péter G, Resnik M, Nemanič J, Raspor P. Indigenous wine killer yeasts and their application as a starter culture in wine fermentation. Food Microbiology, 2001, 18(4): 441-451. |
| [29] |
Zhao JJ, Li Y. Biological properties of killer yeast and its application. Liquor-Making Science & Technology, 2010(9): 68-72.
(in Chinese) 赵静静, 李艳. 嗜杀酵母的生物学特性及应用展望. 酿酒科技, 2010(9): 68-72. |
| [30] |
Wang L, Tan CM, Yu G, Yang XQ, Qi B, Yang SL. Biological properties of killer yeasts and their applications. Journal of Tianjin Agricultural College, 2017, 24(1): 83-87.
(in Chinese) 王麟, 谭春明, 于刚, 杨贤庆, 戚勃, 杨少玲. 嗜杀酵母的生物学特性及其应用. 天津农学院学报, 2017, 24(1): 83-87. |
| [31] | Villalba ML, Sáez JS, Del Monaco S, Lopes CA, Sangorrín MP. TdKT, a new killer toxin produced by Torulaspora delbrueckii effective against wine spoilage yeasts. International Journal of Food Microbiology, 2016, 217: 94-100. |
| [32] | Satora P, Cioch M, Tarko T, Wołkowicz J. Killer strains of Saccharomyces: application for apple wine production. Journal of the Institute of Brewing, 2016, 122(3): 412-421. |
| [33] |
Zhan JH, Yang YH, Tan YL, Kang ZJ. Research advances on killer yeasts and their applications in alcoholic beverage industry. China Brewing, 2007(6): 1-5.
(in Chinese) 詹金花, 杨幼慧, 谭余良, 康增军. 嗜杀酵母的研究进展及其在酿酒工业中的应用. 中国酿造, 2007(6): 1-5. |
| [34] | Todd BEN, Fleet GH, Henschke PA. Promotion of autolysis through the interaction of killer and sensitive yeasts: potential application in sparkling wine production. American Journal of Enology and Viticulture, 2000, 51(1): 65-72. |
 2021, Vol. 61
2021, Vol. 61