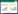中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 王帅涛, 高倩倩, 成娟丽, 林金水. 2021
- Shuaitao Wang, Qianqian Gao, Juanli Cheng, Jinshui Lin. 2021
- 铜绿假单胞菌生物被膜组成及其受群体感应系统和c-di-GMP调控的研究进展
- Regulation of Pseudomonas aeruginosa biofilms by quorum sensing systems and c-di-GMP
- 微生物学报, 61(5): 1106-1122
- Acta Microbiologica Sinica, 61(5): 1106-1122
-
文章历史
- 收稿日期:2020-06-03
- 修回日期:2020-08-28
- 网络出版日期:2020-10-23
2. 陕西省红枣重点实验室(延安大学), 陕西延安 716000
2. Shaanxi Key Laboratory of Chinese Jujube(Yan'an University), Yan'an 716000, Shaanxi Province, China
生物被膜是细菌依附于各种固体表面,由自身产生胞外多糖、基质蛋白和胞外DNA (extracellular DNA,简称eDNA)等大分子物质形成的一种高度结构化的膜状复合物将自身包裹形成的群体结构[1]。包裹在生物被膜中的细菌可以提高对有害环境的抵抗、在营养丰富的区域定殖,促进细菌群体之间的交流等[2]。因此大多数细菌在生命周期中都会形成生物被膜,尤其当病原菌侵染宿主时,由于生物被膜对抗生素和宿主免疫反应的抵抗作用,给治疗带来很大麻烦[1, 3]。据统计,在医院内的病原菌感染有65%–80%都与生物被膜有关[4–5]。体外生物被膜模型的敏感性试验表明,使用抗生素杀菌时,有生物被膜包裹的细菌存活率是浮游细菌的几百倍甚至上千倍[6]。这说明,在临床上采用抗生素治疗很可能是通过杀死浮游细菌来抑制感染症状,但无法根除那些嵌入生物被膜中的细菌,当抗菌药物停止作用时,生物被膜可以作为感染复发的工具。对生物被膜感染的治疗通常会持续到被外科手术从身体中彻底移除为止[7]。
铜绿假单胞菌(Pseudomonas aeruginosa)是一种环境中普遍存在的革兰氏阴性细菌,是人类条件性感染的前三大病原菌之一,它作为重要病原体的主要原因是其对抗生素和消毒剂有超强抗性[8]。对于免疫功能低下、囊性纤维化(cystic fibrosis,CF)、烧伤、开放性骨折或植入医疗器械(如导管)等患者有很强的感染能力[9],占院内感染的10%–20%,并被世界卫生组织列为急需开发新型抗菌药物的人类病原体之一[8, 10]。铜绿假单胞菌可以成功侵染有机体并导致持久性感染,其中一个重要的因素就是其具有形成生物被膜的能力,如囊性纤维化患者的支气管和慢性肺部感染[9–11]。
作为研究细菌生物被膜的模式生物之一,关于铜绿假单胞菌生物被膜的组成、调控、在宿主感染中的作用以及耐药机制等方面均有深入的研究[12–15]。本综述主要概述了铜绿假单胞菌生物被膜的各组成成分及其在生物被膜形成中的重要作用,并进一步阐述了对铜绿假单胞菌生物被膜形成起关键作用的两种调控系统:群体感应系统和c-di-GMP (环二鸟苷酸)系统。
1 铜绿假单胞菌生物被膜的组成成分 1.1 胞外多糖胞外多糖成分是细菌生物被膜最主要的组成成分,不同种类的胞外多糖可以影响最终形成的生物被膜形态,如:粘液型生物被膜、蘑菇状生物被膜以及丝状生物被膜等[16],铜绿假单胞菌成熟蘑菇状生物被膜如图 1所示。胞外多糖成分是生物被膜支撑结构的骨架成分,它可以帮助细菌对营养物质的吸收以及细胞与细胞之间的信号交流等[17–18]。铜绿假单胞菌可以合成至少3种类型的胞外多糖:褐藻多糖、Pel多糖和Psl多糖[19]。在生物被膜的形成过程中,由于不同类型胞外多糖发挥不同的功能,因此不同的生长环境会使铜绿假单胞菌在产生胞外多糖时具有选择性[14–16]。不同的胞外多糖类型也会影响铜绿假单胞菌对抗生素的抵抗能力,影响铜绿假单胞菌感染机体时毒力的发挥以及铜绿假单胞菌和其它细菌形成混合生物被膜的能力等[20]。以下是3种胞外多糖分别在铜绿假单胞菌生物被膜发育中的作用。
|
| 图 1 非粘液型铜绿假单胞菌成熟生物被膜模式图 Figure 1 The model of non-mucous P. aeruginosa mature biofilm. |
1.1.1 褐藻多糖:
铜绿假单胞菌产生的褐藻多糖(alginate)是由β-1-4糖苷键连接的α-L-古洛糖醛酸(α-L-guluronic acid)和β-D-甘露糖酸(β-D-mannuronic acid)组成的一种阴离子多糖[21–22]。由铜绿假单胞菌基因组中12个基因algD、alg8、alg44、algK、algE、algG、algX、algL、algI、algJ、algF和algA组成的操纵子(PA3540-PA3551)负责编码合成[23]。其中AlgA、AlgD和AlgC负责前体GDP-甘露糖醛酸的合成和激活,之后聚合酶Alg8和Alg44将单个GDP-甘露糖醛酸聚合成多聚甘露糖醛酸,AlgI、AlgJ和AlgF将多聚甘露糖醛酸在周质中进行O-乙酰化修饰,然后AlgG将D-甘露糖醛酸转化为L-古洛糖醛酸,最后褐藻多糖裂解酶AlgL将褐藻多糖降解为小分子褐藻多糖,经AlgK和AlgE将周质中褐藻多糖转运至胞外[23]。有研究发现,在人的气道定殖和患有囊性纤维化的个体感染期间,铜绿假单胞菌会过度产生褐藻多糖使其生物被膜表型转化为粘液表型[24]。这是因为粘液型生物被膜比非黏液型生物被膜更能抵抗宿主免疫系统的攻击和抗生素治疗,从而对机体造成持续感染[25–26]。由于褐藻多糖在囊性纤维化病病理学中的重要性使其成为首个被研究的胞外多糖,就目前对铜绿假单胞菌胞外多糖的生物合成和调控机制的认识而言,褐藻多糖仍然是被研究的最清楚的铜绿假单胞菌胞外多糖[27]。
褐藻多糖过度产生导致的粘液型生物被膜主要在铜绿假单胞菌感染机体时出现[28]。囊性纤维化病患肺部的感染,将导致炎症细胞被转移到感染部位,它们释放出的活性氧造成广泛的组织损伤,而铜绿假单胞菌过度产生褐藻多糖可以保护自身免受这种炎症反应的杀伤。因为褐藻多糖可以清除机体释放的自由基,并保护细菌免受活化巨噬细胞的吞噬清除[29–30]。此外在感染期间,铜绿假单胞菌过量产生褐藻多糖可以减少自身合成一些毒力因子如:铁载体、鼠李糖脂等,从而减弱宿主的免疫反应并且促进与其他细菌如金黄色葡萄球菌的共感染[31]。然而,最近研究表明尽管褐藻多糖是铜绿假单胞菌粘液型生物被膜的主要胞外多糖成分,但并不是其生物被膜的必要结构组分[32]。相反,遗传研究表明Psl和Pel多糖才是粘液型铜绿假单胞菌生物被膜中不可或缺的骨架成分,它们可调节大菌落生物被膜的形成[24, 32]。
1.1.2 Psl多糖:Psl多糖是由D-甘露糖(D-mannose)、D-葡萄糖(D-glucose)和L-鼠李糖(L-rhamnose)构成的五糖重复单位组成的中性多糖[33]。由铜绿假单胞菌基因组中pslA、pslB、pslC、pslD、pslE、pslF、pslG、pslH、pslH、pslI、pslJ、pslK、pslL、pslM、pslN、pslO组成的操纵子(PA2231-PA2245)负责编码合成,其详细合成过程还没有被报道[17]。最新研究表明,PslE与PslA,PslD和PslG之间的相互作用,有助于PslD在细胞外膜定位从而将Psl多糖分泌到胞外[34]。铜绿假单胞菌生物被膜内Psl多糖有两种存在形式,其中大分子量的低聚糖重复单位呈螺旋状分布在细胞表面从而在细胞-细胞和细胞-基质表面相互作用中发挥作用[15],而分子量较小的以可溶性状态分布在整个生物被膜基质中[33]。由于Psl多糖的存在,可以将细菌细胞聚集在一起,增加细菌细胞与固体介质的粘附作用,从而促进早期生物被膜微菌落的建立[35]。在生物被膜形成的早期,浮游细菌在固体表面附着时会表现快速附着和慢速附着两种表型,这种独特附着表型是由细菌产生Psl多糖的差异表达介导,表明Psl多糖的差异表达在早期生物被膜的发育和形成中发挥重要作用[36]。随着生物被膜的进一步发育,Psl多糖作为生物被膜的支架发挥作用,它通过增强生物被膜基质的交联和弹性以维持生物被膜的结构和形态[35]。一些研究表明,在抗生物被膜药物的存在下,相比于不能产生Psl多糖的菌株,可以产生Psl多糖的菌株仍然能够有效形成生物被膜,说明Psl多糖对稳定生物被膜结构具有重要作用[37]。
Psl多糖通常是生物被膜内细菌群落抵御抗生素攻击的第一道防线。由于自然界的生物被膜往往是由多种细菌构成的混合生物被膜,所以一些不能产生Psl多糖的细菌可以利用铜绿假单胞菌产生的Psl多糖获得耐受性而保护自身[38]。Psl多糖可以抵抗如阳离子抗菌肽(多粘菌素B)、妥布霉素和环丙沙星等抗菌物质对铜绿假单胞菌的毒害。根据荧光标记多粘菌素B的定位实验结果,发现Psl多糖聚合物可能是通过直接的相互作用吸附小的抗生素分子或降低了抗生素与细胞表面的亲和力,从而增强生物被膜内细菌对抗生素的耐受性[37, 39]。但是Psl多糖的这种保护作用是在生物被膜形成的早期阶段观察到的,随着生物被膜继续发育成为成熟的蘑菇状生物被膜时,这种对抗菌物质的抵抗能力并不会随之增强[15]。此外,在感染铜绿假单胞菌的患者体内发现针对Psl多糖的特异性抗体,表明Psl多糖是铜绿假单胞菌感染的重要生物标志物之一[40]。
近年来,由于Psl多糖水解酶PslG在铜绿假单胞菌生物被膜的破坏和抑制方面的重要作用,因而引起了许多关注。PslG是由pslG基因负责编码的一种属于GH39家族的糖苷水解酶,负责催化Psl多糖的水解[32]。PslG蛋白位于细胞周质空间,并通过其N端跨膜结构域锚定在内膜上,其周质区的活性位点凹槽呈现的镰刀形结构特征与其他GH39家族成员不同[32]。在早期的研究中,通过对pslG的遗传缺失证明它是Psl多糖生物合成所必需的[33]。但是后来的研究表明,敲除pslG导致Psl多糖缺陷的表型并不是由PslG酶活缺失造成的,而是由于pslG基因敲除影响了pslG下游基因的表达,因此Psl多糖的生物合成既不需要PslG也不需要其酶促活性[32]。相比之下,PslG的过度表达可以在短时间内水解Psl多糖从而破坏已经形成的生物被膜,显示出在抗生物被膜方面的重要作用[18, 41]。研究表明,在铜绿假单胞菌中外源添加PslG纯蛋白可以通过水解Psl多糖而降低细菌细胞的粘附能力,并增加细菌的运动速度,从而抑制生物被膜微菌落的形成[35],最终显著增强生物被膜中的细菌对抗生素和巨噬细胞的敏感性,改善小鼠感染模型中生物被膜的清除率[41]。此外,PslG也对其它多种假单胞菌属的细菌均表现出生物被膜抑制和清除活性,表明其在生物医学领域具有潜在的应用价值[41]。
1.1.3 Pel多糖:由于可以产生Pel胞外多糖的细菌在静置培养过程中,其培养物往往会在气-液界面形成生物被膜[17]。因此Pel多糖的合成基因最初在铜绿假单胞菌PA14中使用转座子插入突变,通过在突变体中筛选气-液界面生物被膜缺陷型菌株,从而鉴定了负责合成Pel多糖的操纵子PA3058-PA3064,它由7个相邻基因pelA、pelB、pelC、pelD、pelE、pelF、pelG组成[17, 42]。Pel多糖在最近的研究中被鉴定为由部分去乙酰化的N-乙酰-D-氨基葡萄糖(N-acetyl-D-glucosamine)和N-乙酰-D-半乳糖胺(N-acetyl-D-galactosamine)组成的阳离子多糖[43]。Pel多糖由PelD、PelE、PelF和PelG组成的内膜复合物负责合成,在周质空间经PelA脱乙酰化酶作用下部分去乙酰化而带上正电荷,接着被带负电荷的外膜脂蛋白PelC蛋白吸引,并引导其穿过外膜孔蛋白PelB而分泌到胞外,它可以保护细菌免受氨基糖苷类抗生素的杀害[44]。PelA除了具有脱乙酰化酶活性外,还具有糖苷水解酶活性,能水解Pel多糖[44]。虽然PelA的水解酶活性在Pel多糖生物合成中的作用尚不清楚,但是目前针对它水解破坏Pel多糖的特性,经常与PslG一起被应用于抗生物被膜的研究[18, 44]。
Pel多糖和Psl多糖都可以作为生物被膜基质的关键结构成分,在PAO1中,Psl多糖定位于生物被膜菌落的边缘。在Psl多糖缺失的情况下,生物被膜菌落边缘的Pel产量增加,表明Pel多糖可以在一定程度上与Psl多糖功能互补[15, 43]。然而,与Psl多糖相比,Pel多糖在生物被膜发育过程中并没有起关键作用,而主要在生物被膜发育后期增加生物被膜的厚度和促进生物被膜的扩散[42, 45]。另外,不同于Psl多糖形成的光滑型菌落生物被膜,Pel多糖形成的是皱纹型菌落生物被膜,同时由于Pel多糖与刚果红的结合能力更强而使其形成的皱纹型菌落生物被膜结合更多的刚果红[42]。此外,在铜绿假单胞菌-金黄色葡萄球菌的混合生物被膜模型中,Pel多糖可以将不同种菌株连接在一起,从而促进混合生物被膜的形成[20]。Pel多糖还与生物被膜基质中的另一重要组分eDNA通过正负离子间相互吸引而交联在一起,促进生物被膜的稳定性[43]。还有研究表明,在没有Ⅳ型菌毛的情况下,Pel多糖可以在细菌附着时发挥作用,表明它可以作为一种附着因子来帮助细菌在固体表面定殖[42]。
1.2 基质蛋白生物被膜基质中的蛋白成分对生物被膜的形成和稳定也发挥着重要的作用,这些基质蛋白主要为细胞表面附属物(主要为鞭毛和Ⅳ型菌毛)、细胞粘附素、细胞凝集素、功能性淀粉样蛋白以及一些起非结构功能的蛋白等。细胞表面附属物(主要为鞭毛和Ⅳ型菌毛)通过影响细菌迁移和固体表面附着,在生物被膜的形成时发挥重要作用[46]。铜绿假单胞菌能够通过鞭毛的挥鞭运动在液体中游泳,并能够通过Ⅳ型菌毛的伸展和收缩在固体表面移动。细菌的这种迁移能力使其在发育过程中具有广泛的运动性、竞争性和选择性[46–47]。铜绿假单胞菌PA14形成生物被膜需要鞭毛或鞭毛驱动的运动,而生物被膜微菌落的聚集则需要Ⅳ型菌毛[48]。然而,突变铜绿假单胞菌Ⅵ型分泌系统结构基因icmF3在削弱细菌运动能力的同时,生物被膜形成能力却反而增强[49]。这符合目前普遍接受的一种铜绿假单胞菌生物被膜的发育模型:鞭毛介导细菌向固体表面泳动,Ⅳ型菌毛驱动的沿固体表面的运动导致细胞聚集和小菌落的形成,随后在生物被膜形成的后期为了使其结构更加稳定,细菌在大菌落生物被膜基质中运动能力会被抑制甚至完全丧失,但生物被膜整个形成过程需要细胞间的信号传导[49–50]。
第二信使c-di-GMP在铜绿假单胞菌和其他革兰氏阴性细菌内正调控生物被膜基质蛋白成分的产生[51–52]。通过检测鉴定了铜绿假单胞菌中响应c-di-GMP升高而被诱导转录的基因,虽然其中大多数基因属于pel和psl基因簇[53],但是响应其转录的基因cdrA (PA4625)和cdrB (PA4624)也被证实在生物被膜形成过程中发挥作用。CdrA作为一种细胞粘附素,被外膜转运蛋白CdrB转运到胞外,可促进液体培养中生物被膜的形成和自动聚集;此外通过凝集素染色实验表明,CdrA可以将Psl多糖聚合物交联到细胞上,从而增加生物被膜的结构稳定性,说明CdrA作为一种生物被膜基质蛋白在细胞聚集和生物被膜结构稳定中发挥作用[54]。
作为铜绿假单胞菌毒力因子的一些细胞凝集素也是生物被膜形成所需要的,如凝集素基因lecA (PA-IL)或lecB (PA-IIL)单基因突变体表现出生物被膜缺陷表型[55–56]。LecA对疏水半乳糖苷具有较高的特异性和亲和力,可以在不同细菌细胞表面交联半乳糖苷,从而将细菌连接在一起,促进生物被膜微菌落的形成[55]。生物被膜中,细菌可以通过细胞外膜存在的LecB与Psl多糖结合,使细菌与生物被膜嵌合更加稳定[56]。LecB可以特异性结合L-海藻糖(L-fucose),在细菌细胞外膜存在的LecB通过与碳水化合物配体的结合参与细胞表面粘附[56],如LecB通过介导铜绿假单胞菌粘附于相同或不同物种的细胞上,从而使其在宿主组织上得以定殖或形成单物种或多物种生物被膜。因此通过干扰凝集素介导的细胞聚集和生物被膜的形成,可以有效治疗因生物被膜引起的铜绿假单胞菌感染的问题[57]。此外,从荧光假单胞菌中鉴定出一种功能性淀粉样蛋白,当其过表达时可以显著增强生物被膜的粘附效果。fapC是淀粉样蛋白主要亚基的编码基因,它在假单胞菌属中包括铜绿假单胞菌是保守的。在铜绿假单胞菌中编码FapC的基因位于一个含有6个基因的保守操纵子(fapA-fapF)中。大肠杆菌BL21(DE3)中异源表达fapA-F操纵子导致细菌表现出高度聚集表型,暗示该操纵子可能参与生物被膜的形成[58]。
最近,一种在生物被膜中起非结构作用的蛋白PA2755被首次鉴定,它可以与Psl多糖结合从而抑制铜绿假单胞菌感染期间宿主免疫系统产生的杀菌酶,并保护生物被膜内细胞免受该酶诱导的裂解,这可能代表了一种新的生物被膜保护机制,进一步提高了它们对免疫反应的耐受性[59]。综上,生物被膜内的这些复杂基质蛋白在其形成过程中可以发挥如:帮助细菌定殖、促进细胞聚集、调控细菌代谢、稳定生物被膜结构和参与抵抗宿主免疫反应等功能,对生物被膜的发育和形成至关重要。
1.3 eDNA在许多革兰氏阴性细菌和革兰氏阳性细菌的生物被膜组成和形成过程中,eDNA起着关键作用,它可以作为生物被膜的支撑成分、在营养缺乏时期为生物被膜内细菌提供营养[60]。eDNA的主要结构与其细菌染色体DNA相似,一般被认为是细胞裂解后的DNA残留物[61]。eDNA来源主要有两种途径:细胞裂解释放和非裂解释放。在细胞裂解释放途径中,细胞会产生各种细胞裂解剂,如原噬菌体、自溶蛋白、酶等诱发细胞自溶,从而释放DNA;非裂解释放主要是通过包裹大量DNA的细菌外膜囊泡的释放而产生的[60, 62–63]。在铜绿假单胞菌中,这两种途径都涉及eDNA的产生,但主要来源还是通过细胞自溶释放[64]。有研究表明,铜绿假单胞菌中eDNA的释放受绿脓菌素的调控,它通过诱导过氧化氢(H2O2)介导的细胞裂解来促进eDNA的释放[65]。
eDNA被认为是促进铜绿假单胞菌生物被膜形成的重要因子,参与了细菌细胞的初始附着、细胞-细胞的互连和生物被膜大菌落的形成[66]。脱氧核糖核酸酶Ⅰ是临床上唯一使用的一种破坏铜绿假单胞菌生物被膜的酶。这种治疗酶是通过水解细胞外基质中的eDNA破坏生物被膜。由于eDNA参与了初始生物被膜的建立,未成熟的铜绿假单胞菌生物被膜对脱氧核糖核酸酶Ⅰ的处理比成熟的生物被膜更敏感[67–68]。最近研究发现铜绿假单胞菌产生的绿脓菌素可以插入eDNA的含氮碱基,二者的结合显著影响铜绿假单胞菌细胞表面疏水性,并影响细胞之间的理化相互作用,促进细胞间聚集,并最终促进牢固的生物被膜的形成[65, 69]。
此外,eDNA可以与其他带正电荷的抗菌药物如氨基糖苷类和抗菌肽结合,从而抵御抗生素对细菌的攻击[70–71]。尽管Ca2 +对eDNA的释放没有影响,但带负电荷的eDNA与Ca2+结合,会促进细菌细胞之间的交叉联接,帮助细胞聚集[72]。在囊性纤维化患者气道内发现eDNA可以通过螯合外膜上结合的阳离子而激活铜绿假单胞菌介导抗菌作用的H1-T6SS系统,从而非特异性攻击邻近细菌[73]。eDNA还可以和铜绿假单胞菌Psl多糖结合,形成了eDNA–Psl纤维网,从而为细菌生物被膜提供结构支持和抵抗靶向生物被膜基质的药物的攻击[74]。同时,面对生存压力时,细菌可以利用不同物种eDNA促进混合生物被膜的形成以提高存活率[74]。
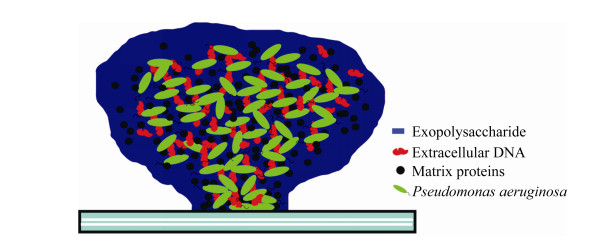
2 生物被膜形成的调控机制 2.1 群体感应系统对生物被膜形成的影响群体感应(quorum sensing)是一种细胞密度依赖的细菌-细胞间信号机制,使细菌能够协调某些基因的表达[75]。信号传递分子通常是被称为自诱导物的小分子,它们可以自由通过细胞膜释放到胞外,当环境中的自诱导物浓度达到阈值即可激活下游特定基因转录[76]。群体感应是细菌群体协调其特定功能的一种方式,例如产生抗生素抗性或引起感染。此外,这种合作不仅仅发生在同一物种内的细菌,对于不同物种细菌也可能共用同一种自诱导物[77–80]。铜绿假单胞菌中至少有四种群体感应系统:las、rhl、pqs和iqs。在每个群体感应系统中,由相应的信号分子与受体蛋白结合,从而激活与毒力因子、次级代谢和生物被膜发育有关的多种基因的转录[78–79],如图 2所示。这4种信号机制在铜绿假单胞菌体内交织成一个复杂的多层次的调控网络,可以调节铜绿假单胞菌基因组中大约12%的基因[80]。由于这种基于细胞密度的细胞间通讯系统在细菌毒力和生物被膜形成的调控中起着关键作用,是目前很有前景的一种防治铜绿假单胞菌感染的药物靶点[78, 80]。
|
| 图 2 铜绿假单胞菌中四种群体感应系统促进生物被膜形成模式图 Figure 2 The model of four quorum sensing systems in Pseudomonas aeruginosa promoting biofilm formation. |
2.1.1 las和rhl系统对生物被膜形成的影响:
las和rhl系统都是使用酰基高丝氨酸内酯(AHL)作为信号分子,与信号分子受体蛋白结合而起调控作用[81]。在las系统中,由LasI合成的小分子OdDHL (N-(3-氧十二烷酰基)-L-高丝氨酸内酯)与其受体蛋白LasR结合形成LasR-OdDHL复合物;在rhl系统中,RhlI负责合成BHL (N-丁酰-L-高丝氨酸内酯),并与其受体蛋白RhlR结合形成RhlR-BHL复合物[80]。LasR-OdDHL和RhlR-BHL复合物与存在于靶基因启动子中的保守las-rhl结合位点结合,从而激活下游基因的转录表达[81]。las系统已被证明可以调节多种毒力因子的表达,如胞外酶(LasB弹性蛋白酶、LasA蛋白酶、碱性蛋白酶)、次级代谢产物(绿脓菌素、氰化氢)、毒素(外毒素A)等[82]。其中一些小分子物质在生物被膜的形成过程中发挥重要作用,如绿脓菌素可以调控eDNA的释放促进生物被膜的形成[70]。在铜绿假单胞菌中,las系统正调控酪氨酸磷酸酶TpbA (PA3885)的产生,该酶通过具有GGDEF结构域的蛋白TpbB (PA1120)的去磷酸化,而抑制Pel多糖的产生[80, 83]。此外,LasR可以与psl操纵子的启动子区域结合,这表明las系统可以调节Psl多糖的表达[45, 84]。
rhl系统可以调控生物表面活性剂—鼠李糖脂的产生,鼠李糖脂在生物被膜形成后期维持细胞聚集体之间的开放空间起到重要作用。因为鼠李糖脂可以促进成熟生物被膜内部腔的形成,允许营养物质和水的适当流通,从而在生物被膜结构中发挥重要作用[85]。它还可防止无氧硝酸盐呼吸过程中一氧化氮(NO)的积累,这可能在临床环境中对维持铜绿假单胞菌生物被膜产生重要作用[86]。rhl系统还可以调控凝集素LecA的产生,LecA通过交联细胞-细胞表面半乳糖苷,帮助形成生物被膜微菌落,并进一步形成成熟的生物被膜[55]。此外,rhlR突变体和rhlI突变体具有完全不同的菌落生物被膜表型,rhlR突变体的菌落生物被膜表面具有明显褶皱,而rhlI突变体的菌落生物被膜表面则是光滑的,同时rhlI突变不影响铜绿假单胞菌的毒力而rhlR突变体则表现出明显的毒力衰减表型。这暗示RhlR可以同时指导RhlI依赖性和RhlI非依赖性的两种方式来控制基因表达,即RhlR响应于一个可替代BHL的新型配体,这种配体在没有RhlI的情况下通过RhlR调控相关基因的表达[87]。随后的研究表明rhlR突变体和rhlI突变体之间菌落生物被膜表型的差异可能与吩嗪类化合物合成量的差异有关,因为rhlR突变使铜绿假单胞菌缺失吩嗪类化合物的合成,而rhlI突变则不影响这一表型;进一步的研究显示rhlI突变后,在不存在BHL信号分子的情况下,RhlR响应于一个由PqsE负责合成的新配体,从而激活吩嗪合成基因的表达,使菌落生物被膜呈光滑表面[87–88]。
2.1.2 pqs系统对生物被膜形成的影响:铜绿假单胞菌pqs系统使用2-烷基-4-喹诺酮(AQ)作为信号分子,主要包括2-庚基-3-羟基-4-喹诺酮(PQS)和2-庚基-4-羟基喹啉(HHQ),这些信号分子在铜绿假单胞菌中由pqsABCDE操纵子和两个独立转录单元pqsH和pqsL负责合成[79]。HHQ是PQS的合成前体,它们共用同一个受体蛋白—PqsR,PqsR–PQS (或PqsR–HHQ)复合物在铜绿假单胞菌中只有一个直接调控位点,即pqsABCDE的启动子区[80, 89]。与rhl系统相似,PqsR也可以不依赖于PQS或HHQ发挥功能,目前已知PqsR可以与铜绿假单胞菌基因组的37个基因位点结合[79–80]。因为pqs突变菌株显示出生物被膜形成被抑制,毒力因子包括绿脓菌素、弹性蛋白酶、凝集素(LecA)和鼠李糖脂等次级代谢物合成减少的表型[90–91],所以pqs系统对生物被膜形成时细菌聚集、生物被膜结构稳定、成熟生物被膜内部腔的形成等具有重要作用。与las和rhl系统不同的是,pqs系统还可以促进eDNA的释放以帮助生物被膜的形成[66]。因此,pqs突变不仅使铜绿假单胞菌生物被膜的发育出现缺陷,也导致形成的生物被膜中eDNA含量显著减少[66, 92]。pqs系统促进铜绿假单胞菌eDNA释放的可能的原因是:(1) pqs系统正调控绿脓菌素的产生,从而诱导过氧化氢(H2O2)介导的细胞裂解来促进铜绿假单胞菌中eDNA的释放[70]。(2) PQS对细胞具有自我毒性作用,在细菌生长后期会导致细菌裂解释放eDNA[93]。此外,PQS还通过介导铁摄取、细胞毒性、外膜囊泡的生物发生和宿主免疫调节等方面的功能为生物被膜群体在营养摄取和降低宿主免疫方面提供帮助[79–80, 94]。
2.1.3 iqs系统对生物被膜形成的影响:铜绿假单胞菌iqs系统也被称为集成群体感应系统,由一个非核糖体肽合酶基因簇ambBCD所编码,以2-(2-羟苯基)-噻唑-4-甲醛(IQS)为信号分子,其受体蛋白还没被鉴定[80, 95]。它可以替代失活的las系统调节下游的pqs、rhl系统,以及协调群体生理和细菌毒力,提高细菌在病原体-宿主相互作用中的竞争力[95]。iqs系统受磷酸盐饥饿的诱导,而磷酸盐耗竭是细菌在宿主感染时的一种常见压力,通过这种整合机制将应激信号和群体感应结合起来,以促进细菌毒力因子的快速产生[96]。iqs系统类似于其他3个群体感应系统,可以调控如绿脓菌素,鼠李糖脂和弹性蛋白酶等毒力因子的产生,对铜绿假单胞菌生物被膜的形成也有很重要的作用[79]。比如,通过研究IQS衍生物对铜绿假单胞菌生物被膜的抑制效果,发现具有4碳链长的芳香族R基团的IQS衍生物对生物被膜形成具有显著的抑制效果,而IQS衍生物的噻唑环是这种抑制作用的关键[97];一种名为B-11的先导化合物在磷酸盐限制条件下通过抑制IQS途径明显下调rhlA和pqsA基因的表达,从而减少了与rhl途径和pqs途径相关的毒力因子,如鼠李糖脂和绿脓菌素的产生[97]。
2.2 c-di-GMP对生物被膜形成的影响环二鸟苷酸(c-di-GMP)首次在木葡糖酸醋杆菌(Gluconacetobacter xylinus)中被鉴定,它可以通过调节纤维素合酶活性来控制该细菌产生纤维素[98],它通常作为第二信使在包括铜绿假单胞菌在内的很多细菌体内广泛存在。c-di-GMP除了作为变构激活剂的作用,还可以将环境信号与基因调控耦合起来[99],如图 3所示。c-di-GMP是由两个GTP分子通过含有GGDEF结构域的二鸟苷酸环化酶(DGC)介导合成,并通过含有EAL或HD-GYP结构域的专一磷酸二酯酶(PDE)被分解为5′-磷酸鸟苷酸-(3′-5′)-鸟苷(pGpG)或鸟苷酸(GMP)[100]。这几个保守结构域对于它们相应的酶促反应是必需的[100]。除了GGDEF,EAL或HD-GYP结构域之外,c-di-GMP代谢酶中还有存在多种特殊结构域,例如信号接收结构域、信号传输结构域以及响应调节结构域,它们很可能参与环境中信号接收和传输[101],并通过将这些信号翻译、转化为酶活性质的改变,从而导致c-di-GMP水平的局部或整体波动,进而调整细胞的行为[101]。

|
| 图 3 c-di-GMP的合成、分解以及在铜绿假单胞菌中的功能 Figure 3 Synthesis and degradation of c-di-GMP and its function in P. aeruginosa. |
在铜绿假单胞菌中,有41种包含这些特殊结构域的蛋白,包括3种HD-GYP结构域蛋白,17种GGDEF结构域蛋白和5种EAL结构域蛋白,以及16种同时具有GGDEF和EAL结构域的蛋白[102–103]。其中含有GGDEF结构域的WspR蛋白,不但参与调节糖原的表达(在铜绿假单胞菌中糖原是合成生物被膜主要结构组分—胞外多糖的前体物质),而且与生物被膜形成所需的菌毛、粘附素的表达有关[104];含有EAL结构域的调节蛋白PvrR可以调节铜绿假单胞菌生物被膜的形成和抗生素耐药性[105];而含HD-GYP结构域的蛋白PA4108和PA4781也可以显著影响铜绿假单胞菌生物被膜的形成和结构组成[103]。c-di-GMP与其效应器(c-di-GMP受体)结合,通过变构改变其效应器的结构并激活其功能,从而发挥对基因表达、代谢酶的活性和生物被膜形成等的调节作用[100]。目前发现有4种c-di-GMP受体:PilZ、FleQ、PelD和Alg44[100]。
PilZ是一种介导蹭行运动(Twiching)的Ⅳ型菌毛形成所必须的,它被c-di-GMP结合而激活,帮助生物被膜微菌落的聚集[106]。Alg44是另一种参与褐藻多糖合成的PilZ家族蛋白,通过响应c-di-GMP而促进褐藻多糖的形成[107]。FleQ是一种结合c-di-GMP的转录因子,一般作为鞭毛合成基因的激活蛋白,在没有c-di-GMP或其它激活物质的情况下也可以作为pel操纵子转录的抑制因子[108]。PelD是铜绿假单胞菌的pel操纵子中编码的一个蛋白,它具有一个类似于GDDEF结构域蛋白中的Ⅰ位点,通过该位点与c-di-GMP结合,从而激活Pel多糖的产生[109]。虽然目前还没有发现与调节Psl多糖合成有关的c-di-GMP受体蛋白,但是有研究发现,随着c-di-GMP浓度升高而被诱导转录的基因中包括psl基因簇中的一些基因[53]。因此被c-di-GMP激活的这些效应器,不但影响着细胞附属物如鞭毛和Ⅳ型菌毛的合成,而且对铜绿假单胞菌的褐藻多糖、Pel多糖和Psl多糖等胞外多糖的合成具有调控作用(图 3),这些功能对于铜绿假单胞菌生物被膜的形成是必不可少的。
3 结论细菌生物被膜的形成是它对外界环境信号做出的极其复杂的响应过程,使用这一防御手段可帮助细菌更好地生存。通过对铜绿假单胞菌生物被膜内各成份的功能及其调控机制的研究,有助于最终找到有效治疗铜绿假单胞菌感染的方法或者对其他细菌生物被膜相关问题的研究提供指导。未来针对生物被膜的研究除了揭示其它可能的基质成分、各组分之间的相互作用及其更精细的调控网络之外,研究的重点还在于寻找更加有效的抗生物被膜药物以及开发新型的临床抗生物被膜疗法。
| [1] | Costerton JW, Stewart PS, Greenberg EP. Bacterial biofilms: a common cause of persistent infections. Science, 1999, 284(5418): 1318-1322. DOI:10.1126/science.284.5418.1318 |
| [2] | Reichhardt C, Parsek MR. Confocal laser scanning microscopy for analysis of Pseudomonas aeruginosa biofilm architecture and matrix localization. Frontiers in Microbiology, 2019, 10: 677. DOI:10.3389/fmicb.2019.00677 |
| [3] | Zhang WP, Ding W, Li YX, Tam C, Bougouffa S, Wang RJ, Pei B, Chiang H, Leung P, Lu YH, Sun J, Fu H, Bajic VB, Liu HB, Webster NS, Qian PY. Marine biofilms constitute a bank of hidden microbial diversity and functional potential. Nature Communications, 2019, 10: 517. DOI:10.1038/s41467-019-08463-z |
| [4] | Kumar A, Alam A, Rani M, Ehtesham NZ, Hasnain SE. Biofilms: survival and defense strategy for pathogens. International Journal of Medical Microbiology, 2017, 307(8): 481-489. DOI:10.1016/j.ijmm.2017.09.016 |
| [5] | Davies D. Understanding biofilm resistance to antibacterial agents. Nature Reviews Drug Discovery, 2003, 2(2): 114-122. DOI:10.1038/nrd1008 |
| [6] | Ceri H, Olson ME, Stremick C, Read RR, Morck D, Buret A. The Calgary biofilm device: new technology for rapid determination of antibiotic susceptibilities of bacterial biofilms. Journal of Clinical Microbiology, 1999, 37(6): 1771-1776. DOI:10.1128/JCM.37.6.1771-1776.1999 |
| [7] | Stewart PS, Costerton JW. Antibiotic resistance of bacteria in biofilms. The Lancet, 2001, 358(9276): 135-138. DOI:10.1016/S0140-6736(01)05321-1 |
| [8] | Azam MW, Khan AU. Updates on the pathogenicity status of Pseudomonas aeruginosa. Drug Discovery Today, 2019, 24(1): 350-359. DOI:10.1016/j.drudis.2018.07.003 |
| [9] | Huang YH, Lin JS, Ma JC, Wang HH. Functional characterization of triclosan-resistant enoyl-acyl-carrier protein reductase (FabV) in Pseudomonas aeruginosa. Frontiers in Microbiology, 2016, 7: 1903. |
| [10] | Fazeli H, Akbari R, Moghim S, Narimani T, Arabestani MR, Ghoddousi AR. Pseudomonas aeruginosa infections in patients, hospital means, and personnel's specimens. Journal of Research in Medical Sciences, 2012, 17(4): 332-337. |
| [11] | Zhang WP, Sun J, Ding W, Lin JS, Tian RM, Lu L, Liu XF, Shen XH, Qian PY. Extracellular matrix-associated proteins form an integral and dynamic system during Pseudomonas aeruginosa biofilm development. Frontiers in Cellular and Infection Microbiology, 2015, 5: 40. |
| [12] | Skariyachan S, Sridhar VS, Packirisamy S, Kumargowda ST, Challapilli SB. Recent perspectives on the molecular basis of biofilm formation by Pseudomonas aeruginosa and approaches for treatment and biofilm dispersal. Folia Microbiologica, 2018, 63(4): 413-432. DOI:10.1007/s12223-018-0585-4 |
| [13] | Bayer AS, Speert DP, Park S, Tu J, Witt M, Nast CC, Norman DC. Functional role of mucoid exopolysaccharide (alginate) in antibiotic-induced and polymorphonuclear leukocyte- mediated killing of Pseudomonas aeruginosa. Infection and Immunity, 1991, 59(1): 302-308. DOI:10.1128/IAI.59.1.302-308.1991 |
| [14] | Ma LY, Conover M, Lu HP, Parsek MR, Bayles K, Wozniak DJ. Assembly and development of the Pseudomonas aeruginosa biofilm matrix. PLoS Pathogens, 2009, 5(3): e1000354. DOI:10.1371/journal.ppat.1000354 |
| [15] | Zhao K, Tseng BS, Beckerman B, Jin F, Gibiansky ML, Harrison JJ, Luijten E, Parsek MR, Wong GCL. Psl trails guide exploration and microcolony formation in Pseudomonas aeruginosa biofilms. Nature, 2013, 497(7449): 388-391. DOI:10.1038/nature12155 |
| [16] | Seviour T, Pijuan M, Nicholson T, Keller J, Yuan ZG. Gel-forming exopolysaccharides explain basic differences between structures of aerobic sludge granules and floccular sludges. Water Research, 2009, 43(18): 4469-4478. DOI:10.1016/j.watres.2009.07.018 |
| [17] | Wu HJ, Wang D, Tang MM, Ma LZ. The advance of assembly of exopolysaccharide Psl biosynthesis machinery in Pseudomonas aeruginosa. Microbiology Open, 2019, 8(10): e857. |
| [18] | Baker P, Hill PJ, Snarr BD, Alnabelseya N, Pestrak MJ, Lee MJ, Jennings LK, Tam J, Melnyk RA, Parsek MR, Sheppard DC, Wozniak DJ, Howell PL. Exopolysaccharide biosynthetic glycoside hydrolases can be utilized to disrupt and prevent Pseudomonas aeruginosa biofilms. Science Advances, 2016, 2(5): e1501632. DOI:10.1126/sciadv.1501632 |
| [19] | Mann EE, Wozniak DJ. Pseudomonas biofilm matrix composition and niche biology. FEMS Microbiology Reviews, 2012, 36(4): 893-916. DOI:10.1111/j.1574-6976.2011.00322.x |
| [20] | Chew SC, Kundukad B, Seviour T, van der Maarel JRC, Yang L, Rice SA, Doyle P, Kjelleberg S. Dynamic remodeling of microbial biofilms by functionally distinct exopolysaccharides. mBio, 2014, 5(4): e01536-14. |
| [21] | Evans LR, Linker A. Production and characterization of the slime polysaccharide of Pseudomonas aeruginosa. Journal of Bacteriology, 1973, 116(2): 915-924. DOI:10.1128/JB.116.2.915-924.1973 |
| [22] | Osman SF, Fett WF, Fishman ML. Exopolysaccharides of the phytopathogen Pseudomonas syringae pv. glycinea. Journal of Bacteriology, 1986, 166(1): 66-71. DOI:10.1128/JB.166.1.66-71.1986 |
| [23] | Franklin MJ, Nivens DE, Weadge JT, Howell PL. Biosynthesis of the Pseudomonas aeruginosa extracellular polysaccharides, alginate, Pel, and Psl. Frontiers in Microbiology, 2011, 2: 167. |
| [24] | Liang Y, Wang HZ, Hong W, Damkiaer S, Jochumsen N, Song ZJ, Givskov M, Høiby N, Molin S. Polysaccharides serve as scaffold of biofilms formed by mucoid Pseudomonas aeruginosa. FEMS Immunology & Medical Microbiology, 2012, 65(2): 366-376. |
| [25] | May TB, Shinabarger D, Maharaj R, Kato J, Chu L, DeVault JD, Roychoudhury S, Zielinski NA, Berry A, Rothmel RK. Alginate synthesis by Pseudomonas aeruginosa: a key pathogenic factor in chronic pulmonary infections of cystic fibrosis patients. Clinical Microbiology Reviews, 1991, 4(2): 191-206. DOI:10.1128/CMR.4.2.191 |
| [26] | Govan JR, Deretic V. Microbial pathogenesis in cystic fibrosis: mucoid Pseudomonas aeruginosa and Burkholderia cepacia. Microbiological Reviews, 1996, 60(3): 539-574. DOI:10.1128/MR.60.3.539-574.1996 |
| [27] | Hay ID, Rehman ZU, Ghafoor A, Rehm BHA. Bacterial biosynthesis of alginates. Journal of Chemical Technology & Biotechnology, 2010, 85(6): 752-759. |
| [28] | Jackson KD, Starkey M, Kremer S, Parsek MR, Wozniak DJ. Identification of psl, a locus encoding a potential exopolysaccharide that is essential for Pseudomonas aeruginosa PAO1 biofilm formation. Journal of Bacteriology, 2004, 186(14): 4466-4475. DOI:10.1128/JB.186.14.4466-4475.2004 |
| [29] | Govan JR, Deretic V. Microbial pathogenesis in cystic fibrosis: mucoid Pseudomonas aeruginosa and Burkholderia cepacia. Microbiology and Molecular Biology Reviews, 1996, 60(3): 539-574. |
| [30] |
Yu S, Ma LY. Iron uptake and biofilm formation in Pseudomonas aeruginosa. Chinese Journal of Biotechnology, 2017, 33(9): 1489-1512.
(in Chinese) 于珊, 马旅雁. 铜绿假单胞菌铁摄取与生物被膜形成研究进展. 生物工程学报, 2017, 33(9): 1489-1512. |
| [31] | Limoli DH, Whitfield GB, Kitao T, Ivey ML, Davis MR Jr, Grahl N, Hogan DA, Rahme LG, Howell PL, O'Toole GA, Goldberg JB. Pseudomonas aeruginosa alginate overproduction promotes coexistence with Staphylococcus aureus in a model of cystic fibrosis respiratory infection. mBio, 2017, 8(2): e00186-17. |
| [32] | Baker P, Whitfield GB, Hill PJ, Little DJ, Pestrak MJ, Robinson H, Wozniak DJ, Howell PL. Characterization of the Pseudomonas aeruginosa glycoside hydrolase PslG reveals that its levels are critical for Psl polysaccharide biosynthesis and biofilm formation. Journal of Biological Chemistry, 2015, 290(47): 28374-28387. DOI:10.1074/jbc.M115.674929 |
| [33] | Byrd MS, Sadovskaya I, Vinogradov E, Lu HP, Sprinkle AB, Richardson SH, Ma LY, Ralston B, Parsek MR, Anderson EM, Lam JS, Wozniak DJ. Genetic and biochemical analyses of the Pseudomonas aeruginosa Psl exopolysaccharide reveal overlapping roles for polysaccharide synthesis enzymes in Psl and LPS production. Molecular Microbiology, 2009, 73(4): 622-638. DOI:10.1111/j.1365-2958.2009.06795.x |
| [34] | Wu HJ, Wang D, Tang MM, Ma LZ. The advance of assembly of exopolysaccharide Psl biosynthesis machinery in Pseudomonas aeruginosa. Microbiology Open, 2019, 8(10): e857. |
| [35] | Zhang JC, He J, Zhai CH, Ma LZ, Gu LC, Zhao K. Effects of PslG on the surface movement of Pseudomonas aeruginosa. Applied and Environmental Microbiology, 2018, 84(13): e00219-18. |
| [36] | Yang S, Cheng XY, Jin ZY, Xia AG, Ni L, Zhang RR, Jin F. Differential production of Psl in planktonic cells leads to two distinctive attachment phenotypes in Pseudomonas aeruginosa. Applied and Environmental Microbiology, 2018, 84(14): e00700-18. |
| [37] | Zegans ME, Wozniak D, Griffin E, Toutain-Kidd CM, Hammond JH, Garfoot A, Lam JS. Pseudomonas aeruginosa exopolysaccharide Psl promotes resistance to the biofilm inhibitor polysorbate 80. Antimicrobial Agents and Chemotherapy, 2012, 56(8): 4112-4122. DOI:10.1128/AAC.00373-12 |
| [38] | Akturk E, Oliveira H, Santos SB, Costa S, Kuyumcu S, Melo LDR, Azeredo J. Synergistic action of phage and antibiotics: parameters to enhance the killing efficacy against mono and dual-species biofilms. Antibiotics, 2019, 8(3): 103. DOI:10.3390/antibiotics8030103 |
| [39] | Billings N, Millan MR, Caldara M, Rusconi R, Tarasova Y, Stocker R, Ribbeck K. The extracellular matrix component Psl provides fast-acting antibiotic defense in Pseudomonas aeruginosa biofilms. PLoS Pathogens, 2013, 9(8): e1003526. DOI:10.1371/journal.ppat.1003526 |
| [40] | DiGiandomenico A, Warrener P, Hamilton M, Guillard S, Ravn P, Minter R, Camara MM, Venkatraman V, MacGill RS, Lin J, Wang Q, Keller AE, Bonnell JC, Tomich M, Jermutus L, McCarthy MP, Melnick DA, Suzich JA, Stover CK. Identification of broadly protective human antibodies to Pseudomonas aeruginosa exopolysaccharide Psl by phenotypic screening. Journal of Experimental Medicine, 2012, 209(7): 1273-1287. DOI:10.1084/jem.20120033 |
| [41] | Yu S, Su TT, Wu HJ, Liu SH, Wang D, Zhao TH, Jin ZJ, Du WB, Zhu MJ, Chua SL, Yang L, Zhu DY, Gu LC, Ma LZ. PslG, a self-produced glycosyl hydrolase, triggers biofilm disassembly by disrupting exopolysaccharide matrix. Cell Research, 2015, 25(12): 1352-1367. DOI:10.1038/cr.2015.129 |
| [42] | Vasseur P, Vallet-Gely I, Soscia C, Genin S, Filloux A. The pel genes of the Pseudomonas aeruginosa PAK strain are involved at early and late stages of biofilm formation. Microbiology, 2005, 151(3): 985-997. DOI:10.1099/mic.0.27410-0 |
| [43] | Jennings LK, Storek KM, Ledvina HE, Coulon C, Marmont LS, Sadovskaya I, Secor PR, Tseng BS, Scian M, Filloux A, Wozniak DJ, Howell PL, Parsek MR. Pel is a cationic exopolysaccharide that cross-links extracellular DNA in the Pseudomonas aeruginosa biofilm matrix. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(36): 11353-11358. DOI:10.1073/pnas.1503058112 |
| [44] | Whitfield GB, Marmont LS, Ostaszewski A, Rich JD, Whitney JC, Parsek MR, Harrison JJ, Howell PL. Pel polysaccharide biosynthesis requires an inner membrane complex comprised of PelD, PelE, PelF, and PelG. Journal of Bacteriology, 2020, 202(8): e00684-19. |
| [45] | Wei Q, Ma LZ. Biofilm matrix and its regulation in Pseudomonas aeruginosa. International Journal of Molecular Sciences, 2013, 14(10): 20983-21005. DOI:10.3390/ijms141020983 |
| [46] | Qi LJ, Christopher GF. Role of flagella, type Ⅳ pili, biosurfactants, and extracellular polymeric substance polysaccharides on the formation of pellicles by Pseudomonas aeruginosa. Langmuir, 2019, 35(15): 5294-5304. DOI:10.1021/acs.langmuir.9b00271 |
| [47] | Semmler ABT, Whitchurch CB, Mattick JS. A re-examination of twitching motility in Pseudomonas aeruginosa. Microbiology, 1999, 145(10): 286-2873. |
| [48] | Heydorn A, Ersbøll B, Kato J, Hentzer M, Parsek MR, Tolker-Nielsen T, Givskov M, Molin S. Statistical analysis of Pseudomonas aeruginosa biofilm development: impact of mutations in genes involved in twitching motility, cell-to-cell signaling, and stationary-phase sigma factor expression. Applied and Environmental Microbiology, 2002, 68(4): 2008-2017. DOI:10.1128/AEM.68.4.2008-2017.2002 |
| [49] | Lin JS, Cheng JL, Chen KQ, Guo CH, Zhang WP, Yang X, Ding W, Ma L, Wang Y, Shen XH. The icmF3 locus is involved in multiple adaptation-and virulence-related characteristics in Pseudomonas aeruginosa PAO1. Frontiers in Cellular and Infection Microbiology, 2015, 5: 70. |
| [50] | Stoodley P, Sauer K, Davies DG, Costerton JW. Biofilms as complex differentiated communities. Annual Review of Microbiology, 2002, 56: 187-209. DOI:10.1146/annurev.micro.56.012302.160705 |
| [51] | Thormann KM, Duttler S, Saville RM, Hyodo M, Shukla S, Hayakawa Y, Spormann AM. Control of formation and cellular detachment from Shewanella oneidensis MR-1 biofilms by cyclic-di-GMP. Journal of Bacteriology, 2006, 188(7): 2681-2691. DOI:10.1128/JB.188.7.2681-2691.2006 |
| [52] | Hickman JW, Tifrea DF, Harwood CS. A chemosensory system that regulates biofilm formation through modulation of cyclic diguanylate levels. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(40): 14422-14427. DOI:10.1073/pnas.0507170102 |
| [53] | Starkey M, Hickman JH, Ma LY, Zhang N, De Long S, Hinz A, Palacios S, Manoil C, Kirisits MJ, Starner TD, Wozniak DJ, Harwood CS, Parsek MR. Pseudomonas aeruginosa rugose small-colony variants have adaptations that likely promote persistence in the cystic fibrosis lung. Journal of Bacteriology, 2009, 191(11): 3492-3503. DOI:10.1128/JB.00119-09 |
| [54] | Borlee BR, Goldman AD, Murakami K, Samudrala R, Wozniak DJ, Parsek MR. Pseudomonas aeruginosa uses a cydic-di- GMP-regulated adhesin to reinforce the biofilm extracellular matrix. Molecular Microbiology, 2010, 75(4): 827-842. DOI:10.1111/j.1365-2958.2009.06991.x |
| [55] | Diggle SP, Stacey RE, Dodd C, Cámara M, Williams P, Winzer K. The galactophilic lectin, LecA, contributes to biofilm development in Pseudomonas aeruginosa. Environmental Microbiology, 2006, 8(6): 1095-1104. DOI:10.1111/j.1462-2920.2006.001001.x |
| [56] | Da Silva DP, Matwichuk ML, Townsend DO, Reichhardt C, Lamba D, Wozniak DJ, Parsek MR. The Pseudomonas aeruginosa lectin LecB binds to the exopolysaccharide Psl and stabilizes the biofilm matrix. Nature Communications, 2019, 10(1): 2183. DOI:10.1038/s41467-019-10201-4 |
| [57] | Azghani AO, Idell S, Bains M, Hancock REW. Pseudomonas aeruginosa outer membrane protein F is an adhesin in bacterial binding to lung epithelial cells in culture. Microbial Pathogenesis, 2002, 33(3): 109-114. DOI:10.1006/mpat.2002.0514 |
| [58] | Dueholm MS, Petersen SV, Sønderkær M, Larsen P, Christiansen G, Hein KL, Enghild JJ, Nielsen JL, Nielsen KL, Nielsen PH, Otzen DE. Functional amyloid in Pseudomonas. Molecular Microbiology, 2010, 77(4): 1009-1020. |
| [59] | Tseng BS, Reichhardt C, Merrihew GE, Araujo-Hernandez SA, Harrison JJ, MacCoss MJ, Parsek MR. A biofilm matrix-associated protease inhibitor protects Pseudomonas aeruginosa from proteolytic attack. mBio, 2018, 9(2): e00543-18. |
| [60] | Tahrioui A, Duchesne R, Bouffartigues E, Rodrigues S, Maillot O, Tortuel D, Hardouin J, Taupin L, Groleau MC, Dufour A, Déziel E, Brenner-Weiss G, Feuilloley M, Orange N, Lesouhaitier O, Cornelis P, Chevalier S. Extracellular DNA release, quorum sensing, and PrrF1/F2 small RNAs are key players in Pseudomonas aeruginosa tobramycin-enhanced biofilm formation. npj Biofilms and Microbiomes, 2019, 5: 15. DOI:10.1038/s41522-019-0088-3 |
| [61] | Böckelmann U, Janke A, Kuhn R, Neu TR, Wecke J, Lawrence JR, Szewzyk U. Bacterial extracellular DNA forming a defined network-like structure. FEMS Microbiology Letters, 2006, 262(1): 31-38. DOI:10.1111/j.1574-6968.2006.00361.x |
| [62] | Das T, Manefield M. Pyocyanin promotes extracellular DNA release in Pseudomonas aeruginosa. PLoS One, 2012, 7(10): e46718. DOI:10.1371/journal.pone.0046718 |
| [63] | Barnes AMT, Ballering KS, Leibman RS, Wells CL, Dunny GM. Enterococcus faecalis produces abundant extracellular structures containing DNA in the absence of cell lysis during early biofilm formation. mBio, 2012, 3(4): e00193-12. |
| [64] | Montanaro L, Poggi A, Visai L, Ravaioli S, Campoccia D, Speziale P, Arciola CR. Extracellular DNA in biofilm. International Journal of Artificial Organs, 2011, 34(9): 824-831. DOI:10.5301/ijao.5000051 |
| [65] | Das T, Ibugo AI, Klare W, Manefield M. Role of pyocyanin and extracellular DNA in facilitating Pseudomonas aeruginosa biofilm formation//Dhanasekaran D and Thajuddin N. Microbial biofilms-importance and applications. London: IntechOpen, 2016: 24–42. |
| [66] | Allesen-Holm M, Barken KB, Yang L, Klausen M, Webb JS, Kjelleberg S, Molin S, Givskov M, Tolker-Nielsen T. A characterization of DNA release in Pseudomonas aeruginosa cultures and biofilms. Molecular Microbiology, 2006, 59(4): 1114-1128. DOI:10.1111/j.1365-2958.2005.05008.x |
| [67] | Hymes SR, Randis TM, Sun TY, Ratner AJ. DNase inhibits Gardnerella vaginalis biofilms in vitro and in vivo. The Journal of Infectious Diseases, 2013, 207(10): 1491-1497. DOI:10.1093/infdis/jit047 |
| [68] | Parks QM, Young RL, Poch KR, Malcolm KC, Vasil ML, Nick JA. Neutrophil enhancement of Pseudomonas aeruginosa biofilm development: Human F-actin and DNA as targets for therapy. Journal of Medical Microbiology, 2009, 58(4): 492-502. DOI:10.1099/jmm.0.005728-0 |
| [69] | Das T, Kutty SK, Tavallaie R, Ibugo AI, Panchompoo J, Sehar S, Aldous L, Yeung AWS, Thomas SR, Kumar N, Gooding JJ, Manefield M. Phenazine virulence factor binding to extracellular DNA is important for Pseudomonas aeruginosa biofilm formation. Scientific Reports, 2015, 5: 8398. DOI:10.1038/srep08398 |
| [70] | Chiang WC, Nilsson M, Jensen PØ, Høiby N, Nielsen TE, Givskov M, Tolker-Nielsen T. Extracellular DNA shields against aminoglycosides in Pseudomonas aeruginosa biofilms. Antimicrobial Agents and Chemotherapy, 2013, 57(5): 2352-2361. DOI:10.1128/AAC.00001-13 |
| [71] | Das T, Sehar S, Manefield M. The roles of extracellular DNA in the structural integrity of extracellular polymeric substance and bacterial biofilm development. Environmental Microbiology Reports, 2013, 5(6): 778-786. DOI:10.1111/1758-2229.12085 |
| [72] | Das T, Sehar S, Koop L, Wong YK, Ahmed S, Siddiqui KS, Manefield M. Influence of calcium in extracellular DNA mediated bacterial aggregation and biofilm formation. PLoS One, 2014, 9(3): e91935. DOI:10.1371/journal.pone.0091935 |
| [73] | Wilton M, Wong MJQ, Tang L, Liang XY, Moore R, Parkins MD, Lewenza S, Dong TG. Chelation of membrane-bound cations by extracellular DNA activates the type Ⅵ secretion system in Pseudomonas aeruginosa. Infection and Immunity, 2016, 84(8): 2355-2361. DOI:10.1128/IAI.00233-16 |
| [74] | Wang SW, Liu X, Liu HS, Zhang L, Guo Y, Yu S, Wozniak DJ, Ma LZ. The exopolysaccharide Psl-eDNA interaction enables the formation of a biofilm skeleton in Pseudomonas aeruginosa. Environmental Microbiology Reports, 2015, 7(2): 330-340. DOI:10.1111/1758-2229.12252 |
| [75] | Yong VFL, Soh MM, Jaggi TK, Mac Aogáin M, Chotirmall SH. The microbial endocrinology of Pseudomonas aeruginosa: inflammatory and immune perspectives. Archivum Immunologiae et Therapiae Experimentalis, 2018, 66(5): 329-339. DOI:10.1007/s00005-018-0510-1 |
| [76] | Rumbaugh KP, Griswold JA, Hamood AN. The role of quorum sensing in the in vivo virulence of Pseudomonas aeruginosa. Microbes and Infection, 2000, 2(14): 1721-1731. DOI:10.1016/S1286-4579(00)01327-7 |
| [77] | Holden MTG, Chhabra SR, De Nys R, Stead P, Bainton NJ, Hill PJ, Manefield M, Kumar N, Labatte M, England D, Rice S, Givskov M, Salmond GPC, Stewart GSAB, Bycroft BW, Kjelleberg S, Williams P. Quorum-sensing cross talk: isolation and chemical characterization of cyclic dipeptides from Pseudomonas aeruginosa and other gram-negative bacteria. Molecular Microbiology, 1999, 33(6): 1254-1266. |
| [78] | Das T, Manoharan A, Whiteley G, Glasbey T, Manos J. Pseudomonas aeruginosa biofilms and infections: roles of extracellular molecules//Yadav MK, Singh BP. New and future developments in microbial biotechnology and bioengineering: microbial biofilms. Amsterdam: Elsevier, 2020: 29–46. |
| [79] | Lin JS, Cheng JL, Wang Y, Shen XH. The Pseudomonas quinolone signal (PQS): not just for quorum sensing anymore. Frontiers in Cellular and Infection Microbiology, 2018, 8: 230. DOI:10.3389/fcimb.2018.00230 |
| [80] | Lin JS, Cheng JL. Quorum sensing in Pseudomonas aeruginosa and its relationship to biofilm development// Rathinam NK, Sani RK. Introduction to biofilm engineering. Washington: American Chemical Society, 2019: 1–16. |
| [81] | Huang H, Shao XL, Xie YP, Wang TT, Zhang YC, Wang X, Deng X. An integrated genomic regulatory network of virulence-related transcriptional factors in Pseudomonas aeruginosa. Nature Communications, 2019, 10(1): 2931. DOI:10.1038/s41467-019-10778-w |
| [82] | Duan K, Surette MG. Environmental regulation of Pseudomonas aeruginosa PAO1 Las and Rhl quorum-sensing systems. Journal of Bacteriology, 2007, 189(13): 4827-4836. DOI:10.1128/JB.00043-07 |
| [83] | Hentzer M, Riedel K, Rasmussen TB, Heydorn A, Bo Andersen J, Parsek MR, Rice SA, Eberl L, Molin S, Høiby N, Kjelleberg S, Givskov M. Inhibition of quorum sensing in Pseudomonas aeruginosa biofilm bacteria by a halogenated furanone compound. Microbiology, 2002, 148(1): 87-102. DOI:10.1099/00221287-148-1-87 |
| [84] | Janssen KH, Diaz MR, Golden M, Graham JW, Sanders W, Wolfgang MC, Yahr TL. Functional analyses of the RsmY and RsmZ small noncoding regulatory RNAs in Pseudomonas aeruginosa. Journal of Bacteriology, 2018, 200(11): e00736-17. |
| [85] | Chrzanowski Ł, Lawniczak Ł, Czaczyk K. Why do microorganisms produce rhamnolipids?. World Journal of Microbiology and Biotechnology, 2012, 28(2): 401-419. DOI:10.1007/s11274-011-0854-8 |
| [86] | Ciofu O, Tolker-Nielsen T, Jensen PØ, Wang HZ, Høiby N. Antimicrobial resistance, respiratory tract infections and role of biofilms in lung infections in cystic fibrosis patients. Advanced Drug Delivery Reviews, 2015, 85: 7-23. DOI:10.1016/j.addr.2014.11.017 |
| [87] | Mukherjee S, Moustafa D, Smith CD, Goldberg JB, Bassler BL. The RhlR quorum-sensing receptor controls Pseudomonas aeruginosa pathogenesis and biofilm development independently of its canonical homoserine lactone autoinducer. PLoS Pathogens, 2017, 13(7): e1006504. DOI:10.1371/journal.ppat.1006504 |
| [88] | Mukherjee S, Moustafa DA, Stergioula V, Smith CD, Goldberg JB, Bassler BL. The PqsE and RhlR proteins are an autoinducer synthase-receptor pair that control virulence and biofilm development in Pseudomonas aeruginosa. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(40): e9411-9418. DOI:10.1073/pnas.1814023115 |
| [89] | Rampioni G, Falcone M, Heeb S, Frangipani E, Fletcher MP, Dubern JF, Visca P, Leoni L, Cámara M, Williams P. Unravelling the genome-wide contributions of specific 2-Alkyl-4-Quinolones and PqsE to quorum sensing in Pseudomonas aeruginosa. PLoS Pathogens, 2016, 12(11): e1006029. DOI:10.1371/journal.ppat.1006029 |
| [90] | Bala A, Kumar L, Chhibber S, Harjai K. Augmentation of virulence related traits of pqs mutants by Pseudomonas quinolone signal through membrane vesicles. Journal of Basic Microbiology, 2015, 55(5): 566-578. DOI:10.1002/jobm.201400377 |
| [91] | Diggle SP, Winzer K, Chhabra SR, Worrall KE, Cámara M, Williams P. The Pseudomonas aeruginosa quinolone signal molecule overcomes the cell density-dependency of the quorum sensing hierarchy, regulates rhl-dependent genes at the onset of stationary phase and can be produced in the absence of LasR. Molecular Microbiology, 2003, 50(1): 29-43. DOI:10.1046/j.1365-2958.2003.03672.x |
| [92] | Yang L, Nilsson M, Gjermansen M, Givskov M, Tolker-Nielsen T. Pyoverdine and PQS mediated subpopulation interactions involved in Pseudomonas aeruginosa biofilm formation. Molecular Microbiology, 2009, 74(6): 1380-1392. DOI:10.1111/j.1365-2958.2009.06934.x |
| [93] | Häussler S, Becker T. The pseudomonas quinolone signal (PQS) balances life and death in Pseudomonas aeruginosa populations. PLoS Pathogens, 2008, 4(9): e1000166. DOI:10.1371/journal.ppat.1000166 |
| [94] | Lin JS, Zhang WP, Cheng JL, Yang X, Zhu KX, Wang Y, Wei GH, Qian PY, Luo ZQ, Shen XH. A Pseudomonas T6SS effector recruits PQS-containing outer membrane vesicles for iron acquisition. Nature Communications, 2017, 8(1): 14888. DOI:10.1038/ncomms14888 |
| [95] | Lee J, Wu JE, Deng YY, Wang J, Wang C, Wang JH, Chang CQ, Dong YH, Williams P, Zhang LH. A cell-cell communication signal integrates quorum sensing and stress response. Nature Chemical Biology, 2013, 9(5): 339-343. DOI:10.1038/nchembio.1225 |
| [96] | Long J, Zaborina O, Holbrook C, Zaborin A, Alverdy J. Depletion of intestinal phosphate after operative injury activates the virulence of P aeruginosa causing lethal gut-derived sepsis. Surgery, 2008, 144(2): 189-197. DOI:10.1016/j.surg.2008.03.045 |
| [97] | Li SG, Chen SY, Fan JJ, Cao Z, Ouyang WH, Tong N, Hu X, Hu J, Li PS, Feng ZF, Huang X, Li YY, Xie MS, He RK, Jian JY, Wu BY, Xu C, Wu WJ, Guo JL, Lin J, Sun PH. Anti-biofilm effect of novel thiazole acid analogs against Pseudomonas aeruginosa through IQS pathways. European Journal of Medicinal Chemistry, 2018, 145: 64-73. DOI:10.1016/j.ejmech.2017.12.076 |
| [98] | Guła G, Dorotkiewicz-Jach A, Korzekwa K, Valvano MA, Drulis-Kawa Z. Complex signaling networks controlling dynamic molecular changes in Pseudomonas aeruginosa biofilm. Current Medicinal Chemistry, 2019, 26(11): 1979-1993. DOI:10.2174/0929867325666180912110151 |
| [99] | Tischler AD, Camilli A. Cyclic diguanylate (c-di-GMP) regulates Vibrio cholerae biofilm formation. Molecular Microbiology, 2004, 53(3): 857-869. DOI:10.1111/j.1365-2958.2004.04155.x |
| [100] | Hengge R. Principles of c-di-GMP signalling in bacteria. Nature Reviews Microbiology, 2009, 7(4): 263-273. DOI:10.1038/nrmicro2109 |
| [101] | Jenal U, Malone J. Mechanisms of cyclic-di-GMP signaling in bacteria. Annual Review of Genetics, 2006, 40(1): 385-407. DOI:10.1146/annurev.genet.40.110405.090423 |
| [102] | Kulesekara H, Lee V, Brencic A, Liberati N, Urbach J, Miyata S, Lee DG, Neely AN, Hyodo M, Hayakawa Y, Ausubel FM, Lory S. Analysis of Pseudomonas aeruginosa diguanylate cyclases and phosphodiesterases reveals a role for bis-(3'-5')-cyclic-GMP in virulence. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(8): 2839-2844. DOI:10.1073/pnas.0511090103 |
| [103] | Ryan RP, Lucey J, O'Donovan K, McCarthy Y, Yang L, Tolker-Nielsen T, Dow JM. HD-GYP domain proteins regulate biofilm formation and virulence in Pseudomonas aeruginosa. Environmental Microbiology, 2009, 11(5): 1126-1136. DOI:10.1111/j.1462-2920.2008.01842.x |
| [104] | D'Argenio DA, Calfee MW, Rainey PB, Pesci EC. Autolysis and autoaggregation in Pseudomonas aeruginosa colony morphology mutants. Journal of Bacteriology, 2002, 184(23): 6481-6489. DOI:10.1128/JB.184.23.6481-6489.2002 |
| [105] | Drenkard E, Ausubel FM. Pseudomonas biofilm formation and antibiotic resistance are linked to phenotypic variation. Nature, 2002, 416(6882): 740-743. DOI:10.1038/416740a |
| [106] | Alm RA, Bodero AJ, Free PD, Mattick JS. Identification of a novel gene, pilZ, essential for type 4 fimbrial biogenesis in Pseudomonas aeruginosa. Journal of Bacteriology, 1996, 178(1): 46-53. DOI:10.1128/JB.178.1.46-53.1996 |
| [107] | Remminghorst U, Rehm BHA. Alg44, a unique protein required for alginate biosynthesis in Pseudomonas aeruginosa. FEBS Letters, 2006, 580(16): 3883-3888. DOI:10.1016/j.febslet.2006.05.077 |
| [108] | Hickman JW, Harwood CS. Identification of FleQ from Pseudomonas aeruginosa as a c-di-GMP-responsive transcription factor. Molecular Microbiology, 2008, 69(2): 376-389. DOI:10.1111/j.1365-2958.2008.06281.x |
| [109] | Lee VT, Matewish JM, Kessler JL, Hyodo M, Hayakawa Y, Lory S. A cyclic-di-GMP receptor required for bacterial exopolysaccharide production. Molecular Microbiology, 2007, 65(6): 1474-1484. DOI:10.1111/j.1365-2958.2007.05879.x |
 2021, Vol. 61
2021, Vol. 61