中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 刘军委, 张璇, 鲍艺萱, 徐鉴昳, 邱吉国, 何健. 2021
- Junwei Liu, Xuan Zhang, Yixuan Bao, Jianyi Xu, Jiguo Qiu, Jian He. 2021
- 厌氧丁草胺降解菌BAD-20的分离鉴定及降解特性研究
- Isolation, identification and degradation characteristics of an anaerobic butachlor-degrading bacterium BAD-20
- 微生物学报, 61(4): 1002-1015
- Acta Microbiologica Sinica, 61(4): 1002-1015
-
文章历史
- 收稿日期:2020-10-16
- 修回日期:2020-12-16
- 网络出版日期:2021-02-23
丁草胺[2-氯-N-(2, 6-二乙基苯基)-N-(丁氧甲基)乙酰胺,butachlor]是氯乙酰胺类除草剂中的主要品种,是我国用量最大的除草剂之一[1]。丁草胺作为选择性芽前除草剂,在直播或移栽水稻田中广泛应用,防除一年生禾本科杂草及某些阔叶杂草[1-3]。丁草胺具有潜在的致畸变和致突变性,被美国环保局定为B-2类致癌物,丁草胺会导致心率紊乱、抽搐、昏迷和呼吸停止[4]。丁草胺对鱼类有很强的毒性,比对哺乳动物的毒性大500–10000倍,会严重危害污染水体中的渔业资源[5]。丁草胺对土壤微生物有明显的抑制作用,显著降低土壤微生物多样性[6-8]。此外,丁草胺使用不当会对农作物产生严重药害,特别是在有机质含量较低的砂质土壤,或者用药量过大、施药后遇持续低温高湿天气时,会严重影响作物的生长[9-12]。因此研究丁草胺在环境中的转化和降解具有重要的意义。
氯乙酰胺类除草剂在土壤中可以通过微生物降解、光解和化学降解而消失,其中微生物降解代谢是土壤中该类除草剂消失的主要因素[13-14]。目前,国内外对氯乙酰胺类除草剂好氧微生物降解的研究比较深入透彻,分离到多株能够好氧降解丁草胺的真菌、放线菌和细菌菌株[15-25]。在降解代谢过程和机制方面,以本课题组对分离到的Sphingomonas sp. DC-6研究最为深入,其代谢氯乙酰胺除草剂的过程和参与的酶和基因已经清楚。甲草胺、乙草胺或丁草胺首先在加氧酶CndABC催化下发生N-脱烷基,脱烷基产物接着在酰胺酶CmeH催化下脱乙酰基生产苯胺衍生物2, 6-二乙基苯胺(DEA)或2-甲基-6-乙基苯胺(MEA),然后DEA或MEA在单加氧酶MeaXY和MeaAB催化下接连在苯环上2次羟基化,然后苯环打开进入TCA循环直至完全降解[26-28]。
此外,在厌氧条件下氯乙酰胺类除草剂也可以被厌氧微生物降解[29-32],叶央芳等[33]利用农药厂厌氧污泥作为接种物富集驯化到一个高效降解丁草胺的富集菌群。最近,本课题组通过富集驯化,利用农药厂污泥作为接种物在厌氧反应器中驯化出能够高效降解乙草胺、丁草胺和甲草胺的厌氧污泥,并通过代谢产物鉴定推测了乙草胺厌氧降解途径[34]。但到目前为止,国内外还没有分离到能够降解氯乙酰胺类除草剂的厌氧菌株,这严重制约了对该类除草剂厌氧降解代谢过程及其遗传生化机制的深入研究。
丁草胺主要应用于水稻田杂草防除,而在水稻种植过程中农田有一段很长的淹水期,而淹水导致水稻田深层土壤处于厌氧状态。因此研究水稻田土壤中厌氧微生物对丁草胺的降解,对于阐明丁草胺在水稻田中的转化降解及其生物学机制具有重要意义。本研究利用水稻田土壤作为接种物,通过富集驯化获得一个高效降解丁草胺的富集液,并从中筛选到一株高效的丁草胺厌氧降解菌BAD-20,研究了该菌的分类地位和对丁草胺的降解特性,利用液相色谱飞行时间质谱联用技术鉴定了丁草胺厌氧降解代谢产物。
1 材料和方法 1.1 材料及培养基丁草胺(纯度97%)、甲草胺(纯度98%)、乙草胺(纯度96%)和丙草胺(纯度96%)购买自百灵威公司(中国,上海)。
厌氧富集驯化培养基(MSM,g/L)[34]:K2HPO4·3H2O 0.35,KH2PO4 0.27,FeSO4·7H2O 0.2,MgCl2·6H2O 0.10,NH4Cl 0.53,CaCl2·2H2O 0.073,柠檬酸钠0.10,丙酮酸钠0.10,刃天青0.001,微量元素复合液1 mL,维生素复合液1 mL,用磷酸盐缓冲液调pH为7.2。
厌氧分离和培养的培养基(PYT,g/L):蛋白胨0.5,酵母浸粉1.0,胰蛋白胨0.5,酸水解酪蛋白1.0,可溶性淀粉1.0,葡萄糖1.0,K2HPO4 0.6,MgSO4 0.2,丙酮酸钠0.6,微量元素复合液1 mL,维生素复合液1 mL,过滤除菌,灭菌后加入,用磷酸盐缓冲液调pH为7.2。固体培养基加入1.7%的琼脂。
培养基除氧:将配置好的培养基煮沸,趁热将30 mL培养基分装于50 mL血清瓶中,塞住胶塞并用铝帽固定,充入N2 30 min,115 ℃高压蒸汽灭菌30 min。灭菌后的培养基应保持无色,如果颜色变为微红色或红色,则说明氧气没有除尽。
除草剂母液(10 g/L):分别称取0.5155 g丁草胺、0.5102 g甲草胺、0.5208 g乙草胺或0.5208 g丙草胺溶于50 mL甲醇中,过滤除菌4 ℃保存备用。
1.2 丁草胺厌氧降解菌的富集分离用来驯化丁草胺厌氧降解菌群的土壤样品采自江苏省南京市江苏省农业科学院水稻试验地(E32°01″N118°52″)。采样时,挖取3–10 cm土壤作为样品,装于无菌塑料袋带回实验室备用。称取采集的土壤样品3 g加入到30 mL含有20 mg/L丁草胺的无氧MSM培养基的血清瓶中,用丁基胶塞封口,然后将血清瓶放入30 ℃恒温摇床150 r/min振荡培养,培养过程中注意观察血清瓶中培养液颜色应保持无色,如果培养基颜色变红则说明血清瓶中混入了氧气。定时取样测定丁草胺的降解效果,当血清瓶中的丁草胺大约降解60%–70%后,取3 mL富集液转接到另一个新鲜的丁草胺MSM培养基的血清瓶中。如此反复转接,直到富集液能够在2周内降解90%的20 mg/L丁草胺时驯化结束。
富集液中丁草胺降解菌的分离在厌氧操作箱(COY-7000220A,COY Laboratory Products Inc.,Michigan,USA)中进行,将富集液梯度稀释后涂布于PYT固体培养基平板上,然后将平板放入厌氧罐中,置于培养箱30 ℃恒温培养7 d,培养过程中注意观察培养基颜色变化,培养基颜色应保持无色状态。挑取平板上的菌落在厌氧培养箱中进一步分离纯化,将纯化的菌落接种到含有20 mg/L丁草胺的MSM培养基中,定时取样测定其降解丁草胺的能力。
1.3 丁草胺厌氧降解菌BAD-20的鉴定16S rRNA基因序列分析及系统进化树构建形态鉴定:将菌株BAD-20划线于PYT固体平板上,于厌氧罐中30 ℃恒温培养3 d,观察菌落形态;将平板上的菌体用磷酸盐缓冲液重悬,用1%锇酸固定后,2%的磷钨酸钠负染,用透射电子显微镜(日立H-7650,日本日立)观察。
碳源利用:分别向去除碳源的MSM培养基(去除丙酮酸钠和柠檬酸钠)中加入酵母膏、蛋白胨、甘氨酸、精氨酸、丝氨酸、苏氨酸、甲酸、乙酸、丙酮酸或乳酸作为营养物质,接种BAD-20菌体,于30 ℃、150 r/min条件下培养,定期观察BAD-20是否生长。
分子生物学鉴定:利用高盐法提取分离菌株的总DNA,利用细菌16S rRNA基因的通用引物对27F (5′-AGAGTTTGATCCTGGCTCAG-3′)和1492R (5′-TACGGCTACCTTGTTACGACTT-3′)[20]进行PCR扩增。PCR体系如下(50 μL):2×Phanta Master Mix (诺维赞公司) 25 μL,模板(40 ng/µL) 1 μL,27F (20 μmol/L) 2 μL,1492R (20 μmol/L) 2 μL,加ddH2O补至50 μL。扩增条件为:95 ℃ 3 min;95 ℃ 15 s,56 ℃ 30 s,72 ℃ 90 s,30个循环;72 ℃ 7 min。PCR产物纯化后,收集1500 bp左右的片段与pMD18-T载体用DNA连接酶连接,然后用热激法转入到大肠杆菌DH5α内,利用含有氨苄青霉素(100 μg/mL)的LB平板筛选阳性克隆子,将阳性克隆子送至南京擎科生物科技有限公司测序。测得的序列在EzBioCloud database (www.ezbiocloud.net)数据库与已报到模式菌株的16S rRNA基因进行同源性分析,选取与其同源性较高的模式菌株相关序列,利用Clustal W软件对齐后,利用MEGA 6.0软件,通过邻接法构建抗性菌株的系统进化树,采用Kimura’s two-parameter model计算遗传距离,Bootstrap值设定为1000[20]。
1.4 影响分离菌株降解丁草胺的各种因素将菌株BAD-20接种于PYT液体培养基中,30 ℃恒温摇床150 r/min厌氧培养至对数生长期后期(OD600约为0.3)作为种子液。将种子液按5%的接种量接种于血清瓶中加入20 mg/L丁草胺的MSM培养基中,于30 ℃、150 r/min摇床内培养10 d。考察不同温度对降解的影响时,设置16、20、25、30、35、40和45 ℃;考察不同pH对降解的影响时,利用缓冲体系50 mmol/L NaH2PO4-Na2HPO4调节不同的pH范围(5.0–9.0)。考察不同盐浓度对降解的影响时,利用NaCl调整培养基盐浓度(0%、0.2%、0.5%、1.0%、1.5%、2.0%、3.0%和5.0%,W/V)。考察氧气对降解的影响时,将血清瓶的橡胶塞换成8层纱布使培养基通入氧气。考察不同底物浓度影响时,丁草胺的浓度分别为10、50、100 mg/L。以上实验分别设置不加菌株BAD-20的空白对照组,处理组和对照组均设置3个重复。
1.5 菌株BAD-20底物谱的测定将培养至对数期后(OD600约为0.3)的菌株BAD-20种子液按5%的接种量接种于30 mL MSM的血清瓶中,然后分别加入各种氯乙酰胺除草剂至终浓度为20 mg/L,于30 ℃、150 r/min摇床内培养,定期取样测定培养液中各种氯乙酰胺除草剂的浓度。
1.6 氯乙酰胺除草剂浓度检测方法及代谢产物鉴定在丁草胺初始浓度为50 mg/L的MSM液体培养基中,加入已制备好的BAD-20种子液1.5 mL (OD600=0.3),使其培养体系为30 mL,于30 ℃、150 r/min条件下培养。定时取样测定丁草胺的浓度,待丁草胺的浓度降解达到80%时,将降解液冷冻干燥,加2 mL甲醇,超声溶解,过0.22 μm有机滤膜。丁草胺(或甲草胺、乙草胺和丙草胺)的检测条件:超高效液相色谱(U3000,Thermo Fisher Scientific,美国);Thermo Fisher Syncronis C18 (5 μm,4.6 mm×250 mm);流动相:乙腈︰水= 90︰10 (V/V);检测波长:215 nm;流速:1.0 mL/min;柱温:40 ℃;进样量:20 μL。
丁草胺代谢产物液相色谱-时间飞行质谱(LC-TOF-MS)的测定条件:LC-20AD(日本岛津),AB Sciex TripleTOF® 5600 (美国AB SCIEX),Phenomenex Kinetex C18色谱柱(2.1 mm×100 mm,2.6 μm);柱温:40 ℃;进样量:10 μL;流速为0.3 mL/min;流动相A为水(0.1%甲酸,V/V),B为甲醇;梯度洗脱程序为:5%甲醇保持0.5 min,甲醇从5%增加到95%用时11 min,然后95%甲醇保持2 min。采用电喷雾离子源(ESI)正离子扫描模式,离子喷雾电压(ISVF) 5500 V,离子源温度(TEM) 550 ℃,雾化气(GS1) 65 psi,辅助气(GS2) 65 psi,气帘气(CUR) 35 psi;质谱数据采集模式为高分辨飞行时间质谱全扫描并同时触发采集高分辨二级质谱(TOF MS IDA MS-MS)模式,TOF-MS扫描范围m/z 50–800,IDA MS-MS扫描范围m/z 50–800,去簇电压(DP) 80 V,MS-MS碰撞能量(CE) 40 eV,扩展碰撞能量(CES) 20 eV。
1.7 数据处理与统计分析降解率计算公式如公式1所示,Cck表示空白对照底物的浓度,Ct表示处理组降解后底物的浓度;一级动力学方程如公式2所示,C0表示初始底物浓度,k表示一级反应速率常数,t表示培养时间;降解半衰期(T1/2):T1/2=(ln2)/k,其含义为底物降解一半所需要的时间。实验中所有数值为每组3个平行的平均值,对不同处理差异显著性的比较采用SPSS 19.0统计学软件进行Duncan检验,P < 0.05为具有统计学差异。

|
公式(1) |
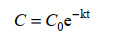
|
公式(2) |
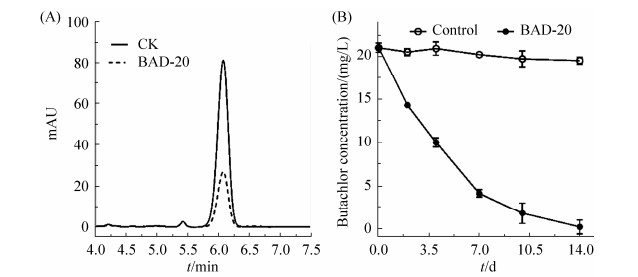
以丁草胺为碳源作为选择压力,以水稻土壤为接种物,富集驯化获得一个丁草胺降解富集液。该富集液能够在2周内降解90%的丁草胺。利用厌氧分离技术,从该富集液中分离纯化到25株厌氧细菌,分别编号为BAD-01到BAD-25。降解试验结果表明菌株BAD-20对丁草胺有很好的降解效果(图 1),在接种菌株BAD-20的培养液中丁草胺吸收峰显著下降(图 1-A),而且降解率随着培养时间延长而增加,在10 d内能够降解90%以上的丁草胺(20 mg/L)(图 1-B)。而其他分离菌株对丁草胺基本没有降解效果(数据未展示)。
|
| 图 1 菌株BAD-20对丁草胺的厌氧降解效果 Figure 1 The anaerobic degradation of butachlor by strain BAD-20. A: HPLC chromatograms of butachlor degradation by strain BAD-20; B: the time-course curve of anaerobic butachlor degradation by strain BAD-20. |
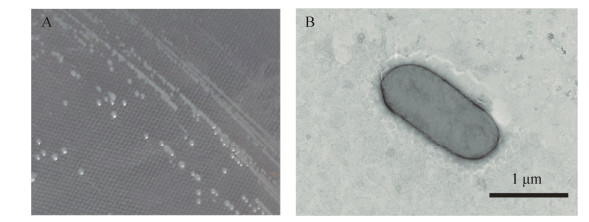
菌株BAD-20在PYT固体培养基30 ℃培养3 d菌落为白色、不透明、凸起、边缘光滑(图 2-A)。用透射电子显微镜观察,BAD-20菌体为杆状,大小为(0.5–0.8) μm×(1.5–1.8) μm,未观察到鞭毛(图 2-B),不能运动,革兰氏染色阴性。菌株BAD-20能利用酵母膏、蛋白胨、甘氨酸、精氨酸、丝氨酸和苏氨酸等蛋白氨基酸类物质生长,能利用甲酸、乙酸、丙酮酸和乳酸作为碳源。
|
| 图 2 菌株BAD-20的菌体形态(A)和透射电镜照片(B) Figure 2 Photograph (A) and transmission electron micrograph (Bar, 1.0 µm) (B) of strain BAD-20. |
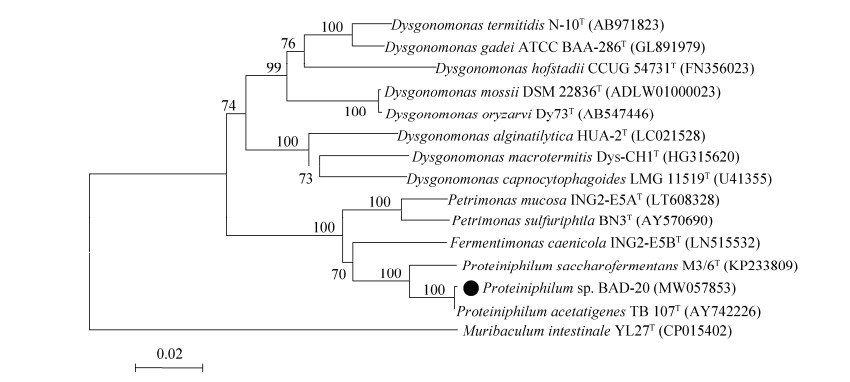
利用细菌16S rRNA基因通用引物扩增该菌16S rRNA基因,测序结果表明BAD-20的16S rRNA基因长度为1492 bp (NCBI的GenBank登录号为MW057853),和Proteiniphilum acetatigenes TB 107T具有99%的一致性,与其形成一个分支,置信度为100% (图 3)。因此,初步将菌株BAD-20鉴定为嗜蛋白属(Proteiniphilum sp.)。
|
| 图 3 菌株BAD-20的16S rRNA基因序列系统发育树 Figure 3 Phylogenetic tree of strain BAD-20 based on 16S rRNA gene sequences. Numbers in parentheses represent the sequences accession number in GenBank; numbers at the nodes indicate the bootstrap values on Neighbor-Joining analysis of 1000 resampled data sets. Bar 0.02 represent sequence divergence. |
2.2 菌株BAD-20降解丁草胺的影响因素
菌株BAD-20降解丁草胺的最适温度为30–35 ℃,10 d对丁草胺降解率达到90%以上;当温度高于35 ℃或低于20 ℃时,对丁草胺的降解速率明显在下降(图 4-A)。菌株BAD-20降解丁草胺的最适pH为7.5–8.0,10 d对丁草胺降解率达到80%以上;当pH高于8.0或低于6.5时,菌株BAD-20对丁草胺的降解速率明显下降(图 4-B)。菌株BAD-20降解丁草胺的最适盐浓度为0–0.5% NaCl,当NaCl浓度超过0.5%,丁草胺的降解效率明显受到抑制(图 4-C)。在有氧条件下,菌株BAD-20完全失去了丁草胺降解能力(图 4-D),说明菌株BAD-20为严格厌氧菌。

|
| 图 4 环境因素对菌株BAD-20降解丁草胺的影响 Figure 4 Effect of environmental factors on degradation of butachlor by strain BAD-20. A: temperature; B: pH; C: NaCl concentration; D: oxygen. All values represent the mean of triplicate samples. Different letters indicate signifificant differences in the degradation of butachlor with respect to temperature (A), pH (B), NaCl concentration (C), and oxygen (D) after incubation for 10 days as determined by one-way AVONA on (P < 0.05). |
图 5结果表明,菌株BAD-20能高效降解高达50 mg/L的丁草胺,而当培养基中丁草胺的起始浓度超过100 mg/L时,菌株BAD-20对丁草胺的降解效率下降,表明高浓度的丁草胺对菌株BAD-20有较强的抑制作用。菌株BAD-20对不同浓度丁草胺的降解动态用一级动力学方程拟合,结果如表 1所示,BAD-20对10 mg/L、50 mg/L和100 mg/L丁草胺的降解半衰期(T1/2)分别为2.49 d、6.78 d和23.17 d,不同初始浓度之间的降解半衰期存在明显的差异(P < 0.05)。

|
| 图 5 丁草胺初始浓度对菌株BAD-20降解的影响 Figure 5 Effect of initial butachlor concentration on its degradation by strain BAD-20. All values represent the mean of triplicate samples. |
| Initial concentration/(mg/L) | Kinetic equation | Correlation coefficient (R2 a) | Rate constant K/[mg/(L·d)] | Half life (T1/2b/d) |
| 10 | C=10.76 e–0.2785t | 0.9880 | 0.2785 | 2.49 a |
| 50 | C=51.01 e–0.1022t | 0.9923 | 0.1022 | 6.78 b |
| 100 | C=100.03 e–0.0299t | 0.9732 | 0.0299 | 23.17 c |
| a: Correlation coefficient, which represents the fitness of the modeling data; b: Duncan test was used to test the difference between the data. Different letters after the same column of data indicated the significant difference between the treatments (P < 0.05). | ||||
2.3 菌株BAD-20对其他主要氯乙酰胺类除草剂的降解
如表 2所示,菌株BAD-20除能降解丁草胺外,还能降解甲草胺、乙草胺和丙草胺等主要氯乙酰胺类除草剂。菌株BAD-20对甲草胺、乙草胺、丙草胺和丁草胺的降解半衰期(T1/2)分别为1.34、2.05、2.78和3.20 d。差异显著性分析表明,菌株对氯乙酰胺类除草剂的降解效率从高到低依次为甲草胺 > 乙草胺 > 丙草胺 > 丁草胺。
| Compoundsa | Structure | Correlation coefficient (R2 b) | Rate constant K/[mg/(L·d)] | Half life (T1/2c/d) |
| Alachlor | 
|
0.9981 | 0.5172 | 1.34 a |
| Acetochlor | 
|
0.9889 | 0.3381 | 2.05 b |
| Pretilachlor | 
|
0.9724 | 0.2493 | 2.78 c |
| Butachlor | 
|
0.9923 | 0.2167 | 3.20 d |
| a: the initial concentration of substrate is 20 mg/L; b: correlation coefficient, which represents the fitness of the modeling data; c: duncan test was used to test the difference between the data. Different letters after the same column of data indicated the significant difference between the treatments (P < 0.05). | ||||
2.4 菌株BAD-20降解丁草胺的代谢产物鉴定
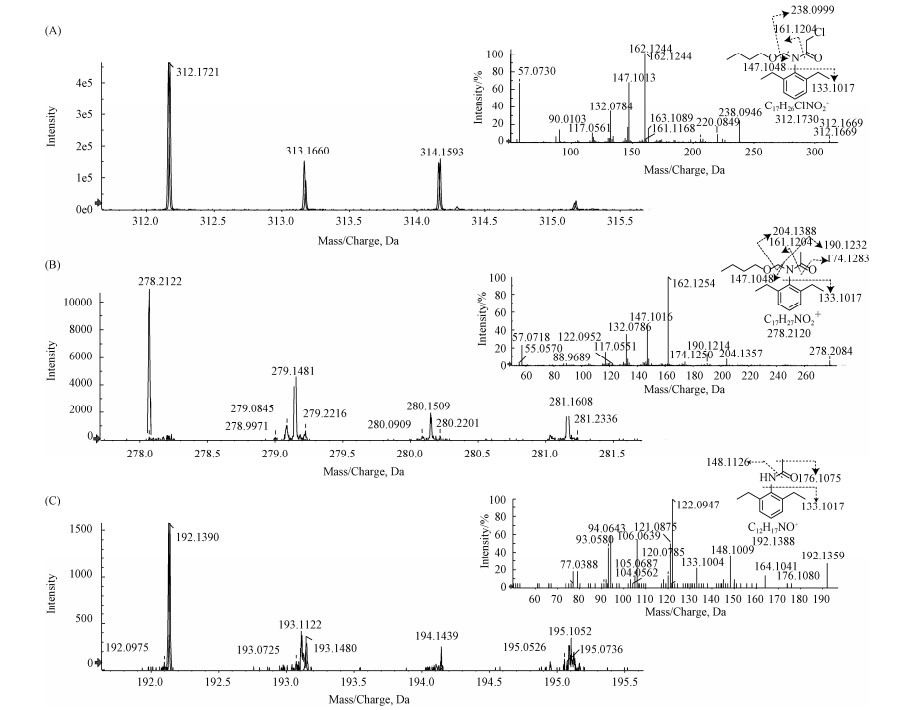
利用LC-TOF-MS检测了菌株BAD-20降解丁草胺的中间代谢产物。结果如图 6所示,在培养液高速离心上清中一共检测3个物质,其中Compound Ⅰ的荷质比(m/z)为312,和丁草胺分子量相同,几个碎片峰荷质比也和丁草胺的分子结构相符合(图 6-A),因此Compound Ⅰ鉴定为底物丁草胺。Compound Ⅱ的荷质比为278,刚好和丁草胺脱去一个氯的产物N-(2, 6-二乙基苯基)-N-(丁氧甲基)乙酰胺(DEPBMA)分子量相同,其几个碎片峰荷质比理论上也符合该产物分子结构(图 6-B),因此Compound Ⅱ鉴定为N-(2, 6-二乙基苯基)-N-(丁氧甲基)乙酰胺(DEBMA)。Compound Ⅲ荷质比为192,刚好和DEBMA脱去N-丁氧甲基的产物分子量相同,其几个碎片峰也与脱该脱N-丁氧甲基产物分子结构相同(图 6-C),因此Compound Ⅲ鉴定为N-(2, 6-二乙基苯基)乙酰胺(DEPA)。
|
| 图 6 利用LC-TOF-MS检测菌株BAD-20降解丁草胺的代谢产物 Figure 6 LC-TOF-MS Mass spectrograms and proposed structures of butachlor and its metabolites by butachlor- degrading strain BAD-20. A: compound Ⅰ: butachlor; B: compound Ⅱ: DEPBMA; C: compound Ⅱ: DEPA. |
3 讨论
丁草胺作为一种主要氯乙酰类除草剂,在农田除草中得到了广泛使用。但丁草胺有潜在的致癌性,对鱼类有高毒性,破坏土壤微生物多样性,而且土壤高残留还会对农作物产生严重药害,因此研究环境中丁草胺的降解规律和机制、开发丁草胺残留污染降解修复技术具有重要的意义。目前对丁草胺好氧微生物降解机制的研究比较深入,其降解过程及参与降解的酶和基因得到了阐明[35]。但是对于丁草胺厌氧降解的研究很少,仅仅获得了能够厌氧降解丁草胺的富集液,而丁草胺厌氧降解菌株分离鉴定及对丁草胺厌氧降解过程和机制方面的研究还未见报道。水稻田淹水时深层土壤处于厌氧状态,而且经常使用丁草胺除草,因此水稻田土壤存在厌氧丁草胺降解菌的生态条件。本研究中我们利用富集驯化技术,以丁草胺为碳源作为选择压力,成功从水稻田土壤中富集分离到一株厌氧丁草胺降解菌BAD-20,鉴定为嗜蛋白质菌属(Proteiniphilum)。嗜蛋白质菌属为一个创建于2005年的比较新的属,到目前共有两个种Proteiniphilum acetatigenes和Proteiniphilum saccharofermentans[36-37],嗜蛋白质菌属的模式菌株Proteiniphilum acetatigenes TB107T (=JCM 12891T= AS 1.5024T)分离自处理啤酒废水的UASB(Up-flow Anaerobic Sludge Bed)厌氧反应器中的颗粒污泥,能够分解利用酵母膏、蛋白胨、甘氨酸、精氨酸、丝氨酸和苏氨酸等蛋白氨基酸类物质生长,但对有机酸利用能力较差,仅能利用丙酮酸生长[36]。而菌株BAD-20除分解利用酵母膏、蛋白胨、甘氨酸和丙酮酸生长外,还能利用甲酸、乙酸和乳酸等有机酸生长,这可能跟菌株BAD-20栖息环境稻田土壤有机酸比较丰富有关;而菌株BAD-20能够降解氯乙酰胺类除草剂则为该属细菌首次报道。
从菌株BAD-20的降解谱可知,该菌能够降解甲草胺、乙草胺和丁草胺等多种氯乙酰胺类除草剂,其降解效率从高到低依次是甲草胺 > 乙草胺 > 丙草胺 > 丁草胺。而通过比较不同氯乙酰胺类除草剂的分子结构,甲草胺、乙草胺、丙草胺和丁草胺分子结构的主要差异之处在于N-取代烷氧基团的长度不同,以及苯环上侧链基团不同(表 2)。通过比较不同氯乙酰胺类除草剂分子结构的差异及菌株BAD-20对不同氯乙酰胺类除草剂降解能力的差异,我们发现氯乙酰胺类除草剂厌氧降解效率与其分子结构有密切关系,即N-取代烷氧基团越长越复杂,其降解效率越低,而苯环上取代基团对降解效率影响不大。甲草胺、乙草胺、丙草胺和丁草胺支链上烷氧基团分别为CH3O-、CH3CH2O-、CH3CH2CH2O-和CH3CH2CH2CH2O-,其长度和复杂程度逐渐增加,而降解效率逐渐降低。N-取代烷氧基团影响降解效率可能的原因是氯乙酰胺除草剂与降解酶作用或结合的关键位置不是苯环,而是N-取代烷氧基团和氯乙酰基,N-取代烷氧基团越大越复杂,会影响降解酶-底物的相互结合,从而影响降解效率。
在好氧菌中,丁草胺的降解上游步骤是在加氧酶CndABC催化下发生N-脱烷基生成N-(2, 6-二2-氯-N-(2, 6-二乙基苯基)乙酰胺,该产物接着在酰胺酶CmeH催化下脱氯乙酰基生成2, 6-二乙基苯胺[26-28]。利用LC-TOF-MS,我们从厌氧菌株BDA-20降解丁草胺的培养液中鉴定到2个代谢产物N-(2, 6-二乙基苯基)-N-(丁氧甲基)乙酰胺(DEPBMA)和N-(2, 6-二乙基苯基)乙酰胺(DEPA)。根据这个产物鉴定结果我们推测菌株BDA-20厌氧降解丁草胺的上游降解步骤为先脱氯再脱N-烷氧烷基(图 7)。这个先脱氯再脱烷基的降解过程完全不同于好氧菌对丁草胺的先脱烷基再脱氯乙酰基降解过程,是一个新的乙草胺降解机制。

|
| 图 7 菌株BAD-20降解丁草胺的代谢途径推测 Figure 7 Proposed anaerobic degradation pathway of butachlor by the strain BAD-20. |
卤代化合物广泛存在于自然界,很多重要的合成化学品也是卤代化合物。一般卤代化合物化学性质非常稳定,难以降解,而脱卤可以提高其可生物降解性。研究表明微生物脱卤机制非常多样化,有氧化脱卤、水解脱卤、还原脱卤和硫醇取代脱卤[38-41]。在厌氧菌中报道比较多的是还原脱卤,有呼吸型还原脱卤和非呼吸型还原脱卤两种类型[40, 42-43]。呼吸型还原脱卤即卤代芳烃在厌氧条件下作为电子呼吸链最终电子受体,因此呼吸型还原脱卤又叫脱卤呼吸。如Dehalobacter restrictus和Dehalospirillum multivorans等能够通过脱卤呼吸将四氯乙烯(Perchloroethylene,PCE)脱去一个氯原子转化为三氯乙烯(Trichloroethylene,TCE)[40, 42]。非呼吸型还原脱卤不与电子呼吸链耦联,其所需的电子由NADPH提供。催化还原型脱卤的关键酶是还原脱卤酶(reductive dehalogenase,RDs),呼吸型位于细胞膜上以方便它们参与膜相关的电子传递—耦合氧化磷酸化,而非呼吸型还原脱卤酶位于细胞质[40, 42-43]。菌株BAD-20到底是通过哪一种脱卤机制脱去丁草胺上的氯原子还不清楚,需要进一步研究。
| [1] | 刘长令. 世界农药大全-除草剂卷. 北京: 化学工业出版社, 2002. |
| [2] |
Jiang ZP, Gu WZ. Research progress of chloroacetamide herbicides. Shandong Pesticide News, 2010(3): 27-28.
(in Chinese) 江忠萍, 谷文喆. 酰胺类除草剂研究进展. 山东农药信息, 2010(3): 27-28. |
| [3] |
Lu MR. Market situation and future prospect of amide herbicides at home and abroad. Pesticide Market News, 2009(16): 11-13.
(in Chinese) 陆明若. 酰胺类除草剂国内外市场状况及未来前景. 农药市场信息, 2009(16): 11-13. |
| [4] | EPA US. Report of the Food Quality Protection Act (FQPA) tolerance reassessment progress and risk management decision (TRED) for acetochlor. Report EPA738-R-00-009. US Environmental Protection Agency. Washington, DC. 2006; http://www.epa.gov/pesticides/reregistration/REDs/acetochlor_tred.pdf. |
| [5] |
Xu HJ, He HB, Wu YY, Liu N, Zhang XD. Environmental behavior and ecological effect of chloroacetamide herbicides in terrestrial ecosystem. Chinese Journal of Soil Science, 2009, 40(5): 1226-1232.
(in Chinese) 徐会娟, 何红波, 武叶叶, 刘宁, 张旭东. 氯乙酰胺类除草剂的环境行为和生态效应研究. 土壤通报, 2009, 40(5): 1226-1232. |
| [6] |
Zhao YH, Mei QZ, Chen MC, Min H. Effect of butachlor on CH4 emission and anaerobes in paddy soil. Acta Microbiologica Sinica, 1997, 37(6): 477-479.
(in Chinese) 赵宇华, 梅其志, 陈美慈, 闵航. 丁草胺对水稻土甲烷释放和厌氧细菌的影响. 微生物学报, 1997, 37(6): 477-479. |
| [7] |
Deng X, Liao XL, Tang QF. Effects of methamidophos and acetochlor on population and activity of methanogens. Rural Eco-Environment, 2004, 20(3): 56-59.
(in Chinese) 邓晓, 廖晓兰, 唐群锋. 甲胺磷和乙草胺对产甲烷菌种群数量及其活性的影响. 农村生态环境, 2004, 20(3): 56-59. |
| [8] | Seghers D, Verthé K, Reheul D, Bulcke R, Siciliano SD, Verstraete W, Top EM. Effect of long-term herbicide applications on the bacterial community structure and function in an agricultural soil. FEMS Microbiology Ecology, 2003, 46(2): 139-146. DOI:10.1016/S0168-6496(03)00205-8 |
| [9] | Abu-Qare AW, Duncan HJ. Herbicide safeners: uses, limitations, metabolism, and mechanisms of action. Chemosphere, 2002, 48(9): 965-974. DOI:10.1016/S0045-6535(02)00185-6 |
| [10] |
Han YJ, Yan CX, He FL, Shi N, Zhao CS. Study of butachlor on safety in early stage of rice. Journal of Northeast Agricultural University, 2007, 38(5): 586-589.
(in Chinese) 韩玉军, 闫春秀, 何付丽, 史娜, 赵长山. 丁草胺对水稻安全性影响的研究. 东北农业大学学报, 2007, 38(5): 586-589. DOI:10.3969/j.issn.1005-9369.2007.05.003 |
| [11] |
Liu WX, Chen YQ, Kou YB, Guo HW, Chang L. Effects of individual and combined pollution of cadmium and butachlor on growth of amaranth. Henan Science, 2009, 27(3): 305-308.
(in Chinese) 刘文霞, 陈元琦, 寇渊博, 郭华武, 常乐. 镉、丁草胺单一及复合污染对苋菜生长的影响. 河南科学, 2009, 27(3): 305-308. DOI:10.3969/j.issn.1004-3918.2009.03.014 |
| [12] |
Liu B, Guan CH, Wang XF, Xie LH. Analyzing the causes of and solutions for crop injury by herbicides commonly used in northeast China. Agrochemicals, 2006, 45(6): 368-373.
(in Chinese) 刘波, 关成宏, 王险峰, 谢丽华. 我国东北地区常见除草剂药害原因分析与解决方法. 农药, 2006, 45(6): 368-373. DOI:10.3969/j.issn.1006-0413.2006.06.003 |
| [13] |
Zheng HH, Ye CM. Hydrolysis of chloroacetanilide herbicidesacetochidr and butachlor. Environmental Chemistry, 2001, 20(2): 168-171.
(in Chinese) 郑和辉, 叶常明. 乙草胺和丁草胺的水解及其动力学. 环境化学, 2001, 20(2): 168-171. |
| [14] |
Zheng HH, Ye CM. Photodegradation of acetochlor and butachlor in soil. Environmental Chemistry, 2002, 21(2): 117-122.
(in Chinese) 郑和辉, 叶常明. 乙草胺和丁草胺在土壤中的紫外光化学降解. 环境化学, 2002, 21(2): 117-122. DOI:10.3321/j.issn:0254-6108.2002.02.003 |
| [15] |
Chu XQ, Pang GH, Fang H, Wang XG, Gao CM, Yu YL. Isolation, identification and characteristics of a butachlor-degrading bacterium. Journal of Agro-Environment Science, 2009, 28(1): 145-150.
(in Chinese) 楚小强, 庞国辉, 方华, 王秀国, 高春明, 虞云龙. 丁草胺降解菌的分离鉴定及降解特性的研究. 农业环境科学学报, 2009, 28(1): 145-150. DOI:10.3321/j.issn:1672-2043.2009.01.027 |
| [16] |
Li C, Gu GB, Liu S. Isolation and its degradability of high efficiency butachlor fungi. Journal of Agro-Environmental Science, 2004, 23(3): 611-614.
(in Chinese) 李川, 古国榜, 柳松. 丁草胺高效真菌的分离及性能研究. 农业环境科学学报, 2004, 23(3): 611-614. DOI:10.3321/j.issn:1672-2043.2004.03.044 |
| [17] |
Li YC, Xiong MH, Xiao J, Li CY. Isolation and characteristics of a butachlor degradation bacterium. Microbiology China, 2009, 36(8): 1178-1182.
(in Chinese) 李艳春, 熊明华, 肖晶, 李春艳. 一株丁草胺降解菌的分离鉴定及其降解特性的研究. 微生物学通报, 2009, 36(8): 1178-1182. |
| [18] |
Ni YY, Zheng JW, Zhang J, Wang BZ, He J, Li SP. Isolation of chloracetanilide herbicides-degrading bacterium Y3B-1 and its degradability to chloracetanilide herbicides. Chinese Journal of Applied & Environmental Biology, 2011, 17(5): 711-716.
(in Chinese) 倪盈盈, 郑金伟, 张隽, 王保站, 何健, 李顺鹏. 氯代酰胺类除草剂降解菌的分离及降解性能. 应用与环境生物学报, 2011, 17(5): 711-716. |
| [19] |
Wu XJ, Yue YD, Hua RM, Tang F. Isolation of effectively butachlor degradating bacterium. Chinese Journal of Applied and Environmental Biology, 2000, 6(6): 593-596.
(in Chinese) 吴新杰, 岳永德, 花日茂, 汤锋. 丁草胺高效降解细菌的分离. 应用与环境生物学报, 2000, 6(6): 593-596. DOI:10.3321/j.issn:1006-687X.2000.06.019 |
| [20] | Chen Q, Zhang J, Wang CH, Jiang J, Kwon SW, Sun LN, Shen WB, He J. Novosphingobium chloroacetimidivorans sp. nov., a chloroacetamide herbicide-degrading bacterium isolated from activated sludge. International Journal of Systematic and Evolutionary Microbiology, 2014, 64(Pt8): 2573-2578. |
| [21] | Chu CW, Chen Q, Wang CH, Wang HM, Sun ZG, He Q, He J, Gu JG. Roseomonas chloroacetimidivorans sp. nov., a chloroacetamide herbicide-degrading bacterium isolated from activated sludge. Antonie Van Leeuwenhoek, 2016, 109(5): 611-618. DOI:10.1007/s10482-016-0664-y |
| [22] |
Chen Q, Yao L, Wang CH, Deng SK, Chu CW, He J. Isolation and characterization of acetochlor-degrading strain Sphingomonas sp. DC-6 and preliminary studies on its metabolic pathway. Journal of Agricultural Science and Technology, 2013, 15(5): 67-74.
(in Chinese) 陈青, 姚利, 王成红, 邓诗凯, 褚翠伟, 何健. 乙草胺降解菌Sphingomonas sp. DC-6的分离鉴定及其代谢途径的初步研究. 中国农业科技导报, 2013, 15(5): 67-74. DOI:10.3969/j.issn.1008-0864.2013.05.10 |
| [23] | Liu HM, Cao L, Lu P, Ni HY, Li YX, Yan X, Hong Q, Li SP. Biodegradation of butachlor by Rhodococcus sp. strain B1 and purification of its hydrolase (ChlH) responsible for N-dealkylation of chloroacetamide herbicides. Journal of Agricultural and Food Chemistry, 2012, 60(50): 12238-12244. DOI:10.1021/jf303936j |
| [24] | Zhang J, Zheng JW, Liang B, Wang CH, Cai S, Ni YY, He J, Li SP. Biodegradation of chloroacetamide herbicides by Paracoccus sp. FLY-8in vitro. Journal of Agricultural and Food Chemistry, 2011, 59(9): 4614-4621. DOI:10.1021/jf104695g |
| [25] | Wang F, Zhou J, Li ZK, Dong WL, Hou Y, Huang Y, Cui ZL. Involvement of the cytochrome P450 system EthBAD in the N-deethoxymethylation of acetochlor by Rhodococcus sp. strain T3-1. Applied and Environmental Microbiology, 2015, 81(6): 2182-2188. DOI:10.1128/AEM.03764-14 |
| [26] | Cheng MG, Meng Q, Yang YJ, Chu CW, Chen Q, Li Y, Cheng D, Hong Q, Yan X, He J. The two-component monooxygenase MeaXY initiates the downstream pathway of chloroacetanilide herbicide catabolism in sphingomonads. Applied and Environmental Microbiology, 2017, 83(7): 1-13. DOI:10.1128/aem.03241-16 |
| [27] | Chen Q, Wang CH, Deng SK, Wu YD, Li Y, Yao L, Jiang JD, Yan X, He J, Li SP. Novel three-component rieske non-heme iron oxygenase system catalyzing theN-dealkylation of chloroacetanilide herbicides in sphingomonads DC-6 and DC-2. Applied and Environmental Microbiology, 2014, 80(16): 5078-5085. DOI:10.1128/AEM.00659-14 |
| [28] | Li Y, Chen Q, Wang CH, Cai S, He J, Huang X, Li SP. Degradation of acetochlor by consortium of two bacterial strains and cloning of a novel amidase gene involved in acetochlor-degrading pathway. Bioresource Technology, 2013, 148: 628-631. DOI:10.1016/j.biortech.2013.09.038 |
| [29] | Loor-Vela SX, Crawford Simmons JJ, Simmons FW, Raskin L. Dissipation of[14C]acetochlor herbicide under anaerobic aquatic conditions in flooded soil microcosms. Journal of Agricultural and Food Chemistry, 2003, 51(23): 6767-6773. DOI:10.1021/jf0341058 |
| [30] | Janniche GS, Mouvet C, Albrechtsen HJ. Acetochlor sorption and degradation in limestone subsurface and aquifers. Pest Management Science, 2010, 66(12): 1287-1297. DOI:10.1002/ps.2011 |
| [31] | Konopka A. Anaerobic degradation of chloroacetanilide herbicides. Applied Microbiology and Biotechnology, 1994, 42(2/3): 440-445. DOI:10.1007/BF00902754 |
| [32] | Seybold CA, Mersie W, McNamee C. Anaerobic degradation of atrazine and metolachlor and metabolite formation in wetland soil and water microcosms. Journal of Environmental Quality, 2001, 30(4): 1271-1277. DOI:10.2134/jeq2001.3041271x |
| [33] |
Ye YF, Min H, Du YF. Anaerobic degradation of butachlor by sulfate reducing bacteria enrichment culture. Acta Scientiae Circumstantiae, 2000, 20(3): 376-378.
(in Chinese) 叶央芳, 闵航, 杜宇峰. 一个硫酸盐还原细菌富集物对丁草胺的厌氧降解. 环境科学学报, 2000, 20(3): 376-378. DOI:10.3321/j.issn:0253-2468.2000.03.026 |
| [34] | Liu JW, Zhang X, Xu JY, Qiu JG, Zhu JC, Cao H, He J. Anaerobic biodegradation of acetochlor by acclimated sludge and its anaerobic catabolic pathway. Science of the Total Environment, 2020, 748: 141122. DOI:10.1016/j.scitotenv.2020.141122 |
| [35] |
Chen Q, Wang HM, Li XH, Wei HF, Su P, He J, Jiang JD. Research progress in microbial degradation of chloroacetanilide herbicides. Chinese Journal of Applied and Environmental Biology, 2019, 25(5): 1252-1260.
(in Chinese) 陈青, 王红妹, 李晓红, 尉鸿飞, 苏朋, 何健, 蒋建东. 氯乙酰胺类除草剂微生物降解研究进展. 应用与环境生物学报, 2019, 25(5): 1252-1260. |
| [36] | Chen SY, Dong XZ. Proteiniphilum acetatigenes gen. nov., sp. nov., from a UASB reactor treating brewery wastewater. International Journal of Systematic and Evolutionary Microbiology, 2005, 55(6): 2257-2261. DOI:10.1099/ijs.0.63807-0 |
| [37] | Hahnke S, Langer T, Koeck DE, Klocke M. Description of Proteiniphilum saccharofermentans sp. nov., Petrimonas mucosa sp. nov. and Fermentimonas caenicola gen. nov., sp. nov., isolated from mesophilic laboratory-scale biogas reactors, and emended description of the genus Proteiniphilum. International Journal of Systematic and Evolutionary Microbiology, 2016, 66(3): 1466-1475. DOI:10.1099/ijsem.0.000902 |
| [38] | Xun L. Purification and characterization of chlorophenol 4-monooxygenase from Burkholderia cepacia AC1100. Journal of Bacteriology, 1996, 178(9): 2645-2649. DOI:10.1128/JB.178.9.2645-2649.1996 |
| [39] | Scholten JD, Chang KH, Babbitt PC, Charest H, Sylvestre M, Dunaway-Mariano D. Novel enzymic hydrolytic dehalogenation of a chlorinated aromatic. Science, 1991, 253(5016): 182-185. DOI:10.1126/science.1853203 |
| [40] | Holliger C, Wohlfarth G, Diekert G. Reductive dechlorination in the energy metabolism of anaerobic bacteria. FEMS Microbiology Reviews, 1998, 22(5): 383-398. DOI:10.1111/j.1574-6976.1998.tb00377.x |
| [41] | la Roche SD, Leisinger T. Sequence analysis and expression of the bacterial dichloromethane dehalogenase structural gene, a member of the glutathione S-transferase supergene family. Journal of Bacteriology, 1990, 172(1): 164-171. DOI:10.1128/JB.172.1.164-171.1990 |
| [42] | Neumann A, Scholz-Muramatsu H, Diekert G. Tetrachloroethene metabolism of Dehalospirillum multivorans. Archives of Microbiology, 1994, 162(4): 295-301. DOI:10.1007/BF00301854 |
| [43] | Chen K, Huang LL, Xu CF, Liu XM, He J, Zinder SH, Li SP, Jiang JD. Molecular characterization of the enzymes involved in the degradation of a brominated aromatic herbicide. Molecular Microbiology, 2013, 89(6): 1121-1139. DOI:10.1111/mmi.12332 |
 2021, Vol. 61
2021, Vol. 61