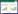中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 杨丽洁, 贾仲君. 2021
- Yang Lijie, Jia Zhongjun. 2021
- “99%难培养”微生物的概念与初步评价:以固氮菌为例
- History and definition of the "99% unculturability paradigm"-case analysis with nitrogen-fixing bacteria
- 微生物学报, 61(4): 903-922
- Acta Microbiologica Sinica, 61(4): 903-922
-
文章历史
- 收稿日期:2020-04-21
- 修回日期:2020-07-13
- 网络出版日期:2020-07-31
2. 中国科学院大学, 北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049, China
过去30年来,微生物学领域最为引人关注的论述常被认为是“绝大多数微生物尚未培养”,或具有一定主观色彩的“99%难培养”。这一论述缺乏严格的实验证据,却被学术界广泛认可,其主要归功于Carl Woese基于核糖体16S rRNA基因的系统发育进化理论及其分类意义[1-2],换言之,提取土壤中所有微生物的基因组DNA,测序16S rRNA基因并开展系统发育同源性分析,通常会发现大量的序列与已知纯培养菌株的16S rRNA亲缘关系较远,被认为代表了尚未培养的微生物,或难培养微生物[3-4],甚至被称为微生物暗物质[5],具有潜在巨大的理论与应用价值,因此,复杂环境如土壤、海洋、人体、动物和植物微生物组的多样性与功能研究,成为过去30年来微生物学的热点和前沿[6-7]。
然而,“99%难培养”本身即为悖论,简言之,地球环境中微生物的物种数量,尚未有共识,具有极大的不确定性,估算误差最高可达1亿倍,从1万到1万亿[8-9]。物种总数量尚不确定的条件下,则无法计算未知微生物的比例。例如,经典的微生物分类学表明,已知的微生物属和种分别为2930属、15448种[10]。但是,基于目前数据库已有的16S rRNA基因序列,模型推测地球最多栖息着1万亿微生物种[8]。但这一模型算法存在较大争议[11],最新的估算则认为全球微生物种的数量约为80万到160万[12]。截止2020年4月10日,NCBI分类学专题报道微生物(细菌和古菌)共计4071属465268种,其中可培养3770属27773种[13],据此,我们推测可培养属和种占所有微生物比例分别为92.6%和5.96%,但这一算法参考价值有限,具有较大的不确定性。同时,“不可培养”的表述并不严谨,其本身即为悖论,更合理的表述是“难培养”或“尚未培养”微生物。因为目前条件下,无法穷尽所有人工培养配方,获得明确实验数据,证明微生物具有“绝对的不可培养性”。
文献分析表明“99%难培养”并未有准确出处[14-15],更多是作为“平板计数异常”的同义词而成为共识,也就是说,针对同一个环境样品,采用显微计数和培养基平板计数两种不同的方法,微生物数量相差最高可达几个数量级,这一现象在1985年被命名为平板计数异常,即“The Great Plate Count Anomaly”[16]。最早的文献报道似乎来自1898年,针对多种病原微生物检测,奥地利科学家Heinrich Winterberg发现平板计数法占显微计数病原菌比例最低约为6%[17]。针对环境样品的分析,1932年俄罗斯科学家围绕自来水消毒后的安全评价文献被广泛引用[18];而土壤最早文献来自1934年英国科学家Henry Thornton的报道,发现草地土壤和农田土壤中,显微计数法是平板计数的117–1780倍,平板形成的菌落CFU (colony forming unit),仅占显微计数所得微生物的0.09%–0.74%[19]。一般认为,造成这种差异的主要原因是培养基无法代表真实的自然环境,导致绝大部分微生物不能在培养基上生长并形成菌落。其他一些原因包括,损伤或死亡细胞较多、细胞聚集成团或吸附于土壤颗粒无法分散[20],或者细胞之间存在共生以及具有活性但无法生长形成菌落等[21]。值得注意的是,传统“99%难培养”概念,通常针对纯培养菌株。然而,目前技术条件下很难获得复杂环境如土壤中所有的纯培养菌株。接种量、传代次数、分离方法、培养时间和营养组成等均可导致特异性分离而遗漏某些微生物。例如,即使从土壤和地下水中分离获得1366株纯菌,仍然仅占显微计数总量的0.1%–1.0%[22]。因此,经典的纯培养菌株分离及表型和形态分类,很难解决平板异常难题[16]。
基于16S rRNA基因的分子系统分类为定量评估“99%难培养”提供了新思路。尽管存在一定争议,16S rRNA基因同源性的系统发育分子分析,事实上提供了一种可操作的微生物分类策略,如分类操作单元Operational Taxonomic Unit (OTU)。通过提取环境中所有微生物的基因组DNA,高通量测序16S rRNA基因并重构物种的系统发育进化关系,即可获得所有微生物的分类信息。这一方法规避了传统富集分离的内在缺陷。例如,常规的细菌培养基会遗漏绝大多数功能菌,如固氮菌、甲烷氧化菌、硝化菌等微生物,特别是化能无机自养菌。这些功能微生物必须采用特殊培养基富集分离,并在专业实验室完成。理论上而言,没有一种培养基可以分离富集所有微生物。因此,针对特定的功能微生物,利用其特异的培养基富集策略,定量评估“99%难培养”概念更具现实意义。然而,迄今尚未有相关报道。
氮是地球所有生命的主要元素,深度发掘可培养固氮菌资源具有重要意义。据估算,全球每年固氮量高达4.15亿吨[23],其中2.9亿吨源自生物固氮[24]。同时,2010年以来,高通量测序已成为一种常规实验手段。据此,我们以固氮菌为例,利用特殊培养基富集可培养固氮菌,结合菌体DNA和土壤DNA提取及分子分类,定量评估可培养富集固氮菌的比例,为“99%难培养”概念提供参考。基本思路如下:(1) 可培养固氮菌的富集。采用常规的固体和液体固氮培养基,传代两次并收集平板表面和液体中所有菌体后,提取DNA开展nifH和16S rRNA基因扩增子测序分析。这些固氮菌富集物在理论上能够最大程度代表潜在的所有可培养纯菌株;(2) 所有固氮菌。直接提取土壤微生物DNA样品,测序nifH基因获得所有固氮菌,同时利用通用引物测序获得所有微生物16S rRNA序列,进一步与Ribosomal Database Project (RDP)数据库中已知固氮菌开展同源性分析,结合可培养固氮菌富集物,获得待测土壤中的所有固氮菌。
1 材料和方法 1.1 土壤样品描述土壤样品采集于中国科学院河南封丘农田生态系统国家野外科学观测试验站(35°00' N,114°24' E),该地区年均气温13.9 ℃,年均降水量615 mm,实行冬小麦和夏玉米轮作制度[25]。供试土壤类型为潮土,由黄河冲积沉积物发育形成。基本理化性质如下:土壤含水量,8.23%;土壤最大持水量(SWHC),47.1%;NH4+-N,3.85 mg/kg;NO3-N,32.4 mg/kg;全氮,1.26 g/kg;全碳,18.6 g/kg;土壤碳氮比,14.8;pH (H2O):7.8。采集0–20 cm新鲜土壤,研磨并过2 mm筛,4 ℃保存。
1.2 可培养固氮菌的富集 1.2.1 培养基成分:利用经典的Modified-NFb无氮培养基[26],研究土壤中可培养固氮菌富集物。液体培养基主要成分包括:苹果酸5 g,葡萄糖2.5 g,蔗糖2.5 g,KH2PO4 0.6 g,K2HPO4·3H2O 0.2 g,MgSO4·7H2O 0.2 g,NaCl 0.1 g,CaCl2 0.02 g,Fe-EDTA (solution 16.4 g/L) 4 mL,KOH 4 g,最后将其定容于1000 mL去离子无菌水,利用KOH调节pH为7.0。固体培养基配方相同,仅额外添加1.5%琼脂。高压灭菌锅121 ℃、20 min灭菌。培养基灭菌后,加入维生素混合液1 mL (生物素10 mg,盐酸吡哆醇20 mg,定容至100 mL,0.22 μm滤膜过滤除菌);微量元素混合液2.0 mL (CuSO4·5H2O 0.04 g;ZnSO4·7H2O 0.12 g;H3BO3 1.40 g;Na2MoO4·2H2O 1.0 g;MnSO4·H2O 1.175 g,定容至1 L,0.22 μm滤膜过滤除菌)。
1.2.2 可培养固氮菌的富集策略:本研究所用的可培养固氮菌并非纯培养菌株,而是常规培养基上可富集的固氮微生物集合。简言之,在固体/液体培养基第一代、第二代中出现的所有微生物菌落集合,可被认为是所有潜在可分离的纯培养菌株。同时,这些微生物也是可利用人工配方营养的固氮菌富集物。
具体操作流程如下:称取土壤样品1 g,溶于100 mL无菌水中后,以200 r/min转速揺菌0.5 h制成土壤悬液作为接种液,通过固体和液体培养基开展可培养固氮菌的富集。所有培养操作步骤均为无菌操作并设置3次生物学重复。
固体可培养固氮菌富集物的操作如下:吸取100 μL土壤悬液并均匀涂布于固体培养基表面,将平板倒置于28 ℃培养箱中培养7 d后,采用10 mL无菌水将培养基表面菌体多次冲洗至50 mL无菌试管制成第一代富集菌液,取1 mL提取DNA。同时,吸取100 μL菌液再次使用稀释涂布平板法接种传代培养7 d后,同样以10 mL无菌水冲洗所有菌体制成第二代富集菌液,取1 mL提取DNA并保存于–20 ℃。
液体可培养固氮菌富集物的操作如下:吸取100 μL土壤悬液接种至100 mL液体培养基后,将培养三角瓶放入28 ℃培养箱中,静置培养7 d后获得第一代菌液,将100 mL第一代富集菌液离心(400×g,10 min)后,弃上清,加入10 mL无菌水,振荡混匀后浓缩为10 mL富集菌液后取1 mL提取DNA。同时,吸取100 μL富集菌液接种至新的100 mL液体培养基中,传代培养7 d后,采用相同方法浓缩为10 mL第二代富集菌液,取1 mL提取DNA并保存于–20 ℃。
1.2.3 稳定性同位素15 N2示踪可培养固氮菌富集物的生长:将30 mL的固体培养基分装于120 mL血清瓶内,接种100 μL第二代固体富集菌液后密封血清瓶,设置15N2-标记组和14N2-对照组。标记组的培养瓶顶空包括30%的15N2(99%原子百分超)、20% O2和50% Ar;对照组则包括30% 14N2、20% O2和50% Ar。所有接种和密封操作均在无菌室完成后,置于28 ℃培养箱7 d,然后打开瓶盖,采用10 mL无菌水反复冲洗固体培养基表面的菌体为富集菌液,取1 mL提取DNA后存于–20 ℃,超高速密度梯度离心分离15N-DNA,验证富集物为固氮菌。所有处理均设置3个生物学重复。
1.3 固氮菌的定量与测序 1.3.1 土壤微生物的DNA提取:土壤样品DNA提取。采用FastDNA Spin Kit for Soil (MP Bio)试剂盒:称取0.5 g土壤样品置于2.0 mL离心管中,加入SPB和MT缓冲液后,利用Fast Prep核酸提取仪以6.0 m/s振荡45 s,14000×g离心15min。转移上清至新2.0 mL离心管中,以PPS沉淀蛋白溶液,Binding Matrix结合DNA,SEWS-M洗涤过滤后,将DNA溶解于50 μL DES缓冲液。微量紫外分光光度计(NanoDrop ND-1000)测定DNA浓度为15.2–56.7 ng/μL,纯度A260/280为1.68–1.92,A260/230为1.52–2.03。同时利用1.2%的琼脂糖凝胶电泳检测DNA完整性。
1.3.2 可培养固氮菌富集物的DNA提取:固体和液体第一代、第二代固氮菌富集物DNA提取。采用E.Z.N.A.Ⓡ Bacterial DNA Kit (Omega)试剂盒:将1 mL富集菌液4000×g离心10 min,吸除上清,加入100 μL TE Buffer重悬,利用Lysozyme、Proteinase K Solution和RNase A裂解细胞,去除蛋白质和RNA,100% DNA Wash Buffer洗涤过滤后,加入50 μL Elution Buffer,过滤收集DNA。DNA浓度为73.4–86.9 ng/μL,纯度A260/280为1.78–1.89,A260/230为1.58–1.96。
1.3.3 实时荧光定量PCR:使用Bio-Rad CFX96实时荧光定量PCR仪,针对土壤DNA、固体和液体固氮菌第一代、第二代富集物菌体DNA以及稳定同位素标记的富集物菌体DNA进行nifH基因和16S rRNA基因定量。
nifH基因定量。采用PolF/PolR引物[27],PolF序列为TGCGAYCCSAARGCBGACTC,PolR序列为ATSGCCATCATYTCRCCGGA。PCR反应体系:10 μL SYBR Premix Ex Taq (TaKaRa),正反向引物各0.5 μL,DNA模板1 μL,8 μL DNase/ RNase-free H2O,PCR扩增程序为:95 ℃ 10 min;94 ℃ 1 min,55 ℃ 30 s,72 ℃ 1 min,39个循环。标准曲线制作:计算含有目标基因片段质粒的摩尔数,然后依次稀释7个浓度梯度,以初始模板DNA的拷贝对数为横坐标,以Ct值为纵坐标,获得标准曲线:Ct=–3.795lgC+44.457(R2=0.999)。
16S rRNA基因定量。采用515F/907R通用引物对[28],515F序列为GTGCCAGCMGCCGCGG;907R序列为CCGTCAATTCMTTTRAGTTT。PCR反应体系与nifH基因相同,PCR扩增程序为:95 ℃ 3 min;95 ℃ 30 s,55 ℃ 30 s,72 ℃ 30 s;39个循环。标准曲线绘制如nifH基因定量所述,Ct=–3.561lgC+27.639(R2=0.998)。
固氮菌同化15N2的证据(可培养富集物15N-DNA分离)。超高速密度梯度离心详细步骤如前所述[29]。简言之:将2 µg DNA与氯化铯溶液混合后,形成初始浮力密度为1.725 g/mL的离心溶液,然后将其封装至5.1 mL超高速离心试管,利用贝克曼Vti65.2转子在速度45 kr/min (190000×g)下20 ℃离心44 h,随后通过自动分离装置将离心管内溶液分为30层不同浮力密度溶液,通过PEG-6000除去氯化铯介质后,采用70%乙醇纯化DNA离心沉淀并溶解于30 μL无菌水,–20 ℃保存。针对分层所得到的15N-DNA进行16S rRNA定量,定量方法如前所述。通过16S rRNA基因在不同浮力密度DNA中的变化规律,即可判定所有固氮菌富集物是否皆同化15N2并合成了生物质DNA。
1.3.4 Illumina Miseq高通量测序:nifH基因扩增子高通量测序。首先利用土壤DNA和固氮菌富集物DNA开展PCR扩增,所用引物PolF/PolR如前所述。PolF的5′端连接6个碱基的barcode序列,用于区分不同的样品。PCR扩增程序为95 ℃ 5 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 45 s,32个循环;72 ℃ 10 min。PCR反应体系为:25 μL Premix Taq (TaKaRa),正反向引物各1 μL,DNA模板2 μL,21 μL DNase/RNase-free H2O。PCR扩增产物在1.8 %的琼脂糖凝胶电泳后,在紫外灯下回收目标产物片段,并通过Agarose Gel DNA Fragment Recovery Kit Ver. 2.0试剂盒(TaKaRa)纯化PCR扩增产物后,溶于30 μL的DNase-free H2O。进一步通过1.2% 琼脂糖凝胶电泳检测PCR产物纯化效果后,利用微量紫外分光光度计(NanoDrop ND-1000)测定纯化后的PCR产物浓度。将所有样品PCR纯化产物按等摩尔数混合后建库和测序(Miseq PE300)。
16S rRNA基因扩增子高通量测序。引物如前所述,其中515F的5′端连接12个碱基的barcode序列,以便于后续识别不同样品的序列。PCR反应体系同nifH基因,扩增程序如下:94 ℃ 3 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 45 s,32个循环;72 ℃ 10 min。PCR产物的纯化、建库及测序过程与nifH基因扩增子相同。
1.4 土壤固氮菌的分子分类 1.4.1 高通量序列分析:背景土壤和传统固体、液体培养基两代富集物合计15个样品(含3个重复)用于高通量测序。nifH基因共计499965条高质量序列,单个样本平均33331条;16S rRNA基因共计386390条高质量序列,单个样本平均25759条。16S rRNA和nifH基因的高通量测序数据由QIIME (Quantitative Insights Into Microbial Ecology,version 1.9.1)分析完成,并使用usearch去除嵌合体。nifH基因的分析过程中,“extract_barcodes.py”指令用于提取barcode,“split_libraries_fastq.py”指令用于分割文库。“pick_otus.py”和“pick_rep_set.py”指令用于OTU聚类和提取代表序列,聚类在95%相似度进行。16S rRNA数据预处理部分同nifH基因,聚类在97%相似度进行。步骤细节参考http://qiime.org/scripts/index.html。
1.4.2 基于nifH和16S rRNA基因的固氮菌分子分类:固氮菌的物种分类。通过提取土壤DNA和固氮菌富集物DNA,利用高通量测序nifH和16S rRNA基因后,与固氮菌数据库比对,通过系统发育分子分类方法,在微生物门、纲、目、科、属水平,获得物种分类信息及其相对丰度。
基于nifH基因的分子分类。将nifH基因代表序列上传RDP网站上的FunGene pipline (http://fungene.cme.msu.edu/FunGenePipeline/pipeline/form.spr)处理进行OTU注释,nifH基因的OTU分类在95%相似度进行,注释参数Percent Identity > 0.8[30]。
基于16S rRNA基因的分子分类。将16S rRNA基因代表序列同RDP数据库进行比对,注释置信度阈值0.8,门、纲、目、科、属分类阈值分别为75.0%、78.5%、82.0%、86.5%、94.5%,某级别无法分类的序列被直接归为该级别的无法分类单元,统一纳入计算[31-32]。同时,本研究利用RDP建立了固氮微生物数据库,针对土壤中所有16S rRNA基因比对后,获得固氮菌序列进行分类。值得注意的是,基于富集物菌体16S rRNA基因比对RDP固氮菌数据库发现,固体和液体培养基中部分微生物菌落16S rRNA基因的亲缘菌株,尚未有固氮生理报道,我们将其定义为未培养固氮菌,并作为可培养固氮菌计算。
1.4.3 可培养固氮菌比例及富集率的计算:固氮菌的物种分类均通过nifH基因和16S rRNA基因分析完成。可培养固氮菌比例按公式(1)计算。

|
公式(1) |
其中土壤中所有固氮菌的物种数量包括两部分:(1) 土壤DNA中的固氮菌物种数量。直接提取土壤DNA,高通量测序nifH和16S rRNA基因,并在微生物门、纲、目、科、属水平,获得土壤中所有固氮菌的物种分类信息;(2) 可培养固氮菌的富集物。固体和液体培养基第一代、第二代中所有菌落富集物的物种分类信息。
可培养固氮菌富集率计算方法如下,基于nifH或16S rRNA基因分类,首先获得土壤DNA中所有固氮菌属的相对丰度;其次获得固体和液体培养基富集物菌落DNA中所有固氮菌属的相对丰度;后者除以前者,即可计算特定固氮菌在培养基中的富集率。富集率 > 1表示该菌属在培养中得到了富集。
1.5 统计分析与数据提交采用SPSS16.0 (SPSS Inc.,USA)对可培养固氮菌的富集率进行t检验,P < 0.05表示显著差异(*);P < 0.001表示极显著差异(**)。
高通量测序数据已提交至The National Center for Biotechnology Information (NCBI)数据库,具体编号为SRR12214932–12214961,其中含30个标签,包括5个处理(土壤、固体和液体培养基的第1、2代)及3个重复的两种基因测序数据。
2 结果和分析 2.1 可培养固氮菌的定量富集规律实时荧光定量PCR分析表明固氮菌在培养基中大量增殖(图 1)。背景土壤中nifH基因数量为2.08×108 copies /g d.w.s (dry weight soil),而根据接种土壤量计算,第一代固体和液体培养富集物nifH基因拷贝数分别为8.79×1012、5.58×1012 copies/g d.w.s,增幅 > 2.5万倍(图 1-A)。土壤中16S rRNA基因为2.11×1010 copies/g d.w.s,第一代固体和液体培养富集物中拷贝数分别为3.73×1013、3.86×1013 copies/g d.w.s,增幅 > 1800倍(图 1-B)。由于第二代的接种液为0.1 mL第一代富集物(含8.79×107 nifH genes),其中理论上仅含1.0×10–6 g土壤(2.08×102 nifH genes),两者相差达4万倍之多,因此接种物主要来自第一代菌体而非土壤,故第二代菌体根据其终体积10 mL计算,在固体和液体培养基中nifH基因分别为1.56×109、6.63×108/mL;16S rRNA基因分别为7.19×109、5.11×109/mL (图 1-B)。同时,土壤中nifH/16S rRNA基因拷贝数的比例仅为(0.982±0.15)%(图 1-C),而在固体和液体第一代培养基中,分别增至(23.9±4.1)%、(17.4±9.6)%,增幅25.5和18.9倍。第二代富集液中该比例未有明显变化,分别为(33.4±11)%、(14.9±2.2)%。

|
| 图 1 土壤及可培养固氮菌富集物的数量变化规律 Figure 1 Qiantification of nifH genes and 16S rRNA genes in N2-fixing enrichenments on solid and liquid medium and in background soil. A: copy number of nifH genes; B: copy number of 16S rRNA genes. Inset figure on the upper right represents the gene copy numbers in the second generation; C: the ratio of nifH to 16S rRNA genes; D: Quantitative distribution of bacterial 16S rRNA across the entire buoyant density of the DNA fractions from N2-fixing enrichenments incubated with either 15N2 or 14N2. 1st Enrich and 2nd Enrich denote the first and second generations of N2-fixing enrichments, respectively. |
针对超高速密度梯度离心后的30个不同浮力密度DNA,定量分析其中的16S rRNA基因拷贝数(图 1-D),结果表明,14N2-对照处理中,16S rRNA基因的最大值位于离心试管上部第20层,DNA浮力密度较小,约为1.718–1.729 g/mL;而15N2-标记处理中,16S rRNA基因的最大值出现在离心试管的下部第16层,浮力密度较大,约为1.730–1.738 g/mL,说明固氮菌富集物同化了15N2并合成了大量基因组DNA,导致超高速离心后,15N-DNA迁移至离心试管下部。
2.2 可培养固氮菌占土壤所有固氮菌的比例如图 2所示,基于nifH基因系统发育分类计算表明,土壤中可培养固氮菌范围为(7.38±1.1)%–(53.8±10)%。以微生物属为例,土壤中所有固氮菌共67属,而固体培养基第一代、第二代菌落富集物分别含有19、14个固氮菌属,占比分别为(28.4±6.3)%和(20.9±6.5)%(图 2-A)。值得注意的是,在更高的微生物分类水平门、纲、目、科,可培养固氮菌的比例逐渐增加,例如,土壤中所有固氮菌共7门,第一和第二代富集物中分别为4门、3门,占比(50.0±10)%、(38.1±10)%(图 2-A)。在更低的OTU种水平下,可培养比例显著降低,第一和第二代固氮菌富集物的比例分别为(8.09±4.2)%、(7.38±1.1)%。液体培养基也得到类似规律(图 2-B)。第一、第二代液体培养基分别含17、15个固氮菌属,占所有固氮菌67属的比例为(25.4±6.5)%、(22.4±4.5)%;在OTU种水平,占比为(13.4±4.6)%和(8.62±3.2)%(图 2-B)。
|
| 图 2 不同微生物分类水平下,土壤中可培养固氮菌富集物占所有固氮菌的比例 Figure 2 The proportion of culturable diazotrophs in soil at different taxonomic levels. The nifH genes-based proportion of culturable diazotroph in soil by solid (A) and liquid (B) medium enrichments; and the 16S rRNA gene-based proportion of culturable diazotroph in soil by solid (C) and liquid (D) medium enrichments. The percent number above the column represents the proportion of culturable diazotrophs at different taxonomic levels. Culturable diazotrophs are defined as all bacteria detected on solid or liquid medium, and abundance and composition was obtained on the basis of nifH and 16S rRNA gene analysis. All diazotrophs are defined as the sum of diazotrophs in soil DNA and bacteria detected on medium. Soil: DNA was directly extracted from soil, and microbial abundance and community composition was obtained by nifH and 16S rRNA gene analysis for phylogenetic classification (the number of taxa shown in the box). The number after "+" represents the number of diazotrophs taxa found only in the culture medium, but not detected in the soil. Solid and Liquid represent Solid and Liquid medium for enrichment of nitrogen-fixing bacteria, respectively. 1st and 2nd enrichment represent the first and second generation. |
基于16S rRNA基因的系统发育分类也得到类似结果,可培养固氮菌占所有固氮菌的比例较高:(31.6±3.4)%–(61.9±15)%。如图 2-C所示,在微生物属水平,土壤中所有固氮菌共255属,第一代、第二代固体培养基分别含有106、100个固氮菌属富集物,占比分别为(41.4±13)%和(39.2±4.2)%。在微生物门水平,第一代、第二代固体可培养固氮菌占比分别为(54.8±8.2)%、(47.6±15.3)%,并且在纲、目、科分类水平未有显著性差异(图 2-C)。液体培养基也得到了类似结果,第一代、第二代液体培养基分别含有82、81个可培养固氮菌属,占所有固氮菌255属的比例分别为(32.3±3.3)%、(31.6±3.4)%(图 2-D)。在更高的微生物分类水平,可培养固氮菌比例呈增加趋势,第一代、第二代液体培养基分别含有9、7固氮菌门,占所有固氮菌门比例分别为(61.9±15)%、(52.4±4.1)%(图 2-D)。
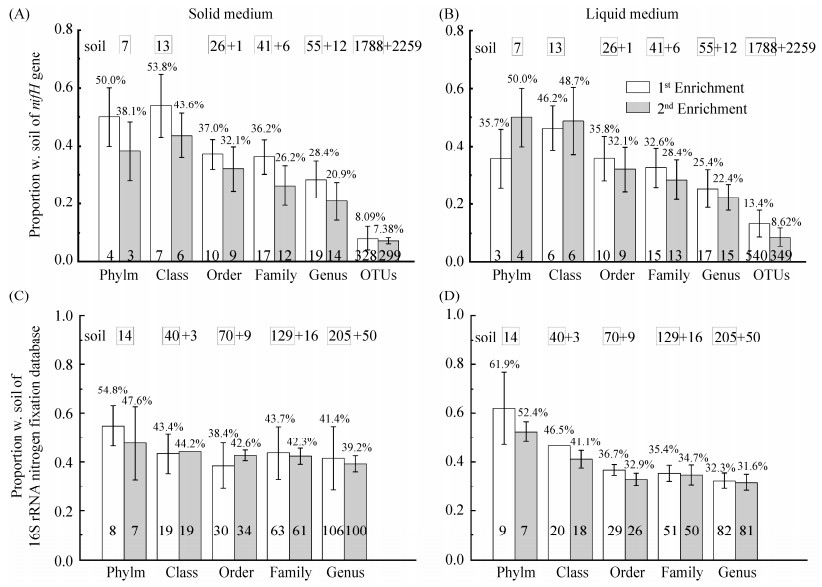
2.3 可培养固氮菌的物种变化规律分子系统分类表明,固体和液体培养基对土壤固氮菌具有极强的选择富集特征(图 3)。基于nifH基因的系统分类发现土壤中仅有7个固氮菌门(图 2-A),其中Proteobacteria丰度高达(90.9±5.7)%,其次为Firmicutes门,占比(0.974±0.20)%;另外5个门占比共计(0.220±0.018)%;值得注意的是,7.89%的土壤nifH基因序列无法分类,可能来自未培养固氮菌(图 3-A)。同时,固体培养基检测到5个固氮菌门富集物,其中Proteobacteria在第一代和第二代丰度分别为(96.4±2.7)%和(98.4±0.89)%,而Firmicutes也小幅度上升,相对丰度分别为(3.02±2.4)%和(1.49±0.90)%,另外3个门的丰度总计仅为0.002%和0.001%。液体培养基则得到完全不同的结果,共计检测到4个门,Firmicutes门是优势固氮菌,在第一代和第二代的丰度分别高达(96.7±0.55)%和(93.5±4.0)%,而Proteobacteria仅为(0.714±0.29)%和(0.65±0.070)%,另外4个门的丰度在第一代和第二代总计分别为0.004%和0.054%(图 3-A)。

|
| 图 3 微生物门和属水平下,土壤中可培养固氮菌的物种组成变化规律 Figure 3 Dynamic changes of diazotrophic communities in soils and enrichments at phylum and genus levels. The nifH gene-based taxonomic composition of diazotrophs in soil and enrichments at levels of phylum (A) and genus (B). The 16S rRNA gene-based taxonomic composition of diazotrophs in soil and enrichments at levels of phylum (C) and genus (D). The underlined taxa indicate that the sequences of these taxa showed high similariry to bacterium in which N2-fixing physiology has not been demonstrated within the underlined phylum and/or genus. All taxa without underlined signs denote at least one of strain within these phylum or genus has shown the physiological capability of N2-fixation. All other labels are the same as Figure 2. |
基于nifH基因的固氮微生物属富集物也得到了类似结果。土壤中所有67个固氮菌属中(图 2-A),我们选择了19个优势属分析(每个样品中的丰度前10属),其相对丰度分别占土壤、固体和液体培养基固氮菌属的89.2%、99.3%–99.8%和94.2%–97.4% (图 3-B)。土壤中Azospirillum占比最高达(47.0±2.9)%,其次为Bradyrhizobium (31.0±2.8)%和Rhizobium属(7.36±0.89)%。固体培养基中,Azotobacter在第一代和第二代占比分别高达96.6%±2.7%和98.2%±0.94%,而在土壤本底中仅为1.71%±0.40%;而原始土壤中丰度最高的Azospirillum和Bradyrhizobium则降至0.234%– 0.289%和0.004%–0.005% (图 3-B)。液体培养基则选择了完全不同的固氮菌,Paenibacillus在土壤中丰度为(0.843±0.140)%,在第一代和第二代中占比分别高达(76.7±3.9)%和(57.4±9.6)%;Clostridium (土壤中0.12%)在第二代显著增加至(21.9±4.0)%和(24.8±7.7)%;同样值得注意的是,土壤中最优势的Bradyrhizobium和Azospirillum丰度急剧下降至0.038%–0.094%和0.002%–0.006% (图 3-B)。
16S rRNA基因的系统分类表明,土壤中大部分固氮菌门均可通过经典的固体和液体培养基富集生长(图 3-C),但多次传代驯化了特定固氮菌,导致部分固氮菌被遗漏。例如,在微生物门水平,第一代和第二代固体培养基富集后,共检测到12种固氮菌门,包括8个门已知可固氮,而4个门尚未有固氮生理报道。已知8个固氮菌门中Proteobacteria丰度最高,在第一代和第二代中占比分别为(69.30±0.87)%和(81.4±3.4)%,其次是Firmicutes占比分别为(26.7±1.0)%和(8.64±3.60)% (图 3-C)。其他4个未知固氮门包括Acidobacteria、Planctomycetes、Chloroflexi、Gemmatimonadetes,这些微生物门在数量上极为弱势,占固体培养基所有固氮菌丰度的0.05%。液体培养基也得到了类似结果,共检测到12种固氮菌门,包括6个已知门,6个未知门。已知固氮菌门中Firmicutes丰度最高[第一代(80.9±13.0)%;第二代(59.8±18)%]、其次分别为Bacteroidetes [第一代(13.0±12.0)%;第二代(19.0±12.0)%]和Proteobacteria (第一代(6.01±0.96)%;第二代(21.1±7.8)%;未知的6个固氮菌门则包括Acidobacteria、Planctomycetes、Chloroflexi、Gemmatimonadetes、Crenarchaeota、Armatimonadetes),它们在数量上极为弱势,占液体培养基所有固氮菌丰度仅为0.01%。值得注意的是,土壤中含有固体和液体培养基上检测到的所有14个固氮菌门,其中8个为已知固氮菌门,占所有固氮菌丰度为79.7%,Firmicutes最高为(36.1±3.4)%,其次分别为Proteobacteria [(27.0±1.6)%]和Actinobacteria [(12.100±0.061)%]。
进一步基于16S rRNA基因分类,发现土壤中所有255个固氮菌属(图 2)也具有类似规律(图 3-D)。根据相对丰度由高到低选择排名前10优势类群,土壤、固体和液体培养基中共计28个属(图 3-D)。固体培养基中共检测到27个优势固氮菌属富集物,已知的固氮菌属共15个,其中Pseudomonas丰度最高[第一代(59.8±1.4)%,第二代(54.6±15)%],其次为Paenibacillus[(23.1±5.6)%,(14.1±0.43)%];而12个属尚未有固氮生理报道,其丰度分别为14.2%和18.0%,丰度较高的包括Lysobacter [第一代(4.52±0.98)%;第二代(0.335±0.230)%、Pseudoxanthomonas [(4.19±1.1)%;(4.030±0.012)%]、Olivibacter [(2.77±1.30)%;(3.43±2.0)%]。液体培养基27个优势固氮菌属富集物中,15个为已知固氮属,12个属尚未有固氮生理报道。15个已知属中,Paenibacillus丰度最高[第一代(31.3±20)%,第二代(27.7±22)%],其次为Sporolactobacillus [(21.5±6.5)%,(13.0±12)%];所有12个未知固氮菌属中丰度较高的分别为Flavobacterium [(15.7±15)%,(30.3±15)%],Stenotrophomonas [(2.74±0.87)%,(5.87±3.5)%]。此外,土壤中含有所有16个已知固氮菌属和12个未有固氮生理特征的属,16个已知固氮菌属占土壤中所有优势固氮菌丰度为70.1%,其中Bacillus占比最高为30.8%±2.8%,其次为Paenibacillus (15.8%±2.3%),Lysobacter (11.7%±1.1%)。
2.4 可培养固氮菌的富集规律nifH基因分析发现土壤中含有67个固氮菌属,经过两代富集后,固体和液体培养基共获得固氮菌富集物39属,其中固体培养基30属,液体培养基23属;固体和液体共有属14个(图 4-A)。16S rRNA基因分析则发现土壤中含有255固氮菌属,培养基两代富集后共计获得248属富集物,其中固体培养基212个属,而液体培养基164个属;固体和液体共有属128个,然而值得注意的是,仅有22个属已有固氮生理报道,其他226属尚未有固氮报道(图 4-A),占可培养属高达91.1%。同时,绝大部分微生物均为数量上占弱势的稀有属,nifH基因分析表明菌体富集物中69.5%固氮菌属丰度 < 0.1%,42.8%的属 < 0.01%;而16S rRNA分析则发现菌体富集物中78.5%固氮菌属丰度 < 0.1%,50.2%的属 < 0.01%。
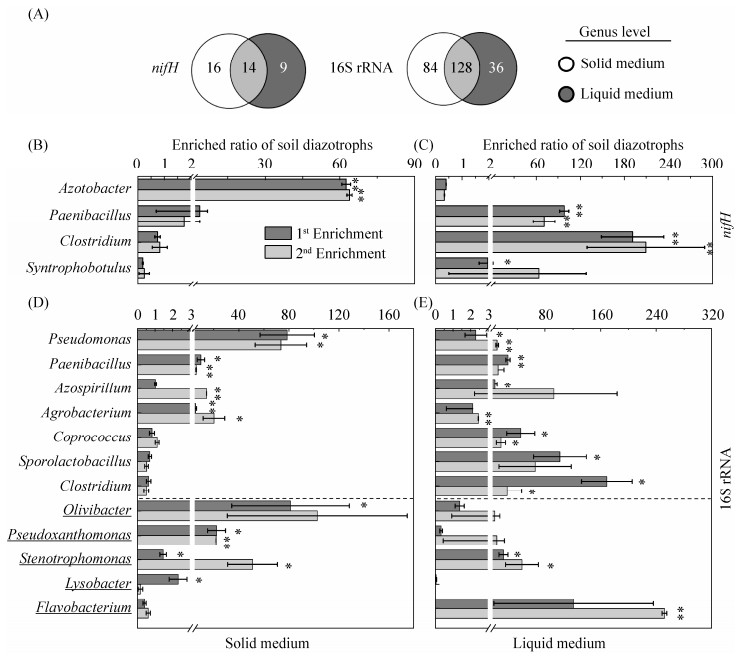
|
| 图 4 土壤固氮菌属在固体和液体培养基中的选择性富集规律 Figure 4 Selective enrichment patterns of culturable diazotrophs in solid and liquid media. A: The common and unique diazotrophs between solid and liquid medium based on nifH and 16S rRNA gene analysis; B: One nitrogen-fixing genus wassignificantly enriched based on analysis of nifH gene in solid media (30 diazotrophs in total) as indicated by the stars above the columns, all other three generas are not in solid medium; C: Three nitrogen-fixing genera were significantly enriched based on analysis of nifH gene in liquid media (23 diazotrophs in total) as indicated by the stars above the columns; D: Eight nitrogen-fixing genera were significantly enriched based on analysis of 16S rRNA gene in soild media (212 nitrogen fixers in total); E: Nine nitrogen-fixing genera were significantly enriched based on analysis of 16S rRNA gene in liquid media (164 nitrogen fixers in total). The underlined genera indicate that the sequences of these genera showed high similariry to bacterium in which N2-fixing physiology has not been demonstrated within the underlined genus. All genera without underlined signs denote at least one of strain within the genus has shown the physiological capability of N2-fixation. All other labels are the same as Figure 2 and Figure 3. |
尽管固体和液体固氮培养基均可富集大量固氮菌,但与背景土壤中固氮菌丰度相比,仅有很少一部分固氮菌被显著富集(图 4)。例如,nifH基因分析表明,固体培养基上的30个属中(图 4-A),仅有Azotobacter属发生了显著富集(图 4-B),其在背景土壤中的丰度为(1.53±0.35)%,而在第一代和第二代固体培养基的丰度分别为(96.6±2.7)%和(98.00±0.94)%,富集率63倍和64倍。液体培养基也得到了类似结果,共计23个固氮菌属富集物中(图 4-A),仅选择性富集了3个固氮菌属(图 4-C),其中Paenibacillus和Clostridium是液体培养基中的优势物种,在土壤中丰度为(0.771± 0.13)%和(0.111±0.035)%,而在第一代培养中丰度分别为(76.6±3.8)%和(21.9±4.0)%,富集倍数高达99.3和192倍,此外,背景土壤中Syntrophothermus丰度仅为0.029%,在第一代培养中显著增加至0.048%,导致其富集率为1.99(图 4-C)。
基于16S rRNA基因的分析也得到类似结果,固体培养基212个固氮菌属富集物中(图 4-A),与土壤中的丰度相比,仅有8个属发生了显著增加(图 4-D),包括4个已知固氮菌属,4个尚未有固氮生理报道的属。已知固氮属中Pseudomonas富集率最高,在土壤中丰度为(0.620±0.14)%,在第一和第二代分别为(49.0±13)%和(45.5±7.1)%,富集率高达79和74倍;另外3个显著富集的已知固氮属分别为Paenibacillus (第一代10倍,第二代6.2倍)、Agrobacterium(5.67倍;20倍)和Azospirillum (第二代15倍),在土壤中的丰度分别为1.89%、0.30%、0.06%。此外,4个未知固氮属的富集倍数分别为Olivibacter (第一代82倍;第二代103倍)、Pseudoxanthomonas (23倍;22倍)、Stenotrophomonas (1.48,51.3)和Lysobacter (第一代2.33),这些未知固氮属在土壤中的丰度分别为0.03%、0.15%、0.11%、1.41%,经过富集培养后在第一代液体培养基中占比共计11.6%,第二代则增至14.5% (图 3-D)。
类似的,液体培养基共计164个固氮属中(图 4-A),与土壤中丰度相比,仅有9个属发生了显著富集(图 4-E),包括7个已知固氮菌属,2个尚未有固氮生理报道的属。已知固氮属中富集率最高的为第一代Clostridium属(169倍),在土壤和培养基中的丰度分别为(0.0980±0.0094)%和(16.6±3.6)%;其他显著富集的6个固氮菌属分别为Sporolactobacillus (102倍;66.2倍)、Coprococcus (45.6;17.1)、Paenibacillus (13.6;12.9)、Azospirillum (8.32;93.2)、Pseudomonas (2.32;11.5)、Agrobacterium (2.16;2.47);它们在土壤中的丰度分别为0.17%、0.06%、1.89%、0.06%、0.62%、0.30%。两种未知固氮属中,Flavobacterium富集率最高(122倍;252倍),其次为Stenotrophomonas (20.6;47.3),它们在土壤中的丰度仅为0.11%,在第二代液体培养基中丰度高达26.8%、5.17%,在所有未知固氮属中丰度最高(图 3-D)。
3 讨论“99%难培养”通常意味着绝大部分微生物尚未获得纯培养。事实上,连续的传代过程中,代时长且生理功能多样的微生物通常会处于竞争劣势,进而被易生长的微生物所淘汰。本研究假设所有可利用人工营养配方的固氮菌均可富集并形成菌落,同时,第一代和第二代培养基的菌落集合,能够在最大程度代表潜在的、可利用人工培养配方的可培养纯菌株。同时,选择第一代和第二代微生物菌落集合可规避连续传代可能引起的多样性丧失。进一步利用固氮菌分子标靶基因nifH和16S rRNA基因引物,高通量测序菌落DNA和土壤DNA并进行比较分析。结果表明,在目前技术条件下,土壤中可培养固氮菌属的比例 > 22.4%。
尽管本研究中可培养固氮菌定义为所有可培养纯菌株的集合,但也不能完全排除个别微生物为非固氮菌或者必须通过互利共生的非固氮菌。研究中所用培养基中未添加无机氮,所有试剂中也未检测到无机氮,稳定同位素示踪也表明固氮菌富集物同化了15N2,同时nifH/16S rRNA的比例在传代中保持相对恒定,表明固氮菌富集物群落组成未发生显著改变。然而,尽管固体培养基菌落的16S rRNA基因与12个门高度同源,但其中4个门尚未有固氮生理报道(Acidobacteria、Planctomycetes、Chloroflexi、Gemmatimonadetes),而液体培养基更检测到非固氮菌Crenarchaeota、Armatimonadetes门的16S rRNA基因同源序列。这些微生物迄今尚未被分离培养的可能原因是,传统微生物分离纯化过程中,平板挑选或液体转接具有极大的随机性,会遗漏代时较长、生长缓慢的非优势固氮菌[26]。同时,这些固氮菌可能属于营养缺陷体,需要与其他细菌共生获得营养物质,如钴胺素、生物素等[33-35]。然而,本研究也不能排除这些微生物是非固氮菌。例如,培养基中的固氮菌可能会分泌15N-化合物,作为营养物促进非固氮菌的生长,同样导致非固氮菌基因组被15N2所标记。事实上,针对西伯利亚落叶松林冻土层土壤的研究发现,将非固氮纯菌株与固氮
菌株混合培养后,可显著增加土壤固氮量[36]。未来通过优化固氮培养基和富集策略,获得这些与非固氮菌16S rRNA基因高度同源的纯菌株具有重要意义。
固体和液体培养中数量上优势的固氮菌属中绝大部分为共有属,而弱势固氮菌中绝大部分为二者的特有属。nifH基因分析表明其中数量上优势(相对丰度排名前十,> 94.2%)的19个属中(图 3-B),固体、液体培养基共有14个属,固体培养基特有4个属,分别为Hyphomicrobium、Burkholderia、Cupriavidus和Pseudomonas (相对丰度由土壤中0.001%–3.760%降为0.0005%–0.0800%),液体培养基特有的1个属Ethanoligenens (相对丰度由土壤中0.001%富集为0.001%–0.030%);数量上弱势的20个属中,未发现共有属,固体培养基特有12个属,液体培养基特有的8个属,弱势菌株丰度均未发生显著富集。16S rRNA基因分析表明数量上优势(相对丰度排名前十,> 81.1%)的28个属中(图 3-D),固体、液体培养基共有属26个属,固体培养基特有1个属Hyphomicrobium (丰度由土壤中0.15%降为(0.001±0.001)%),液体培养基特有的1个属Nitrospira (丰度由土壤中0.33%降为(0.001±0.001)%)。数量上弱势的220个属中,固体、液体培养基共有102个属(土壤中相对丰度总和为0.65%,液体和固体培养基中分别下降为0.13%和0.19%),固体培养基特有82个属,液体培养基特有36个属。这些数量上占弱势、与背景土壤相比未被显著富集但却能在培养基形成菌落生长的固氮菌,是未来定向培养分离固氮菌资源的重要内容。
固体和液体培养基分别富集了不同的固氮菌,造成这一差异的原因可能包括氧气、琼脂、附着物环境差异。在属水平,nifH基因分析表明固体培养基选择了Azotobacter (96±2.94%),而液体培养基选择了Paenibacillus (54.63±9.15)%和Clostridium (27.21±4.59)%。固体培养基中菌落与空气接触,氧气浓度约为19%,而液体培养基中溶解氧浓度通常约为0.1%。已有报道表明Azotobacter具有好氧生长的固氮功能,可能通过细胞团聚形成厌氧微域,防止氧气阻遏固氮酶合成及对固氮活性的影响,而Paenibacillus和Clostridium适应氧气胁迫能力更强。此外,尽管理论上琼脂不可被微生物代谢利用,但也有研究发现中性或碱性条件下,磷酸盐和琼脂混合后经高温灭菌后会产生H2O2双氧水[37-38],而液体培养基中磷酸盐单独加热则不会产生H2O2,H2O2对微生物生长有明显的抑制作用[37],也可能是造成固体培养与液体培养之间差异原因的。然而,这些研究大多针对纯菌株的生理生化和遗传学研究[39],随着高通量测序成本急剧下降,研究氧气对固氮菌富集培养过程的功能调控影响具有重要意义,如胞内生物氧化与ATP合成、特定防氧机制如呼吸保护、构象保护和分隔保护等[40]对氧气的正负反馈机制,以及琼脂的可能利用机制等。此外,固体和液体培养基的环境特征也并不相同,固体平板表面为微生物提供了附着物,更利于可培养固氮微生物群落形成由胞外多聚基质联合的微生物被膜[41],进一步改变固氮菌群落之间的群体感应过程[42],从而影响固氮菌及其共生微生物的生理生态行为。
我们的研究结果为最近关于“99%难培养”的论断提供了明确的实验证据。通过整合已有的文献数据,Martiny推测可培养微生物占比高达52%–74%[43]。针对6个生境中40个微生物群落的Sanger测序16S rRNA基因,每个群落中随机抽取了100条全长序列(约1.5 kb)并与RDP (Ribosome Data Base)数据库中的纯菌株序列比对分析,发现可培养微生物的细胞和物种占比分别高达(52.0± 24)%和(34.9±23.0)%[43]。这一结果引起了较大的争议,Steen等重新分析了该论文的数据,认为可培养微生物占比约为2%–18%[44]。两者的差异主要在于所用分析软件的可靠性、16S rRNA基因数据库的差异、系统发育分类的阈值等。然而,这些研究均未开展实验室富集培养,存在较大不确定性。我们的结果则表明,单纯的16S rRNA基因序列并不能反映生理功能,序列相似度与可培养性之间并没有必然联系,相当高比例的微生物可被培养,但尚未分离获得纯菌株。
我们的结果也表明,传统富集培养与系统发育的耦合分析更有意义。尽管系统发育分类方法具有极强的实践操作性,但其生理生化意义有限。例如,即使同一微生物种的不同菌株,其16S rRNA同源性高于99%,但其表型也可能存在很大差异[45],因此,无论是16S rRNA基因或核心基因组,单纯的DNA序列同源性并不能作为细菌分类的唯一标准[46]。同时,环境样品DNA提取、PCR扩增、文库构建、高通量测序等等一系列分子分析过程,均可能引入大量的误差,例如,尽管本研究所用引物对PolF/PolR特异性高,广泛应用于固氮微生物研究近二十年,却也有一定的偏好性,对一些土壤类型中的固氮菌覆盖度仅为51%[27, 47],同时,16S rRNA相似性 > 97%的所有固氮菌基因组中,43%的nifH基因同源性低于85%[48],而本研究中nifH和16S rRNA基因的固氮菌系统分类结果也并不完全一致。已有研究表明,基于特征性分子标靶基因片段扩增子得到的OTU不足以代表含有该特征片段的所有微生物,特别是稀有物种,其标靶基因序列可能与已知菌株具有较大差异而无法被现有方法检测[49]。事实上,本研究发现相当比例的可培养固氮菌富集物并未在土壤DNA被检测。此外,PCR扩增及高通量测序中也可能产生一些稀有序列变异体,进而高估了物种多样性。经典分类学则能较好地弥补系统分类的缺点,表型分析能够最大程度反映菌株的生理功能,而纯菌株标靶基因和组学数据的积累则可形成完善的数据库,为系统发育分类学推测未培养微生物功能提供依据。例如,在严格固氮培养条件下,研究发现22%纯培养固氮菌未检测到nifH基因,组学分析则能提供关键固氮酶的遗传信息,并与生理特性形成更好的互补[50]。值得注意的是,“不可培养微生物”的表述有待商榷,“尚未培养”或“难培养”微生物更为合理。因为在某个菌株被培养前,无法判断其可培养性,因此,未来应开展更大规模的富集培养与系统分类研究,定量评估高通量测序的系统性误差,构建纯菌株表型和组学特征为核心的数据库,结合系统发育进化分析,更好地促进难培养微生物的分离。
4 结论通过文献整合分析、传统富集培养和标靶基因的系统发育分子分类研究,得到如下结论。
(1) 文献分析表明“99%难培养”即为经典的“平板计数异常”同义词。
(2) “不可培养”微生物的表述有待商榷,“尚未培养”或“难培养”微生物更为合理。因为某个菌株被培养前无法判断其可培养性,特别是绝大多数数量上占弱势的稀有物种,目前条件下无法穷尽所有人工培养配方开展可培养研究。
(3) 土壤中可培养固氮菌占比高达97.3%,但其中91.1%属尚未获得纯培养或固氮生理报道。基于16S rRNA基因分析表明,土壤中固氮菌共计255属,其中248属在固体和液体培养基可形成菌落被培养,但高达226属尚未获得纯培养或固氮生理报道。nifH基因则发现土壤中固氮菌共67属,而39属在固体和液体培养基可形成菌落被培养,占比高达58.2%。
(4) 土壤中绝大部分固氮菌为稀有物种,> 40%的固氮菌属丰度 < 0.01%。基于16S rRNA基因分析表明,固体和液体富集物中78.5%固氮菌属丰度 < 0.1%,50.2%属 < 0.01%;而nifH基因分析发现69.5%固氮菌属丰度 < 0.1%,42.8%的属 < 0.01%。
(5) 传统分离方法遗漏的固氮菌属占比高达95.2%。16S rRNA基因分析发现,与背景土壤中固氮菌相比,固体和液体可培养248属中,仅有12属的丰度统计显著增加;nifH基因分析则发现可培养39属中,仅4属的丰度显著增加。固体和液体培养基具有极强的选择性,分别富集了Proteobacteria和Firmicutes的优势固氮菌门;固体仅富集了Azotobacter优势属,而液体富集了Paenibacillus和Clostridium优势属。连续传代过程中选择性遗漏了大量尚未获得分离的固氮菌。
(6) 亟需发展先进分离方法获得纯培养固氮菌株。本研究仅基于固氮菌这一特定功能群,其他类群的微生物可培养比例可能存在差别,未来应针对生理类群多样的微生物开展更多研究;综合不同学科先进技术,如富集分离、分类鉴定、生理表型和系统发育相关研究,为定向发掘重要微生物资源及功能提供参考。
| [1] | Woese CR, Fox GE. Phylogenetic structure of the prokaryotic domain: the primary kingdoms. Proceedings of the National Academy of Sciences of the United States of America, 1977, 74(11): 5088-5090. DOI:10.1073/pnas.74.11.5088 |
| [2] | Woese CR, Kandler O, Wheelis ML. Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya. Proceedings of the National Academy of Sciences of the United States of America, 1990, 87(12): 4576-4579. DOI:10.1073/pnas.87.12.4576 |
| [3] | Olsen GJ, Lane DJ, Giovannoni SJ, Pace NR, Stahl DA. Microbial ecology and evolution: a ribosomal RNA approach. Annual Review of Microbiology, 1986, 40: 337-365. DOI:10.1146/annurev.mi.40.100186.002005 |
| [4] | Giovannoni SJ, Britschgi TB, Moyer CL, Field KG. Genetic diversity in Sargasso Sea bacterioplankton. Nature, 1990, 345(6270): 60-63. DOI:10.1038/345060a0 |
| [5] | Marcy Y, Ouverney C, Bik EM, Lösekann T, Ivanova N, Martin HG, Szeto E, Platt D, Hugenholtz P, Relman DA, Quake SR. Dissecting biological "dark matter" with single-cell genetic analysis of rare and uncultivated TM7 microbes from the human mouth. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(29): 11889-11894. DOI:10.1073/pnas.0704662104 |
| [6] |
Huang L, Feng XL, Du QS, Dong XZ, Liu SJ, Wen MZ, Dai X. Focusing on key scientific issues of microbiome research in hydrosphere: NSFC major research plan for microbes in hydrosphere. Bulletin of Chinese Academy of Sciences, 2017, 32(3): 266-272.
(in Chinese) 黄力, 冯雪莲, 杜全生, 东秀珠, 刘双江, 温明章, 戴欣. 水圈微生物重大研究计划: 聚焦水圈微生物组研究的核心科学问题. 中国科学院院刊, 2017, 32(3): 266-272. |
| [7] |
Zhu YG, Shen RF, He JZ, Wang YF, Han XG, Jia ZJ. China soil microbiome initiative: progress and perspective. Bulletin of Chinese Academy of Sciences, 2017, 32(6): 554-565.
(in Chinese) 朱永官, 沈仁芳, 贺纪正, 王艳芬, 韩兴国, 贾仲君. 中国土壤微生物组: 进展与展望. 中国科学院院刊, 2017, 32(6): 554-565. |
| [8] | Locey KJ, Lennon JT. Scaling laws predict global microbial diversity. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(21): 5970-5975. DOI:10.1073/pnas.1521291113 |
| [9] | Mora C, Tittensor DP, Adl S, Simpson AGB, Worm B. How many species are there on earth and in the Ocean?. PLoS Biology, 2011, 9(8): e1001127. DOI:10.1371/journal.pbio.1001127 |
| [10] | Parte AC. LPSN-List of Prokaryotic names with Standing in Nomenclature (bacterio. net), 20 years on. International Journal of Systematic and Evolutionary Microbiology, 2018, 68(6): 1825-1829. DOI:10.1099/ijsem.0.002786 |
| [11] | Willis A. Extrapolating abundance curves has no predictive power for estimating microbial biodiversity. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(35): E5096. DOI:10.1073/pnas.1608281113 |
| [12] | Louca S, Mazel F, Doebeli M, Parfrey LW. A census-based estimate of Earth's bacterial and archaeal diversity. PLoS Biology, 2019, 17(2): e3000106. DOI:10.1371/journal.pbio.3000106 |
| [13] | Benson DA, Karsch-Mizrachi I, Lipman DJ, Ostell J, Sayers EW. GenBank. Nucleic Acids Research, 2008, 37(S1): D26-D31. |
| [14] | Amann RI, Ludwig W, Schleifer KH. Phylogenetic identification and in situ detection of individual microbial cells without cultivation. Microbiological Reviews, 1995, 59(1): 143-169. DOI:10.1128/MR.59.1.143-169.1995 |
| [15] | Ward DM, Weller R, Bateson MM. 16S rRNA sequences reveal numerous uncultured microorganisms in a natural community. Nature, 1990, 345(6270): 63-65. DOI:10.1038/345063a0 |
| [16] | Staley JT, Konopka A. Measurement of in situ activities of nonphotosynthetic microorganisms in aquatic and terrestrial habitats. Annual Review of Microbiology, 1985, 39: 321-346. DOI:10.1146/annurev.mi.39.100185.001541 |
| [17] | Winterberg H. Zur Methodik der Bakterienzählung. Zeitschrift für Hygiene und Infektionskrankheiten, 1898, 29(1): 75-93. DOI:10.1007/BF02217377 |
| [18] | Razumov AS. The direct method of calculation of bacteria in water: comparison with the Koch method. Mikrobiologija, 1932, 1: 131-146. |
| [19] | Thornton HG, Gray PHH. The numbers of bacterial cells in field soils, as estimated by the ratio method. Proceedings of the Royal Society B: Biological Sciences, 1934, 115(795): 522-543. |
| [20] | Skinner FA, Jones PCT, Mollison JE. A comparison of a direct- and a plate-counting technique for the quantitative estimation of soil micro-organisms. Microbiology, 1952, 6(3/4): 261-271. |
| [21] | Xu HS, Roberts N, Singleton FL, Attwell RW, Grimes DJ, Colwell RR. Survival and viability of nonculturable Escherichia coli and Vibrio cholerae in the estuarine and marine environment. Microbial Ecology, 1982, 8(4): 313-323. DOI:10.1007/BF02010671 |
| [22] | Kämpfer P, Steiof M, Dott W. Microbiological characterization of a fuel-oil contaminated site including numerical identification of heterotrophic water and soil bacteria. Microbial Ecology, 1991, 21(1): 227-251. DOI:10.1007/BF02539156 |
| [23] | Fowler D, Coyle M, Skiba U, Sutton MA, Cape JN, Reis S, Sheppard LJ, Jenkins A, Grizzetti B, Galloway JN, Vitousek P, Leach A, Bouwman AF, Butterbach-Bahl K, Dentener K, Stevenson D, Amann M, Voss M. The global nitrogen cycle in the twenty-first century. Philosophical Transactions of the Royal Society B: Biological Sciences, 2013, 368(1621): 20130164. DOI:10.1098/rstb.2013.0164 |
| [24] | Vitousek PM, Menge DNL, Reed SC, Cleveland CC. Biological nitrogen fixation: rates, patterns and ecological controls in terrestrial ecosystems. Philosophical Transactions of the Royal Society B: Biological Sciences, 2013, 368(1621): 20130119. DOI:10.1098/rstb.2013.0119 |
| [25] | Zhang JB, Zhu TB, Cai ZC, Qin SW, Müller C. Effects of long-term repeated mineral and organic fertilizer applications on soil nitrogen transformations. European Journal of Soil Science, 2012, 63(1): 75-85. DOI:10.1111/j.1365-2389.2011.01410.x |
| [26] | Baldani JI, Reis VM, Videira SS, Boddey LH, Baldani VLD. The art of isolating nitrogen-fixing bacteria from non-leguminous plants using N-free semi-solid media: a practical guide for microbiologists. Plant and Soil, 2014, 384(1/2): 413-431. DOI:10.1007/s11104-014-2186-6 |
| [27] | Poly F, Monrozier LJ, Bally R. Improvement in the RFLP procedure for studying the diversity of nifH genes in communities of nitrogen fixers in soil. Research in Microbiology, 2001, 152(1): 95-103. DOI:10.1016/S0923-2508(00)01172-4 |
| [28] | Christner BC, Mosley-Thompson E, Thompson LG, Reeve JN. Isolation of bacteria and 16S rDNAs from Lake Vostok accretion ice. Environmental Microbiology, 2001, 3(9): 570-577. DOI:10.1046/j.1462-2920.2001.00226.x |
| [29] |
Jia ZJ. Principle and application of DNA-based stable isotope probing——A review. Acta Microbiologica Sinica, 2011, 51(12): 1585-1594.
(in Chinese) 贾仲君. 稳定性同位素核酸探针技术DNA-SIP原理与应用. 微生物学报, 2011, 51(12): 1585-1594. |
| [30] | Wang Q, Quensen Ⅲ JF, Fish JA, Lee TK, Sun YN, Tiedje JM, Cole JR. Ecological Patterns of nifH Genes in four terrestrial climatic zones explored with targeted metagenomics using FrameBot, a new informatics tool. mBio, 2013, 4(5): e00592-13. |
| [31] | Wang Q, Garrity GM, Tiedje JM, Cole JR. Naive Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy. Applied and Environmental Microbiology, 2007, 73(16): 5261-5267. DOI:10.1128/AEM.00062-07 |
| [32] | Yarza P, Yilmaz P, Pruesse E, Glöckner FO, Ludwig W, Schleifer KH, Whitman WB, Euzéby J, Amann R, Rosselló-Móra R. Uniting the classification of cultured and uncultured bacteria and archaea using 16S rRNA gene sequences. Nature Reviews Microbiology, 2014, 12(9): 635-645. DOI:10.1038/nrmicro3330 |
| [33] | Zengler K, Zaramela LS. The social network of microorganisms-how auxotrophies shape complex communities. Nature Reviews Microbiology, 2018, 16(6): 383-390. DOI:10.1038/s41579-018-0004-5 |
| [34] | Degnan PH, Taga ME, Goodman AL. Vitamin B12 as a modulator of gut microbial ecology. Cell Metabolism, 2014, 20(5): 769-778. DOI:10.1016/j.cmet.2014.10.002 |
| [35] | Mu DS, Liang QY, Wang XM, Lu DC, Shi MJ, Chen GJ, Du ZJ. Metatranscriptomic and comparative genomic insights into resuscitation mechanisms during enrichment culturing. Microbiome, 2018, 6(1): 230. DOI:10.1186/s40168-018-0613-2 |
| [36] | Hara S, Desyatkin RV, Hashidoko Y. Investigation of the mechanisms underlying the high acetylene-reducing activity exhibited by the soil bacterial community from BC2 horizon in the permafrost zone of the East Siberian larch forest bed. Journal of Applied Microbiology, 2014, 116(4): 865-876. DOI:10.1111/jam.12424 |
| [37] | Tanaka T, Kawasaki K, Daimon S, Kitagawa W, Yamamoto K, Tamaki H, Tanaka M, Nakatsu CH, Kamagata Y. A hidden pitfall in the preparation of agar media undermines microorganism cultivability. Applied and Environmental Microbiology, 2014, 80(24): 7659-7666. DOI:10.1128/AEM.02741-14 |
| [38] | Kawasaki K, Kamagata Y. Phosphate-catalyzed hydrogen peroxide formation from agar, gellan, and κ-carrageenan and recovery of microbial cultivability via catalase and pyruvate. Applied and Environmental Microbiology, 2017, 83(21): e01366-17. |
| [39] | Reed SC, Cleveland CC, Townsend AR. Functional ecology of free-living nitrogen fixation: a contemporary perspective. Annual Review of Ecology, Evolution, and Systematics, 2011, 42: 489-512. DOI:10.1146/annurev-ecolsys-102710-145034 |
| [40] | Robson RL, Postgate JR. Oxygen and hydrogen in biological nitrogen fixation. Annual Review of Microbiology, 1980, 34: 183-207. DOI:10.1146/annurev.mi.34.100180.001151 |
| [41] | McDougald D, Rice SA, Barraud N, Steinberg PD, Kjelleberg S. Should we stay or should we go: mechanisms and ecological consequences for biofilm dispersal. Nature Reviews Microbiology, 2012, 10(1): 39-50. DOI:10.1038/nrmicro2695 |
| [42] | Mukherjee S, Bassler BL. Bacterial quorum sensing in complex and dynamically changing environments. Nature Reviews Microbiology, 2019, 17(6): 371-382. DOI:10.1038/s41579-019-0186-5 |
| [43] | Martiny AC. High proportions of bacteria are culturable across major biomes. The ISME Journal, 2019, 13(8): 2125-2128. DOI:10.1038/s41396-019-0410-3 |
| [44] | Steen AD, Crits-Christoph A, Carini P, DeAngelis KM, Fierer N, Lloyd KG, Cameron Thrash J. High proportions of bacteria and archaea across most biomes remain uncultured. The ISME Journal, 2019, 13(12): 3126-3130. DOI:10.1038/s41396-019-0484-y |
| [45] | Sun DL, Jiang X, Wu QL, Zhou NY. Intragenomic heterogeneity of 16s rRNA genes causes overestimation of prokaryotic diversity. Applied and Environmental Microbiology, 2013, 79(19): 5962-5969. DOI:10.1128/AEM.01282-13 |
| [46] | Chun J, Oren A, Ventosa A, Christensen H, Arahal DR, da Costa MS, Rooney AP, Yi HN, Xu XW, De Meyer S, Trujillo ME. Proposed minimal standards for the use of genome data for the taxonomy of prokaryotes. International Journal of Systematic and Evolutionary Microbiology, 2018, 68(1): 461-466. DOI:10.1099/ijsem.0.002516 |
| [47] | Gaby JC, Buckley DH. A comprehensive evaluation of PCR primers to amplify the nifH gene of nitrogenase. PLoS One, 2012, 7(7): e42149. DOI:10.1371/journal.pone.0042149 |
| [48] | Gaby JC, Buckley DH. A comprehensive aligned nifH gene database: a multipurpose tool for studies of nitrogen- fixing bacteria. Database, 2014, 2014: bau001. |
| [49] | Laehnemann D, Borkhardt A, McHardy AC. Denoising DNA deep sequencing data-high-throughput sequencing errors and their correction. Briefings in Bioinformatics, 2016, 17(1): 154-179. DOI:10.1093/bib/bbv029 |
| [50] | Mirza BS, Rodrigues JLM. Development of a direct isolation procedure for free-living diazotrophs under controlled hypoxic conditions. Applied and Environmental Microbiology, 2012, 78(16): 5542-5549. DOI:10.1128/AEM.00714-12 |
 2021, Vol. 61
2021, Vol. 61