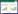中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 杜梦璇, 姜民志, 刘畅, 刘双江. 2021
- Du Mengxuan, Jiang Minzhi, Liu Chang, Liu Shuangjiang. 2021
- 肠道微生物菌株资源库的构建与应用开发
- Gut Microbial BioBanks: Construction and Applications
- 微生物学报, 61(4): 875-890
- Acta Microbiologica Sinica, 61(4): 875-890
-
文章历史
- 收稿日期:2020-11-29
- 修回日期:2021-02-04
- 网络出版日期:2021-02-10
2. 山东大学微生物技术国家重点实验室, 山东 青岛 266237
2. State Key Laboratory of Microbial Technology, Shandong University, Qingdao 266237, Shandong Province, China
肠道微生物组具有群落结构复杂、数量庞大、多样性高、功能丰富等特点,大量的研究表明,肠道微生物组广泛参与宿主的神经、生理、代谢、免疫等各种生命过程和生理活动,与宿主健康息息相关;肠道微生物菌群失去平衡,可以引发机体代谢紊乱和肠道炎症等疾病[1-4]。人体作为宿主,为种类繁多的微生物提供了良好的栖息环境[5]。在不同年龄、体重、性别、地域的健康人群中,肠道微生物组成可以分为3种肠型,即拟杆菌型、普氏菌型及瘤胃球菌型[6]。在肠道环境中,不同肠型的出现与宿主的饮食习惯密切相关,宿主的饮食习惯和食物结构组成以及宿主与肠道微生物的相互作用,决定了肠道中微生物菌群,肠道菌群结构和组成相对稳定。肠道菌群一旦失去平衡,会影响宿主的许多生理功能,其中包含新陈代谢及炎症的发生[3-4],特别是脑–肠轴的提出,拓宽了我们对肠道微生物组成变化引起疾病的认识[7]。鉴于肠道微生物组具有高度复杂性,对其进行研究的策略也是多种多样,根据研究手段不同,大致可分为基于免培养的多组学技术和基于可培养菌株资源两种研究策略。得益于高通量测序和质谱等技术的飞速发展[8],肠道微生物的多组学研究发展迅猛,产生了海量的组学数据,研究人员通过数据挖掘和结果分析,发现了不同人群的菌群差异、以及个人肠道菌群的纵向动态变化等,根据已知功能基因的预测,还可以对肠道微生物宏基因组的功能进行预测。过去的10余年中,尽管此类不依赖微生物培养(免培养技术)的研究是肠道微生物研究的热点,并揭示了肠道菌群与宿主健康和疾病存在着关联性,但此类研究不能揭示肠道菌群与健康或者疾病的因果关系。肠道微生物菌株资源是研究肠道菌群与宿主健康或者疾病的因果关系、揭示肠道微生物与宿主互作的机制必不可少的材料[9-11],正如2016年Vilanova等在其综述文章中描述:仅仅依靠基于免培养的组学技术不足以阐明肠道菌群这样一个复杂群落,还需要通过提高肠道微生物的培养技术和增加可培养菌株资源,将免培养组学技术与基于可培养的研究策略有机结合起来,才能实现肠道微生物组研究的深入发展和对多组学数据的精确解析[12]。近年来肠道微生物大规模分离培养的工作越来越受到研究人员的关注,并取得了良好的进展[13-14];这些研究不仅证明了肠道可培养微生物菌株是研究肠道菌–宿主互作机制、开发利用功能菌株、实现靶向肠道干预宿主健康的资源基础,还是提高宏基因组等组学数据挖掘深入和精度的钥匙[15-16]。本文结合作者对肠道微生物分离培养工作的经验,总结近年来国内外完成的一些具有代表性的肠道微生物大规模分离培养和菌株资源库构建的工作结果,在回顾肠道微生物分离培养技术的研究进展和利用功能菌株靶向调控肠道菌干预疾病的研究实例的基础上,探讨未来培养肠道微生物菌株资源的技术发展方向以及肠道微生物的开发潜力。
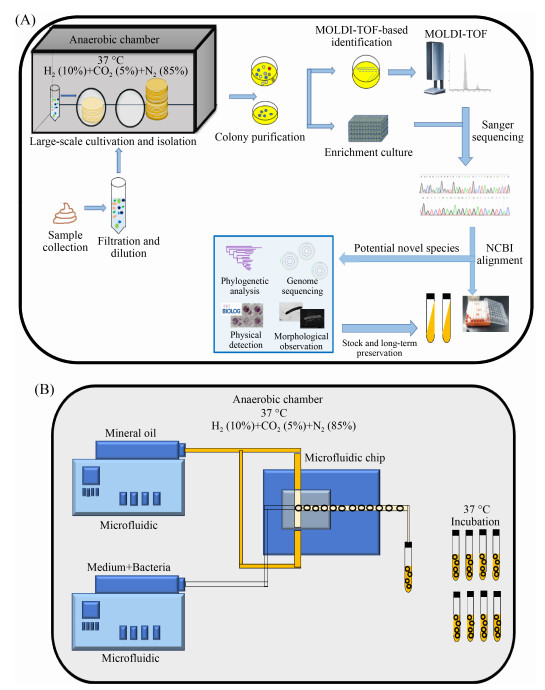
1 肠道微生物大规模分离培养方法的研究进展微生物的大规模分离培养从流程上大致可分为样品前处理、细胞分离与培养、获得菌株的分类学鉴定和菌种保存,如图 1-A。粪便样品前处理的方法根据实验需求而有所不同,常用的前处理方法包括过滤、预培养、酒精处理、热处理等[17-18]。处理后的样品进行菌株的分离培养工作,肠道微生物分离培养目前采用的方法包括传统的梯度稀释平板涂布法和基于微流控的单细胞分离培养方法,2种方法各有优劣。传统的分离培养方法是将前处理样品进行不同浓度梯度的稀释,然后涂布于选取的培养基平板上,培养不同时间之后对平板上生长的单菌落进行挑取和转接扩大培养后进行物种鉴定[19-22]。目前该种方法尽管耗时耗力,但仍然是肠道微生物分离培养最常用的方法之一[23]。Liu等和Zou等运用该分离培养方法对粪便样品进行大规模分离培养,同时通过设计不同的样品方式、培养基组成和培养条件的组合,从粪便中分离获得了丰富多样的肠道微生物[16, 18]。此外,Lagier等用了70种不同的组合条件,分离获得了上千种肠道微生物,并确定了18种最优的培养条件组合[23-25]。另外一种相较于传统平板涂布法具有更高通量的方法是基于微流控高通量的分离培养方法[26],该方法可以实现单细胞水平的分离与培养,可以更好的获得对低丰度稀有菌株的有效分离,并且更容易通过定向设计培养条件实现目标菌株的定向获取,工作原理图如图 1-B所示。微流控分离微生物优点是通过微液滴中的单细胞培养,避免因优势微生物快速生长抢占生态位而导致的慢生长微生物无法形成肉眼可见菌落,甚至可以通过反向基因组学[27],设计筛选目的微生物特异性引物或者探针对其及进行大批量筛选,获得更多未培养肠道微生物[28-29]。但是,该方法同样存在缺点,众所周知复杂环境中存在一些微生物的生长需要依赖于微生物间共生或接触关系,而微流控方法所采用的极限稀释法会使得一部分依赖共生的微生物在单细胞水平缺乏共生对象而无法被分离获取。一直以来,微生物的分离培养受到多种因素的制约,包括复杂且不确定的分离培养条件,缺乏复苏休眠微生物的手段等。为了应对这些问题,创新性的培养方法也层出不穷,除上述介绍的方法外,还有包括基于膜扩散的培养方法和基于细胞分选的培养方法等[30],并且部分技术已经成功用于分离新的微生物。
|
| 图 1 基于传统平板涂布(A)和高通量微流控(B)肠道微生物分离–培养–鉴定流程图 Figure 1 The workflows of Petri dish–based (A) and high–throughput microfluidic–based (B) gut microbial isolation and identification. |
在分离获得纯培养菌株之后,需要对菌株的分类学地位进行鉴定。目前在肠道微生物分离培养最常采用的方法是通过对纯菌株的16S rRNA基因全长进行测序和16S rRNA基因库比对注释[11]。该方法利用27F/1492R通用引物扩增微生物16S rRNA全长并测序,通过与NCBI (https://www.ncbi.nlm.nih.gov/)和EzBiocloud (https://www.ezbiocloud.net/)数据库进行比对,可以获得较权威的物种分类信息。当遇到之前未被鉴定过的潜在新物种时,也可以直接利用其16S rRNA基因全长序列进行系统发育树的构建,描述各潜在新种及其密切相关物种的系统发育,对其分类学地位进行大致的评估。除了利用16S rRNA基因的鉴定方法以外,以Schubert等人为代表的研究团队,也将基质辅助激光解吸/电离飞行时间质谱法(MALDI-TOF MS)[31]用于肠道微生物的大批量鉴定中,其工作原理如图 1-A所示。该种方法通过测定和比对“多肽指纹图谱”,对目标纯培养菌株分类学地位进行初步鉴定。这一方法相较于测序法,具有高通量、经济等优点,通过对初步鉴定结果进行比对筛选后,研究人员可以进一步挑取其中感兴趣的菌株进行鉴定和后续的分析[32]。这种分离鉴定方法已经辅助许多大规模分离培养工作的开展,依赖于该鉴定方法建立起来的人肠道大规模分离培养工作流程也被称作人肠道的“培养组学”[8, 23, 31]。最后要介绍的是一种新型的菌株鉴定方法—拉曼光谱鉴定法,该方法利用微生物中蛋白质、核苷酸、脂质等物质生成的“全生物指纹图谱”[33],进行快速、高效、实时的微生物鉴定[34]。与其他方法相比,拉曼光谱法鉴定微生物的优点在于可以鉴定混合样品。然而,由于拉曼光谱法用于微生物菌株鉴定时间较短,可比对的指纹图谱数据库包含的物种多样性相较于16S rRNA基因数据库和肽指纹图谱库都要低很多,因此鉴定的准确度和鉴定范围都相较于前两种方法差很多,仍需要研究人员通过后续的工作进行不断完善和补充,方能使得该方法得到更好的发展和应用[33]。
目前,还没有专门针对肠道微生物菌种的保藏技术和方法,从粪便中分离获得的纯培养菌株可以通过甘油法、冷冻干燥法或者毛细管法进行长期保存,供后续复苏和开展实验使用。但是,许多从事肠道微生物分离培养的研究者已经发现,传统的甘油法、冷冻干燥等微生物保藏技术,对于肠道微生物并不适用,相当比例的肠道微生物在保藏之后不能复活,研究针对肠道微生物的保藏技术和方法,是一个十分重要和迫切的课题。
2 人的肠道微生物可培养菌株资源现状人的肠道微生物组与人类健康密切相关,一直以来受到研究人员的关注,肠道微生物的大规模分离培养工作也多数集中于对人源样本系统开展[35-36]。对于人肠道微生物的大规模分离培养,早在2010年人类微生物组计划(Human Microbiome Project,HMP)启动之时就已经开始,该计划从人体各部位获取不同微生物进行全基因组测序,目前测序2000多个微生物基因组,其中包括437个人类肠道微生物基因组[37]。2011年,Andrew等在严格厌氧条件下,利用改良GMM培养基,对2个粪便样品中的厌氧微生物进行大规模的分离培养[36]。为避免手动挑取单克隆,研究人员利用稀释高通量写板的方式分离培养微生物,利用大量384孔板对微生物进行分离培养,共获得1170株微生物,分属于15个科、23个属、48个种,用来分析不同微生物对营养物质的利用差异[36]。随后的2012年,Didier R团队对人粪便样本进行了大规模分离培养,研究人员前后采用了212种不同的培养条件,共分离培养了341个菌种,其中一半以上为首次在肠道中分离发现的[38]。随后,该团队又于2016年宣布通过传统平板涂布法结合MALDI–TOF检测的“培养组学”方法,从人粪便样本一共分离获得了1056个物种,其中包含247个新物种[23]。同年,另一个肠道微生物大规模分离培养的重要研究成果由Trevor团队发表,他们通过对粪便样本进行酒精处理来杀死营养细胞,保留芽孢,然后通过在培养基中添加牛黄胆酸盐的方法刺激芽孢萌发的策略,成功分离培养了137株、121种产芽孢肠道菌,其中包含69个首次分离培养的新物种[39]。该种样品处理方法后来被广泛的应用于肠道微生物的分离培养中。该团队又在3年后,对分离培养的菌株资源库做了进一步的扩充,构建了人类肠道细菌培养库(human gastrointestinal bacteria culture collection,HBC),该菌株资源库包含737株来自273个不同物种的菌株资源及其对应的基因组数据,其中包含105个潜在新物种[40]。2019年另一个代表性的肠道微生物分离培养工作是由华大基因研究团队完成的,研究人员通过对155个健康志愿者粪便样品分离培养,从6000多个单菌落中获得了1520分离株,并对全部菌株进行了基因组测序,构建了肠道微生物培养的基因组文集(Culturable Genome Reference,CGR)[16]。作者后续通过对1520株基因组进行16S rRNA序列提取和比对,以98.7%同源性作为分类标准的话,这1520株菌来自于至少319个不同菌种,且其中包含至少141个尚未进行分类学鉴定的物种[16]。同年,Poyet团队通过对11位粪菌移植供体粪便进行微生物分离培养,建立了菌株库BIO-ML (Broad Institute-OpenBiome Microbiome Library)[41]。该库中包含来自106个种的代表性菌株和7758个菌株的基因组框架图[29, 41]。近期,刘双江团队通过从239份中国健康人粪便样本中分离超过15000个分离株,获得400个肠道微生物物种,来自159个属和53个科,其中包含102个新物种,28个新属和3个新科),全部新种进行了全基因组测序、菌种描述和命名,代表400个物种的1170个菌株保存在国内外菌种保藏中心,构建了对学术界开放使用的中华健康人肠道菌株资源库hGMB (hgmb.nmdc.cn)[42]。通过将前文所列举的从2016年至今的,6个肠道微生物资源库进行综合比较,发现这6个代表性分离培养工作,一共获得超过1521个明确人肠道来源的微生物物种,其中1150个物种只出现在其中一个资源库中[42] (图 2)。除了上述较有代表性的菌株资源库以外,研究人员根据各自研究目的的不同,针对人群样本进行肠道微生物的分离培养,例如专门针对毛螺菌科(Lachnospiraceae)[43]和阿克曼菌属(Akkermansia)[41]开展的大规模分离培养和表征工作。这些工作不仅揭示了特定类群菌种的生理功能与生态特征,同时也丰富了对应的肠道菌种资源。据不完全统计,目前已经成功分离培养的肠道微生物约在1500–2000种,而根据作者和其他研究人员对于人肠道微生物组高通量测序数据的整合分析,保守估计仍有50%–70%的肠道微生物物种仍处于未培养状态,肠道微生物分离培养工作仍任重道远[18, 39, 42]。

|
| 图 2 六个近年来具有代表性的人肠道大规模分离培养工作获得菌种的数量和分布情况(根据文献[44]修改) Figure 2 Comparison of 6 representative and large-scale studies on gut microbial cultivation and collections (derived from preprint[44]). SPORE covers 121 species and 18 species are not covered by other 5 studies[39]. BIO-ML covers 106 species and 19 species are not covered by other 5 studies[41]; Culturomics covers 1056 species and 803 species are not covered by other 5 studies[23]; CGR covers 319 species and 127 species are not covered by other 5 studies[16]; HBC covers 236 species and 39 species are not covered by other 5 studies[40]; hGMB covers 400 species and 138 species are not covered by other 5 studies[42]. |
3 模式动物肠道微生物可培养菌株资源现状
小鼠是肠道微生物研究的最常用动物模型之一,大量与肠道菌群相关的研究工作都是利用小鼠模型完成的。然而,很长一段时间里,我们对于小鼠肠道菌群结构和组成、特别是系统的分离培养,缺乏足够的关注。直到2016年,Ilias L等通过对分离培养的小鼠肠道微生物进行系统整合和分析,构建了首个小鼠肠道微生物菌株资源库miBC (www.dsmz.de/miBC)[8]。他们分离挑取培养了超过1500个单克隆,并从中鉴定出了76个不同物种,分属于26个科和5个门,分别为放线菌门(Actinobacteria)、厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、变形菌门(Proteobacteria)和疣微菌门(Verrucomicrobia)。然而,通过对不同模型小鼠肠道16S rRNA基因扩增子数据的综合分析,这76个物种仅覆盖不到10%的物种多样性。这意味着大部分小鼠肠道菌仍处于未培养状态。随后,在2020年,刘双江等人的团队通过一套自主设计的标准化分离培养流程鉴定,对ob/ob小鼠肠道内容物进行大规模分离培养,先后分离并鉴定了1831个分离株,并将其中的244个菌株保藏在国际认可的菌保中心[18]。此次建构的小鼠肠道微生物资源库(mGMB)包括126种微生物及其基因组,其中76种微生物是首次分离培养的新物种,并对这些新物种进行了分类学鉴定和命名。该小鼠菌株资源库覆盖了超过88%小鼠肠道的核心属,其基因组代表了超过52%的小鼠肠道的非冗余功能基因集。mGMB资源库的构建使得可培养的小鼠肠道微生物菌株资源从76种增加到180种,从48个属增加到110个属(图 3),两个小鼠菌株资源库(miBC和mGMB)构成了目前最大的小鼠肠道微生物资源集合,为后续基于小鼠模型开展肠道菌–宿主互作机理研究和功能菌株开发奠定了资源基础。
除小鼠以外,昆虫是自然界中存在数量和种类最大的一类动物,针对昆虫肠道微生物生物分离获得的菌株却数量有限。吕宝乾团队在对椰心叶甲的肠道微生物分离中,利用LB培养基获得22株微生物,分属于5个属3个门,分别为变形菌门、厚壁菌门和放线菌门,其中13株属于变形菌门是占比最高的微生物,而且由于椰心叶甲经常食用纤维素、木聚糖等植物多糖,分离获得的微生物大多也可以降解纤维素和木聚糖等[44]。白蚁是备受关注的一类分解木质纤维素的昆虫,周楠等采用高通量微流控液滴阵列技术,对白蚁肠道微生物进行了培养并获得396株微生物,其中包括Burkholderia、Micrococcus和Dysgonomonas等属的新成员[25]。华中师范大学杨红团队与刘双江研究团队合作,通过利用寡营养成分的培养基,分离获得104株微生物分属于18个属4个门,分别是厚壁菌门、放线菌门、变形菌门和奇异球菌–栖热菌门(Deinococcus–Thermus),包含首次在白蚁肠道中分离得到的Deinococcus[45]和Gryllotalpicola[46]属微生物和另外2株肠道新种[47-48]。Nishiyama T和Manfredi AP团队在唯一碳源条件下从鳞翅目、鞘翅目等成虫肠道微生物进行分离培养,都获得了大量可降解纤维素的菌株,其中Manfredi AP团队共分离233株微生物,分属于12个属4个门,分别是厚壁菌门、放线菌门、变形菌门及拟杆菌门,由于宿主饮食特性,其中45株微生物均可对纤维素进行水解[49-50]。也有很多科学家在体外模拟肠道环境,通过调整环境中的氧分压对微生物进行富集培养后,再进行微生物分离培养,杨红团队在黑胸散白蚁肠道中发现,在严格厌氧条件下分离出的优势微生物是肠杆菌、柠檬酸杆菌、乳球菌等,但在好氧情况下为乳球菌、芽孢杆菌等,并且发现寡营养培养基对白蚁肠道微生物分离多样性具有较好的影响[51]。同样,杨书燕团队在不同肠段分离的微生物,由于氧分压不同,在分离获得342株微生物中,虽然分布于16个属中,但在不同肠段分离的微生物组成和优势微生物也不尽相同[52]。不论是为了分离获得功能微生物还是利用不同条件培养富集后分离微生物,都能使不同的微生物在相应的条件下富集,这也为后续大规模分离培养工作奠定了基础。
相比于陆生生物,针对水生动物肠道微生物的分离培养报道较少。就目前的研究结果而言,水生动物肠道微生物组成主要包含拟杆菌门、厚壁菌门、变形菌门、软壁菌门(Tenericutes)、放线菌门和梭杆菌门[53]。张美玲团队从罗非鱼肠道前肠中分离培养286株微生物,其中6株可以将鱼油或豆油作为唯一碳源,其中5株属于柠檬酸杆菌属(Citrobacter),1株为气单胞菌属(Aeromonas),它可以将豆油和鱼油分别分解成21种脂肪酸和17种脂肪酸[54]。
4 肠道微生物资源的开发与利用正如前言中所论述的,肠道微生物菌株资源对于推动肠道微生物组从数据分析描述型研究向因果机理型和应用开发型研究的过渡与深入发展,是必不可少的。下面作者将通过几个典型的基于菌株资源开展的肠道微生物组研究的例子,展示肠道微生物菌株资源库的应用场景和未来开发潜力。
首先,肠道微生物的分离培养与鉴定工作极大地推动了肠道高通量测序研究的发展。一直以来,肠道菌群的高通量测序研究,无论是宏基因组数据还是扩增子数据,在进行物种分析时,通常需要通过与已知物种信息的基因组或者标志基因的数据库进行比对与分析,实现物种注释[15]。然而在实际操作中,研究人员发现,由于肠道中大量物种处于未培养和未鉴定状态,那么数据库中就缺乏这些微生物对应的物种信息、特征基因和基因组的数据,结果就是在对高通量测序数据进行分析注释时,这些未培养的物种无法被准确注释出来,因而这些未培养物种中的潜在功能,也就不得而知了。解决这一问题的最直接方法是把这些未培养的微生物变得可以培养、获得更多可培养的新物种,包括其对应的物种分类信息、标志基因序列和基因组数据等[16, 55]。举例来说,在小鼠菌株资源库mGMB构建之前,利用LTP数据库(LTP_v132)对随机选取的小鼠肠道16S rRNA基因扩增子进行物种注释时,仅有12.98%±5.07%的reads可以被注释到明确的物种,33.43%±9.52% reads可以被注释到已知属[8]。而当研究人员将mGMB中的76个新物种的16S rRNA序列更新到LTP数据库中之后,发现对于同样的一批扩增子数据进行注释时,有24.37%±6.60%的reads可以被注释到明确的物种,而可以注释到已知属的reads数也提高到了49.46%±9.54%,这一提高,使得更多测序数据被利用和分析,更多的信息可以被挖掘出来[18]。例如,研究人员通过对ob/ob小鼠和野生型小鼠的肠道扩增子数据进行基于注释的差异分析,就发现有3个mGMB鉴定的新属(Columnibacterium gen. nov., Musculibacter gen. nov.和Faeciroseburia gen. nov.)是在ob/ob小鼠肠道中特异性富集的,而有2个新属(Digestivibacterium gen. nov.和Stereobacter gen. nov.)是在野生型小鼠中显著富集的[18]。这些差异菌属背后可能代表了与表型相关联的潜在新功能。此外,前文提到的2019年建立的2个人肠道菌株资源库HBC[41]和CGR[16]也是不约而同的采用大规模分离培养和基因组测序的策略,实现了对于宏基因组数据挖掘和功能分析的精确度和覆盖度的大幅度提高。其中,CGR的工作通过对1520株来自339个不同物种的肠道微生物进行培养和基因组测序,对全球范围内人肠道的宏基因组数据的reads覆盖度从50%提高到了超过70%[16]。而HBC通过培养和测序737株来自273个物种的肠道微生物,将人肠道宏基因组数据的物种注释率相较于Human Microbiome Project (HMP)基因组集提高了61%;通过将培养获得的微生物基因组与原有数据库进行整合,对全球范围内13490份人肠道宏基因组数据中超过81%的属进行明确的注释[41, 56]。
第二,获得更多肠道微生物菌株资源促进了宿主–肠道菌群功能的验证与因果机理的阐明。香港大学于君团队开展了大量基于可培养菌株的因果关系与作用机理研究工作,通过小鼠灌胃模型和细胞模型实验研究,发现厌氧消化链球菌(Peptostreptococcus anaerobius)表面蛋白PCWBR2与CRC细胞表面的整合素α2/β1结合,激活下游的PI3K-Akt-NF-κB信号通路,从而导致结直肠癌发生发展[57]。而最新发表的研究结果证明,癌症患者体内特异性富集的奇异变形菌属可以通过侵润肠道上皮细胞系、增加促炎因子(IL-18、IL-1α)产生,诱导NOD、JAK-STAT、MAPK信号通路活化诱导炎症应答,并加剧DSS处理小鼠的结肠炎症[58]。然而研究表明肠道内丁酸盐浓度的下降与肠道炎症发病存在一定关联,但它们之间是否存在因果关系,Kazuyuki K对8周动脉粥样硬化ApoE–/– GM小鼠接种常见丁酸产生菌群(C. symbiosum、C. comes以及R. torques)或在食物中添加三丁酸甘油酯(TB),两组分别食用HPP饮食和HPP+6%TB,发现丁酸盐可通过减少内毒素血症来抑制动脉粥样硬化的发展,使接种丁酸盐产生菌组小鼠动脉硬化病情发展得到较好控制,并且发现肠道中Roseburia定殖对心血管疾病是有益处的[59]。北京大学第三附属医院段丽萍团队通过对肠应激大鼠(避水应激法)和对照组粪便样品组学分析结果发现,在肠易激组肠道菌群组成上毛螺菌科明显减少,且丁酸盐合成途径富集情况显著降低[60]。为了验证丁酸产生菌是否直接影响大鼠的应激表型,研究人员利用了一株毛螺菌科的丁酸盐产生菌Roseburia hominis对肠易激组进行灌胃补充,补充后发现大鼠盲肠内不仅丁酸盐含量有明显提升,相对灌胃PBS的肠易激组丙酸盐也有明显提升,并且对肠应激症状有所改善[61]。
第三,菌株资源库的构建有利于更多功能益生菌的开发。目前肠道益生菌在物种水平上种类相对较少,根据中国卫生部引发《可用于食品菌种名单》(更新于2018年)中所列,可用于食品的菌种数量为35种。想要从现有框架内发现更多全新功能的益生菌存在一定难度和局限。菌株资源库的构建使得肠道来源的菌株得到了极大的丰富,更多的物种多样性决定了肠道微生物具有更高的功能多样性,研究人员就可以从这些菌株资源库中通过对多组学数据进行分析发掘,进一步发现和鉴定新的功能菌作为益生菌的开发对象。例如,刘宏伟和刘双江研究团队通过分析HMP健康人群和肥胖人群高通量测序数据,发现其中Parabacteroides相对丰度与体质指数(BMI)呈明显负相关,而且该属微生物是人类肠道微生物组成18核心成员之一,并且在肥胖人群中的分析Parabacteroides相对丰度明显下降[62]。随后,研究人员利用mGMB中的一株狄氏副拟杆菌Parabacteroides distasonis (PD)对ob/ob小鼠进行灌胃,结果表明口服PD可改善代谢、控制体重、减少高血糖和降低脂肪肝,是潜在的功能益生菌[62]。PD可通过产生琥珀酸增强肠上皮中的果糖-1, 6-二磷酸酶活性,从而促进肠道糖异生,可通过活化肠道FXR–FGF15通路,恢复肠道屏障完整性,可转化胆汁酸,增加次级胆汁酸石胆酸(LCA)和熊去氧胆酸(UDCA)从而缓解小鼠高血脂[62]。近期,两个研究团队合作,通过对中华健康人肠道菌株资源库hGMB中的一株解木糖拟杆菌进行功能鉴定,发现该菌株灌胃fa/fa大鼠可以显著增加肝脏、血液中叶酸含量,提高叶酸介导的一碳代谢,显示良好的降脂、降糖、抗脂肪肝作用,且该益生效果依赖于解木糖拟杆菌中的叶酸合成途径[62]。该菌株展示出的对肥胖大鼠的脂肪肝缓解效果与口服具有非酒精性脂肪肝干预作用的灵芝杂萜衍生物(GMD)效果类似[63]。
除了上述基于可培养菌株资源所开展的肠道微生物组研究以外,肠道微生物菌株资源库还可以应用于更多的研究场景。例如,现阶段的粪菌移植还滞留在使用供体完整粪便对受体进行移植,在这过程中,并不能有效排除粪便中一些未出现表型的有害生物或者非生物因子,如果这些有害因子随同粪便转入受体体内,可能会带来不良影响。这也更加体现出从复杂的群落中获得纯培养和彻底弄清其中的相互作用机制的重要性,从而达到对症下药,个性配比的目的[41, 55, 64]。肠道微生物菌株资源库的构建为粪菌移植过程中进行人工设计与个性化人工菌群的构建、更多疾病的肠道干预靶点的验证与发现、肠道菌功能代谢产物的发掘与应用以及未知功能基因的研究等,提供了可能性和资源基础。
5 肠道微生物分离培养面临的问题与机遇为探索更多疾病与肠道微生物之间存在的互作机理并实现通过干预肠道菌群组成达到精准医疗的目的,需要对肠道群落有更为全面的了解。这需要通过不懈的努力,将更多“不可培养”微生物培养出来,不断扩充新的可培养菌株资源,从而更深入的了解微生物自身特性及微生物与宿主间作用机制。为完善肠道菌株资源库,许多团队都在探索合适的分离技术和培养手段,包括笔者所在研究组在内的多个研究团队发现在样品前处理阶段,利用酒精处理后可以提高产芽孢菌株的分离培养效果,并且在培养基中添加瘤胃液和羊血可以提高分离培养的多样性和生长效果[18]。不仅如此,Lagier团队利用70种不同培养方案对粪便中微生物进行培养,其中18种被验证非常适用与肠道微生物的培养,而且该团队依靠这种培养策略分离获得大量不同菌株[11]。其中,厌氧环境的维持是至关重要的,这些最佳培养条件的确定使得以后的研究可以获得更多未鉴定微生物。在分离培养微生物的方法上,优化潜力同样是巨大的,从最经典的稀释涂布[39],到目前可以批量分离培养微生物的微流控设备[24, 28],在减少人力、物力资源投入的同时,提高了分离培养的通量。微流控分离微生物优点是通过培养微液滴中唯一的单细胞,避免因优势微生物抢占生态位而不能形成肉眼可见菌落而被忽略的细菌,甚至可以通过反向基因组学[27],通过设计筛选目的微生物特异性引物或者探针对其及进行大批量筛选,有望通过这样大批量筛选的方法可以获得更多HMP中“Most wanted”列表上通过16S rRNA基因扩增子数据生物信息学分析总结的与人类疾病相关且未获得纯培养的肠道微生物,希望以其为指引为未来开展微生物–宿主作用机理研究奠定资源基础[28-29]。但基于微流控的分离培养方法也存在诸多限制因素,根据泊松分布计算微液滴体积和原始样本稀释倍数后,体积是巨大的,如何更有效的检测每个液滴中存在微生物细胞数量,从而更有效的剔除0或 > 1细胞数的微液滴,从而提高仪器及培养空间的利用效率,应该成为下一个微流控分离微生物的方向之一。
根据之前的报道,肠道中未培养的微生物数量是巨大的,随着大规模分离培养的研究不断推进[16, 23, 39, 41],我们面临的问题还包括对新发现的微生物的命名,统一的命名是科学交流的基础,然而,目前通用的国际微生物命名规则和要求,例如期刊International Journal of Systematic and Evolutionary Microbiology (IJSEM)采用多相分类方法,要求对新菌株的生理生态、细胞化学、形态特征、系统发育等进行全面描述,并且要求标准菌株必须保藏在经过认定的2个国家的菌种保藏中心,过程的复杂阻滞了肠道微生物鉴定和命名的发展,使得一些科学家不愿意对分离培养的微生物进行命名和描述。在研究规范的基础上,如何简化物种描述内容和标准菌株的保存流程,急需制定一套合理规范、简洁易行、广为接受的菌种命名和菌株保藏流程,还需要微生物分类学家的努力。另一方面,新种模式菌株需要存放于不同的保种中心,这需要各国间加强相互交流,制定更便捷的邮寄菌株方案及法律法规,尽量降低繁杂且重复的工作,将会对提高新物种描述的效率有极大的帮助。
最后,还需要加强对于肠道微生物保存方法的研究。当前使用的冻干粉或者液氮保藏,有其局限性,例如发现经过保藏之后,相当比例的菌株不能复活。菌株经冻干后的菌粉,十分依赖保护剂类型,且不同菌株对保护剂要求也趋于不同,对该方面的进一步探索也是十分必要的。
综上所述,通过更高效的分离肠道微生物,建立肠道菌株资源库,得到未培养微生物且后续对新培养微生物的命名及保存工作的完善,是目前亟待解决的问题。建立肠道菌株资源库可以更加高效的服务科学研究,进行资源的合理开发与利用,资源库的建立可以促进高通量数据的精准解析与深入挖掘,可以针对靶向肠道的多种疾病干预效果进行有效的评价与实验验证,同时可以进行潜在的益生菌、后生素的开发利用,也可以进行肠道菌与宿主互作相关分子机制的研究,解密肠道中的微生物暗物质,精准的调控肠道菌群,使肠道菌能够更好的服务于人类健康。所以建立分离培养的新方法,创建保藏菌株资源的进技术是保证肠道菌株资源长久发展的基础,是推进菌株资源开发利用的奠基石。希望在今后的科学发展中,可以获得更多的不可培养微生物,深入了解菌株特性及发掘其在疾病治疗方面的潜力,为人类健康保驾护航。
| [1] | Turnbaugh PJ, Hamady M, Yatsunenko T, Cantarel BL, Duncan A, Ley RE, Sogin ML, Jones WJ, Roe BA, Affourtit JP, Egholm M, Henrissat B, Heath AC, Knight R, Gordon JI. A core gut microbiome in obese and lean twins. Nature, 2009, 457(7228): 480-484. DOI:10.1038/nature07540 |
| [2] | Lopez-Siles M, Martinez-Medina M, Abellà C, Busquets D, Sabat-Mir M, Duncan SH, Aldeguer X, Flint HJ, Garcia-Gil LJ. Mucosa-associated Faecalibacterium prausnitzii phylotype richness is reduced in patients with inflammatory bowel disease. Applied and Environmental Microbiology, 2015, 81(21): 7582-7592. DOI:10.1128/AEM.02006-15 |
| [3] | Lamas B, Richard ML, Leducq V, Pham HP, Michel ML, Da Costa G, Bridonneau C, Jegou S, Hoffmann TW, Natividad JM, Brot L, Taleb S, Couturier-Maillard A, Nion-Larmurier I, Merabtene F, Seksik P, Bourrier A, Cosnes J, Ryffel B, Beaugerie L, Launay JM, Langella P, Xavier RJ, Sokol H. CARD9 impacts colitis by altering gut microbiota metabolism of tryptophan into aryl hydrocarbon receptor ligands. Nature Medicine, 2016, 22(6): 598-605. DOI:10.1038/nm.4102 |
| [4] | Pedersen HK, Gudmundsdottir V, Nielsen HB, Hyotylainen T, Nielsen T, Jensen BAH, Forslund K, Hildebrand F, Prifti E, Falony G, Le Chatelier E, Levenez F, Dore J, Mattila I, Plichta DR, Poho P, Hellgren LI, Arumugam M, Sunagawa S, Vieira-Silva S, Jorgensen T, Holm JB, Trost K, Kristiansen K, Brix S, Raes J, Wang J, Hansen T, Bork P, Brunak S, Oresic M, Ehrlich SD, Pedersen O, Consortium M. Human gut microbes impact host serum metabolome and insulin sensitivity. Nature, 2016, 535(7612): 376-381. DOI:10.1038/nature18646 |
| [5] | Sender R, Fuchs S, Milo R. Are we really vastly outnumbered? revisiting the ratio of bacterial to host cells in humans. Cell, 2016, 164(3): 337-340. DOI:10.1016/j.cell.2016.01.013 |
| [6] | Wu GD, Chen J, Hoffmann C, Bittinger K, Chen YY, Keilbaugh SA, Bewtra M, Knights D, Walters WA, Knight R, Sinha R, Gilroy E, Gupta K, Baldassano R, Nessel L, Li H, Bushman FD, Lewis JD. Linking long-term dietary patterns with gut microbial enterotypes. Science, 2011, 334(6052): 105-108. DOI:10.1126/science.1208344 |
| [7] | Torres-Fuentes C, Schellekens H, Dinan TG, Cryan JF. The microbiota-gut-brain axis in obesity. Lancet Gastroenterology & Hepatology, 2017, 2(10): 747-756. |
| [8] | Lagkouvardos I, Pukall R, Abt B, Foesel BU, Meier-Kolthoff JP, Kumar N, Bresciani A, Martinez I, Just S, Ziegler C, Brugiroux S, Garzetti D, Wenning M, Bui TPN, Wang J, Hugenholtz F, Plugge CM, Peterson DA, Hornef MW, Baines JF, Smidt H, Walter J, Kristiansen K, Nielsen HB, Haller D, Overmann J, Stecher B, Clavel T. The Mouse Intestinal Bacterial Collection (miBC) provides host-specific insight into cultured diversity and functional potential of the gut microbiota. Nature Microbiology, 2016, 1(11): 16131. |
| [9] | Eckburg PB, Bik EM, Bernstein CN, Purdom E, Dethlefsen L, Sargent M, Gill SR, Nelson KE, Relman DA. Diversity of the human intestinal microbial flora. Science, 2005, 308(5728): 1635-1638. DOI:10.1126/science.1110591 |
| [10] | Hayashi H, Sakamoto M, Benno Y. Phylogenetic analysis of the human gut microbiota using 16S rDNA clone libraries and strictly anaerobic culture-based methods. Microbiology Immunology, 2002, 46(8): 535-548. DOI:10.1111/j.1348-0421.2002.tb02731.x |
| [11] | Lagier JC, Hugon P, Khelaifia S, Fournier PE, La Scola B, Raoult D. The Rebirth of Culture in Microbiology through the Example of Culturomics To Study Human Gut Microbiota. Clinical Microbiology Reviews, 2015, 28(1): 237-264. DOI:10.1128/CMR.00014-14 |
| [12] | Vilanova C, Porcar M. Are multi-omics enough?. Nature Microbiology, 2016, 1(8): 16101. DOI:10.1038/nmicrobiol.2016.101 |
| [13] | Kaeberlein T, Lewis K, Epstein SS. Isolating "uncultivable" microorganisms in pure culture in a simulated natural environment. Science, 2002, 296(5570): 1127-1129. DOI:10.1126/science.1070633 |
| [14] | Nichols D, Cahoon N, Trakhtenberg EM, Pham L, Mehta A, Belanger A, Kanigan T, Lewis K, Epstein SS. Use of ichip for high-throughput in situ cultivation of "uncultivable" microbial species. Applied and Environmental Microbiology, 2010, 76(8): 2445-2450. DOI:10.1128/AEM.01754-09 |
| [15] | Almeida A, Nayfach S, Boland M, Strozzi F, Beracochea M, Shi ZJ, Pollard KS, Sakharova E, Parks DH, Hugenholtz P, Segata N, Kyrpides NC, Finn RD. A unified catalog of 204, 938 reference genomes from the human gut microbiome. Nature Biotechnology, 2020, 39(1): 105-114. |
| [16] | Zou Y, Xue W, Luo G, Deng Z, Qin P, Guo R, Sun H, Xia Y, Liang S, Dai Y, Wan D, Jiang R, Su L, Feng Q, Jie Z, Guo T, Xia Z, Liu C, Yu J, Lin Y, Tang S, Huo G, Xu X, Hou Y, Liu X, Wang J, Yang H, Kristiansen K, Li J, Jia H, Xiao L. 1, 520 reference genomes from cultivated human gut bacteria enable functional microbiome analyses. Nature Biotechnology, 2019, 37(2): 179-185. DOI:10.1038/s41587-018-0008-8 |
| [17] | Lau JT, Whelan FJ, Herath I, Lee CH, Collins SM, Bercik P, Surette MG. Capturing the diversity of the human gut microbiota through culture-enriched molecular profiling. Genome Medicine, 2016, 8(1): 72. DOI:10.1186/s13073-016-0327-7 |
| [18] | Liu C, Zhou N, Du MX, Sun YT, Wang K, Wang YJ, Li DH, Yu HY, Song Y, Bai BB, Xin Y, Wu L, Jiang CY, Feng J, Xiang H, Zhou Y, Ma J, Wang J, Liu HW, Liu SJ. The mouse gut microbial biobank expands the coverage of cultured bacteria. Nature Communications, 2020, 11(1): 79. DOI:10.1038/s41467-019-13836-5 |
| [19] | Dione N, Khelaifia S, La Scola B, Lagier J, Raoult D. A quasi-universal medium to break the aerobic/anaerobic bacterial culture dichotomy in clinical microbiology. Clinical Microbiology and Infection, 2016, 22(1): 53-58. DOI:10.1016/j.cmi.2015.10.032 |
| [20] | Duncan SH, Hold GL, Harmsen HJM, Stewart CS, Flint HJ. Growth requirements and fermentation products of Fusobacterium prausnitzii, and a proposal to reclassify it as Faecalibacterium prausnitzii gen. nov., comb. nov.. International Journal of Systematic and Evolutionary Microbiology, 2002, 52(Pt 6): 2141-2146. |
| [21] | Bergmanson JPG, Wang E, Gire AI, Osato MS. In vitro effects of medium tonicity, nutrient concentration, and free chlorine content on Acanthamoeba. Contact Lens and Anterior Eye, 2011, 34(4): 164-168. DOI:10.1016/j.clae.2011.02.003 |
| [22] | Chong Y, Lee SY. Comparison of Thioglycollate Media, Modified thioglycollage media, and gam for the cultivation of non-sporeforming anaerobes. Yonsei Medical Journal, 1974, 15(2): 136-146. DOI:10.3349/ymj.1974.15.2.136 |
| [23] | Lagier JC, Khelaifia S, Alou MT, Ndongo S, Dione N, Hugon P, Caputo A, Cadoret F, Traore SI, Seck EH, Dubourg G, Durand G, Mourembou G, Guilhot E, Togo A, Bellali S, Bachar D, Cassir N, Bittar F, Delerce J, Mailhe M, Ricaboni D, Bilen M, Nieko NPMD, Badiane NMD, Valles C, Mouelhi D, Diop K, Million M, Musso D, Abrahao J, Azhar EI, Bibi F, Yasir M, Diallo A, Sokhna C, Djossou F, Vitton V, Robert C, Rolain JM, La Scola B, Fournier PE, Levasseur A, Raoult D. Culture of previously uncultured members of the human gut microbiota by culturomics. Nature Microbiology, 2016, 1(12): 16203. DOI:10.1038/nmicrobiol.2016.203 |
| [24] | Jiang CY, Dong LB, Zhao JK, Hu XF, Shen CH, Qiao YX, Zhang XY, Wang YP, Ismagilov RF, Liu SJ, Du WB. High-throughput single-cell cultivation on microfluidic streak plates. Applied and Environmental Microbiology, 2016, 82(7): 2210-2218. DOI:10.1128/AEM.03588-15 |
| [25] | Zhou N, Sun YT, Chen DW, Du W, Yang H, Liu SJ. Harnessing microfluidic streak plate technique to investigate the gut microbiome of Reticulitermes chinensis. Microbiologyopen, 2019, 8(3): e00654. DOI:10.1002/mbo3.654 |
| [26] | Villa MM, Bloom RJ, Silverman JD, Durand HK, Jiang S, Wu A, Dallow EP, Huang S, You L, David LA. Interindividual variation in dietary carbohydrate metabolism by gut bacteria revealed with droplet microfluidic culture. mSystems, 2020, 5(3): e00864-00819. |
| [27] | Cross KL, Campbell JH, Balachandran M, Campbell AG, Cooper SJ, Griffen A, Heaton M, Joshi S, Klingeman D, Leys E, Yang Z, Parks JM, Podar M. Targeted isolation and cultivation of uncultivated bacteria by reverse genomics. Nature Biotechnology, 2019, 37(11): 1314-1321. DOI:10.1038/s41587-019-0260-6 |
| [28] | Ma L, Kim J, Hatzenpichler R, Karymov MA, Hubert N, Hanan IM, Chang EB, Ismagilov RF. Gene-targeted microfluidic cultivation validated by isolation of a gut bacterium listed in Human Microbiome Project's Most Wanted taxa. Proceeding of the National Academy Science USA, 2014, 111(27): 9768-9773. DOI:10.1073/pnas.1404753111 |
| [29] | Fodor AA, DeSantis TZ, Wylie KM, Badger JH, Ye Y, Hepburn T, Hu P, Sodergren E, Liolios K, Huot-Creasy H, Birren BW, Earl AM. The "most wanted" taxa from the human microbiome for whole genome sequencing. PLoS One, 2012, 7(7): e41294. DOI:10.1371/journal.pone.0041294 |
| [30] |
Liu G, Ren WK, Xiong X, Li TJ, Zhong J, Yin YL. Advances and applications of in vitro models for host-microbe interactions research. Chinese Journal of Animal Nutrition, 2013, 25(8): 1677-1682.
(in Chinese) 刘刚, 任文凯, 熊霞, 李铁军, 钟瑾, 印遇龙. 肠道与微生物相互作用体外研究模型进展与应用. 动物营养学报, 2013, 25(8): 1677-1682. DOI:10.3969/j.issn.1006-267x.2013.08.003 |
| [31] | Schubert S, Kostrzewa M. MALDI-TOF MS in the microbiology laboratory: current trends. Current Issues in Molecular Biology, 2017, 23: 17-20. |
| [32] |
Chen F, Hu BJ, Zhao H. Application of MALDI-TOF MS in direct identification of clinical microbiological samples. Laboratory Medicine, 2015, 30(7): 750-756.
(in Chinese) 陈飞, 胡玢婕, 赵虎. MALDI-TOF MS在临床微生物样本直接检测中的应用. 检验医学, 2015, 30(7): 750-756. DOI:10.3969/j.issn.1673-8640.2015.07.019 |
| [33] | Huang WE, Griffiths RI, Thompson IP, Bailey MJ, Whiteley AS. Raman microscopic analysis of single microbial cells. Analytical Chemistry, 2004, 76(15): 4452-4458. DOI:10.1021/ac049753k |
| [34] | Willemse-Erix HF, Jachtenberg J, Barutçi H, Puppels GJ, van Belkum A, Vos MC, Maquelin K. Proof of principle for successful characterization of methicillin-resistant coagulase-negative staphylococci isolated from skin by use of Raman spectroscopy and pulsed-field gel electrophoresis. Journal of Clinical Microbiology, 2010, 48(3): 736-740. DOI:10.1128/JCM.01153-09 |
| [35] | Qin J, Li R, Raes J, Arumugam M, Burgdorf KS, Manichanh C, Nielsen T, Pons N, Levenez F, Yamada T, Mende DR, Li J, Xu J, Li S, Li D, Cao J, Wang B, Liang H, Zheng H, Xie Y, Tap J, Lepage P, Bertalan M, Batto JM, Hansen T, Le Paslier D, Linneberg A, Nielsen HB, Pelletier E, Renault P, Sicheritz-Ponten T, Turner K, Zhu H, Yu C, Li S, Jian M, Zhou Y, Li Y, Zhang X, Li S, Qin N, Yang H, Wang J, Brunak S, Dore J, Guarner F, Kristiansen K, Pedersen O, Parkhill J, Weissenbach J, Consortium M, Bork P, Ehrlich SD, Wang J. A human gut microbial gene catalogue established by metagenomic sequencing. Nature, 2010, 464(7285): 59-65. DOI:10.1038/nature08821 |
| [36] | Goodman AL, Kallstrom G, Faith JJ, Reyes A, Moore A, Dantas G, Gordon JI. Extensive personal human gut microbiota culture collections characterized and manipulated in gnotobiotic mice. Proceeding of the National Academy Science USA, 2011, 108(15): 6252-6257. DOI:10.1073/pnas.1102938108 |
| [37] | Human Microbiome Jumpstart Reference Strains Consortium, Nelson KE, Weinstock GM, Highlander SK, Worley KC, Creasy HH, Wortman JR, Rusch DB, Mitreva M, Sodergren E, Chinwalla AT, Feldgarden M, Gevers D, Haas BJ, Madupu R, Ward DV, Birren BW, Gibbs RA, Methe B, Petrosino JF, Strausberg RL, Sutton GG, White OR, Wilson RK, Durkin S, Giglio MG, Gujja S, Howarth C, Kodira CD, Kyrpides N, Mehta T, Muzny DM, Pearson M, Pepin K, Pati A, Qin X, Yandava C, Zeng Q, Zhang L, Berlin AM, Chen L, Hepburn TA, Johnson J, McCorrison J, Miller J, Minx P, Nusbaum C, Russ C, Sykes SM, Tomlinson CM, Young S, Warren WC, Badger J, Crabtree J, Markowitz VM, Orvis J, Cree A, Ferriera S, Fulton LL, Fulton RS, Gillis M, Hemphill LD, Joshi V, Kovar C, Torralba M, Wetterstrand KA, Abouellleil A, Wollam AM, Buhay CJ, Ding Y, Dugan S, FitzGerald MG, Holder M, Hostetler J, Clifton SW, Allen-Vercoe E, Earl AM, Farmer CN, Liolios K, Surette MG, Xu Q, Pohl C, Wilczek-Boney K, Zhu D. A catalog of reference genomes from the human microbiome. Science, 2010, 328(5981): 994-999. DOI:10.1126/science.1183605 |
| [38] | La Scola B, Fournier PE, Raoult D. Burden of emerging anaerobes in the MALDI-TOF and 16S rRNA gene sequencing era. Anaerobe, 2011, 17(3): 106-112. DOI:10.1016/j.anaerobe.2011.05.010 |
| [39] | Browne HP, Forster SC, Anonye BO, Kumar N, Neville BA, Stares MD, Goulding D, Lawley TD. Culturing of 'unculturable' human microbiota reveals novel taxa and extensive sporulation. Nature, 2016, 533(7604): 543-546. DOI:10.1038/nature17645 |
| [40] | Forster SC, Kumar N, Anonye BO, Almeida A, Viciani E, Stares MD, Dunn M, Mkandawire TT, Zhu A, Shao Y, Pike LJ, Louie T, Browne HP, Mitchell AL, Neville BA, Finn RD, Lawley TD. A human gut bacterial genome and culture collection for improved metagenomic analyses. Nature Biotechnology, 2019, 37(2): 186-192. DOI:10.1038/s41587-018-0009-7 |
| [41] | Poyet M, Groussin M, Gibbons SM, Avila-Pacheco J, Jiang X, Kearney SM, Perrotta AR, Berdy B, Zhao S, Lieberman TD, Swanson PK, Smith M, Roesemann S, Alexander JE, Rich SA, Livny J, Vlamakis H, Clish C, Bullock K, Deik A, Scott J, Pierce KA, Xavier RJ, Alm EJ. A library of human gut bacterial isolates paired with longitudinal multiomics data enables mechanistic microbiome research. Nature Medicine, 2019, 25(9): 1442-1452. DOI:10.1038/s41591-019-0559-3 |
| [42] | Liu C, Du MX, Abdugheni R, Yu HY, Li DH, Wang YJ, Zhou N, Jiang MZ, Niu PX, Han SS, Chen HH, Shi WY, Wu L, Xin YH, Ma J, Zhou Y, Jiang C, Liu HW, Liu SJ. Enlightening the taxonomy darkness of human gut microbiomes with cultured biobank. Microbiome, 2020. DOI:10.21203/rs.3.rs-74101/v1 |
| [43] | Sorbara MT, Littmann ER, Fontana E, Moody TU, Kohout CE, Gjonbalaj M, Eaton V, Seok R, Leiner IM, Pamer EG. Functional and genomic variation between human-derived isolates of Lachnospiraceae reveals inter- and intra-species diversity. Cell Host & Microbe, 2020, 28(1): 134-146. |
| [44] |
Zhang YL, Lv BQ, Yang F, Tu Y, Jiang FYD, Qi KX, Li ZC. Isolation, identification and functional analysis of intestinal microorganisms of Brontispa longissi Gestro. Chinese Journal of Tropical Crops, 2021. https://kns.cnki.net/kcms/detail/46.1019.S.20200727.1649.004.html (in Chinese). 章雨璐, 吕宝乾, 杨帆, 涂艳, 蒋方一丁, 齐可欣, 李紫城. 椰心叶甲肠道微生物的分离鉴定及功能分析. 热带作物学报, 2021. https://kns.cnki.net/kcms/detail/46.1019.S.20200727.1649.004.html |
| [45] | Chen W, Wang B, Hong H, Yang H, Liu SJ. Deinococcus reticulitermitis sp. nov., isolated from a termite gut. International Journal of Systematic and Evolutionary Microbiology, 2012, 62(Pt 1): 78-83. |
| [46] | Fang H, Lv W, Huang Z, Liu SJ, Yang H. Gryllotalpicola reticulitermitis sp. nov., isolated from a termite gut. International Journal of Systematic and Evolutionary Microbiology, 2015, 65(Pt 1): 85-89. |
| [47] | Yang SY, Zheng Y, Huang Z, Wang XM, Yang H. Lactococcus nasutitermitis sp. nov. isolated from a termite gut. International Journal of Systematic and Evolutionary Microbiology, 2016, 66(1): 518-522. DOI:10.1099/ijsem.0.000743 |
| [48] | Wang XM, Ma S, Yang SY, Peng R, Zheng Y, Yang H. Paenibacillus nasutitermitis sp. nov., isolated from a termite gut. International Journal of Systematic and Evolutionary Microbiology, 2016, 66(2): 901-905. DOI:10.1099/ijsem.0.000807 |
| [49] | Nishiyama T, Ueki A, Kaku N, Ueki K. Clostridium sufflavum sp. nov., isolated from a methanogenic reactor treating cattle waste. International Journal of Systematic and Evolutionary Microbiology, 2009, 59(Pt 5): 981-986. |
| [50] | Manfredi AP, Perotti NI, Martínez MA. Cellulose degrading bacteria isolated from industrial samples and the gut of native insects from Northwest of Argentina. Journal of Basic Microbiology, 2015, 55(12): 1384-1393. DOI:10.1002/jobm.201500269 |
| [51] | Fang H, Chen W, Wang B, Li X, Yang H. Cultivation and characterization of symbiotic bacteria from the gut of Reticulitermes chinensis. Applied Environmental Biotechnology, 2016, 1(1): 3-12. DOI:10.26789/AEB.2016.01.004 |
| [52] | 杨书燕. 海南象白蚁肠道不同部位可培养厌氧及兼性厌氧菌多样性分析与两株乳球菌的鉴定. 华中师范大学硕士论文, 2016. |
| [53] | Rungrassamee W, Klanchui A, Chaiyapechara S, Maibunkaew S, Tangphatsornruang S, Jiravanichpaisal P, Karoonuthaisiri N. Bacterial population in intestines of the black tiger shrimp (Penaeus monodon) under different growth stages. PLoS One, 2013, 8(4): e60802. DOI:10.1371/journal.pone.0060802 |
| [54] | 孙玉红. 鱼肠道油脂分解菌的分离及其在宿主脂类吸收中机制的研究. 华东师范大学硕士论文, 2016. |
| [55] | Lagier JC, Dubourg G, Million M, Cadoret F, Bilen M, Fenollar F, Levasseur A, Rolain JM, Fournier PE, Raoult D. Culturing the human microbiota and culturomics'. Nature Reviews Microbiology, 2018, 16(9): 540-550. DOI:10.1038/s41579-018-0041-0 |
| [56] | Qin J, Li R, Raes J, Arumugam M, Burgdorf KS, Manichanh C, Nielsen T, Pons N, Levenez F, Yamada T, Mende DR, Li J, Xu J, Li S, Li D, Cao J, Wang B, Liang H, Zheng H, Xie Y, Tap J, Lepage P, Bertalan M, Batto JM, Hansen T, Le Paslier D, Linneberg A, Nielsen HB, Pelletier E, Renault P, Sicheritz-Ponten T, Turner K, Zhu H, Yu C, Li S, Jian M, Zhou Y, Li Y, Zhang X, Li S, Qin N, Yang H, Wang J, Brunak S, Doré J, Guarner F, Kristiansen K, Pedersen O, Parkhill J, Weissenbach J, Consortium M, Bork P, Ehrlich SD, Wang J. A human gut microbial gene catalogue established by metagenomic sequencing. Nature, 2010, 464(7285): 59-65. DOI:10.1038/nature08821 |
| [57] | Long X, Wong CC, Tong L, Chu ESH, Ho Szeto C, Go MYY, Coker OO, Chan AWH, Chan FKL, Sung JJY, Yu J. Peptostreptococcus anaerobius promotes colorectal carcinogenesis and modulates tumour immunity. Nature Microbiology, 2019, 4(12): 2319-2330. DOI:10.1038/s41564-019-0541-3 |
| [58] | Zhang JW, Hoedt EC, Liu Q, Berendsen E, Teh JJ, Hamilton A, O'Brien AW, Ching JYL, Wei H, Yang KL, Xu ZL, Wong SH, Mak JWY, Sung JJY, Morrison M, Yu J, Kamm MA, Ng SC. Elucidation of Proteus mirabilis as a key bacterium in Crohn's Disease inflammation. Gastroenterology, 2021, 160(1): 317-330. DOI:10.1053/j.gastro.2020.09.036 |
| [59] | Kasahara K, Krautkramer KA, Org E, Romano KA, Kerby RL, Vivas EI, Mehrabian M, Denu JM, Bäckhed F, Lusis AJ, Rey FE. Interactions between Roseburia intestinalis and diet modulate atherogenesis in a murine model. Nature Microbiology, 2018, 3(12): 1461-1471. DOI:10.1038/s41564-018-0272-x |
| [60] | Zhang J, Song L, Wang Y, Liu C, Zhang L, Zhu S, Liu S, Duan L. Beneficial effect of butyrate-producing Lachnospiraceae on stress-induced visceral hypersensitivity in rats. Journal of Gastroenterology and Hepatology, 2019, 34(8): 1368-1376. DOI:10.1111/jgh.14536 |
| [61] | Tan J, McKenzie C, Potamitis M, Thorburn AN, Mackay CR, Macia L. The role of short-chain fatty acids in health and disease. Advances in Immunology, 2014, 121: 91-119. |
| [62] | Wang K, Liao M, Zhou N, Bao L, Ma K, Zheng Z, Wang Y, Liu C, Wang W, Wang J, Liu SJ, Liu H. Parabacteroides distasonis alleviates obesity and metabolic dysfunctions via production of succinate and secondary bile acids. Cell Reports, 2019, 26(1): 222-235. DOI:10.1016/j.celrep.2018.12.028 |
| [63] | Qiao S, Bao L, Wang K, Sun S, Liao M, Liu C, Zhou N, Ma K, Zhang Y, Chen Y, Liu SJ, Liu H. Activation of a specific gut bacteroides-folate-liver axis benefits for the alleviation of nonalcoholic hepatic steatosis. Cell Reports, 2020, 32(6): 108005. |
| [64] | Gilbert JA, Lynch SV. Community ecology as a framework for human microbiome research. Nature Medicine, 2019, 25(6): 884-889. |
 2021, Vol. 61
2021, Vol. 61