中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 刘庆, 杨蕾蕾, 周宇光, 辛玉华, 东秀珠. 2021
- Qing Liu, Leilei Yang, Yuguang Zhou, Yuhua Xin, Xiuzhu Dong. 2021
- 冰川细菌冷杆菌属的多样性研究进展
- Research progress on the diversity of glacial bacteria Cryobacterium
- 微生物学报, 61(4): 807-815
- Acta Microbiologica Sinica, 61(4): 807-815
-
文章历史
- 收稿日期:2020-12-25
- 修回日期:2021-02-25
- 网络出版日期:2021-02-26
2. 中国科学院微生物研究所, 微生物资源前期开发国家重点实验室, 北京 100101
2. State Key Laboratory of Microbial Resources, Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China
冰川是地球主要的冰冻圈环境之一,是多年降雪积聚形成的自然冰体。我国为低纬度山地冰川最发达的国家,主要分布于山势高大的西部边缘地区。降雪、季风、沉降以及冰川融水径流等将冰川与外界连接,形成一个动态的、特殊的生态系统。冰川中蕴藏着丰富的低温微生物资源。冰川表层环境与外界接触最为密切,其中的微生物常年处于低温、高强度紫外线辐射、周期性冻结-解冻等压力,经历了漫长的适应性进化和遗传多样化过程,在物种、生态、遗传、代谢等多个方面展现出丰富的多样性。根据生长温度特征,低温微生物可分为两类:一类是最高生长温度低于20 ℃的嗜冷微生物;另一类是在0-5 ℃可生长繁殖,最高生长温度高于20 ℃的耐冷微生物[1]。低温微生物产生的不饱和脂肪酸、低温酶等生物活性物质,在食品保健、洗涤剂、环境工程等领域具有广泛的应用前景。冰川环境常见的低温细菌有冷杆菌属(Cryobacterium)、黄杆菌属(Flavobacterium)、节杆菌属(Arthrobacter)、薄层杆菌属(Hymenobacter)、假单胞菌属(Pseudomonas)、鞘氨醇单胞菌属(Sphingomonas)等等,但严格的嗜冷菌多属于Cryobacterium和Flavobacterium等属。
冷杆菌属是革兰氏阳性细菌,被归类于放线菌门(Actinobacteria),放线菌纲(Actinobacteria),微球菌目(Micrococcales)中的微杆菌科(Microbacteriaceae)。1976年,Inoue & Komagata[2]从南极分离出了1株嗜冷细菌“Curtobacterium psychrophilum”,随后Suzuki等[3]对其进行了分类学修订,建立了Cryobacterium (冷杆菌)属。模式菌株为杆状、不产孢、菌落呈鲜艳的红色,是一株生长温度范围在0-18 ℃的嗜冷菌,因此被命名为嗜冷冷杆菌(Cryobacterium psychrophilum)。直到2007年,Zhang等[4]在中国新疆一号冰川发现了该属的第2个物种耐冷冷杆菌(Cryobacterium psychrotolerans)。目前为止,仅有15个有效发表的物种(https://lpsn.dsmz.de),其中8个物种是在2012至2020年间,由本实验室陆续从我国新疆一号冰川、甘肃透明梦珂冰川等低温环境中分离和描述。
1 冷杆菌属的分布冷杆菌属已知物种中,除Cryobacterium tepidiphilum[5]、Cryobacterium soli[6]和Cryobacterium mesophilum[7]来源于土壤或根际环境以外,其余12个种均来源于冰川或极地环境,如Cryobacterium arcticum分离自格陵兰岛[8],Cryobacterium roopkundense分离自喜马拉雅山脉的Roopkund冰川湖畔土壤[9],而C. psychrotolerans[4]、Cryobacterium flavum[10]、Cryobacterium luteum[10]、Cryobacterium levicorallinum[11]、Cryobacterium aureum[12]、Cryobacterium zongtaii[13]、Cryobacterium melibiosiphilum[14]、Cryobacterium breve[15]以及Cryobacterium ruanii[15]均分离自新疆一号冰川或甘肃透明梦珂冰川表层样品。
另外在南北极、青藏高原冻土、冰川、南极湖泊沉积物、积雪、土壤甚至沙漠植物根际土壤中均发现有冷杆菌[16-21]。尽管纯培养研究显示冷杆菌属具有较高的物种多样性[17],但对我国西部6条冰川表层样品的16S rRNA基因扩增子高通量测序,发现该属在冰川环境中的丰度很低,仅占0.02%,应属于稀有类群[20]。
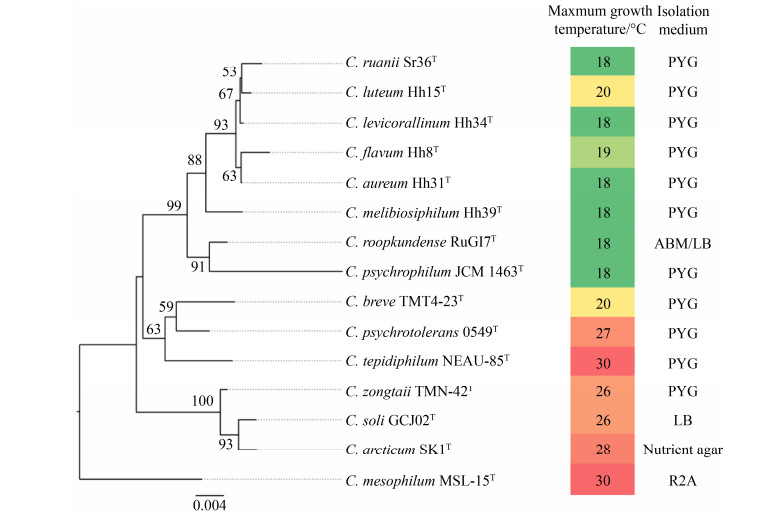
2 冷杆菌属不同种的低温适应性目前,冷杆菌属的15个种中,有9个种的最高生长温度低于20 ℃,属于严格的嗜冷菌,而另外6个种的最高生长温度在26-30 ℃之间(图 1)。从图 1系统发育树可以看出,该属的物种除C. mesophilum外,形成了3个系统发育分支,其中8株嗜冷菌形成一个大的分支,boostrap支持率达到99%;嗜冷种C. breve和2个耐冷物种C. tepidiphilum、C. psychrotolerans形成一个分支;3个耐冷物种C. soli、C. arcticum和C. zongtaii聚为一个分支。该属物种间系统发育关系和生长温度特征表明,该属在低温环境中进化产生了显著的低温适应性差异,因此,是研究低温适应性进化机制的良好材料。
|
| 图 1 基于冷杆菌属16S rRNA基因序列的系统发育树 Figure 1 Phylogenetic tree based on 16S rRNA gene sequence of the genus Cryobacterium. |
在对冷杆菌属物种低温适应性进化的研究中,Liu等[22]发现它们之间的温度耐受性存在梯度差异,而且温度耐受性与系统发育分支的聚类相关;通过全基因组分析发现,其温度耐受性差异与同义密码子突变导致的密码子第3位的GC (GC3)变异显著相关,嗜冷类群(最高生长温度≤20 ℃)偏好使用以A/U结尾的同义密码子;GC3的变异可导致嗜冷类群和耐冷类群的DNA分子具有不同的柔韧性,进而确保DNA以合适的分子动态正常行使功能,而自然选择是推动这种密码子使用偏好的主要进化动力;该研究揭示了冷杆菌菌株对低温适应性进化的分子机制。Liu等[23]通过对分离自青藏高原冰芯样品的冷杆菌和微杆菌科其它属中温菌的比较基因组学研究发现,冷杆菌基因组中与应激反应、运动性和趋化性相关的基因数量显著高于中温菌,而且冷杆菌在经历基因组扩张的进化过程中获得了一些与维生素合成、色素合成、膜转运以及单糖代谢等代谢途径相关的基因,该研究从基因组特征和基因组成的进化角度揭示了冷杆菌对冰川低温、高强度UV辐射等环境的适应机制。
3 冷杆菌的分离培养虽然多数冷杆菌栖息在冰冻圈环境,但该属的菌株均可在营养丰富的培养基中生长。除C. mesophilum MSL-15T的分离培养基为寡营养的R2A以外,其余物种的分离培养基均为PYG或LB等营养较为丰富的培养基(图 1)。本实验室冰川耐低温冷杆菌菌株均在14 ℃条件下,利用PYG培养基(W/V,细菌蛋白胨0.5%,酵母提取物0.02%,葡萄糖0.5%,牛肉膏0.3%,NaCl 0.05%,MgSO4·7H2O 0.15%,pH 7.0)分离得到[20]。Liu等[24]总结,冷杆菌属已知种大多可在2%-3% NaCl (M/V)浓度生长,而冰川环境优势的黄杆菌属菌株往往不能在高于0.5% NaCl (M/V)浓度中生长[25-27]。因此,对冷杆菌的分离培养,可尝试提高培养基中NaCl的浓度,以抑制一些优势类群的生长。
4 冷杆菌属的物种多样性目前GenBank中共有冷杆菌纯培养物的基因组序列109条,去除重复后共99条,其中78条为本实验室分析测定[22]。本研究利用BioSAK工具(https://github.com/songweizhi/BioSAK)在GenBank中获取99条全基因组序列,采用FastANI工具[28]计算菌株两两之间平均核酸序列一致性(average nucleotide identity,ANI)。根据ANI值和聚类分析结果显示,99个全基因组可划分为41个物种(图 2)。说明目前已获得纯培养的冷杆菌属菌株至少有41个种,因此,仍有至少26个新物种有待有效描述。根据基因组ANI值聚类分析,该稀有类群具有较高的系统发育多样性(图 3),一些尚未被鉴定描述的物种具有很高的种内多样性。也说明基于16S rRNA基因序列的已知物种系统发育分析(图 1)远远低估了冷杆菌的物种多样性。

|
| 图 2 Cryobacterium菌株基于全基因组ANI值95%为阈值的物种划分 Figure 2 Species delineation of Cryobacterium spp. based on 95% ANI value of genome sequences. |

|
| 图 3 基于全基因组ANI值的聚类分析 Figure 3 Cluster analysis based on whole genome ANI values. The dotted lines represent 95% and 96% of the ANI values, respectively. |
5 冷杆菌的应用潜力
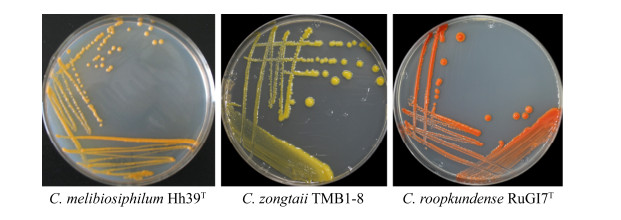
冷杆菌属菌落多呈黄色、橙色、红色等鲜艳颜色(图 4)。Vila等[29]利用高压液相色谱和液相色谱-质谱联用等技术发现,分离自南极的冷杆菌菌株可产类胡萝卜素C.p. 450,因此具有食品、化妆品等工业应用潜力。庹利等[21]在研究罗布泊地区植物根际放线菌时,分离到一株具有抗菌活性的冷杆菌,不仅扩展了我们对于该属生态分布的认识,也表明该属是可产生活性代谢产物的潜在资源。倪永清等[30]在新疆一号冰川底部沉积层样品中分离到2株产低温蛋白酶的Cryobacterium菌株,低温蛋白酶在食品加工、洗涤剂、医用疫苗和生物保鲜等多个方面具有应用优势。Gong等[31]通过对菌株C. soli GCJ02的谷氨酰胺合成酶基因的功能研究发现其具有农业应用潜力。Wang等[32]在一株嗜冷菌Cryobacterium sp. LW097中发现了4种新的低温β-半乳糖苷酶,将这4种β-半乳糖苷酶分别在大肠杆菌中表达纯化后发现,它们在5 ℃时仍然保持着较高的相对酶活性,对低乳糖牛奶生产等奶制品加工领域具有应用前景。因此,对冷杆菌物种资源的挖掘和多样性研究,可为低温酶工程、食品加工、医药卫生等行业奠定基础。
|
| 图 4 冷杆菌属菌株典型菌落颜色示例 Figure 4 Examples of colony color of Cryobacterium spp.. |
6 展望
冰川生态系统中包括了3个生命域的生物。光照、降雪、季风、沉降以及冰川融水径流等使得冰川形成了一个特殊的生态系统,在参与生物地球化学循环过程起到非常重要的作用。栖息在冰川环境中的细菌物种多样性非常高。得益于高通量测序技术的发展,目前冰川环境中细菌的群落组成已逐渐清晰。宏基因组学等非培养技术的使用,不仅可以获取冰川细菌的物种多样性,且有利于对冰川细菌群落动态演替规律及构建机制等生态学问题进行深入研究。但是,组学方法并不能替代基于培养物的生理功能及遗传进化等研究。在一些未培养的细菌类群中,半数以上的基因功能都无法预测,甚至85%的基因无法归类到任何已知的代谢途径[33]。因此,分离培养在微生物研究中仍然具有不可替代的重要性,只有将微生物菌种在实验室培养和保藏,才有可能对微生物资源进行可持续的研究和开发利用。以冷杆菌属为代表的冰川低温细菌,采用低温复苏等培养方法,目前已有数千株纯培养物[17, 20],尚有大量新物种有待研究,新资源挖掘潜力巨大。
冷杆菌属广泛分布于低温环境中,经过漫长的进化,形成了特殊的抗冻生理特征。该属的基因组学研究表明其具有丰富的遗传多样性,但在冰川等低温环境中的进化历史和物种形成机制目前尚不明确。因此,该类群是开展理论和功能研究的宝贵资源。不断挖掘冷杆菌新的物种资源,提高物种描述率,研究其物种进化和形成机制,进一步开发低温生物活性物质,探索适应低温的分子机制,是未来研究的重要方向,能够使我们更加深刻理解冰川环境生态系统。
| [1] | Morita RY. Psychrophilic bacteria. Bacteriological Reviews, 1975, 39(2): 144-167. DOI:10.1128/BR.39.2.144-167.1975 |
| [2] | Inoue K, Komagata K. Taxonomic study on obligately psychrophilic bacteria isolated from Antarctica. The Journal of General and Applied Microbiology, 1976, 22(4): 165-176. DOI:10.2323/jgam.22.165 |
| [3] | Suzuki KI, Sasaki J, Uramoto M, Nakase T, Komagata K. Cryobacterium psychrophilum gen. nov., sp. nov., nom. rev., comb. nov., an obligately psychrophilic actinomycete to accommodate "Curtobacterium psychrophilum" Inoue and Komagata 1976. International Journal of Systematic Bacteriology, 1997, 47(2): 474-478. DOI:10.1099/00207713-47-2-474 |
| [4] | Zhang DC, Wang HX, Cui HL, Yang Y, Liu HC, Dong XZ, Zhou PJ. Cryobacterium psychrotolerans sp. nov., a novel psychrotolerant bacterium isolated from the China No. 1 glacier. International Journal of Systematic and Evolutionary Microbiology, 2007, 57(4): 866-869. DOI:10.1099/ijs.0.64750-0 |
| [5] | Wang Y, Cao P, Sun P, Zhao JW, Sun XJ, Zhang J, Li CX, Xiang WS, Wang XJ. Cryobacterium tepidiphilum sp. nov., isolated from rhizosphere soil of lettuce (var. ramosa Hort.). Antonie Van Leeuwenhoek, 2019, 112(11): 1611-1621. DOI:10.1007/s10482-019-01287-3 |
| [6] | Gong CJ, Lai QL, Cai HF, Jiang YM, Liao H, Liu YF, Xue DS. Cryobacterium soli sp. nov., isolated from forest soil. International Journal of Systematic and Evolutionary Microbiology, 2020, 70(1): 675-679. DOI:10.1099/ijsem.0.003820 |
| [7] | Dastager SG, Lee JC, Ju YJ, Park DJ, Kim CJ. Cryobacterium mesophilum sp. nov., a novel mesophilic bacterium. International Journal of Systematic and Evolutionary Microbiology, 2008, 58(5): 1241-1244. DOI:10.1099/ijs.0.65650-0 |
| [8] | Bajerski F, Ganzert L, Mangelsdorf K, Lipski A, Wagner D. Cryobacterium arcticum sp. nov., a psychrotolerant bacterium from an Arctic soil. International Journal of Systematic and Evolutionary Microbiology, 2011, 61(8): 1849-1853. DOI:10.1099/ijs.0.027128-0 |
| [9] | Reddy GSN, Pradhan S, Manorama R, Shivaji S. Cryobacterium roopkundense sp. nov., a psychrophilic bacterium isolated from glacial soil. International Journal of Systematic and Evolutionary Microbiology, 2010, 60(4): 866-870. DOI:10.1099/ijs.0.011775-0 |
| [10] | Liu Q, Liu HC, Wen Y, Zhou YG, Xin YH. Cryobacterium flavum sp. nov. and Cryobacterium luteum sp. nov., isolated from glacier ice. International Journal of Systematic and Evolutionary Microbiology, 2012, 62(Pt_6): 1296-1299. DOI:10.1099/ijs.0.033738-0 |
| [11] | Liu Q, Liu HC, Zhang JL, Zhou YG, Xin YH. Cryobacterium levicorallinum sp. nov., a psychrophilic bacterium isolated from glacier ice. International Journal of Systematic and Evolutionary Microbiology, 2013, 63(Pt_8): 2819-2822. DOI:10.1099/ijs.0.046896-0 |
| [12] | Liu Q, Xin YH, Chen XL, Liu HC, Zhou YG, Chen WX. Cryobacterium aureum sp. nov., a psychrophilic bacterium isolated from glacier ice collected from the ice tongue surface. International Journal of Systematic and Evolutionary Microbiology, 2018, 68(4): 1173-1176. DOI:10.1099/ijsem.0.002647 |
| [13] | Liu Q, Liu HC, Zhou YG, Xin YH. Genetic diversity of glacier-inhabiting Cryobacterium bacteria in China and description of Cryobacterium zongtaii sp. nov. and Arthrobacter glacialis sp. nov. Systematic and Applied Microbiology, 2019, 42(2): 168-177. DOI:10.1016/j.syapm.2018.10.005 |
| [14] | Liu Q, Tian JH, Liu HC, Zhou YG, Xin YH. Cryobacterium melibiosiphilum sp. nov., a psychrophilic bacterium isolated from glacier ice. International Journal of Systematic and Evolutionary Microbiology, 2019, 69(10): 3276-3280. DOI:10.1099/ijsem.0.003620 |
| [15] | Liu Q, Tian JH, Liu HC, Zhou YG, Xin YH. Cryobacterium ruanii sp. nov. and Cryobacterium breve sp. nov., isolated from glaciers. International Journal of Systematic and Evolutionary Microbiology, 2020, 70(3): 1918-1923. DOI:10.1099/ijsem.0.003994 |
| [16] | Segawa T, Miyamoto K, Ushida K, Agata K, Okada N, Kohshima S. Seasonal change in bacterial flora and biomass in mountain snow from the tateyama mountains, Japan, analyzed by 16S rRNA gene sequencing and real-time PCR. Applied and Environmental Microbiology, 2005, 71(1): 123-130. DOI:10.1128/AEM.71.1.123-130.2005 |
| [17] | Peeters K, Ertz D, Willems A. Culturable bacterial diversity at the Princess Elisabeth Station (Utsteinen, Sør Rondane Mountains, East Antarctica) harbours many new taxa. Systematic and Applied Microbiology, 2011, 34(5): 360-367. DOI:10.1016/j.syapm.2011.02.002 |
| [18] | Shivaji S, Kumari K, Kishore KH, Pindi PK, Rao PS, Radha Srinivas TN, Asthana R, Ravindra R. Vertical distribution of bacteria in a lake sediment from Antarctica by culture-independent and culture-dependent approaches. Research in Microbiology, 2011, 162(2): 191-203. DOI:10.1016/j.resmic.2010.09.020 |
| [19] | Singh P, Singh SM, Dhakephalkar P. Diversity, cold active enzymes and adaptation strategies of bacteria inhabiting glacier cryoconite holes of High Arctic. Extremophiles, 2014, 18(2): 229-242. DOI:10.1007/s00792-013-0609-6 |
| [20] | Liu Q, Zhou YG, Xin YH. High diversity and distinctive community structure of bacteria on glaciers in China revealed by 454 pyrosequencing. Systematic and Applied Microbiology, 2015, 38(8): 578-585. DOI:10.1016/j.syapm.2015.09.005 |
| [21] |
Tuo L, Xugela H, Guo L, Zhang YQ, Tao L, Wang FF, Liu JM, Jiang ZK, Pan Z, Zhang YB, Sun CH. Studies on diversity and bioactivity of rhizosphere actinomycetes from psammophyte in Lop Nor Region. Chinese Journal of Antibiotics, 2012, 37(1): 21-26.
(in Chinese) 庹利, 旭格拉·哈布丁, 郭琳, 张玉琴, 陶玲, 王飞飞, 刘佳萌, 蒋忠科, 潘臻, 张玉彬, 孙承航. 罗布泊地区沙生植物根际放线菌多样性及生物活性的研究. 中国抗生素杂志, 2012, 37(1): 21-26. |
| [22] | Liu Q, Song WZ, Zhou YG, Dong XZ, Xin YH. Phenotypic divergence of thermotolerance: Molecular basis and cold adaptive evolution related to intrinsic DNA flexibility of glacier-inhabiting Cryobacterium strains. Environmental Microbiology, 2020, 22(4): 1409-1420. DOI:10.1111/1462-2920.14957 |
| [23] | Liu YQ, Shen L, Zeng YH, Xing TT, Xu BQ, Wang NL. Genomic insights of Cryobacterium isolated from ice core reveal genome dynamics for adaptation in glacier. Frontiers in Microbiology, 2020, 11: 1530. DOI:10.3389/fmicb.2020.01530 |
| [24] | Liu Q, Zhou YG, Xin YH. Cryobacterium. //Whitman WB. Bergey's Manual of Systematics of Archaea and Bacteria. New York: John Wiley and Sons, 2018. |
| [25] | Yang LL, Liu Q, Liu HC, Zhou YG, Xin YH. Flavobacterium laiguense sp. nov., a psychrophilic bacterium isolated from Laigu glacier on the Tibetan Plateau. International Journal of Systematic and Evolutionary Microbiology, 2019, 69(6): 1821-1825. DOI:10.1099/ijsem.0.003400 |
| [26] | Zhang GQ, Liu Q, Liu HC, Zhou YG, Xin YH. Flavobacterium ranwuense sp. nov., isolated from glacier. International Journal of Systematic and Evolutionary Microbiology, 2019, 69(12): 3812-3817. DOI:10.1099/ijsem.0.003687 |
| [27] | Yang LL, Liu Q, Liu HC, Zhou YG, Xin YH. Flavobacterium psychrotolerans sp. nov., a psychrotolerant bacterium isolated from Renlongba glacier on the Tibetan Plateau. International Journal of Systematic and Evolutionary Microbiology, 2020, 70(2): 1031-1036. DOI:10.1099/ijsem.0.003868 |
| [28] | Jain C, Rodriguez-R LM, Phillippy AM, Konstantinidis KT, Aluru S. High throughput ANI analysis of 90K prokaryotic genomes reveals clear species boundaries. Nature Communications, 2018, 9: 5114. DOI:10.1038/s41467-018-07641-9 |
| [29] | Vila E, Hornero-Méndez D, Azziz G, Lareo C, Saravia V. Carotenoids from heterotrophic bacteria isolated from Fildes Peninsula, King George Island, Antarctica. Biotechnology Reports, 2019, 21: e00306. DOI:10.1016/j.btre.2019.e00306 |
| [30] |
Ni YQ, Gu YL, Shi XW, Zheng XJ, Han L, Zhou H, Cheng GD. Phylogenetic and physiological diversity of cold-adapted bacteria producing protease from sediments of the bottom layer of the Glacier No.1 in the Tianshan Mountains. Acta Microbiologica Sinica, 2013, 53(2): 164-172.
(in Chinese) 倪永清, 顾燕玲, 史学伟, 郑晓吉, 韩亮, 周红, 程国栋. 天山一号冰川底部沉积层产蛋白酶耐低温菌株的筛选及其系统发育. 微生物学报, 2013, 53(2): 164-172. |
| [31] | Gong CJ, You XH, Zhang SY, Xue DS. Functional analysis of a glutamine biosynthesis protein from a psychrotrophic bacterium, Cryobacterium soli GCJ02. Indian Journal of Microbiology, 2020, 60(2): 153-159. DOI:10.1007/s12088-020-00858-7 |
| [32] | Wang L, Mou Y, Guan B, Hu Y, Zhang Y, Zeng J, Ni Y. Genome sequence of the psychrophilic Cryobacterium sp. LW097 and characterization of its four novel cold-adapted β-galactosidases. International Journal of Biological Macromolecules, 2020, 163: 2068-2083. DOI:10.1016/j.ijbiomac.2020.09.100 |
| [33] | Overmann J, Abt B, Sikorski J. Present and future of culturing bacteria. Annual Review of Microbiology, 2017, 71: 711-730. DOI:10.1146/annurev-micro-090816-093449 |
 2021, Vol. 61
2021, Vol. 61