中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 杜宇, 王杰, 蒋海宾, 王秀娜, 范元婵, 范小雪, 祝智威, 隆琦, 张文德, 熊翠玲, 郑燕珍, 付中民, 陈大福, 郭睿. 2021
- Yu Du, Jie Wang, Haibin Jiang, Xiuna Wang, Yuanchan Fan, Xiaoxue Fan, Zhiwei Zhu, Qi Long, Wende Zhang, Cuiling Xiong, Yanzhen Zheng, Zhongmin Fu, Dafu Chen, Rui Guo. 2021
- 基于第三代长读段测序数据解析蜜蜂球囊菌基因的可变剪切与可变腺苷酸化
- Analysis of alternative splicing and polyadenylation of Ascosphaera apis genes based on third-generation long-read sequencing dataset
- 微生物学报, 61(3): 667-682
- Acta Microbiologica Sinica, 61(3): 667-682
-
文章历史
- 收稿日期:2020-05-15
- 修回日期:2020-08-26
- 网络出版日期:2020-12-11
2. 福建农林大学蜂产品加工与应用教育部工程研究中心, 福建 福州 350002;
3. 福建农林大学生命科学学院, 福建 福州 350002;
4. 福建省病原真菌与真菌毒素重点实验室, 福建 福州 350002
2. Engineering Research Center of Processing and Application of Bee Products of Ministry of Education, Fujian Agriculture and Forestry University, Fuzhou 350002, Fujian Province, China;
3. College of Life Sciences, Fujian Agriculture and Forestry University, Fuzhou 350002, Fujian Province, China;
4. Key Laboratory of Pathogenic Fungi and Mycotoxins of Fujian Province, Fuzhou 350002, Fujian Province, China
蜜蜂球囊菌(Ascosphaera apis,简称球囊菌)专性侵染蜜蜂幼虫而引发白垩病,该病可导致成年蜜蜂数量、蜂群群势和蜂产品产量的明显下降,长期困扰养蜂生产[1-2]。球囊菌作为专性寄生蜜蜂幼虫的真菌病原,在与宿主的长期协同进化过程形成了特殊的侵染模式。与球孢白僵菌(Beauveria bassiana)和金龟子绿僵菌(Metarhizium anisopliae)等昆虫病原真菌不同,球囊菌不能通过分泌水解酶以及机械压力从宿主体表进行侵染,其孢子经成年蜜蜂的哺育行为被蜜蜂幼虫经口摄入是其侵染宿主的唯一方式,孢子在宿主肠道内环境的刺激下低水平萌发,并在宿主预蛹期前2 d迅速生长为具有致病性的菌丝体,期间分泌各类几丁质酶、蛋白酶、脂酶及次级代谢物等毒力因子共同抑制宿主的免疫防御系统,进而降解围食膜、肠壁和体壁,最终在预蛹期穿透尾部,逐渐蔓延包裹整个虫尸[3]。Qin等[4]于2006年利用Sanger测序技术对球囊菌的0.5-1A和A10菌株进行了基因组测序和组装,公布了球囊菌的基因序列信息,但作者没有同时公布基因功能注释信息,导致该基因组版本长期无法使用。此后,Shang等[5]利用二代测序技术对球囊菌的ARSEF 7405菌株重新进行了基因组测序和组装,公布了完整的基因序列和功能注释,为球囊菌的组学和分子生物学提供了必要基础。
一个基因转录形成的前体mRNA (pre-mRNA)通过不同的剪切方式产生不同成熟mRNA的过程称之为可变剪切(alternative splicing,AS)[6-7]。通过AS,基因表达出具有不同结构和功能特性的蛋白产物,或相同的细胞出现不同表型,因而能够大幅增加细胞、组织和个体的转录组与蛋白组的复杂性[8-9]。人类[10]、猪[11]和拟南芥(Arabidopsis thaliana)[12]等真核生物的基因普遍发生AS。例如,人类的95%左右的多外显子基因转录形成的pre-mRNA发生AS[10]。大量的研究结果表明AS在真核生物的基因表达调控、生长发育、环境适应、胁迫应答及疾病发生等生物学过程发挥关键作用[6, 8, 12-13]。此前,人们利用二代测序技术对动植物和微生物的AS事件进行了较多研究[14-17]。但由于测序读段长度较短,二代测序技术对剪切异构体(isoform)的发掘能力有限,特别是对于多倍体物种和重复元件丰富的基因组[18]。
对于成熟mRNA,3’端的多聚腺苷酸poly-A尾巴可保护其免受核酸外切酶攻击[19]。此外,poly-A尾巴对成熟mRNA的稳定性、出核运输、细胞质定位及翻译也十分重要[20]。所谓可变多聚腺苷酸化(alternative polyadenylation,APA),是指通过poly-A尾巴的AS,来源于同一基因的不同转录本含有不同长度的3’非翻译区(un-translated region,UTR)[21]。APA已被证实能通过形成含有不同编码区和3’ UTR的转录本增加动植物转录组的多样性,进而通过多种机制对基因表达进行调控[22]。此前,由于基于二代测序得到的短读段无法获得转录本的全长信息,无法对转录本进行APA分析。
近年来,第三代测序技术逐渐兴起并在真核生物的转录组研究领域崭露头角[23-26]。三代测序产生的超长读段不需要拼接,能够精确鉴定同一个基因形成的不同isoform[27-28]。目前,三代测序已成功应用于人、小鼠和高粱等动植物的AS和APA相关研究[28-31]。牛津纳米孔(Oxford Nanopore)测序技术的原理是DNA/RNA双链在马达蛋白的牵引下与镶嵌在生物膜上的Nanopore蛋白结合并解螺旋,然后DNA/RNA链在生物膜两侧电压差的作用下以一定的速率通过Nanopore通道蛋白,因不同碱基化学性质存在差异,故单个碱基或DNA分子通过Nanopore通道时会引起不同电学信号的变化[32],通过对这些信号进行检测及对应即可计算获得相应碱基的类型,从而完成序列的实时测定[33]。笔者所在课题组前期已利用Nanopore测序技术对球囊菌的纯化菌丝(Aam)和纯化孢子(Aas)分别进行测序,分别鉴定到9859和16795条非冗余全长转录本[34];通过对上述全长转录本进行了蛋白功能注释,此外还鉴定到648条高可信度的长链非编码RNA (long non-coding RNA,lncRNA)[35]。菌丝和孢子是球囊菌的两种不同形态,不同性别的球囊菌菌丝通过杂交产孢形成孢子;球囊菌在侵染蜜蜂幼虫的过程中也伴随着孢子的剧烈萌发和菌丝的大量生长[36]。因此,探究球囊菌菌丝和孢子中基因的AS与APA,对于丰富和拓展球囊菌的基础生物学以及深入理解球囊菌生长、发育、生殖和致病性的相关机理具有重要意义。本研究基于已获得的高质量Nanopore长读段测序数据对球囊菌菌丝和孢子中基因的AS和APA行分析和探讨,旨在揭示球囊菌转录组的复杂性,并为深入探究isoform的分子功能提供参考信息和理论依据。
1 材料和方法 1.1 供试球囊菌球囊菌菌株[3, 37-38]由福建农林大学动物科学学院(蜂学学院)蜜蜂保护实验室分离与活化。
1.2 全长转录组数据来源前期研究中,笔者课题组参考已报道的方法[39]在实验室条件下进行球囊菌菌丝和孢子的培养和纯化[40],将保存的球囊菌孢子转接至PDA固体培养基上,置于生化培养箱,在33.0±0.5 ℃条件下培养7 d后,可见培养基平板上层为白色菌丝,覆盖着下层的黑色孢子囊,且白色菌丝较厚较蓬松。为获取培养基表面最上层的白色菌丝,同时避免菌丝与孢子之间的交叉污染,首先在超净台中利用接种环小心将培养基表面最上层的白色菌丝刮至RNA-free的EP管(共计100 mg),并要避免接触与孢子囊接触的菌丝,然后进行显微制片和镜检(不含黑色孢子),即为球囊菌的纯净菌丝;为得到纯净孢子,先通过无菌操作将覆盖在黑色孢子囊上的薄层菌丝刮去,再用另一个干净的接种环将孢子囊刮至干净的EP管;继而按照Jensen等[39]的方法进行差速离心,球囊菌孢子密度较大,沉淀在EP管底部,弃去上清;重复上述差速离心3次,弃去上清;取少量孢子制备孢子悬液,取少量悬液进行显微制片及观察(400倍),视野中可见较多的游离孢子,未见菌丝。收集的纯化菌丝和纯化孢子放置–80 ℃冷冻保存。
利用TRizol试剂盒(Thermo Fisher公司,美国)分别提取Aam和Aas的总RNA;引物退火,利用Maxima H Minus Reverse Transcriptase试剂盒(Thermo Fisher公司,美国)进行反转录,得到的cDNA添加switch oligo,再合成互补链;对DNA进行损伤修复和末端修复,再利用磁珠对cDNA进行纯化[35]。利用Oxford Nanopore PromethION平台对Aam和Aas的cDNA文库分别进行测序,分别测得6321704和6259727条原始读段(raw reads),居中长度(N50)、平均读长和最大读长分别为1094 bp和1157 bp、992 bp和1047 bp以及9421 bp和13060 bp;分别鉴定出9859和16795条非冗余全长转录本,N50、平均长度和最大长度分别为1482 bp和1658 bp、1187 bp和1303 bp以及6472 bp和6815 bp[34]。高质量的长读段测序数据可为本研究中球囊菌基因的AS和APA分析提供可靠的数据支撑。测序原始数据已上传NCBI SRA数据库,获得BioProject号:PRJNA645872。
1.3 共有和特有非冗余全长转录本的表达量计算和数据库注释前期研究中,笔者通过Venn分析筛选出Aam和Aas的6512条共有非冗余全长转录本,以及3347和10283条特有非冗余全长转录本[35]。为了让转录组测序所得片段数目能真实地反映转录本表达水平,首先对样品中的mapped reads的数目进行归一化,然后采用CPM (counts per million)算法计算上述非冗余全长转录本的表达水平。CPM计算公式如下:CPM=reads mapped to transcript/total reads aligned in sample×1000000
利用BLAST工具将上述共有和特有非冗余全长转录本比对GO (Gene Ontology)[41]和KEGG (Kyoto Encyclopedia of Genes and Genomes)[42]数据库比对,以获得功能和通路注释信息。
1.4 基因的AS分析AS的类型主要包括外显子跳跃(skipping exon,SE)、内含子保留(retained intron,RI)、可变5’剪切位点(alternative 5’ splice-site,A5)、可变3’剪切位点(alternative 3’ splice-site,A3)、外显子互斥(mutually exclusive exons,MEE)[17]。参照陈华枝等的方法[43],利用Astalavista软件[44]鉴定球囊菌基因的AS类型,采用默认参数。根据鉴定结果对基因的AS事件类型进行统计,并利用百迈克云平台的相关工具(https://international.biocloud.net/zh/software/tools/pieChart)绘制饼图。参照Foissac等的方法[44],利用IGV (integrative genomics viewer)浏览器对部分isoform的结构进行可视化。
1.5 基因的APA分析参照陈华枝等的方法[43],利用TAPIS pipeline[28]对球囊菌基因的APA位点进行鉴定,采用默认参数。参照Abde等[28]和Chao等[29]方法,通过MEME软件[45]对全部转录本的poly-A剪切位点上游50 bp的序列特征进行分析以鉴定motif,参数设置为:-norc,-meme-minw 6,-meme-maxw 6,-spamo-skip,-fimo-skip。
2 结果和分析 2.1 球囊菌基因的AS数量及类型在Aam中共鉴定到286次AS事件,包括162 (56.64%)次RI,87 (30.42%)次A3,32 (11.19%)次A5和5 (1.75%)次SE (图 1-A、B);AS事件的具体信息详见表 1。不同isoform的表达量介于0.17–76721.37,其中表达量最高和最低的isoform分别为ONT.6598.1 (CPM=76721.37)和ONT.985.8 (CPM=0.17)。

|
| 图 1 球囊菌基因的可变剪切类型统计 Figure 1 Type statistics of AS of A. apis genes. A: Diagram showing five types of AS events; B: Pie chart showing types of AS events of genes in mycelium; C: Pie chart showing types of AS events of genes in spore. Aam represents A. apis mycelium and Aas represents A. apis spore. |
| Type of AS | Start site/bp | End site/bp | Strand | Gene location |
| A3 | 254851 | 254856 | + | AZGZ01000001.1:254525–255804 |
| A3 | 818470 | 818493 | + | AZGZ01000001.1:818161–820873 |
| A3 | 928129 | 928142 | + | AZGZ01000001.1:927581–929692 |
| A3 | 928962 | 928974 | + | AZGZ01000001.1:927581–929692 |
| A3 | 1341809 | 1341810 | + | AZGZ01000001.1:1340323–1343134 |
| A5 | 721398 | 721413 | + | AZGZ01000001.1:720880–722868 |
| A5 | 330633 | 330594 | – | AZGZ01000001.1:329730–330758 |
| A5 | 220693 | 221278 | + | AZGZ01000002.1:220545–222739 |
| A5 | 583693 | 583661 | – | AZGZ01000002.1:582773–583898 |
| A5 | 274350 | 274421 | + | AZGZ01000003.1:273701–275565 |
| SE | 777152 | 777133 | – | AZGZ01000005.1:776046–777410 |
| SE | 754660 | 754643 | – | AZGZ01000007.1:753188–754998 |
| SE | 46883 | 46866 | – | AZGZ01000008.1:45508–47357 |
| SE | 285114 | 285098 | – | AZGZ01000008.1:283981–285853 |
| SE | 302717 | 302423 | – | AZGZ01000011.1:301809–303170 |
| RI | 255071 | 255141 | + | AZGZ01000001.1:254525–255804 |
| RI | 526700 | 526904 | + | AZGZ01000001.1:525686–527670 |
| RI | 528923 | 528984 | + | AZGZ01000001.1:528547–530058 |
| RI | 1077352 | 1077421 | + | AZGZ01000001.1:1076947–1078664 |
| RI | 1185364 | 1185484 | + | AZGZ01000001.1:1184640–1186772 |
在Aas中共鉴定到559次AS事件,包括305 (54.56%)次RI、155 (27.73%)次A3、85 (15.21%)次A5、13 (2.33%)次SE和1 (0.18%)次MEE (图 1-A、C);AS事件的具体信息详见表 2。不同isoform的表达量为0.16–20116.28,其中表达量最高和最低的isoform分别为ONT.7346.1 (CPM=20116.28)和ONT.994.2 (CPM=0.16)。
| Type of AS | Start site/bp | End site/bp | Strand | Gene location |
| A3 | 818470 | 818493 | + | AZGZ01000001.1:818205–820869 |
| A3 | 928129 | 928142 | + | AZGZ01000001.1:927079–929693 |
| A3 | 1402216 | 1402245 | + | AZGZ01000001.1:1401448–1405777 |
| A3 | 1509288 | 1509295 | + | AZGZ01000001.1:1509079–1511363 |
| A3 | 1076025 | 1076021 | – | AZGZ01000001.1:1074627–1076347 |
| A3 | 654903 | 654905 | + | AZGZ01000001.1:648415–655394 |
| A3 | 721398 | 721413 | + | AZGZ01000001.1:720944–722880 |
| A3 | 932019 | 932021 | + | AZGZ01000001.1:930341–933203 |
| A3 | 1509781 | 1509782 | + | AZGZ01000001.1:1509079–1511363 |
| A3 | 376981 | 376980 | – | AZGZ01000001.1:376471–379983 |
| SE | 786654 | 786597 | – | AZGZ01000001.1:786107–786952 |
| SE | 521195 | 521231 | + | AZGZ01000003.1:519787–521776 |
| SE | 698962 | 698863 | – | AZGZ01000007.1:696402–699251 |
| SE | 754660 | 754643 | – | AZGZ01000007.1:753188–754916 |
| SE | 285114 | 285098 | – | AZGZ01000008.1:283988–285853 |
| RI | 125314 | 125451 | + | AZGZ01000001.1:124879–126265 |
| RI | 207699 | 208059 | + | AZGZ01000001.1:207374–209587 |
| RI | 255071 | 255141 | + | AZGZ01000001.1:254245–255805 |
| RI | 528923 | 528984 | + | AZGZ01000001.1:528065–530064 |
| RI | 677524 | 677598 | + | AZGZ01000001.1:677252–678531 |
| MEE | 322090 | 322264 | + | AZGZ01000011.1:321773–323412 |
将本研究鉴定到的全长转录本与球囊菌参考基因组注释基因进行比较,发现多数注释基因的结构并不完整;对于同一注释基因,在球囊菌菌丝中转录形成的isoform与孢子中转录形成的isoform在数量和结构方面均存在差异(图 2-A、B)。此外,还发现部分isoform在参考基因组中没有对应的注释基因(图 2-C),说明现有的参考基因组注释需要进一步完善。

|
| 图 2 球囊菌参考基因组上部分已注释和未注释转录本的IGV浏览器视图 Figure 2 IGV browser view of partial annotated and unannotated transcripts in A. apis reference genome. A: AAZGZ01000001.1:254525–255804W; B: AAZGZ01000005.1:776046–777410C; C: AAZGZ01000001.1: 329730–330758. Black rectangles indicate transcript annotated in reference genome. White rectangles represent scaffolds, and numbers above indicate positions on scaffolds; red and purple rectangles represent exons, and arrows indicate the direction of transcription; red and purple lines represent introns. |
2.2 球囊菌全长转录本数量及功能注释
Aam和Aas的共有非冗余全长转录本有6512条,特有非冗余全长转录本分别为3347和10283条[36]。GO数据库注释结果显示,3526条共有全长转录本可注释到44个功能条目,包括14个细胞组分大类相关条目(细胞膜、细胞器和大分子复合物等),14个分子功能大类相关条目(结构分子活性、电子载体活性和分子功能调节器等),以及16个生物学进程大类相关条目(生殖、发育进程和应激反应等);Aam的1330条特有全长转录本可注释到39个功能条目,其中与细胞组分、分子功能和生物学进程相关的条目分别为13、11和15个;Aas的3865条特有全长转录本可注释到43个功能条目,其中与细胞组分、分子功能和生物学进程相关的条目分别为14、14和15个;进一步分析发现,生物附着条目仅被共有全长转录本所注释,而未被Aam和Aas的特有全长转录本所注释;Aam的特有全长转录本所注释的条目均被Aas的特有全长转录本所注释;拟核、金属蛋白伴侣活性、翻译调节活性、活动分子传感器等条目被Aas的特有全长转录本所注释,而未被Aam的特有全长转录本所注释。
KEGG数据库注释结果显示,3628条共有全长转录本可注释到119条通路,注释数量最多的是抗生素的生物合成(286)、核糖体(231)、碳代谢(142)、氨基酸的生物合成(141)及氧化磷酸化(118);Aam的1469条特有全长转录本可注释到100条通路,注释数量最多的是核糖体(202)、抗生素的生物合成(96)、碳代谢(61)、氨基酸的生物合成(50)及RNA转运(50);Aas的3499条特有全长转录本可注释到117条通路,注释数量最多的是抗生素的生物合成(232)、氨基酸的生物合成(131)、剪接体(111)、碳代谢(106)及核糖体(106);进一步分析发现,黄曲霉毒素的生物合成通路仅被共有全长转录本所注释,而未被Aam和Aas的特有全长转录本所注释;其他多糖降解通路被Aam的特有全长转录本所注释,而未被Aas的特有全长转录本所注释;卟啉和叶绿素代谢、维生素B6代谢、硫中继系统、叶酸的生物合成、单菌霉素的生物合成、阿特拉津的降解、酮体的合成和降解、青霉素和头孢菌素的生物合成、碳青霉烯的生物合成、糖胺聚糖降解、亚麻酸代谢、鞘糖脂的生物合成、内酰胺抗药性、硫胺素新陈代谢、花生四烯酸代谢、亚油酸的新陈代谢、类胡萝卜素的生物合成、长寿调节途径等18条通路被Aas的特有全长转录本所注释,而未被Aam的特有全长转录本所注释。
2.3 球囊菌基因的APA位点数量及上下游序列特征在Aam中共鉴定到2748个基因含有1个及以上的APA位点,其中含有1个APA位点的基因数量最多,达到726 (26.42%)个,含有2个APA位点的基因数量次之,为585 (21.29%)个,另有474 (17.25%)个基因含有5个以上的APA位点(图 3-A)。在Aas中共鉴定到2768个基因含有1个及以上的APA位点,其中含有5个以上APA位点的基因数量最多(1180,42.63%),其次为含有1个APA位点的基因,数量为380 (13.73%)个(图 3-B)。

|
| 图 3 球囊菌菌丝(A)和孢子(B)中含有不同APA位点的基因数量统计 Figure 3 Number statistics of genes with various APA sites in A. apis mycelium (A) and spore (B). The number above each column indicates the total transcripts with a certain number of APA sites. |
进一步分析发现,同一个基因在Aam和Aas中含有不同的APA位点数,例如Aam中有2条clean reads能比对到gene 5,gene 5含有1个APA位点(图 4-A);但Aas中有23条clean reads能比对到gene 5,gene 5含有6个APA位点(图 4-C);Aam中有9条clean reads能比对到gene 8,gene 8含有1个APA位点(图 4-B);但Aas中有33条clean reads能比对到gene 8,gene 8含有5个APA位点(图 4-D)。

|
| 图 4 球囊菌部分基因的3’ UTR含有的APA位点 Figure 4 APA sites within 3' UTRs of partial A. apis genes. A: The number of APA sites within 3' UTRs of A. apis mycelium gene 5; B: The number of APA sites within 3' UTRs of A. apis mycelium gene 8; C: The number of APA sites within 3' UTRs of A. apis spore gene 5; D: The number of APA sites within 3' UTRs of A. apis spore gene 8. |
进一步分析发现,球囊菌全长转录本的3’UTR的上下游表现出明显的碱基倾向性,U在3’UTR的上游富集,而A在3’UTR的下游富集(图 5-A);利用软件在球囊菌全长转录本的poly-A剪切位点上游鉴定到4个motif,分别为UCUCCU、UCUUCU、CCCACC和CCCCCU (图 5-B、C和D)。
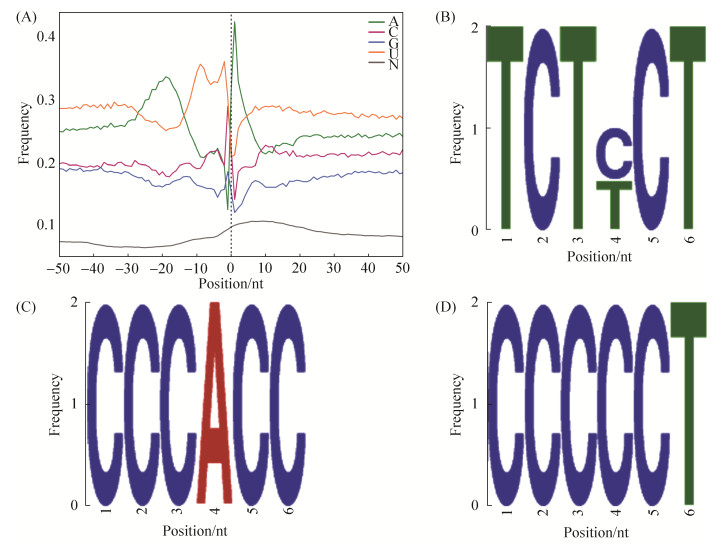
|
| 图 5 球囊菌全长转录本的可变腺苷酸化位点的上下游序列特征 Figure 5 Characteristics of upstream and downstream sequences of A. apis full-length transcripts' APA sites. A: Nucleotide composition around APA sites. B–D: Three motifs identified at 50 nt upstream of APA sites of A. apis full-length transcripts. |
3 讨论
目前,基于三代测序技术的AS和APA研究较少且主要集中在人类和少数模式物种[28-32]。对于球囊菌,AS和APA的相关信息近乎空白。此前,笔者所在课题组基于Nanopore长读段测序数据对蜜蜂的另一种真菌病原东方蜜蜂微孢子虫基因的AS和APA进行全转录组水平的分析和探讨[43],该研究为蜜蜂病原AS和APA研究的首例报道。为开展球囊菌的全长转录组研究,笔者前期利用Nanopore测序技术对Aam和Aas分别进行测序,构建并注释了球囊菌的全长转录组[35]。本研究基于高质量的长读段数据在Aam和Aas中分别鉴定到286和559次AS事件,分别鉴定到2748和2768个基因含有1个及以上的APA位点;球囊菌全长转录本的3’UTR的上下游表现出明显的碱基倾向性,U和A分别富集在3’UTR的上游和下游;此外还在全长转录本的APA位点上游鉴定到4个motif (UCUCCU、UCUUCU、CCCACC和CCCCCU)。本研究鉴定到的isoform和APA位点信息可为球囊菌的基因组[5]和转录组[46-47]注释提供重要补充,也能为其他真菌的相关研究提供有益的参考信息。
本研究中,在孢子中鉴定到的AS事件数量(559次)明显多于菌丝中的AS事件数量(286次),此外孢子中发生APA的基因数量(2768个)稍多于菌丝中发生APA的基因数量(2748个),说明孢子中基因的AS和APA更为活跃。本研究中菌丝和孢子来源于实验室条件下获得的球囊菌纯培养,而非来源于实际的侵染过程;同时考虑到球囊菌在其致病过程历经逃避或抵御宿主免疫反应、摄取宿主营养、干扰宿主生理代谢以及分泌几丁质酶、蛋白酶、脂酶等毒力因子破坏宿主组织结构等复杂过程,菌丝作为球囊菌的生长状态,需要从蜜蜂幼虫宿主体内大量摄取供自身生长发育、合成及分泌各类毒力因子所需的物质和能量,而球囊菌孢子作为病原增殖周期结束的休眠态,其由几丁质和肽聚糖组成的致密孢子壁有利于抵御外界不良环境及宿主天然免疫;推测处于实际侵染过程的球囊菌孢子中基因的AS事件数量及发生APA的基因数量应少于菌丝。笔者课题组前期在东方蜜蜂微孢子虫的基因中仅鉴定到5次AS事件,包括1次A5和4次RI[43],远少于在球囊菌菌丝和孢子中鉴定到的AS事件总数(286次和559次);此外,发生APA的东方蜜蜂微孢子虫基因数量(233个)[43]也远少于球囊菌(2748个和2768个)。微孢子虫作为专性细胞内寄生的单细胞真菌病原,经过与宿主的长期协同进化,其增殖所需物质和能量高度依赖宿主细胞提供[48]。类似于原核生物,微孢子虫的基因组很小,重叠基因和重叠转录本较多,内含子和编码基因总数较少[49]。东方蜜蜂微孢子虫基因组大小仅约为5.69 Mb,编码基因的总数为3209个[50];而球囊菌的基因组大小约为20.31 Mb,包含6442个编码基因[5]。更大的基因组和更多的编码基因导致球囊菌较东方蜜蜂微孢子虫具有更多的AS事件和发生APA的基因。
Pre-mRNA的3’UTR通过多聚腺苷酸化调节RNA结构或RNA与蛋白质的结合,从而控制RNA的翻译效率及稳定性[51]。去腺苷酸化对于mRNA在细胞质中的降解十分重要[52]。随着三代测序技术的快速发展和进步,人类、甘蔗和红车轴草(Trifolium pratense)等动植物基因的APA位点被陆续鉴定出来[28-31]。但对于真菌,基于三代测序技术的全长转录组研究迄今仅有两例报道。Jenjaroenpun等[53]分别利用Nanopore长读段测序技术和PacBio SMRT测序技术对酿酒酵母(Saccharomyces cerevisiae) CEN.PK113-7D菌株进行基因组测序,然后分别完全基于Nanopore测序数据、完全基于PacBio测序数据以及混合Nanopore和PacBio测序数据组装基因组,均能组装出包含16条染色体的完整基因组;此外,作者利用Nanopore MinION平台直接对酿酒酵母进行RNA测序,通过深入分析揭示了酿酒酵母转录组的复杂性,同时还鉴定到大量腺苷酸化的非编码RNA、rRNA、端粒RNA及反义RNA。Bao等[54]基于PacBio SMRT测序技术组装了稻瘟病菌(Magnaporthe oryzae) FJ81278和Guy11菌株的染色体水平基因组,并探讨了转座元件对染色体易位等基因组变异的贡献度。然而上述两项研究均未涉及真菌的APA分析。本研究首次在球囊菌菌丝和孢子中分别鉴定到2748和2768个基因发生APA,说明动物、植物和微生物的基因普遍发生APA。在Aam中,含有1和2个APA位点的基因数量最多,分别占发生APA的基因总数的26.42%和21.29%;但在Aas中,含有5个以上的APA位点的基因数最多,占比高达42.63%。前期研究发现,在东方蜜蜂微孢子虫孢子中,含有1个APA位点的基因数最多(61.37%)[43]。以上结果表明对于不同的蜜蜂真菌病原的孢子,APA的发生特点存在差异;对于同一蜜蜂真菌病原(球囊菌),菌丝和孢子中APA的发生特点也存在差异。本研究发现,球囊菌全长转录本的3’UTR的上下游表现出明显的碱基倾向性,U和A分别在3’UTR的上游和下游富集,与此前其他物种中的相关研究报道一致[22, 28, 44, 54]。此外,在球囊菌全长转录本的上游鉴定到4个motif,分别是UCUCCU、UCUUCU、CCCACC和CCCCCU。AAUAAA是一个在动植物中较为保守的motif,我们前期在东方蜜蜂微孢子虫的全长转录本的APA位点上游也鉴定到了该motif,但本研究在球囊菌中未鉴定到。此前在东方蜜蜂微孢子虫的全长转录本APA位点上游还鉴定到另外2个motif (UGAUGC和GCGACG),与本研究中在球囊菌全长转录本APA位点上游鉴定到的4个motif均不相同。Abdel等[28]曾在甘蔗全长转录本的APA位点上游鉴定到一个motif (UGUA),与本研究鉴定到的4个motif也不相同。以上结果表明球囊菌全长转录本的APA位点上游的motif比较特殊,值得进一步深入研究;不同物种的全长转录本APA位点上游的motif具有种属特异性。
本研究的测序材料是实验室条件下纯培养的球囊菌,而处于侵染过程的球囊菌大量增殖,伴随着基因的活跃表达,发生的AS和APA事件必然与球囊菌纯培养不同。球囊菌孢子随食物被蜜蜂幼虫经口摄入,前中期在幼虫中肠低水平萌发和生长,末期在幼虫后肠暴发式生长,菌丝先后穿透肠壁和体壁并最终包裹整个虫尸[55]。目前尚没有技术手段可以将处于侵染过程的球囊菌菌丝和孢子分离出来。笔者所在课题组前期研究证实,通过将球囊菌侵染的蜜蜂幼虫肠道的二代转录组测序数据连续比对核糖体数据库、宿主基因组和病原基因组,从而获得纯净的球囊菌转录组数据是一种可行的方法[56-57]。下一步将利用Nanopore测序技术对球囊菌侵染的意蜂幼虫和中蜂幼虫肠道进行测序,通过上述方法获得纯净的球囊菌长读段测序数据,一方面分析和探讨球囊菌在侵染过程的AS事件和APA位点的数量及发生特点,另一方面与纯化的球囊菌孢子中基因的AS和APA进行比较分析,进而探究AS和APA在病原侵染不同抗性蜂种过程中的作用。
综上所述,本研究首次对球囊菌基因的AS事件数量和类型、发生APA的基因数量、APA位点上下游序列特征及motif进行全面解析,研究结果不仅揭示了球囊菌转录组的复杂性,为完善现有的参考基因组和转录组注释提供了宝贵信息,也为探究AS和APA在球囊菌菌丝和孢子的基因表达调控中的作用提供了基础。
| [1] | Chen DF, Guo R, Xiong CL, Zheng YZ, Hou CS, Fu ZM. Morphological and molecular identification of chalkbrood disease pathogen Ascosphaera apis in Apis cerana cerana. Journal of Apicultural Research, 2018, 57(4): 516-521. DOI:10.1080/00218839.2018.1475943 |
| [2] |
Guo R, Du Y, Tong XY, Xiong CL, Zheng YZ, Xu GJ, Wang HP, Geng SH, Zhou DD, Guo YL, Wu SZ, Chen DF. Differentially expressed microRNAs and their regulation networks in Apis mellifera ligustica larval gut during the early stage of Ascosphaera apis infection. Scientia Agricultura Sinica, 2019, 52(1): 166-180.
(in Chinese) 郭睿, 杜宇, 童新宇, 熊翠玲, 郑燕珍, 徐国钧, 王海朋, 耿四海, 周丁丁, 郭意龙, 吴素珍, 陈大福. 意大利蜜蜂幼虫肠道在球囊菌侵染前期的差异表达microRNA及其调控网络. 中国农业科学, 2019, 52(1): 166-180. |
| [3] |
Xiong CL, DU Y, Feng RR, Jiang HB, Shi XY, Wang HP, Fan XX, Wang J, Zhu ZW, Fan YC, Chen HZ, Zhou DD, Zheng YZ, Chen DF, Guo R. Differential expression pattern and regulation network of microRNAs in Ascosphaera apis invading Apis cerana cerana 6-day-old larvae. Acta Microbiologica Sinica, 2020, 60(5): 992-1009.
(in Chinese) 熊翠玲, 杜宇, 冯睿蓉, 蒋海宾, 史小玉, 王海朋, 范小雪, 王杰, 祝智威, 范元婵, 陈华枝, 周丁丁, 郑燕珍, 陈大福, 郭睿. 侵染中华蜜蜂6日龄幼虫的蜜蜂球囊菌的微小RNA差异表达谱及调控网络. 微生物学报, 2020, 60(5): 992-1009. |
| [4] | Qin X, Evans JD, Aronstein KA, Murray KD, Weinstock GM. Genome sequences of the honey bee pathogens Paenibacillus larvae and Ascosphaera apis. Insect Molecular Biology, 2006, 15(5): 715-718. DOI:10.1111/j.1365-2583.2006.00694.x |
| [5] | Shang YF, Xiao GH, Zheng P, Cen K, Zhan S, Wang CS. Divergent and convergent evolution of fungal pathogenicity. Genome Biology and Evolution, 2016, 8(5): 1374-1387. DOI:10.1093/gbe/evw082 |
| [6] | Modrek B, Lee C. A genomic view of alternative splicing. Nature Genetics, 2002, 30(1): 13-19. DOI:10.1038/ng0102-13 |
| [7] | Filichkin SA, Hamilton M, Dharmawardhana PD, Singh SK, Sullivan C, Ben-Hur A, Reddy ASN, Jaiswal P. Abiotic stresses modulate landscape of poplar transcriptome via alternative splicing, differential intron retention, and isoform ratio switching. Frontiers in Plant Science, 2018, 9: 5. DOI:10.3389/fpls.2018.00005 |
| [8] | Paronetto MP, Passacantilli I, Sette C. Alternative splicing and cell survival: from tissue homeostasis to disease. Cell Death & Differentiation, 2016, 23(12): 1919-1929. |
| [9] | Wang ET, Sandberg R, Luo SJ, Khrebtukova I, Zhang L, Mayr C, Kingsmore SF, Schroth GP, Burge CB. Alternative isoform regulation in human tissue transcriptomes. Nature, 2008, 456(7221): 470-476. DOI:10.1038/nature07509 |
| [10] | Pan Q, Shai O, Lee LJ, Frey BJ, Blencowe BJ. Deep surveying of alternative splicing complexity in the human transcriptome by high-throughput sequencing. Nature Genetics, 2008, 40(12): 1413-1415. DOI:10.1038/ng.259 |
| [11] | Nygard AB, Cirera S, Gilchrist MJ, Gorodkin J, Jørgensen CB, Fredholm M. A study of alternative splicing in the pig. BMC Research Notes, 2010, 3: 123. DOI:10.1186/1756-0500-3-123 |
| [12] | Mei WB, Boatwright L, Feng GQ, Schnable JC, Barbazuk WB. Evolutionarily conserved alternative splicing across monocots. Genetics, 2017, 207(2): 465-480. DOI:10.1534/genetics.117.300189 |
| [13] | Remy E, Cabrito TR, Baster P, Batista RA, Teixeira MC, Friml J, Sá-Correia I, Duque P. A major facilitator superfamily transporter plays a dual role in polar auxin transport and drought stress tolerance in Arabidopsis. The Plant Cell, 2013, 25(3): 901-926. DOI:10.1105/tpc.113.110353 |
| [14] | Shen YT, Zhou ZK, Wang Z, Li WY, Fang C, Wu M, Ma YM, Liu TF, Kong LA, Peng DL, Tian ZX. Global dissection of alternative splicing in paleopolyploid soybean. The Plant Cell, 2014, 26(3): 996-1008. DOI:10.1105/tpc.114.122739 |
| [15] | Thatcher SR, Zhou WG, Leonard A, Wang BB, Beatty M, Zastrow-Hayes G, Zhao XY, Baumgarten A, Li BL. Genome-wide analysis of alternative splicing in Zea mays: landscape and genetic regulation. The Plant Cell, 2014, 26(9): 3472-3487. DOI:10.1105/tpc.114.130773 |
| [16] |
Guo R, Li L, Xiong CL, Zheng YZ, Fu ZM, Wang HP, Zhao HX, Chen DF. Analysis of the alternatively spliced genes in Apis cerana cerana larval gut under the Ascosphaera apis stress. Journal of Sichuan University (Natural Science Edition), 2018, 55(6): 1313-1318.
(in Chinese) 郭睿, 李龙, 熊翠玲, 郑燕珍, 付中民, 王海朋, 赵红霞, 陈大福. 中华蜜蜂幼虫肠道响应球囊菌胁迫的可变剪切基因分析. 四川大学学报(自然科学版), 2018, 55(6): 1313-1318. |
| [17] |
Guo R, Chen H, Zhang L, Zhao HY, Xiong CL, Zheng YZ, Fu ZM, Liang Q, Chen DF. Analysis of Apis mellifera ligustica larval gut's alternative spliced genes responding to Ascosphaera apis stress. Journal of Shanghai Jiaotong University (Agricultural Science), 2018, 36(3): 1-6.
(in Chinese) 郭睿, 陈恒, 张璐, 赵浩宇, 熊翠玲, 郑燕珍, 付中民, 梁勤, 陈大福. 意大利蜜蜂幼虫肠道响应球囊菌胁迫的可变剪接基因分析. 上海交通大学学报(农业科学版), 2018, 36(3): 1-6. |
| [18] | Steijger T, Abril JF, Engström PG, Kokocinski F, The Rgasp Consortium, Hubbard TJ, Guigó R, Harrow J, Bertone P. Assessment of transcript reconstruction methods for RNA-seq. Nature Methods, 2013, 10(12): 1177-1184. DOI:10.1038/nmeth.2714 |
| [19] | Jackson RJ, Standart N. Do the poly(A) tail and 3' untranslated region control mRNA translation?. Cell, 1990, 62(1): 15-24. DOI:10.1016/0092-8674(90)90235-7 |
| [20] | Naftelberg S, Schor IE, Ast G, Kornblihtt AR. Regulation of alternative splicing through coupling with transcription and chromatin structure. Annual Review of Biochemistry, 2015, 84: 165-198. DOI:10.1146/annurev-biochem-060614-034242 |
| [21] | Shen YJ, Venu RC, Nobuta K, Wu XH, Notibala V, Demirci C, Meyers BC, Wang GL, Ji GL, Li QSQ. Transcriptome dynamics through alternative polyadenylation in developmental and environmental responses in plants revealed by deep sequencing. Genome Research, 2011, 21(9): 1478-1486. DOI:10.1101/gr.114744.110 |
| [22] | Sherstnev A, Duc C, Cole C, Zacharaki V, Hornyik C, Ozsolak F, Milos PM, Barton GJ, Simpson GG. Direct sequencing of Arabidopsis thaliana RNA reveals patterns of cleavage and polyadenylation. Nature Structural & Molecular Biology, 2012, 19(8): 845-852. |
| [23] | Hargreaves AD, Mulley JF. Assessing the utility of the Oxford Nanopore MinION for snake venom gland cDNA sequencing. PeerJ, 2015, 3: e1441. DOI:10.7717/peerj.1441 |
| [24] | Kilianski A, Haas JL, Corriveau EJ, Lien AT, Willis KL, Kadavy DR, Rosenzweig CN, Minot SS. Bacterial and viral identification and differentiation by amplicon sequencing on the MinION nanopore sequencer. Giga Science, 2015, 4(1): 12. DOI:10.1186/s13742-015-0051-z |
| [25] | Bolisetty MT, Rajadinakaran G, Graveley BR. Determining exon connectivity in complex mRNAs by nanopore sequencing. Genome Biology, 2015, 16(1): 204. DOI:10.1186/s13059-015-0777-z |
| [26] | Byrne A, Beaudin AE, Olsen HE, Jain M, Cole C, Palmer T, DuBois RM, Forsberg EC, Akeson M, Vollmers C. Nanopore long-read RNAseq reveals widespread transcriptional variation among the surface receptors of individual B cells. Nature Communications, 2017, 8: 16027. DOI:10.1038/ncomms16027 |
| [27] | Wang B, Tseng E, Regulski M, Clark TA, Hon T, Jiao YP, Lu ZY, Olson A, Stein JC, Ware D. Unveiling the complexity of the maize transcriptome by single-molecule long-read sequencing. Nature Communications, 2016, 7: 11708. DOI:10.1038/ncomms11708 |
| [28] | Abdel-Ghany SE, Hamilton M, Jacobi JL, Ngam P, Devitt N, Schilkey F, Ben-Hur A, Reddy ASN. A survey of the sorghum transcriptome using single-molecule long reads. Nature Communications, 2016, 7: 11706. DOI:10.1038/ncomms11706 |
| [29] | Chao YH, Yuan JB, Li SF, Jia SQ, Han LB, Xu LX. Analysis of transcripts and splice isoforms in red clover (Trifolium pratense L.) by single-molecule long-read sequencing. BMC Plant Biology, 2018, 18(1): 300. DOI:10.1186/s12870-018-1534-8 |
| [30] | Li Y, Fang CC, Fu YH, Hu A, Li CC, Zou C, Li XY, Zhao SH, Zhang CJ, Li CC. A survey of transcriptome complexity in Sus Scrofa using single-molecule long-read sequencing. DNA Research, 2018, 25(4): 421-437. DOI:10.1093/dnares/dsy014 |
| [31] | Workman RE, Tang AD, Tang PS, Jain M, Tyson JR, Razaghi R, Zuzarte PC, Gilpatrick T, Payne A, Quick J, Sadowski N, Holmes N, De Jesus JG, Jones KL, Soulette CM, Snutch TP, Loman N, Paten B, Loose M, Simpson JT, Olsen HE, Brooks AN, Akeson M, Timp W. Nanopore native RNA sequencing of a human poly (A) transcriptome. Nature Methods, 2019, 16(12): 1297-1305. DOI:10.1038/s41592-019-0617-2 |
| [32] | Magi A, Semeraro R, Mingrino A, Giusti B, D'aurizio R. Nanopore sequencing data analysis: state of the art, applications and challenges. Briefings in Bioinformatics, 2018, 19(6): 1256-1272. |
| [33] | Jain M, Olsen HE, Paten B, Akeson M. The Oxford Nanopore MinION: delivery of nanopore sequencing to the genomics community. Genome Biology, 2016, 17: 239. DOI:10.1186/s13059-016-1103-0 |
| [34] | Du Y, Chen HZ, Wang J, Zhu ZW, Xiong CL, Zheng YZ, Chen DF, Guo R. Nanopore long-read transcriptome data of fungal pathogen of chalkbrood disease, Ascosphaera apis. bioRxiv, 2020. |
| [35] |
Du Y, Zhu ZW, Wang J, Wang XN, Jiang HB, Fan YC, Fan XX, Chen HZ, Long Q, Cai ZB, Xiong CL, Zheng YZ, Fu ZM, Chen DF, Guo R. Construction and annotation of Ascosphaera apis full-length trancriptome utilizing Nanopore third-genration long-read sequencing technology. Scientia Agricultura Sinica, 2021, 54(4): 864-876.
(in Chinese) 杜宇, 祝智威, 王杰, 王秀娜, 蒋海宾, 范元婵, 范小雪, 陈华枝, 隆琦, 蔡宗兵, 熊翠玲, 郑燕珍, 付中民, 陈大福, 郭睿. 利用第三代纳米孔长读段测序技术构建和注释蜜蜂球囊菌的全长转录组. 中国农业科学, 2021, 54(4): 864-876. |
| [36] | 梁勤, 陈大福. 蜜蜂保护学. 北京: 中国农业出版社, 2009. |
| [37] |
Du Y, Tong XY, Zhou DD, Chen DF, Xiong CL, Zheng YZ, Xu GJ, Wang HP, Chen HZ, Guo YL, Long Q, Guo R. MicroRNA responses in the larval gut of Apis cerana cerana to Ascosphaera apis stress. Acta Microbiologica Sinica, 2019, 59(9): 1747-1764.
(in Chinese) 杜宇, 童新宇, 周丁丁, 陈大福, 熊翠玲, 郑燕珍, 徐国钧, 王海朋, 陈华枝, 郭意龙, 隆琦, 郭睿. 中华蜜蜂幼虫肠道响应球囊菌胁迫的MicroRNA应答分析. 微生物学报, 2019, 59(9): 1747-1764. |
| [38] |
Guo R, Du Y, Zhou NH, Liu SY, Xiong CL, Zheng YZ, Fu ZM, Xu GJ, Wang HP, Geng SH, Zhou DD, Chen DF. Comprehensive analysis of differentially expressed microRNAs and their target genes in the larval gut of Apis mellifera ligustica during the late stage of Ascosphaera apis stress. Acta Entomologica Sinica, 2019, 62(1): 49-60.
(in Chinese) 郭睿, 杜宇, 周倪红, 刘思亚, 熊翠玲, 郑燕珍, 付中民, 徐国钧, 王海朋, 耿四海, 周丁丁, 陈大福. 意大利蜜蜂幼虫肠道在球囊菌胁迫后期的差异表达微小RNA及其靶基因分析. 昆虫学报, 2019, 62(1): 49-60. |
| [39] | Jensen AB, Aronstein K, Flores JM, Vojvodic S, Palacio MA, Spivak M. Standard methods for fungal brood disease research. Journal of Apicultural Research, 2013, 52(1): 10. |
| [40] |
Jiang HB, Du Y, Fan XX, Wang J, Zhu ZW, Fan YC, Xiong CL, Fu ZM, Xu GJ, Chen DF, Guo R. Comparative transcriptome investigation of Ascosphaera apis spore and mycelium. Journal of Sichuan University (Natural Science Edition), 2020, 57(6): 1177-1185.
(in Chinese) 蒋海宾, 杜宇, 范小雪, 王杰, 祝智威, 范元婵, 熊翠玲, 付中民, 徐国钧, 陈大福, 郭睿. 蜜蜂球囊菌菌丝和孢子的比较转录组分析. 四川大学学报(自然科学版), 2020, 57(6): 1177-1185. |
| [41] | Ashburner M, Ball CA, Blake JA, Botstein D, Butler H, Cherry JM, Davis AP, Dolinski K, Dwight SS, Eppig JT, Harris MA, Hill DP, Issel-Tarver L, Kasarskis A, Lewis S, Matese JC, Richardson JE, Ringwald M, Rubin GM, Sherlock G. Gene ontology: tool for the unification of biology. Nature Genetics, 2000, 25(1): 25-29. DOI:10.1038/75556 |
| [42] | Kanehisa M, Goto S, Kawashima S, Okuno Y, Hattori M. The KEGG resource for deciphering the genome. Nucleic Acids Research, 2004, 32(S1): D277-D280. |
| [43] |
Chen HZ, Fan XX, Fan YC, Wang J, Zhu ZW, Jiang HB, Zhang WD, Long Q, Xiong CL, Zheng YZ, Fu ZM, Chen DF, Guo R. Analysis of alternative splicing and alternative polyadenylation of Nosema ceranae genes. Mycosystema, 2020, 39(12): 1-12. (in Chinese) 陈华枝, 范小雪, 范元婵, 王杰, 祝智威, 蒋海宾, 张文德, 隆琦, 熊翠玲, 郑燕珍, 付中民, 徐国钧, 陈大福, 郭睿. 东方蜜蜂微孢子虫基因的可变剪接及可变腺苷酸化解析. 菌物学报, 2020, 39(12): 1-12. |
| [44] | Foissac S, Sammeth M. Astalavista: dynamic and flexible analysis of alternative splicing events in custom gene datasets. Nucleic Acids Research, 2007, 35(S2): W297-W299. |
| [45] | Bailey TL, Williams N, Misleh C, Li WW. MEME: discovering and analyzing DNA and protein sequence motifs. Nucleic Acids Research, 2006, 34(S2): W369-W373. |
| [46] |
Zhang ZN, Xiong CL, Xu XJ, Huang ZJ, Zheng YZ, Luo Q, Liu M, Li WD, Tong XY, Zhang Q, Liang Q, Guo R, Chen DF. De novo assembly of a reference transcriptome and development of SSR markers for Ascosphaera apis. Acta Entomologica Sinica, 2017, 60(1): 34-44.
(in Chinese) 张曌楠, 熊翠玲, 徐细建, 黄枳腱, 郑燕珍, 骆群, 刘敏, 李汶东, 童新宇, 张琦, 梁勤, 郭睿, 陈大福. 蜜蜂球囊菌的参考转录组de novo组装及SSR分子标记开发. 昆虫学报, 2017, 60(1): 34-44. |
| [47] | Chen DF, Du Y, Fan XX, Zhu ZW, Jiang HB, Wang J, Fan YC, Chen HZ, Zhou DD, Xiong CL, Zheng YZ, Xu XJ, Luo Q, Guo R. Reconstruction and functional annotation of Ascosphaera apis full-length transcriptome utilizing PacBio long reads combined with Illumina short reads. Journal of Invertebrate Pathology, 2020(176): 107475. |
| [48] | Burri L, Williams BAP, Bursac D, Lithgow T, Keeling PJ. Microsporidian mitosomes retain elements of the general mitochondrial targeting system. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(43): 15916-15920. DOI:10.1073/pnas.0604109103 |
| [49] | Cornman RS, Chen YP, Schatz MC, Street C, Zhao Y, Desany B, Egholm M, Hutchison S, Pettis JS, Ian Lipkin W, Evans JD. Genomic analyses of the microsporidian Nosema ceranae, an emergent pathogen of honey bees. PLoS Pathogens, 2009, 5(6): e1000466. DOI:10.1371/journal.ppat.1000466 |
| [50] | Pelin A, Selman M, Aris-Brosou S, Farinelli L, Corradi N. Genome analyses suggest the presence of polyploidy and recent human-driven expansions in eight global populations of the honeybee pathogen Nosema ceranae. Environmental Microbiology, 2015, 17(11): 4443-4448. DOI:10.1111/1462-2920.12883 |
| [51] | Yi H, Park J, Ha MJ, Lim J, Chang H, Kim VN. PABP cooperates with the CCR4-NOT complex to promote mRNA deadenylation and block precocious decay. Molecular Cell, 2018, 70(6): 1081-1088. DOI:10.1016/j.molcel.2018.05.009 |
| [52] | Wu XH, Liu M, Downie B, Liang C, Ji GL, Li QSQ, Hunt AG. Genome-wide landscape of polyadenylation in Arabidopsis provides evidence for extensive alternative polyadenylation. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(30): 12533-12538. DOI:10.1073/pnas.1019732108 |
| [53] | Jenjaroenpun P, Wongsurawat T, Pereira R, Patumcharoenpol P, Ussery DW, Nielsen J, Nookaew I. Complete genomic and transcriptional landscape analysis using third-generation sequencing: a case study of Saccharomyces cerevisiae CEN. PK113-7D. Nucleic Acids Research, 2018, 46(7): e38. DOI:10.1093/nar/gky014 |
| [54] | Bao JD, Chen ML, Zhong ZH, Tang W, Lin LY, Zhang XT, Jiang HL, Zhang DY, Miao CY, Tang HB, Zhang JS, Lu GD, Ming R, Norvienyeku J, Wang BH, Wang ZH. PacBio sequencing reveals transposable elements as a key contributor to genomic plasticity and virulence variation in Magnaporthe oryzae. Molecular Plant, 2017, 10(11): 1465-1468. DOI:10.1016/j.molp.2017.08.008 |
| [55] |
Li JH, Zheng ZY, Chen DF, Liang Q. Factors influencing Ascosphaera apis infection on honeybee larvae and observation on the infection process. Acta Entomologica Sinica, 2012, 55(7): 790-797.
(in Chinese) 李江红, 郑志阳, 陈大福, 梁勤. 影响蜜蜂球囊菌侵染蜜蜂幼虫的因素及侵染过程观察. 昆虫学报, 2012, 55(7): 790-797. |
| [56] |
Chen DF, Guo R, Xiong CL, Liang Q, Zheng YZ, Xu XJ, Huang ZJ, Zhang ZN, Zhang L, Li WD, Tong XY, Xi WJ. Transcriptomic analysis of Ascosphaera apis stressing larval gut of Apis mellifera ligustica. Acta Entomologica Sinica, 2017, 60(4): 401-411.
(in Chinese) 陈大福, 郭睿, 熊翠玲, 梁勤, 郑燕珍, 徐细建, 黄枳腱, 张曌楠, 张璐, 李汶东, 童新宇, 席伟军. 胁迫意大利蜜蜂幼虫肠道的球囊菌的转录组分析. 昆虫学报, 2017, 60(4): 401-411. |
| [57] |
Guo R, Chen DF, Huang ZJ, Liang Q, Xiong CL, Xu XJ, Zheng YZ, Zhang ZN, Xie YL, Tong XY, Hou ZX, Jiang LL, Dao C. Transcriptome analysis of Ascosphaera apis stressing larval gut of Apis cerana cerana. Acta Microbiologica Sinica, 2017, 57(12): 1865-1878.
(in Chinese) 郭睿, 陈大福, 黄枳腱, 梁勤, 熊翠玲, 徐细建, 郑燕珍, 张曌楠, 解彦玲, 童新宇, 侯志贤, 江亮亮, 刀晨. 球囊菌胁迫中华蜜蜂幼虫肠道过程中病原的转录组学研究. 微生物学报, 2017, 57(12): 1865-1878. |
 2021, Vol. 61
2021, Vol. 61