中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 任思羽, 程新宽, 张宇辉, 庄建文, 马龙. 2021
- Siyu Ren, Xinkuan Cheng, Yuhui Zhang, Jianwen Zhuang, Long Ma. 2021
- 氟化天然产物生物合成的研究进展
- Advances in biosynthesis of fluorinated products
- 微生物学报, 61(3): 524-538
- Acta Microbiologica Sinica, 61(3): 524-538
-
文章历史
- 收稿日期:2020-05-04
- 修回日期:2020-05-28
- 网络出版日期:2020-07-31
氟元素(F,元素周期表第9),在地球上所有元素中的天然存在比值排名第24位,在地壳中的所有卤素元素(F、Cl、Br、I、At、Ts)中天然存在比值排名最高,然而以可溶性氟离子(F-)的存在形式却较为稀有。研究表明,在海洋中常见的卤素离子氯离子(Cl-)含量为19000 mg/L,溴离子(Br-)的含量为70 mg/L,但丰度最高的氟离子含量仅为1.3 mg/L[1]。另外,氟元素在水溶液中极易被溶剂化从而导致其生物活性大大降低,与其他卤素相比,很难经卤素过氧化物酶氧化进而生成高附加值的有机物。这两个主要原因造成了氟元素的生物利用率受到极大限制,在氟生物化学的进化速度上也极其缓慢[2]。尽管天然的氟化有机物极其稀有,并且已开发的资源十分有限。但氟化物的价值却十分巨大,将氟原子引入到有机分子中受到越来越多的关注,因为有机氟化物可以被广泛应用于生物有机化学、药物化学和生物材料等领域,尤其在医药领域,氟元素的引入能显著地改善化合物的理化性质并增强药物的活性。据统计,世界上最畅销的药物中,约30%的药品中含有氟元素,例如氟化可的松、阿托伐他汀钙、兰索拉唑、诺氟沙星和依地普仑等[3-5]。虽然在有机分子中引入氟元素已经引起了科研工作者的关注,但通过合成化学的方法在分子中引入氟原子依然相当困难,存在反应条件剧烈、能耗高和产率低等不足[6-7]。与化学法相比,生物法由于反应温和性、特异选择性和更环保的特性,在氟化天然产物的合成研究中已发挥着越来越重要的作用。
目前科学家们的研究主要集中于通过计算生物学、分子生物学、合成生物学以及生物化学等手段,在温和反应条件下,将C-F键选择性地引入到高附加值化合物中,从而形成具有不同结构和活性的生物分子,并将其应用到各个领域。尽管如此,利用生物法选择性地合成氟化产物依然极具挑战性[6]。因此,本文从氟化天然产物合成的不同方面进行综述,将有助于氟化生物化学的发展,推动氟化天然产物选择性合成的研究。
1 氟化天然产物及氟化酶的发现天然产物是生物活性化合物的丰富来源,它们在新药和农作物保护产品的探索和开发中发挥了相当大的作用。据统计到目前为止,被发现并鉴定出的天然产物已超过130000种[8],其中包括超过5000种的卤素取代有机物[9],但仅有6种天然氟化有机物被发现[10-12](图 1)。1943年,Marais等人在Dichapetalum cymosum (毒鼠子科植物,D. cymosum)中发现了氟化乙酸,这是自然界中首次发现氟化天然产物[13]。之后的几十年时间里,科研人员又陆续发现了氟化核苷、ω-氟油酸、(2R, 3R)-氟化柠檬酸和氟化苏氨酸4种新型氟化天然产物。值得注意的是,本课题组在Streptomyces sp. MA37(MA37链霉菌)中也发现了一种新型氟化天然产物-(2R3S4S)-5-氟-2, 3, 4-三羟基戊酸(5-FHPA)[14]。

|
| 图 1 已知的氟化天然产物 Figure 1 The fluorinated natural products that have been identified. |
目前为止,有机氟化物的合成主要有化学法和生物法。在化学法中,氟元素的引入主要在亲核、亲电、自由基、氟甲基化试剂和过渡金属催化的氟代烷基化反应中进行[15-16]。而生物法则是以酶作为反应催化剂,基于酶法的生物催化反应不仅满足了高度选择性、安全性和可持续性的工业需求,而且与化学法相比,生物法在原子经济性和环保方面均具有独特的优势[17-18]。生物法中以氟化酶最具代表性,但是自然界中的氟化酶极为罕见,2002年第一个天然存在的氟化酶在Streptomyces cattleya (卡特利链霉菌,S. cattleya)中被发现,它能够催化无机F-和S-腺苷-L-甲硫氨酸(SAM)发生SN2生物亲核反应,生成5'-氟代脱氧腺苷(5'-FDA)和L-甲硫氨酸(图 2)[19-20]。
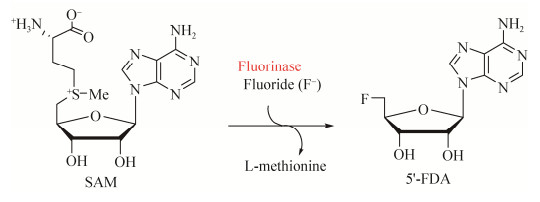
|
| 图 2 S. cattleya来源氟化酶介导的SAM到5'-FDA反应 Figure 2 The fluorinase enzyme from S. cattleya mediates the conversion of S-adenosyl-L-methionine (SAM) to 5'-fluorodeoxy adenosine (5'-FDA). |
之后十几年的时间里,S. cattleya来源的氟化酶一直是唯一的已知氟化酶。因此,在2014年到2016年间,我们通过基因挖掘手段,陆续从Streptomyces xinghaiensis (星海链霉菌,S. xinghaiensis)、Streptomyces sp. MA37、Norcardia brasiliensis (巴西诺卡菌,N. brasiliensis)和Actinoplanes sp. N902-109 (N902-109放线菌)等微生物中,鉴定出了4种不同的新型氟化酶,丰富了氟生物化学的酶库[14, 21-23]。尽管科研人员的探索步伐从未停止,但直到最近,来源于Actinopolyspora mzabensis (嗜盐放线菌,A. mzabensis)的第6个氟化酶才被分离鉴定出来,也首次实现了氟化酶由包涵体重新折叠成可溶性蛋白的转换[24],这也是目前为止,自然界中仅有的6个氟化酶,相信随着分析技术以及氟生物技术的发展,将来会有越来越多的氟化酶被发现并用于氟化天然产物的合成。
2 氟化天然产物的合成通路最早对氟化天然产物合成通路进行研究的是英国圣安德鲁斯大学的David O’Hagan教授,并于2002年在Nature杂志发表文章,报道了在S. cattleya体内存在一条氟化代谢通路,这条通路可以利用无机氟离子合成有机氟化天然产物[20]。经过近十年的努力,David O’Hagan教授及其团队成功地解析出S. cattleya中一段长约10 kb的基因簇(图 3),这段基因簇与S. cattleya中氟化天然产物合成通路(图 4)密切相关,并负责氟化天然产物的合成与分泌[25-27]。简而言之,无机氟离子在S. cattleya体内经由5步“链锁式”的酶催化反应,最终生成剧毒物质氟化乙酸(fluoroacetate)和抗生素氟化苏氨酸(4-fluorothreonine)[28]。链霉菌在固体平板培养基上生长时,有发育良好的分枝菌丝,常产生各种水溶性或脂溶性的色素,S. cattleya产生的脂溶性色素会使菌丝颜色呈现浅粉紫色带有杂白色(并非由氟化物引起)。在液体培养基中加入氟离子,经过一段时间(~10 d)的培养,这两种最终代谢产物(氟化乙酸和氟化苏氨酸)可以通过氟化NMR (nuclear magnetic resonance,19F-NMR)被检测到(图 5)。而氟化乙酸也可以被转化为氟化乙酰辅酶A (fluoroacetyl-CoA),其作为柠檬酸合成酶(citrate synthase)的底物,生成的氟化柠檬酸可通过抑制顺乌头酸酶(aconitase)活性而阻断TCA循环,因而具有强烈毒性。英国剑桥大学Jonathan Spencer博士研究组发现flk基因(图 3,绿色标记) 编码硫酯酶(thioesterase),此酶可催化氟化乙酰辅酶A的水解,从而解除氟化乙酸的毒性而获得自身免疫[26]。
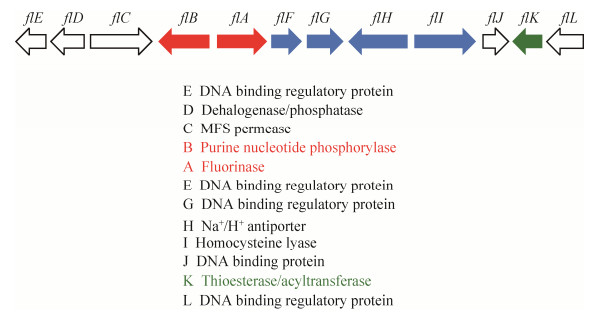
|
| 图 3 S. cattleya中氟化产物的生物合成的基因簇 Figure 3 Gene cluster for biosynthesis of fluorinated natural products in S. cattleya. |
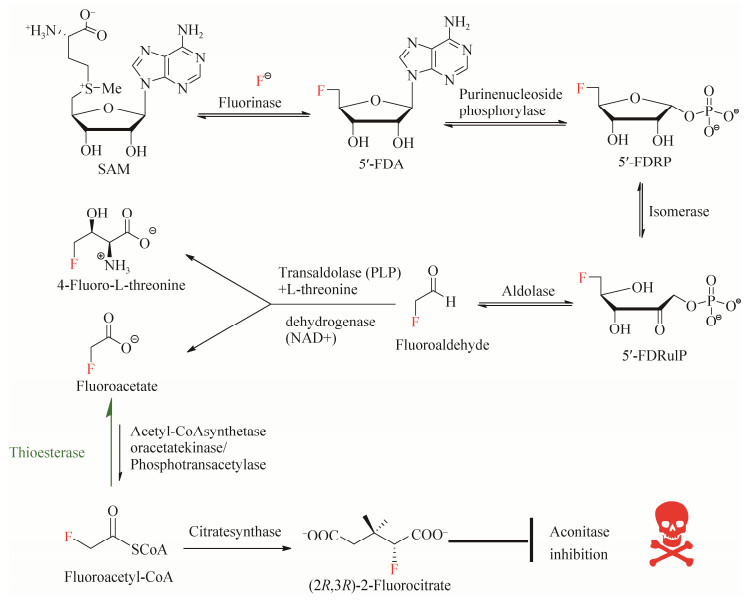
|
| 图 4 S. cattleya中氟化天然产物的生物合成通路 Figure 4 Biosynthetic pathway of fluorinated natural products in S. cattleya. |
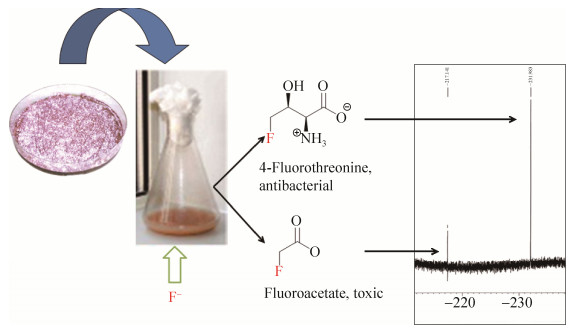
|
| 图 5 S. cattleya的培养和氟化代谢产物的鉴定 Figure 5 S. cattleya was cultivated and its fluorinated metabolite was identified. |
在这条氟化天然产物合成通路中,一些新型的酶也陆续被鉴定,这些酶能够催化氟化底物,被统称为有机氟化物酶系,包括催化5'-FDRP (5-fluoro-5-deoxy-D-ribose-1-phosphate)形成5'-FDRulP (5-fluoro-5-deoxy-D-ribulose-1-phosphate)的异构酶、催化氟化乙醛(fluoroaldehyde)形成氟化苏氨酸的转醛缩酶(transaldolase)以及催化氟化乙醛形成氟化乙酸的醛脱氢酶(aldehyde dehydrogenase)。其中,氟化酶(fluorinase)是这条氟化天然产物合成通路中催化第一步反应的酶,催化SN2亲核取代反应,可以将无机氟离子引入到小分子物质中,从而生成含有C-F的有机氟化物,实现了无机氟离子向有机氟的生物转换,即以无机氟离子和S-腺苷基甲硫氨酸(S-adenosyl-L-methionine,SAM)两种物质为反应底物,以无机氟离子作为亲核试剂进攻SAM核糖上的5'-C,同时脱去SAM上的甲硫氨酸基团,生成产物5'-氟脱氧腺苷(5'-fluorodeoxyadenosine,5'-FDA)[27]。同样令人兴奋的是,David O’Hagan教授在2004年发现了世界上第一个基于原子水平的氟化酶晶体结构并发表至Nature杂志。
在2014年之前,只有S. cattleya中的氟化酶和氟化代谢通路被报道,本课题组一直致力于微生物氟生物化学的系统研究,之后又在非洲加纳的土壤里新分离出来的一株链霉菌Streptomyces sp. MA37,全基因组测序和生物信息学的比对表明是新的菌株。经过发酵培养,发现这种细菌能够产生多种未知的氟化天然产物,新的氟化产物不仅意味着新的化学结构,也意味着新的可能的氟化合成代谢途径。经过鉴定是一种结构全新的氟化天然产物,这是近30年来发现的首个氟化天然产物;并发现了一条新的微生物氟化物合成通路[14]。
此外,Streptomyces calvus (秃裸链霉菌,S. calvus)是另一个产生天然氟化代谢物的菌株,氟化核苷是由S. calvus产生的一种具有抗菌活性的氟化天然产物。但与S. cattleya氟化代谢途径不同,尽管S. calvus全基因组信息已经被解析,但尚未发现任何氟化酶的基因,因此推断S. calvus可能以一种不同于以往的方式催化形成C-F键,也启示我们S. calvus体内可能存在新型的氟化酶和新型氟化物代谢通路[29-30]。通过对S. calvus代谢途径的分析,除了检测到氟化乙酸、氟化苏氨酸和氟戊酸三种氟化产物外,还周期性地检测到两种新型的4'-氟-3'-氧葡萄糖基代谢物(F-Mets Ⅰ和Ⅱ),这两种物质在氟化核苷合成前大量积累,随着氟化核苷的生成后逐渐消失,表明F-Met Ⅰ和F-Met Ⅱ是天然产物氟化核苷的中间体(图 6)。在S. calvus的基因组中分析到UDP-葡萄糖依赖葡萄糖基转移酶基因(nucGT)和糖苷酶基因(nucGS),进一步验证了nucGS和nucGT在氟化核苷生物合成中的作用。将nucGT和nucGS这两个基因敲除后,氟化产物随即消失,这证明了2个酶均参与了氟化核苷的生物合成[31]。此外,通过同位素标记的方法,将放射性标记的[2H1]-(1R, 2R)和[2H1]-(1S, 2R)甘油掺入到S. calvus氟化核苷的合成中,表明S. calvus体内氟代核苷的生物合成是通过磷酸戊糖途径[32]。类似的氟代核苷合成路径也在Streptomyces asterosporus (星状链霉菌,S. asterosporus)体内被证实[33]。
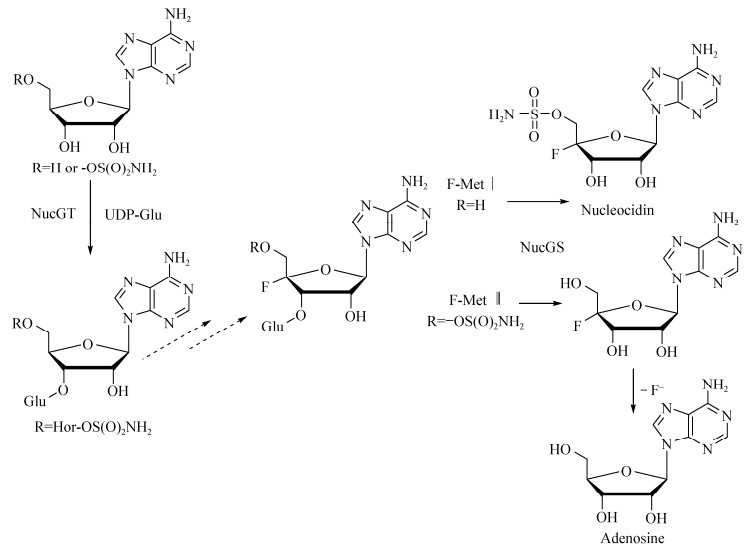
|
| 图 6 S. calvus中氟代核苷的生物合成 Figure 6 Biosynthesis of fluorinated nucleoside in S. calvus. |
3 氟化天然产物合成机制的意义
在合成生物学中,科学家们经常使用改造后的代谢通路用作建筑模块,以此来构建细胞工厂[34],这一技术吸引了越来越多的学者们致力于改造氟化天然产物的合成通路。2013年底,美国Michelle C. Y. Chang教授及其团队在Science杂志发表文章,报道了在已知的氟化天然产物合成通路的基础上,可以将氟化乙酸生成氟乙酰辅酶A这一步反应作为一个建筑模块实现与其他合成通路进行“嫁接”,选择性地将氟原子引入到天然产物中,例如聚酮类、类异戊二烯类、甾族化合物和生物碱等[35],并且这种“嫁接”方式不会改变物质原有的碳骨架。此外,对氟化天然产物合成通路颇有造诣的美国教授Bradely Moore及其团队发现,在Salinispora tropica (海洋放线菌,S. tropica)中存在一种氯化酶,并且S.tropica的氯化天然产物代谢通路中的氯化酶基因与氟化代谢通路中的氟化酶基因高度兼容,借此Bradely Moore团队成功地将S. cattleya中的氟化酶的基因“移植”到S. tropica中,“置换”了S. tropica中原有的氯化酶,相应地,S. tropica中本应由氯化酶催化生成的Salinosporamide A被“置换”后的氟化酶催化生成“氟化版本”的Salinosporamide A[36]。与原产物Salinosporamide A相比(注:Salinosporamide A已于2014年被美国FDA批准成为治疗多发性骨髓瘤的孤儿药,并于同年在欧洲药品管理局EMA获批),“氟化版本”的Salinosporamide A蛋白酶抑制活性大大提高(图 7-A)。在某些细菌体内,烯丙基丙二酰辅酶A (allylmalonyl-CoA)可以作为大环内酯类免疫抑制剂FK506生物合成的延伸单元。同时,这种可以应用特殊延伸单元的细菌底物谱相对广泛,因此科学家们在微生物Streptomyces sp. KCTC 11604BP (链霉菌KCTC 11604BP)体内,将4-氟化丁烯酸酯(4-fluorocrotonate)作为合成前体,利用基因敲除,其allylmalonyl-CoA合成途径被阻断,从而被转化为氟化乙基丙二酰辅酶A (fluoroethylmalonyl-CoA),并参与到Streptomyces sp. KCTC 11604BP体内的天然产物代谢途径,生成最终产物“氟化版本”FK506 (图 7-B)[37]。

|
| 图 7 应用氟化通路和酶工程进行合成生物学的研究 Figure 7 The application of fluorination pathway and enzyme engineering in the study of synthetic biology. |
微生物的分布广泛且种类多样,与动物和植物相比,其丰富的生物多样性更适合于从其次生代谢产物中寻找具有潜在生物活性的先导化合物[38-39]。在此背景下,微生物体内的氟化天然产物代谢途径中,从次生氟化代谢产物中有望获得许多潜在的活性物质,以此发现具有生物活性的氟化有机化合物。革兰氏阳性原核细菌中的放线菌门是微生物药物研究的一个重要来源,主要有放线菌属(Actinomyces)、诺卡氏菌属(Nocardia)和链霉菌属(Streptomyces)等,而链霉菌属是放线菌门中最大的菌属,占比接近70%[40],链霉菌属可以产生大量结构和生物活性各异的次级代谢产物,其代谢产物具有抗细菌、抗真菌、抗病毒、免疫调节剂和抗肿瘤等临床功能。以临床使用的抗生素为例,包括新霉素、链霉素和氯霉素等约2/3的天然抗生素均来自链霉菌[41]。链霉菌属是活性天然产物的来源之一,这吸引了越来越多的研究人员参与到链霉菌属的活性天然产物开发中,其中氟化天然产物合成通路中次级代谢产物的研究最受瞩目[42-44]。
4 氟化酶的进化及氟化物的合成尽管氟化酶应用于氟化天然产物的生物合成领域已经引起了相当大的关注,但是由于底物谱窄和活性低的缺点,其应用受到了极大的限制[45]。因此,为了提高氟化酶对非天然底物5'-氯5'-脱氧腺苷(5'-ClDA)的催化活性,定向进化技术第一次被应用于提高氟化酶对非天然底物的催化效率[46],为后续通过理性设计进一步改造氟化酶打下了基础。此外,氟化酶还可以催化非天然底物5'-ClDA和L-Met进行“转卤素反应”生成5'-FDA,实现了氯元素向氟元素的转化[47-48]。除了氯元素,对于其他卤素取代的脱氧腺苷,在适宜的温度和pH条件下,氟化酶也能进行一步法催化其与F-反应生成5'-FDA[49-50]。根据氟化酶催化底物的混杂性,设计了5'-氯5'-脱氧腺苷单元(ClDA)、PEG间隔单元(PEG)和前列腺特异性膜抗原结合单元(GUL)组成的新型底物ClDA-PEG-GUL,利用氟化酶催化ClDA-PEG-GUL生成FDA-PEG-GUL,不仅扩展了该酶接受底物的范围,而且酶法合成的[18F] FDA-PEG-GUL使其成为用于诊断前列腺癌症的新型PET示踪剂[51]。
为了进一步扩大合成有机氟化物的范围,另类的“氟化产物合成酶”被引入到生物催化中。例如,从Streptomyces sp. MA37中新发现了一种金属依赖型的4-氟苏氨酸转醛酶,可以用于不同构型氟代β-羟基-α-氨基酸的合成[52]。另一种可以进行其他构型氟化产物合成的酶是氟化乙酸脱卤酶RPA1163,借助计算机辅助的半理性设计,成功提高了氟化乙酸脱卤酶RPA1163的催化效率,使其可以高效地催化α-氟代羧酸消旋体生成光学纯的(R)-α-氟代羧酸[53]。此外,由于羟醛缩合反应可以将氟取代的有毒酸类物质转化为高附加值的氟化物[54],因而利用类型Ⅱ醛缩酶催化氟化丙酮酸加成到各种醛中,合成新型氟酸和酯类的衍生物,可用于合成糖、氨基酸和其他有价值手性结构单元的氟化类似物前体[55]。为了进一步提高氟化酶作为生物催化剂的催化效率和稳定性,我们利用自组装短肽标签与目的蛋白结合,促使目的蛋白自组装形成具有纳米尺寸的蛋白质聚集体。其原理是选择3种不同的自组装短肽标签,分别与S. xinghaiensis来源的氟化酶结合后,形成新型的人工氟化酶,将自组装短肽标签应用于氟化酶上,不仅提高了氟化酶的催化效率,实现了5'-FDA的高效合成,并且提高了新型氟化酶的稳定性和可重复利用性(图 8)[56]。生物矿化技术也被用于改善氟化酶的生物合成能力,将氟化酶与氟化的羟基磷灰石组装,同样形成具有纳米结构的新型氟化酶,表现出比游离氟化酶更高的催化效率和稳定性[57]。除了体外生物催化合成氟化有机物,利用全细胞反应的体内生物合成途径也被建立起来,将S. cattleya来源的氟化酶整合到大肠杆菌体内后,通过敲除编码嘌呤核苷磷酸化酶(该酶会降解5'-FDA)的基因以及表达编码跨膜SAM转运蛋白的基因,构建了新型氟化物合成细胞工厂,可以利用SAM和氟离子高效合成氟化产物5'-FDA[58]。
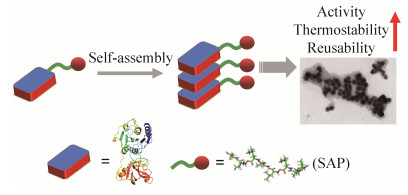
|
| 图 8 新型氟化酶聚集体组装过程 Figure 8 Assembly process of new fluorinase aggregates. |
5 氟化酶及氟化物的应用
在临床疾病的诊断技术中,PET (positron emission tomography,正电子发射断层扫描)作为一种先进的核医学影像技术具有无法取代的地位。在许多大型医院,PET已被用于心血管疾病、神经退行性疾病以及癌症的早期检测中[59-61]。氟-18 (18F)放射性同位素的半衰期大约为110 min,可以用作放射性示踪剂,这种分子探针在PET技术中得到了广泛的应用[62]。传统18F-标记PET示踪剂的合成,通常采用化学合成手段,对葡萄糖分子进行放射性同位素氟-(18F)的标记,这种方式需要多步反应或极端的反应条件,存在选择性差、产率低且不安全经济等不足[63]。与之相比,氟化酶作为一种生物催化剂,可以实现在水溶液中直接快速地进行放射性同位素氟-(18F)的标记,其特有的反应条件温和、检测成本廉价、标记操作简单等优点,使之极有潜力取代传统的化学合成标记法。David O’Hagan教授及其团队开辟了PET在实际应用上的新思路,利用氟化酶能够使用无机氟离子(F-)作为底物的这一重要特性,催化SAM合成[18F]-FDA,然后借助其他有机氟化酶系的催化,一共发现了5条合成放射性同位素标记示踪剂的通路(图 9)[64-67]。Ⅰ和Ⅱ两条通路是碱基交换反应,分别形成[18F]-嘌呤和[18F]-氟尿嘧啶。其中,Ⅰ为嘌呤核苷酸磷酸化酶催化;Ⅱ为嘧啶核苷酸磷酸化酶催化;Ⅲ为Kuhn-Roth氧化(CrO3/H2SO4)催化。通路Ⅳ和Ⅴ分别是腺苷酸脱氨酶和嘌呤核苷酸磷酸化酶催化。相比于其他4条合成通路,第5条通路发展了两代,第一代途径需要两步反应合成[18F]-FDR (5'-氟脱氧核糖),而第二代途径可以通过核苷水解酶(TvNH)直接将[18F]-FDA转换为[18F]-FDR。随后,[18F]-FDR可以通过缀合反应掺入靶向肽(例如RGD肽)[68-69]。同时,小鼠实验表明,与传统的放射性同位素标记的示踪剂[18F]-FDG (氟化脱氧葡萄糖)相比,氟化脱氧核糖的显示效果更好。
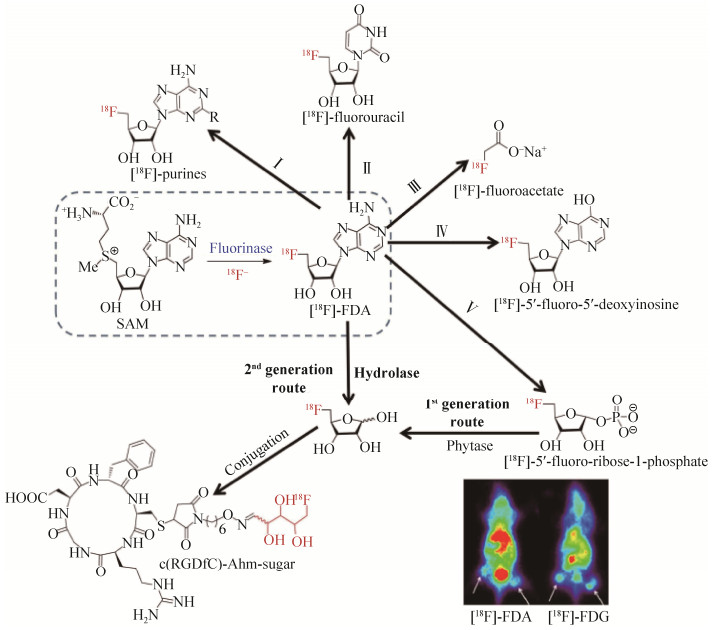
|
| 图 9 基于氟化酶的18F放射性示踪剂 Figure 9 18F labeled radiotracers based on fluorianse. |
6 总结与展望
氟元素作为一种能够赋予有机物特殊性质的卤素,含氟化合物的发现与合成对于生物有机化学、材料学、药物化学等学科的工业化应用具有重要意义。然而,自然界中已发现的氟化天然产物极其稀少,并且现有的氟化生物技术还无法克服氟元素的生物利用率低以及选择性掺入的问题。对微生物体内氟化天然产物合成通路的解析以及次级代谢产物机制的研究,将促使越来越多的人能够了解氟生物化学,同时也能使科学家们更加深入地理解,自然界中存在的无机氟离子是如何经过微生物体内的代谢途径生成新的C-F键最终合成氟化天然有机产物。对现有氟化物生物合成技术的研究,也将有助于人们利用理解和运用绿色节能的生物催化技术,用于氟化物的人工合成。相关科学问题的解析是科研人员研究氟化天然产物合成的基础,随着氟化生物技术这一概念的提出,将进一步推动新型氟化物的发现,尤其是氟化物生物合成的发展。
本文综述了氟化天然产物生物合成的研究进展,针对氟生物技术中氟元素利用率和选择性氟化的挑战性问题,未来利用生物法合成氟化物的研究重点主要在以下几个方面:一、利用基因挖掘和宏基因组技术寻找新型可以催化合成氟化物的酶;二、借助计算生物学和蛋白质工程手段对现有的氟化酶进行定向进化,使其可以合成多种复杂的氟化物;三、对氟化物的代谢通路进行解析,研究生物合成氟化物的新途径;四、利用现有的氟化酶与其他功能的酶偶联克服氟化产物种类少的问题。
| [1] | Dickson AG, Goyet C. Handbook of Methods for the Analysis of the Various Parameters of the Carbon Dioxide System in Sea Water. Version 2. Washington, DC: Oak Ridge National Lab, 1994. |
| [2] | Kirk KL. Fluorination in medicinal chemistry: methods, strategies, and recent developments. Organic Process Research & Development, 2008, 12(2): 305-321. |
| [3] | Isanbor C, O'Hagan D. Fluorine in medicinal chemistry: a review of anti-cancer agents. Journal of Fluorine Chemistry, 2006, 127(3): 303-319. DOI:10.1016/j.jfluchem.2006.01.011 |
| [4] | O'Hagan D. Fluorine in health care: organofluorine containing blockbuster drugs. Journal of Fluorine Chemistry, 2010, 131(11): 1071-1081. DOI:10.1016/j.jfluchem.2010.03.003 |
| [5] | Hagmann WK. The many roles for fluorine in medicinal chemistry. Journal of Medicinal Chemistry, 2008, 51(15): 4359-4369. DOI:10.1021/jm800219f |
| [6] | Wu LR, Maglangit F, Deng H. Fluorine biocatalysis. Current Opinion in Chemical Biology, 2020, 55: 119-126. DOI:10.1016/j.cbpa.2020.01.004 |
| [7] | Deng H, O'Hagan D. The fluorinase, the chlorinase and the duf-62 enzymes. Current Opinion in Chemical Biology, 2008, 12(5): 582-592. DOI:10.1016/j.cbpa.2008.06.036 |
| [8] | Henkel T, Brunne RM, Müller H, Reichel F. Statistical investigation into the structural complementarity of natural products and synthetic compounds. Angewandte Chemie International Edition, 1999, 38(5): 643-647. DOI:10.1002/(SICI)1521-3773(19990301)38:5<643::AID-ANIE643>3.0.CO;2-G |
| [9] | Gribble GW. Occurrence of halogenated alkaloids. The Alkaloids: Chemistry and Biology, 2012, 71: 1-165. |
| [10] | Carvalho MF, Oliveira RS. Natural production of fluorinated compounds and biotechnological prospects of the fluorinase enzyme. Critical Reviews in Biotechnology, 2017, 37(7): 880-897. DOI:10.1080/07388551.2016.1267109 |
| [11] | Deng H, O'Hagan D, Schaffrath C. Fluorometabolite biosynthesis and the fluorinase from Streptomyces cattleya. Natural Product Reports, 2004, 21(6): 773. DOI:10.1039/b415087m |
| [12] | Harper DB, O'Hagan D. The fluorinated natural products. Natural Product Reports, 1994, 11: 123-133. DOI:10.1039/np9941100123 |
| [13] | Marais JSC, Du TPJ. The isolation of the toxic principle "potassium cymonate" from "Gifblaar" Dichapetalum cymosum (Hook) Engl. Onderstepoort Journal Veterinary Science and Animal Industry, 1943, 18: 203-206. |
| [14] | Ma L, Bartholome A, Tong MH, Qin ZW, Yu Y, Shepherd T, Kyeremeh K, Deng H, O'Hagan D. Identification of a fluorometabolite from Streptomyces sp. MA37:(2R3S4S)-5-fluoro-2, 3, 4-trihydroxypentanoic acid. Chemical Science, 2015, 6(2): 1414-1419. DOI:10.1039/C4SC03540B |
| [15] | Ni CF, Hu JB. The unique fluorine effects in organic reactions: recent facts and insights into fluoroalkylations. Chemical Society Reviews, 2016, 45(20): 5441-5454. DOI:10.1039/C6CS00351F |
| [16] | Reichel M, Karaghiosoff K. Reagents for selective fluoromethylation: a challenge in organofluorine chemistry. Angewandte Chemie International Edition, 2020, 59(30): 12268-12281. DOI:10.1002/anie.201913175 |
| [17] | Truppo MD. Biocatalysis in the pharmaceutical industry: the need for speed. ACS Medicinal Chemistry Letters, 2017, 8(5): 476-480. DOI:10.1021/acsmedchemlett.7b00114 |
| [18] | Fryszkowska A, Devine PN. Biocatalysis in drug discovery and development. Current Opinion in Chemical Biology, 2020, 55: 151-160. DOI:10.1016/j.cbpa.2020.01.012 |
| [19] | Dong CJ, Huang FL, Deng H, Schaffrath C, Spencer JB, O'Hagan D, Naismith JH. Crystal structure and mechanism of a bacterial fluorinating enzyme. Nature, 2004, 427(6974): 561-565. DOI:10.1038/nature02280 |
| [20] | O'Hagan D, Schaffrath C, Cobb SL, Hamilton JTG, Murphy CD. Biosynthesis of an organofluorine molecule. Nature, 2002, 416(6878): 279. |
| [21] | Ma L, Li YF, Meng LP, Deng H, Li YY, Zhang Q, Diao AP. Biological fluorination from the sea: discovery of a SAM-dependent nucleophilic fluorinating enzyme from the marine-derived bacterium Streptomyces xinghaiensis NRRL B24674. RSC Advances, 2016, 6(32): 27047-27051. DOI:10.1039/C6RA00100A |
| [22] | Deng H, Ma L, Bandaranayaka N, Qin ZW, Mann G, Kyeremeh K, Yu Y, Shepherd T, Naismith JH, O'Hagan D. Identification of Fluorinases from Streptomyces sp. MA37, Norcardia brasiliensis, and Actinoplanes sp. N902-109 by genome mining. ChemBioChem, 2014, 105(3): 364-368. |
| [23] | Huang S, Ma L, Tong MH, Yu Y, O'Hagan D, Deng H. Fluoroacetate biosynthesis from the marine-derived bacterium Streptomyces xinghaiensis NRRL B-24674. Organic & Biomolecular Chemistry, 2014, 12(27): 4828-4831. |
| [24] | Sooklal SA, De Koning C, Brady D, Rumbold K. Identification and characterisation of a fluorinase from Actinopolyspora mzabensis. Protein Expression and Purification, 2020, 166: 105508. DOI:10.1016/j.pep.2019.105508 |
| [25] | Murphy CD, O'Hagan D, Schaffrath C. Identification of a PLP-dependent threonine transaldolase: a novel enzyme involved in 4-fluorothreonine biosynthesis in Streptomyces cattleya. Angewandte Chemie International Edition, 2001, 40(23): 4479-4481. DOI:10.1002/1521-3773(20011203)40:23<4479::AID-ANIE4479>3.0.CO;2-1 |
| [26] | Huang FL, Haydock SF, Spiteller D, Mironenko T, Li TL, O'Hagan D, Leadlay PF, Spencer JB. The gene cluster for fluorometabolite biosynthesis in Streptomyces cattleya: a thioesterase confers resistance to fluoroacetyl-coenzyme A. Chemistry & Biology, 2006, 13(5): 475-484. |
| [27] | O'Hagan D, Deng H. Enzymatic fluorination and biotechnological developments of the fluorinase. Chemical Reviews, 2015, 115(2): 634-649. DOI:10.1021/cr500209t |
| [28] | Deng H, Cross SM, McGlinchey RP, Hamilton JTG, O'Hagan D. In vitro reconstituted biotransformation of 4-fluorothreonine from fluoride ion: application of the fluorinase. Chemistry & Biology, 2008, 15(12): 1268-1276. |
| [29] | Bartholomé A, Janso JE, Reilly U, O'Hagan D. Fluorometabolite biosynthesis: isotopically labelled glycerol incorporations into the antibiotic nucleocidin in Streptomyces calvus. Organic & Biomolecular Chemistry, 2017, 15(1): 61-64. |
| [30] | Zhu XM, Hackl S, Thaker MN, Kalan L, Weber C, Urgast DS, Krupp EM, Brewer A, Vanner S, Szawiola A, Yim G, Feldmann J, Bechthold A, Wright GD, Zechel DL. Biosynthesis of the fluorinated natural product nucleocidin in Streptomyces calvus is dependent on the bldA-Specified Leu-tRNAUUA molecule. ChemBioChem, 2015, 16(17): 2498-2506. DOI:10.1002/cbic.201500402 |
| [31] | Feng X, Bello D, Lowe PT, Clark J, O'Hagan D. Two 3'-O-β-glucosylated nucleoside fluorometabolites related to nucleocidin in Streptomyces calvus. Chemical Science, 2019, 10(41): 9501-9505. DOI:10.1039/C9SC03374B |
| [32] | Feng X, Maharik NA, Bartholomé A, Janso JE, Reilly U, O'Hagan D. Incorporation of[2H1]-(1R, 2R)-and[2H1]-(1S, 2R)-glycerols into the antibiotic nucleocidin in Streptomyces calvus. Organic & Biomolecular Chemistry, 2017, 15(38): 8006-8008. |
| [33] | Zhang SY, Klementz D, Zhu J, Makitrynskyy R, Pasternak ARO, Günther S, Zechel DL, Bechthold A. Genome mining reveals the origin of a bald phenotype and a cryptic nucleocidin gene cluster in Streptomyces asterosporus DSM 41452. Journal of Biotechnology, 2019, 292: 23-31. DOI:10.1016/j.jbiotec.2018.12.016 |
| [34] | O'Hagan D, Harper DB. Fluorine-containing natural products. Journal of Fluorine Chemistry, 1999, 100(1/2): 127-133. |
| [35] | Walker MC, Thuronyi BW, Charkoudian LK, Lowry B, Khosla C, Chang MCY. Expanding the fluorine chemistry of living systems using engineered polyketide synthase pathways. Science, 2013, 341(6150): 1089-1094. DOI:10.1126/science.1242345 |
| [36] | Eustáquio AS, O'Hagan D, Moore BS. Engineering fluorometabolite production: fluorinase expression in Salinispora tropica yields fluorosalinosporamide. Journal of Natural Products, 2010, 73(3): 378-382. DOI:10.1021/np900719u |
| [37] | Mo SJ, Kim DH, Lee JH, Park JW, Basnet DB, Ban YH, Yoo YJ, Chen SW, Park SR, Choi EA, Kim E, Jin YY, Lee SK, Park JY, Liu Y, Lee MO, Lee KS, Kim SJ, Kim D, Park BC, Lee SG, Kwon HJ, Suh JW, Moore BS, Lim SK, Yoon YJ. Biosynthesis of the Allylmalonyl-CoA extender unit for the FK506 Polyketide synthase proceeds through a dedicated polyketide synthase and facilitates the mutasynthesis of analogues. Journal of the American Chemical Society, 2011, 133(4): 976-985. DOI:10.1021/ja108399b |
| [38] | Demain AL, Sanchez S. Microbial drug discovery: 80 years of progress. The Journal of Antibiotics, 2009, 62(1): 5-16. DOI:10.1038/ja.2008.16 |
| [39] | Harvey AL, Edrada-Ebel RA, Quinn RJ. The re-emergence of natural products for drug discovery in the genomics era. Nature Reviews Drug Discovery, 2015, 14(2): 111-129. DOI:10.1038/nrd4510 |
| [40] | Anderson AS, Wellington EM. The taxonomy of Streptomyces and related genera. International Journal of Systematic and Evolutionary Microbiology, 2001, 51(3): 797-814. DOI:10.1099/00207713-51-3-797 |
| [41] | Newman DJ, Cragg GM. Natural products as sources of new drugs over the 30 years from 1981 to 2010. Journal of Natural Products, 2012, 75(3): 311-335. DOI:10.1021/np200906s |
| [42] |
Tang XM, Xu J. Advance in research on the halogenase from microorganism. Chinese Journal of Antibiotics, 2008, 33(11): 641-644, 677.
(in Chinese) 唐晓敏, 徐俊. 微生物卤代酶研究进展. 中国抗生素杂志, 2008, 33(11): 641-644, 677. |
| [43] |
Gao P, Xi LJ, Piao YH, Ruan JS, Huang Y. Analysis of the halogenase gene in actinomycetes from different habitats and its implications for halometabolite discovery. Acta Microbiologica Sinica, 2009, 49(10): 1367-1373.
(in Chinese) 高鹏, 郗丽君, 朴玉华, 阮继生, 黄英. 不同生境放线菌的卤化酶基因分析及其对卤代产物筛选的意义. 微生物学报, 2009, 49(10): 1367-1373. |
| [44] |
Li H, Zhu L, Chen DJ. Recent developments of FADH2- dependent halogenases involved in the biosynthesis of antibiotics. Chinese Journal of Antibiotics, 2010, 35(1): 1-6.
(in Chinese) 李航, 朱丽, 陈代杰. 参与抗生素生物合成的FADH2依赖型卤化酶研究进展. 中国抗生素杂志, 2010, 35(1): 1-6. |
| [45] | Tong W, Huang Q, Li M, Wang JB. Enzyme-catalyzed C-F bond formation and cleavage. Bioresources and Bioprocessing, 2019, 6(1): 46. DOI:10.1186/s40643-019-0280-6 |
| [46] | Sun HH, Yeo WL, Lim YH, Chew X, Smith DJ, Xue B, Chan KP, Robinson RC, Robins EG, Zhao HM, Ang EL. Directed evolution of a fluorinase for improved fluorination efficiency with a non-native substrate. Angewandte Chemie, 2016, 128(46): 14489-14492. DOI:10.1002/ange.201606722 |
| [47] | Sun H, Zhao H, Ang EL. A coupled chlorinase-fluorinase system with a high efficiency of trans-halogenation and a shared substrate tolerance. Chemical Communications, 2018, 54(68): 9458-9461. DOI:10.1039/C8CC04436H |
| [48] | Lowe PT, Cobb SL, O'Hagan D. An enzymatic Finkelstein reaction: fluorinase catalyses direct halogen exchange. Organic & Biomolecular Chemistry, 2019, 17(32): 7493-7496. |
| [49] | Thompson S, Onega M, Ashworth S, Fleming IN, Passchier J, O'Hagan D. A two-step fluorinase enzyme mediated 18F labelling of an RGD peptide for positron emission tomography. Chemical Communications, 2015, 51(70): 13542-13545. DOI:10.1039/C5CC05013H |
| [50] | Lowe PT, Dall'Angelo S, Devine A, Zanda M, O'Hagan D. Enzymatic fluorination of biotin and tetrazine conjugates for pretargeting approaches to positron emission tomography imaging. ChemBioChem, 2018, 19(18): 1969-1978. DOI:10.1002/cbic.201800234 |
| [51] | Lowe PT, Dall'Angelo S, Fleming IN, Piras M, Zanda M, O'Hagan D. Enzymatic radiosynthesis of a 18F-Glu-Ureido-Lys ligand for the prostate-specific membrane antigen (PSMA). Organic & Biomolecular Chemistry, 2019, 17(6): 1480-1486. |
| [52] | Wu LR, Tong MH, Raab A, Fang Q, Wang S, Kyeremeh K, Yu Y, Deng H. An unusual metal-bound 4-fluorothreonine transaldolase from Streptomyces sp. MA37 catalyses promiscuous transaldol reactions. Applied Microbiology and Biotechnology, 2020, 104(9): 3885-3896. DOI:10.1007/s00253-020-10497-z |
| [53] | Zhang HX, Tian SX, Yue Y, Li M, Tong W, Xu GY, Chen B, Ma M, Li YW, Wang JB. Semirational design of fluoroacetate dehalogenase RPA1163 for kinetic resolution of α-Fluorocarboxylic acids on a gram scale. ACS Catalysis, 2020, 10(5): 3143-3151. DOI:10.1021/acscatal.9b04804 |
| [54] | Saadi J, Wennemers H. Enantioselective aldol reactions with masked fluoroacetates. Nature Chemistry, 2016, 8(3): 276-280. DOI:10.1038/nchem.2437 |
| [55] | Fang J, Hait D, Head-Gordon M, Chang MCY. Chemoenzymatic platform for synthesis of chiral organofluorines based on type Ⅱ aldolases. Angewandte Chemie International Edition, 2019, 58(34): 11841-11845. DOI:10.1002/anie.201906805 |
| [56] | Tu CH, Zhou J, Peng L, Man SL, Ma L. Self-assembled nano-aggregates of fluorinases demonstrate enhanced enzymatic activity, thermostability and reusability. Biomaterials Science, 2020, 8(2): 648-656. DOI:10.1039/C9BM00402E |
| [57] | Li NN, Hu BJ, Wang AM, Li HM, Yin YC, Mao TY, Xie T. Facile bioinspired preparation of Fluorinase@Fluoridated hydroxyapatite nanoflowers for the biosynthesis of 5'-fluorodeoxy adenosine. Sustainability, 2020, 12(1): 431. DOI:10.3390/su12010431 |
| [58] | Markakis K, Lowe PT, Davison-Gates L, O'Hagan D, Rosser SJ, Elfick A. An engineered E. coli strain for direct in vivo fluorination. ChemBioChem, 2020, 21(13): 1856-1860. DOI:10.1002/cbic.202000051 |
| [59] | Yamaga LYI, Thom AF, Wagner J, Baroni RH, Hidal JT, Funari MG. The effect of catecholamines on the glucose uptake in brown adipose tissue demonstrated by 18F-FDG PET/CT in a patient with adrenal pheochromocytoma. European Journal of Nuclear Medicine and Molecular Imaging, 2008, 35(2): 446-447. DOI:10.1007/s00259-007-0538-7 |
| [60] | Sharma R, Aboagye E. Development of radiotracers for oncology-the interface with pharmacology. British Journal of Pharmacology, 2011, 163(8): 1565-1585. DOI:10.1111/j.1476-5381.2010.01160.x |
| [61] | Soussan M, Hyafil F. Can FDG-PET imaging play a role in guiding indications to endovascular treatments in patients presenting acute aortic syndromes?. Journal of Nuclear Cardiology, 2019, 26(6): 642-644. |
| [62] | Cai LS, Lu SY, Pike VW. Chemistry with[18F]fluoride ion. European Journal of Organic Chemistry, 2008, 2008(17): 2853-2873. DOI:10.1002/ejoc.200800114 |
| [63] | Clark J, O'Hagan D. Strategies for radiolabelling antibody, antibody fragments and affibodies with fluorine-18 as tracers for positron emission tomography (PET). Journal of Fluorine Chemistry, 2017, 203: 31-46. DOI:10.1016/j.jfluchem.2017.08.001 |
| [64] | Thompson S, Zhang QZ, Onega M, McMahon S, Fleming I, Ashworth S, Naismith JH, Passchier J, O'Hagan D. A localized tolerance in the substrate specificity of the fluorinase enzyme enables "Last-Step" 18F fluorination of a RGD peptide under ambient aqueous conditions. Angewandte Chemie International Edition, 2014, 53(34): 8913-8918. DOI:10.1002/anie.201403345 |
| [65] | Li XG, Domarkas J, O'Hagan D. Fluorinase mediated chemoenzymatic synthesis of[18F]-fluoroacetate. Chemical Communications, 2010, 46(41): 7819-7821. DOI:10.1039/c0cc02264k |
| [66] | Dall'Angelo S, Bandaranayaka N, Windhorst AD, Vugts DJ, van der Born D, Onega M, Schweiger LF, Zanda M, O'Hagan D. Tumour imaging by Positron Emission Tomography using fluorinase generated 5-[18F] fluoro-5-deoxyribose as a novel tracer. Nuclear Medicine and Biology, 2013, 40(4): 464-470. DOI:10.1016/j.nucmedbio.2013.02.006 |
| [67] | Winkler M, Domarkas J, Schweiger LF, O'Hagan D. Fluorinase-coupled base swaps: synthesis of[18F]-5'-Deoxy-5'-fluorouridines. Angewandte Chemie International Edition, 2008, 47(52): 10141-10143. DOI:10.1002/anie.200804040 |
| [68] | Li XG, Autio A, Ahtinen H, Helariutta K, Liljenbäck H, Jalkanen S, Roivainen A, Airaksinen AJ. Translating the concept of peptidelabeling with 5-deoxy-5-[18F]fluororibose into preclinical practice: 18F-labeling of Siglec-9 peptide for PET imaging of inflammation. Chemical Communications, 2013, 49(35): 3682-3684. DOI:10.1039/C3CC40738A |
| [69] | Dall'Angelo S, Zhang QZ, Fleming IN, Piras M, Schweiger LF, O'Hagan D, Zanda M. Efficient bioconjugation of 5-fluoro-5-deoxy-ribose (FDR) to RGD peptides for positron emission tomography (PET) imaging of αvβ3 integrin receptor. Organic & Biomolecular Chemistry, 2013, 11(27): 4551-4558. |
 2021, Vol. 61
2021, Vol. 61