中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 吴颖, 陈鸿迅, 张立奎. 2021
- Ying Wu, Hongxun Chen, Likui Zhang. 2021
- 极端嗜热古菌8oxoG DNA糖苷酶的研究进展
- Research progress in 8oxoG DNA glycosylases of hyperthermophilic archaea
- 微生物学报, 61(3): 491-500
- Acta Microbiologica Sinica, 61(3): 491-500
-
文章历史
- 收稿日期:2020-04-05
- 修回日期:2020-05-20
- 网络出版日期:2020-08-07
2. 扬州大学广陵学院, 江苏 扬州 225128
2. College of Guangling, Yangzhou University, Yangzhou 225128, Jiangsu Province, China
20世纪90年代,Woese等人提出了三域学说,将古菌放在了与细菌和真核生物同等的分类地位,从而把生命划分为真核生物、细菌和古菌[1]。随着宏基因组学和单细胞基因组技术的运用,人们发现古菌广泛地分布在自然界的各类环境中。目前,古菌分为4个超家族:TACK (Thaumarchaeota、Aigarchaeota、Crenarchaeota和Korarchaeota)、Asgard (Lokiarchaeota、Thorarchaeota、Heimdallarchaeota和Odinarchaeota)、DPANN (Diapherotrites、Parvarchaeota、Aenigmarchaeota、Nanohaloarchaeota和Nanoarchaeota)和Euryarchaeota[2]。新发现的古菌改变了生命系统发育树,扩展了人们对其多样性的认识。古菌细胞内蕴藏着大量未知的生物学过程和功能,它们不仅是研究生命基本规律、极限适应能力、生命起源与演化等的重要研究对象,还有着不可估量的生物技术开发前景。因此,开展古菌的研究具有重要的科学意义和应用价值。
基因组DNA中碱基很容易受到活性氧(Reactive oxygen species,ROS)的攻击使之变成损伤碱基,进而影响到基因组的完整性和稳定性,并可能进一步导致疾病的发生[3-4]。在ROS引起损伤类型中,8oxoG是最为常见的DNA氧化性损伤碱基,主要通过鸟嘌呤碱基的氧化所产生(图 1-A)[5]。研究发现,每一个哺乳动物细胞中每天会产生约100000个8oxoG[6]。通常,8oxoG具有两种构象(图 1-B):一种是反式构象,能够与胞嘧啶形成Watson-Crick配对(8oxoG: C);另一种是顺式构象,能够与腺嘌呤形成Hoogsteen配对(8oxoG: A)[7]。如果8oxoG形成8oxoG: A配对,经过复制后,将会形成A: T配对,从而引起GC→TA突变(图 1-C)[8]。另一方面,DNA聚合酶在DNA中腺嘌呤对应的位置掺入8oxodGTP (由dGTP氧化产生),经过一轮复制后,会导致AT→CG突变[9]。因此,8oxoG会影响细胞基因组的稳定性和完整性,进而对细胞是有害的。
为了修复DNA中的8oxoG,细胞已经演化出碱基切除修复途径(Base excision repair,BER)[10]。研究发现,8oxoG DNA糖苷酶(8-oxoguanine DNA glycosylases,OGG)为BER途径中的第一个酶,其广泛分布在细菌、古菌和真核生物[11]。最新的研究发现,OGG在控制肝脏的糖异生、检测基因组的氧化损伤、疾病诊断等方面起着重要作用[4, 12-13]。无论是源自细菌、真核生物还是古菌的OGG均为双功能酶,即具有DNA糖苷酶活性和AP (apurinic/apyrimidinic)裂合酶活性。双功能OGG作用于含有8oxoG的DNA的催化机理如图 2所示[14]:首先OGG利用DNA糖苷酶活性切割8oxoG与DNA脱氧核糖之间的N-C糖苷键,去除DNA中的8oxoG,形成AP位点,进而形成Schiff碱基中间体;然后利用其AP裂合酶活性,OGG在AP位点处切割DNA的3'-5'磷酸二酯键,通过β-消除反应,产生带有3'-α, β-不饱和醛和5'-磷酸的缺口DNA,或者通过β-和δ-消除反应,产生带有3'-磷酸和5'-磷酸的缺口DNA[15-16]。
古菌在DNA复制和DNA修复方面类似于真核生物,是真核生物DNA复制和DNA修复理想的简化版本。高温环境加快了DNA中碱基的自发水解和氧化,造成DNA的损伤。因此,极端嗜热古菌基因组面临着高温环境所带来的严峻挑战。但是,极端嗜热古菌与常温微生物具有相似的突变频率[17],这暗示着前者具有较强的DNA修复能力。极端嗜热古菌基因组显示,它们均编码OGG。目前,对于细菌和真核生物的OGG已经进行了大量的研究,但是极端嗜热古菌OGG的研究相对较少。本文总结了极端嗜热古菌OGG的研究进展,并对今后的研究进行了一些展望。
1 8oxoG DNA糖苷酶的分类在细菌中,负责去除DNA中8oxoG的DNA糖苷酶为甲酰胺基嘧啶-DNA糖基化酶(formamidopyrimidine-DNA glycosylase,Fpg)。研究发现,Fpg切割含有8oxoG: C配对的dsDNA,通过β, δ-消除反应产生带有3'-磷酸和5'-磷酸的缺口DNA[18]。而在真核生物、古菌和少数细菌中,识别和切除8oxoG的DNA糖苷酶为OGG。大多数真核生物和古菌的OGG切割含有8oxoG: C配对的dsDNA,并通过β-消除反应产生带有3'-α, β-不饱和醛和5'-磷酸的缺口DNA。目前,OGG分为3个家族:OGG1、OGG2和AGOG (archaeal GO glycosylase)[18]。表 1总结了3个家族OGG的异同点。氨基酸序列比对和晶体结构显示,FpG和OGG成员均具有螺旋-发夹-螺旋(helix-hairpin-helix, HhH) 结构,属于HhH-GPD (a glycine/proline-rich sequence followed by an absolutely conserved aspartate,富含甘氨酸/脯氨酸和高度保守的天冬氨酸序列) 超家族[18]。
| Family | OGG1 | OGG2 | AGOG |
| Member source | Bacteria and eukaryotes | Archaea and bacteria | Archaea |
| Number of conserved motifs | 3 | 2 | 2 |
| Amino acid sequence similarity | Low | Low | Low |
| Member size | Major difference | Constant | Minor difference |
| HhH motif | Yes | Yes | Yesa |
| Crystal structure | Similar | Similar | Similar |
| Catalytic mechanism | Same | Same | Same |
| a: AGOG has a distinct HhH motif from OGG1 and OGG2. | |||
OGG1家族成员最多,包括已研究清楚的人类OGG1 (h-OGG1)[19-26]和细菌Clostridium acetobutylicum OGG1 (Cac-OGG)[27]。尽管OGG1成员之间大小不同,但是它们具有相似的空间结构,该结构由3个结构域组成。OGG2家族成员主要包括源自古菌和少数细菌的糖苷酶,例如Methanococcus jannaschii OGG (Mja-OGG)[28]、Sulfolobus solfataricus OGG (Sso-OGG)[29]、Archaeoglobus fulgidus OGG (Afu-OGG)[30]、Thermoplasma volcanium OGG (Tvo-OGG)[31]和细菌Thermotoga maritima OGG (Tma-OGG)[32]。在空间结构方面,OGG2酶不同于OGG1,只具有2个结构域,缺少OGG1酶的A-结构域。另外,OGG2家族成员大小恒定(约207氨基酸),并且对DNA底物的严谨性比OGG1成员更低。
作为第三个家族,AGOG成员只源自于古菌。目前,已报道的AGOG只有3个,分别是:Pyrobaculum aerophilum OGG (Pae-OGG)[33]、Thermococcus gammatolerans (Tga-OGG)[34]和Thermococcus kodakarensi OGG (Tko-AGOG)[14]。尽管AGOG具有与OGG2相似的结构域,但是它们的HhH结构域与OGG1和OGG2家族的HhH结构域不同。
除了具有HhH结构域的核心序列之外,3个OGG家族成员中氨基酸序列的相似性非常低[18],例如,OGG2酶与OGG1酶的氨基酸序列相似性只有13%-19%。甚至OGG1成员之间的氨基酸序列相似性也非常低,比如Cac-OGG1与h-OGG1只具有28%氨基酸序列的等同性。尽管OGG存在氨基酸序列的差异性,但是它们具有相似的空间结构、保守的催化位点和相似的活性中心,这都暗示着它们具有相同的催化机理。
2 极端嗜热古菌8oxoG DNA糖苷酶如前文所述,源自于极端嗜热古菌的8oxoG DNA糖苷酶分别属于OGG2家族和AGOG家族,它们在氨基酸组成和HhH结构等方面存在明显差异。第一个被报道的古菌OGG源自极端嗜热古菌M. jannaschii,属于OGG2家族[28]。Mja-OGG与酵母菌OGG1 (y-OGG1)和h-OGG1在氨基酸序列方面具有非常低的相似性。Mja-OGG具有DNA糖苷酶和DNA裂合酶的活性,并且表现出对含有8oxoG: C配对的DNA非常强的偏好性,这不同于真核生物的OGG[28]。突变分析结果表明,Mja-OGG突变体K129S失去了切割DNA的活性。序列比对结果显示,Mja-OGG中的K129对应y-OGG1中的K241。研究发现,y-OGG1中的K241能够与DNA中的糖形成Schiff碱基,它是该酶发挥裂合酶活性所必需的[35]。因此,Mja-OGG中的K129是该酶的关键氨基酸残基之一。
Faucher等解析了Mja-OGG和Sso-OGG的晶体结构[29]。通过与OGG1和AGOG空间结构比较,可以推测Mja-OGG中C末端的K207是该酶区分G和8oxoG的关键氨基酸残基,后续的突变分析结果证实了这一推测。另外,Faucher等还解析了Mja-OGG与含有8oxoG的DNA形成复合物的晶体结构[36],进一步显示了该酶的K207是OGG2家族成员中保守的氨基酸残基,具有识别8oxoG的功能。此外,该复合物的晶体结构还揭示了该酶通过构象变化结合DNA,这与OGG1酶结合DNA的方式相似。目前,Sso-OGG的生化性质和催化机理尚未被研究。
源自极端嗜热古菌A. fulgidus的OGG也是双功能酶,即具有DNA糖苷酶和AP裂合酶的活性。该酶的最适pH为8.5,最适温度为60 ℃,不能耐受80 ℃,但是能够耐受500 mmol/L的盐[30]。进一步的生化性质研究表明,Afu-OGG能够有效地切割含有8oxoG: C和8oxoG: G配对的DNA,但是对于含有8-oxoG: T和8oxoG: A配对的DNA的切割效率非常低。尽管Afu-OGG对含有8oxoG的DNA的切割催化机理与h-OGG1相似,但是该酶识别8oxoG的机制和对底物的专一性不同于h-OGG1。
嗜酸热古菌T. volcanium的OGG与Mja-OGG和Sso-OGG序列同源性较高。生化性质的研究结果表明,Tvo-OGG的最适反应pH为7.5,其最适反应温度为60 ℃[31]。该酶也是具有DNA糖苷酶活性和AP裂合酶活性的双功能酶,切割含有8oxoG的DNA。但是,Tvo-OGG不能切除尿嘧啶: G配对中的尿嘧啶和T: G配对中的T。
极端嗜热古菌P. aerophilum的OGG也属于AGOG家族,能够有效地切除DNA双链和单链中的8oxoG[33]。Lingaraju等解析了Pae-AGOG和该酶与8oxoG形成复合物的晶体结构[37]。尽管Pae-AGOG具有OGG中保守的HhH结构域,但是该酶的HhH结构不同于其他OGG的HhH结构,这暗示着该酶作用于DNA的方式不同于其他OGG。Pae-AGOG通过氢键和碱基堆积力识别8oxoG,这不同于h-OGG1,但是两者可能具有相似的催化机理。
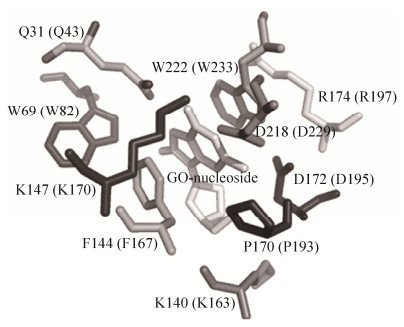
突变分析的结果表明,Pae-AGOG中的W222、W69、K147、D218和N31是该酶识别8oxoG的关键氨基酸残基,而K140和D172参与该酶切除8oxoG (图 3)[38]。另外,Pae-AGOG突变体D172Q失去了切割活性,而对应的h-OGG1突变体仍具有切割活性。进一步的研究发现,Pae-AGOG突变体K140Q、D172N和D172Q结合含有8oxoG的ssDNA比结合含有8oxoG: C配对的dsDNA具有更高的亲和力。因此,Pae-AGOG具有独特的HhH结构,暗示着该酶具有不同于其他OGG的功能。
作为超嗜热古菌的模式生物[39],极端嗜热古菌T. kodakarensis OGG的催化机理、动力学和底物专一性最近也得到了研究[14]。最新的研究表明,Tko-AGOG是极端嗜热古菌T. kodakarensis细胞中重要的OGG,但是其并非为细胞所必需的[14]。另外,Tko-AGOG介导的完整BER途径得到了阐明,即在Tko-AGOG、AP核酸内切酶、DNA聚合酶B和DNA连接酶的作用下修复DNA中的8oxoG[14]。
3 极端嗜热耐辐射古菌T. gammatolerans 8oxoG DNA糖苷酶极端嗜热耐辐射古菌T. gammatolerans分离于加利福尼亚湾的热液口,其最适生长温度为88 ℃,最适生长pH为6.0[40]。研究发现,古菌T. gammatolerans能够耐受5.0 kGy剂量的γ辐射,是迄今为止最耐辐射的古菌[40]。目前,关于耐辐射古菌DNA修复机制的研究越来越多[41],但是对古菌T. gammatolerans DNA修复机制仍然知之甚少。T. gammatolerans的基因组测序已完成,其G+C含量为51.3%[42],编码一种古菌8oxoG DNA糖苷酶(Tga-AGOG)。Barbier等发现Tga-AGOG能够在高温条件下切除DNA中的GO[34],但是关于该酶的生化性质和催化机理尚未进行研究。
我们实验室最近克隆表达并纯化了Tga-AGOG[43]。对其生化性质的研究发现,Tga-AGOG能够在45-95 ℃范围内切割含有8oxoG的DNA,其最适反应温度为85 ℃,最适反应pH为7.0-8.5。该酶的切割活性不依赖二价金属离子,但Cu2+和Co2+会抑制其活性。在高盐条件下,该酶活性受到显著抑制。我们还发现Tga-AGOG能够切除DNA中的8oxoG,并通过β-消除反应产生带有3'-α, β-不饱和醛和5'-磷酸的缺口DNA。进一步的研究发现,Tga-AGOG切割含有8oxoG的dsDNA的效率不同,其切割效率的顺序为8oxoG: C > 8oxoG: T > 8oxoG: A > 8oxoG: G。
Pae-AGOG与8oxoG形成复合物的晶体结构显示(图 3),除了前文讨论的氨基酸残基之外,该酶的F144、P170和R174接近于8oxoG,但是这三个氨基酸残基的功能尚不清楚。氨基酸序列比对显示,Tga-AGOG的F167、P193和R197对应于Pae-AGOG的F144、P170和R174。突变分析结果表明,R197是Tga-AGOG切割含有8oxoG的DNA的关键氨基酸残基之一,而F167和P193不参与切割含有8oxoG的DNA,从而完善了AGOG识别和切割DNA中的8oxoG的分子机制。更为重要的是,我们研究了Tga-AGOG在不同温度下切割含有8oxoG的DNA的反应速率,从而揭示了该酶切除DNA中的8oxoG所需的激活自由能(16.9±0.9 kcal/mol)。
4 总结和展望鸟嘌呤被ROS氧化生成8oxoG,是常见的DNA损伤碱基之一。为了修复DNA中的8oxoG,细胞内的OGG能够识别和切割DNA中的8oxoG,从而启动BER途径。古菌广泛地分布在自然界的各种环境中,其基因组均编码至少一种8oxoG DNA糖苷酶。AGOG含有OGG中保守的HhH结构域,具有糖苷酶和AP-裂合酶两种活性,从而能够及时地识别和切除DNA中的8oxoG,避免基因组中8oxoG的积累。
AGOG能够切除DNA中的8oxoG,并在8oxoG所在的DNA单链上形成一个缺口,而这个缺口被其他核酸代谢酶或蛋白所利用,进而完成后续的修复。目前,AGOG切割含有8oxoG的DNA所形成的缺口,在古菌细胞内会被哪些核酸代谢酶所利用;古菌细胞内存在哪些蛋白能够与AGOG相互作用以及如何相互作用共同完成修复DNA中的8oxoG,都是值得深入探讨的科学问题。
尽管古菌AGOG驱动的BER途径对于修复DNA中的8oxoG起着重要的作用,但是古菌细胞中是否存在修复DNA中的8oxoG的其他修复途径,尚未清楚。古菌,特别是极端嗜热古菌,编码多种修复型核酸内切酶[44]。最近,我们发现极端嗜热古菌T. gammatolerans NucS核酸内切酶能够切割含有尿嘧啶和次黄嘌呤的DNA[45],并且还能切割含有8oxoG的DNA(尚未发表),暗示着该酶介导的修复可能是修复DNA中8oxoG的一种新的替代途径。目前,极端嗜热古菌NucS核酸内切酶切割含有8oxoG的催化机制需要进一步研究。
| [1] | Woese CR, Kandler O, Wheelis ML. Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya. Proceedings of the National Academy of Sciences of the United States of America, 1990, 87(12): 4576-4579. DOI:10.1073/pnas.87.12.4576 |
| [2] | Baker BJ, De Anda V, Seitz KW, Dombrowski N, Santoro AE, Lloyd KG. Diversity, ecology and evolution of Archaea. Nature Microbiology, 2020, 5(7): 887-900. DOI:10.1038/s41564-020-0715-z |
| [3] | Harman D. Role of free radicals in aging and disease. Annals of the New York Academy of Sciences, 1992, 673(S1): 126-141. |
| [4] | Sliwinska A, Kwiatkowski D, Czarny P, Toma M, Wigner P, Drzewoski J, Fabianowska-Majewska K, Szemraj J, Maes M, Galecki P, Sliwinski T. The levels of 7, 8-dihydrodeoxyguanosine (8-oxoG) and 8-oxoguanine DNA glycosylase 1(OGG1)-A potential diagnostic biomarkers of Alzheimer's disease. Journal of the Neurological Sciences, 2016, 368: 155-159. DOI:10.1016/j.jns.2016.07.008 |
| [5] | Grollman AP, Moriya M. Mutagenesis by 8-oxoguanine: an enemy within. Trends in Genetics, 1993, 9(7): 246-249. DOI:10.1016/0168-9525(93)90089-Z |
| [6] | Park EM, Shigenaga MK, Degan P, Korn TS, Kitzler JW, Wehr CM, Kolachana P, Ames BN. Assay of excised oxidative DNA lesions: isolation of 8-oxoguanine and its nucleoside derivatives from biological fluids with a monoclonal antibody column. Proceedings of the National Academy of Sciences of the United States of America, 1992, 89(8): 3375-3379. DOI:10.1073/pnas.89.8.3375 |
| [7] | Kuchino Y, Mori F, Kasai H, Inoue H, Iwai S, Miura K, Ohtsuka E, Nishimura S. Misreading of DNA templates containing 8-hydroxydeoxyguanosine at the modified base and at adjacent residues. Nature, 1987, 327(6117): 77-79. DOI:10.1038/327077a0 |
| [8] | Shibutani S, Takeshita M, Grollman AP. Insertion of specific bases during DNA synthesis past the oxidation-damaged base 8-oxodG. Nature, 1991, 349(6308): 431-434. DOI:10.1038/349431a0 |
| [9] | Shimizu M, Gruz P, Kamiya H, Kim SR, Pisani FM, Masutani C, Kanke Y, Harashima H, Hanaoka F, Nohmi T. Erroneous incorporation of oxidized DNA precursors by Y-family DNA polymerases. EMBO Reports, 2003, 4(3): 269-273. DOI:10.1038/sj.embor.embor765 |
| [10] | Wallace SS. Base excision repair: a critical player in many games. DNA Repair, 2014, 19: 14-26. DOI:10.1016/j.dnarep.2014.03.030 |
| [11] | Hazra TK, Hill JW, Izumi T, Mitra S. Multiple DNA glycosylases for repair of 8-oxoguanine and their potential in vivo functions. Progress in Nucleic Acid Research and Molecular Biology, 2001, 68: 193-205. |
| [12] | Fang YX, Zou P. Genome-wide mapping of oxidative DNA damage via engineering of 8-oxoguanine DNA glycosylase. Biochemistry, 2020, 59(1): 85-89. DOI:10.1021/acs.biochem.9b00782 |
| [13] | Scheffler K, Rachek L, You PP, Rowe AD, Wang W, Kuśnierczyk A, Kittelsen L, Bjørås M, Eide L. 8-oxoguanine DNA glycosylase (Ogg1) controls hepatic gluconeogenesis. DNA Repair, 2018, 61: 56-62. DOI:10.1016/j.dnarep.2017.11.008 |
| [14] | Gehring AM, Zatopek KM, Burkhart BW, Potapov V, Santangelo TJ, Gardner AF. Biochemical reconstitution and genetic characterization of the major oxidative damage base excision DNA repair pathway in Thermococcus kodakarensis. DNA Repair, 2020, 86: 102767. DOI:10.1016/j.dnarep.2019.102767 |
| [15] | Schermerhorn KM, Delaney S. A chemical and kinetic perspective on base excision repair of DNA. Accounts of Chemical Research, 2014, 47(4): 1238-1246. DOI:10.1021/ar400275a |
| [16] | Sun B, Latham KA, Dodson ML, Lloyd RS. Studies on the catalytic mechanism of five DNA glycosylases: probing for enzyme-DNA imino intermediates. The Journal of Biological Chemistry, 1995, 270(33): 19501-19508. DOI:10.1074/jbc.270.33.19501 |
| [17] | Grogan DW, Carver GT, Drake JW. Genetic fidelity under harsh conditions: analysis of spontaneous mutation in the thermoacidophilic archaeon Sulfolobus acidocaldarius. Proceedings of the National Academy of Sciences of the United States of America, 2001, 98(14): 7928-7933. DOI:10.1073/pnas.141113098 |
| [18] | Faucher F, Doublie S, Jia ZC. 8-oxoguanine DNA glycosylases: one lesion, three subfamilies. International Journal of Molecular Sciences, 2012, 13(6): 6711-6729. DOI:10.3390/ijms13066711 |
| [19] | Šebera J, Hattori Y, Sato D, Řeha D, Nencka R, Kohno T, Kojima C, Tanaka Y, Sychrovský V. The mechanism of the glycosylase reaction with hOGG1 base-excision repair enzyme: concerted effect of Lys249 and Asp268 during excision of 8-oxoguanine. Nucleic Acids Research, 2017, 45(9): 5231-5242. DOI:10.1093/nar/gkx157 |
| [20] | Popov AV, Yudkina AV, Vorobjev YN, Zharkov DO. Catalytically competent conformation of the active site of human 8-oxoguanine-DNA glycosylase. Biochemistry (Moscow), 2020, 85(2): 192-204. DOI:10.1134/S0006297920020066 |
| [21] | Tyugashev TE, Vorobjev YN, Kuznetsova AA, Lukina MV, Kuznetsov NA, Fedorova OS. Roles of active-site amino acid residues in specific recognition of DNA lesions by human 8-oxoguanine-DNA glycosylase (OGG1). The Journal of Physical Chemistry B, 2019, 123(23): 4878-4887. DOI:10.1021/acs.jpcb.9b02949 |
| [22] | Shinmura K, Kasai H, Sasaki A, Sugimura H, Yokota J. 8-hydroxyguanine (7, 8-dihydro-8-oxoguanine) DNA glycosylase and AP lyase activities of hOGG1 protein and their substrate specificity. Mutation Research/DNA Repair, 1997, 385(1): 75-82. DOI:10.1016/S0921-8777(97)00041-4 |
| [23] | Arai K, Morishita K, Shinmura K, Kohno T, Kim SR, Nohmi T, Taniwaki M, Ohwada S, Yokota J. Cloning of a human homolog of the yeast OGG1 gene that is involved in the repair of oxidative DNA damage. Oncogene, 1997, 14(23): 2857-2861. DOI:10.1038/sj.onc.1201139 |
| [24] | Bjørås M, Luna L, Johnsen B, Hoff E, Haug T, Rognes T, Seeberg E. Opposite base-dependent reactions of a human base excision repair enzyme on DNA containing 7, 8-dihydro-8-oxoguanine and abasic sites. The EMBO Journal, 1997, 16(20): 6314-6322. DOI:10.1093/emboj/16.20.6314 |
| [25] | Radicella JP, Dherin C, Desmaze C, Fox MS, Boiteux S. Cloning and characterization of hOGG1, a human homolog of the OGG1 gene of Saccharomyces cerevisiae. Proceedings of the National Academy of Sciences of the United States of America, 1997, 94(15): 8010-8015. DOI:10.1073/pnas.94.15.8010 |
| [26] | Rosenquist TA, Zharkov DO, Grollman AP. Cloning and characterization of a mammalian 8-oxoguanine DNA glycosylase. Proceedings of the National Academy of Sciences of the United States of America, 1997, 94(14): 7429-7434. DOI:10.1073/pnas.94.14.7429 |
| [27] | Faucher F, Robey-Bond SM, Wallace SS, Doublie S. Structural characterization of Clostridium acetobutylicum 8-oxoguanine DNA glycosylase in its apo form and in complex with 8-oxodeoxyguanosine. Journal of Molecular Biology, 2009, 387(3): 669-679. DOI:10.1016/j.jmb.2009.01.067 |
| [28] | Gogos A, Clarke ND. Characterization of an 8-oxoguanine DNA glycosylase from Methanococcus jannaschii. The Journal of Biological Chemistry, 1999, 274(43): 30447-30450. DOI:10.1074/jbc.274.43.30447 |
| [29] | Faucher F, Duclos S, Bandaru V, Wallace SS, Doublié S. Crystal structures of two archaeal 8-oxoguanine DNA glycosylases provide structural insight into guanine/8-oxoguanine distinction. Structure, 2009, 17(5): 703-712. DOI:10.1016/j.str.2009.03.007 |
| [30] | Chung JH, Suh MJ, Park YI, Tainer JA, Han YS. Repair activities of 8-oxoguanine DNA glycosylase from Archaeoglobus fulgidus, a hyperthermophilic archaeon. Mutation Research/DNA Repair, 2001, 486(2): 99-111. DOI:10.1016/S0921-8777(01)00081-7 |
| [31] | Fujii M, Hata C, Ukita M, Fukushima C, Matsuura C, Kawashima-Ohya Y, Tomobe K, Kawashima T. Characterization of a thermostable 8-oxoguanine DNA glycosylase specific for GO/N mismatches from the thermoacidophilic archaeon Thermoplasma volcanium. Archaea, 2016, 2016: 8734894. |
| [32] | Im EK, Hong CH, Back JH, Han YS, Chung JH. Functional identification of an 8-oxoguanine specific endonuclease from Thermotoga maritima. Korean Society for Biochemistry and Molecular Biology, 2005, 38(6): 676-682. |
| [33] | Sartori AA, Lingaraju GM, Hunziker P, Winkler FK, Jiricny J. Pa-AGOG, the founding member of a new family of archaeal 8-oxoguanine DNA-glycosylases. Nucleic Acids Research, 2014, 32(22): 6531-6539. |
| [34] | Barbier E, Lagorce A, Hachemi A, Dutertre M, Gorlas A, Morand L, Saint-Pierre C, Ravanat JL, Douki T, Armengaud J, Gasparutto D, Confalonieri F, Breton J. Oxidative DNA damage and repair in the radioresistant archaeon Thermococcus gammatolerans. Chemical Research in Toxicology, 2016, 29(11): 1796-1809. DOI:10.1021/acs.chemrestox.6b00128 |
| [35] | Nash HM, Bruner SD, Scharer OD, Kawate T, Addona TA, Spooner E, Lane WS, Verdine GL. Cloning of a yeast 8-oxoguanine DNA glycosylase reveals the existence of a base-excision DNA-repair protein superfamily. Current Biology, 1996, 6(8): 968-980. DOI:10.1016/S0960-9822(02)00641-3 |
| [36] | Faucher F, Wallace SS, Doublié S. The C-terminal lysine of Ogg2 DNA glycosylases is a major molecular determinant for guanine/8-oxoguanine distinction. Journal of Molecular Biology, 2010, 397(1): 46-56. DOI:10.1016/j.jmb.2010.01.024 |
| [37] | Lingaraju GM, Sartori AA, Kostrewa D, Prota AE, Jiricny J, Winkler FK. A DNA glycosylase from Pyrobaculum aerophilum with an 8-oxoguanine binding mode and a noncanonical helix-hairpin-helix structure. Structure, 2005, 13(1): 87-98. DOI:10.1016/j.str.2004.10.011 |
| [38] | Lingaraju GM, Prota AE, Winkler FK. Mutational studies of Pa- AGOG DNA glycosylase from the hyperthermophilic crenarchaeon Pyrobaculum aerophilum. DNA Repair, 2009, 8(7): 857-864. DOI:10.1016/j.dnarep.2009.03.009 |
| [39] | Atomi H, Reeve J. Microbe profile: Thermococcus kodakarensis: the model hyperthermophilic archaeon. Microbiology, 2019, 165(11): 1166-1168. DOI:10.1099/mic.0.000839 |
| [40] | Jolivet E, L'Haridon S, Corre E, Forterre P, Prieur D. Thermococcus gammatolerans sp. nov., a hyperthermophilic archaeon from a deep-sea hydrothermal vent that resists ionizing radiation. International Journal of Systematic and Evolutionary Microbiology, 2003, 53(3): 847-851. DOI:10.1099/ijs.0.02503-0 |
| [41] | Ishino Y, Narumi I. DNA repair in hyperthermophilic and hyperradioresistant microorganisms. Current Opinion in Microbiology, 2015, 25: 103-112. DOI:10.1016/j.mib.2015.05.010 |
| [42] | Zivanovic Y, Armengaud J, Lagorce A, Leplat C, Guérin P, Dutertre M, Anthouard V, Forterre P, Wincker P, Confalonieri F. Genome analysis and genome-wide proteomics of Thermococcusgammatolerans, the most radioresistant organism known amongst the Archaea. Genome Biology, 2009, 10(6): R70. DOI:10.1186/gb-2009-10-6-r70 |
| [43] | Zhang LK, Li YT, Shi HQ, Zhang D, Yang ZH, Oger P, Zheng JT. Biochemical characterization and mutational studies of the 8-oxoguanine DNA glycosylase from the hyperthermophilic and radioresistant archaeon Thermococcus gammatolerans. Applied Microbiology and Biotechnology, 2019, 103(19): 8021-8033. DOI:10.1007/s00253-019-10031-w |
| [44] |
Li YT, Shi HQ, Zhang LK. Research progress of hyperthermophilic archaeal DNA repair endonucleases. Acta Microbiologica Sinica, 2019, 59(10): 1889-1896.
(in Chinese) 李玉婷, 史昊强, 张立奎. 极端嗜热古菌DNA修复核酸内切酶的研究进展. 微生物学报, 2019, 59(10): 1889-1896. |
| [45] | Zhang LK, Shi HQ, Gan Q, Wang YX, Wu M, Yang ZH, Oger P, Zheng JT. An alternative pathway for repair of deaminated bases in DNA triggered by archaeal NucS endonuclease. DNA Repair, 2020, 85: 102734. DOI:10.1016/j.dnarep.2019.102734 |
 2021, Vol. 61
2021, Vol. 61