中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 刘京伟, 李香真, 姚敏杰. 2021
- Jingwei Liu, Xiangzhen Li, Minjie Yao. 2021
- 植物根际微生物群落构建的研究进展
- Research progress on assembly of plant rhizosphere microbial community
- 微生物学报, 61(2): 231-246
- Acta Microbiologica Sinica, 61(2): 231-246
-
文章历史
- 收稿日期:2020-03-16
- 修回日期:2020-09-12
- 网络出版日期:2020-09-22
2. 中国科学院大学, 北京 100049;
3. 福建农林大学资源与环境学院土壤修复福建省高校工程研究中心, 福建 福州 350002
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. Engineering Research Center of Soil Remediation of Fujian Province University, College of Resources and Environment, Fujian Agriculture and Forestry University, Fuzhou 350002, Fujian Province, China
土壤是地球表面的一层疏松的物质,由矿物质、有机物质、水分、空气等组成,这些物质构成了微生物的天然培养基,通常一克土壤中有数亿以上的微生物细胞[1]。德国科学家Hiltner于1904年首次提出了不同于原土体的根际土壤的概念[2],即受植物根系影响的、距离根表 1–2 mm的土壤,其理化性质、营养物质的含量和微生物的组成与非根际土壤有所差异。如根系生长过程中通过影响土壤容重、孔隙度等因素改变了土壤结构,与非根际土壤相比,根际土壤物理结构表现出更强的稳定性[3]。植物生长过程中释放20%–60%的光合产物,如糖、有机酸、氨基酸及死亡的边缘细胞进入根际微域[4],根系分泌物中各种水解酶在碳、氮、磷、硫循环中发挥了重要作用,改变了根际土壤养分浓度;此外,根系分泌的黏液中含有表面活性剂,如卵磷脂,可以通过减小土壤溶液的表面张力使根际土壤中更多的水被释放出来,从而改变根际土壤的释水特性[5]。根际土壤养分含量和理化性质的变化影响根际土壤微生物的活动进而影响植物生长。总之,根际是土壤-微生物-植物间相互作用的重要界面,是养分或者有害物质从土壤进入植物的重要门户和通道。本文将从根际微生物群落的研究现状、研究方法、影响因素、微生物-植物互作机制和根际微生物的应用研究进展5个方面进行综述。
1 根际微生物群落的研究现状植物根际是根系自身生命活动和代谢对土壤影响最直接、最强烈的区域。土壤中细菌、真菌等微生物受到植物根系分泌物的吸引富集在根际土壤中,在植物生长发育过程中发挥了重要作用。植物根际土壤中微生物的数量比非根际土壤中多几倍至几十倍,但从非根际土壤到根际土壤,细菌群落的多样性呈下降趋势[6]。植物根际土壤和非根际土壤中微生物群落结构的差异,很大程度上归因于植物对微生物群落的强烈选择或抑制作用。非根际土壤中寡营养型细菌如酸杆菌门(Acidobacteria)在数量上占绝对优势,植物根际土壤中的微生物群落以生长速度更快的富营养型细菌为主,如变形菌门(Proteobacteria)、拟杆菌门(Bacteriodetes)、厚壁菌门(Firmicutes)、放线菌门(Actinobacteria)等[7],它们可以释放并帮助根系吸收植物可利用的钾、磷和其他微量营养元素[8]。
植物根际微生物群落的装配是由微生物、植物宿主和环境之间复杂的相互作用控制的,根际微生物群落处于动态波动中,相互发生作用,从而构成了复杂的根际微生物网络[9]。Xu等(2018)在研究不同地理区域柑橘根际和非根际土壤微生物群落结构和功能组成发现,柑橘根际的核心微生物菌群包括假单胞菌(Pseudomonas)、农杆菌(Agrobacterium)、贪铜菌(Cupriavidus)、根瘤菌(Rhizobium)、伯克氏菌(Burkholderia)、纤维弧菌(Cellvibrio)、鞘氨醇单胞菌(Sphingomonas)和贪噬菌(Variovorax)等,并具备调节微生物-植物和微生物-微生物相互作用,营养获取以及促进植物生长等功能特征[10]。Gao等(2020)通过研究高粱生长发育和干旱胁迫对其根际微生物组的影响,发现高粱对土壤真菌物种库具有强烈的筛选作用,高粱根际土壤中丛枝菌根真菌(Arbuscular mycorrhizal fungi,AMF)、担子菌酵母(Basidiomycetous yeast)、病原菌和内生真菌的多样性增加,而腐生菌的多样性降低。高粱生长早期,真菌群落的随机性过程发挥了重要作用;在生长后期随着干旱的胁迫,真菌群落的随机性过程减弱,植物寄主对真菌的群落构建起到了强烈的选择作用[11]。在被不同植物选择的微生物类群中,如假单胞菌和木霉菌(Trichoderma),或多或少地存在于各种植物根际,表明这些微生物菌群可能是根际微生物的共有菌群,这种微生物的群落组成易趋向于随机性过程的组装。有些植物可以通过根系分泌物和作为信号的化合物从土壤库中招募特定的微生物到根际,如豆科植物以黄酮类化合物为化学引诱物吸引根瘤菌[12];或者通过不同养分资源(如C、N、P等养分循环)对微生物群落进行选择作用,根际微生物群落组装趋于确定性过程,形成根际特定的生态位来促进植物生长发育[13]。
1.1 碳循环相关的根际微生物与非根际土壤相比,植物的可溶性根系分泌物为根际微生物提供了丰富的有效性碳源,植物根系分泌物的输入和凋落物分解等过程相互作用引起的根际激发效应还能刺激微生物的生命活动,对根际微生物的群落结构和生态功能具有重大影响。一些细菌如变形菌门、拟杆菌门(Bacteroides)、放线菌门,真菌如子囊菌(Ascomycetes)和菌根真菌如AMF、杜鹃花类菌根(Ericoid mycorrhiza)等对碳源的趋化反应是其在根部定殖的第一步[14-15]。植物根际糖类、氨基酸等易被微生物同化,形成了碳源比较丰富的环境,拟杆菌门和变形菌门优先定殖在植物根部[15]。此外,一些低分子量含碳化合物如二羧酸通过酸化根际土壤,导致β-变形菌(Betaproteobacteria)和放线菌的相对丰度增加[14]。Kraft等(2018)研究证明,菌根真菌在植物根际碳分配方面起着重要的作用,与菌根共生的植物向根外释放的碳源大部分优先分配给菌根真菌,此过程在较短时间内完成后,菌根真菌逐渐将这些碳释放到根际,供微生物代谢利用[16]。
1.2 氮循环相关的根际微生物生物固氮主要由共生固氮微生物和非共生固氮微生物参与,共生固氮微生物与一些植物互利共生,如根瘤菌在豆科植物的根上形成特殊的根瘤,并固定氮以交换寄主植物的碳。豆科植物固定的铵盐释放到周围土壤中还能被氨氧化细菌及亚硝酸盐氧化菌转化为亚硝酸盐和硝酸盐,从而推动根际土壤中的氮循环过程[17]。Dardanelli等(2008)的研究发现,固氮螺菌属(Azospirillum)通过增加根瘤菌的感染部位促进根瘤菌的结瘤能力[18]。当土壤中氮素供应不足时,大多数植物会与外生菌根真菌形成共生体,通过菌丝吸收土壤氮,将其运入植物根部并获得植物糖作为回报[19]。此外,土壤中非共生固氮菌群在氮素循环过程中也起着重要作用,如广泛生活在禾本科植物根际的固氮螺菌属能在微氧条件下固定N2,这些微生物合成的激素影响宿主根系代谢和增殖,从而提高植物对养分的吸收和利用[20]。土壤中的氮以有机氮、铵态氮和硝态氮等不同形式存在,其中铵态氮可以通过硝化作用迅速转变为硝态氮,而土壤中的硝态氮易淋溶或通过反硝化作用损失。为了减少氮素流失,一些植物根系分泌物可以通过抑制氨氧化微生物活性从而抑制土壤硝化过程,提高土壤氮素利用率。如高粱根际释放的生物硝化抑制剂樱花素(Sakuranetin)和高粱酮(Sorgoleone)可以抑制硝化酶的活性,对羟基苯丙酸甲酯(MHPP)抑制氨氧化微生物的潜力[21]。
1.3 磷循环相关的根际微生物磷是植物生长的关键营养元素之一,尽管大多数土壤中总磷含量并不一定低,但植物根部只能吸收可溶性磷,而其他形态的磷如有机磷和矿物磷等并不能被植物直接吸收利用。AMF是辅助植物获取磷元素的主要微生物之一,AMF可以在植物的根系上延伸,通过拓宽植物获取磷的范围以及产生磷酸酶等方式促进植物利用土壤中的磷[22]。AMF还可以利用菌丝渗出物提供的C源吸引土壤中的磷酸盐溶解细菌(PSB),这些细菌富集到AMF菌丝表面,使磷酸酶的活性增强[23]。与没有AMF群落共生的植物相比,接种AMF菌根群落的植物平均多吸收44%的磷[24]。Philippot等(2013)的研究证明,超过80%的维管植物与AMF共生,宿主植物通过为AMF提供C源以满足其生长发育过程中对磷的需求[25],但土壤中磷含量较高时,植物可以在没有AMF共生的条件下获得最佳的养分供应[26]。此外,土壤中的一些微生物,如芽孢杆菌(Bacillus)、假单胞菌等还可以通过产生有机酸或磷酸酶将矿物质磷溶解为植物能够吸收利用的正磷酸盐。
2 根际微生物群落的研究方法 2.1 根箱法研究根际微生物将与植物相连的细根(直径小于2 mm)导入装有本地土壤或非本地土壤的聚乙烯根箱中,培养后进行破坏性取样。Ding等(2019)利用根箱法研究落叶松和樟子松的根际效应,发现土壤不同指标的根际效应间存在强烈的正相关关系,如H+浓度的根际效应和土壤有机碳、总氮和微生物生物量等存在显著的正相关关系[27]。该方法将加入活根的根箱中所有土壤定义为根际土壤,不加活根的对照箱中的所有土壤则为非根际土壤,但实际上并非所有根箱里的土壤都受到活根的显著影响,从而低估了植物根系对土壤的影响[28]。此外,植物生长从土壤中吸取水分,而根箱中根际土壤含水量降低,进而抑制微生物活动,从而影响测定的根际效应[29]。在研究土壤碳矿化速率的过程中,利用根箱法可以在不破坏完整土壤和根系时测定土壤呼吸产生的CO2。根箱法还可以与同位素示踪技术联用,从而非破坏性测定根际激发效应[30]。
2.2 根际微生物的同位素技术应用稳定同位素探测技术利用13C、15N、18O标记的底物,使利用这些底物的微生物的DNA、磷脂被标记,从而分析植物和微生物之间的相互作用。如在长期施肥对植物-真菌相互作用的研究中,通过DNA稳定同位素探测技术(DNA-SIP)追踪从植物到根际土壤的13C运动,并利用13C标记的DNA的高通量测序来表征真菌群落,最终发现长期施氮能够改变小麦13C标记的根际真菌群落组成,但不会改变其多样性[31]。为了研究秸秆还田对消耗玉米根系分泌物的微生物的影响,采用13CO2脉冲标记和磷脂脂肪酸稳定同位素探针技术(PLFA-SIP),发现添加秸秆后玉米根际微生物对根系分泌物的同化作用降低[32]。
2.3 根际微生物的高通量测序由于土壤微生物的多样性以及根际土壤自身的复杂性,传统的微生物分析方法无法对大多数微生物进行深入研究。高通量测序技术的发展为研究土壤微生物群落提供了强有力的支撑,从而使我们可以直接研究根际微生物群落的结构和功能,而无需分离培养菌株。Li等(2014)利用16S rRNA基因的焦磷酸测序法对不同生长阶段玉米根际细菌进行动态检测,发现Massila属在苗期是优势菌群,推测该菌为r-strategist,能够快速利用根系分泌的氨基酸等小分子有机物[33]。在番茄根际微生物对青枯菌(Ralstonia)的抗性作用研究中,通过16S rRNA基因测序技术检测番茄感病品种和抗病品种的根际微生物群落结构,发现这两个品种根际微生物群落结构存在明显差异,黄杆菌在抗病种根系的相对丰度远高于感病种。但在后续的培养实验中,得到的黄杆菌分离株对青枯菌并没有抗性作用。进一步通过宏基因组技术分析黄杆菌单菌基因组,最终拼接组装得到黄杆菌TRG1,发现它能缓解植株的疾病症状,提高番茄抗青枯病的能力[34]。为了研究干旱对高粱根际微生物群落成的影响,Xu等(2018)通过16S扩增子和宏转录组测序等研究,发现干旱不仅会延迟高粱早期根系微生物群落的发展,导致革兰氏阳性菌的丰度和活性增加;还对根际相关微生物群落的转录活性也具有显著影响,使碳水化合物和氨基酸代谢和转运相关的基因在干旱下表达增加[35]。
2.4 根际微生物的定量研究方法利用高通量测序技术可以检测植物根际微生物的相对丰度,但无法评估微生物相对于宿主的总量。因此,也就无法评估根际某些微生物的富集是由于其绝对丰度的增加还是由于其他优势类群丰度的减少造成的。为了打破这一局限,建立了可以确定微生物总量和个体微生物相对于植物宿主丰度的宿主相关定量分析方法(host-associated quantitative abundance profiling,HA-QAP)[36]。这种研究方法基于人工合成的spile-in质粒,对样本中细菌和真菌reads数量标准化,计算土壤样品中细菌的绝对丰度。如在干旱胁迫下水稻植株和根腐病小麦根际微生物研究中,检测了微生物相对于宿主植物的总量,而且在不影响微生物相对丰度的前提下,研究了个体微生物相对于植物宿主的定殖量,从而反映了根际微生物群落相对于宿主的变化及微生物间互作关系,发现根系微生物总量增加是根腐病患病小麦以及处于干旱胁迫的水稻的一个关键特征[37]。此外,还可以利用表型微阵列技术(phenotype microarray technology)准确地确定简单群落中每个物种的绝对丰度。为了研究植物根际微生物之间的相互作用,Niu等(2017)构建了一个由7种细菌组成的极简并具有代表性的合成细菌群落来研究微生物之间的相互作用,使用表型微阵列技术通过选择培养的方法跟踪每种细菌的丰度,发现阴沟肠杆菌(Enterobacter cloacae,E. cloacae)在这个简化的微生物群落组装中至关重要。在没有E. cloacae的情况下极小短小杆菌(Curtobacterium pusillum,C. pusillum)物种丰度增加,而其他物种则在群落中消失,这表明E. cloacae是这7个物种构成的简化微生物组中的核心微生物,证明了微生物之间相互作用的重要性和微生物组中关键物种的存在[38]。
2.5 根际微生物的高通量分离培养高通量分离培养技术解决了土壤和植物微生物群落研究中无法系统地分离培养微生物群落的局限,获取根际土壤中的大量微生物菌株。这项技术主要使用Trypic Soy Broth Medium (TSB Medium)、R2A medium等相关培养基以及限制稀释方法,并辅以细胞分选和菌落挑选使菌株生长在96孔板上,然后基于两步条码法系统鉴定分离菌株[39]。如在人工重组拟南芥根际微生物群落的实验中,分离了根际土壤中64%的菌株,突破了传统的只有不到1%的微生物可以被分离培养的认知,大大提高了微生物的鉴定效率。在水稻根际微生物群落成与氮肥利用效率关系的研究中,利用改进后的微生物高通量分离培养技术,分离培养了水稻根系70%的细菌种类,为根际微生物群落与水稻互作及功能的研究奠定了基础[40]。通过高通量分离培养技术获得根际微生物群落中的菌群,然后重新构建植物根际微生物群落,推动了根际微生物群落由描述性研究向功能性研究的转变[41]。
2.6 根际微生物-植物互作机制的析因方法根际微生物与植物相互作用,影响植物的生长发育。随着基因组学、代谢组学的发展,人们研究了植物的生理指标和根系分泌物等对根际微生物群落的影响,但此类研究多为描述性研究,不能揭示根际微生物群落与植物之间相互作用的功能和机理。人工构建合成群落通过降低微生物群落的复杂性,可以让我们深入研究植物对微生物群落的调控作用以及微生物群落对植物生长发育的影响。在研究籼稻和粳稻根际微生物群落时发现,籼稻根际比粳稻根际富集了更多参与氮循环的微生物类群,这些微生物分解有机氮能力较强,使得籼稻利用有机氮的效率提高;进一步研究发现,这种差异与水稻氮转运和感知基因NRT1.1B在粳稻和籼稻之间的自然变异相关。为了研究籼稻和粳稻根际富集细菌与氮代谢的关系,Zhang等(2019)通过从籼稻和粳稻根际分离纯化的菌株进行人工构建合成群落,发现在补充有机氮源时籼稻富集菌对水稻生长促进作用更明显[40]。在研究拟南芥三萜化合物对根际微生物群落的调控作用中,通过解析拟南芥三萜基因簇的代谢通路,阐明了其根系特异表达的代谢网络,并利用体外培养实验,测试了纯化的拟南芥根系三萜化合物和具有代表性的三萜化合物对从拟南芥根系土壤中分离出的19株细菌菌株活性的影响,结果证明,三萜类化合物调控拟南芥特异性微生物群落的组成,使其能够根据自身需要来塑造和调整根内和根周围的微生物群落[42]。
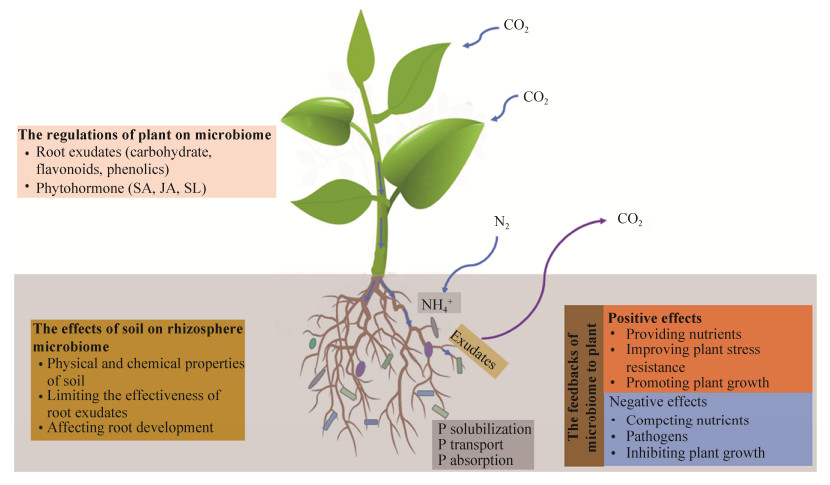
3 根际微生物群落的影响因素植物根际微生物的群落构建受多种生物及非生物因素的影响。不同地区土壤的理化性质宏观地影响了该地区土壤中的微生物类群,而植物的种类和生长阶段等因素决定了哪些微生物能够在根际富集[43]。植物根系通过改变根际环境、分泌代谢产物和激素等,在根际创造独特的生态位,逐渐改变土壤微生物群落。在研究土壤类型和植物种类对荧光假单胞菌(Pseudomonas fluorescens)多样性的影响时发现,土壤类型对荧光假单胞菌多样性的影响更显著[44]。而对生长在不同土壤类型中的小麦和三叶草根际微生物群落的研究表明,植物种类对根际微生物群落有重要影响,不同土壤类型对根际微生物群落无显著影响,这表明在决定根际土壤微生物群落结构的众多因素中,生物或非生物因素都发挥了重要的作用,具体哪种因素起决定性作用因土壤类型和植物种类而异[45](图 1),哪种因素的环境筛选作用(environmental filtering)越强,对微生物群落构建的相对贡献可能会越大。
|
| 图 1 根际微生物、植物和土壤间的相互作用 Figure 1 Interactions among rhizosphere microorganisms, plants and soil. SA (salicylic acid), JA (jasmonic acid), SL (strigolactones). |
3.1 根系分泌物对根际微生物的影响
根系分泌物是植物通过根系的不同部位释放到根际土壤中复杂有机物质的总称,这些物质通过细胞运输并分泌到根际周围,为植物根系微生物创造了独特的生存环境[46]。植物通过根系分泌物调控根际环境来促进根际微生物的生长,从而提高植物在特定生态环境中的适应性(表 1)。Li等(2014)对玉米根际微生物的代谢能力研究中发现,玉米根际土壤中含有相对丰度较高的与C、N循环相关的微生物菌群,参与分解有机酸、糖、氨基酸、纤维素和芳香族化合物等,说明这些物质是影响根际微生物群落形成的驱动力[47]。值得注意的是,植物根系分泌物的组成不是均匀或静态的,而是取决于植物种类、发育阶段、根系性状和土壤类型等因素。
| Root exudates | Effects on microbial communities | Influence Mechanism | References |
| Carbohydrates and amino acids | Stimulate positive chemotaxis of bacteria | Provide effective carbon and nitrogen sources for microorganisms in rhizosphere | [61, 15, 49] |
| Methionine, glycine | Positive chemotactic effect on Enterobacter | Different concentrations of methionine with positive chemotaxis have no effects on the growth dynamics, phenolase activity and phenolase efficiency of bacteria. However, lysine with negative chemotaxis delays bacterial growth at high concentration and inhibits the activity of phenolase. With the decrease of lysine concentration, various inhibitory effects are weakened accordingly | [62-63] |
| Lysine, cystine | Negative chemotactic effect on Enterobacter | ||
| Citric acid | Enrichment of fluorescent Pseudomonas | Low molecular weight organic acid as carbon source | [64, 49, 24] |
| Benzoic acid, coumarin | Stimulate spore germination, hypha branching and root colonization | Phenols are necessary carbon and nitrogen sources for some microorganisms | [65-66] |
| P-hydroxybenzoic acid | Inhibits the growth of bacteria and actinomycetes in soil | Phenol hydroxyl has toxic effects on bacteria and actinomycetes in soil | [64] |
| Vanillic acid, ferulic acid | Promote fungal growth at low concentrations; inhibit fungal growth at high concentrations | Affect the developmental conditions of microbial communities and produce selective stimulation or inhibition | [57, 67-68] |
| Strigolactone | Induces arbuscular mycorrhizal fungi branching | Induces the branching of fungal hyphae, changes the physiological status of fungi and activates fungal mitochondria | [36, 42, 69] |
| Salicylic acid | Mainly inhibits Actinobacteria and enrich Proteobacteria | Immune signal or carbon source | [43-44, 70] |
| Jasmonic acid | Inhibits bacteria that promote plant growth, such as Bacillus, Pseudomonas, and enriches bacteria for biological control | Changes the composition of carbon-containing compounds released from plant roots | [45, 71] |
3.1.1 碳水化合物和氨基酸对根际微生物的影响: 碳水化合物和氨基酸类物质为根际土壤中的微生物提供有效的C源和N源。通过研究从植物根际分离的菌株对根系分泌物的利用情况,发现这些根际分离的菌株主要利用氨基酸、糖类物质和核苷酸。根系分泌的碳水化合物和氨基酸等物质会刺激根表面植物根际促生菌(PGPR)的趋化性,在种类和数量上影响着土壤微生物种群在根际的重新组合(assembly)。Chaparro (2013)和Zhalnina (2018)的研究表明,在拟南芥(Arabidopsis thaliana)生长发育过程中,糖和糖醇的分泌水平在早期较高,吸引了大量不同种类的微生物在根际定殖;而随着植物年龄的增长,氨基酸的分泌水平增加,为根际土壤中能增强植物先天免疫的有益微生物群落提供了底物[48-49]。通过对不同植物抗病品种和感病品种的根系分泌物比较发现,根系分泌物中氨基酸可能作为营养源,通过改变根际微生物群落组成进而抑制或刺激病原菌的生长[50]。也就是说,糖类物质作为一种简单的碳源可能不具备对特定微生物的选择性,而氨基酸类物质是一些特定微生物所偏好的碳源,能够选择某些特定的微生物进行定殖或富集。
3.1.2 黄酮类化合物对根际微生物的影响: 黄酮类化合物是豆科植物根系分泌物中的常见成分,而且构成了根系分泌物的很大一部分[51],被认为是许多植物-微生物相互作用中的关键信号分子。首先,类黄酮类物质可以作为碳源被部分微生物降解,从而对根际土壤中微生物群落结构产生影响;其次,黄酮类化合物还可以作为化学诱导剂吸引根瘤菌,提高根瘤菌中nod基因的表达,促进结瘤的过程[52-53],如木犀草素和芹菜素,已被证明能在根瘤菌中引起强烈的趋化反应;然而,为了维持nod基因相对丰度在最佳水平,并防止植物引起的防御反应,一些黄酮类化合物表现出对某些根瘤菌基因的抑制,如异黄酮类(香豆素和苜蓿素)和香豆雌酚已被证明可以抑制苜蓿根瘤菌(Rhizobium meliloti)中的nod基因[54]。在磷胁迫环境下诱导产生的异黄酮类还能刺激菌根真菌孢子的萌发、土壤中菌丝分枝和根系定殖[55]。此外,黄酮类化合物中的抗菌毒素,如异黄酮是豆科植物中一类主要的植物抗菌毒素,能破坏病原菌细胞膜上的ATP酶和线粒体呼吸电子传递体[56]。
3.1.3 酚酸类物质对根际微生物的影响: 从根系分泌物中发现的酚酸类化合物,如阿魏酸、苯甲酸、肉桂酸和皂角苷,都对根际土壤微生物生长起着调控作用。这些酚酸类化合物大部分是三羧酸循环的中间体,对根际微环境和根际微生物的活力影响很大。具体而言,植物根系分泌物中的酚酸类物质通过改变土壤养分、pH值及化感作用等理化性质,显著影响根际土壤微生物的生物量、多样性和群落结构,选择性地增强根际土壤中特殊的微生物种类[57]。如花生根系分泌物中的苯甲酸增加了根际土壤中伯克霍氏菌(Burkholderia spp.)的相对丰度[58]。西瓜根系分泌物中的阿魏酸可以促进尖孢镰刀菌(Fusarium oxysporum)孢子的形成和萌发[59]。此外,酚酸类物质还能抑制微生物产生挥发性脂肪酸等物质,并减少微生物对生长介质的消耗,而自由态的酚酸类物质进入根际土壤后,可能被根际微生物当作碳源和能量利用,比如在C限制条件下酚酸可以为固氮菌提供碳源[60]。
3.1.4 信号物质对根际微生物的影响: 植物细胞在特定环境信号的诱导下产生植物激素,不仅能够调控植物的生长发育与分化,还能对植物根际的微生物群落产生影响。当土壤中的养分受到限制时,植物根部合成并释放独脚金内酯,吸引土壤中的AMF并与植物建立共生关系[72-73],为根际土壤提供磷素。此外,植物防御性激素无论是作为生长信号还是作为碳源都可能影响根际微生物的群落结构。当植物受到病原体侵染后,植物根部释放的激素及依赖植物激素的次级代谢产物,在调节植物免疫系统和根际微生物群落间的关系中发挥了重要作用。如水杨酸信号的改变导致植物根系微生物群落中放线菌等特定细菌群落的相对丰度改变;当水杨酸缺失时,植物根部核心细菌群落发生显著改变[71]。植物茉莉酸信号通路的激活显著影响根际细菌群落的组成,主要起生物抑制作用的细菌,如产生杀虫剂毒素和抗菌素的细菌得到富集[62-74]。由此可以看出,植物的免疫系统可能通过植物激素及其信号传导直接影响着根际微生物群落的组成。
3.2 土壤物理性质对根际微生物的影响植物根系的生长和吸水能力直接影响根际土壤的物理性质。植物根系生长产生的压力会改变根际土壤孔隙度、土壤强度等物理性质进而影响根际微生物的群落组成。田间实验证明,土壤强度的提高增加了小麦根际细菌的数量,土壤结构的变化也是影响根际根霉菌(Rhizoctonia solani)的重要因素[75]。植物通过根际界面吸收水分还可以对土壤湿度产生影响,土壤湿度对植物根际土壤气体和液体扩散速率起主导作用,并影响氧气和营养基质的有效性[76]。土壤湿度的增加会降低气体扩散速率,通过限制氧等主要电子受体的供应,直接影响微生物的生理状态和活动;增加土壤湿度同时也会增加液体扩散速度,为微生物提供可溶性有机碳等关键营养基质[77]。
3.3 土壤化学性质对根际微生物的影响土壤pH值是改变细菌多样性和群落组成的一个主要原因,而环境pH值偏离中性不利于微生物的生存,这可能与微生物本身对土壤pH值的适应范围较窄有关。植物根系释放的有机酸、二羧酸、二氧化碳等使植物根际土壤酸化至少2个pH单位[78]。土壤酸化后会降低微生物生物量和微生物活性。如鹰嘴豆根际土壤pH值从5降至4.3,微生物活性被抑制[79]。根系分泌物二羧酸通过酸化土壤增强了土壤中正磷酸盐和微量营养元素的可用性,导致β-变形菌和放线菌的相对丰度增加[14]。与非根际土壤相比,植物根系向根际土壤释放了更多的含碳化合物和其他不易分解的根沉积物,而土壤中的微生物大多处于“寡营养”状态,由于微生物对根分泌物(主要是糖、氨基酸和有机酸)的趋化性,微生物被吸引到根际。Blagodatskaya等(2014)的研究证明根际土壤中微生物总生物量比非根际土壤中微生物总生物量高14%–31%[80]。由于质子和其他离子在植物根部的进出,植物的根也会产生外部电流或电场[81]。研究证明,植物根部任何根区的伤口都表现出大量的正电离子向内流动的现象,造成伤口部位的局部阴性[82]。通过检测大麦根伤口的感应电场,发现电流在30 min内迅速减小至不可检测的水平,感应电场的大小并不稳定,甚至还可能出现极性的反转[83-84]。与此对应的是,根部受伤后,立即有大量的真菌游动孢子向伤口移动,并在30 min内,孢子向伤口游动的百分比下降到初始值的52%[83]。已经证实在离根面(< 500 μm)相对较短的距离内,电化学梯度能将游动孢子吸引至特定区域[83],但尚不清楚土壤微生物对化学信号或电信号的感知能否影响其成为病原体或共生体。此外,植物还可以通过调节基因转录和释放特定的根系分泌物来影响根际微生物群落。如植物根系分泌物中各种水解酶在碳、氮、磷、硫等元素循环中发挥了重要作用,通过改变土壤养分浓度影响根际微生物群落组成[85]。
4 根际微生物-植物互作机制植物根际微生物群落的组成是由非生物环境因素及其生物群落之间的复杂的相互作用决定的。土壤复杂的理化特性影响植物的生长发育,进而影响根际微生物群落的组成。此外,非根际土壤微生物群落通过影响植物根际微生物群落组成间接影响了植物与其根际微生物群落互作。自然环境中,根际的多个物种界间相互关系的复杂性使阐明植物-微生物的互作机制具有挑战性。
4.1 土壤通过影响植物间接影响根际微生物土壤复杂的理化特性还可以影响植物的生长发育,进而影响根际微生物群落的组成。例如,植物根系在土壤中的生长状态,除了与根系的特性有关,还常受到土壤环境条件的影响[75, 86]。土壤的理化性质,如水分、养分的有效性还可以调节植物的光合速率和生长,进而通过诱导根系分泌物模式的变化来调节根际微生物群落的组成。在干旱条件下高粱根系富集放线菌,Xu等(2018)进一步研究发现,高粱根系在干旱条件下会产生过量的分泌物G3P,相应的根际土壤中放线菌与G3P相关的ABC转运蛋白及其代谢途径活性增强,也就是说,放线菌能够利用植物根系分泌的G3P从而使其在根际富集[35]。当土壤中磷含量过低时,豆科植物会通过分泌异黄酮类物质刺激菌根真菌孢子的萌发[12]。
4.2 土壤微生物之间的相互作用对根际微生物群落的影响非根际土壤中存在大量具有根际适应能力的微生物菌群,对根际微生物群落结构产生影响。通过研究不同土壤类型中生长的沙苔草根际和非根际微生物群落组成,发现沙苔草根际细菌群落结构在很大程度上是由非根际土壤的细菌群落组成决定的,并没有发现沙苔草会优先选择某些有害或有益的微生物群落[87]。也就是说,非根际土壤中微生物群落组成在根际微生物群落的构建中发挥了重要作用。相比于非根际土壤,根际微域的微生物细胞密度更高,根际微生物之间的相互作用更复杂,并且根际微生物之间的相互作用大多是相互促进的,表明根际微生物有更大的互利共生潜力[8]。如在营养受限制的条件下,根瘤菌和AMF与豆科植物相互作用,通过提供不同的养分促进植物生长,间接影响了豆科植物根际微生物的群落组成[88]。
5 根际微生物的应用研究进展植物根际微生物既包含促进植物生长的有益微生物,又包含抑制植物生长的有害微生物。有益的根际微生物帮助植物获取养分,提高植物抗逆性。有害的根际微生物与植物竞争土壤中的养分或者通过根感染植物,抑制植物的健康生长。
5.1 根际微生物对植物的促生作用植物根际土壤中存在的根际促生菌可以通过两种不同的方式来促进植物的生长发育。其一,根际微生物可以通过分泌植物生长激素、脱落酸等直接刺激植物生长,如节杆菌(Arthrobacter)和荧光假单胞菌等通过分泌植物激素(如细胞分裂素)来促进植物根系发育,从而提高植物吸收营养物质和水分的能力[89]。根际微生物的代谢物质还可以影响植物信号的产生和代谢过程,从而改变根系分泌物的成分并诱导释放更有利的分泌物,导致在植物根系富集植物促生菌[90]。例如,豆科植物根际的黄杆菌属(Flavobacterium)通过影响黄酮类化合物的渗出来增强固氮菌的固氮能力[91]。拟南芥在受到病原菌Pseudomonas syringae感染后,根系会分泌大量的苹果酸,并通过苹果酸招募枯草芽孢杆菌FB17(Bacillus subtilis FB17),从而激活植物免疫系统[50]。其二,根际微生物可以促进环境营养元素如氮、磷、铁的摄取。例如,根瘤菌通过与豆科植物建立共生关系将空气中的氮转化为植物能够吸收利用的有机氮;土壤中AMF还可以通过促进根瘤的生长间接提高生物固氮的能力;此外菌根真菌还能直接活化土壤中植物不能主动吸收的氮、磷元素[92]。总之,根际促生菌可以通过调节植物激素、改善营养获取等多种机制与植物相互作用并影响植物生长。
5.2 根际微生物提高植物的抗逆性植物生长过程中受到多种生物或非生物因素的胁迫,其中盐胁迫是植物生长过程中最常见的非生物胁迫因子之一,对植物的光合作用和代谢功能等各方面都有不利影响[93]。根际促生菌可通过各种直接和间接机制促进盐胁迫下的植物生长。如枯草芽孢杆菌通过产生挥发性有机化合物下调拟南芥幼苗根系高亲和性钾离子转运蛋白(HKT1)的表达,限制钠离子进入根系[94]。拟南芥和小麦等植物的根际促生菌可以产生主动结合根系周围钠离子的胞外多糖来减少植物中钠离子的含量,或者诱导植物产生脯氨酸以及抗ROS酶来减轻盐分对植物的胁迫作用[95-96]。此外,根际促生菌在盐胁迫下形成的生物膜可以有效缓解土壤盐分过高对植物产生的影响[97]。
除了非生物因素的影响,自然界的植物在生长过程中会受到各种病原菌的入侵。特异性抑病型土壤可以帮助植物抵御病原菌的入侵,如小麦全蚀病[98]、马铃薯赤霉病[99]和番茄青枯病[34]等相应的特异性抑病型土壤被陆续发现。通过进一步的研究发现,抑病型土壤的抑病能力与土壤中特定有益微生物的富集有关,富集的有益微生物可以直接产生抑制病原菌的抗菌化合物或者激活植物的免疫系统间接地协助植物抵御病原菌。如枯草芽孢杆菌通过分泌激素类物质、多种酶和抗菌素类物质促进植物生长,增强抗病性[50]。
5.3 根际微生物对植物的抑制作用植物根际土壤中也包含土著的病原微生物,它们可以引起植物病害或者产生有毒物质,从而对植物生长产生不利影响。Li等(2014)发现在花生根系分泌物的介导下,连作花生根际土壤伯克氏菌属(Burkholderia spp.)及潜在的病原性镰刀菌属(Fusarium)增加,而有益菌群丰度降低,从而导致花生连作障碍[64]。除此之外,病毒还可能以真菌等作为载体进入植物根部组织,并通过植物的根部感染植物[100]。根际微生物除了直接对植物生长产生消极影响外,还可以与植物竞争土壤溶液中的养分,间接地影响植物的生长。在营养极度贫乏的土壤环境中,根际微生物与植物对N的竞争尤为明显[101]。通过同位素标记法分别检测植物及其根际微生物对N的捕获能力,发现提高根际微生物的生物量会增加微生物对氮元素的捕获,而减少了植物对氮元素的吸收[102]。此外,一些微生物还能将土壤中的氮转化为可溶性的形式,如硝化细菌产生的硝态氮,很容易进入地下水或地表径流而损失,从而降低了植物对营养物质的可获得性,间接降低了植物的生产力[103]。
6 展望植物根际微生物在植物生长发育过程中发挥了重要作用。植物根系和土壤是影响微生物群落的两个主要因素,但目前的研究往往基于相对简化的生态体系,而对大尺度、复杂生态体系根际微生物的了解还不全面。此外,在自然环境中植物根际常常存在微生物间跨界相互作用的复杂关系,为了充分了解植物根际微生物群落,可以采用合成群落的方法研究植物宿主相关微生物类群之间的关系。虽然大多数土壤微生物仍然未被分离和详细描述,但是可以根据生物标记物基因的序列对其进行分类,并利用基因组信息注释菌种的功能属性。但是,这需要更多土壤菌种的基因组信息和更加高效准确的生物信息学方法,目前三代测序技术的发展可以加快这一研究的进程。
植物与微生物之间的相互作用机制复杂,还有很多问题不清楚。理论上讲,植物分泌的每一种特有化合物,或者植物对根际微环境的影响,都应该会引起特定菌群的响应,反之亦然,每种特定菌群的变化,可能都会反馈影响植物。需要利用合成生物学方法、微生物高通量分离技术等,从分子、生理、生态水平上研究植物-微生物之间的互作机制。例如,通过研究根瘤菌-豆科植物的共生模式,探索豆科植物中根瘤菌产生化学信号的天然合成路径,再将合成化学信号的传导途径转基因到其他植物上,使植物能够控制其根际微生物,从而为植物生长提供氮源。从农业发展的角度考虑,选育那些具有高效养分转化及抗病抗逆性强的根际微生物群落的品种,可以减少农药化肥的使用,有利于生态农业的发展。水生植物根际微生物群落是甲烷产生的重要来源,筛选具有甲烷释放量低的微生物群落的水稻品种,大面积种植这样的品种有利于减少甲烷释放,缓解温室效应。除了微生物,植物根际也存在其他生物,如原生生物、无脊椎动物等,这些生物及其与根际微生物的相互作用,与植物健康和营养的关系,也是值得研究的重要课题。在农业生产中,如何通过农业生产措施,有效维持植物健康的根际微生物群落,从而提高植物抗病、抗逆性、养分利用效率等,是未来需要大力研究的方向。
| [1] | Serna-Chavez HM, Fierer N, van Bodegom PM. Global drivers and patterns of microbial abundance in soil. Global Ecology and Biogeography, 2013, 22(10): 1162-1172. DOI:10.1111/geb.12070 |
| [2] | Hartmann A, Rothballer M, Schmid M. Lorenz hiltner, a pioneer in rhizosphere microbial ecology and soil bacteriology research. Plant and Soil, 2008, 312(1/2): 7-14. |
| [3] | Hinsinger P, Gobran GR, Gregory PJ, Wenzel WW. Rhizosphere geometry and heterogeneity arising from root-mediated physical and chemical processes. New Phytologist, 2005, 168(2): 293-303. DOI:10.1111/j.1469-8137.2005.01512.x |
| [4] | van Dam NM, Bouwmeester HJ. Metabolomics in the rhizosphere:tapping into belowground chemical communication. Trends in Plant Science, 2016, 21(3): 256-265. DOI:10.1016/j.tplants.2016.01.008 |
| [5] | Read DB, Bengough AG, Gregory PJ, Crawford JW, Robinson D, Scrimgeour CM, Young IM, Zhang K, Zhang XX. Plant roots release phospholipid surfactants that modify the physical and chemical properties of soil. New Phytologist, 2003, 157(2): 315-326. DOI:10.1046/j.1469-8137.2003.00665.x |
| [6] | Bulgarelli D, Garrido-Oter R, Müench PC, Weiman A, Dröge J, Pan Y, McHardy AC, Schulze-Lefert P. Structure and function of the bacterial root microbiota in wild and domesticated barley. Cell Host & Microbe, 2015, 17(3): 392-403. |
| [7] | Tian P, Razavi BS, Zhang XC, Wang QK, Blagodatskaya E. Microbial growth and enzyme kinetics in rhizosphere hotspots are modulated by soil organics and nutrient availability. Soil Biology and Biochemistry, 2020, 141: 107662. DOI:10.1016/j.soilbio.2019.107662 |
| [8] | Zhang RF, Vivanco JM, Shen QR. The unseen rhizosphere root-soil-microbe interactions for crop production. Current Opinion in Microbiology, 2017, 37: 8-14. DOI:10.1016/j.mib.2017.03.008 |
| [9] | Trivedi P, Leach JE, Tringe SG, Sa TM, Singh BK. Plant-microbiome interactions:from community assembly to plant health. Nature Reviews Microbiology, 2020, 18(11): 607-621. DOI:10.1038/s41579-020-0412-1 |
| [10] | Xu J, Zhang YZ, Zhang PF, Trivedi P, Riera N, Wang YY, Liu X, Fan GY, Tang JL, Coletta-Filho HD, Cubero J, Deng XL, Ancona V, Lu ZJ, Zhong BL, Roper MC, Capote N, Catara V, Pietersen G, Vernière C, Al-Sadi AM, Li L, Yang F, Xu X, Wang J, Yang HM, Jin T, Wang N. The structure and function of the global citrus rhizosphere microbiome. Nature Communications, 2018, 9(1): 4894. DOI:10.1038/s41467-018-07343-2 |
| [11] | Gao C, Montoya L, Xu L, Madera M, Hollingsworth J, Purdom E, Singan V, Vogel J, Hutmacher RB, Dahlberg JA, Coleman-Derr D, Lemaux PG, Taylor JW. Fungal community assembly in drought-stressed sorghum shows stochasticity, selection, and universal ecological dynamics. Nature Communications, 2020, 11(1): 34. DOI:10.1038/s41467-019-13913-9 |
| [12] | Hassan S, Mathesius U. The role of flavonoids in root-rhizosphere signalling:opportunities and challenges for improving plant-microbe interactions. Journal of Experimental Botany, 2012, 63(9): 3429-3444. DOI:10.1093/jxb/err430 |
| [13] | Vandenkoornhuyse P, Quaiser A, Duhamel M, Le Van A, Dufresne A. The importance of the microbiome of the plant holobiont. New Phytologist, 2015, 206(4): 1196-1206. DOI:10.1111/nph.13312 |
| [14] | Eilers KG, Lauber CL, Knight R, Fierer N. Shifts in bacterial community structure associated with inputs of low molecular weight carbon compounds to soil. Soil Biology and Biochemistry, 2010, 42(6): 896-903. DOI:10.1016/j.soilbio.2010.02.003 |
| [15] | Sánchez-Cañizares C, Jorrín B, Poole PS, Tkacz A. Understanding the holobiont:the interdependence of plants and their microbiome. Current Opinion in Microbiology, 2017, 38: 188-196. DOI:10.1016/j.mib.2017.07.001 |
| [16] | Kraft B, Strous M, Tegetmeyer HE. Microbial nitrate respiration-Genes, enzymes and environmental distribution. Journal of Biotechnology, 2011, 155(1): 104-117. DOI:10.1016/j.jbiotec.2010.12.025 |
| [17] | Kuypers MMM, Marchant HK, Kartal B. The microbial nitrogen-cycling network. Nature Reviews Microbiology, 2018, 16(5): 263-276. DOI:10.1038/nrmicro.2018.9 |
| [18] | Dardanelli MS, de Córdoba FJF, Espuny MR, Carvajal MAR, Díaz MES, Gil Serrano AM, Okon Y, Megías M. Effect of Azospirillum brasilense coinoculated with rhizobium on Phaseolus vulgaris flavonoids and nod factor production under salt stress. Soil Biology and Biochemistry, 2008, 40(11): 2713-2721. DOI:10.1016/j.soilbio.2008.06.016 |
| [19] | Lambers H, Mougel C, Jaillard B, Hinsinger P. Plant-microbe-soil interactions in the rhizosphere:an evolutionary perspective. Plant and Soil, 2009, 321(1/2): 83-115. |
| [20] | Hayat R, Ali S, Amara U, Khalid R, Ahmed I. Soil beneficial bacteria and their role in plant growth promotion:a review. Annals of Microbiology, 2010, 60(4): 579-598. DOI:10.1007/s13213-010-0117-1 |
| [21] | Subbarao GV, Nakahara K, Ishikawa T, Ono H, Yoshida M, Yoshihashi T, Zhu YY, Zakir HAKM, Deshpande SP, Hash CT, Sahrawat KL. Biological nitrification inhibition (BNI) activity in sorghum and its characterization. Plant and Soil, 2013, 366(1/2): 243-259. |
| [22] | Rakiami A, Bechtaoui N, Tahiri AI, Anli M, Meddich A, Oufdou K. Use of rhizobacteria and mycorrhizae consortium in the open field as a strategy for improving crop nutrition, productivity and soil fertility. Frontiers in Microbiology, 2019, 10: 1106. DOI:10.3389/fmicb.2019.01106 |
| [23] | Zhang L, Xu MG, Liu Y, Zhang FS, Hodge A, Feng G. Carbon and phosphorus exchange may enable cooperation between an arbuscular mycorrhizal fungus and a phosphate-solubilizing bacterium. New Phytologist, 2016, 210(3): 1022-1032. DOI:10.1111/nph.13838 |
| [24] | Pérez-Jaramillo JE, Mendes R, Raaijmakers JM. Impact of plant domestication on rhizosphere microbiome assembly and functions. Plant Molecular Biology, 2016, 90(6): 635-644. DOI:10.1007/s11103-015-0337-7 |
| [25] | Philippot L, Raaijmakers JM, Lemanceau P, van der Putten WH. Going back to the roots:the microbial ecology of the rhizosphere. Nature Reviews Microbiology, 2013, 11(11): 789-799. DOI:10.1038/nrmicro3109 |
| [26] | Ferrol N, Azcón-Aguilar C, Pérez-Tienda J. Review:arbuscular mycorrhizas as key players in sustainable plant phosphorus acquisition:an overview on the mechanisms involved. Plant Science, 2019, 280: 441-447. DOI:10.1016/j.plantsci.2018.11.011 |
| [27] | Ding ZJ, Tang M, Chen X, Yin LM, Gui HC, Zhu B. Measuring rhizosphere effects of two tree species in a temperate forest:a comprehensive method comparison. Rhizosphere, 2019, 10: 100153. DOI:10.1016/j.rhisph.2019.100153 |
| [28] | Zhu B, Panke-Buisse K, Kao-Kniffin J. Nitrogen fertilization has minimal influence on rhizosphere effects of smooth crabgrass (Digitaria ischaemum) and bermudagrass (Cynodon dactylon). Journal of Plant Ecology, 2015, 8(4): 390-400. DOI:10.1093/jpe/rtu034 |
| [29] | Moyano FE, Manzoni S, Chenu C. Responses of soil heterotrophic respiration to moisture availability:an exploration of processes and models. Soil Biology and Biochemistry, 2013, 59: 72-85. DOI:10.1016/j.soilbio.2013.01.002 |
| [30] | Tang M, Cheng WX, Zeng H, Zhu B. Light intensity controls rhizosphere respiration rate and rhizosphere priming effect of soybean and sunflower. Rhizosphere, 2019, 9: 97-105. DOI:10.1016/j.rhisph.2018.12.002 |
| [31] | Wang QF, Ma MC, Jiang X, Zhou BK, Guan DW, Cao FM, Chen SF, Li J. Long-term N fertilization altered 13C-labeled fungal community composition but not diversity in wheat rhizosphere of Chinese black soil. Soil Biology and Biochemistry, 2019, 135: 117-126. DOI:10.1016/j.soilbio.2019.04.009 |
| [32] | Maarastawi SA, Frindte K, Bodelier PLE, Knief C. Rice straw serves as additional carbon source for rhizosphere microorganisms and reduces root exudate consumption. Soil Biology and Biochemistry, 2019, 135: 235-238. DOI:10.1016/j.soilbio.2019.05.007 |
| [33] | Li XZ, Rui JP, Mao YJ, Yannarell A, Mackie R. Dynamics of the bacterial community structure in the rhizosphere of a maize cultivar. Soil Biology and Biochemistry, 2014, 68: 392-401. DOI:10.1016/j.soilbio.2013.10.017 |
| [34] | Kwak MJ, Kong HG, Choi K, Kwon SK, Song JY, Lee J, Lee PA, Choi SY, Seo M, Lee HJ, Jung EJ, Park H, Roy N, Kim H, Lee MM, Rubin EM, Lee SW, Kim JF. Rhizosphere microbiome structure alters to enable wilt resistance in tomato. Nature Biotechnology, 2018, 36(11): 1100-1109. DOI:10.1038/nbt.4232 |
| [35] | Xu L, Naylor D, Dong ZB, Simmons T, Pierroz G, Hixson KK, Kim YM, Zink EM, Engbrecht KM, Wang Y, Gao C, DeGraaf S, Madera MA, Sievert JA, Hollingsworth J, Birdseye D, Scheller HV, Hutmacher R, Dahlberg J, Jansson C, Taylor JW, Lemaux PG, Coleman-Derr D. Drought delays development of the sorghum root microbiome and enriches for monoderm bacteria. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(18): E4284-E4293. DOI:10.1073/pnas.1717308115 |
| [36] | Tkacz A, Hortala M, Poole PS. Absolute quantitation of microbiota abundance in environmental samples. Microbiome, 2018, 6(1): 110. DOI:10.1186/s40168-018-0491-7 |
| [37] | Guo XX, Zhang XN, Qin Y, Liu YX, Zhang JY, Zhang N, Wu K, Qu BY, He ZS, Wang X, Zhang XJ, Hacquard S, Fu XD, Bai Y. Host-associated quantitative abundance profiling reveals the microbial load variation of root microbiome. Plant Communications, 2020, 1(1): 100003. DOI:10.1016/j.xplc.2019.100003 |
| [38] | Niu B, Paulson JN, Zheng XQ, Kolter R. Simplified and representative bacterial community of maize roots. Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(12): E2450-E2459. DOI:10.1073/pnas.1616148114 |
| [39] | Liu YX, Qin Y, Bai Y. Reductionist synthetic community approaches in root microbiome research. Current Opinion in Microbiology, 2019, 49: 97-102. DOI:10.1016/j.mib.2019.10.010 |
| [40] | Zhang JY, Liu YX, Zhang N, Hu B, Jin T, Xu HR, Qin Y, Yan PX, Zhang XN, Guo XX, Hui J, Cao SY, Wang X, Wang C, Wang H, Qu BY, Fan GY, Yuan LX, Garrido-Oter R, Chu CC, Bai Y. Nrt1.1B is associated with root microbiota composition and nitrogen use in field-grown rice. Nature Biotechnology, 2019, 37(6): 676-684. DOI:10.1038/s41587-019-0104-4 |
| [41] | Bai Y, Müller DB, Srinivas G, Garrido-Oter R, Potthoff E, Rott M, Dombrowski N, Münch PC, Spaepen S, Remus-Emsermann M, Hüttel B, McHardy AC, Vorholt JA, Schulze-Lefert P. Functional overlap of the Arabidopsis leaf and root microbiota. Nature, 2015, 528(7582): 364-369. DOI:10.1038/nature16192 |
| [42] | Huang AC, Jiang T, Liu YX, Bai YC, Reed J, Qu BY, Goossens A, Nützmann HW, Bai Y, Osbourn A. A specialized metabolic network selectively modulates Arabidopsis root microbiota. Science, 2019, 364(6440): eaau6389. DOI:10.1126/science.aau6389 |
| [43] | Walters WA, Jin Z, Youngblut N, Wallace JG, Sutter J, Zhang W, González-Peña A, Peiffer J, Koren O, Shi QJ, Knight R, del Rio TG, Tringe SG, Buckler ES, Dangl JL, Ley RE. Large-scale replicated field study of maize rhizosphere identifies heritable microbes. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(28): 7368-7373. DOI:10.1073/pnas.1800918115 |
| [44] | Gaiero JR, McCall CA, Thompson KA, Day NJ, Best AS, Dunfield KE. Inside the root microbiome:bacterial root endophytes and plant growth promotion. American Journal of Botany, 2013, 100(9): 1738-1750. DOI:10.3732/ajb.1200572 |
| [45] | Chaparro JM, Badri DV, Vivanco JM. Rhizosphere microbiome assemblage is affected by plant development. The ISME Journal, 2014, 8(4): 790-803. DOI:10.1038/ismej.2013.196 |
| [46] | Hu LF, Robert CAM, Cadot S, Zhang X, Ye M, Li BB, Manzo D, Chervet N, Steinger T, van der Heijden MGA, Schlaeppi K, Erb M. Root exudate metabolites drive plant-soil feedbacks on growth and defense by shaping the rhizosphere microbiota. Nature Communications, 2018, 9(1): 2738. DOI:10.1038/s41467-018-05122-7 |
| [47] | Li XZ, Rui JP, Xiong JB, Li JB, He ZL, Zhou JZ, Yannarell AC, Mackie RI. Functional potential of soil microbial communities in the maize rhizosphere. PLoS One, 2014, 9(11): e112609. DOI:10.1371/journal.pone.0112609 |
| [48] | Chaparro JM, Badri DV, Bakker MG, Sugiyama A, Manter DK, Vivanco JM. Root exudation of phytochemicals in arabidopsis follows specific patterns that are developmentally programmed and correlate with soil microbial functions. PLoS One, 2013, 8(2): e55731. DOI:10.1371/journal.pone.0055731 |
| [49] | Zhalnina K, Louie KB, Hao Z, Mansoori N, da Rocha UN, Shi SJ, Cho H, Karaoz U, Loqué D, Bowen BP, Firestone MK, Northen TR, Brodie EL. Dynamic root exudate chemistry and microbial substrate preferences drive patterns in rhizosphere microbial community assembly. Nature Microbiology, 2018, 3(4): 470-480. DOI:10.1038/s41564-018-0129-3 |
| [50] | Yuan J, Zhao J, Wen T, Zhao ML, Li R, Goossens P, Huang QW, Bai Y, Vivanco JM, Kowalchuk GA, Berendsen RL, Shen QR. Root exudates drive the soil-borne legacy of aboveground pathogen infection. Microbiome, 2018, 6(1): 156. DOI:10.1186/s40168-018-0537-x |
| [51] | Cesco S, Neumann G, Tomasi N, Pinton R, Weisskopf L. Release of plant-borne flavonoids into the rhizosphere and their role in plant nutrition. Plant and Soil, 2010, 329(1/2): 1-25. |
| [52] | Maj D, Wielbo J, Marek-Kozaczuk M, Skorupska A. Response to flavonoids as a factor influencing competitiveness and symbiotic activity of Rhizobium leguminosarum. Microbiological Research, 2010, 165(1): 50-60. DOI:10.1016/j.micres.2008.06.002 |
| [53] | Moscatiello R, Squartini A, Mariani P, Navazio L. Flavonoid-induced calcium signalling in Rhizobium leguminosarum bv. viciae. New Phytologist, 2010, 188(3): 814-823. DOI:10.1111/j.1469-8137.2010.03411.x |
| [54] | Zuanazzi JAS, Clergeot PH, Quirion JC, Husson HP, Kondorosi A, Ratet P. Production of Sinorhizobium meliloti nod gene activator and repressor flavonoids from Medicago sativa roots. Molecular Plant-Microbe Interactions, 1998, 11(8): 784-794. DOI:10.1094/MPMI.1998.11.8.784 |
| [55] | Kikuchi K, Matsushita N, Suzuki K, Hogetsu T. Flavonoids induce germination of basidiospores of the ectomycorrhizal fungus Suillus bovinus. Mycorrhiza, 2007, 17(7): 563-570. DOI:10.1007/s00572-007-0131-8 |
| [56] | Harrison MJ, Dixon RA. Isoflavonoid accumulation and expression of defense gene transcripts during the establishment of vesicular-arbuscular mycorrhizal associations in roots of Medicago truncatula. Molecular Plant-Microbe Interactions, 1993, 6(5): 643-654. DOI:10.1094/MPMI-6-643 |
| [57] | Badri DV, Chaparro JM, Zhang RF, Shen QR, Vivanco JM. Application of natural blends of phytochemicals derived from the root exudates of arabidopsis to the soil reveal that phenolic-related compounds predominantly modulate the soil microbiome. Journal of Biological Chemistry, 2013, 288(7): 4502-4512. DOI:10.1074/jbc.M112.433300 |
| [58] | Liu JG, Li XG, Jia ZJ, Zhang TL, Wang XX. Effect of benzoic acid on soil microbial communities associated with soilborne peanut diseases. Applied Soil Ecology, 2017, 110: 34-42. DOI:10.1016/j.apsoil.2016.11.001 |
| [59] | Hao WY, Ren LX, Ran W, Shen QR. Allelopathic effects of root exudates from watermelon and rice plants on Fusarium oxysporum f. sp. niveum. Plant and Soil, 2010, 366(1/2): 485-497. |
| [60] | Mandal SM, Chakraborty D, Dey S. Phenolic acids act as signaling molecules in plant-microbe symbioses. Plant Signaling & Behavior, 2010, 5(4): 359-368. |
| [61] | Somers E, Vanderleyden J, Srinivasan M. Rhizosphere bacterial signalling:a love parade beneath our feet. Critical Reviews in Microbiology, 2004, 30(4): 205-240. DOI:10.1080/10408410490468786 |
| [62] | Carvalhais LC, Dennis PG, Badri DV, Kidd BN, Vivanco JM, Schenk PM. Linking jasmonic acid signaling, root exudates, and rhizosphere microbiomes. Molecular Plant-Microbe Interactions, 2015, 28(9): 1049-1058. DOI:10.1094/MPMI-01-15-0016-R |
| [63] | Czaban W, Rasmussen J, Laursen BB, Vidkjær NH, Sapkota R, Nicolaisen M, Fomsgaard IS. Multiple effects of secondary metabolites on amino acid cycling in white clover rhizosphere. Soil Biology and Biochemistry, 2018, 123: 54-63. DOI:10.1016/j.soilbio.2018.04.012 |
| [64] | Li XG, Ding CF, Hua K, Zhang TL, Zhang YN, Zhao L, Yang YR, Liu JG, Wang XX. Soil sickness of peanuts is attributable to modifications in soil microbes induced by peanut root exudates rather than to direct allelopathy. Soil Biology and Biochemistry, 2014, 78: 149-159. DOI:10.1016/j.soilbio.2014.07.019 |
| [65] | Scheffknecht S, Mammerler R, Steinkellner S, Vierheilig H. Root exudates of mycorrhizal tomato plants exhibit a different effect on microconidia germination of Fusarium oxysporum f. sp. lycopersici than root exudates from non-mycorrhizal tomato plants. Mycorrhiza, 2006, 16(5): 365-370. DOI:10.1007/s00572-006-0048-7 |
| [66] | Souto XC, Chiapusio G, Pellissier F. Relationships between phenolics and soil microorganisms in spruce forests:significance for natural regeneration. Journal of Chemical Ecology, 2000, 26(9): 2025-2034. DOI:10.1023/A:1005504029243 |
| [67] | Qu XH, Wang JG. Effect of amendments with different phenolic acids on soil microbial biomass, activity, and community diversity. Applied Soil Ecology, 2008, 39(2): 172-179. DOI:10.1016/j.apsoil.2007.12.007 |
| [68] | Zhou XG, Wu FZ. P-coumaric acid influenced cucumber rhizosphere soil microbial communities and the growth of Fusarium oxysporum f. sp. cucumerinum owen. PLoS One, 2012, 7(10): e48288. DOI:10.1371/journal.pone.0048288 |
| [69] | Rasmann S, Turlings TCJ. Root signals that mediate mutualistic interactions in the rhizosphere. Current Opinion in Plant Biology, 2016, 32: 62-68. DOI:10.1016/j.pbi.2016.06.017 |
| [70] | Lebeis SL, Paredes SH, Lundberg DS, Breakfield N, Gehring J, McDonald M, Malfatti S, del Rio TG, Jones CD, Tringe SG, Dangl JL. Salicylic acid modulates colonization of the root microbiome by specific bacterial taxa. Science, 2015, 349(6250): 860-864. DOI:10.1126/science.aaa8764 |
| [71] | Li XG, Jousset A, de Boer W, Carrión VJ, Zhang TL, Wang XX, Kuramae EE. Legacy of land use history determines reprogramming of plant physiology by soil microbiome. The ISME Journal, 2019, 13(3): 738-751. DOI:10.1038/s41396-018-0300-0 |
| [72] | Bednarek P, Kwon C, Schulze-Lefert P. Not a peripheral issue:secretion in plant-microbe interactions. Current Opinion in Plant Biology, 2010, 13(4): 378-387. DOI:10.1016/j.pbi.2010.05.002 |
| [73] | Akiyama K, Matsuzaki K, Hayashi H. Plant sesquiterpenes induce hyphal branching in arbuscular mycorrhizal fungi. Nature, 2005, 435(7043): 824-827. DOI:10.1038/nature03608 |
| [74] | Carvalhais LC, Dennis PG, Badri DV, Tyson GW, Vivanco JM, Schenk PM. Activation of the jasmonic acid plant defence pathway alters the composition of rhizosphere bacterial communities. PLoS One, 2013, 8(2): e56457. DOI:10.1371/journal.pone.0056457 |
| [75] | Neumann G, Bott S, Ohler MA, Mock HP, Lippmann R, Grosch R, Smalla K. Root exudation and root development of lettuce (Lactuca sativa L. cv. Tizian) as affected by different soils. Frontiers in Microbiology, 2014, 5: 1-6. |
| [76] | Doussan C, Pagès L, Pierret A. Soil exploration and resource acquisition by plant roots:an architectural and modelling point of view. Agronomie, 2003, 23(5/6): 419-431. |
| [77] | Banerjee S, Helgason B, Wang LF, Winsley T, Ferrari BC, Siciliano SD. Legacy effects of soil moisture on microbial community structure and N2O emissions. Soil Biology and Biochemistry, 2016, 95: 40-50. DOI:10.1016/j.soilbio.2015.12.004 |
| [78] | Hacquard S, Garrido-Oter R, González A, Spaepen S, Ackermann G, Lebeis S, McHardy AC, Dangl JL, Knight R, Ley R, Schulze-Lefert P. Microbiota and host nutrition across plant and animal kingdoms. Cell Host & Microbe, 2015, 17(5): 603-616. |
| [79] | Rousk J, Bååth E, Brookes PC, Lauber CL, Lozupone C, Caporaso JG, Knight R, Fierer N. Soil bacterial and fungal communities across a ph gradient in an arable soil. The ISME Journal, 2010, 4(10): 1340-1351. DOI:10.1038/ismej.2010.58 |
| [80] | Blagodatskaya E, Blagodatsky S, Anderson TH, Kuzyakov Y. Microbial growth and carbon use efficiency in the rhizosphere and root-free soil. PLoS One, 2014, 9(4): e93282. DOI:10.1371/journal.pone.0093282 |
| [81] | Živanović BD. Surface tip-to-base Ca2+ and H+ ionic fluxes are involved in apical growth and graviperception of the Phycomyces stage I sporangiophore. Planta, 2012, 236(6): 1817-1829. DOI:10.1007/s00425-012-1738-3 |
| [82] | Bais HP, Weir TL, Perry LG, Gilroy S, Vivanco JM. The role of root exudates in rhizosphere interactions with plants and other organisms. Annual Review of Plant Biology, 2006, 57: 233-266. DOI:10.1146/annurev.arplant.57.032905.105159 |
| [83] | van West P, Morris BM, Reid B, Appiah AA, Osborne MC, Campbell TA, Shepherd SJ, Gow NAR. Oomycete plant pathogens use electric fields to target roots. Molecular Plant-Microbe Interactions, 2002, 15(8): 790-798. DOI:10.1094/MPMI.2002.15.8.790 |
| [84] | Robinson KR, Messerli MA. Left/right, up/down:The role of endogenous electrical fields as directional signals in development, repair and invasion. Bioessays, 2003, 25(8): 759-766. DOI:10.1002/bies.10307 |
| [85] | George TS, Turner BL, Gregory PJ, Cade-Menun BJ, Richardson AE. Depletion of organic phosphorus from oxisols in relation to phosphatase activities in the rhizosphere. European Journal of Soil Science, 2006, 57(1): 47-57. DOI:10.1111/j.1365-2389.2006.00767.x |
| [86] | Schreiter S, Ding GC, Heuer H, Neumann G, Sandmann M, Grosch R, Kropf S, Smalla K. Effect of the soil type on the microbiome in the rhizosphere of field-grown lettuce. Frontiers in Microbiology, 2014, 5: 144. |
| [87] | de Ridder-Duine AS, Kowalchuk GA, Gunnewiek PJA, Smant W, van Veen JA, de Boer W. Rhizosphere bacterial community composition in natural stands of Carex arenaria (sand sedge) is determined by bulk soil community composition. Soil Biology and Biochemistry, 2005, 37(2): 349-357. DOI:10.1016/j.soilbio.2004.08.005 |
| [88] | van der Heijden MGA, de Bruin S, Luckerhoff L, van Logtestijn RSP, Schlaeppi K. A widespread plant-fungal-bacterial symbiosis promotes plant biodiversity, plant nutrition and seedling recruitment. The ISME Journal, 2016, 10(2): 389-399. DOI:10.1038/ismej.2015.120 |
| [89] | Kang SM, Khan AL, Waqas M, You YH, Kim JH, Kim JG, Hamayun M, Lee IJ. Plant growth-promoting rhizobacteria reduce adverse effects of salinity and osmotic stress by regulating phytohormones and antioxidants in Cucumis sativus. Journal of Plant Interactions, 2014, 9(1): 673-682. DOI:10.1080/17429145.2014.894587 |
| [90] | Kim YC, Leveau J, Gardener BBM, Pierson EA, Pierson LS III, Ryu CM. The multifactorial basis for plant health promotion by plant-associated bacteria. Applied and Environmental Microbiology, 2011, 77(5): 1548-1555. DOI:10.1128/AEM.01867-10 |
| [91] | Dardanelli MS, Manyani H, González-Barroso S, Rodríguez-Carvajal MA, Gil-Serrano AM, Espuny MR, López-Baena FJ, Bellogín RA, Megías M, Ollero FJ. Effect of the presence of the plant growth promoting rhizobacterium (PGPR) Chryseobacterium balustinum Aur9 and salt stress in the pattern of flavonoids exuded by soybean roots. Plant and Soil, 2010, 328(1/2): 483-493. |
| [92] | Scheublin TR, Ridgway KP, Young JPW, van der Heijden MGA. Nonlegumes, legumes, and root nodules harbor different arbuscular mycorrhizal fungal communities. Applied and Environmental Microbiology, 2004, 70(10): 6240-6246. DOI:10.1128/AEM.70.10.6240-6246.2004 |
| [93] | Deinlein U, Stephan AB, Horie T, Luo W, Xu GH, Schroeder JI. Plant salt-tolerance mechanisms. Trends in Plant Science, 2014, 19(6): 371-379. DOI:10.1016/j.tplants.2014.02.001 |
| [94] | Zhang HM, Kim MS, Sun Y, Dowd SE, Shi HZ, Paré PW. Soil bacteria confer plant salt tolerance by tissue-specific regulation of the sodium transporter HKT1. Molecular Plant-Microbe Interactions, 2008, 21(6): 737-744. DOI:10.1094/MPMI-21-6-0737 |
| [95] | Choudhury FK, Rivero RM, Blumwald E, Mittler R. Reactive oxygen species, abiotic stress and stress combination. The Plant Journal, 2017, 90(5): 856-867. DOI:10.1111/tpj.13299 |
| [96] | Kumari S, Vaishnav A, Jain S, Varma A, Choudhary DK. Bacterial-mediated induction of systemic tolerance to salinity with expression of stress alleviating enzymes in soybean (Glycine max l. Merrill). Journal of Plant Growth Regulation, 2015, 34(3): 558-573. DOI:10.1007/s00344-015-9490-0 |
| [97] | Kasim WA, Gaafar RM, Abou-Ali RM, Omar MN, Hewait HM. Effect of biofilm forming plant growth promoting rhizobacteria on salinity tolerance in barley. Annals of Agricultural Sciences, 2016, 61(2): 217-227. DOI:10.1016/j.aoas.2016.07.003 |
| [98] | Weller DM, Raaijmakers JM, Gardener BBM, Thomashow LS. Microbial populations responsible for specific soil suppressiveness to plant pathogens. Annual Review of Phytopathology, 2002, 40: 309-348. DOI:10.1146/annurev.phyto.40.030402.110010 |
| [99] | Rosenzweig N, Tiedje JM, Quensen JF Ⅲ, Meng QX, Hao JJJ. Microbial communities associated with potato common scab-suppressive soil determined by pyrosequencing analyses. Plant Disease, 2012, 96(5): 718-725. DOI:10.1094/PDIS-07-11-0571 |
| [100] | Macfarlane SA. Molecular determinants of the transmission of plant viruses by nematodes. Molecular Plant Pathology, 2003, 4(3): 211-215. DOI:10.1046/j.1364-3703.2003.00164.x |
| [101] | Xu XL, Ouyang H, Richter A, Wanek W, Cao GM, Kuzyakov Y. Spatio-temporal variations determine plant-microbe competition for inorganic nitrogen in an alpine meadow. Journal of Ecology, 2011, 99(2): 563-571. |
| [102] | van der Heijden MGA, Bardgett RD, van Straalen NM. The unseen majority:soil microbes as drivers of plant diversity and productivity in terrestrial ecosystems. Ecology Letters, 2008, 11(3): 296-310. DOI:10.1111/j.1461-0248.2007.01139.x |
| [103] | Ding JT, Xi BD, Gao RT, He LS, Liu HL, Dai XL, Yu YJ. Identifying diffused nitrate sources in a stream in an agricultural field using a dual isotopic approach. Science of the Total Environment, 2014, 484: 10-18. DOI:10.1016/j.scitotenv.2014.03.018 |
 2021, Vol. 61
2021, Vol. 61