中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 牛婷婷, 周雪洁, 余小波, 蔡宜生, 陈静, 王瑞, 付爽, 栾闯, 何剑为. 2021
- Tingting Niu, Xuejie Zhou, Xiaobo Yu, Yisheng Cai, Jing Chen, Rui Wang, Shuang Fu, Chuang Yi, Jianwei He. 2021
- 过表达PDI对毕赤酵母分泌淀粉样蛋白的影响——以胱抑素为例
- Effect of PDI overexpressing on secretion of exogenous amyloid protein in Pichia pastoris—take cystatin as an example
- 微生物学报, 61(10): 3315-3327
- Acta Microbiologica Sinica, 61(10): 3315-3327
-
文章历史
- 收稿日期:2021-01-23
- 修回日期:2021-03-30
- 网络出版日期:2021-08-06
巴斯德毕赤酵母(Komagataella phaffii, 原命名为Pichia pastoris)在表达外源蛋白时可将甲醇作为唯一的碳源和能源来诱导和调控外源基因的表达。毕赤酵母表达系统具有大肠杆菌表达系统的诸多优势, 同时又具有高等真核表达系统的许多优点:如蛋白加工、折叠、翻译后修饰等[1];其胞外蛋白分泌的非常少, 有利于后续对外源蛋白的分离和纯化;通过使用工业生物反应器, 可以从较小的培养体积大规模地生产目的蛋白质。因此, 毕赤酵母已成为比较理想的外源蛋白表达宿主之一, 且已被成功地应用于工业、生物制药及疫苗研究领域[2]。
蛋白质二硫键异构酶(protein disulfide isomerases, PDI)是内质网中与折叠相关的负责二硫键形成的多功能蛋白, 它既可以催化新生肽链形成二硫键, 又能够帮助蛋白质折叠形成其正确的天然构象[3]。酵母的PDI与哺乳动物PDI结构相似, 全长PDI分子结构在空间上呈“U”型, 具有a、b、b′、a′、c功能结构域, “U”型结构的底部由b和b′结构域组成, 通过其疏水表面结合底物。“U”型结构的两个侧臂由a和a′结构域组成, 主要起催化功能[4]。由于“U”型结构的内表面富集疏水残基, 有利于其与错误折叠蛋白之间发生相互作用。
鸡胱抑素C (chicken cystatin C, cC), 即鸡半胱氨酸蛋白酶抑制剂, 是人胱抑素C (human cystatin C, HCC)的同源蛋白, 二者的氨基酸序列、结构、生理特性等都具有高度的同源性[5]。HCC在体内易形成二聚体或寡聚体, 导致其半胱氨酸蛋白酶抑制剂的活性丧失, 而进一步的寡聚化能形成多聚体最终形成成熟的淀粉样纤维, 高度稳定的淀粉样纤维沉积在脑动脉血管壁直接导致了脑淀粉样血管病的发生[6-7]。cC大小约为14 kDa, 由116个氨基酸组成, 是一种低分子量蛋白。cC在生理条件下以单体形式存在, 更易于分离出蛋白单体, 并具有更高的热力学稳定性, 因此在进行实验操作时具有更高的可行性和便利性。在科研中也常用cC来替代HCC作为淀粉样蛋白的模型蛋白进行科学研究[8-9]。
有关PDI调控外源新生蛋白表达的相关研究显示:在敲除PDI同源蛋白Eps1的酿酒酵母中表达外源淀粉样蛋白时, 外源蛋白的胞外分泌量和胞内驻留量出现显著变化[10];同时, 毕赤酵母在表达外源淀粉样蛋白时存在二硫键未正确形成的问题, 干扰细胞自身蛋白的正确折叠, 误折叠蛋白的积累会导致折叠胁迫[11]。折叠胁迫会改变细胞的正常代谢, 降低细胞的抗逆性, 甚至造成细胞的死亡[12]。大量研究表明, 毕赤酵母中PDI的过表达可以促进毕赤酵母分泌外源重组蛋白, 如PDI的过表达使得抗体蛋白2F5 Fab在Pichia pastoris的表达水平提高了2倍[13]。虽然通过与PDI的共表达提高了外源蛋白的表达水平和活性, 但在真核细胞中, 由于蛋白质折叠是一个复杂的多步骤过程, PDI帮助新生蛋白质合成与折叠的调控机制以及在蛋白分泌网络中与PDI相关的调控因子仍不明确。
本研究通过构建PDI过表达菌株, 研究PDI过表达对外源淀粉样蛋白cC在毕赤酵母中合成、折叠及分泌的影响。结合RNA-Seq组学方法与高通量分析手段比较PDI过表达菌株与对照菌株, 揭示RNA转录本在特定时刻的存在情况和数量的多少。根据基因在不同菌株中的不同表达量进行差异基因表达分析、筛选、功能注释等生物学信息分析, 以期识别显著上调/下调的基因, 在表达宿主全基因组范围内挖掘改造宿主分泌途径的新靶点。
1 材料和方法 1.1 菌种和质粒毕赤酵母GS115 (his4)菌株由福州大学刘树滔教授惠赠。酵母表达载体pPICZαA购自Invitrogen公司。酵母胞内表达载体pPIC3.5K购自武汉淼灵生物科技有限公司。
1.2 培养基和菌体的培养条件PDI相关重组DNA操作在大肠杆菌菌株(DH5α, Invitrogen)中进行, 该菌株在37 ℃于LB培养基中培养。在我们先前的工作中, 已经构建了质粒pPICZαA-cC并将其转化到大肠杆菌菌株(Top10, Invitrogen)中。酵母细胞在YPD (1%酵母提取物, 2%蛋白胨和2%葡萄糖)中于30 ℃培养至对数生长期。携带外源基因的重组酵母细胞在培养基YPM [1%酵母提取物, 2%蛋白胨, 0.5%甲醇(V/V)]中诱导表达72 h。
1.3 表达载体和过表达菌株的构建根据已经发表的GenBank序列号EU805807的酵母PDI基因序列设计引物5′-TACGTAACCA TGCAATTCAACTGGGATATT-3′ (正向, 下划线为SnaB Ⅰ位点)和引物5′-GCGGCCGCTTAAAGC TCGTCGTGAGCGTC-3′ (反向, 下划线为Not Ⅰ位点)从Pichia pastoris X33 (Invitrogen)的基因组DNA扩增出来。扩增的PDI片段被SnaB I/Not I (TaKaRa)酶切后与载体pPIC3.5K连接, 构建pPIC3.5K-PDI重组载体。将含有cC编码基因的重组载体pT-cC (前期构建)用Xho I/Xba I (Thermo Fisher)酶切后与载体pPICZαA连接, 构建pPICZαA-cC重组载体。通过电穿孔将Sac Ⅰ线性化的质粒pPICZαA-cC转化到毕赤酵母GS115细胞中, 并在含有100 μg/mL Zeocin (Invitrogen)的YPDS平板(1%酵母提取物, 2%蛋白胨, 2%葡萄糖, 1 mol/L山梨醇和2%琼脂粉)中筛选重组子。同时, 采用同样方法将线性化的pPIC3.5K-PDI也转化到毕赤酵母GS115细胞中, 并在MD平板上选择重组子。选择后的酵母细胞的阳性转化子即为Mut+ (EasySelect™ Pichia表达试剂盒)。
1.4 蛋白表达分析将在YPM培养基诱导72 h的酵母细胞于4 ℃、12000 r/min离心30 min后收集上清即为胞外蛋白样品。应用酵母蛋白质提取试剂(TaKaRa)从酵母细胞中提取胞内蛋白样品。在变性条件下用15% SDS聚丙烯酰胺凝胶电泳(PAGE)检测cC蛋白, 用12% PAGE观察检测PDI蛋白表达情况。将凝胶用考马斯亮蓝显示蛋白条带, 然后将其转移到PVDF膜(Millipore)上进行蛋白质印迹分析。1:2000稀释度的PDI一抗(ABclonal)用于检测PDI, 1:2000稀释度的cC一抗(自制)检测cC。二抗为1:5000稀释度的HRP-conjugated Goat Anti-Rabbit IgG。使用Image J软件分析PVDF膜上的条带, 并使用Graphpad Prism 5.0处理生成的数据。
1.5 Total RNA样品抽提和检测当酵母生长至OD600=2.5, 处于对数生长期时, 依据酵母RNA提取试剂盒Yeast RNAiso Kit (TaKaRa)操作说明提取酵母Total RNA。
1.6 高通量RNA-Seq测序使用NanoDrop 2000测量RNA浓度, 之后应用Agilent Bioanalyzer 2100系统的RNA Nano 6000测定试剂盒评估RNA的完整性, 后进行转录组测序的文库制备。根据厂家说明, 将文库制备物在Illumina Hiseq Xten平台上测序, 并产生配对末端读数。此实验委托北京百迈克生物科技有限公司操作。
1.7 实时荧光定量qRT-PCR将提取的总RNA作为模板, 使用反转录试剂盒(RT Master Mix, TaKaRa)合成第一条链cDNA, 具体操作步骤如产品说明书所描述。按照荧光定量试剂盒(SYBR Premix Ex TaqⅡ)说明书配置荧光定量PCR体系的混合液, 并且依据酶的特性设定荧光定量PCR循环条件, 进行荧光定量PCR实验。
2 结果和分析 2.1 GS115毕赤酵母菌株PDI过表达系统的建立已有文献报道, 存在于内质网并且与蛋白折叠相关的分子伴侣中, PDI的过表达可以增加毕赤酵母的分泌外源蛋白的表达量[13]。因此, 我们研究使用过表达PDI的方法来提高外源淀粉样蛋白cC的表达量。为了建立用于表达分泌型cC的PDI过表达酵母菌株, 我们首先选择pPIC3.5K作为胞内表达载体用于胞内表达PDI, 构建了过表达PDI的重组毕赤酵母菌株GS115-PDI, 然后选择pPICZαA载体进行cC的胞外分泌蛋白表达, 构建了过表达PDI状态下的重组毕赤酵母菌株GS115-PDI-cC。
2.2 PDI的过表达增加了外源淀粉样蛋白cC的表达由于醇氧化酶启动子(AOX1)受甲醇诱导, 利用甲醇可诱导AOX1连接的外源蛋白编码基因的表达, 通过蛋白质免疫印迹法检测诱导后PDI在各个菌株中的表达情况, 结果见图 1, 过表达酵母菌株中PDI蛋白的含量显著高于对照菌株, 表明成功构建了PDI过表达的毕赤酵母菌株。然后, 我们使用SDS-PAGE方法分析了每个菌株中胞内和胞外蛋白的表达情况, 发现胞内和胞外蛋白都显著增加(图 2)。同时, 使用蛋白质免疫印迹法检测每个菌株中表达的cC蛋白量, 结果表明, 过表达PDI菌株中胞内和胞外的cC的表达量显著增加, 并且胞内cC二聚体也明显增加(图 3)。以上结果表明, PDI的过表达显著增加了毕赤酵母GS115中外源淀粉样蛋白cC的表达量。
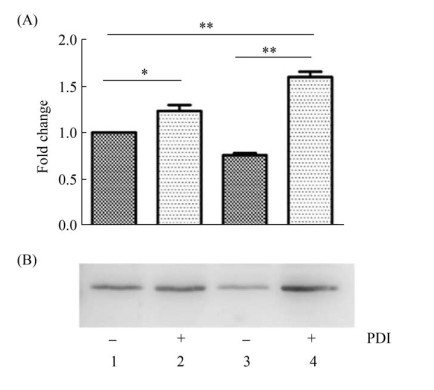
|
| 图 1 Western blotting验证毕赤酵母GS115中PDI的表达水平 Figure 1 Western blotting verifies the expression level of PDI in Pichia pastoris GS115. +: the plasmid carrying PDI gene is transformed into Pichia pastoris GS115 and the PDI protein is successfully overexpressed; -: the PDI gene is not overexpressed in Pichia pastoris GS115. The abscissa of the Figure 1-A corresponds to the band of every lane in Figure 1-B. |

|
| 图 2 各菌株细胞内总蛋白(A)与细胞外分泌蛋白(B)的SDS-PAGE凝胶成像图 Figure 2 SDS-PAGE gel image of total intracellular protein (A) and extracellular secretion protein (B) of each strain. +: the plasmid carrying this gene is transformed into Pichia pastoris GS115; -: the vector carrying the target gene is not transfected into Pichia pastoris GS115 relatively. PDI: disulfide bond isomerase; cC: chicken cystatin. |
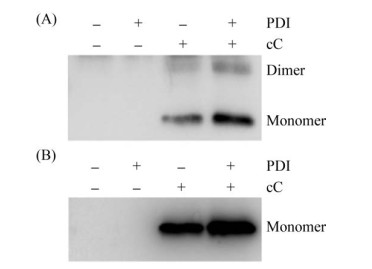
|
| 图 3 Western blotting分析诱导72 h后cC蛋白表达情况 Figure 3 Western blotting analysis of the expression of cC protein 72 h after induction. +: the plasmid carrying this gene is transformed into the host (Pichia pastoris GS115); -: the target gene is not transfected into the host strain. |
2.3 高通量RNA-Seq测序
已有研究表明基因表达存在着生物变异性[14-15]。为了寻找真正差异表达的基因, 需要考虑并解决由于生物变异引起的表达差异。目前最常用且最有效的方法是在实验设计中设立生物学重复。重复条件限制越严格, 重复样品数目越多, 寻找到的差异表达基因越可靠[16]。研究表明, 皮尔逊相关系数r常作为生物学重复相关性的评估指标[17]。r2越接近1, 说明2个重复样品相关性越强[18]。因此, 我们对菌株GS115、GS115-PDI、GS115-cC、GS115-PDI-cC每个菌株进行3次重复共完成了12个样品的转录组测序分析。分析样本之间的相关系数如图 4所示, 从图中可以看到2个相同样品的相关性r2值都接近1, 说明样品的重复性达到了生物学统计的标准, 可以用于筛选差异表达基因。

|
| 图 4 两两样品的表达量相关性热图 Figure 4 Heat map of expression level correlation of pairwise samples. |
2.4 利用RNA测序技术筛选并注释GS115-cC与GS115-PDI-cC比较之后的差异表达基因
如表 1、图 5所示, 通过比较不同菌株, 我们鉴定出了数千个差异表达基因。但是对于只属于在过表达PDI时影响毕赤酵母GS115宿主菌株增加cC表达量的基因, 我们最终筛选出373个差异表达基因, 其中上调和下调的基因分别有191和182个。注释到KEGG数据库的差异表达基因(DEG)的结果[19]如图 6所示, 有122个基因成功注释到KEGG数据库中, 其中上、下调的基因分别有52和70个。然而, 并不是所有这些差异表达基因都参与蛋白质的运输和分泌以及内质网质量控制系统。通过对图 6的分析, 我们发现有24个DEG参与蛋白质翻译通路, 12个DEG参与蛋白质的运输和分解代谢, 21个DEG参与蛋白质折叠、分选和降解的途径。基因差异表达结果显示, 大部分参与翻译通路以及大部分参与蛋白质运输和分解代谢的DEG表达下调, 暗示着蛋白质过量表达会消耗宿主细胞可利用的碳源和能量, 因此会给宿主带来代谢负担。而参与蛋白质折叠、分选和降解通路的DEG表达上调。这暗示着由于PDI本身是一种存在于内质网中的分子伴侣, 它能够帮助蛋白质正确折叠防止那些错误折叠蛋白被蛋白质降解系统降解, 从而来增加外源蛋白的表达量。在这些DEG中, 我们进一步在蛋白质折叠及内质网应激途径进行差异基因的再次筛选, 如表 2所示, 我们选取到了参与上述两途径的8个DEG。
| Group | Strains comparison | Samples in RNA-seq |
| Group0(G0) | GS115 VS GS115-PDI | T1, T2, T3 VS T4, T5, T6 |
| Group1(G1) | GS115 VS GS115-cC | T1, T2, T3 VS T7, T8, T9 |
| Group2(G2) | GS115 VS GS115-PDI-cC | T1, T2, T3 VS T10, T11, T12 |
| Group3(G3) | GS115-PDI VS GS115-PDI-cC | T4, T5, T6 VS T10, T11, T12 |
| Group4(G4) | GS115-cC VS GS115-PDI-cC | T7, T8, T9 VS T10, T11, T12 |
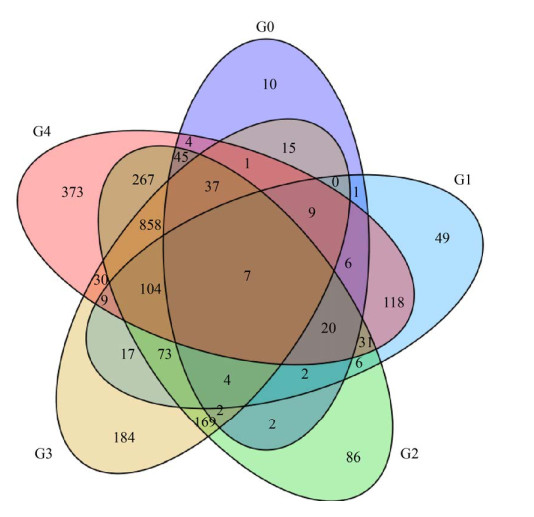
|
| 图 5 不同菌株比较组差异表达基因(DEG)维恩图 Figure 5 Differentially expressed genes (DEG) Venn diagram of different strains comparison group. |

|
| 图 6 差异表达基因KEGG分类图 Figure 6 Differentially expressed genes KEGG classification map. The ordinate is the name of the KEGG metabolic pathway, the abscissa is the ratio of the number of genes annotated to the pathway and their number to the total number of annotated genes. |
| Gene ID | Gene name | GenBank | Regulation | KEGG pathway annotation |
| Gene 263 | PAS_chr1-3_0261 | XM_002489561.1 | Up | Endocytosis (ko04144) |
| Gene 3092 | PAS_chr3_1165 | XM_002492390.1 | Up | Endocytosis (ko04144) |
| Gene 3874 | PAS_chr3_0974 | XM_002493172.1 | Up | Endocytosis (ko04144) |
| Gene 3122 | PAS_chr3_0249 | XM_002492420.1 | Up | Protein processing in endoplasmic reticulum (ko04141) |
| Gene 4006 | PAS_chr3_1104 | XM_002493304.1 | Up | Protein processing in endoplasmic reticulum (ko04141) |
| Gene 463 | PAS_chr1-1_0160 | XM_002489761.1 | Down | Protein processing in endoplasmic reticulum (ko04141) |
| Gene 160 | PAS_chr1-3_0161 | XM_002489458.1 | Up | SNARE interactions in vesicular transport (ko04130) |
| Gene 3814 | PAS_chr3_1238 | XM_002493112.1 | Up | SNARE interactions in vesicular transport (ko04130) |
2.5 差异基因的再次验证
我们使用qRT-PCR的方法对表 2中的DEG进行再次验证, 证实这些基因在分子水平上是否出现了相同的上下调的变化情况。表 3列出目的基因引物序列。如图 7-A所示, 在内吞通路中的基因(PAS_chr1-3_0261, PAS_chr3_1165, PAS_ chr3_0974)均上调。图 7-B所示功能未知的基因(PAS_chr3_0249)和参与内质网蛋白加工的基因(PAS_chr3_1104)上调。从qRT-PCR结果可知, 这与我们表 4的RNA-seq结果一致。因此, 我们再一次在分子水平上验证了差异表达基因的变化情况。我们认为对差异表达基因的再次鉴定是基于客观的实验数据, 鉴定出的差异表达基因具有一定的研究价值, 可以作进一步分析。
| Gene ID | Gene name | GenBank | Sequences of primer |
| Gene 263 | PAS_chr1-3_0261 | XM_002489561.1 | F: GGTAGAGGGGCAAGAACATGAG R: TGGGCTGAGTAGCAGATGGTG |
| Gene 3092 | PAS_chr3_1165 | XM_002492390.1 | F: ATCGGTTTCGTCACTGTTTCTC R: ACTGGATTTCACTGGCTTGTTC |
| Gene 3874 | PAS_chr3_0974 | XM_002493172.1 | F: GGAGAAGGGTGGTAATGAACG R: GAGACTGTCCCGAATGGTCAG |
| Gene 3122 | PAS_chr3_0249 | XM_002492420.1 | F: TTCGCCCTCCTGAACTGATAG R: CCTGGGGAACTACGGTCTTG |
| Gene 4006 | PAS_chr3_1104 | XM_002493304.1 | F: GACCAATCGTCGTTTAGAGCTAC R: CTGCATTCTGTCTTGTCGTCC |
| Gene 463 | PAS_chr1-1_0160 | XM_002489761.1 | F: AGTTTTATGCCGATTGGTGTTC R: TGAGTATTTGTTAGCATCAATGGC |
| Gene 160 | PAS_chr1-3_0161 | XM_002489458.1 | F: TAGAATCGCAAAATGAGGACG R: TTTCTTAGCCATTACCATCATCC |
| Gene 3814 | PAS_chr3_1238 | XM_002493112.1 | F: GGCGAACCTACCCTCATAACAC R: TGGCTTCATTTCTATTGCATCTC |
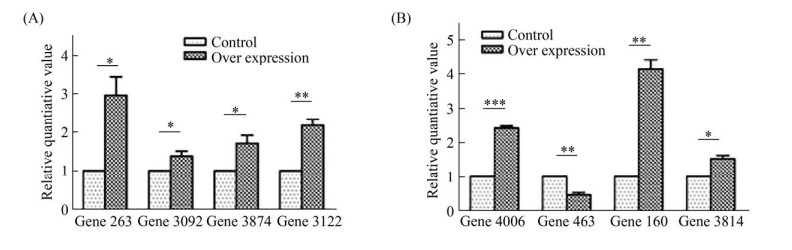
|
| 图 7 关键差异基因相对荧光定量柱状图 Figure 7 Relative fluorescence quantitative histogram of key differential genes. The control group is the GS115-cC strain. The overexpression group represents the GS115-PDI-cC strain. |
| Gene ID | Gene name | Log2FC | FDR | Regulation | ΔCt/ Over-expression | ΔCt/Control | 2-ΔΔCt | Regulation |
| Gene 263 | PAS_chr1-3_0261 | 1.907035171 | 2.17E-85 | Up | -0.657 | 0.893 | 2.954 | Up |
| Gene 3092 | PAS_chr3_1165 | 1.038464554 | 2.21E-21 | Up | -0.107 | 0.353 | 1.380 | Up |
| Gene 3874 | PAS_chr3_0974 | 1.054612732 | 2.46E-15 | Up | 1.230 | 2.000 | 1.713 | Up |
| Gene 3122 | PAS_chr3_0249 | 1.056165135 | 1.68E-22 | Up | -3.257 | -2.137 | 2.177 | Up |
| Gene 4006 | PAS_chr3_1104 | 1.032749024 | 1.48E-99 | Up | 1.793 | 3.063 | 2.412 | Up |
| Gene 463 | PAS_chr1-1_0160 | -1.181488455 | 7.85E-46 | Down | 0.59 | -0.503 | 0.472 | Down |
| Gene 160 | PAS_chr1-3_0161 | 1.047977247 | 1.42E-07 | Up | 1.227 | 3.273 | 4.138 | Up |
| Gene 3814 | PAS_chr3_1238 | 1.046077293 | 2.14E-42 | Up | 1.140 | 1.743 | 1.522 | Up |
| FC: fold change; FDR: false discovery rate. | ||||||||
3 讨论
毕赤酵母在表达外源蛋白时可将甲醇作为唯一的碳源来严格控制外源基因的表达[20]。但是对淀粉样蛋白进行表达时, 由于其易于聚集, 所以此系统对其表达的结果不是很理想[21]。内质网是合成、加工蛋白质的主要场所, 当内质网中存在大量未正确折叠的蛋白时, 会引起UPR反应, 启动蛋白质降解途径使其降解[22]。PDI是存在于内质网中的氧化还原酶, 它既能够帮助蛋白质形成二硫键又可以促使蛋白正确折叠[23]。本研究通过构建过表达PDI的毕赤酵母菌株, 在蛋白水平上证明了过表达PDI的菌株在表达蛋白cC时, 无论在胞内还是胞外, cC表达量都显著高于PDI正常水平的毕赤酵母菌株。这表明过表达PDI的毕赤酵母菌株能显著提高cC的表达量。
基于以上蛋白水平的表达结果, 应用高通量测序平台并采用转录组深度测序分析手段, 初步挖掘出部分在蛋白折叠、修饰、转运与分泌过程中的潜在参与基因。通过与正常PDI水平的毕赤酵母表达cC比较, 当过表达PDI时, 我们筛选到了373个发生了上调或下调的基因。进一步将这些基因注释到KEGG途径中后发现它们主要集中在蛋白质翻译修饰、内质网加工、未折叠蛋白反应(unfolded protein response, UPR)等途径。这个结果表明当过表达PDI时, 酵母细胞会通过调节这些差异基因的表达, 对新生的大量未正确折叠的蛋白和错误折叠的蛋白进行必要的辅助折叠和修饰, 并启动UPR对错误折叠的蛋白质进行降解, 以适应折叠胁迫对于细胞表达外源蛋白的微环境造成不利影响[11]。这些连锁反应不仅提高了蛋白的正确折叠效率, 也提高了外源蛋白的表达量和分泌量。另一方面, 折叠胁迫会改变细胞的正常代谢, 降低细胞的抗逆性, 甚至造成细胞的死亡[12]。因此本研究中筛选到的候选基因也为折叠胁迫相关细胞信号通路、途径的深入分析提供了实验数据上的有力支撑。
综上, 对差异表达基因的进一步挖掘分析可能为寻找降低淀粉样蛋白在胞内的聚集、提高毕赤酵母中外源淀粉样蛋白表达效率的方法提供新思路, 同时也为发现新的有关淀粉样蛋白沉积疾病的药物提供线索, 为改造高效过表达淀粉样蛋白的工程酵母菌株奠定基础。
| [1] | Karbalaei M, Rezaee SA, Farsiani H. Pichia pastoris: a highly successful expression system for optimal synthesis of heterologous proteins. Journal of Cellular Physiology, 2020, 235(9): 5867-5881. DOI:10.1002/jcp.29583 |
| [2] | Macauley-Patrick S, Fazenda ML, McNeil B, Harvey LM. Heterologous protein production using the Pichia pastoris expression system. Yeast, 2005, 22(4): 249-270. DOI:10.1002/yea.1208 |
| [3] | Ellgaard L, Ruddock LW. The human protein disulphide isomerase family: substrate interactions and functional properties. EMBO Reports, 2005, 6(1): 28-32. DOI:10.1038/sj.embor.7400311 |
| [4] | Tian G, Xiang S, Noiva R, Lennarz WJ, Schindelin H. The crystal structure of yeast protein disulfide isomerase suggests cooperativity between its active sites. Cell, 2006, 124(1): 61-73. DOI:10.1016/j.cell.2005.10.044 |
| [5] | Bode W, Engh R, Musil D, Thiele U, Huber R, Karshikov A, Brzin J, Kos J, Turk V. The 2.0 A X-ray crystal structure of chicken egg white cystatin and its possible mode of interaction with cysteine proteinases. The EMBO Journal, 1988, 7(8): 2593-2599. DOI:10.1002/j.1460-2075.1988.tb03109.x |
| [6] | Janowski R, Abrahamson M, Grubb A, Jaskolski M. Domain swapping in N-truncated human cystatin C. Journal of Molecular Biology, 2004, 341(1): 151-160. DOI:10.1016/j.jmb.2004.06.013 |
| [7] | Janowski R, Kozak M, Abrahamson M, Grubb A, Jaskolski M. 3D domain-swapped human cystatin C with amyloidlike intermolecular beta-sheets. Proteins, 2005, 61(3): 570-578. DOI:10.1002/prot.20633 |
| [8] | Mori K. Tripartite management of unfolded proteins in the endoplasmic Reticulum. Cell, 2000, 101(5): 451-454. DOI:10.1016/S0092-8674(00)80855-7 |
| [9] | Higgins DR, Cregg JM. Introduction to Pichia pastoris. Methods in Molecular Biology: Clifton, N J, 1998, 103: 1-15. |
| [10] | He JW, Sakamoto T, Song YT, Saito A, Harada A, Azakami H, Kato A. Effect of EPS1 gene deletion in Saccharomyces cerevisiae on the secretion of foreign proteins which have disulfide bridges. FEBS Letters, 2005, 579(11): 2277-2283. DOI:10.1016/j.febslet.2005.03.019 |
| [11] | Gasser B, Saloheimo M, Rinas U, Dragosits M, Rodríguez-Carmona E, Baumann K, Giuliani M, Parrilli E, Branduardi P, Lang C, Porro D, Ferrer P, Tutino ML, Mattanovich D, Villaverde A. Protein folding and conformational stress in microbial cells producing recombinant proteins: a host comparative overview. Microbial Cell Factories, 2008, 7: 11. DOI:10.1186/1475-2859-7-11 |
| [12] | Zhu TC, Guo MJ, Zhuang YP, Chu J, Zhang SL. Understanding the effect of foreign gene dosage on the physiology of Pichia pastoris by transcriptional analysis of key genes. Applied Microbiology and Biotechnology, 2011, 89(4): 1127-1135. DOI:10.1007/s00253-010-2944-1 |
| [13] | Gasser B, Maurer M, Gach J, Kunert R, Mattanovich D. Engineering of Pichia pastoris for improved production of antibody fragments. Biotechnology and Bioengineering, 2006, 94(2): 353-361. DOI:10.1002/bit.20851 |
| [14] | Inan M, Aryasomayajula D, Sinha J, Meagher MM. Enhancement of protein secretion in Pichia pastoris by overexpression of protein disulfide isomerase. Biotechnology and Bioengineering, 2006, 93(4): 771-778. DOI:10.1002/bit.20762 |
| [15] | Elowitz MB. Stochastic gene expression in a single cell. Science, 2002, 297(5584): 1183-1186. DOI:10.1126/science.1070919 |
| [16] | Hansen KD, Wu ZJ, Irizarry RA, Leek JT. Sequencing technology does not eliminate biological variability. Nature Biotechnology, 2011, 29(7): 572-573. DOI:10.1038/nbt.1910 |
| [17] | Robasky K, Lewis NE, Church GM. The role of replicates for error mitigation in next-generation sequencing. Nature Reviews Genetics, 2014, 15(1): 56-62. DOI:10.1038/nrg3655 |
| [18] | Schulze SK, Kanwar R, Gölzenleuchter M, Therneau TM, Beutler AS. SERE: single-parameter quality control and sample comparison for RNA-Seq. BMC Genomics, 2012, 13: 524. DOI:10.1186/1471-2164-13-524 |
| [19] | Kanehisa M, Araki M, Goto S, Hattori M, Hirakawa M, Itoh M, Katayama T, Kawashima S, Okuda S, Tokimatsu T, Yamanishi Y. KEGG for linking genomes to life and the environment. Nucleic Acids Research, 2008, 36(suppl_1): D480-D484. |
| [20] | Daly R, Hearn MTW. Expression of heterologous proteins in Pichia pastoris: a useful experimental tool in protein engineering and production. Journal of Molecular Recognition, 2005, 18(2): 119-138. DOI:10.1002/jmr.687 |
| [21] | Montoliu-Gaya L, Esquerda-Canals G, Bronsoms S, Villegas S. Production of an anti-Aβ antibody fragment in Pichia pastoris and in vitro and in vivo validation of its therapeutic effect. PLoS ONE, 2017, 12(8): e0181480. DOI:10.1371/journal.pone.0181480 |
| [22] | Guan B, Chen FX, Su S, Duan ZY, Chen Y, Li HZ, Jin J. Effects of co-overexpression of secretion helper factors on the secretion of a HSA fusion protein (IL2-HSA) in Pichia pastoris. Yeast, 2016, 33(11): 587-600. DOI:10.1002/yea.3183 |
| [23] | Cheng H, Wang L, Wang CC. Domain a' of protein disulfide isomerase plays key role in inhibiting α-synuclein fibril formation. Cell Stress and Chaperones, 2010, 15(4): 415-421. DOI:10.1007/s12192-009-0157-2 |
 2021, Vol. 61
2021, Vol. 61




